Method Article
Genoma Edición y diferenciación dirigida de hPSCs para interrogar Determinantes linaje en el Desarrollo Humano de páncreas
* Estos autores han contribuido por igual
En este artículo
Resumen
Protocolos para generar HPSC líneas mutantes que utilizan la plataforma iCRISPR y se diferencien en células hPSCs ß-como sensibles a glucosa se describen. La combinación de la tecnología de edición del genoma con la diferenciación dirigida HPSC proporciona una potente plataforma para el análisis sistemático de la función de los factores determinantes del linaje en el desarrollo humano y la progresión de la enfermedad.
Resumen
Interrogar a la función de genes en auto-renovación o diferenciación de las células madre pluripotentes humanas (hPSCs) ofrece una plataforma valiosa para comprender el desarrollo humano y la disección de los mecanismos de la enfermedad en un plato. Para sacar provecho de esta aplicación potencial requiere herramientas de edición de genoma eficientes para generar mutantes HPSC en los genes asociados a la enfermedad, así como los protocolos de diferenciación in vitro HPSC para producir tipos de células relevantes de la enfermedad que recapitular estrechamente sus homólogos en vivo. Una plataforma de edición de genoma eficiente para hPSCs llamado iCRISPR se ha desarrollado a través de la focalización mediada por TALEN de un casete de expresión en el locus Cas9 AAVS1. Aquí, se describen los protocolos para la generación de líneas inducibles Cas9 HPSC utilizando células cultivadas en un medio químicamente definido y una condición libre de alimentador. Los procedimientos detallados para utilizar el sistema iCRISPR para la eliminación de genes o alteraciones genéticas precisas en hPSCs, ya sea a través de nen homóloga extremo de unión (NHEJ) o a través de alteraciones de nucleótidos precisas utilizando una plantilla de reparación de homología dirigida (HDR), respectivamente, están incluidos. Estos procedimientos técnicos incluyen descripciones del diseño, la producción y la transfección de ARN guía CRISPR (gRNAs); la medición de la tasa de mutación CRISPR por ensayos T7E1 o RFLP; y la creación y validación de líneas mutantes clonales. Por último, los procedimientos para la diferenciación de crónica HPSC en células beta pancreáticas-como sensibles a glucosa al imitar en el desarrollo embrionario del páncreas vivo. La combinación de la tecnología iCRISPR con la diferenciación dirigida HPSC permite el estudio sistemático de la función de genes para mejorar nuestra comprensión de los mecanismos de desarrollo de la diabetes y las enfermedades del páncreas.
Introducción
Las células madre pluripotentes humanas (hPSCs) tienen la capacidad tanto de auto-renovarse y dar lugar a todos los derivados de los tres linajes germinales embrionarias. Ellos proporcionan un recurso valioso para la terapia de reemplazo celular y la modelización de enfermedades, al servir como una plataforma única para recapitular los procesos celulares en un contexto de desarrollo humano. Son también una fuente de células para los análisis experimentales escalables y de alto rendimiento. Sin embargo, el progreso ha sido limitado debido a dos retos principales: la falta de herramientas de modificación genética eficientes y la dificultad para recapitular los pasos del desarrollo embrionario complejos en una placa de cultivo.
La modificación genética es una herramienta indispensable para estudiar la función de genes en el desarrollo normal y la enfermedad. Sin embargo, mientras gen clásica estrategias selectivas mediante recombinación homóloga han demostrado ser una herramienta poderosa para diseccionar la función de genes en células madre embrionarias de ratón (mESCs) 1, este enfoqueha sido extremadamente ineficiente cuando se aplica a hPSCs 2, 3. La reciente adhesión a paso ligero de nucleasas programables, específicas del lugar de la naturaleza para uso en laboratorio, incluyendo nucleasas de dedos de zinc (ZFNs), transcripción activador de nucleasas efectoras (Talens) y los agrupados repeticiones palindrómicas cortas espaciadas regularmente (CRISPR) / CRISPR-asociado (Cas) sistemas 4, significa que la ingeniería del genoma se ha convertido en una tarea mucho más fácil en una amplia gama de organismos y líneas de células, incluso en hPSCs. Estas herramientas de edición gen se aprovechan del hecho de que las nucleasas quiméricas, tales como la endonucleasa de Cas9 pueden permitir una amplia gama de modificaciones genéticas mediante la inducción de roturas de doble cadena (DSBs) en lugares precisos, la activación de la maquinaria de reparación del ADN endógeno para activar o bien no homóloga terminar (NHEJ) o la reparación homología dirigida (HDR). Ambos mecanismos pueden ser explotados para la manipulación genética mediante la inducción de either inserción aleatoria y mutaciones de deleción (indeles; vía NHEJ), para crear mutaciones de desplazamiento del marco que anulan alelos de genes, o sustituciones de nucleótidos precisas (vía HDR), para recapitular las mutaciones de los pacientes para el modelado de la enfermedad humana o para corregir una mutación causante de la enfermedad para la terapia génica .
CRISPR / ingeniería genómica mediada por Cas requiere dos componentes: la constante de Cas9 endonucleasa de ARN guiada necesaria para la escisión de ADN y un ARN CRISPR variable (crRNA) y dúplex de activación trans (tracrRNA) que especifica de reconocimiento de diana de ADN. El dúplex crRNA / tracrRNA se puede reemplazar con una sola guía de ARN quimérico (gRNA), que se han encontrado para trabajar más eficientemente 5, 6, 7. Mientras que el sistema / Cas9 CRISPR se ha adaptado para los organismos más experimentales y líneas celulares, la entrega y la expresión de Cas9 y gRNA varía significativamente y necesita ser optimizado aún más a Achivíspera de edición genoma eficiente en muchos sistemas, incluyendo hPSCs 8. Una plataforma genoma de edición eficiente, iCRISPR, se ha establecido en 5 hPSCs. En este sistema, un enfoque mediada por TALEN se ha utilizado para orientar ambos alelos del AAVS1 "transgén puerto seguro locus" en trans, un alelo con un transactivador inverso controlado por tetraciclina (M2rtTA) y la otra con un elemento de respuesta de tetraciclina (TRE ) que dirige la expresión de Cas9 (iCas9) en los hPSCs. En líneas clonales establecidas (iCas9 hPSCs), Cas9 es altamente expresado con el tratamiento doxiciclina. Mientras tanto, debido a su pequeño tamaño (100 nt), gRNAs individuales o múltiples se pueden entregar fácilmente en iCas9 hPSCs con alta eficiencia y pueden dirigir Cas9 para la escisión específica del sitio, lo que permite eficiente interrupción de genes mediada por NHEJ, así como mediada por HDR modificaciones de nucleótidos precisas en presencia de plantillas de donantes cortas de ADN de cadena sencilla (ssDNA). El sistema puede ser iCRISPRutilizado con éxito para generar un panel de líneas HPSC que imitan la enfermedad con bialélicas (homocigotas o heterocigotas compuestas) o mutaciones heterocigotas de pérdida de función en los genes de desarrollo importantes 5, 9. Aunque varios grupos han informado de edición génica eficaz usando CRISPR / Cas en hPSCs, el éxito sigue siendo limitada a un pequeño número de laboratorios tecnológicamente adeptos. La plataforma iCRISPR ofrece una solución eficiente y simple para la edición gen rutina por investigadores de diferentes niveles de habilidad, y que ya ha sido utilizado en una serie de estudios publicados por nuestro grupo y otros 9, 10, 11, 12. Este enfoque también se ha ampliado al silenciamiento inducible basado en la expresión de dCas9-KRAB 13.
Junto con los avances en la tecno-edición genomalogía, mejoras significativas también se han logrado en HPSC mantenimiento y diferenciación dirigida. Las condiciones de cultivo para hPSCs han evolucionado a partir de las condiciones medias del ratón irradiado de fibroblastos embrionarios (IMEF) alimentador dependiente de condiciones libres de alimentador de los componentes de la matriz extracelular definidos, ya partir de formulaciones de medios complejos de constitución química definida 14. Estas mejoras han reducido la variabilidad en hPSCs debido a las diferencias de lote a lote en la preparación de los componentes de recambio y suero nocaut IMEF, y por lo tanto proporcionar un entorno más reproducible para la diferenciación HPSC. Mientras tanto, un mejor conocimiento de las vías que regulan el desarrollo embrionario humano, así como los descubrimientos de exámenes de drogas de alto rendimiento de señalización, se han llevado a mejorar los protocolos de diferenciación 15, 16, 17, 18. Estos protocolos se asemejan más en vivo pasos de desarrollo y generar tipos celulares que recapitular estrechamente sus homólogos en vivo. Para la diferenciación HPSC en el linaje de páncreas, protocolos iniciales imitaban el desarrollo de páncreas principios relativamente bien, pero finalmente generada células ß polyhormonal que eran de fenotipos fetales inmaduros y respondieron mal a la estimulación de la glucosa. Los avances recientes 16, 17, 19, 20 han permitido la generación de las células beta pancreáticas-como sensibles a glucosa, lo que nos permitirá investigar los acontecimientos posteriores, tales como la formación y la maduración de las células ß más monohormonal.
A continuación, detallamos la aplicación de las líneas del genoma modificado para el estudio del desarrollo de páncreas mediante la combinación del sistema de iCRISPR con la plataforma basada en HPSC in vitro diferenciación hacia pancr respuesta a la glucosacélulas ß-como eatic. Este acoplamiento de potentes herramientas de edición genoma con un protocolo mejorado la diferenciación HPSC no sólo ofrece la velocidad y la escala necesaria para satisfacer la creciente demanda de la validación de la causalidad de la enfermedad, sino que también permite manipulaciones genéticas sofisticadas para realizar nuevas investigaciones mecanicistas en el control transcripcional que subyacen el desarrollo normal y la enfermedad 9 .
Protocolo
Este protocolo se basa en nuestro trabajo con líneas HPSC H1, HUES8, y MEL-1 en la condición químicamente definido y libre de alimentador (Por favor, vea la Tabla Material y Equipo). Para otras líneas HPSC o hPSCs mantenidas en diferentes condiciones de cultivo, se recomienda una mayor optimización.
1. HPSC Cultura en el Estado de constitución química definida y libre de alimentador
- Adaptar la cultura HPSC en alimentadores IMEF a la condición libre de alimentador. En los casos en que las células congeladas no sobreviven bien cuando se recuperan directamente en la condición libre de alimentador, recuperar las células en condiciones IMEF primero y luego adaptarse a la condición libre de alimentador.
NOTA: En general, se necesitan 2 pasajes para hPSCs cultivadas en alimentadores IMEF que adaptarse a la condición de libre de alimentador. - Cambiar el medio cada día y el paso de los hPSCs cuando las células han alcanzado ~ 80% de confluencia. En general, hPSCs paso a ~ 1: 6 - 1:15 proporciones de cada 4 - 6 días. Añadir 10 mM inhibidor ROCA Y-27632 WHen la descongelación o pases las células.
- Antes de la siembra de las hPSCs, placas de cultivo pre-capa con 5 mg / ml (1 ml / 10 cm 2) truncada forma humana recombinante de vitronectina (VTN) durante al menos 1 h a temperatura ambiente (RT). Además, preparar el medio químicamente definido completa mediante la adición de suplemento en el medio basal.
- Retirar el medio de cultivo, lavar las células una vez con PBS sin Ca2 + y Mg2 +, y el tratamiento de las células con EDTA 0,5 mM para ~ 2-5 min a TA.
- Aspirar el EDTA antes de que las colonias se han desprendido. Con pipeteo suave, dispersar las colonias HPSC en trozos pequeños y resuspender las células en medio completo.
- Recoge los hPSCs disociados y centrifugar las células a 200 xg durante 5 min. Volver a suspender las hPSCs sedimentadas en el medio completo y sembrar las células en placas recubiertas con VTN.
2. Generación de Líneas iCas9 HPSC
- Orden y amplificar los plásmidos siguientes: AAVS1-TALEN-L, T-AAVS1ALEN-R, AAVS1-Neo-M2rtTA, y AAVS1-Puro-iCas9.
NOTA: Para evitar eventos inesperados de recombinación, usar células competentes Stbl3 recombinación deficiente para la transformación y la amplificación de los plásmidos a 30 ° C. - Por lo general, la preparación de los hPSCs en un plato de 10 cm (~ 1 x 10 7 células si ~ 80% de confluencia) para un experimento de orientación.
NOTA: Dado que la electroporación generalmente causa la muerte celular significativa y génica mediada por la orientación TALEN en el locus AAVS1 requiere la selección de antibióticos, tiene que ser cabeza de serie para identificar correctamente las células específicas de un número relativamente grande de células. Se recomienda Optimización para la concentración del fármaco para cada línea celular y cada condición de cultivo. - En el Día -1, (el día antes de la electroporación), añadir inhibidor Rock 10 mM durante el cambio de medios de comunicación.
- En el Día 0, (el día de la electroporación), preparar placas recubiertas con VTN con antelación.
- Disociar las hPSCs en células individuales USIng 1x reactivo de disociación (consulte la Tabla Material y Equipo). En pocas palabras, eliminar el medio de cultivo, se lavan las células una vez con PBS sin Ca2 + y Mg2 +, y el tratamiento de las células con el reactivo de disociación 1x a 37 ° C durante ~ 3 min. Aspirar el reactivo de disociación antes se han desprendido las células. Con pipeteo suave, dispersar las hPSCs en una suspensión de una sola célula en 10,5 ml de medio completo.
- Tomar 0,5 ml de suspensión de células para contar el número de células utilizando un contador de células automatizado. Sedimentar las hPSCs a 200 xg durante 5 min y resuspender las células en frío (4 ° C) PBS a 12,5 x 10 6 células / ml.
- Añadir los plásmidos (véase la Tabla 1) en 800 l de suspensión HPSC (12,5 x 10 6 células / ml) y mezclar bien. Transferir la mezcla a una cubeta de electroporación de 0,4-cm y mantener en hielo durante ~ 5 min.
- Electroporar las células usando un sistema de electroporación a 250 V y 500 mF; las constantes de tiempo observed después de la electroporación es normalmente 9 - 13 ms.
- Después de la electroporación, la transferencia de las células a un tubo cónico de 15 ml con 5 ml de medio completo pre-calentado. Crítico para el éxito de la focalización: Use hPSCs proliferantes saludable y manejar las células con mucho cuidado cuando se transfieren, resuspensión, y en placas de las células después de la electroporación.
- Sedimentar las células a 200 xg durante 5 min. Resuspender las células en 10 ml de medio completo con 10 mM inhibidor de la ROCA y la placa 1, 2,5, y 5 x 10 6 células en cada uno de los platos de tres VTN-revestido, de 10 cm; esto asegura que al menos una de las placas tendrán suficiente de colonias a una densidad clonal de una sola célula para el picking de colonia.
- En el Día 1 (el día después de la electroporación), cambiar el medio.
- En los días 2-5, inicio de la selección de neomicina cuando las células son ~ 60% de confluencia. Cambiar el medio a diario con 500 mg / ml de G418 sulfato; la muerte celular significativa debido a selection se observa típicamente 2 días después de la selección con G418.
- En el Día 6, cambiar el medio sin selección de antibióticos.
- En los días 7-9, iniciar la selección de puromicina. Cambiar el medio diariamente con 1 mg de dihidrocloruro de puromicina / mL; muerte celular significativa debe observarse el día siguiente.
- En el Día 10, empezar a cambiar el medio todos los días sin selección de antibióticos hasta que las colonias de una sola célula HPSC llegar a 1 - 2 mm de diámetro.
NOTA: Por lo general, 50 colonias en una placa de 10 cm se observan con 2,5 x 10 6 hPSCs plateado en el día 0. - Recoger 12 - 24 colonias bajo un microscopio estereoscópico. Mecánicamente desagregar las colonias HPSC en trozos pequeños (~ 10 piezas por colonia) utilizando una aguja de 23 G (una punta de pipeta de 200 l es también fino) y transfieren las células directamente en revestidas-VTN placas de 24 pocillos.
- Cambiar el medio todos los días hasta que las células se hacen confluentes. El paso de las células en cada pocillo de las placas de 24 pocillos enduplicar los pocillos de placas de 6 pocillos.
- Cuando las células se hacen confluentes en las placas de 6 pocillos, utilizar un pocillo de una solución madre congelada y el otro bien para la extracción de ADN genómico para una caracterización adicional.
- Caracterizar y validar las líneas iCas9 establecidos por PCR genotipo, transferencia de Southern, análisis de RT-qPCR, cariotipo, y el ensayo de pluripotencia. Consulte a Zhu et al. 21 para los procedimientos experimentales detallados.
3. Generación de HPSC mutante Líneas Uso del sistema de iCRISPR
- Generación de líneas knockout HPSC
- diseño y producción gRNA
- Elige regiones diana en el gen de interés para maximizar la posibilidad de interrumpir la función de proteína de tipo salvaje. Para los genes bien anotado, elegir una región diana cadena arriba de un dominio funcional esencial. Alternativamente, gRNAs diseño para apuntar a una región aguas abajo del codón de inicio. Elija por lo menos 2 diferentesregiones de un gen de interés.
- gRNAs de diseño utilizando la herramienta de diseño en línea de CRISPR (http://crispr.mit.edu). Para cada región diana, diseño 3 gRNAs con bajo potencial fuera de los objetivos y utilizar el que tiene la más alta eficiencia de focalización para la generación clonal línea mutante 22.
NOTA: Con el fin de lograr la eficiencia edición de alta genoma, se recomienda para entregar gRNA como oligos de ARN en lugar de ADN como un plásmido debido a la mayor eficiencia de transfección de ARN pequeños en comparación con plásmidos en la experiencia pasada. - Pedido 120 nucleótidos (nt) oligos de ADN que contienen la secuencia del promotor T7, la secuencia variable de 20-nt crRNA reconocimiento (N) 20 (no incluyen la secuencia PAM), y la secuencia guía quimérico constante. Diluir los oligos a 100 mM solución madre en ddH2O y preparar 250 nM como solución de trabajo.
NOTA: TAATACGACTCACTATAGGG (N) 20 GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTT - PCR-amplificar los oligos utilizando los cebadores T7F y TracrR (véase la Tabla 2) para producir la plantilla de ADN de doble cadena (dsDNA) para gRNA transcripción in vitro (IVT). Utilice 50 l de mezcla de reacción de PCR (véase la Tabla 3) y las condiciones del ciclo de PCR (ver Tabla 4).
- Utilice un kit de transcripción T7 de alto rendimiento para vitro gRNA transcripción en con la plantilla amplificado por PCR en 20 l, vitro gRNA mezcla en la transcripción (véase la Tabla 5) según las instrucciones del fabricante. Purificar los productos gRNA utilizando el kit de transcripción de limpieza según las instrucciones del fabricante.
- Eluir gRNAs siguiendo el protocolo de purificación de alto rendimiento según las instrucciones del fabricante (típicamente ~ 50 - 100 g) en 100 l de tampón de elución. Ajustar la concentración de 320 ng / l (10 M) cuando sea posible y se almacena a -80 y# 176; C hasta su uso.
- PCR y secuenciación de Sanger primer diseño
- Diseño y validación de los cebadores de PCR que amplifican la región de destino, con tamaños de productos típicamente van desde ~ 500 - 1.000 pb.
- Diseño Sanger cebadores de secuenciación de unión internamente a los productos de la PCR para permitir la secuenciación directa de los productos de PCR sin purificación.
- transfección gRNA en iCas9 hPSCs
- En el Día -1, tratar las células iCas9 con 2 mg / ml de doxiciclina 24 h antes de la transfección gRNA.
- En el Día 0 de gRNA transfección, preparar placas recubiertas con VTN con antelación.
- Disociar las células iCas9 en células individuales utilizando el reactivo de disociación 1x, como se describe en el paso 2.5.
- Sedimentar las hPSCs a 200 xg durante 5 min y resuspender las células en ~ 0,5 x 10 6 células / ml en medio completo suplementado con 2 mg / ml de doxiciclina y el inhibidor de Rock 10 mM.
- Plate 0,5 ml de las células resuspendidas en los pocillos individuales de placas de 24 pocillos. Preparar pozos adicionales para servir como controles no transfectadas.
- Para cada gRNA, hacer las siguientes mezclas de transfección: Mezclar A, 50 l de medio con suero reducido + 1 l de gRNA (10 M); Mezcla B, 50 l de medio con suero reducido + 3 l de reactivo de transfección.
- Combine la mezcla A y B para la fabricación de 100 l mezcla. Incubar durante 5 min a TA. Añadir 50 l de la mezcla a las células en los pocillos duplicados de las placas de 24 pocillos y se mezcla bien.
- En el Día 1, lleve a cabo una segunda transfección si es necesario para aumentar aún más la eficiencia de orientación. De lo contrario, cambiar el medio sin doxiciclina.
- En los días 2 - 3, cambiar el medio día.
- En el Día 4, extraer el ADN genómico de un pocillo de cada célula de control transfectadas y no transfectadas utilizando un kit de extracción de ADN. Ajustar la concentración de 50 ng / μL.
- PCR-amplificar las regiones diana que flanquean las secuencias de direccionamiento gRNA y estimar la eficiencia de edición utilizando digestión con endonucleasa de T7 (T7EI) o el ensayo de longitud de fragmentos de restricción polimórficos (RFLP), como se describe anteriormente 5.
- ensayo de T7EI
- PCR-amplificar la región diana usando los cebadores diseñados y validados en el paso 3.1.
- Preparar las mezclas (véase la Tabla 6) y llevar a cabo la desnaturalización del ADN y la hibridación de los productos de PCR usando las condiciones descritas en la Tabla 7.
NOTA: En base a nuestra experiencia, por lo general no es necesario purificar los productos de PCR para el ensayo T7EI utilizando nuestra condición de la PCR. Sin embargo, la purificación podría ser beneficioso en otras condiciones. - Realizar una digestión T7EI a 37 ° C durante 30 min utilizando 10 l de desnaturalizado y se hibridó producto de PCR y 0,2 l (2 U) de T7E1 (10 U / l).
- resolve las muestras de PCR digeridos con T7E1 por electroforesis en gel. ImageJ utilizar para determinar las intensidades de banda relativas de corte y el ADN sin cortar. Calcular la frecuencia indel usando la fórmula: (1 - (√ (1- (b + c)) / (a + b + c))) x 100, donde a es la intensidad del producto no digerido PCR y son b y c las intensidades de los productos escindidos-T7E1.
- ensayo de RFLP
Nota: en los casos en que un sitio de restricción está en estrecha proximidad (<5 pb) a un sitio de escisión Cas9 (3 pb 5 'de la secuencia PAM), un ensayo de RFLP se puede realizar para cuantificar la frecuencia indel.- Utilizar los mismos productos de PCR como se describe en el paso 3.1.4.1.
- Se digiere el producto de PCR con una enzima de restricción que contiene un sitio de restricción en estrecha proximidad al sitio de escisión Cas9.
- Resolver las muestras de PCR digeridos mediante electroforesis en gel. ImageJ utilizar para determinar las intensidades de banda relativas de corte y el ADN sin cortar. Calcular la indelfrecuencia utilizando la fórmula: a / (a + b + c) x 100, donde a es la intensidad del producto no digerido PCR y b y c son las intensidades de los productos digeridos.
- Establecimiento de líneas mutantes clonales
NOTA: la edición del Genoma en hPSCs utilizando el sistema de iCRISPR con gRNA transfección es altamente eficiente, y no se necesita la selección de antibióticos. Para establecer líneas clonales, es necesario para sembrar las células a una densidad relativamente baja para garantizar la formación de colonias derivadas de una sola célula.- Identificar gRNAs con la más alta eficiencia de edición (utilizando el T7EI o ensayo RFLP) y con una buena supervivencia de las células. Utilizar el duplicado correspondiente también para el establecimiento de la línea mutante clonal.
- Disociar el hPSCs en una suspensión de una sola célula usando el reactivo de disociación 1x, como se describe en el paso 2.5. Replate 500, 1.000, y 2.000 células en cada uno de los platos tres VTN-revestido, de 10 cm.
- Cambiar el medio de u diariaasta el de una sola célula colonias alcanzan ~ 2 mm de diámetro.
- Recoger 24 - 48 colonias para cada gRNA, en función de la estimación de la eficiencia de la focalización por la T7EI y / o ensayo de RFLP. Mecánicamente desagregar cada colonia en trozos pequeños (~ 10 piezas por colonia) utilizando una aguja 23-G (una punta de pipeta de 200 l también es bien) y replate las células por duplicado, placas de 96 pocillos recubiertas con VTN. Use un plato para la extracción de ADN genómico y la secuenciación de Sanger y la otra placa para una mayor expansión.
- Cuando las células en las placas de 96 pocillos se han convertido en confluentes, extraer el ADN genómico (sin extracción con fenol / cloroformo) usando un protocolo simple, como se indica a continuación.
- Se elimina el medio y se lavan las células una vez con PBS sin Ca2 + y Mg2 +. Añadir 50 l de tampón de lisis (5 l de proteinasa K (10 mg / ml), 5 l de tampón de PCR 10x, y 40 l de ddH 2 O) a cada pocillo de una placa de 96 pocillos. Sellar la placa con una lámina adhesiva y Incubadoraste durante la noche a 55 ° C.
- El día siguiente, la transferencia de los lisados de células en una placa de PCR de 96 pocillos e incubar durante 10 min a 99 ° C en un termociclador para inactivar la proteinasa K.
- PCR-amplificar la región diana usando los mismos cebadores que para la T7EI o ensayo de RFLP, usando 1 l de lisado de células como una plantilla.
- Utilice 1 l del producto de PCR para la secuenciación de Sanger con una unión internamente al producto de la PCR del cebador.
- Amplificar los clones con mutaciones de desplazamiento del marco de indel existencias congeladas. Además, amplificar un par de clones de tipo salvaje del mismo experimento orientación para servir como líneas de control isogénicas para experimentos adicionales.
- diseño y producción gRNA
- Generación de líneas mutantes con alteraciones de nucleótidos precisas
NOTA: En comparación con los mutantes knockout generados por extremos no homólogos (NHEJ), alteración de nucleótidos preciso puede lograrse a través de homología dirigida reparación (HDR) en presencia de ADN repaiplantillas r. Tales alteraciones de nucleótidos precisas permiten la generación de mutaciones específicas para cada paciente en hPSCs de tipo salvaje y para la corrección de mutaciones en células iPS derivadas de pacientes.- Diseño de ssDNA como una plantilla de HDR
- Diseño y producción de 2 - 3 gRNAs en las proximidades de una mutación específica del paciente, tal como se describe en el paso 3.1.1.
- Diseño de un ADN monocatenario (ssDNA) que contiene la mutación específica del paciente flanqueado por ~ 40 - 80 nt de homología en cada lado como una plantilla de HDR.
- Para reducir de corte adicional después de la reparación correcta, introducir una mutación silenciosa a la plantilla ssDNA en la región dentro de la secuencia de reconocimiento gRNA y en estrecha proximidad a la secuencia de PAM o en la secuencia de PAM sí mismo, si es posible.
- Si es posible, el diseño de la mutación silenciosa para introducir un nuevo sitio de restricción de la digestión, así por lo que se puede utilizar para estimar la eficiencia de reparación utilizando el ensayo de RFLP.
- GRNA / ssDNA co-transfección y el establecimiento de líneas clonales
- Realizar la co-transfección de gRNA / ssDNA en células iCas9, como se describe en el paso 3.1.3, con mezclas de transfección A y B. Para cada gRNA y control no transfectadas, transfectar las células en pocillos duplicados de placas de 24 pocillos. Mezcla A: 50 l de medio de suero reducido + 1 l de gRNA (10 M) + 2 l de ssDNA (10 mM). Mezcla B: 50 l de medio con suero reducido + 3 l de reactivo de transfección.
- Después de la transfección, se extrajo el ADN genómico de un pocillo de cada célula de control transfectadas y no transfectadas y estimar la eficiencia de reparación utilizando la T7EI y / o ensayo de RFLP.
- Identificar la mezcla gRNA / ssDNA con la más alta eficiencia de reparación y una buena supervivencia de las células. Utilizar el duplicado correspondiente también para el establecimiento de la línea mutante clonal.
- Recoger 48 - 96 colonias, dependiendo de la estimación de la eficacia de dirección por el T7EI y / o culo RFLPsí. En general, la eficiencia de mutación mediada por HDR es menor que knockout mutación y por lo tanto más colonias deben ser recogidos.
- Secuencia, ampliar y validar las líneas clonales, tal como se describe en el paso 3.1.6.
- Diseño de ssDNA como una plantilla de HDR
4. En Vitro HPSC diferenciación en células ß pancreáticas sensibles a glucosa
NOTA: En la diferenciación in vitro de mutantes HPSC en tipos celulares relevantes de la enfermedad proporciona una plataforma para el modelado de la enfermedad en un plato. El siguiente protocolo se centra en la diferenciación in vitro de hPSCs en células ß pancreáticas sensibles a glucosa para estudios de desarrollo y diabéticos pancreáticas 9, 16, 17.
- la diferenciación HPSC en endodermo definitivo
- Mantener mutantes HPSC y líneas de tipo salvaje de control en el estado químicamente definido y para los enlaces de conexión, como se ha descritod en el paso 1.
- Para preparar los hPSCs para la diferenciación, disociar las hPSCs usando el reactivo de disociación 1x y dispersarlos en suspensión de una sola célula en medio completo.
- Sedimentar las células a 200 xg durante 5 min y volver a suspender las células en medio completo con el inhibidor de Rock 10 mM. Contar el número de células y sembrar las células en ~ 1,4 x 10 5 células / cm2 en placas recubiertas con VTN.
- Cambiar el medio 24 h después de la siembra.
- En el Día 0, iniciar la diferenciación después de 48 h, cuando las células han llegado a ~ 80% de confluencia.
NOTA: Para lograr una alta eficiencia de diferenciación de páncreas, la optimización de la densidad de siembra y el nivel de confluencia a las 48 h se recomienda para cada línea individual. - Aspirar el medio HPSC y enjuagar las células una vez con PBS sin Ca2 + y Mg2 +.
- Cambiar el medio a día 0 (D0) medio de diferenciación.
- En los Días 1 - 2, cambiar el diferenciación medio día, de acuerdo a las recetas de la Tabla 8.
- En el Día 3, examinar los marcadores endodermo definitivo sox17, FoxA2, y CXCR4 por la tinción de inmunofluorescencia y análisis de citometría de flujo.
- La diferenciación endodermo definitivo en progenitoras de páncreas
- En los días 3-9, continuar la diferenciación endodermo definitivo hacia el linaje pancreático cambiando el medio a diario, según las recetas de la Tabla 9.
- En el Día 7, examinar la temprana progenitor de páncreas (PP1) marcador PDX1 por tinción de inmunofluorescencia y citometría de flujo análisis.
- En el día 10, después de examinar la progenitora pancreática (PP2) y marcadores PDX1 Nkx6.1. Mientras tanto, se preparan para transferir las células a PP2 interfase aire-líquido para su posterior diferenciación en células endocrinas pancreáticas.
- Endocrina pancreática difierenciación en la interfase aire-líquido
- El tratamiento de las células con el inhibidor PP2 Rock 10 M 4 h antes de la disociación.
- Retirar el medio y lavar las células una vez con PBS sin Ca2 + y Mg2 +.
- Añadir 2 ml de reactivo de disociación 1x a las células PP2 en una placa de 10 cm y se incuba a 37 ° C durante 2-3 min.
- Aspirar el reactivo de disociación antes se han desprendido las células. Añadir 10 ml de medio BLAR y dispersar las células PP2 en células individuales pipeteando suavemente hacia arriba y hacia abajo.
- Recoger la suspensión de una sola célula. Contar el número de células y de pellets a 200 xg durante 5 min.
- Resuspender el sedimento celular en ~ 0,5 x 10 5 células / l en medio de diferenciación S5 y la mancha de 5 - 10 l de células por punto en un filtro transwell inserto. Colocar 10 - 15 puntos en una pieza de inserción de 6 pocillos y ~ 100 puntos en un inserto de 10-cm.
- Añadir medio S5 a la parte inferior de cada inserción transwell, ~ 1,5 ml para las inserciones 6 pocillos y ~ 8 ml fo inserciones de 10 cm.
- Cambiar el medio a diario con las recetas de la Tabla 10.
- Examine páncreas marcadores endocrinos PDX1, Nkx6.1 NeuroD1, NKX2.2, insulina y glucagón en el día 34 por la tinción de inmunofluorescencia y análisis de citometría de flujo 17.
- Examinar la función de células β-como derivados de HPSC con un ensayo de secreción de insulina estimulada por glucosa (GSIS) 16, 17.
Resultados
HPSC química definida y libre de alimentador de Cultura adaptación y mantenimiento
hPSCs cultivadas en alimentadores IMEF pueden adaptarse rápidamente a las placas recubiertas con VTN en la condición de cultivo libre de alimentador. La misma relación de división como de cultivo normal alimentador IMEF se puede utilizar durante la adaptación. La figura 1A muestra morfologías típicas de hPSCs en los alimentadores IMEF en medio a base de KSR y sobre una superficie recubierta con VTN en medio libre de alimentador. La Figura 1B muestra un cambio morfológico típico y el crecimiento de colonias HPSC durante la primera paso de adaptación (4 días). Las células pueden ser más passaged o congelados para futuros experimentos. Realizar 2 - 3 pasajes de la cultura de adaptación antes de comenzar los experimentos de diferenciación. Cariotipo También se recomienda después de la adaptación, aunque no hemos observado anomalías del cariotipo durante la fase de adaptación.
"Fo: keep-together.within-page =" 1 "> AAVS1 mediada por TALEN Generación de Líneas iCas9 HPSC través de la orientaciónComo se indica anteriormente, hPSCs se sometieron a electroporación con un par de AAVS1 plásmidos TALEN y Cas9 y M2rtTA plásmidos 5. Figura 2A y B muestran vector donante de diseño detallado y todo el procedimiento para generar líneas clonales iCas9 HPSC orientación. Después de la selección de antibióticos, los clones derivados de una sola célula que muestran un tamaño adecuado y morfología típica HPSC estaban listos para ser recogidos (10 - 12 días después de la electroporación, la Figura 2C). Por lo general, ~ 50% de los clones están dirigidos correctamente sin integraciones aleatorias, según lo verificado por Southern Blot (Figura 2D) 5. integración aleatoria podría causar la expresión de fugas Cas9 en ausencia de tratamiento doxiciclina.
M eficiente genéticaODIFICACIÓN en hPSCs utilizando la plataforma iCRISPR
En establecida iCas9 hPSCs, Cas9 se expresa con el tratamiento con doxiciclina y guió a su locus diana por las gRNAs transfectadas, donde genera DSB. En la ausencia de un modelo de reparación, la reparación del ADN a través de NHEJ genera indeles, que a menudo resultan en la alteración del gen o nocaut. En presencia de una plantilla de reparación (por ejemplo, un donante de ssDNA), HDR se puede emplear para las alteraciones genéticas precisas, tales como la generación de una mutación específica del paciente en un fondo HPSC de tipo salvaje o la corrección de una variante genética asociada a la enfermedad en el paciente iPS derivadas (Figura 3A). Se tarda aproximadamente 1 mes para generar líneas mutantes clonados utilizando el sistema de iCRISPR. Después de la inducción iCas9 y transfección gRNA, se utilizaron ensayos de T7EI y / o RFLP para evaluar la eficiencia de corte Cas9, y las células transfectadas se sembraron más tarde como células individuales en placas de 10 cm a baja densidad (~ 500 - 2, 000células / placa de 10 cm). 10 - 12 días más tarde, los clones derivados de una sola célula se colocaron en los pocillos de una placa de 96 pocillos para la expansión y la caracterización adicional (es decir, el genotipado y Western Blot) (Figura 3B). Dado que la eliminación de genes mediada por iCRISPR es altamente eficiente, ningún proceso de selección está implicado, y por lo general 20 - 50% mutantes bialélicos se puede conseguir fácilmente (Figura 3C) 5. Para alteraciones genéticas eficientes y precisos, gRNAs se co-transfectaron con un donante de ssDNA que lleva la alteración de la secuencia específica (por un cambio pequeño pero específica de secuencia del genoma). A menudo, para evitar la re-corte en alelos modificados, se recomienda incluir una mutación silenciosa en las proximidades de o en la secuencia de PAM (Figura 3D). Con este sistema, se consiguen ~ 10% de los clones que llevan la modificación del genoma HDR mediada deseada sin alteraciones adicionales en ambos alelos (Figuras 3E). El rápido y precise modificación del genoma HPSC utilizando la plataforma iCRISPR nos permite realizar de forma rápida y eficiente líneas HPSC que sirven como modelos para el estudio del desarrollo humano y la enfermedad.
La diferenciación eficiente de hPSCs hacia las células ß-como respuesta a la glucosa
Los recientes progresos en la diferenciación de páncreas HPSC ha permitido el desarrollo de protocolos que recapitular de cerca el desarrollo embrionario del páncreas. HPSCs indiferenciadas primera diferenciadas en el endodermo definitivo, luego en PDX1 + progenitores pancreáticos tempranos (PP1) y más tarde células progenitoras pancreáticas (PP2) PDX1 + Nkx6.1 +, y, finalmente, en respuesta a la glucosa de células 16, 17, 19 β-como, 20 , 23, 24. estos protocoles se optimizaron además para generar de forma fiable las células ß-como sensibles a glucosa + 9 PDX1 Nkx6.1 + progenitores pancreáticos y. La Figura 4A muestra los suplementos químicos detallados utilizados en cada etapa de la diferenciación. Por lo general, ~ 80% de confluencia 2 días después de que el recubrimiento inicial de células es ideal para el inicio de la diferenciación de HUES8 hPSCs (Figura 4B). Probar varias diferentes densidades de siembra es muy recomendable para descubrir la condición óptima para cada línea celular específica. Por lo general, al menos 75% FOXA2 + SOX17 pueden lograrse + células en la DE etapa y 40% PDX1 + células Nkx6.1 + en la etapa PP2 (Figura 4C). En la etapa S5, la presencia de una forma aura-como todo el agregado celular es un indicador de la buena supervivencia de las células, que es importante para la diferenciación adicional a las células ß-como CPEP + sensibles a glucosa Nkx6.1 + (Figura 4D ). Este protocolo es útil para el estudio de páncreas d humana esarrollo y la enfermedad en un plato.

Figura 1. HPSC química definida y mantenimiento libre de alimentador y Cultura Adaptación del IMEF alimentadores. (A) Imágenes representativas de hPSCs cultivaron en alimentador IMEF o en VTN en el día 4, listo para ser dividido. (B) la morfología típica de hPSCs durante el primer paso de la adaptación en cultivo libre de alimentador de un alimentador IMEF. Día 4 hPSCs cultivadas en alimentadores IMEF se dividieron y se sembraron en placas recubiertas con VTN en medio definido químicamente con un inhibidor de roca. La relación de división era la misma que la relación de división habitual para el cultivo en condiciones de alimentación IMEF. El medio se cambió cada día sin inhibidor de la roca. Haga clic aquí para ver una versión más grande de esta figura.
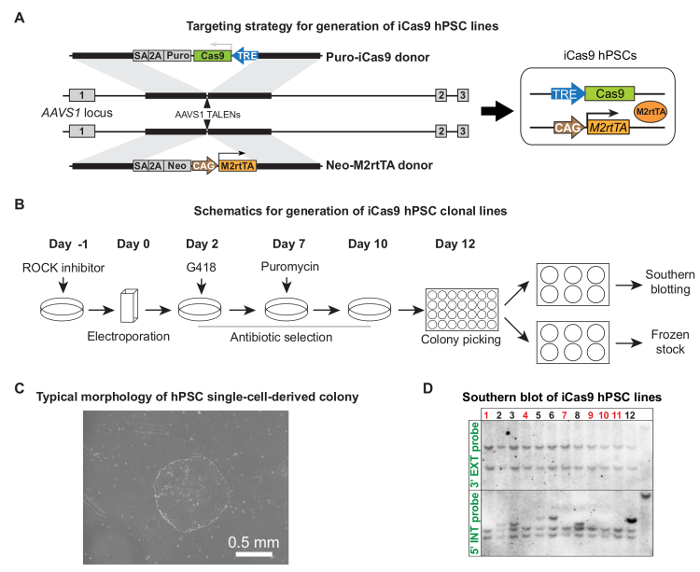
Figura 2. Generación de Líneas iCas9 HPSC. (A) Orientación de la estrategia para la generación de líneas iCas9 HPSC. Puro-Cas9 donante y donante Neo-M2rtTA fueron atacados en el locus AAVS1 humano por un par de AAVS1 Talens. (B) Esquema de todo el proceso de orientación, incluyendo la electroporación, la selección de antibióticos, la recolección y la expansión de colonias, y de caracterización de líneas de células. (C) en los clones de una sola célula Representante listo para ser recogido en torno a los 10 - 12 días después de la electroporación. (D) Los ejemplos de Southern blot para identificar clones correctamente orientados sin la integración adicional de dos plásmidos donantes. Los clones correctos están marcados en rojo. (A) y (D) se adaptaron a partir de Referencia 5, con el permiso.ig2large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
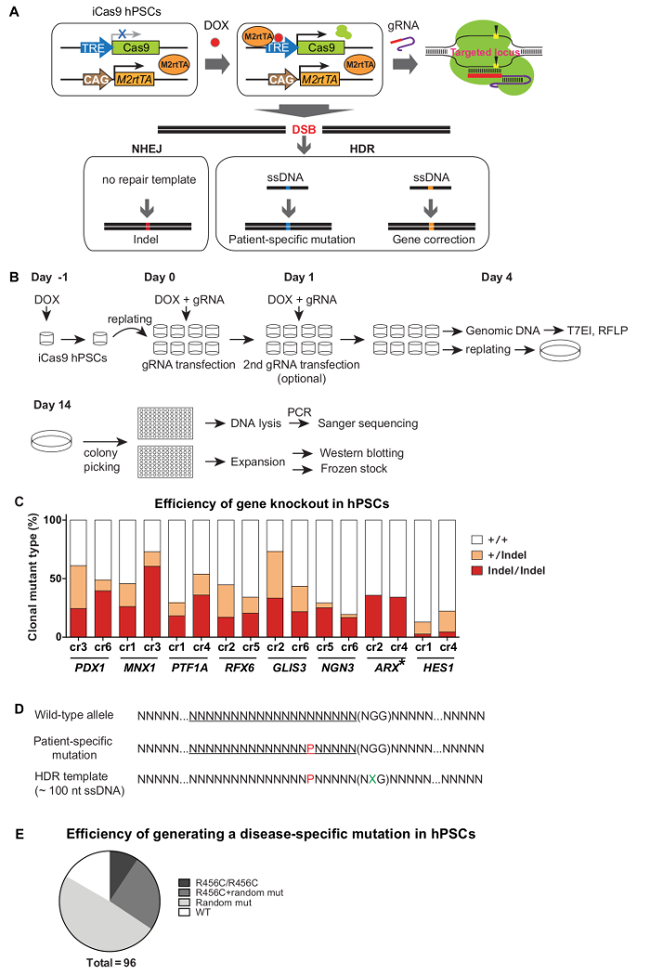
Figura 3. Modificación genética eficiente en hPSCs Uso del sistema de iCRISPR. Esquemas (A) para la generación de mutantes knockout de genes o la modificación genética precisa utilizando el sistema iCRISPR. (B) procedimiento de focalización y el establecimiento línea clonal. La eficiencia de eliminación directa (C) Gen través de NHEJ en hPSCs 9. (D) Diseño de un ssDNA que lleva una modificación de nucleótido específica. P, en rojo: la mutación específica del paciente; X, en verde: mutación silenciosa. (E) Eficiencia de la modificación de nucleótidos precisa mediada HDR. Una mutación R456C precisa (causada por un nucleótido C específica> T mutación) en el locus GATA6 se introdujo usando el sistema de iCRISPR.(Consulte la Referencia 5 para obtener información más detallada). (C) y (E) han sido adaptados de Referencia 9 y Referencia 5, respectivamente, con los permisos. Haga clic aquí para ver una versión más grande de esta figura.
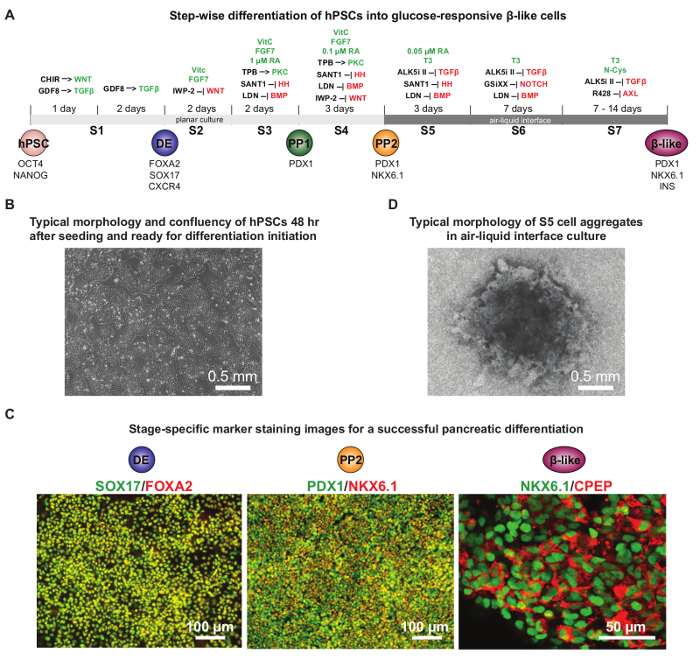
Figura 4. Dirigido HPSC diferenciación en células beta pancreáticas-como respuesta a la glucosa. Esquemas (A) del protocolo de diferenciación detallada, con productos químicos complementados en cada etapa de la diferenciación. las vías de señalización que se activan o inhiben durante la diferenciación se destacan en verde o rojo, respectivamente. CHIR: inhibidor de GSK3; GDF8: el crecimiento factor de diferenciación de 8 o miostatina, un miembro de la familia de proteínas TGF-beta; HH: erizo; DE: endodermo definitivo; PP1:PDX1 + progenitora pancreática temprana; PP2: PDX1 + Nkx6.1 + más tarde progenitor de páncreas. (B) de confluencia Típica (70 - 80%) de hPSCs 48 h después de la siembra cuando esté listo para la iniciación diferenciación. (C) Las imágenes de tinción de inmunofluorescencia representativo que muestra la diferenciación éxito en el endodermo definitivo (DE) etapa (FOXA2 y SOX17 co-tinción), etapa progenitor de páncreas (PP2: PDX1 y Nkx6.1 co-tinción), y respuesta a la glucosa β- como etapa de célula (Nkx6.1 y c-péptido co-tinción). En general, para lograr más de 10% de células CPEP + Nkx6.1 + en la etapa de célula β-como, al menos 75 + células DE sox17% FOXA2 + y 40% PDX1 + se requieren células Nkx6.1 + PP2 en las fases correspondientes. (D) la morfología típica de un agregado celular en la etapa S5 en una membrana de filtro de inserto en la cultura interfase aire-líquido. Las células supervivientes han migrado al centro del agregado (en la oscuridad) y se dejó una estructura similar aura en el borde. (A ) y (D) se adaptó de Referencia 9 con permisos. Haga clic aquí para ver una versión más grande de esta figura.
| plásmido | Cantidad |
| AAVS1-TALEN-L | 5 g |
| AAVS1-TALEN-R | 5 g |
| AAVS1-Puro-iCas9 | 40 g |
| AAVS1-Neo-M2rtTA | 40 g |
tabla 1
| Cebador | Secuencia |
| T7F | TAATACGACTCACTATAGGG |
| TracrR | AAAAGCACCGACTCGGTGCC |
Tabla 2
| Componente | Cantidad |
| ddH2O | 35,5 l |
| tampón de reacción 5x PCR | 10 l |
| mezcla de dNTP (25 mM) | 0,5 l |
| T7F (10 M) | 1,25 l |
| TracrR (10 M) | 1,25 l |
| T7-gRNA plantilla IVT (250 nM) | 1 l |
| ADN polimerasa | 0,5 l |
| mezcla total de reacción de PCR | 50 l |
Tabla 3
| Condiciones de ciclos de PCR | |||
| número de ciclo | Desnaturalizar | Recocer | Ampliar |
| 1 | 94 ° C, 2 min | ||
| 2-31 | 94 ºC, 20 s | 60 ºC, 20 s | 72 ° C, 1 min |
| 32 | 72 ° C, 2 min | ||
Tabla 4
| Componente | Cantidad |
| T7 ATP | 2 l |
| CTP T7 | 2 l |
| T7 GTP | 2 l |
| UTP T7 | 2 l |
| T7 tampón 10x | 2 l |
| mezcla de enzimas T7 | 2 l |
| PCR amplificado plantilla | 8 l |
| Total vitro gRNA mezcla de transcripción in | 20 l |
| Se incuba a 37 ° C durante 6 horas hasta toda la noche |
Tabla 5
| Componente | Cantidad (l) |
| No purificado producto de PCR | 8 |
| Buffer 2 10x | 2 |
| El agua destilada (dH2O) | 10 |
Tabla 6
| Desnaturalización del ADN y la hibridación de las condiciones del ciclo | ||
| Temperatura | Duración | Las condiciones del termociclador |
| 95 ° C | 10 minutos | |
| 85 ° C | 1 minuto | Rampa a 85 ° C a 2 ° C / s |
| 75 ° C | 1 minuto | Rampa a 75 ° C a 0.3 ° C / s |
| 65 ° C | 1 minuto | Rampa a 65 ° C a 0.3 ° C / s |
| 55 ° C | 1 minuto | Rampa a 55 ° C a 0.3 ° C / s |
| 45 ° C | 1 minuto | Rampa a 45 ° C a 0.3 ° C / s |
| 35 ° C | 1 minuto | Rampa a 35 ° C a 0.3 ° C / s |
| 25 ° C | 1 minuto | Rampa a 25 ° C a 0.3 ° C / s |
| 4 ° C | Sostener | |
Tabla 7
| Escenario | Día | Medios de comunicación | Suplemento | |
| S1 | D0 | S1 | GDF8 100 ng / mL | CHIR-99021 3 M |
| d1 | S1 | GDF8 100 ng / mL | CHIR-99021 0,3 M | |
| d2 | S1 | GDF8 100 ng / mL | ||
| Medios S1: MCDB 131 + 1x suplemento de L-glutamina + 0,5% de BSA + 1,5 g / L de NaHCO 3 + mM Glucosa 10 | ||||
Tabla 8
| Escenario | Día | Medios de comunicación | Suplemento | |||||||
| S2 | D3-D4 | S1 | LAA 0,25 mM | FGF7 50 ng / mL | IWP-2 2,5 M | |||||
| S3 | d5 d6 | S3 | LAA 0,25 mM | FGF7 50 ng / mL | SANT-1 0,25 M | REAL ACADEMIA DE BELLAS ARTES 1 M | LDN 100 nM | TPB 200 nM | ITS-X 1: 200 | |
| S4 | D7-D9 | S3 | LAA 0,25 mM | FGF7 2 ng / mL | SANT-1 0,25 M | REAL ACADEMIA DE BELLAS ARTES 0.1 M | LDN 200 nM | TPB 100 nM | ITS-X 1: 200 | IWP-2 2,5 M |
| Medios S3: MCDB 131 + 1x suplemento de L-glutamina + 2% BSA + 2,5 g / L de NaHCO 3 + mM Glucosa 10 | ||||||||||
Tabla 9
| Escenario | Día | Medios de comunicación | Suplemento | |||||||
| S5 | d10-d12 | S5 | T3 1 M | ALK5i II 10 mM | Sant-1 0,25 M | RA 0,05 M | LDN 100 nM | ITS-X 1: 200 | ZnSO4 10 M | Heparina 10 mg / ml |
| S6 | d19 d13- | S5 | T3 1 M | ALK5i II 10 mM | GSiXX 100 nM | LDN 100 nM | ITS-X 1: 200 | ZnSO4 10 M | Heparina 10 mg / ml | |
| S7 | d33 d20- | S5 | T3 1 M | ALK5i II 10 mM | N-Cys 1 mM | Trolox 10 M | R428 2 M | ITS-X 1: 200 | ZnSO4 10 M | Heparina 10 mg / ml |
| Medios S5: BLAR + 1x L-glutamina suplemento + 2% BSA + 1,5 g / L de NaHCO 3 + glucosa 20 mM | ||||||||||
Tabla 10
Discusión
Consideraciones de tiempo para la generación de mutantes Líneas
A pesar de los recientes enfoques basados en sistemas / Cas CRISPR para la edición del genoma han dado lugar a una selección adecuada, una plataforma más eficaz y universal sería preferible que los análisis a gran escala de la función génica. La plataforma iCRISPR ofrece un método rápido y eficiente para introducir mutaciones en cualquier gen de interés 5, 9. En primer lugar, el método de síntesis gRNA basado en la PCR permite la producción de cientos de gRNAs en formato dispuestos en un día sin las etapas de clonación que consumen mucho tiempo. En segundo lugar, con la expresión inducible por doxiciclina Cas9 en iCas9 hPSCs, la etapa que implica la transfección gRNA requiere sólo una cantidad mínima de trabajo, y por lo tanto, múltiples gRNA experimentos de orientación puede llevarse a cabo al mismo tiempo. En tercer lugar, debido a la alta orientación eficiencias alcanzables con nuestro sistema, el análisis de ~ 24 - 48 colonias por gRNA transfectadas deben ser suffciente para establecer monoallelic múltiple y líneas mutantes bialélicos para un único gen, aunque las eficiencias varían en función del locus diana. Dado que es factible para una persona capacitada para recoger mecánicamente 384 colonias (placas de 4 x 96 pocillos) en una sola sesión, la cual debe tener ~ 4 h bajo un microscopio de disección, una persona capacitada puede esperar para generar líneas mutantes que afectan a 12 genes dentro de 12 meses. La generación de mutantes aerodinámico HPSC en poco tiempo permite el análisis sistemático de una gran variedad de factores de transcripción y / o componentes de la ruta de señalización que interactúan entre sí, la regulación del proceso de desarrollo 9. Además, eficiente focalización gen multiplexado también abre la puerta a la investigación de las interacciones genéticas subyacentes rasgos humanos complejos.
Generación de modificaciones genéticas precisas
La derivación de células iPS específicas para cada paciente de un tipo de células somáticas de fácil acceso s y la diferenciación en tipos celulares relevantes de la enfermedad ofrecen una gran oportunidad para la validación funcional de las mutaciones asociadas a la enfermedad. Sin embargo, debido a la considerable variabilidad en el fondo genética entre los individuos, las comparaciones directas entre iPSCs de pacientes y de donantes sanos pueden no permitir a uno distinguir fenotipos de la enfermedad de los efectos de fondo. Por lo tanto, es necesario generar células iPS de control isogénicas mediante la corrección de la mutación de la enfermedad de nuevo a la secuencia de tipo salvaje o para introducir las mutaciones específicas para cada paciente en un fondo HPSC de tipo salvaje, tal como se propone por otros 25, 26. Además de las mutaciones (null) pérdida de la función, se puede ahora diseccionar más precisamente mecanismos de la enfermedad a través de la introducción de unos pacientes específicos de secuencia de las alteraciones en el locus endógeno en hPSCs, que incluye un paciente hypermorphic, hypomorphic, neomorphic, o dominante negativo mutaciones.
ntent "> modificación genética Precise emplea HDR para la reparación de DSB en presencia de una plantilla de reparación. Ya que es mucho menos eficiente que la reparación del ADN mediada por NHEJ, un plásmido donante que contiene la mutación específica del paciente, un casete de selección de drogas, y brazos de homología se ha utilizado previamente como la plantilla de reparación 2. Después de la selección de medicamentos y la verificación de la mutación específica del paciente, un segundo paso es generalmente necesario para eliminar el casete de selección de fármacos. Mientras que los más utilizados sistemas Cre-loxP y FLP-FRT dejan atrás secuencia residual en el locus endógeno, el uso de piggyBac transposón ha permitido la eliminación perfecta de la casete de selección de drogas 27. Más recientemente, las plantillas de ssDNA cortos también se han demostrado para apoyar HDR eficiente con endonucleasas de ADN de ingeniería 28. en comparación con un plásmido donante , ssDNA se puede sintetizar directamente y por lo tanto evita la etapa de clonación de tiempo. A continuación, tiene seren demostrado que, por co-transfección de gRNA y una plantilla de ssDNA para inducir la reparación de homología dirigida, iCRISPR se puede utilizar para introducir modificaciones de nucleótidos específicas con alta eficiencia. Esto es crítico para la disección no sólo el papel de nucleótidos esenciales dentro de los dominios funcionales de proteínas, sino también para el modelado de las mutaciones de enfermedades humanas y potencialmente corrección de estas mutaciones asociadas a la enfermedad para la intervención terapéutica. Debido al gran número de loci de susceptibilidad que están asociados cada uno con múltiples variantes de la secuencia, el modelado de enfermedades complejas y multigénicas como la diabetes ha sido un reto para los genetistas. Como la plataforma iCRISPR es permisiva para la rápida generación de una serie alélica o para la orientación del gen multiplexable, se puede facilitar la investigación de múltiples loci asociados a la enfermedad, ya sea individualmente o en combinación con fondos isogénicas.Orientación de Eficiencias y fuera de objetivo efectos
Tenemos encontrado una buena correlación entre los resultados del ensayo de RFLP T7E1 y y el número de líneas mutantes identificados por secuenciación. Esto pone de relieve la importancia de realizar estos ensayos en paralelo con el establecimiento de líneas clonales. Si bien las eficiencias de orientación alcanzado puede variar dependiendo de los loci genómico, en la mayoría de los experimentos de gen de la orientación individuales, 20-60% de los clones fueron encontrados con ambos alelos mutados (incluyendo dentro del marco y mutaciones frameshift) 9. En los casos en que se realizó la orientación gen multiplexado, triples clones mutantes bialélicos con 5 a 10% de eficiencia se obtuvieron 5. mediada por ssDNA HDR de varios genes también se realizaron para obtener alteraciones genéticas precisas, con la eficiencia de la obtención de homocigotos ronda en los clones que van de 1 - 10% 5. Trabajar con CRISPR / Cas en hPSCs, ninguna mutación en los sitios potenciales de fuera de objetivo que no comparten la misma secuencia diana gRNA aún no se han detectadolass = "xref"> 5, 9, 12. Secuenciación del genoma completo realizado en un estudio reciente también ha fallado en identificar mutaciones sustanciales fuera de objetivo en líneas clonales HPSC generada usando CRISPR / Cas 29. Sin embargo, para minimizar cualquier efecto potencial de confundir fenotipos introducidas por mutaciones en los sitios de efectos fuera de la meta, se sugiere para generar líneas mutantes independientes usando al menos dos gRNAs independientes dirigidas a diferentes secuencias dentro del mismo gen. fenotipos similares observados en múltiples líneas generadas usando diferentes gRNAs podría descartar principalmente la posibilidad de que el fenotipo viene de un efecto desviado.
Alimentador dependiente frente al cultivo independiente y Orientación
Los métodos tradicionales para el cultivo HPSC implican su mantenimiento y expansión de células alimentadoras en medios que contienen suero o suero de reemplazo que incluye animales prodpro-, tales como albúmina de suero bovino. células alimentadoras, suero, suero de recambio, y la albúmina todos contienen componentes complejos, no definidos y muestran una considerable variabilidad lote. La adaptación a la condición libre de alimentador y definido químicamente reduce significativamente los esfuerzos para el mantenimiento HPSC y, más importante, aumenta la consistencia de la diferenciación de los experimentos. En la actualidad, existen muy pocos estudios que describen los procedimientos de edición del genoma en hPSCs cultivadas en condiciones de cultivo completamente definidas 30. Hemos encontrado mayor eficiencia cuando la orientación CRISPR gRNA transfección y la deposición clonal se realizaron en condiciones libres de alimentador en comparación con las condiciones de cultivo de alimentación dependiente. Creemos que esto es debido al aumento de la supervivencia celular después de la transfección y la siembra de células individuales para la formación de colonias. Por otra parte, han sido previamente demostrado células alimentadoras para secuestrar los reactivos de transfección, lo que reduce la eficiencia de transfección.
CombinatorioEdición genoma con diferenciación dirigida
Protocolos de diferenciación anteriores han dado sólo una pequeña fracción de las células positivas para insulina, la mayoría de los cuales eran polyhormonal y se parecía a las células endocrinas fetales 23. El progreso reciente ha permitido la diferenciación de hPSCs en células más maduras sensibles a glucosa beta-como 16, 17, 19, 20. Podemos obtener de manera rutinaria las células del endodermo definitivo al menos el 75%, el 40% de las células beta-como sensibles a glucosa PDX1 + + Nkx6.1 progenitoras pancreáticas, y alrededor de 20% Nkx6.1 + + utilizando CPEP HUES8 hPSCs 9. Cuando se combina con el sistema de edición genoma iCRISPR, este protocolo de diferenciación más robusto ha facilitado el análisis de factores de transcripción que son cruciales para las etapas progenitoras pancreáticas endocrinas y de la diferenciación de páncreas. Esto haráEs posible, en futuros estudios, para examinar un gran número de genes candidatos enfermedad para la validación funcional y la investigación sobre los mecanismos que subyacen a la diabetes 9.
Las aplicaciones futuras o llegar
Nuestro sistema iCRISPR pueden facilitar la generación de las modificaciones que darán más complejas, tales como la creación de alelos reportero a través de la orientación de genes usando plantillas de ADN de gran donantes etiquetas de codificación de proteínas o reporteros fluorescentes 12 HDR-mediada. Hemos demostrado que, debido a la alta CRISPR orientación eficiencias logradas en el sistema, este proceso se puede realizar en hPSCs, sin la necesidad de una selección adicional de drogas 12. Por otra parte, el gen de multiplexado de direccionamiento puede ser utilizado para investigar las interacciones genéticas de la enfermedad humana compleja subyacente, como se muestra en nuestro estudio reciente, que creemos que es el primer ejemplo de este tipo de trabajo 31. iCRISPR también se puede utilizar para comprender el control de regulación de genes mediante la creación de deleciones tanto en los ARN no codificantes o en regiones reguladoras de genes, tales como promotores y potenciadores. Para generar eficientemente mutantes reguladores utilizando iCRISPR, gRNAs pueden ser diseñados para interrumpir el sitio de unión de una proteína de unión al ADN, incluyendo, pero no restringidos a la maquinaria transcripcional basal o un factor de transcripción específico de tejido. ssDNA HDR plantilla mediada también puede utilizarse para mutar los sitios específicos de unión a proteínas. Finalmente, prevemos que una optimización adicional permitiría el uso de la plataforma en iCRISPR hPSCs para análisis genéticos de rendimiento más altos de fenotipos de pluripotencia o fenotipos de la enfermedad cuando se combina con un protocolo de diferenciación in vitro. Estos estudios iCRISPR mediada podrá permitir que sea más rápida identificación de los genes y los estudios de su relevancia funcional asociados a la enfermedad candidatos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado en parte por el NIH / NIDDK (R01DK096239) y el estado de Nueva York Stem Cell Science (NYSTEM C029156). ZZ fue apoyada por la beca postdoctoral NYSTEM del Centro de Biología de Células Madre del Instituto Sloan Kettering.
Materiales
| Name | Company | Catalog Number | Comments |
| Chemically defined medium (E8) | Thermo Fisher Scientific | A1517001 | Essential 8 basal medium and Essential 8 supplement included |
| Truncated recombinant human form of vitronectin | Thermo Fisher Scientific | A14700 | |
| ROCK inhibitor Y-27632 | Selleck Chemicals | S1049 | |
| Dissociation reagent (TrypLE Select enzyme) | Thermo Fisher Scientific | 12563029 | 1x, animal origin free, recombinant enzyme |
| G418 Sulfate | Thermo Fisher Scientific | 10131035 | Geneticin Selective Antibiotic |
| Puromycin dihydrochloride | Sigma-Aldrich | P8833 | Puromycin Selective Antibiotic |
| DNA Polymerase (Herculase II Fusion) | Agilent Technologies | 600679 | PCR kit |
| MEGAshortscript T7 Transcription kit | Thermo Fisher Scientific | AM1354 | |
| MEGAclear Transcription Clean-Up Kit | Thermo Fisher Scientific | AM1908 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| Opti-MEM medium | Thermo Fisher Scientific | 31985062 | Reduced Serum Medium |
| Lipofectamine RNAiMAX Transfection Reagent | Thermo Fisher Scientific | 13778150 | |
| DNeasy Blood & Tissue Kit | QIAGEN | 69504 | Genomic DNA extraction kit |
| Proteinase K | Roche | 3115879001 | |
| MCDB 131 medium | Thermo Fisher Scientific | 10372-019 | |
| BLAR medium | Thermo Fisher Scientific | Custom-made with a published formulation (Rezania et al., 2014) | |
| L-glutamine supplement (GlutaMAX) | Thermo Fisher Scientific | 35050061 | |
| NaHCO3 | Thermo Fisher Scientific | 144-55-8 | |
| Glucose | Sigma-Aldrich | G8769 | |
| BSA | LAMPIRE Biological Laboratories | 7500855 | Fatty acid free |
| GDF8 | PeproTech | 120-00 | |
| CHIR-99021 | Stemgent | 04-0004 | GSK-3 inhibitor |
| L-Ascorbic acid | Sigma-Aldrich | A4544 | Vitamin C |
| FGF7 | R&D Systems | 251-KG | |
| SANT1 | Tocris Bioscience | 1974 | Hedgehog inhibitor |
| RA | Sigma-Aldrich | R2625 | Retinoic acid |
| LDN | Stemgent | 04-0019 | BMP inhibitor |
| IWP-2 | Tocris Bioscience | 3533 | Wnt antagonist |
| ITS-X | Thermo Fisher Scientific | 51500-056 | |
| TPB | EMD Millipore | 565740-1MG | PKC activator |
| 3,3’,5-Triiodo-L-thyronine (T3) | Sigma-Aldrich | T6397 | |
| ALK5i II | Enzo Life Sciences | ALX-270-445 | ALK5 inhibitor II |
| ZnSO4 | Sigma-Aldrich | Z0251 | |
| Heparin | Sigma-Aldrich | H3149 | |
| GSiXX | EMD Millipore | 565789 | Gamma secretase inhibitor XX, NOTCH signaling inhibitor |
| N-Cys (N-acetyl cysteine) | Sigma-Aldrich | A9165 | |
| Trolox | EMD Millipore | 648471 | Vitamin E analogue |
| R428 | Selleck Chemicals | S2841 | AXL receptor tyrosine kinase inhibitor |
| 24 mm Transwell with insert | Corning Life Sciences | 3414 | |
| Gene Pulser Xcell Electroporation System | Bio-Rad | 1652660 | |
| 0.4 cm Electroporation Cuvettes | Bio-Rad | 1652081 | |
| AAVS1-TALEN-L | Addgene | 59025 | |
| AAVS1-TALEN-R | Addgene | 59026 | |
| AAVS1-Neo-M2rtTA | Addgene | 60843 | |
| AAVS1-Puro-iCas9 | Addgene | 58409 | |
| T7 Endonuclease I | NEB | M0302L | |
| Buffer 2 | NEB | B7002S | NEBuffer 2 |
| Long oligonucleotide | Eton Bioscience or IDT | ||
| SOX17 antibody | R&D Systems | AF1924 | 1:500 |
| FOXA2 antibody | Millipore | 07-633 | 1:100 |
| CXCR4-APC antibody | R&D Systems | FAB170A | 1:25 |
| PDX1 antibody | R&D Systems | AF2419 | 1:500 |
| NKX6.1 antibody | DSHB | F55A12 | 1:500 |
| NKX2.2 antibody | DSHB | 74.5A5 | 1:100 |
| NEUROD1 antibody | Santa Cruz Biotechnology | sc-1084 | 1:100 |
| Insulin antibody | Dako | A0564 | 1:2,000 |
| C-peptide antibody | DSHB | GN-ID4-c | 1:2,000 |
| Glucagon antibody | Sigma-Aldrich | G2654 | 1:1,000 |
Referencias
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6 (6), 507-512 (2005).
- Zwaka, T. P., Thomson, J. A. Homologous recombination in human embryonic stem cells. Nat Biotechnol. 21 (3), 319-321 (2003).
- Eiges, R., et al. Establishment of human embryonic stem cell-transfected clones carrying a marker for undifferentiated cells. Curr Biol. 11 (7), 514-518 (2001).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- González, F., et al. An iCRISPR Platform for Rapid, Multiplexable, and Inducible Genome Editing in Human Pluripotent Stem Cells. Cell Stem Cell. 15 (2), 215-226 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Braam, S. R., et al. Improved genetic manipulation of human embryonic stem cells. Nat Methods. 5 (5), 389-392 (2008).
- Zhu, Z., et al. Genome Editing of Lineage Determinants in Human Pluripotent Stem Cells Reveals Mechanisms of Pancreatic Development and Diabetes. Cell Stem Cell. 18 (6), 755-768 (2016).
- Kotini, A. G., et al. Functional analysis of a chromosomal deletion associated with myelodysplastic syndromes using isogenic human induced pluripotent stem cells. Nat Biotechnol. 33 (6), 646-655 (2015).
- Carlson-Stevermer, J., et al. High-Content Analysis of CRISPR-Cas9 Gene-Edited Human Embryonic Stem Cells. Stem Cell Reports. 6 (1), 109-120 (2016).
- Zhu, Z., Verma, N., Gonzalez, F., Shi, Z. D., Huangfu, D. A CRISPR/Cas-Mediated Selection-free Knockin Strategy in Human Embryonic Stem Cells. Stem Cell Reports. 4 (6), 1103-1111 (2015).
- Mandegar, M. A., et al. CRISPR Interference Efficiently Induces Specific and Reversible Gene Silencing in Human iPSCs. Cell Stem Cell. 18 (4), 541-553 (2016).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Methods. 8 (5), 424-429 (2011).
- Rezania, A., et al. Production of functional glucagon-secreting alpha-cells from human embryonic stem cells. Diabetes. 60 (1), 239-247 (2011).
- Pagliuca, F. W., et al. Generation of functional human pancreatic beta cells in vitro. Cell. 159 (2), 428-439 (2014).
- Rezania, A., et al. Reversal of diabetes with insulin-producing cells derived in vitro from human pluripotent stem cells. Nat Biotechnol. 32 (11), 1121-1133 (2014).
- Chen, S., et al. A small molecule that directs differentiation of human ESCs into the pancreatic lineage. Nat Chem Biol. 5 (4), 258-265 (2009).
- Nostro, M. C., et al. Efficient generation of NKX6-1+ pancreatic progenitors from multiple human pluripotent stem cell lines. Stem Cell Reports. 4 (4), 591-604 (2015).
- Russ, H. A., et al. Controlled induction of human pancreatic progenitors produces functional beta-like cells in vitro. EMBO J. 34 (13), 1759-1772 (2015).
- Zhu, Z., Gonzalez, F., Huangfu, D. The iCRISPR platform for rapid genome editing in human pluripotent stem cells. Methods Enzymol. 546, 215-250 (2014).
- Soh, C. L., Huangfu, D. CRISPR/Cas9-Mediated Mutagenesis of Human Pluripotent Stem Cells in Defined Xeno-Free E8 Medium. Methods in Molecular Biology. 1498, (2017).
- D'Amour, K. A., et al. Production of pancreatic hormone-expressing endocrine cells from human embryonic stem cells. Nat Biotechnol. 24 (11), 1392-1401 (2006).
- Kelly, O. G., et al. Cell-surface markers for the isolation of pancreatic cell types derived from human embryonic stem cells. Nat Biotechnol. 29 (8), 750-756 (2011).
- Musunuru, K. Genome editing of human pluripotent stem cells to generate human cellular disease models. Dis Model Mech. 6 (4), 896-904 (2013).
- Hockemeyer, D., Jaenisch, R. Induced Pluripotent Stem Cells Meet Genome Editing. Cell Stem Cell. 18 (5), 573-586 (2016).
- Yusa, K., et al. Targeted gene correction of alpha1-antitrypsin deficiency in induced pluripotent stem cells. Nature. 478 (7369), 391-394 (2011).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Veres, A., et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 15 (1), 27-30 (2014).
- Huang, X., et al. Production of Gene-Corrected Adult Beta Globin Protein in Human Erythrocytes Differentiated from Patient iPSCs After Genome Editing of the Sickle Point Mutation. Stem Cells. 33 (5), 1470-1479 (2015).
- Shi, Z. D., et al. Genome Editing in hPSCs Reveals GATA6 Haploinsufficiency and a Genetic Interaction with GATA4 in Human Pancreatic Development. Cell Stem Cell. , (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados