Method Article
Genome Editing und gerichteten Differenzierung von hPSCs zur Abfrage Lineage Bestimmungsfaktoren im Bereich Human Pancreatic Entwicklung
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Protokolle HPSC Mutantenlinien mit der iCRISPR Plattform und zu differenzieren hPSCs in Glukose-responsive β-ähnlichen Zellen beschrieben zu erzeugen. Genom Bearbeitungstechnologie mit HPSC gerichtete Differenzierung Die Kombination bietet eine leistungsfähige Plattform für die systematische Analyse der Rolle von Lineage Determinanten in der menschlichen Entwicklung und Progression der Erkrankung.
Zusammenfassung
Abfrage Genfunktion in sich selbst erneuernden oder humanen pluripotenten Stammzellen (hPSCs) Differenzierung bietet eine wertvolle Plattform in Richtung der menschlichen Entwicklung zu verstehen und Mechanismen Krankheit in einer Schale zu sezieren. Um dieses Potenzial Anwendung nutzen erfordert effiziente Genom-Editing - Tools HPSC Mutanten in krankheitsassoziierten Genen, sowie in - vitro - HPSC Differenzierung Protokolle krankheitsrelevanten Zelltypen produzieren , die eng rekapitulieren ihre in - vivo - Pendants zu erzeugen. Eine effiziente Genom-Editing - Plattform für hPSCs namens iCRISPR wurde durch die TALEN-vermittelte Targeting einer Cas9 Expressionskassette im AAVS1 Locus entwickelt. Hierbei sind die Protokolle für die Erzeugung von induzierbaren Cas9 HPSC Linien Zellen kultiviert in einem chemisch definierten Medium und einem Zubringer freien Zustand verwenden beschrieben. Detaillierte Verfahren für die Verwendung des iCRISPR System für Gen-Knockout oder präzise genetische Veränderungen in hPSCs, entweder durch nauf homologe Ende (NHEJ) oder über präzise Nukleotidveränderungen Verbinden einer Homologie gerichteten Reparatur (HDR) Vorlage jeweils enthalten sind. Diese technischen Verfahren sind Beschreibungen der Konstruktion, Produktion, und die Transfektion von CRISPR Führung RNAs (gRNAs); die Messung der Geschwindigkeit CRISPR Mutation durch T7E1 oder RFLP-Tests; und die Etablierung und Validierung von klonalen Mutantenlinien. Schließlich haben wir Chronik Verfahren zur HPSC Differenzierung in Glukose-responsive Pankreas - β-ähnliche Zellen durch in - vivo - Pankreas - Embryonalentwicklung nachahmt. iCRISPR Technologie mit gerichtetem HPSC Differenzierung Kombination ermöglicht die systematische Untersuchung von Genfunktionen unser Verständnis von Pankreas-Entwicklung und Diabetes Krankheitsmechanismen zu fördern.
Einleitung
Humanen pluripotenten Stammzellen (hPSCs) haben die Fähigkeit, sowohl selbst erneuern und verursachen alle Derivate der drei embryonalen Keimlinien. Sie bieten eine wertvolle Ressource für die Zell-Ersatz-Therapie und Krankheitsmodelle, indem sie als einzigartige Plattform dient, zelluläre Prozesse in einem menschlichen Entwicklungskontext zu rekapitulieren. Sie sind auch eine Quelle von Versuchszellen für skalierbare, Hochdurchsatz-Analyse. Jedoch wurden Fortschritte aufgrund von zwei wesentlichen Herausforderungen beschränkt: den Mangel an effizienten genetischen Modifikation Werkzeuge und die Schwierigkeit, in die komplexen embryonalen Entwicklungsschritte in einer Kulturschale zu rekapitulieren.
Genetische Veränderung ist ein unverzichtbares Werkzeug Genfunktion in der normalen Entwicklung und Krankheit zu untersuchen. Während jedoch die klassische Gen Ansätze durch homologe Rekombination gezielt bewährt haben ein mächtiges Werkzeug zu sezieren Genfunktion in embryonalen Stammzellen der Maus (mESCs) 1, dieser Ansatz zu seinwar äußerst ineffizient , wenn 3 bis hPSCs 2, angewendet. Die jüngste flotten Beitritt von programmierbaren, ortsspezifische Nukleasen von der Natur zu den Laboreinsatz, einschließlich Zink-Finger-Nukleasen (ZFNs), Transkriptionsaktivator- artigen Effektor Nukleasen (Talens) und den Cluster regelmäßig kurze Palindrom Wiederholungen (CRISPR) voneinander beabstandete / CRISPR-assoziierten (Cas) -Systeme 4, bedeutet , dass Genomtechnik eine viel einfachere Aufgabe , in einem breiten Spektrum von Organismen und Zelllinien geworden ist, einschließlich in hPSCs. Diese Gen-Editing-Tools nutzen die Tatsache, dass chimären Nukleasen wie die Cas9 Endonuklease eine ganze Reihe von genetischen Veränderungen durch Induktion Doppelstrangbrüche (DSBs) an genauen Stellen erlauben kann, die Auslösung der endogenen DNA-Reparatur-Maschinen zu aktivieren entweder nicht-homologe Ende Beitritt (NHEJ) oder Homologie gerichtete Reparatur (HDR). Beide Mechanismen können durch Induzieren eith zur genetischen Manipulation ausnutzener zufällige Insertion und Deletionsmutationen (indels; über NHEJ), Frameshift - Mutationen zu erzeugen , die Gen - Allele zunichte machen, oder präzise Nukleotidsubstitutionen (via HDR), Patienten Mutationen für die menschliche Krankheitsmodelle zu rekapitulieren oder eine krankheitsverursachende Mutation für die Gentherapie zu korrigieren .
CRISPR / Cas-vermittelte Genomtechnik erfordert zwei Komponenten: die Konstante RNA-gesteuerte Cas9 Endonuklease, die für DNA-Spaltung und einer variablen CRISPR RNA (crRNA) und trans-Aktivierung (tracrRNA) Duplex-DNA, die Zielerkennung angibt. Die crRNA / tracrRNA Duplex kann mit einem einzigen chimären Führung RNA (gRNA) ersetzt werden, die gefunden wurden , effizienter 5, 6, 7 zu arbeiten. Während die CRISPR / Cas9 System hat zu den meisten experimentellen Organismen und Zelllinien angepasst worden ist, ändert sich die Lieferung und die Expression von Cas9 und gRNA signifikant und werden muss, weiter zu Achi optimiertVorabend effiziente Genom Bearbeitung in vielen Systemen, einschließlich hPSCs 8. Eine effiziente Genom-Editing - Plattform, iCRISPR wurde 5 in hPSCs etabliert. In diesem System wird ein TALEN-vermittelte Ansatz verwendet wurde , in trans, ein Allel mit einem Reverse - Tetracyclin-kontrollierten Transaktivator (M2rtTA) und die andere mit einem Tetracyclin - Response - Element beide Allele des "Transgen sicheren Hafen locus" AAVS1 Ziel (TRE ) in den hPSCs die Expression von Cas9 (iCas9) fahren. In etablierten Klonlinien (iCas9 hPSCs) wird Cas9 hoch mit Doxycyclin Behandlung ausgedrückt. Inzwischen aufgrund seiner geringen Größe (100 nt), können einzelne oder mehrere gRNAs leicht in iCas9 hPSCs mit hohem Wirkungsgrad geliefert werden und Cas9 für die ortsspezifische Spaltung anweisen kann, eine effiziente NHEJ-vermittelten Gen-Unterbrechung sowie HDR-vermittelte genaue Nucleotid Modifikationen in Gegenwart von kurzen einsträngigen DNA (ssDNA) Donator-Vorlagen. Das iCRISPR System kannverwendet , um erfolgreich erzeugen eine Gruppe von krankheits nachahmt HPSC Linien mit biallelic (homozygot oder heterozygot) oder heterozygot loss-of-function Mutationen in wichtigen Entwicklungsgene 5, 9. Während eine Anzahl von Gruppen effiziente Gen Bearbeitung unter Verwendung CRISPR / Cas in hPSCs berichtet haben, bleibt der Erfolg auf eine kleine Anzahl von technologisch Adept Laboratorien beschränkt. Die iCRISPR Plattform bietet eine effiziente und dennoch einfache Lösung für Routine - Gen Bearbeitung von Forschern unterschiedlicher Schwierigkeitsstufen, und es wurde bereits in einer Reihe von veröffentlichten Studien von unserer Gruppe und andere 9, 10, 11, 12 verwendet. Dieser Ansatz wurde auch erweitert weiter induzierbaren silencing basierend auf der Expression von dCas9-KRAB 13.
Zusammen mit dem Fortschritt in der Genombearbeitung Technologie, haben erhebliche Verbesserungen auch in HPSC Wartung und gerichtet Differenzierung erreicht. Die Kulturbedingungen für hPSCs haben aus bestrahlten embryonalen Maus - Fibroblasten (Imef) Feeder abhängig Feeder freien Bedingungen auf definierte extrazellulären Matrixkomponenten entwickelt und von komplexen Medien Formulierungen chemisch Medium Bedingungen 14 definiert. Derartige Verbesserungen haben die Variabilität in hPSCs aufgrund von Charge zu Charge unterschiedliche Imef Vorbereitung und knockout Serumersatzkomponenten reduziert und somit eine reproduzierbare Umgebung für HPSC Differenzierung bereitzustellen. Inzwischen verbesserte Kenntnis der Signalwege menschlichen embryonalen Entwicklung , die Errichtung sowie Entdeckungen von Hochdurchsatz - Wirkstoffscreenings, haben dazu geführt , zu einer verbesserten Differenzierung Protokolle 15, 16, 17, 18. Diese Protokolle mehr imitieren eng in vivo Entwicklungsschritte und Zelltypen zu erzeugen , die eng ihre in - vivo - Pendants rekapitulieren. Für HPSC Differenzierung in den Pankreas Abstammung, anfängliche Protokolle nachgeahmt frühen Pankreasentwicklung relativ gut, aber schließlich polyhormonal β-Zellen erzeugt, die von unreifen fetalen Phänotypen waren und reagiert schlecht auf die Glukose-Stimulation. Jüngsten Fortschritte 16, 17, 19, 20 sind für die Erzeugung von Glukose-responsive pankreatischen β-ähnlichen Zellen erlaubt, die es uns ermöglichen, spätere Ereignisse zu untersuchen, wie die Bildung und die weitere Reifung von monohormonal β - Zellen.
Hier stellen wir detailliert die Anwendung von Genom-modifizierten Zeilen für die Untersuchung der Pankreasentwicklung durch die iCRISPR System kombiniert mit der HPSC basierten in vitro Differenzierung in Richtung Plattform Glukose-responsive pancreatic β-ähnliche Zellen. Diese Kopplung von leistungsfähigen Genom - Editing - Tools mit einer verbesserten HPSC Differenzierung Protokoll bietet nicht nur die Geschwindigkeit und das Ausmaß notwendig , um die wachsende Nachfrage nach Validierung Krankheit Kausalität gerecht zu werden, sondern ermöglicht auch anspruchsvolle genetische Manipulationen für weitere mechanistische Untersuchungen zur Transkriptionskontrolle zugrunde liegenden normalen Entwicklung und Krankheit 9 .
Protokoll
Dieses Protokoll basiert auf unserer Arbeit mit HPSC Linien H1, HUES8 und MEL-1 in der chemisch definierten und Feederfreien Zustand (Bitte beachten Sie die Material- und Ausstattungstabelle). Für andere HPSC Linien oder hPSCs in unterschiedlichen Kulturbedingungen gehalten wird, wird eine weitere Optimierung empfohlen.
1. HPSC Kultur in dem chemisch definierten und Feeder freien Zustand
- Passen Sie die HPSC Kultur auf Imef Zubringer zum Zubringer freien Zustand. In Fällen, in denen gefrorenen Zellen nicht gut überleben, wenn sie direkt in den Einzug freien Zustand wiederhergestellt, erholen Zellen in Imef Zustand zuerst und dann an die Speisefreien Zustand anzupassen.
HINWEIS: In der Regel dauert es 2 Durchgänge für hPSCs auf Imef Speiser kultiviert zur zufuhrfreien Zustand angepasst werden. - Ändern Sie den Medium jeden Tag und Passage die hPSCs, wenn die Zellen ~ 80% Konfluenz erreicht haben. Im allgemeinen Passage hPSCs bei ~ 1: 6 - 01.15 Verhältnisse alle 4 - 6 Tage. In 10 uM ROCK-Inhibitor Y-27632 when Auftauen oder die Zellen Passagierung.
- Stand der hPSCs, pre-coat - Kulturschalen mit 5 ug / ml (1 ml / 10 cm 2) trunkierten rekombinanten menschlichen Form von Vitronectin (VTN) für mindestens 1 h bei Raumtemperatur (RT) zum Säen. Auch bereiten vollständig chemisch definierte Medium durch Ergänzung in das Basismedium hinzugefügt wird.
- Entfernen Sie das Kulturmedium, waschen Sie die Zellen einmal mit PBS ohne Ca 2+ und Mg 2+, und behandeln die Zellen mit 0,5 mM EDTA für ~ 2-5 min bei RT.
- Saugen Sie das EDTA, bevor die Kolonien abgelöst haben. Mit sanften Pipettieren, zerstreuen die HPSC Kolonien in kleine Stücke und Resuspendieren der Zellen in komplettem Medium.
- Sammeln Sie die distanzierte hPSCs und drehen Sie die Zellen bei 200 xg für 5 min nach unten. Resuspendieren der pelletierten hPSCs im vollständigen Medium und die Zellen auf VTN beschichteten Platten Samen.
2. Erzeugung von iCas9 HPSC Linien
- Bestellen und verstärken die folgenden Plasmide: AAVS1-TALEN-L, AAVS1-TALEN-R, AAVS1-Neo-M2rtTA und AAVS1-Puro-iCas9.
HINWEIS: unerwartete Rekombinationsereignisse zu vermeiden, verwenden Rekombination-defizienten Stbl3 kompetente Zellen für die Transformation und Amplifikation von Plasmiden bei 30 ° C. - Typischerweise bereiten die hPSCs in einem 10-cm - Schale (~ 1 x 10 7 Zellen , wenn ~ 80% konfluent) für ein Targeting - Experiment.
Hinweis: Da in der Regel die Elektroporation verursacht signifikante Zelltod und TALEN-vermittelten Gen in dem Locus AAVS1 Targeting antibiotische Selektion erfordert, muss eine relativ große Anzahl von Zellen ausgesät werden korrekt angezielten Zellen zu identifizieren. Optimierung für die Arzneimittelkonzentrationen für jede Zelllinie und jeder Kulturbedingung empfohlen. - Am Tag -1 (dem Tag vor der Elektroporation), 10 & mgr; M ROCK - Inhibitor während der Medienwechsel hinzufügen.
- Am Tag 0 (Tag der Elektroporation), bereiten VTN beschichteten Platten im Voraus.
- Distanzieren die hPSCs in einzelne Zellen using 1x Dissoziation Reagenz (bitte das Material und Ausrüstung siehe Tabelle). Kurz gesagt, entfernen Sie das Kulturmedium, um die Zellen einmal mit PBS ohne Ca 2+ und Mg 2+ waschen und die Zellen mit Reagenz 1x Dissoziation bei 37 ° C für ca. 3 min zu behandeln. Saugen Sie das Dissoziation Reagenz, bevor die Zellen abgelöst haben. Mit sanften Pipettieren, zerstreuen die hPSCs in eine Einzelzellsuspension in 10,5 ml Vollmedium.
- Nehmen Sie 0,5 ml Zellsuspension die Zellzahl unter Verwendung eines automatisierten Zellzähler zu zählen. Pellet die hPSCs bei 200 xg für 5 Minuten und Resuspendieren der Zellen in kaltem (4ºC) PBS bei 12,5 x 10 6 Zellen / mL.
- Fügen Sie die Plasmide (siehe Tabelle 1) in 800-ul HPSC Suspension (12,5 x 10 6 Zellen / ml) und gut mischen. Das Gemisch wird in einer 0,4-cm Elektroporationsküvette und auf Eis halten für ca. 5 min.
- Elektroporation der Zellen eine Elektroporation System bei 250 V und 500 uF unter Verwendung; die Zeitkonstante observed nach der Elektroporation ist in der Regel von 9 bis 13 ms.
- Nach der Elektroporation übertragen die Zellen auf eine 15-ml konischen Röhrchen mit 5 ml vorgewärmtem Komplettmedium. Entscheidend für erfolgreiches Targeting: Verwenden Sie gesunden wuchernden hPSCs und die Zellen sehr schonend behandeln , wenn die Übertragung, Resuspendieren und Ausplattieren der Zellen nach der Elektroporation.
- Pelletieren Sie die Zellen bei 200 · g für 5 min. Resuspendieren der Zellen in 10 ml komplettem Medium mit 10 uM ROCK - Inhibitor und der Platte 1, 2,5 und 5 x 10 6 Zellen auf jeder der drei VTN-beschichteten 10-cm - Schalen; Dies stellt sicher, dass für die Kolonie picking mindestens eine der Platten eine ausreichende klonale Kolonien Dichte Einzelzell haben.
- Am 1. Tag (der Tag nach der Elektroporation), das Medium ändern.
- An den Tagen 2 bis 5, Neomycinselektion beginnen , wenn die Zellen sind ~ 60% konfluent. Ändern Sie das Medium täglich mit 500 ug / ml G418-Sulfat; signifikante Zelltod aufgrund Selection typischerweise 2 Tage nach der G418-Selektion beobachtet.
- Am 6. Tag, ändern Sie das Medium ohne Antibiotika - Selektion.
- An den Tagen 7-9, Puromycin - Selektion starten. Ändern Sie das Medium täglich mit 1 ug / ml Puromycin Dihydrochlorid; signifikanten Zelltod sollte am nächsten Tag beobachtet werden.
- Am 10. Tag, starten Sie das Medium täglich ohne Antibiotika - Selektion zu ändern , bis die HPSC Einzelzellkolonien erreichen 1 bis 2 mm im Durchmesser.
HINWEIS: In der Regel 50 Kolonien in einem 10-cm - Schale mit 2,5 × 10 6 hPSCs beobachtet plattiert werden am Tag 0. - Pick-12-24 Kolonien unter einem Stereomikroskop. Mechanisch die Kolonien in kleine Stücke HPSC disaggregieren (~ 10 Stück pro Kolonie) eine 23-G-Nadel (eine 200-ul Pipettenspitze ist auch in Ordnung) und übertragen Sie die Zellen direkt in VTN-beschichteten 24-Well-Platten.
- Ändern Sie das Medium täglich, bis die Zellen konfluent werden. Passage der Zellen in jeder Vertiefung der 24-Well-Platten inzwei Vertiefungen von Platten mit 6 Vertiefungen.
- Wenn die Zellen konfluent werden in den 6-Well-Platten, verwenden Sie eine gut für eine gefrorene Lager und die anderen auch für die genomische DNA-Extraktion zur weiteren Charakterisierung.
- Charakterisieren und validieren die etablierten iCas9 Linien, die durch PCR-Genotypisierung, Southern-Blot, RT-qPCR-Analyse, Karyotypisierung und Pluripotenz-Test. Wenden Sie sich an Zhu et al. 21 für detaillierte experimentelle Verfahren.
3. Erzeugung von HPSC Mutant Zeilen über den iCRISPR-System
- Die Erzeugung von HPSC Knockout Linien
- gRNA Design und Produktion
- Wählen Sie Zielregionen in dem Gen von Interesse, die Möglichkeit zu maximieren zu stören Wildtyp-Protein-Funktion. Für gut kommentierte Gene, ein Zielgebiet vor einer wesentlichen Funktions Domäne wählen. Alternativ Design gRNAs einen Bereich stromabwärts des Startcodons abzuzielen. Wählen mindestens 2 verschiedeneRegionen für ein Gen von Interesse.
- Design gRNAs die Online-CRISPR-Design-Tool mit (http://crispr.mit.edu). Für jede Zielregion, Design 3 gRNAs mit niedrigem Potential off-Ziele und die mit der höchsten Ziel Effizienz verwenden 22 klonalen Mutantenlinie zu erzeugen.
ANMERKUNG: Um eine hohe Genom Bearbeitungseffizienz zu erreichen, wird empfohlen, gRNA als RNA-Oligos statt als Plasmid-DNA aufgrund der höheren Transfektionseffizienz von kleinen RNAs im Vergleich zu Plasmiden, in vergangenen Erfahrung zu liefern. - Bestellen 120 - Nukleotid (nt) DNA - Oligos die T7 Promotorsequenz, die variable 20-nt crRNA Erkennungssequenz (N) 20 (nicht das PAM - Sequenz), und die Konstante chimäre Leitsequenz enthält. Verdünnen Sie die Oligos bis 100 & mgr; M Stammlösung in ddH 2 O und bereiten 250 nM als Lösung arbeiten.
HINWEIS: TAATACGACTCACTATAGGG (N) 20 GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTT - PCR-Amplifikation die Oligos die T7F und TracrR Primer verwendet (siehe Tabelle 2) , um die doppelsträngige DNA (dsDNA) Vorlage für gRNA in - vitro - Transkription (IVT) zu erzeugen. Verwenden Sie 50 & mgr; l PCR - Reaktionsgemisch (siehe Tabelle 3) und PCR - Zyklusbedingungen (siehe Tabelle 4).
- Verwenden Sie ein High-Yield - T7 Transcription Kit für die in vitro gRNA Transkription mit der PCR-amplifizierte Matrize in 20 & mgr; l, in vitro gRNA Transkription Mix (siehe Tabelle 5) gemäß den Anweisungen des Herstellers. Reinige die gRNA Produkte die Transkription clean-up-Kit gemäß den Anweisungen des Herstellers verwendet wird.
- Eluieren gRNAs nach dem Hochdurchsatz-Reinigungsprotokoll gemäß den Anweisungen des Herstellers (typischerweise ~ 50 bis 100 & mgr; g) in 100 & mgr; l Elutionspuffer. Stellen Sie die Konzentration auf 320 ng / & mgr; l (10 & mgr; M), wenn möglich, und bei -80 &# 176; C bis zur Verwendung.
- PCR und Sanger - Sequenzierung Primer - Design
- Entwicklung und Validierung PCR-Primer, die Zielregion zu verstärken, mit Produktgrößen typischerweise im Bereich von ~ 500 - 1000 bp.
- Design Sanger-Sequenzierungsprimer intern an die PCR-Produkte Bindung ohne Reinigung direkte Sequenzierung der PCR-Produkte zu ermöglichen.
- gRNA Transfektion in iCas9 hPSCs
- Am Tag -1, iCas9 Zellen mit 2 & mgr; g / ml Doxycyclin 24 h vor der Transfektion gRNA behandeln.
- Am Tag 0 von gRNA Transfektion vorbereiten VTN beschichteten Platten im Voraus.
- Distanzieren iCas9 Zellen in einzelne Zellen 1x Dissoziation Reagenz, wie 2,5 in Schritt beschrieben.
- Pellet die hPSCs bei 200 xg für 5 Minuten und Resuspendieren der Zellen bei ~ 0,5 × 10 6 Zellen / ml in Komplettmedium , ergänzt mit 2 & mgr; g / ml Doxycyclin und 10 uM ROCK - Inhibitor.
- Platte 0,5 ml der resuspendierten Zellen in einzelnen Vertiefungen von Platten mit 24 Vertiefungen. Bereiten Sie weitere Quellen als nicht-transfizierten Kontrollen zu dienen.
- Für jede gRNA, die folgenden Transfektion Mischungen: Mischung A, 50 & mgr; l reduzierten Serum-Medium + 1 ul gRNA (10 & mgr; M); Mischung B wurden 50 ul reduzierten Serummedium + 3 & mgr; l Transfektionsreagenz.
- Kombinieren Mix A und B zu 100 & mgr; l Mischung herzustellen. Inkubieren für 5 min bei RT. Je 50 ul der Mischung zu den Zellen in den doppelten Wells der 24-Well-Platten und gut mischen.
- Am Tag 1 eine zweite Transfektion durchführen , wenn erforderlich , um die Targeting - Effizienz zu steigern. Andernfalls ändern das Medium ohne Doxycyclin.
- An den Tagen 2 - 3, wobei das Medium täglich ändern.
- Am Tag 4, extrahieren genomische DNA aus einer Vertiefung jeder transfizierten und Kontrollzelle nicht transfizierten eine DNA - Extraktion - Kit. Stellen Sie die Konzentration auf 50 ng / μL.
- PCR-Amplifikation der Zielregionen , die gRNA Targeting - Sequenzen flankieren , und schätzen die Bearbeitungseffizienz unter Verwendung von T7 - Endonuklease I (T7EI) Verdauung oder die RFLP (RFLP) Assay, wie zuvor 5 beschrieben.
- T7EI Assay
- PCR-Amplifikation der Zielregion der Primer entwickelt und validiert in Schritt 3.1 verwendet wird.
- Herstellung der Gemische (siehe Tabelle 6) und DNA - Denaturierung und Hybridisierung der PCR - Produkte durchzuführen unter Verwendung umrissenen Bedingungen in Tabelle 7.
HINWEIS: Aufgrund unserer Erfahrung, es ist in der Regel nicht notwendig, PCR-Produkte für den T7EI Assay zu reinigen, wenn unsere PCR-Bedingungen verwendet wird. Jedoch könnte die Reinigung in anderen Bedingungen vorteilhaft sein. - Durchführen einer T7EI Verdau bei 37 ° C für 30 min unter Verwendung von 10 & mgr; l des denaturierten und hybridisierten PCR-Produkt und 0,2 & mgr; l (2 U) von T7E1 (10 U / ul).
- Resolve die T7E1-verdaute PCR-Proben durch Gelelektrophorese. Verwenden Sie ImageJ die relativen Bandintensitäten von geschnittenen und ungeschnittenen DNA zu bestimmen. Berechne die indel Frequenz unter Verwendung der Formel: (1 - (√ (1- (b + c)) / (a + b + c))) x 100, wobei A die Intensität des unverdauten PCR Produkts und b und c die Intensitäten der T7E1 gespaltenen Produkte.
- RFLP - Assay
HINWEIS: In Fällen, in denen eine Restriktionsstelle in der Nähe ist (<5 bp) zu einem Cas9 Spaltungsstelle (3 bp 5 'der PAM-Sequenz) kann ein RFLP-Assay die indel Frequenz zu quantifizieren durchgeführt werden.- Verwenden Sie die gleichen PCR-Produkte, wie in Schritt 3.1.4.1 beschrieben.
- Digest das PCR-Produkt mit einem Restriktionsenzym, das eine Restriktionsstelle in der Nähe des Cas9 Spaltungsstelle enthält.
- Die verdauten PCR-Proben durch Gelelektrophorese. Verwenden Sie ImageJ die relativen Bandintensitäten von geschnittenen und ungeschnittenen DNA zu bestimmen. Berechnen Sie die indelFrequenz unter Verwendung der Formel: a / (a + b + c) x 100, wobei A die Intensität des unverdauten PCR - Produkt ist und b und c sind die Intensitäten der verdauten Produkte.
- Etablierung von klonalen Mutantenlinien
HINWEIS: Genome Bearbeitung in hPSCs die iCRISPR System mit gRNA Transfektion unter Verwendung ist sehr leistungsfähig, und keine Antibiotika-Selektion benötigt. Um klonale Linien herzustellen, ist es notwendig, Zellen mit einer relativ niedrigen Dichte auf Saatgut, um die Bildung von einzelnen Zellen abgeleiteten Kolonien zu gewährleisten.- Identifizieren gRNAs mit der höchsten Bearbeitungseffizienz und mit guter das Überleben der Zellen (die T7EI oder RFLP-Assay verwendet wird). Verwenden Sie die entsprechenden Duplikat gut für klonalen Mutantenlinie Establishment.
- Dissoziieren die hPSCs in eine Einzelzellsuspension 1x Dissoziation Reagens, wie 2,5 in Schritt beschrieben. Ausstrich 500, 1.000, und 2.000 Zellen auf jeder der drei VTN-beschichteten 10-cm-Schalen.
- Ändern Sie das Medium täglich until Einzelzellkolonien erreichen ~ 2 mm im Durchmesser.
- Pick 24 bis 48 Kolonien pro gRNA, abhängig von der Schätzung der Zieleffizienz durch die T7EI und / oder RFLP-Assay. Mechanisch jede Kolonie in kleine Stücke zerlegen (~ 10 Stück pro Kolonie) unter Verwendung einer 23-G-Nadel (eine 200-ul Pipettenspitze ist auch in Ordnung) und Ausstrich die Zellen in doppelter VTN-beschichteten 96-Well-Platten. Verwenden Sie eine Platte für die genomische DNA-Extraktion und Sanger-Sequenzierung und die andere Platte für die weitere Expansion.
- Wenn die Zellen in den 96-Well-Platten konfluenter geworden sind, extrahieren die genomische DNA (ohne Phenol / Chloroform-Extraktion) ein einfaches Protokoll verwendet, wie unten beschrieben.
- Entfernen Sie das Medium und die Zellen werden einmal mit PBS ohne Ca 2+ und Mg 2+. Zugabe von 50 & mgr; l Lyse - Puffer (5 & mgr; l Proteinase K (10 mg / mL), 5 & mgr; l PCR - Puffer 10x und 40 & mgr; l ddH 2 O) in jede Vertiefung einer 96-Well - Platte. Verschließen Sie die Platte mit einer Klebefolie und Incubate über Nacht bei 55 ° C.
- Am nächsten Tag übertragen die Zelllysate in einer 96-Well PCR-Platte und Inkubation für 10 min bei 99 ° C in einem Thermocycler die Proteinase K zu inaktivieren
- PCR-Amplifizierung der Zielregion der gleichen Primer wie für die T7EI oder RFLP-Assay unter Verwendung von 1 & mgr; l Zelllysat als Vorlage verwendet wird.
- Verwenden 1 & mgr; l des PCR-Produkts für die Sanger-Sequenzierung mit einem Primer intern mit dem PCR-Produkts binden.
- Amplify die Klone mit Frameshift indel Mutationen für gefrorene Bestände. Außerdem verstärken ein paar Wildtyp-Klone, die aus dem gleichen Targeting Experiment als isogene Steuerleitungen für weitere Experimente dienen.
- gRNA Design und Produktion
- Die Erzeugung von Mutantenlinien mit präzisen Nukleotidveränderungen
HINWEIS: Im Vergleich mit Knockout-Mutanten, die durch nicht-homologe Endverbindung (NHEJ), exakte Nucleotid-Veränderung kann durch Homologie-directed Reparatur (HDR) in Gegenwart von DNA erreicht werden repair Vorlagen. Eine solche präzise Nukleotidveränderungen ermöglichen die Erzeugung von patientenspezifischen Mutationen in Wildtyp hPSCs und zur Korrektur von Mutationen in Patienten stamm iPSCs.- Gestaltung von ssDNA als HDR - Vorlage
- Design und produzieren 2 - 3 gRNAs in unmittelbarer Nähe zu einer patientenspezifische Mutation, wie in Schritt 3.1.1 beschrieben.
- Entwerfen Sie ein Einzelstrang-DNA (ssDNA), um die patientenspezifische Mutation enthält, flankiert von ~ 40-80 nt der Homologie auf jeder Seite als HDR-Vorlage.
- Um zusätzliche Schneiden nach der richtigen Reparatur zu verringern, führen eine stille Mutation in die ssDNA Template im Bereich innerhalb der gRNA Erkennungssequenz und in der Nähe des PAM-Sequenz oder in der PAM Sequenz selbst, falls möglich.
- Wenn möglich, gestalten die stille Mutation eine neuartige Restriktionsverdau Ort als auch einzuführen, so dass es verwendet werden kann, um die Reparatur Effizienz mit Hilfe der RFLP-Test zu schätzen.
- GRNA / ssDNA Co-Transfektion und die Einrichtung von Klonlinien
- Führen Sie die Cotransfektion von gRNA / ssDNA in iCas9 Zellen, wie in Schritt 3.1.3 mit Transfektion Mischungen A und B. Für jede gRNA und nicht-transfizierten Kontroll beschrieben, zu transfizieren, die Zellen in doppelten Vertiefungen von Platten mit 24 Vertiefungen. Mix A: 50 & mgr; l reduziertem Serum-Medium + 1 & mgr; l gRNA (10 & mgr; M) + 2 & mgr; l ssDNA (10 uM). Mix B: 50 & mgr; l reduziert Serum-Medium + 3 ul Transfektionsreagenz.
- Nach der Transfektion extrahieren genomische DNA aus einer Vertiefung jeder transfizierten und Kontrollzelle nicht transfizierten und schätzen die Reparatureffizienz der T7EI und / oder RFLP Assay.
- Identifizieren Sie die gRNA / ssDNA Mischung mit der höchsten Reparatureffizienz und gute Überleben der Zelle. Verwenden Sie die entsprechenden Duplikat gut für klonalen Mutantenlinie Establishment.
- Pick 48-96 Kolonien in Abhängigkeit von der Schätzung der Zieleffizienz durch die T7EI und / oder RFLP assay. Im allgemeinen ist die Effizienz der HDR-vermittelter Mutation niedriger als Knockout-Mutation und somit mehr Kolonien müssen kommissioniert werden.
- Sequenz, erweitern und Klonlinien validieren, wie in Schritt 3.1.6 beschrieben.
- Gestaltung von ssDNA als HDR - Vorlage
4. In - vitro - HPSC Differenzierung in Glukose-responsive Bauchspeicheldrüsen β - Zellen
HINWEIS: In vitro Differenzierung von HPSC Mutanten in krankheitsrelevanten Zelltypen für Krankheitsmodelle in einer Schale , eine Plattform zur Verfügung stellt. Das folgende Protokoll konzentriert sich auf die in vitro Differenzierung von hPSCs in Glukose-responsive pankreatischen β - Zellen bei Bauchspeicheldrüsenentwicklungs und diabetischer Studien 9, 16, 17.
- HPSC Differenzierung in endgültige Endoderm
- Pflegen HPSC Mutanten und Wildtyp-Steuerleitungen in dem chemisch definierten und Feederfreien Zustand, wie beschreibend in Schritt 1.
- Um die hPSCs zur Differenzierung vorzubereiten, distanzieren die hPSCs 1x Dissoziation Reagenz und verteilen sie in Einzelzellsuspension in komplettem Medium.
- Pellet die Zellen bei 200 xg für 5 min und erneut die Zellen in komplettem Medium mit 10 & mgr; M ROCK-Inhibitor. Zählen der Zellanzahl und Samen der Zellen bei ~ 1,4 x 10 5 Zellen / cm 2 auf VTN beschichteten Platten.
- Ändern Sie das Medium 24 h nach dem Aussäen.
- Am Tag 0 beginnen die Differenzierung nach 48 h, wenn die Zellen ~ 80% Konfluenz erreicht haben.
HINWEIS: Zur Erzielung einer hohen Pankreas Differenzierung Effizienz, Optimierung der Aussaatdichte und das Niveau der Konfluenz bei 48 h wird für jede einzelne Zeile zu empfehlen. - Saugen Sie das HPSC Medium und spülen Sie einmal die Zellen mit PBS ohne Ca 2+ und Mg 2+.
- Ändern Sie das Medium zu einer Differenzierung Tag 0 (d0) Medium.
- An den Tagen 1 bis 2, ändern Sie die differentiation Medium täglich, nach den Rezepten in Tabelle 8.
- Am Tag 3, die endgültigen endoderm Marker SOX17, FOXA2 und CXCR4 durch Immunfluoreszenzanfärbung untersuchen und Zytometrie Analyse fließen.
- Definitive endoderm Differenzierung in Pankreas - Vorläufer
- An den Tagen 3 bis 9, weiterhin definitive Endoderm Differenzierung in Richtung der pankreatischen lineage durch das Medium täglich ändern, entsprechend den Rezepturen in Tabelle 9.
- Am 7. Tag untersuchen die frühen Pankreas - Vorläufer (PP1) Marker PDX1 durch Immunfluoreszenzanfärbung und Durchflusszytometrie - Analyse.
- Am 10. Tag, so prüft die später Pankreas - Vorläufer (PP2) Marker PDX1 und NKX6.1. Inzwischen bereiten PP2 Zellen an der Luft-Flüssigkeit-Grenzfläche für eine weitere Differenzierung in endokrinen Pankreaszellen zu übertragen.
- Pankreatischen endokrinen unterscheidenrenzierung in der Luft-Flüssigkeit-Grenzfläche
- Behandeln Sie die PP2 Zellen mit 10 & mgr; M ROCK-Inhibitor 4 h vor der Dissoziation.
- Entfernen Sie das Medium und spülen Sie einmal die Zellen mit PBS ohne Ca 2+ und Mg 2+.
- In 2 ml 1x Dissoziation Reagenz zu PP2 Zellen in einer 10-cm-Schale und Inkubation bei 37 ° C für 2 - 3 min.
- Saugen Sie das Dissoziation Reagenz, bevor die Zellen abgelöst haben. In 10 ml Blar Medium und verteilen die PP2 Zellen in einzelne Zellen durch vorsichtiges Pipettieren nach oben und unten.
- Sammeln Sie die Einzelzellsuspension. Zählen Sie die Zellzahl und Pellet bei 200 xg für 5 min.
- Zellpellet bei ~ 0,5 x 10 5 Zellen / ul in S5 Differenzierungsmedium und Spot von 5 bis 10 & mgr; l der Zellen pro Punkt auf einem Transwell - Filtereinsatz. Legen Sie 10 bis 15 Punkte in einer 6-Well-Einsatz und ~ 100 Punkte in einem 10-cm Einsatz.
- Hinzufügen S5 Medium zu dem Boden jeder Transwell-Einsatz, ~ 1,5 ml für 6-well-Einsätzen und ~ 8 ml foder 10-cm-Einsätzen.
- Ändern Sie das Medium täglich mit den Rezepten in der Tabelle 10.
- Untersuchen pankreatische endokrine Marker PDX1, NKX6.1 NEUROD1, NKX2.2, Insulin und Glucagon an Tag 34 durch Immunfluoreszenzfärbung und Durchflusszytometrie - Analyse 17.
- Untersuchen HPSC abgeleitetes β-ähnlichen Zellfunktion mit einem Glukose-stimulierte Insulinsekretion (GSIS) Assay 16, 17.
Ergebnisse
HPSC chemisch definiertem und Feeder-freie Kultur Anpassung und Wartung
hPSCs kultiviert auf Imef Zuführungen können schnell auf VTN beschichteten Platten im Speiser freien Kulturbedingungen angepasst werden. Das gleiche Teilungsverhältnis wie normale Imef Feeder Kultur kann bei der Anpassung verwendet werden. 1A zeigt typische Morphologien von hPSCs auf Imef Zubringer in KSR-basierten Medium und auf einer VTN beschichtete Oberfläche in Feeder-freiem Medium. 1B zeigt eine typische morphologische Veränderung und das Wachstum von Kolonien HPSC während des ersten Durchgangs der Anpassung (4 Tage). Die Zellen können weiter agierten oder für zukünftige Experimente eingefroren. Führen Sie 2 - 3 Passagen der Anpassung Kultur vor Differenzierung Experimente beginnen. Karyotyping wird auch nach der Anpassung empfohlen, obwohl wir nicht Karyotyp Anomalien während der Anpassungsphase beobachtet haben.
"Fo: keep-together.within-page =" 1 "> Generation von iCas9 HPSC Linien durch TALEN-vermittelte AAVS1 TargetingWie zuvor gezeigt, hPSCs wurden mit einem Paar von AAVS1 TALEN Plasmiden und Cas9 und M2rtTA Plasmiden 5 elektroporiert. 2A und B zeigt detaillierte Spender Vektor - Design und die gesamte Prozedur Targeting iCas9 HPSC Klonlinien zu erzeugen. Nach Antibiotika - Selektion wurden Einzelzell-abgeleiteten Klone ausreichende Größe und typische HPSC Morphologie Anzeige bereit , abgeholt werden (10 - 12 Tage nach der Elektroporation, 2C). Regel ~ 50% der Klone richtig ohne Zufallsintegrationen gezielte, als verifiziert durch Southern Blotting (2D) 5. Zufällige Integration kann dazu führen leaky Expression Cas9 in Abwesenheit von Doxycyclin-Behandlung.
Effiziente Genetische MNDERUNG in hPSCs die iCRISPR Plattform
In etablierten iCas9 hPSCs wird Cas9 mit Doxycyclin Behandlung exprimiert und auf seine Ziel-Locus durch die transfizierten gRNAs geführt, wo sie DSBs erzeugt. In Abwesenheit eines Reparaturschablone, DNA-Reparatur durch NHEJ erzeugt indels, die oft in Genzerstörung oder knockout führen. In Gegenwart eines Reparaturschablone (beispielsweise ein ssDNA - Donor) kann HDR zur präzisen genetischen Veränderungen eingesetzt werden, wie beispielsweise eine patientenspezifische Mutation in einem Wildtyp - Hintergrund HPSC Erzeugung oder eine krankheitsassoziierten genetischen Variante in Patienten- Korrigieren abgeleitet iPSCs (3A). Es dauert ca. 1 Monat klonalen Mutantenlinien mit dem iCRISPR System erzeugen. Nach iCas9 Induktion und gRNA Transfektion T7EI und / oder RFLP-Tests wurden verwendet Cas9 Schneidleistung und die transfizierten Zellen zu beurteilen, wurden ausgesät später als einzelne Zellen in 10-cm-Schalen mit geringer Dichte (~ 500-2, 000Zellen / 10 cm-Schale). 10 - 12 Tage später, single-cell-derived Klone wurden in die Vertiefungen einer 96-Well - Platte für die Expansion und weitere Charakterisierung (dh Genotypisierung und Western - Blotting) , (3B) angeordnet ist . Da die iCRISPR vermittelte Gen - Knockout sehr leistungsfähig ist, wird kein Auswahlprozess beteiligt sind , und in der Regel 20-50% biallelischen Mutanten leicht erreicht werden kann (3C) 5. Für eine effiziente und präzise genetische Veränderungen, gRNAs sind cotransfiziert mit einem ssDNA Spender die spezifische Sequenzänderung trägt (für eine kleine, aber gezielte Veränderung der Genomsequenz). Oft Nachschneiden in modifizierten Allele zu verhindern, ist es empfehlenswert , eine stille Mutation in unmittelbarer Nähe zu oder in der PAM - Sequenz (3D) aufzunehmen. Mit diesem System ~ 10% der Klone die gewünschte HDR-vermittelten Genoms Modifikation ohne zusätzliche Änderungen in beiden Allelen tragen, erreicht werden (3E). Die schnelle und precise Modifikation des HPSC Genoms die iCRISPR-Plattform ermöglicht es uns, schnell und effizient HPSC Linien machen, die für das Studium der menschlichen Entwicklung und Krankheit als Vorbild dienen.
Effiziente Differenzierung von hPSCs zu Glucose ansprechenden β-ähnlichen Zellen
Die jüngsten Fortschritte in HPSC pankreatischen Differenzierung hat die Entwicklung von Protokollen erlaubt, die eng Bauchspeicheldrüsen Embryonalentwicklung rekapitulieren. Undifferenzierten hPSCs zuerst in die definitive Endoderm differenziert, dann in PDX1 + frühen pankreatischen Progenitoren (PP1) und PDX1 + NKX6.1 + später Pankreas - Vorläuferzellen (PP2) und schließlich in Glukose-responsive β-ähnlichen Zellen 16, 17, 19, 20 , 23, 24. Diese Protocols wurden weiter optimiert , zuverlässig PDX1 + NKX6.1 + Pankreas - Vorläufern und auf Glucose reagierenden β-wie 9 - Zellen zu erzeugen. 4A zeigt die detaillierten chemische Zusätze in jeder Phase der Differenzierung verwendet. Üblicherweise ~ 80% Konfluenz 2 Tage nach der anfänglichen Zell plating ist ideal für den Start der Differenzierung von HUES8 hPSCs (4B). verschiedene Einsaatdichten Testen wird dringend empfohlen, die optimierte Bedingung für jede spezifische Zelllinie zu entdecken. Normalerweise mindestens 75% FOXA2 + SOX17 + Zellen in der DE Stufe und 40% PDX1 + NKX6.1 + Zellen an der PP2 Stufe erreicht werden kann (4C). Am S5 Stufe die Anwesenheit einer Aura artigen Form um die Zellaggregat ist ein Indikator für die gute Überleben der Zellen, die für die weitere Differenzierung zu NKX6.1 + CPEP + Glukose-responsive β-ähnlichen Zellen wichtig ist (4D ). Dieses Protokoll ist nützlich für das Studium menschlicher Pankreas-d evelopment und Krankheit in einer Schale.

Abbildung 1. HPSC chemisch definiertem und Feeder freie Wartung und Kultur Anpassung von Imef Feeders. (A) Repräsentative Bilder von hPSCs kultiviert auf Imef Einzug oder auf VTN am Tag 4, bereit , aufgeteilt werden. (B) Typische Morphologie von hPSCs beim ersten Durchgang der Anpassung in Feeder-freie Kultur von einem Imef Einzug. Tag 4 hPSCs kultiviert auf Imef Speiser wurden in chemisch definiertem Medium mit ROCK-Inhibitor auf VTN beschichteten Platten aufgeteilt und plattiert. Das Teilungsverhältnis war das gleiche wie das übliche Teilungsverhältnis für in Imef feeder Bedingungen kultiviert werden. Das Medium wurde jeden Tag ohne ROCK-Inhibitor verändert. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
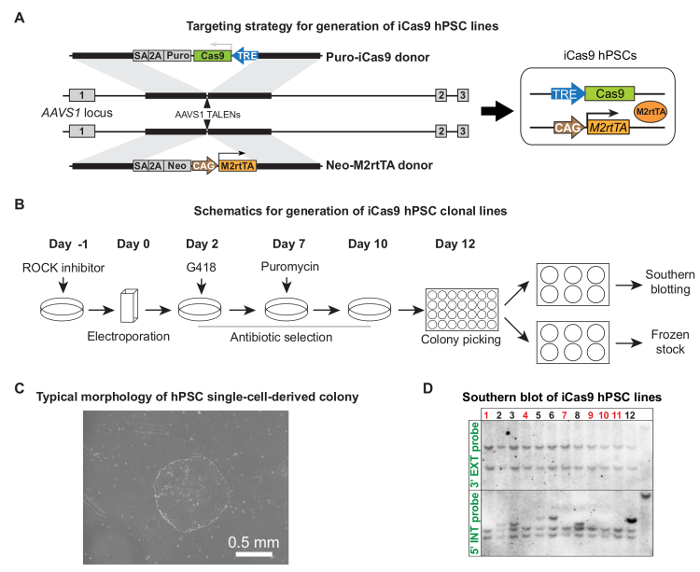
Abbildung 2. Erzeugung von iCas9 HPSC Lines. (A) Targeting - Strategie für die Erzeugung von iCas9 HPSC Linien. Puro-Cas9 Donor- und Neo-M2rtTA Spender wurden durch ein Paar von AAVS1 TALENS in den menschlichen AAVS1 locus abgezielt. (B) Schema des gesamten Targeting - Prozess, einschließlich Elektroporation, Antibiotika - Selektion, Kolonie Kommissionierung und Expansion und Zelllinie Charakterisierung. 12 Tage nach der Elektroporation - (C) Repräsentative Einzelzellen - Klon bereit , auf rund 10 abgeholt werden. (D) Southern - Blot - Beispiele für ohne die zusätzliche Integration von zwei Donorplasmiden richtig gezielt Klone identifiziert. Korrekte Klone sind rot markiert. (A) und (D) wurden von der Referenz 5, mit der Erlaubnis angepasst.ig2large.jpg "target =" _ blank "> Bitte hier klicken, um eine größere Version dieser Figur zu sehen.
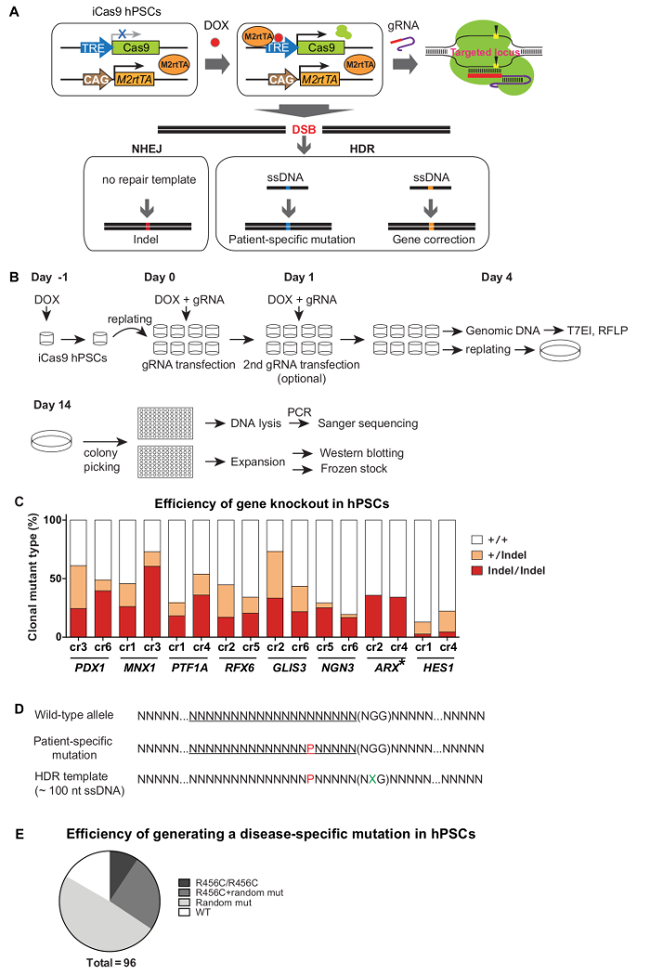
Abbildung 3. Effiziente Genmanipulation in hPSCs Mit dem iCRISPR - System. (A) Schema für die Erzeugung von Gen - Knockout - Mutanten oder präzise Genveränderungen das iCRISPR System. (B) Targeting Verfahren und klonalen Linie Einrichtung. (C) Gen - Knockout - Effizienz durch NHEJ in hPSCs 9. (D) Entwurf eines ssDNA eine spezifische Nucleotid - Modifikation trägt. P, in rot: patientenspezifische Mutation; X, in grün: stille Mutation. (E) Die Effizienz des HDR-vermittelte präzise Nucleotidmodifizierung. Eine genaue R456C - Mutation (verursacht durch eine spezifische Nukleotid C> T - Mutation) im GATA6 Locus wurde mit der iCRISPR - System eingeführt.(Siehe 5 für nähere Informationen zu einem Referenz). (C) und (E) wurden von Referenz angepasst 9 und Referenz 5 jeweils mit Berechtigungen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
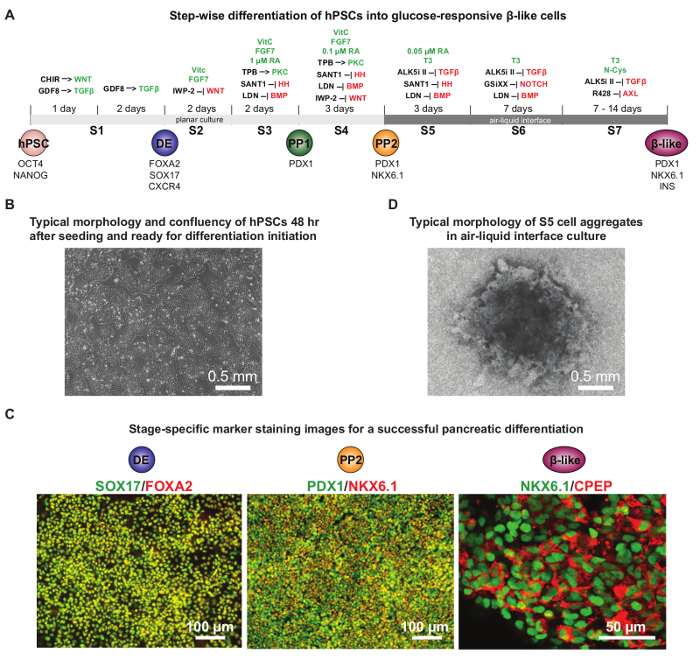
Abbildung 4. Directed HPSC Differenzierung in Glucose reagierenden Bauchspeicheldrüsen β-ähnliche Zellen. (A) Schematische Darstellung der detaillierten Differenzierung Protokoll, mit Chemikalien in jeder Phase der Differenzierung ergänzt. Signalwege, die während der Differenzierung aktiviert oder gehemmt werden, sind in grün oder rot markiert sind. CHIR: GSK3-Inhibitor; GDF8: Wachstumsdifferenzierungsfaktor 8 oder Myostatin, ein TGF-beta-Protein, ein Familienmitglied; HH: Igel; DE: Definitive endoderm; PP1:PDX1 + frühen Pankreas-Vorläufer; PP2: PDX1 + NKX6.1 + später Pankreas-Vorläufer. (B) Typische Konfluenz (70-80%) von hPSCs 48 h nach dem Aussäen , wenn Sie bereit für die Differenzierung Initiation. (C) Repräsentative Immunfluoreszenzanfärbung Bilder die erfolgreiche Differenzierung im Rahmen der endgültigen endoderm (DE) Stufe (FOXA2 und SOX17 Co-Färbung), Pankreas - Vorläuferstufe (PP2: PDX1 und NKX6.1 Co-Färbung) zeigt, und auf Glucose reagierenden β- wie Zellstadium (NKX6.1 und C-Peptid Co-Färbung). In der Regel mehr als 10% NKX6.1 + CPEP + Zellen an der β-ähnlichen Zellstadium, mindestens 75% FOXA2 + + SOX17 DE-Zellen und 40% PDX1 + NKX6.1 + PP2 Zellen an den entsprechenden Stufen erforderlich sind, zu erreichen. (D) Typische Morphologie eines Zellaggregats am S5 Stufe an einem Einsatz Filtermembran in der Luft-Flüssigkeit - Grenzfläche Kultur. Die überlebenden Zellen wurden in die Mitte des Aggregats (in dunkel) migriert und links eine Aura artige Struktur auf dem Rand. (A ) und (D) wurden von der Referenz 9 mit Berechtigungen angepasst. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
| Plasmid | Menge |
| AAVS1-TALEN-L | 5 ug |
| AAVS1-TALEN-R | 5 ug |
| AAVS1-Puro-iCas9 | 40 & mgr; g |
| AAVS1-Neo-M2rtTA | 40 & mgr; g |
Tabelle 1
| Grundierung | Sequenz |
| T7F | TAATACGACTCACTATAGGG |
| TracrR | AAAAGCACCGACTCGGTGCC |
Tabelle 2
| Komponente | Menge |
| ddH 2 O | 35,5 ul |
| 5x PCR-Reaktionspuffer | 10 & mgr; l |
| dNTP-Mix (25 mM) | 0,5 ul |
| T7F (10 uM) | 1,25 & mgr; l |
| TracrR (10 uM) | 1,25 & mgr; l |
| T7-gRNA IVT-Vorlage (250 nM) | 1 ul |
| DNA-Polymerase | 0,5 ul |
| Gesamt-PCR-Reaktionsgemisch | 50 ul |
Tisch 3
| PCR - Zyklusbedingungen | |||
| Zyklusnummer | Denaturieren | anneal | Erweitern |
| 1 | 94 ºC, 2 min | ||
| 31.02 | 94 ºC, 20 s | 60 ºC, 20 s | 72 ºC, 1 min |
| 32 | 72 ºC, 2 min | ||
Tabelle 4
| Komponente | Menge |
| T7 ATP | 2 ul |
| T7 CTP | 2 ul |
| T7 GTP | 2 ul |
| T7 UTP | 2 ul |
| T7 10x Puffer | 2 ul |
| T7 Enzymmischung | 2 ul |
| PCR amplifizierten Template | 8 ul |
| Insgesamt in vitro gRNA Transkription Mix | 20 uL |
| über Nacht für 6 h bei 37 ºC inkubieren, um |
Tabelle 5
| Komponente | Menge (ul) |
| Ungereinigtem PCR Produkt | 8 |
| Puffer 2 10x | 2 |
| Destilliertes Wasser (dH 2 O) | 10 |
Tabelle 6
| DNA - Denaturierung und Hybridisierung Zyklusbedingungen | ||
| Temperatur | Dauer | Thermocycler Bedingungen |
| 95 ° C | 10 Minuten | |
| 85 ° C | 1 Minute | Rampe auf 85 ° C bei 2 ° C / s |
| 75 ° C | 1 Minute | Rampe auf 75 ° C bei 0,3 ° C / s |
| 65 ° C | 1 Minute | Rampe auf 65 ° C bei 0,3 ° C / s |
| 55 ° C | 1 Minute | Rampe auf 55 ° C bei 0,3 ° C / s |
| 45 ° C | 1 Minute | Rampe auf 45 ° C bei 0,3 ° C / s |
| 35 ° C | 1 Minute | Rampe auf 35 ° C bei 0,3 ° C / s |
| 25 ° C | 1 Minute | Rampe auf 25 ° C bei 0,3 ° C / s |
| 4 ° C | Halt | |
Tabelle 7
| Stufe | Tag | Medien | Ergänzen | |
| S1 | d0 | S1 | GDF8 100 ng / mL | CHIR-99021 3 & mgr; M |
| d1 | S1 | GDF8 100 ng / mL | CHIR-99021 0,3 & mgr; M | |
| d2 | S1 | GDF8 100 ng / mL | ||
| S1 Medium: MCDB 131 + 1x L-Glutaminergänzung + 0,5% BSA + 1,5 g / L NaHCO 3 + 10 mM Glucose | ||||
Tabelle 8
| Stufe | Tag | Medien | Ergänzen | |||||||
| S2 | d3-d4 | S1 | LAA 0,25 mM | FGF7 50 ng / ml | IWP-2 2,5 & mgr; M | |||||
| S3 | d5-d6 | S3 | LAA 0,25 mM | FGF7 50 ng / ml | SANT-1 0,25 & mgr; M | RA 1 uM | LDN 100 nM | TPB 200 nM | ITS-X 1: 200 | |
| S4 | d7-D9 | S3 | LAA 0,25 mM | FGF7 2 ng / ml | SANT-1 0,25 & mgr; M | RA 0,1 uM | LDN 200 nM | TPB 100 nM | ITS-X 1: 200 | IWP-2 2,5 & mgr; M |
| S3 Medien: MCDB 131 + 1x L-Glutamin Ergänzung + 2% BSA + 2,5 g / l NaHCO 3 + 10 mM Glucose | ||||||||||
Tabelle 9
| Stufe | Tag | Medien | Ergänzen | |||||||
| S5 | d10-d12 | S5 | T3 1 uM | ALK5i II 10 & mgr; M | SANT-1 0,25 uM | RA 0,05 uM | LDN 100 nM | ITS-X 1: 200 | ZnSO4 10 & mgr; M | Heparin 10 ug / ml |
| S6 | D13-St d19 | S5 | T3 1 uM | ALK5i II 10 & mgr; M | GSiXX 100 nM | LDN 100 nM | ITS-X 1: 200 | ZnSO4 10 & mgr; M | Heparin 10 ug / ml | |
| S7 | d20- d33 | S5 | T3 1 uM | ALK5i II 10 & mgr; M | N-Cys 1mM | Trolox 10 uM | R428 2 uM | ITS-X 1: 200 | ZnSO4 10 & mgr; M | Heparin 10 ug / ml |
| S5 Medien: Blar + 1x L-Glutamin Ergänzung + 2% BSA + 1,5 g / l NaHCO 3 + 20 mM Glucose | ||||||||||
Tabelle 10
Diskussion
Zeit Überlegungen zur Erzeugung von Mutantenlinien
Obwohl die jüngsten Ansätze auf Basis von CRISPR / Cas-Systeme für die Genom Bearbeitung zur erfolgreichen Ausrichtung geführt haben, ein wirksamer und universelle Plattform würde für größere Analysen von Genfunktionen vorzuziehen. Die iCRISPR Plattform bietet eine schnelle und effiziente Methode Mutationen zu jedem Gen von Interesse 5, 9 einzuführen. Erstens erlaubt die PCR-basierte gRNA Syntheseverfahren die Herstellung von Hunderten von gRNAs in Array-Format an einem Tag ohne die zeitraubende Klonierungsschritte. Zweitens mit Doxycyclin-induzierbaren Expression in Cas9 iCas9 hPSCs erfordert der Schritt, der eine gRNA Transfektion nur eine minimale Menge an Arbeit, und somit können mehrere gRNA Targeting-Experimente gleichzeitig durchgeführt werden. Drittens, aufgrund der hohen Wirkungsgrade erreichbar Targeting mit unserem System die Analyse von ~ 24 bis 48 Kolonien pro gRNA sollte transfiziert ausr seiniziente mehrere monoallelic und biallelic Mutantenlinien für ein einzelnes Gen zu schaffen, obwohl die Effizienz auf dem Zielort variieren. Da es mechanisch für eine ausgebildete Person möglich ist, zu 384 Kolonien Pick (4 x 96-Well-Platten) in einer Sitzung, die sollte nehmen ~ 4 h unter einem Binokular kann eine ausgebildete Person zu erwarten Mutantenlinien zu erzeugen, innerhalb beeinflussen 12 Gene 12 Monate. Die schlanke Generation von HPSC Mutanten in kurzer Zeit ermöglicht die systematische Analyse einer Reihe von Transkriptionsfaktoren und / oder Signalwegs - Komponenten , die miteinander in Wechselwirkung treten, den Entwicklungsprozess regulieren 9. Zusätzlich öffnet effiziente Multiplex-Gen-Targeting auch die Tür genetischen Interaktionen zugrunde liegenden komplexen menschlichen Eigenschaften zu untersuchen.
Die Erzeugung von Precise Genveränderungen
Die Ableitung von patientenspezifischen iPS-Zellen aus leicht zugänglichen somatischen Zelltyp s und die Differenzierung in krankheitsrelevanten Zelltypen bieten eine große Chance für die funktionale Validierung von krankheitsassoziierten Mutationen. Aufgrund der beträchtlichen Variabilität der genetischen Hintergrund zwischen Individuen, direkte Vergleiche zwischen iPSCs von Patienten und von gesunden Spendern kann nicht zulassen, dass eine Krankheit Phänotypen von Hintergrundeffekten zu unterscheiden. Daher ist es notwendig , durch eine Korrektur der Krankheitsmutation zurück in die Wildtyp - Sequenz isogenen Kontrolle iPSCs zu erzeugen oder die Patienten-spezifischen Mutationen in einem Wildtyp HPSC Hintergrund vorstellen, wie 26 andere 25, vorgeschlagen. Neben loss-of-function (null) Mutationen, kann man nun genauer Krankheitsmechanismen durch die Einführung eines Patienten-spezifischen Sequenzveränderungen in den endogenen Lokus in hPSCs einschließlich hypermorphic, hypomorphen, neomorphe oder dominant-negative Patienten sezieren Mutationen.
ntent "> Precise genetische Modifikation HDR beschäftigt für DSB-Reparatur in Gegenwart eines Reparaturschablone. Da es viel weniger effizient als NHEJ-vermittelter DNA-Reparatur ist ein Donorplasmid die patientenspezifische Mutation, eine Arzneimittelselektionskassette, und Homologiearme enthalten, zuvor wurde als Reparaturschablone 2 verwendet. nach der Medikamentenauswahl und Überprüfung der patientenspezifischen Mutation in einem zweiten Schritt wird im allgemeinen benötigt , um die Arzneimittelselektionskassette zu entfernen. Während das am häufigsten verwendete Cre-loxP und FLP-FRT - Systeme hinterlassen Restsequenz in den endogenen Lokus, die Verwendung von piggyBac Transposon wurde zur nahtlosen Entfernung der Arzneimittelselektionskassette erlaubt 27. in neuerer Zeit haben kurze ssDNA templates auch effizient HDR zu unterstützen mit technisch DNA - Endonukleasen 28 gezeigt. im Vergleich zu einem Donorplasmid können ssDNA direkt synthetisiert werden und umgeht so die zeitraub Klonierungsschritt. Hier hat es sein,dass de gezeigt durch Cotransfektion gRNA und eine ssDNA Vorlage Homologie gerichtete Reparatur zu induzieren, können iCRISPR verwendet werden, um spezifische Nukleotidmodifikationen mit hoher Effizienz einzuführen. Dies ist wichtig, nicht nur für die Rolle der essentiellen Nukleotide innerhalb Protein funktionellen Domänen sezieren, aber auch menschliche Krankheit Mutationen zur Modellierung und möglicherweise diese krankheitsassoziierten Mutationen für die therapeutische Intervention zu korrigieren. Aufgrund der großen Anzahl von Suszeptibilitätsloci, die jeweils mit mehreren Sequenzvarianten sind, komplex und multigene Krankheiten wie Diabetes Modellierung ist eine Herausforderung für Genetiker gewesen. Da die iCRISPR Plattform für die schnelle Erzeugung einer allelischen Serie oder für Gen-Targeting multiplex permissive ist, kann es die Untersuchung mehrerer krankheitsassoziierter loci erleichtern entweder einzeln oder in Kombination mit isogenen Hintergründen.Targeting Effizienzen und Off-Target-Effekte
Wir haben eine gute Korrelation zwischen dem T7E1 und RFLP Untersuchungsergebnisse und der Anzahl der Mutantenlinien gefunden durch Sequenzierung identifiziert. Dies unterstreicht die Bedeutung dieser Tests parallel mit der Einrichtung von klonalen Linien durchführen. Während die Zielwirkungsgrade in Abhängigkeit von der genomischen Loci variieren kann erreicht werden , in den meisten Ein-Gen-Targeting - Experimente, 20-60% der Klone mit beide Allele mutiert gefunden wurden (einschließlich in-frame und Rasterverschiebungsmutationen) 9. In Fällen , in denen Multiplex - Gen - Targeting durchgeführt wurde, Dreier- biallelic mutierten Klone mit 5-10% Wirkungsgrad wurden 5 erhalten. ssDNA-vermittelte HDR mehrerer Gene wurden auch genaue genetische Veränderungen durchgeführt zu erhalten, mit der Effizienz des Erhaltens homozygote Knock-in - Klone im Bereich von 1 5 - 10%. Arbeiten mit CRISPR / Cas in hPSCs, alle Mutationen in potenziellen Off-Target-Sites, die sind noch nicht die gleiche gRNA Zielsequenz teilen zu erfasselass = "xref"> 5, 9, 12. Whole-Genom - Sequenzierung in einer kürzlich durchgeführte Studie hat es versäumt , auch erhebliche Off-Target - Mutationen in klonalen HPSC Linien zu identifizieren , erzeugt unter Verwendung von CRISPR / Cas 29. Trotzdem jede mögliche Wirkung von Verwechselung Phänotypen durch Mutationen an Off-Target-Effekt Stellen eingeführt zu minimieren, wird vorgeschlagen, mindestens zwei unabhängige gRNAs Targeting verschiedenen Sequenzen innerhalb des gleichen Gens unter Verwendung unabhängiger Mutantenlinien zu erzeugen. Ähnlich wie in mehreren Linien beobachteten Phänotypen erzeugt unterschiedliche gRNAs mit könnte die Möglichkeit ausschließen, vor allem, dass der Phänotyp von einem Off-Target-Effekt kommt.
Feeder-abhängige gegen unabhängige Kultur und Targeting
Herkömmliche Methoden zur HPSC Kultur beinhalten ihre Wartung und Erweiterung auf Feeder-Zellen in Medien, die Serum oder Serumersatz, der tierische prod enthältdukten, wie Rinderserumalbumin. Feeder-Zellen, Serum, Serum-Ersatz, und alle Albumin enthalten komplexe, nicht definierten Komponenten und zeigen erhebliche Chargenvariabilität. Die Anpassung an die Feederfrei und chemisch definierten Zustand erheblich reduziert den Aufwand für die HPSC Wartung und, was noch wichtiger ist, die Konsistenz der Differenzierungsexperimente erhöht. Derzeit gibt es nur sehr wenige Studien , die Genombearbeitungsverfahren auf hPSCs kultiviert in vollständig definierten Kulturbedingungen 30 beschreiben. Wir haben höhere CRISPR gefunden Effizienzen bei gRNA Transfektion und klonalen Ablagerung Targeting in Feeder freien Bedingungen durchgeführt wurden, im Vergleich zu Feeder-abhängige Kulturbedingungen. Wir glauben, dass dies zu einem erhöhten Überleben der Zelle nach der Transfektion durch und Einzelzellaussaat für die Koloniebildung. Außerdem haben Nährzellen zuvor gezeigt worden, Transfektionsreagenzien zu maskieren, wodurch die Transfektionseffizienz zu senken.
VereinigungGenome Editing mit gerichteten Differenzierung
Vorherige Differenzierungs Protokolle nur einen geringen Anteil an Insulin-positive Zellen ergab, von denen die meisten waren polyhormonal und ähnelten fötalen endokrinen Zellen 23. Die jüngsten Fortschritte hat die Differenzierung von hPSCs in reiferen auf Glucose reagierenden Beta-ähnlichen Zellen erlaubt 16, 17, 19, 20. Wir können routinemäßig mindestens 75% endgültige Entodermzellen, 40% PDX1 + NKX6.1 + Pankreas - Vorläufern, und rund 20% NKX6.1 + CPEP + Glukose-responsive Beta-ähnliche Zellen erhalten 9 HUES8 hPSCs verwenden. Wenn es mit dem iCRISPR Genom Editing-System kombiniert, das robustere Differenzierung Protokoll hat die Analyse von Transkriptionsfaktoren erleichtert, die auf die Differenzierung Pankreas-Vorläufer und endokrine Stadien der Pankreas entscheidend sind. Dadurch wirdes möglich, in zukünftigen Studien, eine große Anzahl von Kandidatenkrankheitsgene für die funktionelle Überprüfung und Untersuchung der Mechanismen zu untersuchen , die 9 - Diabetes zu Grunde liegen.
Zukünftige Anwendungen oder Wegbeschreibungen
Unser iCRISPR System die Erzeugung komplexer genomischer Veränderungen, wie zum Beispiel die Schaffung von Reporter - Allele durch HDR-vermittelte Gen - Targeting mit langen Spender - DNA - Matrizen - Codierung Protein - Tags oder fluoreszierenden Reporter können erleichtern 12. Wir haben gezeigt, dass aufgrund des hohen Wirkungsgrade erreicht CRISPR im System Targeting kann dieser Prozess in hPSCs durchgeführt werden, ohne die Notwendigkeit für weitere Wirkstoffselektion 12. Weiterhin kann Targeting gemultiplexten Gen verwendet werden , um genetische Wechselwirkungen zugrunde liegenden komplexen menschlichen Erkrankung zu untersuchen, wie in unserer Studie gezeigt, die wir 31 ist das erste Beispiel für eine solche Arbeit glauben. iCRISPR kann auch durch die Schaffung von Löschungen regulatorischen Kontrolle zu verstehen Gen verwendet werden, entweder in nicht-kodierenden RNAs oder in der Gen-regulatorische Regionen wie Promotoren und Enhancer. Um effizient zu erzeugen regulatorische Mutanten unter Verwendung iCRISPR kann gRNAs werden entwickelt, um die Bindungsstelle eines DNA-bindenden Proteins zu stören, einschließlich, aber nicht auf die basale Transkriptionsmaschinerie oder eines gewebespezifischen Transkriptionsfaktor beschränkt. ssDNA template-vermittelte HDR kann auch zu mutieren spezifische Protein-Bindungsstellen verwendet werden. Schließlich sehen wir , dass eine weitere Optimierung der Nutzung der iCRISPR Plattform in hPSCs für höheren Durchsatz genetische Analysen von Pluripotenz Phänotypen oder Krankheitsphänotypen ermöglichen würde , wenn sie mit einem in vitro Differenzierung Protokoll kombiniert. Diese iCRISPR-vermittelte Studien können für die schnellere Identifizierung von Kandidatenkrankheitsassoziierten Genen und Studien über ihre funktionelle Bedeutung ermöglichen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde teilweise durch die NIH / NIDDK (R01DK096239) und New York State Stem Cell Science (NYSTEM C029156) gefördert. ZZ wurde von der NYSTEM Postdoc-Stipendium aus dem Zentrum für Stammzellbiologie des Sloan Kettering Institute unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemically defined medium (E8) | Thermo Fisher Scientific | A1517001 | Essential 8 basal medium and Essential 8 supplement included |
| Truncated recombinant human form of vitronectin | Thermo Fisher Scientific | A14700 | |
| ROCK inhibitor Y-27632 | Selleck Chemicals | S1049 | |
| Dissociation reagent (TrypLE Select enzyme) | Thermo Fisher Scientific | 12563029 | 1x, animal origin free, recombinant enzyme |
| G418 Sulfate | Thermo Fisher Scientific | 10131035 | Geneticin Selective Antibiotic |
| Puromycin dihydrochloride | Sigma-Aldrich | P8833 | Puromycin Selective Antibiotic |
| DNA Polymerase (Herculase II Fusion) | Agilent Technologies | 600679 | PCR kit |
| MEGAshortscript T7 Transcription kit | Thermo Fisher Scientific | AM1354 | |
| MEGAclear Transcription Clean-Up Kit | Thermo Fisher Scientific | AM1908 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| Opti-MEM medium | Thermo Fisher Scientific | 31985062 | Reduced Serum Medium |
| Lipofectamine RNAiMAX Transfection Reagent | Thermo Fisher Scientific | 13778150 | |
| DNeasy Blood & Tissue Kit | QIAGEN | 69504 | Genomic DNA extraction kit |
| Proteinase K | Roche | 3115879001 | |
| MCDB 131 medium | Thermo Fisher Scientific | 10372-019 | |
| BLAR medium | Thermo Fisher Scientific | Custom-made with a published formulation (Rezania et al., 2014) | |
| L-glutamine supplement (GlutaMAX) | Thermo Fisher Scientific | 35050061 | |
| NaHCO3 | Thermo Fisher Scientific | 144-55-8 | |
| Glucose | Sigma-Aldrich | G8769 | |
| BSA | LAMPIRE Biological Laboratories | 7500855 | Fatty acid free |
| GDF8 | PeproTech | 120-00 | |
| CHIR-99021 | Stemgent | 04-0004 | GSK-3 inhibitor |
| L-Ascorbic acid | Sigma-Aldrich | A4544 | Vitamin C |
| FGF7 | R&D Systems | 251-KG | |
| SANT1 | Tocris Bioscience | 1974 | Hedgehog inhibitor |
| RA | Sigma-Aldrich | R2625 | Retinoic acid |
| LDN | Stemgent | 04-0019 | BMP inhibitor |
| IWP-2 | Tocris Bioscience | 3533 | Wnt antagonist |
| ITS-X | Thermo Fisher Scientific | 51500-056 | |
| TPB | EMD Millipore | 565740-1MG | PKC activator |
| 3,3’,5-Triiodo-L-thyronine (T3) | Sigma-Aldrich | T6397 | |
| ALK5i II | Enzo Life Sciences | ALX-270-445 | ALK5 inhibitor II |
| ZnSO4 | Sigma-Aldrich | Z0251 | |
| Heparin | Sigma-Aldrich | H3149 | |
| GSiXX | EMD Millipore | 565789 | Gamma secretase inhibitor XX, NOTCH signaling inhibitor |
| N-Cys (N-acetyl cysteine) | Sigma-Aldrich | A9165 | |
| Trolox | EMD Millipore | 648471 | Vitamin E analogue |
| R428 | Selleck Chemicals | S2841 | AXL receptor tyrosine kinase inhibitor |
| 24 mm Transwell with insert | Corning Life Sciences | 3414 | |
| Gene Pulser Xcell Electroporation System | Bio-Rad | 1652660 | |
| 0.4 cm Electroporation Cuvettes | Bio-Rad | 1652081 | |
| AAVS1-TALEN-L | Addgene | 59025 | |
| AAVS1-TALEN-R | Addgene | 59026 | |
| AAVS1-Neo-M2rtTA | Addgene | 60843 | |
| AAVS1-Puro-iCas9 | Addgene | 58409 | |
| T7 Endonuclease I | NEB | M0302L | |
| Buffer 2 | NEB | B7002S | NEBuffer 2 |
| Long oligonucleotide | Eton Bioscience or IDT | ||
| SOX17 antibody | R&D Systems | AF1924 | 1:500 |
| FOXA2 antibody | Millipore | 07-633 | 1:100 |
| CXCR4-APC antibody | R&D Systems | FAB170A | 1:25 |
| PDX1 antibody | R&D Systems | AF2419 | 1:500 |
| NKX6.1 antibody | DSHB | F55A12 | 1:500 |
| NKX2.2 antibody | DSHB | 74.5A5 | 1:100 |
| NEUROD1 antibody | Santa Cruz Biotechnology | sc-1084 | 1:100 |
| Insulin antibody | Dako | A0564 | 1:2,000 |
| C-peptide antibody | DSHB | GN-ID4-c | 1:2,000 |
| Glucagon antibody | Sigma-Aldrich | G2654 | 1:1,000 |
Referenzen
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6 (6), 507-512 (2005).
- Zwaka, T. P., Thomson, J. A. Homologous recombination in human embryonic stem cells. Nat Biotechnol. 21 (3), 319-321 (2003).
- Eiges, R., et al. Establishment of human embryonic stem cell-transfected clones carrying a marker for undifferentiated cells. Curr Biol. 11 (7), 514-518 (2001).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- González, F., et al. An iCRISPR Platform for Rapid, Multiplexable, and Inducible Genome Editing in Human Pluripotent Stem Cells. Cell Stem Cell. 15 (2), 215-226 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Braam, S. R., et al. Improved genetic manipulation of human embryonic stem cells. Nat Methods. 5 (5), 389-392 (2008).
- Zhu, Z., et al. Genome Editing of Lineage Determinants in Human Pluripotent Stem Cells Reveals Mechanisms of Pancreatic Development and Diabetes. Cell Stem Cell. 18 (6), 755-768 (2016).
- Kotini, A. G., et al. Functional analysis of a chromosomal deletion associated with myelodysplastic syndromes using isogenic human induced pluripotent stem cells. Nat Biotechnol. 33 (6), 646-655 (2015).
- Carlson-Stevermer, J., et al. High-Content Analysis of CRISPR-Cas9 Gene-Edited Human Embryonic Stem Cells. Stem Cell Reports. 6 (1), 109-120 (2016).
- Zhu, Z., Verma, N., Gonzalez, F., Shi, Z. D., Huangfu, D. A CRISPR/Cas-Mediated Selection-free Knockin Strategy in Human Embryonic Stem Cells. Stem Cell Reports. 4 (6), 1103-1111 (2015).
- Mandegar, M. A., et al. CRISPR Interference Efficiently Induces Specific and Reversible Gene Silencing in Human iPSCs. Cell Stem Cell. 18 (4), 541-553 (2016).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Methods. 8 (5), 424-429 (2011).
- Rezania, A., et al. Production of functional glucagon-secreting alpha-cells from human embryonic stem cells. Diabetes. 60 (1), 239-247 (2011).
- Pagliuca, F. W., et al. Generation of functional human pancreatic beta cells in vitro. Cell. 159 (2), 428-439 (2014).
- Rezania, A., et al. Reversal of diabetes with insulin-producing cells derived in vitro from human pluripotent stem cells. Nat Biotechnol. 32 (11), 1121-1133 (2014).
- Chen, S., et al. A small molecule that directs differentiation of human ESCs into the pancreatic lineage. Nat Chem Biol. 5 (4), 258-265 (2009).
- Nostro, M. C., et al. Efficient generation of NKX6-1+ pancreatic progenitors from multiple human pluripotent stem cell lines. Stem Cell Reports. 4 (4), 591-604 (2015).
- Russ, H. A., et al. Controlled induction of human pancreatic progenitors produces functional beta-like cells in vitro. EMBO J. 34 (13), 1759-1772 (2015).
- Zhu, Z., Gonzalez, F., Huangfu, D. The iCRISPR platform for rapid genome editing in human pluripotent stem cells. Methods Enzymol. 546, 215-250 (2014).
- Soh, C. L., Huangfu, D. CRISPR/Cas9-Mediated Mutagenesis of Human Pluripotent Stem Cells in Defined Xeno-Free E8 Medium. Methods in Molecular Biology. 1498, (2017).
- D'Amour, K. A., et al. Production of pancreatic hormone-expressing endocrine cells from human embryonic stem cells. Nat Biotechnol. 24 (11), 1392-1401 (2006).
- Kelly, O. G., et al. Cell-surface markers for the isolation of pancreatic cell types derived from human embryonic stem cells. Nat Biotechnol. 29 (8), 750-756 (2011).
- Musunuru, K. Genome editing of human pluripotent stem cells to generate human cellular disease models. Dis Model Mech. 6 (4), 896-904 (2013).
- Hockemeyer, D., Jaenisch, R. Induced Pluripotent Stem Cells Meet Genome Editing. Cell Stem Cell. 18 (5), 573-586 (2016).
- Yusa, K., et al. Targeted gene correction of alpha1-antitrypsin deficiency in induced pluripotent stem cells. Nature. 478 (7369), 391-394 (2011).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Veres, A., et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 15 (1), 27-30 (2014).
- Huang, X., et al. Production of Gene-Corrected Adult Beta Globin Protein in Human Erythrocytes Differentiated from Patient iPSCs After Genome Editing of the Sickle Point Mutation. Stem Cells. 33 (5), 1470-1479 (2015).
- Shi, Z. D., et al. Genome Editing in hPSCs Reveals GATA6 Haploinsufficiency and a Genetic Interaction with GATA4 in Human Pancreatic Development. Cell Stem Cell. , (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten