このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
β-アレプチンの並列尋問2 PRESTOタンゴアッセイを用いたGPCR規模でのリガンドスクリーニングの募集
要約
GPCRは、魅力的な薬剤の標的であることを考えると、GPCRリガンドスクリーニングは、リード化合物の同定や脱孤児化研究に不可欠です。これらの取り組みに向けて、TEVベースのレポーターアッセイを用いて約300 GPCRで一過性β-arrestin2募集の同時プロファイリングに使用されるオープンソースのリソースプラットフォームPRESTO-Tangoについて説明します。
要約
細胞シグナル伝達経路の範囲の最大かつ最も汎用性の高い遺伝子スーパーファミリーおよびメディエーターとして、Gタンパク質共役受容体(GPCRs)は製薬業界にとって最も有望な標的の1つである。Ergoは、創薬やGPCR薬理学と成果を操作するための遠隔操作ツールを表すため、GPCRリガンドスクリーニングアッセイの設計、実施、最適化が重要です。これまで、Gタンパク質依存アッセイは、リガンド誘導事象を検出し、二次メッセンジャーの生成を定量化するこの分野を代表して行っていました。しかし、機能的選択性の出現以来、他のいくつかのGタンパク質非依存経路およびGタンパク質依存アッセイに関連する限界に対する意識の向上と同様に、代替の作成に向けてより大きな推進があるGPCRリガンドスクリーニングアッセイ。この取り組みに向けて、我々は、そのような資源の1つであるPRESTOタンゴプラットフォーム、ヒトGPCR-omeの並列かつ同時の尋問を可能にするルシファーゼレポーターベースのシステムの適用について述べている。技術的にも経済的にも実現不可能。Gタンパク質独立型β-arrestin2採用アッセイに基づいて、GPDRにおけるβ-arrestin2媒介性人身売買およびシグナリングの普遍性により、PRESTO-TANGOは約100人を含む約300の非嗅覚ヒトGPCRsを研究するための適当なツールとなる孤児受容体。PRESTOタンゴの感度と堅牢性は、既知の薬物の新しいGPCR標的を発見したり、孤児受容体の新しいリガンドを発見するために使用される複合ライブラリを使用して、一次ハイスループットスクリーンに適しています。
概要
Gタンパク質共役受容体(GPCRs)は、膜貫通タンパク質の最大かつ最も多様なファミリーを構成し、細胞とその環境間の通信インターフェースとして動作する1。GPCRsの多様性は、神経伝達物質からヌクレオチド、ペプチドから光子、および多くのリガンドの多様な配列を検出する能力と、細胞増殖、移動、分化、アポトーシス、細胞焼成などに関与する多数の下流シグナル伝達カスケードを調節する能力によって強調される。彼らの普遍性と多くの生理学的プロセスへの関与を考慮すると、この受容体ファミリーは、現在入手可能な処方薬の3分の1以上がGPCRs4を標的としているという事実によって示される、最も治療上の重要さである。しかし、これらの既存の治療法は、スーパーファミリーの小さなサブセット(推定10%)のみを標的とし、多くのGPCRsの薬理学は未解明のままである。さらに、100以上のGPCRsが、内因性リガンド5と一致していないとして、孤児受容体として存在する。したがって、GPCRリガンドスクリーニングは、リード発見と最適化への道を開き、おそらく臨床試験段階に道を開くため、脱ファン化と医薬品開発において重要です。
GPCRリガンドスクリーニング法は、従来、Gタンパク質依存性またはGタンパク質独立性機能アッセイ6の2つのカテゴリーのうちの1つに分類されている。GPCRシグナル伝達は、Gαサブユニット7に結合したGDPに対するGTPの交換によって活性化されるヘテロトリメリックGタンパク質(Gαβγ)によって調節される。活性化された受容体からのシグナルは、cAMP、カルシウム、DAG、およびIP3などの二次メッセンジャーを介してGタンパク質によって伝達され、下流エフェクター8で下流シグナル伝達を媒介する。Gタンパク質シグナル伝達の機能的な結果の性質は、受容体活性化を反映する細胞ベースのアッセイを作成するために利用されてきました。Gタンパク質シグナル伝達における近位(直接)または遠位(間接)事象を測定するこれらの方法は、GPCRリガンドスクリーニングに最も頻繁に使用され、主に脱孤児化研究6で採用されている。GPCR媒介Gタンパク質活性化を直接測定するアッセイの例としては、[35S]GTPγS結合アッセイが挙げられる。 Gαサブユニットに対する放射標識および非加水分解性GTPアナログと、Förster/生物発光共鳴エネルギー伝達(FRET/BRET、それぞれ)プローブとGPCR-GαおよびGα/Gγ相互作用を測定し、9年、10年にわたってより多くの牽引力を得ている。遠位イベントを監視するアッセイは、GPCRプロファイリングに最も一般的に使用されるツールです。例えば、cAMPおよびIP1/3アッセイは、Gタンパク質依存性二次メッセンジャーの細胞内蓄積を測定する一方で、[Ca2+]Gタンパク質活性化に関与する特異的応答要素を含むフラックスおよびレポーターアッセイ(CRE、NFAT-RE、SRE、SRF-RE)シグナルカスケード11をさらに下流の事象を調べる。前述のアッセイのほとんどはハイスループットレベルで実行できますが、 かなり敏感であり、特定のアッセイ固有の利点を誇る(例えば、GTPγS結合の場合の完全/部分的なアゴニスト、中立的アンタゴニストおよび逆アゴニスト間の差別、または[Ca2+]およびIP1/3)6などの生細胞上のアッセイ機能性6は、残念ながら既存のG依存性タンパク質方法は存在しない。これは主に、複数のGタンパク質サブファミリーがGPCBにネイティブに結合し、いくつかのカスケードでシグナル伝達を行い、オーファンGPDRで未知のGタンパク質結合を生じるためです。この問題を軽減するために、アッセイは、cAMPのような単一の共通シグナル伝達リードアウトを介して無差別Gタンパク質結合を強制するために開発された、およびCa2+、それらのほとんどは低スループット12であるが。
GPCRのライフサイクルの重要な側面は、Gタンパク質依存性シグナル伝達の終結であり、Gタンパク質の解離を誘導するβ-アスタミンの募集を通じて大部分が起こり、最終的にはクラトリンコーティングされた内在化を標的とする受容体を脱感作する。β-アレスティンの最もユビキタスに発現されるアイソフォームは、非視覚的β-アレスティン1およびβ-アレスティン2であり、また、それぞれ14のアレスティン-2及びアレスティン-3と示される。GPCRリガンドスクリーニングに新たな次元を追加するGタンパク質独立細胞ベースアッセイを入力してください。受容体の密売、標識のない全細胞、およびβ-アレプチンの募集アッセイは、すべて注目すべき例である。GPCR人身売買アッセイは、フルオロフォア標識リガンドまたは受容体15を標的とする共内在化抗体を採用する一方、ラベルフリーの全細胞アッセイは、リガンド結合によって誘導される細胞変化を電気信号または光学信号16などの定量可能な出力に変換するバイオセンサーを使用する。特に、典型的なGPCR-β-アレッシン相互作用は、機能アッセイ17のレパートリーにおける魅力的なツールとしてβ-アレッシン募集アッセイをファッションする。タンゴシステムは、わずか10年前にバルネアらによって最初に開発され、 3つの外因性遺伝要素の導入を伴う:β-arrestin2とタバコエッチウイルスプロテアーゼ(TEVp)からなるタンパク質融合、タバコエッチングを介してGPCRにつながれるテトラサイクリントランス活性化因子(tTA) ウイルスプロテアーゼ切断部位(TEVcs)と、逮捕を促進するためにV2バソプレシン受容体(V2尾)のC末語からの配列が先行し、その転写が誘発されるレポータールシメラーゼ遺伝子tTA転写因子は核への転座、β-アレプチン2募集後の膜アンカーから解放される(図1)18。GPCR活性化およびβ-arrestin2募集の定量的測定値は、その後、発光のための読み取りによって決定することができる。顕著な違いは、受容体の密売およびラベルフリーの全細胞法は比較的低いスループットであるが、タンゴは、標的受容体に特異的な選択的読み出しおよびシグナル統合による感受性を含むいくつかの利点を有し、これはより大きなスケール18でのリガンドスクリーニングに適した候補となる。
これらの戦略的特徴を考慮して、KroezeらはPRESTOタンゴ(パラレル受容体オーム発現および転写出力タンゴによるスクリーニング)を開発し、タンゴアプローチを使用して薬物可能GPCR-omeを並列かつ同時にプロファイルするハイスループットオープンソースプラットフォーム19を開発した。ほぼすべてのGPDRにβ-arrestin2の「無差別」募集を利用して、PRESTO-Tangoは細胞ベースの機能的アッセイの面で初めてのものである。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 一次スクリーニング:細胞培養およびプレートシード
- ポリL-リジン(PLL)コーティングされたプレートを調製するために、電子マルチチャネルピペットまたは試薬ディスペンサーを使用して白または黒の384ウェル光学底板のPLLの20 μg/mLストック溶液の20 μL/ウェルを分配する。プレートを室温で0.5~2時間インキュベートします。
注: 黒い 384 ウェル プレートを使用する場合は、白いプレートと比較してバックグラウンド信号が低いことを期待してください。隣接するウェル間の発光のブリードスルーを減らすために、ブラックプレートをお勧めします。 - コーティングされたプレートを保存し、余分なPLLを洗い流し、それを流しの上にフリックしてPLLを取り除き、ペーパータオルの上で乾かして、電子マルチチャネルピペットまたは試薬ディスペンサーを使用して抗生物質抗ミキミティックの希釈された1x溶液の40 μL/ウェルを加えます。PLLコーティングされたプレートは、プレートの播種の準備ができるまで4°Cで保管してください。
- HTLA細胞(リチャード・アクセル博士が親切に提供)を維持するヒト胚性腎臓細胞株(HEK293T)は、β-アレスティン2-TEVおよびtTA駆動ルシファーゼ-完全なダルベッコの修飾イーグル培地(DMEM)で胎児の血清の5%を補い、 牛子牛血清5%、ピューロマイシン2.5μg/mL、ハイグロマイシン50μg/mL、100 U/mLペニシリン、および100μg/mLストレプトマイシンを5%CO2を含む加湿インキュベーターで37°Cで。
- HTLA細胞を150mmの皿で培養し、希釈係数1:10で週2回、最適な細胞通過数5~25で細胞を通過させます。プライマリ画面の規模に応じて、150 mmの皿が384ウェルプレートシードの日にコンフルエントであることを確認してください。
注: HTLA 細胞の使用法が 25 より大きいと生存率が低下し、最適ではない結果が得られる可能性があります。 - プライマリスクリーン用にHTLA細胞をシードするには、1xリン酸緩衝生理食塩水(PBS)pH 7.4でコンフルエント150mm皿をそっと洗い流します。約6 mLのトリプシン/0.53 mM EDTAを持つ細胞を取り外し、少なくとも同量の完全なダルベックコの改変イーグル培地(DMEM)を含む遠心管に移してトリプシンを中和する。
- HTLA細胞を500xgで3分間スピンダウンし、完全なDMEM中の0.22 x 106細胞/mLの密度で細胞ペレットを再懸濁し、2.5μg/mLのピューロマイシンおよび50μg/mLのハイグロマイシンの添加を省略して、トランスフェクション効果を低下させる可能性があります。
- 必要な384ウェルPLLコーティングプレートを37°Cでインキュベートし、細胞を播種する前に温めます。384ウェルPLLコーティングプレートから1x抗生物質抗ミキティックの貯蔵溶液を取り除き、プレートをシンクの上にフリックし、ペーパータオルの上にテーピングして乾燥させます。
- 電子マルチチャネルピペットを使用して0.22 x 106セル/mL HTLA懸濁液の45 μLを分配することにより、最終的な密度10,000セル/ウェルで384ウェルPLLコーティングプレートに細胞をシードします。一晩で37°Cでプレートをインキュベートする。当日トランスフェクションが好ましい場合は、16,000個の細胞/ウェルの密度で細胞をシードし、4時間後にトランスフェクションを行う。
注:高いトランスフェクション効率のために、50-70%の細胞の合流度が最適です。
2. 一次スクリーニング:DNAプレート調製およびトランスフェクション
- 図2に示すようにトランスフェクション用の384ウェルDNAソースプレートを調製するには、目的のGPCR-Tango構造をコードするプラスミドcDNAを96ウェルプレートに、異なるGPCR/ウェルで配布します。プラスミドDNAは、50 ng/μLの濃度で0.1xトリスEDTA(TE)バッファーに懸濁する必要があります。
注:96ウェルDNAプレートは-20°Cで密閉保存し、複数のスクリーニング実験に再利用することができます。GPCR-タンゴコンストラクトをコードするすべてのcDNAは市販されており(材料表を参照)、pcDNA3.1ネオマイシンプラスミドでクローン化されています。PRESTOタンゴGPCRキットは、それぞれ80個のGPCR、陰性対照として空のベクターを持つウェルのカップル、ドーパミン受容体D2(DRD2)を保持する陽性対照井戸、およびトランスフェクション効率を追跡するために蛍光タンパク質(YFP)をコードするプラスミドを運ぶウェルを含む4つの96ウェルプレートで構成されています。 - マルチチャネルピペットを使用して、96ウェルから384ウェルDNAソースプレートに手動でDNA溶液を移し、384ウェルあたり10 μLを追加します。実験の各条件が四重化でアッセイされることを確実にするために、96ウェルDNAプレート(行A-DまたはE-H)の半分は、各GPCRを2つの象限(第1象限=-化合物、第2象限=+化合物)に分配することによって、完全な384ウェルプレートを覆う。
- Jordan et al.20:0.1x TE バッファー (1 mM Tris-HCl および 0.1 mM EDTA) で説明されているように、リン酸カルシウム沈殿法に必要な次のトランスフェクション試薬を組み立てます。2.5 M CaCl2ソリューション;2xヘップスバッファー、pH 7.05(50 mM HEPES、280 mM NaCl、1.5 mM Na2HPO4)。ろ過によりすべての溶液を殺菌し、4 °Cで保管してください。トランスフェクションの日は、試薬が使用前に室温に達することを可能にする。
- 0.1x TE (1:8 希釈) で 2.5 M CaCl2ストック溶液を希釈し、0.313 M CaCl2と渦の最終濃度にします。0.313 M CaCl2の40 μLを384ウェルDNAソースプレートに移し、ハンドヘルドマルチチャンネルピペットまたは自動ベンチトップ384チャンネルピペットで上下にピペットして混合します。
- 384ウェルDNAソースプレートに2x Hepesバッファーの50 μLを追加し、上下にピペットして再び混ぜ、1分間放置します。各384ウェルは、試験する必要がある化合物の数に応じて、9つの384ウェルプレートのトランスフェクションのための十分な量のDNA/トランスフェクション混合物を有する。384ウェルDNAソースプレートから播種されたHTLA細胞にDNA/トランスフェクション混合物の10 μLを移し、37°Cでプレートを一晩インキュベートします。
3. 一次スクリーニング:細胞刺激
- 24時間後、384ウェルプレートを流しの上にそっとフリックしてペーパータオルの上、または吸引器の頭部でテーピングして、トランスフェクトされた細胞培地をデカントします。40 μLの飢餓培地(DMEMは1%透析したウシ血清(dFBS)と1x抗生物質/抗ミコティック剤を添加し、細胞に直接触れないように注意する。
- 目的の化合物のピペット 20 μL を 3 倍の濃度 (細胞板内の薬物の最終濃度は 1x)、交互の行に (+) 刺激を与え、(-)化合物を含まない交互の行に対する車両バッファーの 20 μL。5%CO2で37°Cのセルプレートを戻し、少なくとも16時間インキュベートする。
4. 一次審査:発光読み取り
- ベーカーとボイス21から変更されたGlo試薬を準備する: 108 mM Tris–HCl;42 mM Tris-Base, 75 mM NaCl, 3 mM MgCl2, 5 mM ジチオスレイトール (DTT), 0.2 mM 補酵素 A, 0.14 mg/ml D-ルシフェリン, 1.1 mM ATP, 0.25% v/v トリトン X-100, 2 mM ハイドロスルフィット.
注:試薬のストック溶液は、D-ルシフェリンを除いて、事前に作ることができます。ブラックプレートを使用した場合、D-ルシフェリンの量を0.25mg/mLまで増やすことができます。 - 刺激の後の16-24時間で、384ウェルプレートを流しの上にそっとフリックし、ペーパータオルの上にテーピングすることによって、トランスフェクトされた細胞培地をデカントします。Glo試薬を20 μL/wellに加え、室温でプレートを5〜20分間インキュベートします。
5. 一次審査:データ分析
- 保存したファイルをルミネセンス カウンタからスプレッドシートとしてエクスポートします。結果は相対発光単位(RLU)で記録されます。384ウェルプレートのレイアウトに基づいて、次の式を使用して各受容体の活性化(折り畳み変化)を計算します。
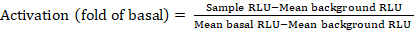
注:ここでサンプルRLUは、刺激された(+化合物)象限の4つの複製井戸のそれぞれの値を指し、平均背景RLUはプレート上の陰性対照の平均であり、平均基底RLUは、その同じ受容体の未処理の象限の平均を指します(- 化合物)。さらに、4つのデータポイントの標準偏差を計算して、結果の品質を検証します。変形性を是正するために、フォールドの平均に対して log2 変換を実行することをお勧めします。log2 ベースは、正のヒットを識別するのに役立つ実用的な選択肢です。経験的に正のヒットしきい値を設定します。いくつかの受容体は、2倍の増加と完全なアゴニストを持つ他の人のために最大40倍の増加まで低く持つことができることに留意する必要があります。 - 結果に基づいて、二次スクリーニングの潜在的な陽性ヒットであるGPCBを選択します。
6. 二次スクリーニング:細胞の播種およびトランスフェクション
- 完全培地(4.55 x 105/mL)の11mLで5 x 106細胞の総細胞密度で100mm皿のHTLA細胞をサブ培養し、37°Cで24時間培養します。当日トランスフェクションが好ましい場合、細胞の濃度が7.5 x106の細胞で種子を入れ、4時間後にトランスフェクションを行う。
- 室温でリン酸カルシウム沈殿に必要な試薬を事前に温めます。0.1x TE バッファの 450 μL と 2.5 M CaCl2の 50 μL と素早く渦を組み合わせます。これらの量は、それが保持する成長培地の量に基づいて、100ミリメートル皿に固有です。
- チューブ内で、TE/CaCl2溶液の500 μLをGPCR cDNAおよび渦の10 μgに加えます。チューブに500 μLの2x Hepesバッファー溶液を加え、激しく振り(渦を起こさない)、1分間インキュベートします。
注:蛍光タンパク質をコードするプラスミドの1μg(YFP、mCherryなど)は、GPCR cDNAの9 μgを合計10 μgで共にトランスフェクトすることができます。蛍光タンパク質はトランスフェクション効率を追跡するために使用され、この最小量はアッセイを妨げない。 - 短いインキュベーションの直後に、1 mL溶液を細胞に滴下して分配する。プレートをゆっくりと前後に揺らし、沈殿物を均等に分配し、プレートを旋回しないように注意し、37°Cで24時間インキュベートします。
- 翌日、蛍光細胞のイメージャー下で蛍光タンパク質の発現を見てトランスフェクション効率を観察する。50%を超えるトランスフェクションが理想的です。
- 必要な384ウェルPLLコーティングプレートを37°Cのインキュベーターにインキュベーターでインキュベートし、細胞を播種する前に温めます。384ウェルPLLコーティングプレートから1x抗生物質抗ミキティックの貯蔵溶液を取り除き、プレートをシンクの上にフリックし、ペーパータオルの上にテーピングして乾燥させます。
- トランスフェクトされた細胞をVersene溶液(1X PBS、pH 7.4;0.53 mM EDTA)で穏やかに洗い、0.05%トリプシン/0.53 mM EDTAの3 mLを皿に加えて取り外します。少なくとも同量の完全なDMEMを含む遠心管に内容物を移してトリプシンを中和する。
- 500 x gで細胞を 3 分間回転させ、飢えた培地で 0.4 x 106細胞/mL の密度でセルを再懸濁します。電子マルチチャネルパイプを使用して細胞懸濁液の45 μLを分配することにより、25,000セル/ウェルの最終密度で384ウェルPLLコーティングプレートに細胞をシードします。プレートを37°Cに最低4時間戻し、細胞が適切に井戸に付着してから刺激に進みます。
7. 二次スクリーニング:16点(半log)線量曲線のための薬物プレート調製
- 96ウェルプレートに、図4に示すように、プレートの最後の行(行H)を除く1X HBSS薬物緩衝液(1xハンクのバランス塩溶液[HBSS]、20 mM HEPES pH 7.4、1x抗生物質抗抗抗抗毒素)の270μLを加える。
注:ペプチド、コロイド分子、水溶性化合物の場合、0.1~1%BSAの添加が推奨されます。薬物の酸化を防ぐために、アスコルビン酸を0.01%まで添加することもできる。 - 薬物ストックから、最終的な3倍濃度(細胞板中の薬物の最終濃度は1倍となる)を計算して、薬物溶液(「高」濃度と称する)を調製する。例として、10 μMの最高濃度の線量応答曲線の場合は、30 μMで「高」濃度を調製します。行Hの井戸に「高い」濃度のピペット300 μL。
- 別のチューブでは、「高」濃度を3.16(ハーフログ)で割った「低」濃度を調製します。前の例に基づいて、「低」濃度は9.49 μMになります。「ハイ」ウェルに隣接する行Hのウェルに「低」濃度のピペット300 μL。
注: 行 H に必要な 96 ウェルの合計数は、細胞の数と刺激条件によって異なります。4つの井戸(2つの「高」と2つの「低」)は、全体の384ウェルプレートを刺激するための十分な薬物溶液を持つことになります。 - 「高」と「低」のウェルから前の行(行G)に30 μLの薬物溶液をピペット化し、手動で上下にピペット処理するか、推奨通りに「ピペットとミックス」機能を備えた電子マルチチャンネルピペットを使用して混合してシリアル希釈を行います。希釈の間のヒントを捨てながら、最初の最も希釈された行(行A)までこのステップを繰り返します。
注: 必要に応じて、シリアル希釈は、行 A の前に停止することができ、薬物のない内部コントロール、つまり「真のゼロ」を表します。 - 図4を参考に、96ウェルプレートから前に播種された384ウェルプレートの行A-Oに「低」カラム希釈液の20μLをピペット化し、さらに「ハイ」カラム希釈液の20μLをウェルB-Pに20μLずつインキュベートして、最低16時間でプレートを37°Cにインキュベートして、トランスフェクトされた細胞を刺激します。
8. 二次スクリーニング:発光読み取りとデータ分析
- 刺激の後の16-24時間で、384ウェルプレートを流しの上にそっとフリックし、ペーパータオルの上にテーピングすることによって、トランスフェクトされた細胞培地をデカントします。Glo試薬を20 μL/wellに加え、室温でプレートを5〜20分間インキュベートします。
- 保存したファイルをルミネセンス カウンタからスプレッドシートとしてエクスポートします。結果は相対発光単位(RLU)で記録されます。384 ウェル プレートのデータを統計ソフトウェアに転送し、非線形回帰曲線適合の組み込み XY 分析を使用して結果を分析します。組み込みの3-パラメータ線量応答刺激機能「Log(アゴニスト)対応答(3つのパラメータ)」を選択し、

注:ここでは、上と下はY軸の単位でプラトー、それぞれ最大応答と基底レベル、EC50は、上と下の間に50%の応答を生成するアゴニストの濃度であり、Xはアゴニストの対数濃度を指します。このモデルは、線量応答曲線の標準ヒル勾配が 1 であると仮定します。
Access restricted. Please log in or start a trial to view this content.
結果
本明細書で提示されたPRESTOタンゴプロトコルを用いて、クロマフィン顆粒(CG)抽出物を168の非嗅覚GPCR標的に対してスクリーニングし、その大半は孤児受容体である。上記の抽出物のプロファイリングは、選択された受容体におけるβ-arrestin2動員を調べることによって、Barneaららによって設計された原理に基づいて行われた(図1)。目的のGPCRのプラスミドc...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
立体構造の動的GPCRsは、信号伝達の大国です。これらのヘプタケリカル受容体の結合ポケットの物理化学的性質と、それらの生理学的関連性は、GPCRリガンドスクリーニングツールの必要性を強調している。上記のように、PRESTOタンゴアッセイは迅速で敏感でユーザーフレンドリーであり、医薬品開発に適しています。このアッセイはアゴニスト誘導活性化を測定するだけでなく、アンタゴニ?...
Access restricted. Please log in or start a trial to view this content.
開示事項
オートアワーは競合する利益を宣言しません。
謝辞
この研究は、カナダ保健研究所(CIHR助成金#MOP142219)によって支援されました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 384 Well Optical Bottom Plates, Polystyrene Polymer Base, Cell Culture Treated, BLACK, with lid, Sterile | NUNC | 12-566 | |
| 384 Well Optical Bottom Plates, Polystyrene Polymer Base, Cell Culture Treated, White, with lid, Sterile | NUNC | 12-566-1 | |
| 384 Well Round Bottom, Polypropylene, Non-Treated, Blue, non-sterile, without lid | ThermoFisher | 12-565-390 | |
| Antibiotic-Antimycotic | Wisent | 450-115-EL | |
| D-Luciferin, sodium salt | GoldBio | LUCNA | |
| DMEM with L-Glutamine, 4.5g/L Glucose and Sodium Pyruvate | Corning | 10-013-CV | |
| Eppendorf Xplorer, 12-channel, variable, 15–300 µL | Eppendorf | 4861000155 | |
| Eppendorf Xplorer, 12-channel, variable, 5–100 µL | Eppendorf | 4861000139 | |
| Matrix Platemate 2x3 | ThermoFisher | 801-10001 | |
| MicroBeta 1450 Wallac | Perkin Elmer | ||
| Penicilin-Streptomycin | Wisent | 450-201-EL | |
| Poly-L-Lysine hydrobromide | Millipore-Sigma | P2636-500MG | |
| Roth Lab PRESTO-Tango GPCR Kit | Addgene | Kit #1000000068 |
参考文献
- Liapakis, G., et al. The G-protein coupled receptor family: actors with many faces. Current pharmaceutical design. 18 (2), 175-185 (2012).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nature Reviews Molecular Cell Biology. 3 (9), 639-650 (2002).
- Kroeze, W. K., Sheffler, D. J., Roth, B. L. G-protein-coupled receptors at a glance. Journal of cell science. 116, Pt 24 4867-4869 (2003).
- Rask-Andersen, M., Almén, M. S., Schiöth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Ngo, T., et al. Identifying ligands at orphan GPCRs: current status using structure-based approaches. British journal of pharmacology. 173 (20), 2934-2951 (2016).
- Zhang, R., Xie, X. Tools for GPCR drug discovery. Acta pharmacologica Sinica. 33 (3), 372-384 (2012).
- Kimple, A. J., Bosch, D. E., Giguere, P. M., Siderovski, D. P. Regulators of G-Protein Signaling and Their G Substrates: Promises and Challenges in Their Use as Drug Discovery Targets. Pharmacological Reviews. 63 (3), 728-749 (2011).
- Wettschureck, N., Offermanns, S. Mammalian G Proteins and Their Cell Type Specific Functions. Physiological Reviews. 85 (4), 1159-1204 (2005).
- Denis, C., Saulière, A., Galandrin, S., Sénard, J. -M., Galés, C. Probing heterotrimeric G protein activation: applications to biased ligands. Current Pharmaceutical Design. 18 (2), 128-144 (2012).
- Yin, H., et al. Lipid G protein-coupled receptor ligand identification using beta-arrestin PathHunter assay. The Journal of Biological Chemistry. 284 (18), 12328-12338 (2009).
- Cheng, Z., et al. Luciferase Reporter Assay System for Deciphering GPCR Pathways. Current Chemical Genomics. 4, 84-91 (2010).
- Roth, B. L., Kroeze, W. K. Integrated Approaches for Genome-wide Interrogation of the Druggable Non-olfactory G Protein-coupled Receptor Superfamily. The Journal of Biological Chemistry. 290 (32), 19471-19477 (2015).
- Jean-Charles, P. -Y., Kaur, S., Shenoy, S. K. G Protein-Coupled Receptor Signaling Through β-Arrestin-Dependent Mechanisms. Journal of Cardiovascular Pharmacology. 70 (3), 142-158 (2017).
- Smith, J. S., Rajagopal, S. The β-Arrestins: Multifunctional Regulators of G Protein-coupled Receptors. The Journal of Biological Chemistry. 291 (17), 8969-8977 (2016).
- Böhme, I., Beck-Sickinger, A. G. Illuminating the life of GPCRs. Cell Communication and Signaling. 7 (1), 16(2009).
- Fang, Y. Label-Free Receptor Assays. Drug discovery today. Technologies. 7 (1), 5-11 (2011).
- Wang, T., et al. Measurement of β-Arrestin Recruitment for GPCR Targets. Assay Guidance Manual. Eli Lilly & Company and the National Center for Advancing Translational Sciences. , (2004).
- Barnea, G., et al. The genetic design of signaling cascades to record receptor activation. Proceedings of the National Academy of Sciences of the United States of America. 105 (1), 64-69 (2008).
- Kroeze, W. K., et al. PRESTO-Tango as an open-source resource for interrogation of the druggable human GPCRome. Nature Structural & Molecular Biology. 22 (5), 362-369 (2015).
- Jordan, M., Schallhorn, A., Wurm, F. M. Transfecting Mammalian Cells: Optimization of Critical Parameters Affecting Calcium-Phosphate Precipitate Formation. Nucleic Acids Research. 24 (4), 596-601 (1996).
- Baker, J. M., Boyce, F. M. High-throughput functional screening using a homemade dual-glow luciferase assay. Journal of Visualized Experiments. (88), 50282(2014).
- Li, H., Eishingdrelo, A., Kongsamut, S., Eishingdrelo, H. G-protein-coupled receptors mediate 14-3-3 signal transduction. Signal Transduction and Targeted Therapy. 1 (1), 16018(2016).
- Walther, C., Ferguson, S. S. G. Minireview: Role of intracellular scaffolding proteins in the regulation of endocrine G protein-coupled receptor signaling. Molecular Endocrinology. 29 (6), Baltimore, Md. 814-830 (2015).
- Gurevich, E. V., Benovic, J. L., Gurevich, V. V. Arrestin2 expression selectively increases during neural differentiation. Journal of Neurochemistry. 91 (6), 1404-1416 (2004).
- Shaikh, S., Nicholson, L. F. B. Optimization of the Tet-On system for inducible expression of RAGE. Journal of Biomolecular Techniques JBT. 17 (4), 283-292 (2006).
- Blau, H. M., Rossi, F. M. Tet B or not tet B: advances in tetracycline-inducible gene expression. Proceedings of the National Academy of Sciences of the United States of America. 96 (3), 797-799 (1999).
- Roche, O., et al. Development of a Virtual Screening Method for Identification of "Frequent Hitters" in Compound Libraries. Journal of Medicinal Chemistry. 45 (1), 137-142 (2002).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved