È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Interrogatorio parallelo del reclutamento di z-Arrestin2 per lo screening dei ligandi su una scala a livello GPCR utilizzando PRESTO-Tango Assay
In questo articolo
Riepilogo
Dato che i GPCR sono interessanti obiettivi drogabili, lo screening dei ligandi GPCR è quindi indispensabile per l'identificazione dei composti di piombo e per gli studi di deorfanotrizzazione. A fronte di questi sforzi, descriviamo PRESTO-Tango, una piattaforma di risorse open source utilizzata per la profilazione simultanea del reclutamento transitorio di z-arrestin2 a circa 300 GPCR utilizzando un test reporter basato su TEV.
Abstract
Essendo la più grande e versatile superfamiglia genica e mediatori di una serie di vie di segnalazione cellulare, i recettori accoppiati alle proteine G (GPCR) rappresentano uno degli obiettivi più promettenti per l'industria farmaceutica. Ergo, la progettazione, l'implementazione e l'ottimizzazione dei saggi di screening dei ligandi GPCR è fondamentale, in quanto rappresentano strumenti di controllo remoto per la scoperta di farmaci e per la manipolazione della farmacologia e dei risultati del GPCR. In passato, i saggi dipendenti dalla proteina G caratterizzavano quest'area di ricerca, rilevando eventi indotti dal ligando e quantificando la generazione di messaggeri secondari. Tuttavia, dall'avvento della selettività funzionale, così come una maggiore consapevolezza di diversi altri percorsi indipendenti dalla proteina G e dei limiti associati ai saggi dipendenti dalla proteina G, c'è una maggiore spinta verso la creazione di alternative saggi di screening dei ligandi GPCR. A questo scopo, descriviamo l'applicazione di una di queste risorse, la piattaforma PRESTO-Tango, un sistema di giornalista luciferata luciferasi che consente l'interrogatorio parallelo e simultaneo del GPCR-ome umano, un'impresa che in precedenza era considerata tecnicamente ed economicamente irrealizzabile. Sulla base di un saggio di reclutamento indipendente indipendente dalla proteina G-arrestin2, l'universalità della tratta mediata da z-arrestin2 e della segnalazione presso i GPCR rende PRESTO-TANGO uno strumento adatto per studiare circa 300 GPD umani non olfattivi, tra cui circa 100 recettori orfani. La sensibilità e la robustezza di PRESTO-Tango lo rendono adatto per schermi primari ad alto rendimento utilizzando librerie composte, impiegati per scoprire nuovi obiettivi GPCR per farmaci noti o per scoprire nuovi ligandi per i recettori orfani.
Introduzione
I recettori accoppiati alle proteine G (GPCR) costituiscono la più grande e diversificata famiglia di proteine transmembrane, operando come interfacce di comunicazione tra una cellula e il suo ambiente1. La versatilità dei GPR è evidenziata dalla loro capacità di rilevare una vasta gamma di ligandi – dai neurotrasmettitori ai nucleotidi, peptidi ai fotoni, e molti altri - così come la loro capacità di regolare numerose cascate di segnalazione a valle coinvolte nella crescita cellulare, migrazione, differenziazione, apoptosi, cottura cellulare, ecc2,3. Considerando la loro ubiquità e il coinvolgimento in una moltitudine di processi fisiologici, questa famiglia di recettori è della massima importanza terapeutica, dimostrare dal fatto che più di un terzo dei farmaci prescritti attualmente disponibili si rivolgono a GPCR4. Tuttavia, queste terapie esistenti si rivolgono solo a un piccolo sottoinsieme della superfamiglia (circa il 10%) e la farmacologia di molti GPCR rimane non chiarita. Inoltre, più di 100 GP esistono come recettori orfani, in quanto non sono stati abbinati a un ligando endogeno5. Pertanto, lo screening dei ligandi GPCR è fondamentale per la deorfamentizzazione e lo sviluppo di farmaci, in quanto spiana la strada verso la scoperta e l'ottimizzazione dei condotti e, eventualmente, verso la fase di sperimentazione clinica.
I metodi per lo screening dei ligandi GPCR sono tradizionalmente rientrati in una delle due categorie, le analisi funzionali indipendenti dalla proteina G o dalla proteina G6. La segnalazione di GPCR è regolata da proteine G eterotrimerici (G , g , che vengono attivate dallo scambio di GTP con il PIL vincolato alla sottounità7 diG . I segnali provenienti dal recettore attivato vengono trasdotti dalle proteine G tramite messaggeri secondari, come cAMP, Calcio, DAG e IP3, per mediare la segnalazione a valle agli effetti a valle8. La natura delle conseguenze funzionali della segnalazione delle proteine G è stata sfruttata per creare analisi basate sulle cellule che riflettono l'attivazione del recettore. Questi metodi, che misurano gli eventi prossimali (diretti) o distali (indiretti) nella segnalazione delle proteine G, sono più frequentemente utilizzati per lo screening dei ligandi GPCR e sono stati principalmente impiegati negli studi di deorfanotrizzazione6. Esempi di saggi che misurano direttamente l'attivazione delle proteine G mediate da GPCR includono il saggio vincolante [35S]GTP che misura il legame di un analogo GTP radioetichettato e non idrolizzabile alla sottounità di G, e il trasferimento di energia per la risonanza di F.risster/bioluminescenza (FRET/BRET, rispettivamente) sonde per monitorare le interazioni GPCR-G . I saggi che monitorano gli eventi distale sono gli strumenti più comunemente utilizzati per la profilazione GPCR; ad esempio, i saggi cAMP e IP1/3 misurano l'accumulo intracellulare di messaggeri secondari dipendenti dalla proteina G, mentre il flusso [Ca2]e i saggi di reporter che coinvolgono elementi di risposta specifici implicati nell'attivazione della proteina G (CRE, NFAT-RE, SRE, SRF-RE) esaminano ulteriormente gli eventi a valle della cascata di segnalazione11. Mentre la maggior parte dei suddetti saggi può essere eseguita a un livello di sono abbastanza sensibili, e vantano alcuni vantaggi specifici per l'analisi (ad esempio, la discriminazione tra agonisti completi/parziali, antagonisti neutri e agonisti inversi nel caso della rilegatura GTPS, o la funzionalità di analisi su cellule vive come [Ca2 ]e IP1/3)6, purtroppo non ci sono metodi dipendenti g-proteina esistenti che si adattino all'interrogatorio dell'intero GPCR-ome farmacologico. Ciò è in gran parte dovuto all'accoppiamento nativo di più sottofamiglie di proteine G ai GPCR, con conseguente segnalazione in diverse cascate e l'accoppiamento sconosciuto delle proteine G agli ORfani GPCR. Per mitigare questo problema, sono stati sviluppati saggi per forzare l'accoppiamento promiscuo di proteine G attraverso un'unica lettura comune di segnalazione, come il cAMP, e Ca2,anche se la maggior parte di essi sono a basso consumo12.
Un aspetto importante del ciclo di vita del GPCR è la cessazione della segnalazione dipendente dalle proteine G, che si verifica in gran parte attraverso il reclutamento di arresti z che induce ladissociazione della proteina G, e infine desensibilizzare il recettore, che è mirato per l'internalizzazione rivestita di clatra1in13 . Le isoforme più onnipresentisamente espresse di z-arrestin sono le non visive z-arrestin1 e z-arrestin2, indicate rispettivamente come arrestin-2 e arrestin-3, rispettivamente14. Inserisci i saggi basati sulle cellule indipendenti della proteina G, che aggiungono una nuova dimensione allo screening dei ligandi GPCR; il traffico di recettori, l'intera cellula senza etichetta e i saggi di reclutamento di arresto z sono tutti esempi degni di nota. I saggi di traffico di GPCR utilizzano ligandi con etichetta fluoroforo o anticorpi co-internalizzati che colpiscono il recettore15, mentre i saggi interi cellulari privi di etichetta utilizzano biosensori che traducono i cambiamenti cellulari indotti dal legame del ligando in output quantificabili, come i segnali elettrici o ottici16. In particolare, le interazioni per eccellenza nel GPCR- Il sistema Tango, sviluppato per la prima volta da Barnea et al. solo dieci anni fa, comporta l'introduzione di tre elementi genetici esogeni: una fusione proteica costituita da z-arrestin2 con proteasi del virus dell'incisione del tabacco (TEVp), un transattivatore di tetraciclina (tTA) legato a un GPCR tramite un virus dell'incisione del tabacco (TEVp), un transattivatore di tetraciclina (tTA) legato a un GPCR tramite un virus dell'incisione del tabacco sito di scissione di scissione (TEVcs) ed è preceduta da una sequenza del capoluogo C del recettore V2pressin (coda V2) per promuovere il reclutamento di arrestate, e un gene reporter luciferasi la cui trascrizione è innescata dal TTA fattore di trascrizione traslocazione al nucleo, che viene liberato dall'ancoraggio della membrana in seguito al reclutamento di z-arrestin2 (Figura 1)18. Le letture quantitative dell'attivazione della GPCR e del reclutamento di z-arrestin2 possono essere successivamente determinate leggendo la luminescenza. Una notevole distinzione è che mentre il traffico di recettori e i metodi interi cellulari privi di etichette sono relativamente a bassa velocità effettiva, il Tango ha diversi vantaggi, tra cui la lettura selettiva che è specifica per il recettore bersaglio e la sensibilità a causa dell'integrazione del segnale, che lo rendono un candidato adatto per lo screening dei ligandi su scala più ampia18.
In considerazione di queste caratteristiche strategiche, Kroeze e altri hanno sviluppato PRESTO-Tango (Parallel Receptor-ome Expression and Screening via Transcriptional Output-Tango), una piattaforma open source ad alta velocità che utilizza l'approccio Tango per profilare il GPCR-ome drogabile in modo parallelo e simultaneo19. Sfruttando il reclutamento "promiscuo" di z-arrestin2 in quasi tutti i GPR, PRESTO-Tango è il primo nel suo genere in termini di analisi funzionali basate sulle cellule, consentendo uno screening rapido "di primo round" di piccoli composti molecolari in quasi tutti i GPL non olfattivi, compresi gli orfani, indipendentedall'accoppiamento della sottofamiglia della proteina G.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Screening primario: coltura cellulare e semidici di placche
- Per preparare piastre rivestite in poli-L-lisina (PLL), erogare 20 gradi/pozzetto di una soluzione di riserva di 25 g/mL di PLL in piastre ottiche bianche o nere con un pipetta multicanale elettronico o un dispenser reagente. Incubare le piastre a temperatura ambiente per 0,5–2 h.
NOTA: Se si utilizzano le piastre nere 384-well, si aspettano che il segnale di sfondo sia inferiore rispetto alle piastre bianche. Si raccomanda nocedi nere per ridurre la smarginatura della luminescenza tra pozzi adiacenti. - Per conservare le piastre rivestite e lavare via il PLL in eccesso, rimuovere il PLL facendolo scorrere sul lavandino, toccare asciugare su un tovagliolo di carta e aggiungere 40 -L/pozza di soluzione 1x diluita di antibiotico-antimiotica utilizzando una pipetta multicanale elettronica o un dispenser reagente. Conservare le piastre rivestite di PLL a 4 gradi centigradi fino a quando non sono pronte per la semina delle piastre.
- Mantenere le cellule HTLA (gentilmente fornite dal Dr. Richard Axel) – una linea cellulare renale embrionale umana (HEK293T) che esprime stabilmente il 5% di fetale Bovine Serum, Il 5% del siero di vitello bovino, il 5,5 g/mL di puromicina, 50 g/mL di igromycin, 100 U/mL penicillina, e 100 g/mL streptomycin a 37 gradi centigradi in un incubatore umidizzato contenente 5% CO2.
- Coltura cellule HTLA in piatti 150 mm e passare le cellule due volte a settimana ad un fattore di diluizione di 1:10, con numero di passaggio cellulare ottimale di 5-25. Assicurarsi che un numero sufficiente di piatti da 150 mm sia confluente il giorno della semina a piastre 384-well, a seconda della scala dello schermo primario.
NOTA: l'uso di cellule HTLA superiori al passaggio 25 può comportare una riduzione della redditività, producendo risultati non ottimali. - Per semine di cellule HTLA per lo schermo primario, sciacquare delicatamente le parabole confluenti da 150 mm con 1x salina con buffer fosfato (PBS), pH 7.4. Staccare le cellule con circa 6 mL di 0,05% Trypsin/0,53 mM EDTA e trasferirle su un tubo di centrifuga contenente almeno la stessa quantità di Dulbecco del mezzo Eagle modificato (DMEM) di Dulbecco per neutralizzare la trypsina.
- Abbassare le cellule HTLA a 500 x g per 3 min e sospendere nuovamente il pellet cellulare ad una densità di 0,22 x 106 cellule/mL in DMEM completo, omettendo l'aggiunta di 2,5 g/mL di metina e 50 g/mL di igromicina in quanto possono diminuire l'efficacia della trasparenza.
- Incubare le necessarie piastre rivestite di PLL a 37 gradi per riscaldarle prima di seminare le cellule. Rimuovere la soluzione di stoccaggio di 1x antibiotic-antimycotic dalle lastre rivestite di 384 pozze ll facendo scorrere la piastra sul lavandino e toccandola su un tovagliolo di carta per asciugarla.
- Le cellule di semi nelle lastre rivestite PLL a 384 pozzetti a densità finale di 10.000 cellule/pozzo, erogando 45 -L delle sospensioni 0,22 x 106 celle/mL HTLA utilizzando un pipet multicanale elettronico. Incubare le piastre a 37 gradi durante la notte. Se si preferisce una trasfezione nello stesso giorno, le cellule di semi ad una densità di 16.000 cellule/bene ed eseguono la trasfezione 4 h più tardi.
NOTA: per un'elevata efficienza di trasfezione, la confluenza cellulare del 50-70% è ottimale.
2. Screening primario: preparazione della piastra del DNA e trasfetazioni
- Per preparare la lastra di origine del DNA di 384 pozzetto per la trasfezione, come illustrato nella Figura 2,distribuire i cDNA plasmid che codificano i costrutti GPCR-Tango di interesse in una lastra di 96 pozze, con un GPCR/well diverso. Il DNA del plasmide deve essere sospeso nel buffer Tris-EDTA (TE) da 0,1x ad una concentrazione di 50 ng/L.
NOTA: Le lastre di DNA del 96 po' possono essere sigillate e conservate a -20 gradi centigradi e riutilizzate per più esperimenti di screening. Tutti i costrutti GPCR-Tango di codifica cDNA sono disponibili in commercio (vedere la Tabella dei materiali) e sono clonati nel plasmide neomicina pcDNA3.1. Il PRESTO-Tango GPCR Kit è costituito da quattro piastre da 96 pozzetti, che includono 80 GPCR ciascuno, un paio di pozzi con un vettore vuoto come controlli negativi, e pozzi di controllo positivi che detengono il recettore della dopamina D2 (DRD2), e pozzi che trasportano una codifica plasmide una proteina fluorescente (YFP) per monitorare l'efficienza della trasfezione. - Utilizzando una pipetta multicanale, trasferire manualmente la soluzione del DNA dal pozzo 96 alla piastra di origine del DNA di 384 pozze, aggiungendo 10 L per 384-pozzetti. Per garantire che ogni condizione dell'esperimento sia analizzata in quadruplicato, metà della piastra di DNA a 96 ben (righe A-D o E-H) coprirà una piastra completa di 384 pozze didistribuzione di ogni GPCR in due quadranti (primo quadrante - - composto, secondo quadrante ) composto), in modo che lo stesso GPCR sarà trafitto in 8 pozzi della piastra 384-well (vedi Figura 2 come guida).
- Assemblare i seguenti reagenti di trasfezione necessari per il metodo di precipitazione del fosfato di calcio, come descritto da Jordan et al.20: 0.1x buffer TE (1 mM Tris-HCl e 0,1 mM EDTA); Soluzione CaCl2 da 2,5 M; 2x buffer di epes, pH 7,05 (50 mM HEPES, 280 mM NaCl, 1,5 mM Na2HPO4). Sterilizzare tutte le soluzioni filtrando e conservare a 4 gradi centigradi. Il giorno della trasfezione, consentire ai reagenti di raggiungere la temperatura ambiente prima dell'uso.
- Diluire la soluzione di 2,5 M CaCl2 in 0.1x TE (diluizione 1:8) per una concentrazione finale di 0.313 M CaCl2 e vortice. Trasferire 40 L di 0,313 M CaCl2 alla piastra di origine del DNA 384-well e mescolare pipetting su e giù con una pipetta multicanale portatile o un banco automatico 384-channel pipettor.
- Aggiungere 50 lof2x buffer di Hepes alla piastra di origine del DNA 384-well, mescolare di nuovo pipeting su e giù e lasciare riposare per 1 min; ogni 384-po 'di una quantità adeguata di miscela di DNA/trasfezione per la trasfezione di nove piastre 384-bene, a seconda del numero di composti che devono essere testati. Trasferire 10 L della miscela DNA/trasfezione dalla piastra di origine del DNA a 384 pozzetti alle cellule HTLA dei semi e incubare le piastre durante la notte a 37 gradi centigradi.
3. Screening primario: Stimolazione cellulare
- Ventiquattro ore dopo, decantare il supporto cellulare trascurato facendo scorrere delicatamente la piastra 384-well sopra il lavandino e toccandolo su un asciugamano di carta, o con una testa di aspiratore. Aggiungere lentamente 40 ll di supporti affamati (DMEM integrato con 1% diallyzed siero bovino fetale (dFBS) e 1x antibiotico/antimicotico), facendo attenzione a evitare di toccare direttamente le cellule.
- Pipet 20 - L of the compound of interest at a 3x concentration (la concentrazione finale del farmaco nella piastra cellulare sarà 1x) nelle file alternate con stimolazione (z) e 20 -L di cuscinetto del veicolo per le righe alternate senza (-) composto. Riportare la piastra cellulare a 37 gradi centigradi nel 5% di CO2 e incubare per almeno 16 h.
4. Screening primario: lettura della luminescenza
- Preparare il reagente Glo, modificato da Baker e Boyce21: 108 mM Tris–HCl; 42 mM Tris-Base, 75 mM NaCl, 3 mM MgCl2, 5 mM Dithiothrei-tol (DTT), 0,2 mM Coenzyme A, 0.14 mg/ml D-Luciferin, 1.1 mM ATP, 0.25% v/v Triton X-100, 2 mM Sodium
NOTA: le soluzioni di riserva dei reagenti possono essere fatte in anticipo, ad eccezione di D-Luciferin, che viene sempre appena aggiunto al reagente Glo nella sua forma in polvere. Se sono state utilizzate piastre nere, la quantità di D-Luciferin può essere aumentata fino a 0,25 mg/mL. - A 16-24 h dopo la stimolazione, decantare il supporto cellulare trascurato facendo scorrere delicatamente la piastra 384-po'sul lavandino e toccandola su un tovagliolo di carta. Aggiungete 20 gradi di reagente Glo e incubate la piastra a temperatura ambiente per 5-20 min. Leggete le piastre utilizzando un contatore di luminescenza a microlamila, con un tempo di integrazione di 1 s/well.
5. Screening primario: analisi dei dati
- Esportare i file salvati dal contatore della luminescenza come foglio di calcolo; i risultati saranno registrati in unità di luminescenza relative (RLU). In base al layout della piastra 384-pozze, calcolare l'attivazione (cambiamento di piega) di ogni recettore utilizzando la seguente formula:
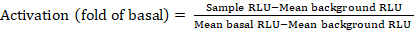
NOTA: Qui Sample RLU si riferisce al valore di ciascuno dei quattro pozzi replicati del quadrante stimolato (composto), RLU sfondo medio è la media dei controlli negativi sulla piastra, e basale medio RLU si riferisce alla media del quadrante non trattato di quello stesso recettore (- composto). Inoltre, calcolare la deviazione standard dei 4 punti dati per verificare la qualità dei risultati. Si consiglia di eseguire una trasformazione log2 sulla media delle modifiche di piegatura per rettificare qualsiasi eteroskedasticity; la base log2 è una scelta pratica per aiutare a identificare i risultati positivi. Empiricamente impostare le soglie di successo positivo; va notato che alcuni recettori possono avere basso come aumento di 2 volte e fino a 40 volte aumento per gli altri con agonista completa. - In base ai risultati, selezionare i GPCr che sono potenziali risultati positivi per lo screening secondario.
6. Screening secondario: semina cellulare e trasfetazioni
- Cellule HTLA di sottocoltura in piatti da 100 mm ad una densità cellulare totale di 5 x 106 cellule in 11 mL di supporti completi (4,55 x 105/mL) e incubare a 37 s per 24 h. Se si preferisce una trasfezione nello stesso giorno, le cellule di semi ad una densità di 7,5 x 106 cellule ed eseguono la trasfezione 4 h in un secondo momento.
- Preriscaldare i reagenti necessari per le precipitazioni di fosfato di calcio a temperatura ambiente. Unire 450 l l di 0,1 x di TE tampone con 50 , 2,5 M CaCl2 e rapidamente vortice; queste quantità sono specifiche per un piatto da 100 mm, in base al volume del mezzo di crescita che detiene.
- In un tubo, aggiungere 500 l della soluzione TE/CaCl2 a 10 g di GPCR cDNA e vortice. Aggiungere nel tubo 500 l di 2x soluzione cuscinetto per il tampone di epes, agitare vigorosamente (non vortice) e incubare per 1 min.
N.: 1 g di qualsiasi plasmide che codifica una proteina fluorescente (ad esempio YFP, mCherry, ecc.) può essere co-transfett con 9 g di CDNA GPCR per un totale di 10 g. La proteina fluorescente viene utilizzata per monitorare l'efficienza della trasfezione, e questa quantità minima non interferirà con il saggio. - Subito dopo la breve incubazione, dispensare la soluzione da 1 mL gocciola sulle cellule. Per sfoltire delicatamente la piastra avanti e indietro per distribuire uniformemente il precipitato, facendo attenzione a non ruotare la piastra, e incubare a 37 gradi centigradi per 24 ore.
- Il giorno seguente, osservare l'efficienza della trasfezione osservando l'espressione della proteina fluorescente sotto un imager cellulare fluorescente; trasfettazioni superiori al 50% di copertura sono l'ideale.
- Incubare le necessarie 384 catrine rivestite di PLL nell'incubatrice a 37 gradi centigradi per scaldarla prima di seminare le cellule. Rimuovere la soluzione di stoccaggio di 1x antibiotic-antimycotic dalle lastre rivestite di 384 pozze ll facendo scorrere la piastra sul lavandino e toccandola su un tovagliolo di carta per asciugarla.
- Sciacquare delicatamente le cellule trasinfettate con la soluzione Versene (1X PBS, pH 7.4; 0,53 mM EDTA) e staccarsi aggiungendo 3 mL di 0,05% di trypsin/0,53 mM EDTA al piatto. Trasferire il contenuto in un tubo di centrifuga contenente almeno una quantità uguale di DMEM completo per neutralizzare la trypsin.
- Ruotare le celle a 500 x g per 3 min e risospendere le celle ad una densità di 0,4 x 106 cellule / mL in supporti affamati. Le cellule di semi nelle lastre rivestite in PLL a 384 pozzetti rivestiti in PLL ad una densità finale di 25.000 cellule/bene erogando 45 ll della sospensione cellulare utilizzando un pipet multicanale elettronico. Riportare le piastre a 37 gradi centigradi per un minimo di 4 h, consentendo alle cellule di attaccarsi correttamente ai pozzi prima di procedere alla stimolazione.
7. Screening secondario: Preparazione della piastra di droga per la curva di dose a 16 punti (mezzo tronco)
- In una piastra di 96 pozze, aggiungere 270 l di 1X buffer di droga HBSS (1x Hank's Balanced Salt Solution [HBSS], 20 mM HEPES pH 7.4, 1x antibiotic-antimycotic), escludendo l'ultima riga (riga H) della piastra, come mostrato nella Figura 4.
NOTA: Per peptidi, molecole colloidali e composti scarsamente solubili in acqua, viene suggerita l'aggiunta di 0,1-1% di BSA. Per prevenire l'ossidazione dei farmaci, può essere aggiunto anche fino allo 0,01% acido ascorbico. - Dal broto del farmaco, preparare una soluzione farmacologica (indicata come la concentrazione "Alta") calcolando una concentrazione finale di 3x (la concentrazione finale del farmaco nella piastra cellulare sarà 1x). Ad esempio, per una curva dose-risposta con una concentrazione più alta di 10 M, preparare la concentrazione "Alta" a 30 M. Pipet 300 -L di concentrazione "Alta" in pozzi nella fila H.
- In un altro tubo, preparare la concentrazione "Bassa", che rappresenta la concentrazione "Alta" divisa per 3.16 (mezzo-log). Sulla base dell'esempio precedente, la concentrazione "Bassa" sarebbe di 9,49 M. Pipet 300 L di concentrazione "Bassa" in pozzi nella fila H, adiacenti ai pozzi "Alto".
NOTA: Il numero totale di 96-pozzi necessari nella fila H dipenderà dal numero di cellule e dalle condizioni di stimolazione. Quattro pozzi (due "Alto" e due "Basso") avrà un'ampia soluzione farmacologica per stimolare un intero 384 bene piatto. - Eseguire una diluizione seriale con il pipettaggio di 30 l di soluzione farmacologica dai pozzetti "Alto" e "Basso" della riga H alla riga precedente (riga G) e mescolare pipettando manualmente su e giù, o come consigliato, utilizzando una pipetta elettronica multicanale con la funzione "Pipette and Mix". Ripetere questo passaggio fino a quando la prima e più diluita riga (riga A), scartando le punte tra le diluizioni.
NOTA: se lo si desidera, le diluizioni seriali possono essere interrotte prima della riga A, che rappresenta un controllo interno senza alcun farmaco, in altre parole, un "true zero". - Utilizzando la Figura 4 come riferimento, stimolare le cellule trasfette con la pipipipenza di 20 gradi l della colonna "Bassa" diluizioni dalla piastra del pozzo 96 alle file A-O della piastra precedentemente semistata 384-well, così come 20L delle diluizioni di colonna "Alta" ai pozzi B-P. Incubare la piastra a 37 gradi centigradi per un minimo di 16 h.
8. Screening secondario: lettura della luminescenza e analisi dei dati
- A 16-24 h dopo la stimolazione, decantare il supporto cellulare trascurato facendo scorrere delicatamente la piastra 384-po'sul lavandino e toccandola su un tovagliolo di carta. Aggiungete 20 gradi di reagente Glo e incubate la piastra a temperatura ambiente per 5-20 min. Leggete le piastre utilizzando un contatore di luminescenza a microlamila, con un tempo di integrazione di 1 s/well.
- Esportare i file salvati dal contatore della luminescenza come foglio di calcolo; i risultati saranno registrati in unità di luminescenza relative (RLU). Trasferisci i dati della piastra 384-po ' a un software di statistiche per analizzare i risultati utilizzando la sua analisi XY integrata per adattarsi alla curva di regressione non lineare. Selezionare la funzione di stimolazione dose-risposta a 3 parametri integrata "Log(agonist) vs risposta (tre parametri)",

NOTA: Qui Top e Bottom sono altipiani nelle unità dell'asse Y, rispettivamente la risposta massima e il livello basale, EC50 è la concentrazione dell'agonista che genera una risposta del 50% tra Superiore e Inferiore, e X si riferisce alla concentrazione di log dell'agonista. Questo modello presuppone che la curva dose-risposta abbia una pendenza Hill standard di 1.
Access restricted. Please log in or start a trial to view this content.
Risultati
Utilizzando il protocollo PRESTO-Tango presentato nel presente documento, un estratto di granulo cromaffina (CG) è stato esaminato contro 168 obiettivi GPCR non olfattivi, con la maggior parte dei quali sono recettori orfani. La profilazione di tale estratto è stata eseguita esaminando la mobilitazione di arresto z2 presso i recettori scelti, sulla base del principio progettato da Barnea et al.18 (Figura 1). Plasmid cDNA dei GPCR di interesse è stato preso dal kit ...
Access restricted. Please log in or start a trial to view this content.
Discussione
I GPCR conformazionalmente dinamici sono centrali elettriche di trasduzione del segnale. Le proprietà fisiochimiche delle tasche di legame di questi recettori eptahelici, così come la loro rilevanza fisiologica sottolineano la necessità di strumenti di screening dei ligandi GPCR. Come presentato in precedenza, l'analisi PRESTO-Tango è rapida, sensibile e user-friendly, prestandosi allo sviluppo di farmaci. Non solo questo saggio misura l'attivazione indotta dagli agonisti, ma può anche essere utilizzata per quantifi...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli authour non dichiarano interessi concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto dai Canadian Institutes of Health Research (CIHR grant #MOP142219).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 384 Well Optical Bottom Plates, Polystyrene Polymer Base, Cell Culture Treated, BLACK, with lid, Sterile | NUNC | 12-566 | |
| 384 Well Optical Bottom Plates, Polystyrene Polymer Base, Cell Culture Treated, White, with lid, Sterile | NUNC | 12-566-1 | |
| 384 Well Round Bottom, Polypropylene, Non-Treated, Blue, non-sterile, without lid | ThermoFisher | 12-565-390 | |
| Antibiotic-Antimycotic | Wisent | 450-115-EL | |
| D-Luciferin, sodium salt | GoldBio | LUCNA | |
| DMEM with L-Glutamine, 4.5g/L Glucose and Sodium Pyruvate | Corning | 10-013-CV | |
| Eppendorf Xplorer, 12-channel, variable, 15–300 µL | Eppendorf | 4861000155 | |
| Eppendorf Xplorer, 12-channel, variable, 5–100 µL | Eppendorf | 4861000139 | |
| Matrix Platemate 2x3 | ThermoFisher | 801-10001 | |
| MicroBeta 1450 Wallac | Perkin Elmer | ||
| Penicilin-Streptomycin | Wisent | 450-201-EL | |
| Poly-L-Lysine hydrobromide | Millipore-Sigma | P2636-500MG | |
| Roth Lab PRESTO-Tango GPCR Kit | Addgene | Kit #1000000068 |
Riferimenti
- Liapakis, G., et al. The G-protein coupled receptor family: actors with many faces. Current pharmaceutical design. 18 (2), 175-185 (2012).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nature Reviews Molecular Cell Biology. 3 (9), 639-650 (2002).
- Kroeze, W. K., Sheffler, D. J., Roth, B. L. G-protein-coupled receptors at a glance. Journal of cell science. 116, Pt 24 4867-4869 (2003).
- Rask-Andersen, M., Almén, M. S., Schiöth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Ngo, T., et al. Identifying ligands at orphan GPCRs: current status using structure-based approaches. British journal of pharmacology. 173 (20), 2934-2951 (2016).
- Zhang, R., Xie, X. Tools for GPCR drug discovery. Acta pharmacologica Sinica. 33 (3), 372-384 (2012).
- Kimple, A. J., Bosch, D. E., Giguere, P. M., Siderovski, D. P. Regulators of G-Protein Signaling and Their G Substrates: Promises and Challenges in Their Use as Drug Discovery Targets. Pharmacological Reviews. 63 (3), 728-749 (2011).
- Wettschureck, N., Offermanns, S. Mammalian G Proteins and Their Cell Type Specific Functions. Physiological Reviews. 85 (4), 1159-1204 (2005).
- Denis, C., Saulière, A., Galandrin, S., Sénard, J. -M., Galés, C. Probing heterotrimeric G protein activation: applications to biased ligands. Current Pharmaceutical Design. 18 (2), 128-144 (2012).
- Yin, H., et al. Lipid G protein-coupled receptor ligand identification using beta-arrestin PathHunter assay. The Journal of Biological Chemistry. 284 (18), 12328-12338 (2009).
- Cheng, Z., et al. Luciferase Reporter Assay System for Deciphering GPCR Pathways. Current Chemical Genomics. 4, 84-91 (2010).
- Roth, B. L., Kroeze, W. K. Integrated Approaches for Genome-wide Interrogation of the Druggable Non-olfactory G Protein-coupled Receptor Superfamily. The Journal of Biological Chemistry. 290 (32), 19471-19477 (2015).
- Jean-Charles, P. -Y., Kaur, S., Shenoy, S. K. G Protein-Coupled Receptor Signaling Through β-Arrestin-Dependent Mechanisms. Journal of Cardiovascular Pharmacology. 70 (3), 142-158 (2017).
- Smith, J. S., Rajagopal, S. The β-Arrestins: Multifunctional Regulators of G Protein-coupled Receptors. The Journal of Biological Chemistry. 291 (17), 8969-8977 (2016).
- Böhme, I., Beck-Sickinger, A. G. Illuminating the life of GPCRs. Cell Communication and Signaling. 7 (1), 16(2009).
- Fang, Y. Label-Free Receptor Assays. Drug discovery today. Technologies. 7 (1), 5-11 (2011).
- Wang, T., et al. Measurement of β-Arrestin Recruitment for GPCR Targets. Assay Guidance Manual. Eli Lilly & Company and the National Center for Advancing Translational Sciences. , (2004).
- Barnea, G., et al. The genetic design of signaling cascades to record receptor activation. Proceedings of the National Academy of Sciences of the United States of America. 105 (1), 64-69 (2008).
- Kroeze, W. K., et al. PRESTO-Tango as an open-source resource for interrogation of the druggable human GPCRome. Nature Structural & Molecular Biology. 22 (5), 362-369 (2015).
- Jordan, M., Schallhorn, A., Wurm, F. M. Transfecting Mammalian Cells: Optimization of Critical Parameters Affecting Calcium-Phosphate Precipitate Formation. Nucleic Acids Research. 24 (4), 596-601 (1996).
- Baker, J. M., Boyce, F. M. High-throughput functional screening using a homemade dual-glow luciferase assay. Journal of Visualized Experiments. (88), 50282(2014).
- Li, H., Eishingdrelo, A., Kongsamut, S., Eishingdrelo, H. G-protein-coupled receptors mediate 14-3-3 signal transduction. Signal Transduction and Targeted Therapy. 1 (1), 16018(2016).
- Walther, C., Ferguson, S. S. G. Minireview: Role of intracellular scaffolding proteins in the regulation of endocrine G protein-coupled receptor signaling. Molecular Endocrinology. 29 (6), Baltimore, Md. 814-830 (2015).
- Gurevich, E. V., Benovic, J. L., Gurevich, V. V. Arrestin2 expression selectively increases during neural differentiation. Journal of Neurochemistry. 91 (6), 1404-1416 (2004).
- Shaikh, S., Nicholson, L. F. B. Optimization of the Tet-On system for inducible expression of RAGE. Journal of Biomolecular Techniques JBT. 17 (4), 283-292 (2006).
- Blau, H. M., Rossi, F. M. Tet B or not tet B: advances in tetracycline-inducible gene expression. Proceedings of the National Academy of Sciences of the United States of America. 96 (3), 797-799 (1999).
- Roche, O., et al. Development of a Virtual Screening Method for Identification of "Frequent Hitters" in Compound Libraries. Journal of Medicinal Chemistry. 45 (1), 137-142 (2002).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon