JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
PRESTO-탱고 분석기를 사용하여 GPCR 전체 규모의 리간드 스크리닝을 위한 β-Arrestin2 모집의 병렬 심문
요약
GPCRs가 매력적인 약물 성 표적이라는 점을 감안할 때, GPCR 리간드 스크리닝은 따라서 납 화합물의 식별및 탈판화 연구에 필수적입니다. 이러한 노력에 대해, 우리는 PRESTO-탱고, TEV 기반 기자 분석기를 사용하여 약 300 GPCRs에서 과도 β arrestin2 모집의 동시 프로파일링에 사용되는 오픈 소스 자원 플랫폼을 설명합니다.
초록
세포 신호 경로의 영역의 가장 크고 가장 다양한 유전자 슈퍼 패밀리 및 중재자로서, G 단백질 결합 수용체 (GPCRs)는 제약 산업에 대한 가장 유망한 목표 중 하나를 나타냅니다. GpCR 리간드 스크리닝 분석의 설계, 구현 및 최적화는 신약 발견및 GPCR 약리학 및 결과 조작을 위한 원격 제어 도구를 나타내므로 매우 중요합니다. 과거에, G 단백질 의존적인 희구한 조사는 연구의 이 지역을, 리간드 유도한 사건을 검출하고 이차 메신저의 생성을 정량화했습니다. 그러나, 기능적 선택성의 출현 이후, 뿐만 아니라 여러 다른 G 단백질 독립적 인 경로의 증가 인식과 G 단백질 의존적 인 반응성 반응성 반응성 반응제와 관련된 제한, 대안의 생성을 향한 더 큰 추진이있다 GPCR 리간드 스크리닝 검거 검사. 이러한 노력에 대해, 우리는 이러한 자원 중 하나의 응용 프로그램을 설명, PRESTO-탱고 플랫폼, 인간 GPCR-ome의 병렬 및 동시 심문을 가능하게 하는 luciferase 기자 기반 시스템, 이전에 고려 된 위업 기술적, 경제적으로 실현 불가능합니다. G-단백질 독립적인 β-arrestin2 모집 분석에 근거하여, GPCRs에서 β-arrestin2 중재된 인신 매매 및 신호의 보편성은 PRESTO-TANGO를 대략 100를 포함하여 대략 300의 비 후각 인간 GPCRs를 공부하기위한 적당한 공구를 만듭니다 고아 수용체. PRESTO-Tango의 감도와 견고성은 알려진 약물에 대한 새로운 GPCR 표적을 발견하거나 고아 수용체에 대한 새로운 리간드를 발견하기 위해 사용되는 화합물 라이브러리를 사용하여 1 차적인 고처리량 스크린에 적합합니다.
서문
G 단백질 결합 수용체 (GPCRs)는 세포와 그 환경 사이의 통신 인터페이스로 작동하는 가장 크고 가장 다양한 세포 막 단백질 제품군을 구성합니다1. GPCRs의 다양성은 신경 전달 물질에서 뉴클레오티드, 펩티드, 그리고 더 많은 -뿐만 아니라 세포 성장에 관련된 수많은 다운 스트림 신호 캐시드를 조절하는 능력뿐만 아니라, 세포 성장, 이동, 분화, 세포 발사, 등 등 리간드의 다양한 배열을 감지 할 수있는 능력에 의해 강조된다2,3. 생리적 과정의 무리에 그들의 보편성과 관여를 고려, 이 수용체 가족은 현재 유효한 처방 된 약물의 3 분의 1 이상이 GPCRs4를대상으로한다는 사실에 의해 전시, 가장 치료 중요성이다. 그러나, 이러한 기존 치료는 슈퍼 패밀리의 작은 하위 집합을 대상으로 (추정 된 10%), 그리고 많은 GPCRs의 약리학 은 elucidated 남아. 더욱이, 100개 이상의 GPCR은 내인성 리간드5와일치하지 않았기 때문에 고아 수용체로서 존재한다. 따라서, GPCR 리간드 스크리닝은 납 발견 및 최적화를 향한 길을 열어주며, 아마도 임상 시험 단계로 가는 데 있어 매우 중요합니다.
GPCR 리간드 스크리닝을 위한 방법은 전통적으로 G 단백질 의존성 또는 G-단백질 독립적인 기능성 검사법6의두 가지 범주 중 하나에 빠졌다. GPCR 신호는 이종성 G 단백질(Gαβγ)에 의해 조절되며, 이는 Gα 서브유닛7에결합된 GDP에 대한 GTP의 교환에 의해 활성화된다. 활성화된 수용체로부터의 신호는 cAMP, 칼슘, DAG 및 IP3와 같은 보조 메신저를 통해 G-단백질에 의해 변환되어 다운스트림 이펙터에서 다운스트림 신호를중재한다 8. G 단백질 신호의 기능적 결과의 본질은 수용체 활성화를 반영하는 세포 기지를 둔 분석문을 만들기 위하여 이용되었습니다. 이러한 방법은, G-단백질 신호에서 근위(direct) 또는 원위(direct) 사건을 측정하는, GPCR 리간드 스크리닝에 가장 빈번하게 사용되며 주로 탈판화 연구에서 사용되어 왔다6. GPCR 매개 G 단백질 활성화를 직접 측정하는 분석법의 예는 [35S]GTPγS 결합 분석, 이는 Gα 소단위로 방사성 표지 및 비 가수분해성 GTP 아날로그의 결합을 측정하고, Förster/bio luminescence 공명 에너지 전달(FRET/BRET, 각각) 프로브를 측정하여 GPCR-Gα 및 Gα/Gγ 상호작용을 모니터링하는 프로브로, 이는9,10년동안 더 많은 견인력을 얻고 있다. 말단 이벤트를 모니터링하는 assays는 GPCR 프로파일링에 가장 일반적으로 사용되는 도구입니다. 예를 들어, cAMP 및 IP1/3 측정은 G-단백질 의존이 되는 이차 메신저의 세포내 축적을 측정하는 반면, [Ca2+]플럭스 및 리포터 어설션은 G-단백질 활성화에 연루된 특정 반응 요소(CRE, NFAT-RE, SRE, SRF-RE)가 시그널링캐스케이드(11)를더 하류로 검사하는 경우이다. 전술한 대부분의 검사는 처리량이 높은 수준에서 수행될 수 있지만, 상당히 민감하고, 특정 분석특이 특이적 이점(예를 들어, GTPγS 결합의 경우 전체/부분 작용제, 중성 길항제 및 역 작용제 간의 차별, 또는 [Ca2+ ]및 IP1/3)와 같은 라이브 세포에 대한 분석 기능6,불행히도 전체 약물 의 심문에 부합하는 기존의 G-단백질 의존적 방법이 없다. 이것은 GPCRs에 다중 G 단백질 하위 가족의 네이티브 결합에 크게 기인합니다, 고아 GPCRs에 몇몇 폭포 및 알려지지 않은 G 단백질 결합에 신호의 결과로. 이 문제점을 완화하기 위하여는, asays는 cAMP와 같은 단 하나 일반적인 신호 판독을 통해 무차별적인 G 단백질 결합을 강제하기 위하여 개발되었습니다, 및Ca2+,이기는 하지만 그들 대부분은 낮은 처리량12입니다.
GPCR 수명 주기의 중요한 양상은 G-단백질-의존성 신호전달의 종결이며, 이는 G-단백질의 해리를 유도하는 β-arrestins의 모집을 통해 상당 부분 발생하고, 궁극적으로 는 클라트린 코팅 내재화를 대상으로 하는 수용체를 둔감화한다13. β-arrestin의 가장 유비쿼터스 표현된 이소폼은 비시각적 β-arrestin1 및 β-arrestin2, 또한 체포-2 및 체포-3로 각각14로표기된다. GPCR 리간드 스크리닝에 새로운 차원을 추가하는 G-단백질 독립적인 세포 기반 검혈을 입력합니다; 수용체 인신 매매, 라벨없는 전체 세포, β-arrestin 모집 세포는 모두 주목할만한 예입니다. GPCR trafficking assays employ fluorophore-labeled ligands or co-internalized antibodies targeting the receptor15, whereas label-free whole cell assays use biosensors which translate cellular changes induced by ligand binding into quantifiable outputs, such as electrical or optical signals16. 특히, 전형적인 GPCR-β-arrestin 상호작용은 β-arrestin 모집 분석법의 레퍼토리에서 매력적인도구로서 17. 불과 10년 전, 바네아 외가 처음 개발한 탱고 시스템, 담배 에칭 바이러스 프로테아제 (TEVp)와 β-arrestin2로 구성된 단백질 융합 , 담배 등 통해 GPCR에 묶여테트라 사이클린 형질 전환기 (tTA)의 세 가지 외인성 유전 요소의 도입을 포함한다 바이러스 프로테아제 분열 부위(TEVcs)는 V2 바소프레신 수용체(V2 tail)의 C-종단으로부터의 서열에 선행되어 체포모집을 촉진하고, 그의 전사가 발동되는 기자 루시퍼라제 유전자는 tTA 전사 인자는 β-arrestin2 모집에 따라 막 앵커링으로부터 해방되는 핵으로전좌(도1)18. GPCR 활성화 및 β-arrestin2 모집의 정량 적 판독은 발광을 위해 판독에 의해 이후에 결정될 수 있다. 주목할 만한 구별은 수용체 인신 매매 및 라벨없는 전체 세포 방법이 상대적으로 낮은 처리량이지만, 탱고는 신호 통합으로 인한 표적 수용체 및 감도에 특이적인 선택적 판독을 포함하여 몇 가지 장점을 가지고 있으며, 이는 더 큰 규모의 리간드 스크리닝에 적합한후보(18)이다.
이러한 전략적 특징을 고려하여, 크로에즈 등은 PRESTO-Tango(병렬 수용체-오메 발현 및 전사 출력 탱고를 통한 스크리닝)를 개발했으며, 탱고 접근법을 사용하여 약물성 GPCR-ome을 병렬 및 동시 방식으로 프로파일링하는 고처리량 오픈 소스플랫폼19를개발했다. 거의 모든 GPCRs에 β-arrestin2의 "무차별"모집을 이용, PRESTO-탱고는 세포 기반 기능 분석의 측면에서 최초의 종류입니다, 거의 모든 비 후각 GPCRs에서 작은 분자 화합물의 빠른 "첫 번째 라운드"스크리닝을 가능하게, 고아를 포함, G-단백질 하위 가족 커플링과 무관.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 1 차 선별 : 세포 배양 및 플레이트 시딩
- 폴리-L-리신(PLL) 코팅 플레이트를 준비하려면 전자 멀티채널 파이펫 또는 시약 디스펜서를 사용하여 흰색 또는 검은색 384웰 광학 바닥 플레이트에 PLL의 25 μg/mL 스톡 솔루션의 20 μL/well을 분배합니다. 0.5-2 시간 동안 실온에서 플레이트를 배양하십시오.
참고: 검정색 384웰 플레이트를 사용하는 경우 흰색 플레이트에 비해 배경 신호가 낮아질 것으로 예상합니다. 검은 판은 인접한 우물 사이의 발광의 출혈을 줄이기 위해 권장됩니다. - 코팅된 플레이트를 보존하고 여분의 PLL을 씻어내려면 싱크대 위로 가볍게 치우고 종이 타월 위에 두드려 PLL을 제거하고 전자 멀티채널 파이펫 또는 시약 디스펜서를 사용하여 희석된 1x 용액 의 40 μL/well을 추가합니다. 플레이트 파종을 위해 준비가 될 때까지 PLL 코팅 플레이트를 4°C에서 보관하십시오.
- HTLA 세포 유지 (리처드 악셀 박사에 의해 친절하게 제공)-인간 배아 신장 세포주 (HEK293T) 안정적으로 β-arrestin2-TEV 및 tTA 구동 루시퍼라제를 발현 -완전한 덜베코의 수정 된 독수리의 배지 (DMEM) 태아 소 소 세럼의 5 %로 보충, 소 송아지 세럼의 5%, 2.5 μg/mL의 푸로마이신, 50 μg/mL의 히그로마이신, 100 U/mL 페니실린, 37°C에서 100 μg/mL 스트렙토마이신을 함유하는 가습 된 인큐베이터에서 5%CO2를함유하고 있다.
- 배양 HTLA 세포는 150 mm 접시에 있고 1:10의 희석 인자에서 일주일에 두 번 세포를 통과시키고, 최적의 세포 전달 수 5-25의 수를 갖는다. 1차 화면의 규모에 따라 384웰 플레이트 파종의 날에 충분한 수의 150mm 접시가 동시제공되도록 하십시오.
참고: 통로 25보다 큰 HTLA 세포를 사용하면 생존율이 저하되어 최적이 아닌 결과를 얻을 수 있습니다. - 1차 스크린을 위한 HTLA 세포를 시드하려면, 1x 인산완충식염수(PBS), pH 7.4로 150mm 접시(들)를 부드럽게 헹구십시오. 약 6 mL의 트립신/0.53 mM EDTA로 세포를 분리하고, 트립신을 중화시키기 위해 완전한 덜벡코의 변형된 이글 배지(DMEM)를 적어도 동일한 양을 포함하는 원심분리관으로 이송한다.
- HTLA 세포를 500 x g에서 3분 동안 스핀다운하고 완전한 DMEM에서 0.22 x 106 세포/mL의 밀도로 세포 펠릿을 다시 일시 중단하고, 2.5 μg/mL의 퓨로마이신과 50 μg/mL의 보습 효능을 감소시킬 수 있습니다.
- 37°C에서 필요한 384웰 PLL 코팅 플레이트를 배양하여 파종 세포 전에 따뜻하게 합니다. 384웰 PLL 코팅 플레이트에서 1x 항생제 항균제의 저장 용액을 싱크대 위로 가볍게 치고 종이 타월 위에 테이핑하여 건조시다.
- 종자 세포는 전자 멀티채널 파이펫을 사용하여 0.22 x 106 셀/mL HTLA 현탁액의 45 μL을 분배함으로써 10,000개의 최종 밀도에서 384웰 PLL 코팅 플레이트내로 코팅되었습니다. 하룻밤 동안 37 °C에서 플레이트를 배양합니다. 당일 형질감염이 바람직한 경우, 종자 세포는 16,000 개의 밀도에서 잘 및 형질 감염을 수행 4 시간 후.
참고: 높은 형질 감염 효율을 위해 50-70% 세포 융합이 최적입니다.
2. 1 차 선별 : DNA 판 준비 및 형질 전환
- 그림 2에나타난 바와 같이 형질을 위한 384웰 DNA 소스 플레이트를 준비하기 위해, 다른 GPCR/well을 가진 96웰 플레이트에 관심 있는 GPCR-탱고 구조를 인코딩하는 플라스미드 cdDNA를 분배한다. 플라스미드 DNA는 50 ng/μL의 농도에서 0.1x Tris-EDTA(TE) 완충액으로 중단되어야 합니다.
참고: 96웰 DNA 플레이트는 -20°C에서 밀봉 및 보관할 수 있으며, 여러 스크리닝 실험에 재사용할 수 있습니다. GPCR-탱고 구문을 인코딩하는 모든 cDNA는 시판되고(재료 표참조) pcDNA3.1 네오마이신 플라스미드에서 복제됩니다. PRESTO-Tango GPCR 키트는 각각 80개의 GPRS, 음의 대조군으로 빈 벡터가 있는 웰 몇 개, 도파민 수용체 D2(DRD2)를 보유하는 양성 대조군 웰, 형광 효율을 추적하기 위해 형광 단백질(YFP)을 코딩하는 플라스미드를 운반하는 웰을 포함하는 4개의 96웰 플레이트로 구성됩니다. - 다중 채널 파이펫을 사용하여 96 웰에서 384 웰 DNA 소스 플레이트로 DNA 용액을 수동으로 이송하고 384 웰 당 10 μL을 추가합니다. 실험의 각 조건이 4중 으로 분석되도록 하기 위해, 96웰 DNA 플레이트(행 A-D 또는 E-H)의 절반은 2개의 사분면(제1 사분면 =- 화합물, 제2 사분면 =+ 화합물)에 각 GPCR을 분배함으로써 전체 384웰 플레이트를 커버할 것이며, 그러한 GPCR이 384-도2의 8웰에서 형질화될 것이다(도2 참조).
- 요르단 외20: 0.1x TE 완충액 (1 mM Tris-HCl 및 0.1 mM EDTA)에 의해 설명된 바와 같이 칼슘 인산염 침전 방법에 필요한 다음과 같은 형질 감염 시약을 조립한다; 2.5 M CaCl2 용액; 2x 헤페스 버퍼, pH 7.05 (50 mM HEPES, 280 mM NaCl, 1.5 mM Na2HPO4). 여과하여 모든 솔루션을 살균하고 4 °C에 보관하십시오. 형질전환의 날, 시약이 사용하기 전에 실온에 도달 할 수 있습니다.
- 2.5M CaCl2 스톡 용액을 0.1x TE(1:8 희석)로 희석하여 0.313 M CaCl2 및 와류의 최종 농도로 희석합니다. 40 μL의 0.313 M CaCl2를 384웰 DNA 소스 플레이트에 옮기고 핸드헬드 멀티채널 파이펫 또는 자동 벤치탑 384채널 파이펫으로 위아래로 파이펫팅하여 혼합합니다.
- 384 웰 DNA 소스 플레이트에 2x Hepes 버퍼 50 μL을 추가하고 위아래로 파이펫팅하여 다시 혼합하고 1 분 동안 방치하십시오. 각 384-well은 시험해야 하는 화합물의 수에 따라 9개의 384웰 플레이트의 형질감염에 대해 적절한 양의 DNA/형질감염 혼합물을 갖게 됩니다. 384웰 DNA 소스 플레이트로부터 DNA/형질감염 혼합물의 10 μL을 시드HTLA 세포로 옮기고 37°C에서 밤새 플레이트를 배양한다.
3. 1 차 스크리닝: 세포 자극
- 24시간 후, 384웰 플레이트를 싱크대 위로 부드럽게 쓸어넘기고 종이 타월 위에 테이핑하거나 흡입기 헤드로 테이핑하여 형질감염된 세포 매체를 디펜더합니다. 40 μL의 굶주림 을 천천히 추가하십시오 (DMEM은 1 % 투석 된 태아 소 혈청 (dFBS)과 1 x 항생제 / 항균제로 보충되어 세포를 직접 만지지 않도록주의하십시오.
- 관심 있는 화합물의 피펫 20 μL은 3배 농도(세포 플레이트 내의 약물의 최종 농도는 1x가 될 것이다)를 (+) 자극을 가진 교대행으로, 및 (-) 화합물없이 교대행에 대한 비히클 버퍼의 20 μL이다. 5%CO2에서 37°C에서 세포 판을 반환하고 적어도 16 시간 동안 배양한다.
4. 1차 검진: 발광 독서
- 베이커와 보이스21에서개질된 글로 시약을 준비한다: 108 mM 트리스-HCl; 42 mM 트리스 베이스, 75 mM NaCl, 3 mM MgCl2, 5 mM 디티오트레이 톨 (DTT), 0.2 mM 코엔자임 A, 0.14 mg/ ml D-루시페린, 1.1 mM ATP, 0.25% v/v 트리톤 X-100, 2 mM 하이드로핏.
참고 : 시약의 재고 용액은 항상 분말 형태로 Glo 시약에 신선하게 첨가되는 D-Luciferin을 제외하고 사전에 만들 수 있습니다. 블랙 플레이트를 사용 하는 경우, D-루시퍼린의 양을 증가 시킬 수 있다 0.25 mg/mL. - 자극 후 16-24 시간에서, 부드럽게 싱크대 위에 384 웰 플레이트를 가볍게 가볍게 가볍게 하고 종이 타월 위에 테이핑하여 형질 감염 된 세포 매체를 decant. Glo 시약 의 20 μL / well을 추가하고 5-20 분 동안 실온에서 플레이트를 배양하십시오.
5. 1차 선별: 데이터 분석
- 발광 카운터에서 저장된 파일을 스프레드시트로 내보냅니다. 결과는 상대 발광 단위(RLU)로 기록됩니다. 384 웰 플레이트의 레이아웃에 기초하여, 다음 공식을 사용하여 각 수용체의 활성화 (접기 변화)를 계산한다:
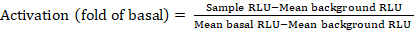
참고: 여기서 샘플 RLU는 자극된(+ 화합물) 사분면의 4개의 복제 웰각각의 값을 의미하며, 평균 배경 RLU는 플레이트상에서 의 음의 대조군의 평균이며, 평균 기저 RLU는 동일한 수용체의 처리되지 않은 사분면의 평균을 의미한다(- 화합물)을 참조하십시오. 또한 4개의 데이터 점의 표준 편차를 계산하여 결과의 품질을 확인합니다. 이종을 정류하기 위해 접기 변경의 평균에 log2 변환을 수행하는 것이 좋습니다. log2 베이스는 포지티브 적중을 식별하는 데 도움이 되는 실용적인 선택입니다. 경험적으로 양수 적중 임계값을 설정; 그것은 주목 해야 한다 일부 수용 체로 낮은 가질 수 있다 2 배 증가 및 최대 40 배 증가 전체 주 작동 근을 가진 다른 사람에 대 한. - 결과에 따라 보조 스크리닝에 대한 잠재적 인 긍정적 인 적중인 GPCR을 선택하십시오.
6. 이차 스크리닝: 세포 종자 및 형질 전환
- 100 mm 의 서브 배양 HTLA 세포는 완전한 배지의 11 mL에서 5 x 106 세포의 총 세포 밀도에서 요리및 24 시간 동안 37 °C에서 배양한다. 당일 형질감염이 바람직한 경우, 종자 세포는 7.5 x 106 세포의 밀도로 추하4시간 후 수행한다.
- 실온에서 칼슘 인산염 침전에 필요한 시약을 미리 따뜻하게 합니다. 450 μL의 0.1x TE 버퍼를 2.5 M CaCl2의 50 μL과 결합하고 신속하게 소용돌이; 이 양은 보유 하는 성장 매체의 볼륨에 따라 하나의 100 mm 접시에 대 한 특정.
- 튜브에 TE/CaCl2 용액 500 μL을 GPCR cDNA 및 와류 10 μg에 추가합니다. 튜브에 2x Hepes 버퍼 용액 500 μL을 넣고 힘차게 흔들고 (소용돌이하지 않음), 1 분 동안 배양하십시오.
참고: 형광 단백질(예: YFP, mCherry 등)을 코딩하는 임의의 플라스미드의 1 μg는 총 10 μg에 대해 9 μg의 GPCR cDNA와 함께 공동 트랜스펙트될 수 있다. 형광 단백질은 형질 감염 효율을 추적하는 데 사용되며,이 최소량은 분석기를 방해하지 않습니다. - 짧은 배양 직후, 1 mL 용액을 세포에 떨어 뜨려 분배하십시오. 플레이트를 앞뒤로 부드럽게 흔들어 침전량을 고르게 분배하고, 플레이트를 소용돌이치지 않도록 주의하고, 24시간 동안 37°C에서 배양한다.
- 다음 날, 형광 세포 이미저 하에서 형광 단백질의 발현을 보면서 형질감염 효율을 관찰하는; 50% 커버리지를 초과하는 형질전환이 이상적입니다.
- 37°C에서 인큐베이터에서 필요한 384웰 PLL 코팅 플레이트(들)를 배양하여 파종 세포 전에 따뜻하게 한다. 384웰 PLL 코팅 플레이트에서 1x 항생제 항균제의 저장 용액을 싱크대 위로 가볍게 치고 종이 타월 위에 테이핑하여 건조시다.
- 형질감염된 세포를 Versene 용액(1X PBS, pH 7.4; 0.53 mM EDTA)으로 부드럽게 헹구고 접시에 0.05% 트립신/0.53 mM EDTA의 3 mL을 추가하여 분리합니다. 내용물들을 트립신을 중화시키기 위해 적어도 동일한 양의 완전한 DMEM을 함유하는 원심분리기 튜브로 옮김을 전달한다.
- 500 x g에서 3 분 동안 세포를 스핀 다운하고 굶주린 매체에서 0.4 x 106 세포 / mL의 밀도로 세포를 다시 일시 중단하십시오. 종자 세포는 전자 다중채널 파이펫을 사용하여 세포 현탁액의 45 μL을 분배함으로써 25,000 셀/웰의 최종 밀도에서 384웰 PLL 코팅 플레이트내로. 플레이트를 최소 4시간 동안 37°C로 되돌려 세포가 웰에 적절하게 부착한 후 자극을 진행합니다.
7. 이차 스크리닝 : 16 점 (반 로그) 용량 곡선을위한 약물 판 준비
- 도 4에나타낸 바와 같이, 플레이트의 마지막 행(행 H)을 제외한 96웰 플레이트에서, 1X HBSS 약물 완충액(1x 행크의 균형 잡힌 염액 [HBSS]), 20 mM HEPES pH 7.4, 1x 항생제 항균제의 270 μL을 첨가한다.
참고 : 펩티드, 콜로이드 분자 및 수용성 화합물의 경우 0.1-1 % BSA를 첨가하는 것이 좋습니다. 약물 산화를 방지하기 위해 최대 0.01 %의 아스코르브 산을 첨가 할 수 있습니다. - 약물 스톡으로부터, 최종 3x 농도(세포 판 내의 약물의 최종 농도는 1배)를 계산하여 약물 용액("높은" 농도라고 함)을 준비한다. 일례로, 가장 높은 농도로 10 μM의 투여량-반응 곡선의 경우, 30 μM에서 "높은" 농도를 준비한다. "높음" 농도의 파이펫 300 μL을 H 행의 우물로.
- 다른 튜브에서, 3.16(반로그)으로 나눈 "높음" 농도를 나타내는 "낮음" 농도를 준비한다. 이전 예에 따라 "낮음" 농도는 9.49 μM입니다. "높음" 웰에 인접한 행 H의 웰로의 "낮은" 농도의 파이펫 300 μL.
참고: 행 H에 필요한 96웰의 총 수는 세포 수와 자극 조건에 따라 달라집니다. 4개의 웰(2개의 "높음"과 2개의 "낮음")은 전체 384 웰 플레이트를 자극하는 충분한 약물 용액을 갖습니다. - "높음" 및 "로우" 행 H의 30 μL 의 약물 용액을 이전 행(row G)으로 파이펫팅하여 직렬 희석을 수행하고 "파이펫 및 믹스" 기능을 갖춘 전자 멀티채널 파이펫을 사용하여 수동으로 위아래로 파이펫팅하거나 권장대로 혼합합니다. 희석 사이의 팁을 삭제하면서 첫 번째 및 가장 희석 된 행 (행 A)까지이 단계를 반복합니다.
참고: 원하는 경우 직렬 희석을 A 행 앞에 중지할 수 있으며, 이는 약물이 없는 내부 제어를 나타내는 것, 즉 "true zero"입니다. - 도 4를 참고로, 96웰 플레이트로부터 20 μL의 "로우" 컬럼 희석을 배관하여 형질감염된 세포를 자극하여 이전에 시드된 384웰 플레이트의 A-O행뿐만 아니라 20 μL의 "높음" 컬럼 희석을 37°C에서 37°C에서 배양하여 최소 16시간 동안 플레이트를 인큐베이트한다.
8. 보조 스크리닝: 발광 판독 및 데이터 분석
- 자극 후 16-24 시간에서, 부드럽게 싱크대 위에 384 웰 플레이트를 가볍게 가볍게 가볍게 하고 종이 타월 위에 테이핑하여 형질 감염 된 세포 매체를 decant. Glo 시약 의 20 μL / well을 추가하고 5-20 분 동안 실온에서 플레이트를 배양하십시오.
- 발광 카운터에서 저장된 파일을 스프레드시트로 내보냅니다. 결과는 상대 발광 단위(RLU)로 기록됩니다. 384웰 플레이트의 데이터를 통계 소프트웨어로 전송하여 비선형 회귀 곡선 맞춤을 위해 내장된 XY 해석을 사용하여 결과를 분석합니다. 내장된 3파라미터 용량-응답 자극 기능 "로그(agonist) vs. 응답(3개의 파라미터)",

참고: 여기서 상부와 하부는 Y축의 단위에서 고원이며, 각각 최대 응답 및 기저 수준, EC50은 상하 간에 50% 응답을 생성하는 작용제의 농도이며, X는 작용제의 로그 농도를 의미한다. 이 모델은 용량 응답 곡선의 표준 언덕 경사가 1이라고 가정합니다.
Access restricted. Please log in or start a trial to view this content.
결과
본 원에 제시된 PRESTO-탱고 프로토콜을 사용하여, 크로마핀 과립(CG) 추출물을 168개의 비후각 GPCR 표적에 대해 스크리닝하였고, 대다수는 고아 수용체이다. 상기 추출물의 프로파일링은 바네아 외18(도 1)에의해 설계된 원리에 기초하여 선택된 수용체에서 β-arrestin2 동원을 조사함으로써 수행되었다. 관심 있는 GPCRs의 플라스미드 cDNA는 PRESTO-탱고 GPCR 키트에서 ...
Access restricted. Please log in or start a trial to view this content.
토론
형태역학적 GPCR은 신호 변환의 강국입니다. 이러한 heptahelical 수용체의 결합 포켓의 생리화학적 특성뿐만 아니라 그들의 생리학적 관련성은 GPCR 리간드 스크리닝 도구의 필요성을 강조한다. 위에서 제시한 바와 같이, PRESTO-탱고 분석은 신속하고 민감하며 사용자 친화적이며 약물 개발에 자신을 빌려주고 있습니다. 이 분석은 작용제 유도 활성화를 측정할 뿐만 아니라 길항제 및 알로스테리컬
Access restricted. Please log in or start a trial to view this content.
공개
authours는 경쟁적인 이익을 선언하지 않습니다.
감사의 말
이 작품은 건강 연구의 캐나다 학회에 의해 지원되었다 (CIHR 교부금 #MOP142219).
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 384 Well Optical Bottom Plates, Polystyrene Polymer Base, Cell Culture Treated, BLACK, with lid, Sterile | NUNC | 12-566 | |
| 384 Well Optical Bottom Plates, Polystyrene Polymer Base, Cell Culture Treated, White, with lid, Sterile | NUNC | 12-566-1 | |
| 384 Well Round Bottom, Polypropylene, Non-Treated, Blue, non-sterile, without lid | ThermoFisher | 12-565-390 | |
| Antibiotic-Antimycotic | Wisent | 450-115-EL | |
| D-Luciferin, sodium salt | GoldBio | LUCNA | |
| DMEM with L-Glutamine, 4.5g/L Glucose and Sodium Pyruvate | Corning | 10-013-CV | |
| Eppendorf Xplorer, 12-channel, variable, 15–300 µL | Eppendorf | 4861000155 | |
| Eppendorf Xplorer, 12-channel, variable, 5–100 µL | Eppendorf | 4861000139 | |
| Matrix Platemate 2x3 | ThermoFisher | 801-10001 | |
| MicroBeta 1450 Wallac | Perkin Elmer | ||
| Penicilin-Streptomycin | Wisent | 450-201-EL | |
| Poly-L-Lysine hydrobromide | Millipore-Sigma | P2636-500MG | |
| Roth Lab PRESTO-Tango GPCR Kit | Addgene | Kit #1000000068 |
참고문헌
- Liapakis, G., et al. The G-protein coupled receptor family: actors with many faces. Current pharmaceutical design. 18 (2), 175-185 (2012).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nature Reviews Molecular Cell Biology. 3 (9), 639-650 (2002).
- Kroeze, W. K., Sheffler, D. J., Roth, B. L. G-protein-coupled receptors at a glance. Journal of cell science. 116, Pt 24 4867-4869 (2003).
- Rask-Andersen, M., Almén, M. S., Schiöth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Ngo, T., et al. Identifying ligands at orphan GPCRs: current status using structure-based approaches. British journal of pharmacology. 173 (20), 2934-2951 (2016).
- Zhang, R., Xie, X. Tools for GPCR drug discovery. Acta pharmacologica Sinica. 33 (3), 372-384 (2012).
- Kimple, A. J., Bosch, D. E., Giguere, P. M., Siderovski, D. P. Regulators of G-Protein Signaling and Their G Substrates: Promises and Challenges in Their Use as Drug Discovery Targets. Pharmacological Reviews. 63 (3), 728-749 (2011).
- Wettschureck, N., Offermanns, S. Mammalian G Proteins and Their Cell Type Specific Functions. Physiological Reviews. 85 (4), 1159-1204 (2005).
- Denis, C., Saulière, A., Galandrin, S., Sénard, J. -M., Galés, C. Probing heterotrimeric G protein activation: applications to biased ligands. Current Pharmaceutical Design. 18 (2), 128-144 (2012).
- Yin, H., et al. Lipid G protein-coupled receptor ligand identification using beta-arrestin PathHunter assay. The Journal of Biological Chemistry. 284 (18), 12328-12338 (2009).
- Cheng, Z., et al. Luciferase Reporter Assay System for Deciphering GPCR Pathways. Current Chemical Genomics. 4, 84-91 (2010).
- Roth, B. L., Kroeze, W. K. Integrated Approaches for Genome-wide Interrogation of the Druggable Non-olfactory G Protein-coupled Receptor Superfamily. The Journal of Biological Chemistry. 290 (32), 19471-19477 (2015).
- Jean-Charles, P. -Y., Kaur, S., Shenoy, S. K. G Protein-Coupled Receptor Signaling Through β-Arrestin-Dependent Mechanisms. Journal of Cardiovascular Pharmacology. 70 (3), 142-158 (2017).
- Smith, J. S., Rajagopal, S. The β-Arrestins: Multifunctional Regulators of G Protein-coupled Receptors. The Journal of Biological Chemistry. 291 (17), 8969-8977 (2016).
- Böhme, I., Beck-Sickinger, A. G. Illuminating the life of GPCRs. Cell Communication and Signaling. 7 (1), 16(2009).
- Fang, Y. Label-Free Receptor Assays. Drug discovery today. Technologies. 7 (1), 5-11 (2011).
- Wang, T., et al. Measurement of β-Arrestin Recruitment for GPCR Targets. Assay Guidance Manual. Eli Lilly & Company and the National Center for Advancing Translational Sciences. , (2004).
- Barnea, G., et al. The genetic design of signaling cascades to record receptor activation. Proceedings of the National Academy of Sciences of the United States of America. 105 (1), 64-69 (2008).
- Kroeze, W. K., et al. PRESTO-Tango as an open-source resource for interrogation of the druggable human GPCRome. Nature Structural & Molecular Biology. 22 (5), 362-369 (2015).
- Jordan, M., Schallhorn, A., Wurm, F. M. Transfecting Mammalian Cells: Optimization of Critical Parameters Affecting Calcium-Phosphate Precipitate Formation. Nucleic Acids Research. 24 (4), 596-601 (1996).
- Baker, J. M., Boyce, F. M. High-throughput functional screening using a homemade dual-glow luciferase assay. Journal of Visualized Experiments. (88), 50282(2014).
- Li, H., Eishingdrelo, A., Kongsamut, S., Eishingdrelo, H. G-protein-coupled receptors mediate 14-3-3 signal transduction. Signal Transduction and Targeted Therapy. 1 (1), 16018(2016).
- Walther, C., Ferguson, S. S. G. Minireview: Role of intracellular scaffolding proteins in the regulation of endocrine G protein-coupled receptor signaling. Molecular Endocrinology. 29 (6), Baltimore, Md. 814-830 (2015).
- Gurevich, E. V., Benovic, J. L., Gurevich, V. V. Arrestin2 expression selectively increases during neural differentiation. Journal of Neurochemistry. 91 (6), 1404-1416 (2004).
- Shaikh, S., Nicholson, L. F. B. Optimization of the Tet-On system for inducible expression of RAGE. Journal of Biomolecular Techniques JBT. 17 (4), 283-292 (2006).
- Blau, H. M., Rossi, F. M. Tet B or not tet B: advances in tetracycline-inducible gene expression. Proceedings of the National Academy of Sciences of the United States of America. 96 (3), 797-799 (1999).
- Roche, O., et al. Development of a Virtual Screening Method for Identification of "Frequent Hitters" in Compound Libraries. Journal of Medicinal Chemistry. 45 (1), 137-142 (2002).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유