Method Article
ナノ流体技術を用いた単一およびバルク C.エレガンス サンプルにおける高スループット定量RT-PCR
* これらの著者は同等に貢献しました
要約
本稿では、単一またはバルク C.エレガンス サンプル中の遺伝子発現レベルの迅速かつ信頼性の高い決定のためのハイスループットプロトコルについて説明する。このプロトコルはRNAの単離を必要とせず、サンプルから直接cDNAを生成します。高スループットの多重化されたナノ流体リアルタイムqPCRプラットフォームと一緒に使用できます。
要約
本論文では、高速、堅牢、高感度の カエノハブディティス・エレガンス に対するハイスループット逆転写定量PCR(RT-qPCR)アッセイを紹介する。このプロトコルは、単一ワームまたはバルクサンプルから遺伝子発現の正確な測定を取得します。ここで示すプロトコルは、ナノ流体RT-qPCRプラットフォームに結合された相補DNA(cDNA)調製のための既存の方法の新しい適応を提供する。このプロトコルの最初の部分は「ワームからCTへ」と名付けられ、事前のmRNA分離を必要とせずに線虫から直接cDNAを産生することができます。3.5時間で96ワームからcDNAを調製することで、実験的なスループットを向上させます。プロトコルの 2 番目の部分では、既存のナノ流体技術を使用して、cDNA 上で高スループット RT-qPCR を実行します。本論文では、2つの異なるナノ流体チップ(最初のラン96サンプルと96ターゲット)を評価し、約1.5日間のベンチワークで9,216回の反応を示します。2番目のチップタイプは6つの12 x 12アレイで構成され、864個の反応が得られます。ここで、ワーム-to-CT法は、単一ワームおよびバルクサンプルからヒートショックタンパク質をコードする遺伝子のmRNAレベルを定量することによって実証される。提供されるプライマーの広範なリストは C.エレガンス ゲノム内のコード遺伝子の大部分のために処理されたRNAを増幅するように設計されている。
概要
単一細胞RNAシーケンシングおよびqPCRの最適化により、転写パルスまたはバーストが細胞当たりのRNA分子数に大きなばらつきを生じさせる可能性が明らかになった。さらに、これらの技術は、標準的なバルクトランスクリプト法測定によって以前に見逃された実質的な細胞異質性を明らかにした。文脈に応じて、いくつかの単細胞転写変動は、組織の混合細胞組成によって引き起こされる。しかし、同じ環境下で増殖した等元性細胞集団においても、転写ヘテロジニティ2,3が広く存在する。この「生物学的変動」は、細菌から人間まで、細胞ネットワークのユビキタスな特性としてますます同定されています。いくつかのケースでは、開発、癌の進行、HIVの遅延、および化学療法4、5への応答に触れ合った結果を持つことができます。
線虫カエノハブディティスエレガンスは、個人間の生物学的変動の原因と結果を研究するための理想的な特徴を持つユニークなモデル生物です。これらの線虫は959細胞からなる単純なモデル生物であり、その透明なキューティクルは、インビボイメージング研究6に適している。C.エレガンスは主に自己受精を通して繁殖する雌雄同体の種である。これは、等原性の実験室株をもたらした。等原性および制御された培養条件にもかかわらず、多くの形質および転写物は、個体間で可変であり、確率的または微小環境的な違いが、個体間の不均一性に寄与することを示唆する。遺伝子発現におけるこのような変動は、突然変異の浸透の変動、生存、発達タイミング、および胎児性7、8、9を含む複数のフィットネスの結果を有する。これらの特徴により、単一ワーム研究は生物全体の生物学的変動を研究する前例のない機会を提供する。
この分野では、単一ワームレベルでのトランスクリプトの正確な検出のための技術を開発し最適化することが必要です。単一ワームRNAシーケンシング10、単離組織11からのRNAシーケンシング、単一細胞シーケンシング12などの新しい技術がC.エレガンスに利用可能になりました。しかし、主な課題は依然として残っている:相互性を監視する場合、弱く発現した遺伝子はしばしば検出可能なレベル13を下回る。これは、少量の出発物質から分離された稀な転写物に特に関連し、平均発現と技術的差異との間に確立された逆の関係があり、しばしば希少なトランスクリプトが統計的カットオフ13を下回る原因となる。ハイスループット多重qPCR技術の最適化は、哺乳類の単細胞研究に有用であることが証明されており、特に希少転写物14,15の発現を研究する場合である。このテクノロジは、他の単一ワーム テクノロジのベンチマークおよび検証の目的にも使用できます。
ワームからCTは、単一ワームcDNA調製のために、細胞生物学の研究で使用されるキットから適応された高速で堅牢な方法です。この方法と組み合わせたcDNAを多重化したナノ流体qPCR技術と結合させたのは、より高い実験スループット、より広いダイナミックレンジの検出を提供し、単細胞目的14,15のために検証されたからである。また、この cDNA 調製は、標準 PCR 技術での使用にも適用できます。スループットは2つの方法で増加します:まず、cDNA調製は、従来のグアニジウム・チオシアネート・フェノール-クロロホルム抽出よりも速く、信頼性が高く、ワームはリシスバッファーに直接添加され、容易に分解可能なRNAの直接単離をスキップします。第2に、ナノ流体技術を活用することで、同時に実行できるサンプルとターゲットの数が大幅に増加します。本稿では、シングル・アレイ・チップとマルチアレイ・チップの2つのチップを比較します。シングルアレイチップは、96個の単一ワームと96のプライマーセットを実行でき、実験ごとに9,216回の反応を得ることができます。標準の qPCR 技術を使用して同様のスループットを実現するには、96 ウェルプレートを使用して 96 個の個別の qPCR 実験が必要になります。より小型で柔軟性の高いマルチアレイチップは、6個の12 x 12アレイで構成され、864個の反応が得られます。この方法の優れた信頼性と感度は、ナノ流体技術と予備増幅工程の導入によって高められています。本論文で紹介する方法は、生物学的分散を抽出する最先端の統計アルゴリズムと共に使用することを意図している。この記事では、単一ワームとバッチワームの両方のサンプルに対する迅速な cDNA 準備と高スループット qPCR のプロトコルを紹介します。アルゴリズムは他の場所で公開されます。このプロトコルでは、実験の前に各チップの構成を準備する必要があります。表1および表2は、マルチアレイチップとシングルアレイチップに対するこれらの計画の例をそれぞれ示しています。図 1 で説明した Worm-to-CT プロトコルの概要と、図 2のマルチアレイ チップとシングル アレイ チップの実行の概要も示します。
プロトコル
注: このプロトコルを通して 、カエノハブディティス・エレガンス は「ワーム」または「ワーム」と呼ばれます。 様々なC.エレガン株 は、オンラインデータベースを介して、またはモデル生物を使用するラボに直接連絡することによって注文することができます。このプロトコルのパートI(セクション1-3)では、ワームからCTへのプロトコルを介したcDNAの準備について説明します。このプロトコルのパート II (セクション 4-13) は、Fluidigm16によって開発されたプロトコルから適応した、ナノ流体を使用して高スループット RT-qPCR を実行する方法を説明します。このプロトコルは、先に定義された2種類のナノ流体チップ、96個のターゲットを96サンプル(合計9,216 RT-qPCR反応)に監視できるシングルアレイチップ、または12ターゲットx12サンプルのサブユニットとして機能するマルチアレイチップの使用に適用されます。すべてのマルチアレイチップには、6つの独立したアレイが含まれています。たとえば、マルチアレイチップ全体を使用すると、72個のターゲットx 12サンプル(またはその逆)、または36ターゲットx 24サンプル(またはその逆)を監視できます。このプロトコルで使用される材料の詳細については、 資料表を参照してください。
1. RT-qPCR プライマー検証
注意:リアルタイムプライマーは、当初MIQUEガイドライン17によって発行された推奨プロパティに基づいて設計されました。処理されたRNAに特異的なプライマーを作るために、製品は、少なくとも1つのスプライス接合の両側に結合した2つのプライマーになるように設計した。適切なプライマーの要件には、グアニンおよびシトシン含量20%、融解温度58-60°C、プライマー対≤0.5°Cの融解温度の差、70~120bpの製品長が含まれていました。生成されるプライマーの配列は 補足表 1に記載されています。プライマーの生成に使用するスクリプトのオープンソースコードは、https://github.com/s-andrews/wormrtpcr にあります。スプライスサイトを持つトランスクリプト用のプライマーペアは、イントロンに隣接する2つのエキソンに横たわるように設計されたが、NCBIプライマーブラストソフトウェア18を使用して、スプライスバリアント特異的になるように設計されていない。この研究では、プライマーセットを C.エレガンス ゲノムに対して爆破し、オフターゲットの相補性をテストした。
- RT-qPCRプライマーのデータベースからプライマーを取り出す(補足表1)。あるいは、NCBI プライマー ブラスト18などのオンライン ツールを使用して qPCR プライマー ペアを設計します。
- qPCR 標準曲線を実行して、標準バルク qPCR 技術19および MIQUE ガイドライン17,20に従ってプライマーの各ペアの特異性と PCR 効率をモニタリングします。
注: R2 > 0.98 のプライマー ペアと PCR 効率は 85~ 115% の場合にのみ使用してください。この研究で使用したプライマーの配列、PCR効率、およびR2 について、 表3に詳述する。
2. ワームからCTへのワームリシス
- 細菌の芝生から新鮮な種なしのNGMプレートにワームを選び、ワームがその動きを通してワームから細菌のほとんどを取り除くために5分間プレートの周りを移動できるようにします。
注:細菌の芝生と成長条件は、実験計画によって異なります。ここで発表された実験では、OP50 大腸菌 を播種した6cmのNGMプレートが必要であり、20°CインキュベーターでL4.9のステージに成長した目的のワームが含まれる。 - RNaseフリーフードでは、12.5 μLの2x RTバッファ、1.25 μLの20倍RT酵素バッファー、および1サンプル当たり0.25 μLのヌクレアーゼフリー水からなるマスターミックスを準備します。ステップ2.8から各サンプルの11 μLに14 μLのマスターミックスを加えます。
- PCRストリップの蓋を逆さまにして、分解スコープのプラットフォームに置き、化合物顕微鏡下のドーム型PCRチューブキャップに10μLのリシスミックスを加えます。
注: サンプルが 1 つか 2 つしかない場合は、キャップが開く可能性が減るため、少なくとも 4 つのチューブを含む PCR ストリップを使用することをおし、その後のフリーズ融解のステップで開きます。あるいは、ゴムバンドを使用してキャップを所定の位置に保持することができます。その場合、チューブが移動するたびにキャップが適切に閉じられていることを確認します。 - 細菌汚染を避けるために、ライシスミックスを含む蓋の各スロットにワームを「すくい」(すなわち、ピックで下のワームを捕まえる)で、プレートからワームを選びます。チューブを閉じ、テーブルトップマイクロ遠心分離機(材料表)を使用して5s回転してから、液体窒素を充填したデュワーフラスコに入れてください。
注: 15 ~ 30 個のワームは、一括実験に使用し、1 つのワームを測定する場合は 1 つのワームを使用する必要があります。
注意:液体窒素を取り扱う場合は、皮膚や目との接触が深刻な凍傷を引き起こす可能性があるため、Cryo-手袋と保護アイウェアを着用し、標準的な衣類規制に準拠しています。 - PCRチューブを液体窒素と~40°Cの水浴との間で転写して10倍凍結解凍します。サンプルが完全に凍結していることを確認するために、液体窒素にチューブを最低5 s放置します。サンプルが解凍されるまで、チューブを水浴中に残します。RNA分解につながるため、長期間放置しないでください。
注:サンプルが約-200°Cに凍結され、RNAの分解を減らすので、チューブは液体窒素に長期間放置することができます。しかし、液体窒素が急速に蒸発するため、これはプロトコル停止点であってはなりません。 - サーマルミキサー(材料表)でサンプルを混合し、4°Cに設定し、20~30分回転して~1,800rpmで回転させます。
- サンプルが混合されている間、氷の上で停止溶液を解凍します。
- テーブルトップのマイクロ遠心分離機(材料表)を使用してサンプルをスピンダウンし、各チューブに1μLのストップ溶液を追加します。
注:サンプルはRNAを逆転写する前に、-80°Cで最大1週間放置することができます(セクション3)。
3. 逆転写
注:単一ワームの逆転写については、ここに示す結果は、ナノ流体チップを備えた試薬を使用して生成されました( 図1のオプション2)。 図1 のオプション2で強調された試薬は、プールされたサンプルの逆転写にも使用されました。どちらの方法も、さまざまなサンプルタイプで同じ意味で動作します。
- 単一ワームの逆転写
- RNaseフリーフードで、1.25 μLの逆転写ミックス(材料表)を新しいPCRチューブに加えます。
注:多くのサンプルがある場合は、96ウェルPCRプレートと自動ピペットを使用することができます。メーカーのプロトコルには、1つのサンプルあたり1 μLが使用できると書かれています。 - 溶解液を5μLとし、ステップ2.8から溶液ミックスを停止し、逆転写ミックスを含む新鮮なPCRチューブに添加します。
注:メーカーのプロトコルでは、1 μLのRNA(2.5 pg/μL~250 ng/μL)を反応ごとに使用できると定めています。1プレート当たりの負のRT制御は、逆転写ミックスを5μLのlysedサンプルと1.25 μLのRNAAseフリー水に置き換えることによって追加できます。 - サーボサイクラーで逆転写プログラムを使用してサンプルを実行します:25°Cは5分間、42°Cは30分、85°Cは5分、∞は4°Cです。
注:生成されたcDNAは、高スループットのqPCRを使用して増幅およびデータ収集に進む前に-20°Cで保存することができます。
- RNaseフリーフードで、1.25 μLの逆転写ミックス(材料表)を新しいPCRチューブに加えます。
- バルク サンプルの逆転写 (15-30 ワーム)
- RNaseフリーフードでは、12.5 μLの2x RTバッファ、1.25 μLの20倍RT酵素バッファー、および1サンプル当たり0.25 μLのヌクレアーゼフリー水からなるマスターミックスを準備します。11 μLの溶解溶液に14 μLのマスターミックスを加え、ステップ2.8から溶液ミックスを停止します。
注:多数のサンプルを扱う場合、これは96ウェルプレートで行うことができます。 - 次の逆転写プログラムを使用してサーモサイクラーを介してサンプルを実行します: 37 °C 60 分, 5 分の 95 °C, ∞の場合は 4 °C.
- 製造したcDNA 1:4をヌクレアーゼを含まない水で希釈します。
注:一般的に、製品は25 μLの最終容積に来て、その場合は75 μLを追加する必要があります。しかし、凝縮のために、最終的な体積は変化する可能性があります。したがって、最終的な溶液で1:4の比率を作るように調整してください。この希釈手順は、単一ワームに対して qPCR を実行する場合には適用されません。生成されたcDNAは、高スループットのqPCRを使用して増幅およびデータ収集に進む前に-20°Cで保存することができます。
- RNaseフリーフードでは、12.5 μLの2x RTバッファ、1.25 μLの20倍RT酵素バッファー、および1サンプル当たり0.25 μLのヌクレアーゼフリー水からなるマスターミックスを準備します。11 μLの溶解溶液に14 μLのマスターミックスを加え、ステップ2.8から溶液ミックスを停止します。
4. マルチプレックスプライマーミックスの準備
- 50 μM 最終濃度のプライマーの各ペアに対して、フォワード/リバース(F/R)プライマーストックを準備します。同じボリュームの順方向プライマーとリバースプライマーをそれぞれ100 μMに混ぜます。
- テストするプライマーペアごとに、50 μM F/R プライマーストックの 1 μL を組み合わせます。DNA懸濁液バッファーを合計容量100 μLまで追加します。
注:ここでのストックプライマー濃度は、メーカーのプロトコル16に記載されているものとは異なりますが、同じ最終濃度の500 nMを保持します。
5. ターゲット特定の予備増幅
- 1 μLのプリアンプ化マスターミックス(材料表)、プールプライマーミックスの0.5 μL(ステップ4.2)、および10%の全体余剰体積を含む反応ごとに2.25μLのヌクレアーゼフリー水を含むマスターミックスを準備します。
- 96ウェルプレートでは、マスターのアリコート3.75 μLが、サンプル数の実行に必要な数のウェルに混入します。
- ステップ3.1.3または3.2.3で生成された目的のcDNA溶液の1.25 μLを各ウェルに追加します。
- プレートを96ウェルシーリングテープ、短い渦、遠心分離機で卓上プレートスピナーで覆います。サーモサイクラーに移し、次のプログラムを実行します:2分間95°C、15sの95°Cで15サイクル、60°Cでアニーリング/延長4分、∞4°C。
注:メーカーは、プリアンプリフェケーション反応16のために10〜20サイクルから推奨しています。このプロトコルは、標的遺伝子の発現レベルに応じて10サイクルまたは15サイクルを推奨する。
6. エキソヌクレアーゼI治療
注: これは、未組み込みのプライマーを予備化から除去する方法です。
- エキソヌクレアーゼIの0.2 μLのエキソヌクレアスI反応バッファー(材料表)、エキソヌクレアーゼIの0.4 μLを20 U/μL(材料表)、1サンプル当たり1.4μLのヌクレアーゼフリー水で調製します。氷の上にすべての試薬、特にエキソヌクレアーゼIを保管してください。
- 96ウェルプレート(ステップ5.4)をサーモサイクラーから取り出し、卓上プレートスピナーで遠心分離機を取り出し、シールを慎重に取り外します。各プレアンプ化反応に2μLのエキソヌクレアーゼをミックスする。再シール、遠心分離機、および次のプログラムを使用してサーボサイクラーに96ウェルプレートを戻します:30分間37°C、15分間80°C、∞の場合は4°C。
- サーモサイクラーからサンプルを取り出し、1x Tris EDTAバッファ(材料表)の18 μLを加えて1:5を希釈します。
メモ:cDNAサンプルは-20°Cに保ち、後で使用することができます。メーカーのプロトコルは、対象となるターゲットの発現レベルに応じて、この段階16で5倍、10倍、または20倍の希釈の可能性を示唆しています。
7. アッセイミックスの準備
注:アッセイミックスは、ウェルがナノ流体チップと同じ間隔を持ち、読み込みが容易になるため、384ウェルプレートで調製できます。
- マルチアレイチップ用アッセイミックスの準備
- 2xアッセイローディング試薬2μL(材料表)と1.6μLのDNA懸濁液緩衝液(材料表)からなるマスターミックスを、準備計画に従って各ウェルに用意します。このマスターミックスのアリコート3.6 μLは、384ウェルプレートに分かれています。
- ステップ4.1で準備した50μM F/Rプライマーストックの0.4 μLを、準備された計画に従って適切なウェルに追加します。
注:これは1 μLの余剰と、井戸ごとのアッセイミックスの合計4 μLを提供します。
- シングル・アレイ・チップ用のアッセイ・ミックスの準備
- 調製計画に従って、3μLの2xアッセイローディング試薬と2.4μLのDNA懸濁液バッファーからなるマスターミックスを準備します。このマスターミックスのアリコート5.4 μLは、マークされた384ウェルプレートに十分に混ぜます。
- 準備された計画に従って、50 μM F/R プライマーストックの 0.6 μL を適切なウェルに追加します。
注:これは1つの井戸ごとのアッセイミックスの合計6 μLを提供し、1 μLの余剰を提供します。
8. サンプルミックスの準備
注:サンプルミックスは、事前に1日まで調製し、4°Cで保存することができます。
- マルチアレイチップのサンプルの準備
- 2x蛍光プローブスーパーミックス2μLのサンプルマスターミックスを調製し、低ROX(材料表)、サンプル試薬0.2μL(材料表)をサンプルあたりご用意ください。このミックスの2.2 μLをマークされた384ウェルプレートに分配します。
注:メーカーは、サンプル試薬16をボルテックスしないことをお勧めします。 - ピペット1.8μLの各プリアンプ化されたエキソヌクレアーゼは、ステップ3.2.3から適切なウェルにサンプルを処理した。
注:合計4 μLで、余剰は1μLです。
- 2x蛍光プローブスーパーミックス2μLのサンプルマスターミックスを調製し、低ROX(材料表)、サンプル試薬0.2μL(材料表)をサンプルあたりご用意ください。このミックスの2.2 μLをマークされた384ウェルプレートに分配します。
- シングル・アレイ・チップ用サンプルの準備
- 低ROX(材料表)を用いた3μLの蛍光プローブスーパーミックスと、サンプルあたり20倍のDNA結合染料サンプルローディング試薬(表)の0.3 μLからなるサンプルマスターミックスを準備します。このミックスの3.3 μLをマークされた384ウェルプレートに分配します。
- ピペット2.7μLの各プリアンプとエキソヌクレアーゼは、ステップ6.3から適切なウェルにサンプルを処理し、準備された計画に従って。
注:合計6 μLで、余剰は1μLです。サンプルなしで実行するウェルがある場合、これらはcDNAの代わりにサンプルマスターミックスと2.7 μLの水をロードする必要があります。これは両方のチップタイプに推奨されます。チップのグリッドを検出するために、機械はすべての入口に低いROXを必要とします。
9. ナノ流体チップのプライミング
注: マルチアレイチップは、初回実行時にのみプライミングする必要があります。同じチップの後続の実行がある場合、このステージはスキップできます。これらの手順は、両方のチップタイプで同じです。
- ゆっくりと慎重に、含まれているシリンジから制御ライン流体の完全な150 μLをチップのアキュムレータに注入します。45°の角度でチップを保持し、シリンジの先端を押さえて、こぼれを避けて、制御液体がチップに触れないようにします。
- チップの底面から青い保護フィルムを取り外します。
- チップをナノ流体 PCR プライミングマシン (材料表) に挿入し、バーコードを外側に向けます。15~20 分かかる'Prime (153x)' スクリプトを実行します。
メモ:この段階でナノ流体サーモサイクラー(材料表)をオンにします。
10. ナノ流体チップのロード
- ロードが行われると、バリアプラグを順番に取り外します。これにより、井戸の誤ロードの可能性が低くなります。
- マルチアレイチップに3μL、または各プライマーアッセイのシングルアレイチップに5μLを転送し、調製した計画に従ってナノ流体チップの対応する入口にサンプルミックスを行います。バブルを導入しないようにしてください。
注:プライマーなしで実行される井戸がある場合は、プライマーのボリュームを水に置き換えて、マスターミックスをロードすることが重要です。これは両方のチップタイプに適用されます。この段階では、チップを暗い表面に置くのが簡単になり、井戸がより簡単に見えるようにすることができます。
11. ナノ流体チップの実行
注: マルチアレイ チップを初めて実行する場合は、[ ツール] を選択してトラッキング ファイルを設定| Flex Six Usage Trackingをクリックし、[ 新規] をクリックしてファイル名を入力し、場所を選択してから [完了] をクリックします。
- データ収集ソフトウェアを開きます。[ 新しい実行の開始] をクリックします。バーコードを外側に向けてナノ流体サーモサイクラーに取り込んだチップを置きます。
- 必要に応じてプロジェクト設定を選択し、[ 次へ ]をクリック| 読み込み。マルチアレイチップをロードする場合は、実行するパーティション(アレイ)を選択します。
- アプリケーションの [参照プローブ] を選択し、アプリケーションの種類を [遺伝子表現] に変更し、パッシブ参照を [ROX]に変更します。 [単一プローブアッセイ] を選択し、プローブタイプを Eva Greenに変更して、[ 次へ] をクリックします。
- マルチアレイ チップを実行する熱サイクル プロトコル GE FLEX 6 Fast PCR+Melt v1 を選択するか、シングルアレイ チップを実行するプロトコル GE 96.96 Fast PCR+Melt v2 を選択します。
- [自動露出]が選択されていることを確認し、[ファイル名を指定して実行を開始] をクリックします。
12. ポストチップラン
注:このセクションは、チップ全体を使用しない場合にのみ、マルチアレイチップに必要です。
- ナノ流体サーモサイクラーからチップを取り出し、ナノ流体PCRプライミングマシンにロードし、5分続くポストラン(153x)スクリプトを実行します。
- 個人参照に使用するプラグにラベルを付けます。
注:チップは室温で保存でき、チップ上の残りのアレイは2ヶ月以内に実行できます。
13. データのクリーンアップと分析
- データを開くには、 "リアルタイム PCR 解析" ソフトウェア (材料表) を参照してください。テストしたすべてのプライマーペアの融解ピーク温度を確認します。所定のプライマー対に対して、複数の融解温度ピークを示す結果を排除する。
注:複数のピークは、プライマーペアがダイマーを形成するとき、またはプールされたプライマーミックス内の他のプライマーとのターゲットプライマーの相互作用から、時折しか現れません。 - データを「ヒートマップ」スプレッドシートファイルとしてエクスポートし、失敗したサンプルやプライマーを排除します。
- 標準のデルタCt法21を使用してデータを分析します。統計的評価のために、相対式レベルで一方向の分散分析を行う。
結果
cDNA調製方法としてのワーム-CTの検証
ワーム対CTプロトコルが有効なcDNA抽出方法であるかどうかを試験するために、標準的なグアニジウム・チオシアネート・フェノール-クロロホルム抽出方法と比較した。結果は 図3に示されており、cDNAは標準のグアニジウム・チオシアネート・フェノール-クロロホルム抽出技術22 およびワームツーCT法を用いた30ワームから平均1,000ワームから調製された。試料を同時に熱衝撃(34°Cで30分)とした。世界的に、全RNAの 100ng当たりのhsp-70 mRNA発現レベルは、両方の方法を用いて比較可能であった。しかしながら 、hsp-70 発現が最も高い場合(すなわち、N2ではヒートショック後)発現レベルがワーム-ct法と共に高く、感度が向上したことを示す。
hsf-1(sy441)23におけるhsp発現の予想減少が予想されるかどうかを判断するために、分子シャペロン23,24の主転写調節因子における突然変異を再現することができ、短い熱ショックに続く転写シャペロン誘導を比較した。両方の方法でhsp-70誘導の減少はhsf-1(sy441)動物で検出された。これは、変異型hsf-1(sy441)動物がHSF-1のトランス活性化ドメインにおける切り捨てによるシャペロン誘導能の低下を示すため、予想された。グアニジウム・チオシアネート・フェノール-クロロホルム抽出hsp70の場合、野生型動物と比較して対照と比較して82.7%、ワームからCTに対して92.3%減少した(図3)。結果は両方の方法で比較可能であり、以前のレポート23と同等であった。これらの結果は、ワームツーCT法が標準的なcDNA合成技術に代わる有効な代替手段であることを示している。
mRNA標的の増幅に用いられるナノ流体PCRプラットフォームの検証
転写増幅のためのナノ流体qPCRを用いた結果の一貫性をテストするために、ワーム-CTバルク法から得られたPCR結果を、標準qPCRシステム(材料表)とマルチアレイチップを用いたナノ流体qPCRシステムの両方で比較した。3つの異なる遺伝子の発現の折り目変化を、野生型と比較して、dbl-1(dbl-1(nk3))25でヌルアレーレを持つ動物で、sma-3(図4A)、sma-10(図4B)、およびdnj-26を監視した。Dbl-1は、骨形態形成タンパク質(BMP)シグナル伝達経路の唯一のリガンドをコードする。sma-3およびsma-10は、BMPシグナル伝達カスケードの主要成分であるSMADオルソローグをコードする遺伝子である。Dnj-26は、BMPシグナル伝達の標的である分子シャペロンをコードする。これらの結果は、2つの方法の結果を比較する折り目変化にほとんどまたは全く違いを示さないため、sma-3、sma-10、およびdnj-26の場合、それぞれ有意なP値は0.3113、0.2635、および0.3481で示されない。 これらの結果は、バルクサンプルに適用されるワーム-to-CT法が、少数のワームからRNAを抽出する効率的かつ迅速な方法であり、標準PCRシステムまたは高スループットナノ流体ベースのqPCRプラットフォームと組み合わせた場合に信頼性の高いデータを提供することを示しています。
一回のワームから得られた平均を用いてバルクサンプルによって得られた発現レベルの比較
相対発現レベルは、バルクサンプル(25ワーム)から得られたcDNAまたは平均36個の単一ワームサンプルから計算した(図5)。cDNAはいずれもワームツーCT法を用いて取得し、ナノ流体PCR技術を用いて増幅した。A–C図5に観察されるように、試験されたすべてのシャペロン(すなわち、hsp16.1、F44E5.4、hsp-70)について、同等の発現レベルを検出した。 これらの結果は、単一ワームから得られたパラメータが信頼できる状態であることを示しています。
ワーム--CT結合ナノ流体技術の応用による単一ワーム遺伝子発現パラメータの推定
シングルアレイチップは、96個の個々のサンプルで最大96のターゲットトランスクリプトを監視できるため、単一ワーム間の転写表現の個々の変動を監視するのに適しています。図6Aは、短い熱ショック後の単一ワームからの複数のhsp転写物の平均発現を示す代表的な結果を示す。図に示すように、転写物の発現のばらつきは遺伝子によって大きく異なった(図6A)。さらなる洞察を得るために、変動係数(CV)を、標準偏差を式レベル26の平均で割って計算した(図6B)。CV値が以前に別の方法で推定された3つの遺伝子を監視した(未発表データ)。2つの安定した転写物(ife-1およびY45F10D.4)と1つの変数(nlp-2927)は、予想される変動性を示した。また、このグラフは、変動性値と式レベル26の間の既知の逆関係を明確に示しています(図6B)。
一括サンプルを使用する際に再現性を確保するために、技術的な複製が最も重要です。しかし、これは必ずしも単一細胞実験14、15、28の場合には当てはまらない。単一ワームサンプルを使用する場合にパラメータ推定に技術的な複製を使用する必要があるかどうかを判断するために、短い熱ショックの後に28個の個々のワームが収穫され、技術的な三重体を使用して処理されました。三重化(図7、技術CVの青い点)で得られた単一ワームデータから算出されたCV値と、個々のワームから得られたすべての転写物(図7の赤い点、生物学的変動性)について比較した。テストされたすべてのトランスクリプトについて、技術的なCVは生物学的CVよりも低く、技術的トリクセートはパラメータ推定に必要でないことを示した。技術的な複製が必要とされないという事実は、品質を損なうことなく実験のスループットを増加させます。

図 1: ワームツーCT プロトコルの概要
この図は、Worm-to-CT プロトコルを使用してワームを実行するために必要なさまざまな手順の概要を示しています。逆転写のステップでは、2 つのオプションの方法が示されています。これらは、どちらのタイプのチップでも交換可能な方法です。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:ナノ流体qPCRの調製と実行の概要
この図は、マルチアレイチップとシングルアレイチップを使用したナノ流体qPCRシステムの実行準備を示しています。 この図の大きなバージョンを表示するには、ここをクリックしてください。
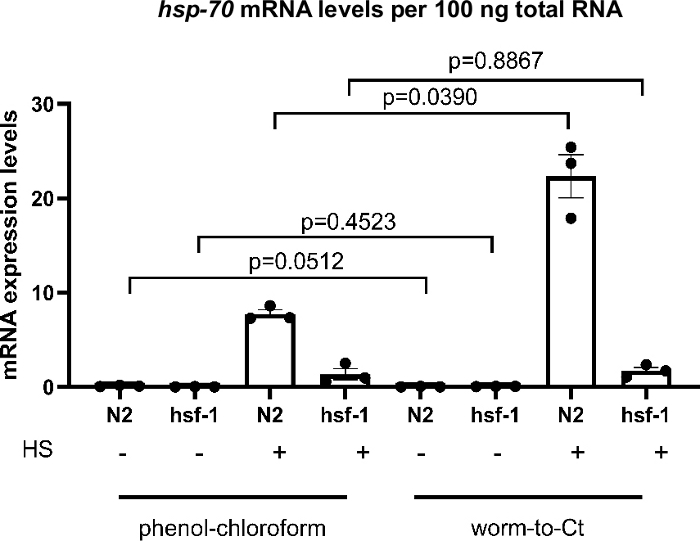
図 3: バルク サンプル上のワームから CT へのプロトコルが、信頼できる結果を提供しました。
バルクサンプル上の通常のグアニジウム・チオシアネート・フェノール・クロロホルム抽出22とのワーム対CTプロトコルの比較。hsf-1(sy441)変異体23における以前の知見と一致して、熱ショックに応答したhsp転写物のレベルは低下した。上記のヒストグラムは、34°Cで30分の短い熱ショック(-)の不在時のhsp-70の誘導を示しています。 cDNAは、1,000ワーム(左)に適用されたグアニジウム・チオシアネート・フェノール・クロロホルム抽出を用いて、または30個のプールワームに適用されたワームからCTへの方法を用いて得られた(右)。各方法で得られた全RNAの100ng当たりhsp-70の発現レベルを比較した。予想通り、hsf-1(sy441)では、熱ショックに応答したhsp-70の転写誘導は、グアニジウム・チオシアネート・フェノールクロロホルムを用いて82.7%有意に減少し、ワーム-to-CT法を用いて92.3%減少した。標的遺伝子からのmRNAレベルを、3つのハウスキーピング遺伝子cdc-42、pmp-3、およびire-1の平均に対して正規化した。 各ドットは、生物学的複製を表す。データは、パラメトリック分析に必要な規則を満たさなかったため、統計分析のためにログ変換されました。統計分析は、シダックの多重比較検定を使用して、RM-One-way ANOVAを使用して行われました。ワイルドタイプ = N2, hsf-1 = hsf-1 (sy441)棒は平均の標準誤差を示します。この図の大きなバージョンを表示するには、ここをクリックしてください。
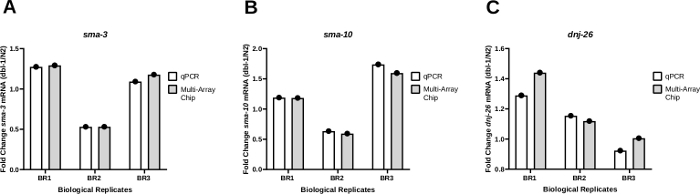
図4:発現パターンは、標準qPCRとナノ流体qPCR系の間で一貫していた。
(A)sma-3(A)、sma-10(B)またはdnj-26(C)mRNAの発現レベルは、野生型株(N2)およびdbl-1(nk3)ノックアウト株25からワーム-CTを介して生成されたcDNAの3つの生物学的複製から、通常のqPCRおよびナノ流体qPCR(マルチアレイチップ)を介して決定された。 デルタCt法21を用いて各株についてmRNA発現レベルを相対的に求めた。次に、dbl-1(nk3)ワームで得られた発現レベルをN2株の対応するmRNAレベルで割ることによって折り畳み変化を求めた。パネルAに示すように、パターンは個々の生物学的複製の両方の方法について一貫していた。(B) および (C) は、 sma-10およびdnj-26 mRNA レベルの場合は、 それぞれ (A) と同じです。標的mRNAレベルは、ハウスキーピング遺伝子cdc-42およびpmp-3に対して正規化した。統計解析は、標準qPCRを介して生成された3つの生物学的複製の結果とナノ流体qPCRを介して生成されたものを比較する対t検定を用いて、各遺伝子について計算した。これらの比較のP値は、それぞれ0.3113,0.2635,0.3481,sma-10,dnj-26であった。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図 5: ワーム対 CT メソッドをバルク サンプルまたは単一ワームに対して使用すると、ワームごとに正規化された場合と同様のレベルの表現が提供されました。
(A) hsp-16.1/11、 (B) F44E5.4, および(C) hsp-70 (C12C8.1) の発現レベルを、熱ショックがない状態で若年成動物で分析した。ワームごとにデータを正規化した場合、両方の方法を使用して各トランスクリプトについてワームごとに得られるレベル間に有意な差はなかった。標的遺伝子からのmRNAレベルを、cdc-42、pmp-3、およびire-1の3つのハウスキーピング遺伝子の平均に対して正規化した。棒は平均の標準誤差を表します。統計 = ペアの t 検定。この図の大きなバージョンを表示するには、ここをクリックしてください。
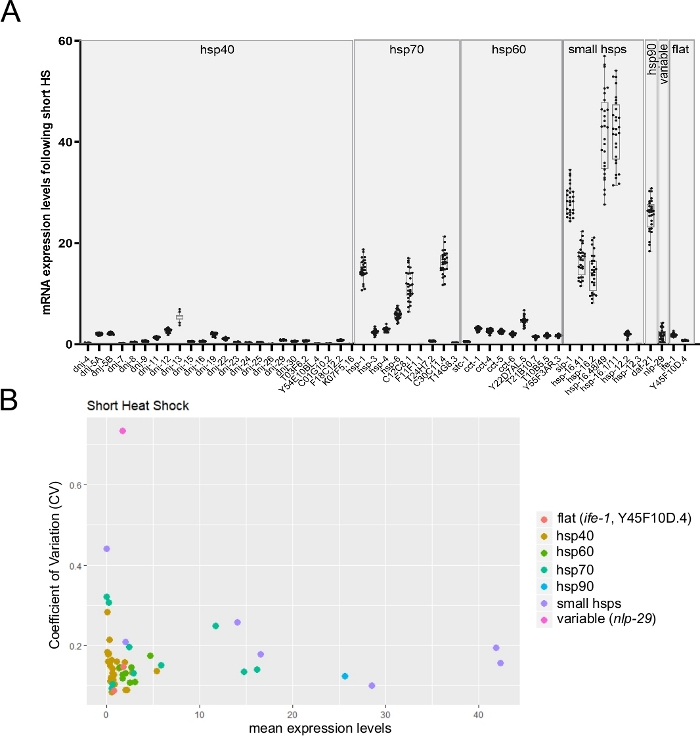
図 6: ワームから CT への方法を使用する単一ワームの高スループット RT-qPCR は、遺伝子発現における個人間の変動を監視できます。
(A)短い熱ショック(34°Cで30分)に曝された場合に得られた53の転写物の平均発現レベル。箱ひげ図は、個々のワームからの平均mRNA発現の分布を表します(個々のワームごとに平均3つの技術的複製が使用されました)。ドットは、28 個の個別ワームの式レベルを表します。標的遺伝子からのmRNAレベルを、cdc-42、pmp-3、およびire-1の3つのハウスキーピング遺伝子の平均に対して正規化した。(B)短い熱ショックへの曝露後の53個の転写物に対する平均mRNA発現の関数としての変動係数26(CV)を、28個の個別の動物から計算した(パネルBに示す生データ)。トランスクリプトのセットには、変数nlp-29トランスクリプト27と 2 つの安定したトランスクリプト(ife-1およびY45F10D.4;未公開データ) が含まれています。CVは、平均に対する標準偏差の比率です。このCVは、個々のワーム間の転写発現における個人間変動を推定するために利用された。予想通り、平均発現レベルの低下に伴って個人間変動が拡大した。この図の大きなバージョンを表示するには、ここをクリックしてください。
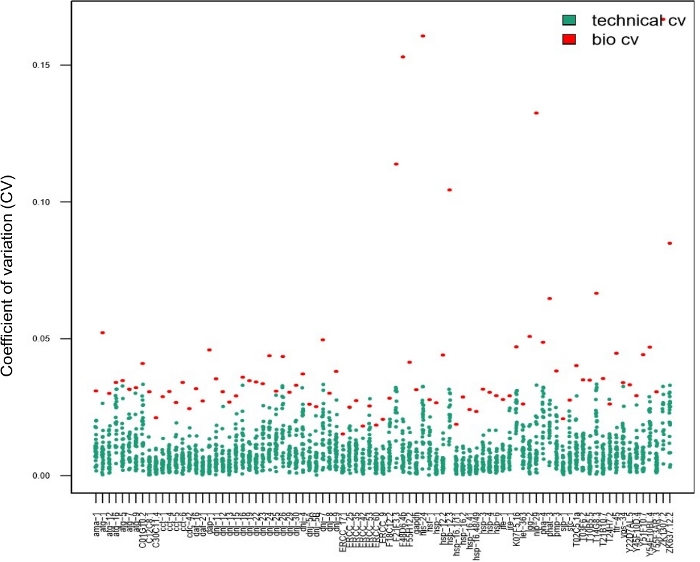
図7:ナノ流体チップを用いた遺伝子発現の個々の変動性を解析する際に技術的複製は必要なかった。
このグラフに示されたデータは、短い熱ショック(34°Cで30分)に続く28個のワームで得られた。各赤い点は、28個のワーム(バイオCV)間でアッセイされた1つの転写物に対する平均転写発現レベルの変動係数(CV)を表す。各青い点は、1つのワームから得られた3つの技術的複製の間の発現レベルのCVを表し、トランスクリプトごとにアッセイ(技術的CV)を示す。このグラフは、技術的な変動(技術的複製間)が生物学的変動性(個々のワーム間)よりもはるかに低いことを示し、単一ワームにおける遺伝子発現をアッシングする際にナノ流体遺伝子発現アレイ上で技術的複製を行う必要がないということを示している。この図の大きなバージョンを表示するには、ここをクリックしてください。
表 1: マルチアレイ チップのレイアウトを計画する 上の表は、マルチアレイチップの実行を計画する際に利用できるシンプルなレイアウトを示しています。左側には、対象となるプライマー ターゲットで埋める必要があるスペースがあり、右側には目的のサンプルを埋める必要があるスペースがあります。各アッセイとサンプルアレイは、チップを介して数的にペア化されます。 こちらの表をダウンロードしてください。
表 2: シングル・アレイ・チップのレイアウトを計画する。 上の表は、シングル・アレイ・チップの実行を計画する際に使用できるシンプルなレイアウトを示しています。左側には、対象となるプライマーターゲットで埋める必要があるスペースがあり、右側には目的のサンプルで埋める必要があるスペースがあります。 こちらの表をダウンロードしてください。
表3:本研究で用いたRT-qPCRプライマーのリストこちらの表をダウンロードしてください。
補足表1:RT-qPCRプライマーのデータベースからのプライマーこちらの表をダウンロードしてください。
ディスカッション
本論文では、ワーム-to-CTプロトコルは、単一ワームまたは小さなワームプールからRNAを抽出するための迅速かつ効率的な方法であることが示されている。ナノ流体システムによって提供されるハイスループットは、個人間変動測定の定量化に最適です。さらに、この方法の高感度により、単一ワームRNA-seq技術9を用いた場合の検出以下に下がる低レベルで発現した遺伝子の検出が可能となる。
単一ワームからcDNAを準備する方法の選択を検討する場合。Ly et al.29は、プロテクチクル消化のためにプロテナーゼKに依存するプロトコルを最適化した。キューティクルは、ワームからの分子の分離のための主要なハードルであり、プロテアーゼKはそれを壊す効果的な方法を提供します。しかし、リテナーゼKは、逆転写に酵素を使用できるように熱不活性化する必要があります。Lyらは96°Cに10分曝露を用いたが、RNAは容易に分解性であるため、このステップはこのプロトコルでは回避された。プロテナーゼKを使用する代わりに、キューティクルを壊すために繰り返し凍結融解サイクルを使用した。凍結融解は、ワームごとにより多くのRNAを単離することができるので、キューティクルを壊す効果的な方法です。Ly et al. 報告では、1ワーム当たり全RNAはプロテナーゼKを用いて35ngであり、このプロトコルはワーム当たりのRNA全体の51.75 ng±6.74 SEMを得る。熱暴露の回避と予備化ステップは、標準的なプロトコルと比較してワームからCTへのダイナミックな検出範囲を広げるようです。Ly et al. hsp-16.2の場合は 21.1 ± 0.15、ヒートショック後のhsp-70の場合は 0.17 ± 22.8 の絶対Ct値を報告する。同じ熱衝撃条件(30°Cで1h)を使用して、このプロトコルはhsp-16.2の場合は0.57±17.93±、hsp-70の場合は21.13±0.33の絶対Ct値を取得します。 これは、凍結融解法がより高いRNAの収率を提供し、低発現転写物に対してより適切であることを示している。
ナノ流体システムは、特定のターゲットトランスクリプトのセットを調査し、より小さい(マルチアレイチップ)またはより大きな(シングルアレイチップ)数のサンプルを使用して実験の規模に適応させる場合に理想的です。単一のワームで発現されるすべての転写物の公平な画像を得るために、明らかな選択はRNAシーケンシングを使用することです。しかし、実験の焦点が小さいがまだ比較的大きな標的遺伝子セットである場合、研究者がナノ流体PCRマシンにアクセスできれば、このプロトコルを利用する方が費用対効果が高い。ナノ流体系試薬とシングルアレイチップのコストはワームあたり約£13と見積もられていますが、シングルワームシーケンシングの試薬のコストは、シーケンシングコストを含まないワームあたり約£60になります。
使用するPCRプラットフォームを検討する場合、ワーム-to-CT法をナノ流体qPCRに結合すると、時間とスループットに関して利点があります。9,216 RT-qPCRの結果は約2日間の作業で得ることができますが、標準的なqPCRプラットフォームを使用して同じ数のターゲットの増幅は、1日に4プレートを実行し、96ウェルプレートアッセイを使用して約5作業週間かかります。ただし、テスト対象の数が少ない場合は、標準の qPCR マシンと組み合わせた Worm-to-CT を使用する方がコスト効率が高くなります。シングルアレイチップは再実行できないため、空の井戸を実行するとコスト効率が低下します。
この方法の1つの制限は、多重工程中のプライマー-プライマーダイマーの形成の可能性であるが、これは1%未満の場合に起こる。ワーム対CTプロトコルは効率的であり、単一のワームに適用すると信頼性の高い結果を提供しますが、収穫工程中にワームがキャップまたはチューブの上部に閉じ込められたままの場合に対応する可能性が高い、約5%の故障率があります。
この多目的で信頼性の高い方法を組み合わせることで、より標準的な技術に比べてスループットと感度が向上します。この方法は、高スループットスクリーンの検証に非常に有用であり、単一ワーム遺伝子発現レベルを監視または検証するのに最適です。この方法は、単離組織からの遺伝子発現量などの他の困難な手法に適用することができます。例えば、腸、生殖腺、FACsによって単離された細胞などの完全な組織の単離は、RNAシーケンシング実験を行うのに十分な材料を提供する。しかし、限られた量の材料は、多くの場合、まれなトランスクリプトの定量を排除する重複読み取りにつながります。このシナリオでは、ナノ流体工学ベースの技術を使用すると、実験に対する感度を高め、研究者がそれらの組織または細胞内のすべてのトランスクリプトのサブセットのみを監視する必要がある場合は、コスト効率を高める必要があります。
開示事項
著者らは開示するものは何もない。
謝辞
シャーリーン・マードックとバブラハム研究所の支援に感謝します。JLPはウェルカムトラスト(093970/Z/10/Z)によってサポートされ、OCはERC 638426とBBSRC[BBS/ E/ B000C0426]によってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| 100µM stock primers for genes of interest. | |||
| 20X DNA Binding Dye | Fluidigm | 100-7609 | for 96.96 chip (Single-Array Chip) |
| 2X Assay Loading Reagent | Fluidigm | 100-7611 | |
| 384 well plates | |||
| 8-strip PCR tubes | |||
| 96 well ice block | |||
| 96 well plates | |||
| 96.96 Dynamic Array IFC | Fluidigm | BMK-M-96.96 | Referenced throughout the manuscript as "Single-Array Chip" |
| 96-well sealing tape | |||
| BioMark & EP1 Software | Fluidigm | 101-6793 | Contains: Biomark HD Data Collection software, Real-Time PCR Analysis software, SNP Genotyping Analysis software, Digital PCR Analysis software, Melt Curve Analysis software |
| BioMark or BioMark HD system | Fluidigm | 100-2451 K1 | Referenced throughout the manuscript as "nanofluidics thermocycler" |
| Control Line Fluid Kit for 96.96 and FlexSix IFCs | Fluidigm | 89000021 | Referenced throughout manuscript as "control line fluid" |
| DNA Suspension buffer | TEKnova | T0021 | |
| domed PCR caps | |||
| Exonuclease I | New England BioLabs | M0293L | |
| Exonuclease I reaction buffer | New England BioLabs | B0293S | |
| FLEXsix DELTAgene Sample Reagent | Fluidigm | 100-7673 | referenced throughout manuscript as "sample reagent" |
| FLEXsix Gene Expression IFC | Fluidigm | 100-6308 | referenced throughout manuscript as "Multi-Array Chip" |
| Fluidigm Real-Time PCR Analysis User Guide | Fluidigm | 68000088 | This is the protocol used as a basis for section 2 of the protocol, referenced within the manuscript. |
| IFC Controller MX | Fluidigm | 68000112 I1 | Referenced throughout the manuscript as "nanofluidics PCR priming machine" |
| IFC Controllers SOFTWEAR | Fluidigm | 100-2297 | Contains all softwear and scripts required for running the IFC controller MX |
| liquid nitrogen in dewar | |||
| Microcentrifuge | StarLab | C1301B-230V | Used to spin down PCR tube strips in the protocol. |
| Nuclease Free Water | |||
| plate spinner | Labnet | K4050725 | mini plate spinner, mps 1000. referenced through the protocol as "tabletop plate spinner" |
| Power SYBR Green Cells-to-Ct kit | invitrogen | 4402955 | This kit is that which is adapted for use in nematodes in the Worm-to-CT protocol. Contense: Store Stop Solution, DNase I and 20X RT Enzyme Mix at -20°C. Store Lysis Solution, 2X SYBR RT Buffer (referenced throughout the manuscript as RT buffer), and Power SYBR®Green PCR Master Mix (refferenced througout the manuscript as PCR Master Mix). This Kit is for 400 reactions, kits also available for 40 and 100 reactions. |
| PreAmp Master Mix | Fluidigm | 100-5580, 100-5581 | |
| Reverse Transcription Master Mix—480 reactions | Fluidigm | 100-6299 | One tube also available for 96 reactions (PN100-6298) |
| Rnase Free water | |||
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad Laboratories | 172-5211 | referenced throughout the manuscript as "fluorescent probe supermix" |
| Standard 96 and 384-well Thermal Cycler | |||
| TE Buffer (10mM Tris, pH8.0, 1.0mM EDTA | TEKnova | T0224 | Referenced throughout the manuscript as Tris-EDTA buffer |
| ThermoMixer C | Eppendorf | 5382000031 | Referenced throughout the text as "thermal mixer" |
| Trizol | Thermo-Fisher | 15596026 | Referenced through the protocol as "guanidium thiocyanate-phenol-chloroform" |
| Warm water bath |
参考文献
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biology. 4 (10), 309 (2006).
- Semrau, S., van Oudenaarden, A. Study lineage decision-making in vitro: emerging concepts and novel tools. Annual Review of Cell Developmental Biology. 31, 317-345 (2015).
- Kelsey, G., Stegle, O., Reik, W. Single-cell epigenomics: Recording the past and predicting the future. Science. 358 (6359), 69-75 (2017).
- Eling, N., Morgan, M. D., Marioni, J. C. Challenges in measuring and understanding biological noise. Nature Review Genetics. 20 (9), 536-548 (2019).
- Kaufmann, B. B., van Oudenaarden, A. Stochastic gene expression: from single molecules to the proteome. Current Opinion in Genetics & Development. 17 (2), 107-112 (2007).
- Corsi, A. K., Wightman, B., Chalfie, M. A Transparent window into biology: A primer on Caenorhabditis elegans. WormBook. , (2015).
- Casanueva, M. O., Burga, A., Lehner, B. Fitness trade-offs and environmentally induced mutation buffering in isogenic C. elegans. Science. 335 (6064), 82-85 (2012).
- Raj, A., Rifkin, A. A., Andersen, E., van Oudenaarden, A. Variability in gene expression underlies incomplete penetrance. Nature. 463 (7283), 913-918 (2010).
- Perez, M. F., Francesconi, M., Hidalgo-Carcedo, C., Lehner, B. Maternal age generates phenotypic variation in Caenorhabditis elegans. Nature. 552 (7683), 106-109 (2017).
- Serra, L., et al. Adapting the Smart-seq2 Protocol for Robust Single Worm RNA-seq. Bio-protocol. 8 (4), 2729 (2018).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Biology. 14 (8), 1007559 (2018).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nature Review Genetics. 16 (3), 133-145 (2015).
- Ståhlberg, A., Kubista, M. The workflow of single-cell expression profiling using quantitative real-time PCR. Expert Review of Molecular Diagnostics. 14 (3), 323-331 (2014).
- Livak, K. J., et al. Methods for qPCR gene expression profiling applied to 1440 lymphoblastoid single cells. Methods. 59 (1), 71-79 (2013).
- Fluidigm. . Real-Time PCR Analysis User Guide. , (2018).
- Huggett, J. F., et al. The digital MIQE guidelines: Minimum Information for Publication of Quantitative Digital PCR Experiments. Clinical Chemistry. 59 (6), 892-902 (2013).
- . U. S. National Library of Reference. Primer Blast Available from: https://www.ncbi.nlm.nih.gov/tools/primer-blast/ (2020)
- Bustin, S. A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems. Journal of Molecular Endocrinology. 29 (1), 23-39 (2002).
- Nolan, T., Huggett, J., Sanchez, E. . Good Practice guide for the application of quantitative PCR (qPCR), LGC. , (2013).
- Yuan, J. S., Reed, A., Chen, F., Stewart, C. N. Statistical analysis of real-time PCR data. BMC Bioinformatics. 22 (7), 85 (2006).
- He, F. Total RNA Extraction from C. elegans. Bio-protocol. Bio-101. 47, (2011).
- Hajdu-Cronin, Y. M., Chen, W. J., Sternberg, P. W. The L-Type Cyclin CYL-1 and the Heat-Shock-Factor HSF-1 Are Required for Heat-Shock-Induced Protein Expression in Caenorhabditis elegans. Genetics. 168 (8), 1937-1949 (2004).
- Douglas, P. M., et al. Heterotypic Signals from Neural HSF-1 Separate Thermotolerance from Longevity. Cell Reports. 12 (7), 1196-1204 (2015).
- Zhang, X., Zhang, Y. Dbl-1 a TGF-beta is essential for C. elegans aversive olfactory learning. Proceedings of the National Academy of Sciences of the United States of America. 109 (42), 17081-17086 (2012).
- Sørensen, J. B. The Use and Misuse of the Coefficient of Variation in Organizational Demography Research. Sociological Methods & Research. 30 (4), 475-491 (2002).
- Pujol, N. Distinct innate immune responses to infection and wounding in the C. elegans epidermis. Current Biology. 18 (7), 481-489 (2008).
- Brennecke, P., et al. Accounting for technical noise in single-cell RNA-seq experiments. Nature Methods. 10 (11), 1093-1095 (2013).
- Ly, K., Reid, S. J., Snell, R. J. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 7 (2), 59-63 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved