Method Article
Rt-PCR quantitatif à haut débit dans les échantillons de C. elegans en vrac et en vrac à l’aide de la technologie nanofluidique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Dans cet article, un protocole à haut débit pour une détermination rapide et fiable des niveaux d’expression des gènes dans des échantillons de C. elegans en vrac ou en vrac est décrit. Ce protocole n’exige pas l’isolement d’ARN et produit l’ADND directement à partir d’échantillons. Il peut être utilisé avec des plates-formes qPCR nanofluidiques multiplexées à haut débit.
Résumé
Cet article présente un essai quantitatif de transcription inverse à haut débit PCR (RT-qPCR) pour Caenorhabditis elegans qui est rapide, robuste, et très sensible. Ce protocole obtient des mesures précises de l’expression des gènes à partir de vers simples ou d’échantillons en vrac. Le protocole présenté ici fournit une nouvelle adaptation des méthodes existantes pour la préparation complémentaire de l’ADN (CDNA) couplée à une plate-forme nanofluidique RT-qPCR. La première partie de ce protocole, appelée « Ver-to-CT », permet la production d’ADNd directement à partir de nématodes sans avoir besoin d’isolement antérieur de l’ARNm. Il augmente le débit expérimental en permettant la préparation de l’ADND à partir de 96 vers en 3,5 h. La deuxième partie du protocole utilise la technologie nanofluidique existante pour exécuter rt-qPCR à haut débit sur l’ADND. Cet article évalue deux puces nanofluidiques différentes : la première exécute 96 échantillons et 96 cibles, ce qui entraîne 9 216 réactions en environ 1,5 jour de travail sur le banc. Le deuxième type de puce se compose de six tableaux de 12 x 12, ce qui entraîne 864 réactions. Ici, la méthode ver-to-CT est démontrée en quantifiant les niveaux d’ARNm des gènes codant les protéines de choc thermique à partir de vers simples et d’échantillons en vrac. Une longue liste d’amorces conçues pour amplifier l’ARN traité pour la majorité des gènes codants dans le génome de C. elegans est fournie.
Introduction
L’optimisation du séquençage de l’ARN unicelliste et qPCR a révélé que les impulsions transcriptionnelles ou les éclats peuvent conduire à une variation massive du nombre de molécules d’ARN par cellule1. En outre, ces technologies ont découvert l’hétérogénéité cellulaire substantielle précédemment manquée par les mesures transcriptomiques en vrac standard. Selon le contexte, une certaine variabilité transcriptionnelle à cellules simples est causée par une composition cellulaire mixte des tissus. Cependant, même dans les populations de cellules isogéniques cultivées dans le même environnement, il existe une hétérogénéité transcriptionnellegénéralisée 2,3. Cette « variabilité biologique » est de plus en plus identifiée comme une propriété omniprésente des réseaux cellulaires, des bactéries à l’homme. Dans certains cas, il peut avoir des conséquences phénotypiques dans le développement, la progression du cancer, la latence du VIH et laréponse à la chimiothérapie 4,5.
Le nématode Caenorhabditis elegans est un organisme modèle unique avec des caractéristiques idéales pour étudier les causes et les conséquences de la variabilité biologique entre les individus. Ces nématodes sont un organisme modèle simple composé de 959 cellules, et leur cuticule transparente les rend agréables pour les études d’imagerie in vivo6. C. elegans est une espèce hermaphroditique qui se reproduit principalement par auto fertilisation; il en est résulté des souches isogéniques en laboratoire. Malgré l’isogénicité et les conditions de culture contrôlées, de nombreux phénotypes et transcriptions sont variables d’un individus à l’autre, ce qui suggère que les différences stochastiques ou microenvironnementales contribuent à l’hétérogénéitéchez les individus 7,8. Une telle variabilité dans l’expression des gènes a de multiples conséquences de forme physique, y compris la variabilité dans la pénétration des mutations, la survie, le moment du développement, et lafécondité 7,8,9. En raison de ces caractéristiques, les études sur les vers uniques offrent l’occasion sans précédent d’étudier la variabilité biologique dans tout un organisme.
Il y a un besoin fondamental sur le terrain de développer et d’optimiser des technologies pour la détection précise des transcriptions à un niveau d’un seul ver. Les nouvelles technologies, telles que le séquençage de l’ARN à un ver10,le séquençage de l’ARN à partir de tissusisolés 11,et le séquençage à cellulessimples 12 sont maintenant disponibles pour C. elegans. Cependant, un défi majeur demeure : lors de la surveillance de l’interindividualité, les gènes faiblement exprimés tombent souvent en dessous des niveauxdétectables 13. Ceci est particulièrement pertinent pour les transcriptions rares isolées de petites quantités de matériel de départ, car il existe une relation bien établie et inverse entre l’expression moyenne et la variance technique, ce qui fait souvent tomber de rares transcriptions en dessous des seuilsstatistiques 13. L’optimisation des technologies qPCR multiplexées à haut débit s’est avérée utile pour les études sur les mammifères unicell cellules, en particulier lors de l’étude de l’expression de transcriptionsrares 14,15. Cette technologie peut également être utilisée à des fins d’analyse comparative et de validation d’autres technologies à vers unique.
Worm-to-CT est une méthode rapide et robuste adaptée d’un kit utilisé dans les études de biologie cellulaire, pour la préparation de l’ADN à un seul ver. cDNA obtenu par cette méthode couplée à la technologie qPCR nanofluidique multiplexée a été choisi parce qu’il fournit un débit expérimental plus élevé, une gamme dynamique plus large de détection et a été validé à des fins unicelliques14,15. La préparation de l’ADND décrite est également applicable aux technologies PCR standard. Le débit est augmenté de deux façons : premièrement, la préparation de l’ADN est plus rapide et plus fiable que l’extraction traditionnelle de thiocyanate-phénol-chloroforme de guanidium, parce que les vers sont directement ajoutés au tampon de lyse, en sautant l’isolement direct de l’ARN facilement dégradable. Deuxièmement, l’utilisation de technologies nanofluidiques augmente considérablement le nombre d’échantillons et de cibles qui peuvent être exécutés simultanément. Dans ce document, deux puces sont comparées : une puce à un seul tableau et une puce multi-tableaux. Une puce à réseau unique peut exécuter 96 vers simples et 96 ensembles d’amorce, ce qui entraîne 9 216 réactions par expérience. Pour obtenir un débit similaire à l’aide des technologies qPCR standard, il faudrait 96 expériences qPCR distinctes, utilisant 96 plaques de puits. La puce multi-tableaux plus petite et plus flexible se compose de six tableaux de 12 x 12, ce qui entraîne 864 réactions. La fiabilité et la sensibilité supérieures de la méthode sont renforcées par la technologie nanofluidique et par l’introduction d’une étape de préamplification. La méthode présentée dans cet article est destinée à être utilisée avec un algorithme statistique de pointe pour extraire la variance biologique. Cet article présente le protocole pour la préparation rapide de cDNA et le qPCR à haut débit pour les échantillons de ver simple et de ver de lot ; l’algorithme sera publié ailleurs. Pour ce protocole, l’organisation de chaque puce doit être préparée avant l’expérience. Les tableaux 1 et 2 montrent des exemples de ces plans pour une puce multi-tableaux et à un seul tableau, respectivement. Il y a également des aperçus du protocole Worm-to-CT détaillés à la figure 1 et exécutant les puces multi-tableaux et mono-tableaux de la figure 2.
Protocole
NOTE: Tout au long de ce protocole Caenorhabditis elegans est appelé « ver » ou « vers ». Une variété de souches C. elegans peuvent être commandées par le biais de bases de données en ligne ou en contactant directement les laboratoires qui utilisent l’organisme modèle. La partie I de ce protocole (sections 1−3) décrit la préparation de l’ADNT par le protocole Ver-to-CT. La partie II de ce protocole (sections 4−13) décrit l’exécution de RT-qPCR à haut débit à l’aide de nanofluidiques, adapté d’un protocole développé par Fluidigm16. Ce protocole s’applique à l’utilisation des deux types de puces nanofluidiques définies précédemment, la puce à réseau unique, qui peut surveiller 96 cibles en 96 échantillons (9 216 réactions RT-qPCR au total), ou la puce multi-tableaux, qui fonctionne comme sous-unité de 12 échantillons cibles x 12. Chaque puce multi-tableaux contient six tableaux indépendants qui peuvent être exécutés ensemble ou séparément. Par exemple, l’utilisation d’une puce multi-table entière peut surveiller 72 cibles x 12 échantillons (ou vice versa), ou 36 cibles x 24 échantillons (ou vice versa). Pour de plus amples renseignements sur l’un ou l’autre des documents utilisés dans ce protocole, consultez le Tableau des matériaux.
1. Validation de l’amorce RT-qPCR
REMARQUE : Les amorces en temps réel ont été conçues en fonction des propriétés recommandées initialement émises par les lignes directrices du MIQUE17. Pour rendre les amorces spécifiques à l’ARN transformé, les produits ont été conçus de telle sorte que les deux amorces sont liées de chaque côté d’au moins une jonction d’épissage. Les exigences relatives aux amorces appropriées comprenaient une teneur en guanine et cytosine de 20 %−80 %, une température de fusion de 58−60 °C, une différence de température de fusion entre les paires d’amorce de ≤0,5 °C et une longueur de produit de 70 à 120 pb. La séquence des amorces générées se trouve dans le tableau supplémentaire 1. Le code open source pour les scripts utilisés pour générer les amorces peut être trouvé à https://github.com/s-andrews/wormrtpcr. Paires d’amorce pour les transcriptions avec des sites d’épissage ont été conçus de sorte qu’ils se trouvent dans deux exons flanquant un intron, mais n’ont pas été conçus pour être épissage-variante spécifique, en utilisant NCBI Primer Blast logiciel18. Pour cette étude, les ensembles d’amorce ont été soufflés contre le génome de C. elegans pour tester toute complémentarité hors cible.
- Récupérez les amorces de la base de données des amorces RT-qPCR( Tableau supplémentaire 1). Alternativement, concevoir des paires d’amorce qPCR en utilisant des outils en ligne tels que NCBI Primer Blast18.
- Effectuez une courbe standard qPCR pour surveiller la spécificité et l’efficacité du PCR pour chaque paire d’amorce en utilisant les techniques qPCR en vrac standard19 et en suivant les directives MIQUE17,20.
REMARQUE : Seules les paires d’amorce avec R2 > 0,98 et l’efficacité pcr entre 85% et 115% devraient être utilisées. La séquence, l’efficacité du PCR et le R2 pour les amorces utilisées dans cette étude sont détaillés dans le tableau 3.
2. Lyse de ver par worm-to-CT
- Choisissez les vers de leur pelouse bactérienne sur une plaque NGM fraîche et non enseillée et laissez les vers se déplacer autour de la plaque pendant 5 min pour éliminer la plupart des bactéries du ver par son mouvement.
REMARQUE : La pelouse bactérienne et les conditions de croissance varieront selon la conception expérimentale. Les expériences présentées ici nécessitent des plaques NGM de 6 cm ensemencées avec OP50 Escherichia coli avec des vers d’intérêt cultivés au stade L4.9 dans un incubateur de 20 °C. - Dans une hotte exempte de RNase, préparez un mélange principal composé de 12,5 μL de tampon RT 2x, de 1,25 μL de tampon enzymatique RT 20x et de 0,25 μL d’eau sans nucléase par échantillon. Ajouter 14 μL de mélange principal à 11 μL de chaque échantillon à partir de l’étape 2.8.
- Placez le couvercle d’une bande PCR à l’envers sur la plate-forme de la portée de dissection et ajoutez 10 μL du mélange de lyse aux bouchons de tube PCR bombés sous un microscope composé.
REMARQUE : Avec seulement un ou deux échantillons, il est préférable d’utiliser une bande PCR contenant au moins quatre tubes, car cela réduit le risque que les bouchons s’ouvrent dans les étapes subséquentes de gel-dégel. Alternativement, des élastiques peuvent être utilisés pour maintenir les bouchons en place. Dans ce cas, assurez-vous que les bouchons sont correctement fermés chaque fois que les tubes sont transférés. - Choisissez les vers de la plaque dans chaque fente du couvercle contenant le mélange de lyse en les « ramassant » (c.-à-d. attraper les vers en dessous avec la pioche) pour éviter la contamination bactérienne. Fermez les tubes et faites-les tourner vers le bas pendant 5 s à l’aide d’une microcentrifugeuse de table (Table of Materials) avant de les placer dans un flacon Dewar rempli d’azote liquide.
REMARQUE : Entre 15 et 30 vers devraient être utilisés pour des expériences en vrac et 1 ver pour les mesures d’un seul ver.
MISE EN GARDE : Lorsque vous manipulez de l’azote liquide, portez des cryo-gants ainsi que des lunettes de protection et respectez les règlements vestimentaires standard, car le contact avec la peau ou les yeux peut causer de graves blessures causées par des engelures. - Décongeler-décongeler les tubes PCR 10x en les transférant entre de l’azote liquide et un bain d’eau d’environ 40 °C. Laissez les tubes dans l’azote liquide pendant au moins 5 s pour vous assurer que les échantillons sont complètement congelés. Laissez les tubes dans le bain d’eau jusqu’à ce que les échantillons dégèlent. Ne partez pas plus longtemps, car cela entraîne une dégradation de l’ARN.
REMARQUE : Les tubes peuvent être laissés dans de l’azote liquide pendant une période prolongée, car les échantillons sont congelés à environ -200 °C, ce qui réduit la dégradation de l’ARN. Cela ne devrait toutefois pas être un point d’arrêt protocolaire, car l’azote liquide s’évapore rapidement. - Mélanger les échantillons sur un mélangeur thermique (Table of Materials) fixé à 4 °C pendant 20−30 min en rotation à ~1 800 rpm.
- Pendant que les échantillons sont mélangés, décongelez la solution d’arrêt sur la glace.
- Faites tourner les échantillons vers le bas à l’aide d’une microcentrifugeuse de table(Tableau des matériaux)et ajoutez 1 μL de solution d’arrêt à chaque tube.
REMARQUE : Les échantillons peuvent être laissés à -80 °C jusqu’à 1 semaine avant de transcrire l’ARN (section 3).
3. Transcription inversée
REMARQUE : Pour la transcription inversée des vers simples, les résultats présentés ici ont été générés à l’aide des réaugmentés fournis avec les puces nanofluidiques (option 2 de la figure 1). Les reagents mis en évidence dans l’option 2 de la figure 1 ont également été utilisés pour la transcription inversée d’échantillons mis en commun. L’une ou l’autre méthode fonctionne de façon interchangeable pour les différents types d’échantillons.
- Transcription inversée de vers simples
- Dans une hotte sans RNase, ajouter 1,25 μL de mélange de transcription inversée(Table of Materials)à un tube PCR frais.
REMARQUE : Une plaque PCR de puits 96 et une pipette automatique peuvent être utilisées s’il y a beaucoup d’échantillons. Le protocole du fabricant stipule que 1 μL peut être utilisé par échantillon. - Prenez 5 μL de la solution de lyse et arrêtez le mélange de solution de l’étape 2.8 et ajoutez-le au tube PCR frais contenant le mélange de transcription inverse.
REMARQUE : Le protocole du fabricant stipule que 1 μL d’ARN (2,5 pg/μL-250 ng/μL) peut être utilisé par réaction. Un contrôle rt négatif par plaque peut être ajouté en remplaçant le mélange de transcription inverse par 5 μL d’échantillon lysé et 1,25 μL d’eau sans RNAse. - Exécutez les échantillons à l’aide du programme de transcription inverse suivant sur un thermocycleur : 25 °C pendant 5 min, 42 °C pendant 30 min, 85 °C pendant 5 min et 4 °C pendant ∞.
REMARQUE : L’ADNc produite peut être stockée à -20 °C avant de passer à l’amplification et à la collecte de données à l’aide d’un QPCR à haut débit.
- Dans une hotte sans RNase, ajouter 1,25 μL de mélange de transcription inversée(Table of Materials)à un tube PCR frais.
- Transcription inversée pour les échantillons en vrac (15−30 vers)
- Dans une hotte exempte de RNase, préparez un mélange principal composé de 12,5 μL de tampon RT 2x, de 1,25 μL de tampon enzymatique RT 20x et de 0,25 μL d’eau sans nucléase par échantillon. Ajouter 14 μL de mélange principal à 11 μL de solution de lyse et arrêter le mélange de solutions à partir de l’étape 2.8.
REMARQUE : Lorsqu’il s’agit d’un grand nombre d’échantillons, cela peut être effectué dans 96 plaques de puits. - Exécutez les échantillons à travers un thermocycleur en utilisant le programme de transcription inverse suivant : 37 °C pendant 60 min, 95 °C pendant 5 min et 4 °C pour ∞.
- Diluer le cDNA produit 1:4 dans de l’eau sans nucléase.
REMARQUE : En général, les produits arrivent à un volume final de 25 μL, auquel cas 75 μL doivent être ajoutés. Cependant, en raison de la condensation, le volume final peut varier. Par conséquent, ajustez-vous en conséquence pour faire un rapport de 1:4 dans la solution finale. Cette étape de dilution ne s’applique pas si vous effectuez qPCR sur les vers simples. L’ADNc produite peut être stockée à -20 °C avant de passer à l’amplification et à la collecte de données à l’aide d’un QPCR à haut débit.
- Dans une hotte exempte de RNase, préparez un mélange principal composé de 12,5 μL de tampon RT 2x, de 1,25 μL de tampon enzymatique RT 20x et de 0,25 μL d’eau sans nucléase par échantillon. Ajouter 14 μL de mélange principal à 11 μL de solution de lyse et arrêter le mélange de solutions à partir de l’étape 2.8.
4. Préparation du mélange d’amorce multiplexe
- Préparer un stock d’amorce avant/arrière (F/R) pour chaque paire d’amorce à une concentration finale de 50 μM. Mélanger le même volume d’amorce avant et arrière à 100 μM chacun.
- Combinez 1 μL de 50 μM de stock d’amorce F/R pour que chaque paire d’amorce soit testée. Ajouter le tampon de suspension d’ADN jusqu’à un volume total de 100 μL.
REMARQUE : Les concentrations d’amorce des stocks diffèrent ici de celles décrites dans le protocole16 du fabricant, mais conservent la même concentration finale de 500 nM.
5. Cibler la préamplification spécifique
- Préparez un mélange maître contenant 1 μL de mastermix de préamplification(tableau des matériaux),0,5 μL du mélange d’amorce mis en commun (étape 4.2), et 2,25 μL d’eau sans nucléase par réaction avec un volume excédentaire global de 10 %.
- Dans une plaque de puits de 96, aliquot 3,75 μL du mélange principal dans autant de puits que nécessaire pour le nombre d’échantillons à exécuter.
- Ajouter 1,25 μL des solutions d’intérêt cDNA générées à l’étape 3.1.3 ou 3.2.3 à chaque puits.
- Couvrir la plaque de 96 ruban adhésif bien scellant, brièvement vortex et centrifugeuse à l’aide d’une essoreuse de plaque de table. Transfert à un thermocycleur et exécuter le programme suivant: 95 °C pendant 2 min, 15 cycles de dénaturation à 95 °C pour 15 s, annealing / extension à 60 °C pendant 4 min, et 4 °C pour ∞.
REMARQUE : Le fabricant recommande à partir de 10−20 cycles pour la réaction de préamplification16. Ce protocole recommande 10 ou 15 cycles selon les niveaux d’expression des gènes cibles.
6. Traitement Exonuclease I
REMARQUE : Il s’agit d’éliminer les amorces non incorporées de la préamplification.
- Préparer un mélange exonucléase I contenant 0,2 μL d’exonucléase I tampon de réaction (Tableau des matériaux), 0,4 μL d’exonuclease I à 20 U/μL ( Tableau desmatériaux), et 1,4 μL d’eau sans nucléase par échantillon. Gardez tous les reagents sur la glace, en particulier l’exonucléase I.
- Retirez la plaque de puits 96 (étape 5.4) du thermocycleur, centrifugeuse avec la essoreuse de plaque de table, et retirez soigneusement le joint. Ajouter 2 μL de l’exonucléase que je mélange à chaque réaction de préamplification. Reseal, centrifugeuse, et remettre la plaque de puits 96 dans le thermocycleur en utilisant le programme suivant: 37 °C pendant 30 min, 80 °C pendant 15 min, et 4 °C pour ∞.
- Sortez les échantillons du thermocycleur et diluez-les 1:5 en ajoutant 18 μL de tampon 1x Tris EDTA (Tableau des matériaux).
REMARQUE : Il est possible de maintenir les échantillons d’ADN à -20 °C pour une utilisation ultérieure. Le protocole du fabricant suggère des dilutions potentielles de 5x, 10x ou 20x à cestade 16,selon le niveau d’expression des cibles d’intérêt.
7. Préparation des mélanges d’essai
REMARQUE : Les mélanges d’analyse peuvent être préparés dans 384 plaques de puits, car les puits ont le même espacement que les copeaux nanofluidiques, ce qui facilite le chargement.
- Préparation de mélanges d’analyse pour une puce multi-tableaux
- Préparer un mélange principal composé de 2 μL de réagenage de chargement de 2 x essai(tableau des matériaux)et de 1,6 μL de tampon de suspension d’ADN(tableau des matériaux)pour chaque puits selon le plan préparé. Aliquot 3,6 μL de ce mélange maître par puits dans une plaque de puits 384.
- Ajouter 0,4 μL du stock d’amorce F/R de 50 μM préparé à l’étape 4.1 aux puits appropriés selon le plan préparé.
REMARQUE : Cela donne un total de 4 μL de mélange d’analyse par puits, avec un excédent de 1 μL.
- Préparation des mélanges d’analyse pour une puce à un seul tableau
- Préparer un mélange principal composé de 3 μL de réageni de chargement d’analyse de 2 x et de 2,4 μL de tampon de suspension d’ADN pour chaque puits selon le plan préparé. Aliquot 5,4 μL de ce mélange maître par puits dans une plaque de puits marquée 384.
- Ajouter 0,6 μL du stock d’amorce F/R de 50 μM aux puits appropriés selon le plan préparé.
REMARQUE : Cela donne un total de 6 μL de mélange d’analyse par puits, avec un excédent de 1 μL.
8. Préparation des mélanges d’échantillons
REMARQUE : Les mélanges d’échantillons peuvent être préparés jusqu’à 1 jour à l’avance et conservés à 4 °C.
- Préparation d’échantillons pour une puce multi-tableaux
- Préparer un mélange maître d’échantillon composé de 2 μL de supermixe fluorescent de sonde 2 x avec un faible ROX (Tableau des matériaux) et 0,2 μL de réageni d’échantillon(tableau des matériaux)par échantillon. Distribuez 2,2 μL de ce mélange dans la plaque de puits marquée 384.
REMARQUE : Le fabricant recommande de ne pas vortexer le réaccente del’échantillon 16. - Pipette 1,8 μL de chaque échantillon préampliifié et exonucléase J’ai traité l’échantillon de l’étape 3.2.3 dans les puits appropriés selon le plan préparé.
REMARQUE : Cela donne un total de 4 μL, avec un excédent de 1 μL.
- Préparer un mélange maître d’échantillon composé de 2 μL de supermixe fluorescent de sonde 2 x avec un faible ROX (Tableau des matériaux) et 0,2 μL de réageni d’échantillon(tableau des matériaux)par échantillon. Distribuez 2,2 μL de ce mélange dans la plaque de puits marquée 384.
- Préparation d’échantillons pour une puce à réseau unique
- Préparer un mélange maître d’échantillon composé de 3 μL de supermixe fluorescent de sonde 2x avec un faible ROX (Tableau des matériaux) et 0,3 μL de 20 x échantillon de colorant liant l’ADN reagent de chargement ( Tableau desmatériaux) par échantillon. Distribuez 3,3 μL de ce mélange dans la plaque de puits marquée 384.
- Pipette 2,7 μL de chaque échantillon préampliifié et exonucléase J’ai traité l’échantillon de l’étape 6.3 dans les puits appropriés selon le plan préparé.
REMARQUE : Cela donne un total de 6 μL, avec un excédent de 1 μL. S’il y a des puits à faire fonctionner sans échantillon, ceux-ci doivent être chargés d’un mélange maître d’échantillon et de 2,7 μL d’eau au lieu de l’ADND. Ceci est recommandé pour les deux types de puces. La machine a besoin d’un ROX faible dans chaque entrée afin de détecter la grille de la puce.
9. Amorçage de la puce nanofluidique
REMARQUE : Une puce multi-tableaux n’a besoin d’être amorcée qu’à la première manche. S’il y a des courses ultérieures de la même puce, cette étape peut être ignorée. Ces étapes sont les mêmes pour les deux types de puces.
- Lentement et soigneusement, injecter le plein 150 μL du fluide de la ligne de commande à partir des seringues incluses dans les accumulateurs de la puce. Assurez-vous qu’aucun liquide de commande ne touche la puce en tenant la puce à un angle de 45° et en retenant l’extrémité de la seringue pour éviter le déversement.
- Retirez le film de protection bleu du bas de la puce.
- Placez la puce dans la machine d’amorçage PCR nanofluidique(Table of Materials),avec le code à barres orienté vers l’extérieur. Exécutez lescript ' Prime (153x),quiprend ~15-20 min.
REMARQUE: Allumez le thermocycleur nanofluidique (Table of Materials) à ce stade, parce que la caméra prend environ 10 min pour refroidir à moins de 0 °C.
10. Chargement de la puce nanofluidique
- Retirez les bouchons de barrière séquentiellement au fur et à mesure que le chargement a lieu. Cela réduit le risque de déchargement des puits.
- Transférez soit 3 μL pour une puce multi-table, soit 5 μL pour une puce à un seul tableau de chaque mélange d’essai d’amorce et des mélanges d’échantillons aux entrées correspondantes de la puce nanofluidique selon le plan préparé. Assurez-vous de ne pas introduire de bulles, ce qui peut rendre le volume total transféré plus petit que souhaité.
REMARQUE : S’il y a des puits qui seront exécutés sans amorce, il est important qu’ils soient chargés avec le mélange principal, remplaçant le volume d’amorce avec de l’eau. Cela s’applique aux deux types de puces. À ce stade, il peut être plus facile de placer la puce sur une surface sombre, ce qui permettra de voir les puits plus facilement.
11. Exécution de la puce nanofluidique
REMARQUE : La première fois que vous exécutant une puce multi-tableaux, configurez le fichier de suivi en sélectionnant Tools | Flex Six Utilisation Tracking, cliquez sur Nouveau, entrez un nom de fichier, et sélectionnez un emplacement avant de cliquer sur Done.
- Ouvrez le logiciel de collecte de données. Cliquez sur Démarrer new run. Placez la puce chargée dans le thermocycleur nanofluidique avec le code à barres orienté vers l’extérieur.
- Choisissez le paramètre de projet s’il y a lieu, puis cliquez sur | Charge. Si vous chargez une puce multi-tableaux, sélectionnez les partitions (tableaux) à exécuter.
- Sélectionnez les sondes de référence d’application,puis modifiez le type d’application en Expressiongénétique et modifiez la référence passive à ROX. Sélectionnez l’analyse de sonde unique,changer le type de sonde en Eva Green, et cliquez sur Suivant.
- Sélectionnez le protocole de cycle thermique GE FLEX six Fast PCR+Melt v1 pour exécuter une puce multi-table, ou le protocole GE 96.96 Fast PCR+Melt v2 pour exécuter une puce à réseau unique.
- Confirmez que l’exposition automatique est sélectionnée et cliquez sur Démarrer la course.
12. Course de jetons postaux
REMARQUE : Cette section n’est nécessaire que pour les puces multi-tableaux lorsque vous n’utilisez pas la puce entière.
- Sortez la puce du thermocycleur nanofluidique et chargez-la dans la machine d’amorçage PCR nanofluidique et exécutez le script Post Run (153x), qui dure 5 min.
- Étiquetez les bouchons utilisés pour la référence personnelle.
REMARQUE : La puce peut maintenant être stockée à température ambiante et les tableaux restants sur la puce peuvent être exécutés dans les 2 mois.
13. Nettoyage et analyse des données
- Ouvrez les données dans lelogiciel « Analyse PCR entemps réel »(Tableau des matériaux). Vérifiez la température de pointe de fusion pour chaque paire d’amorce testée. Éliminez les résultats présentant plus d’un pic de température de fusion, pour une paire d’amorce donnée.
REMARQUE : Plusieurs pics n’apparaissent qu’occasionnellement, vraisemblablement lorsque les paires d’amorces forment des dimers, ou à partir d’interactions d’amorces cibles avec d’autres amorces dans le mélange d’amorce mis en commun. - Exportez les données sous la forme d’un fichier de feuille de calcul « heatmap » et éliminez les échantillons ou amorce échoués.
- Analyser les données à l’aide de la méthode Delta-Ct standard21. Pour l’évaluation statistique effectuer anova à sens unique sur les niveaux d’expression relatifs.
Résultats
Validation du ver au CT comme méthode de préparation de l’ADNC
Pour vérifier si le protocole de ver-à-CT est une méthode valide d’extraction de cDNA, il a été comparé aux méthodes standards d’extraction de thiocyanate-phénol-chloroforme de guanidium. Les résultats sont présentés à la figure 3, où l’ADNC a été préparée à partir d’une moyenne d’environ 1 000 vers utilisant des techniques standard d’extraction du thiocyanate-phénol-chloroforme22 et de 30 vers utilisant la méthode ver-to-CT. Les échantillons ont été choqués simultanément par la chaleur (30 min à 34 °C). À l’échelle mondiale, les niveaux d’expression de l’ARNm hsp-70 par 100 ng d’ARN total étaient comparables en utilisant les deux méthodes. Toutefois, dans le cas de l’expression hsp-70 la plus élevée (c.-à-d. dans le N2 suivant le choc thermique), les niveaux d’expression étaient plus élevés avec la méthode du ver au CT, ce qui indique une sensibilité accrue.
Pour déterminer si une diminution prévue de l’expression de hsp dans hsf-1 (sy441)23,une mutation dans le régulateur transcriptionnel principal des chaperons moléculaires23,24,pourrait être reproduite, induction transcriptionnelle de chaperon suivant un choc thermique bref a été comparée. Avec les deux méthodes une diminution de l’induction de hsp-70 a été détectée chez les animaux hsf-1 (sy441). On s’y attendait, parce que les animaux mutants hsf-1 (sy441) présentent une capacité diminuée d’induire des chaperons en raison d’une troncature dans le domaine de transactivation de HSF-1. Dans le cas du guanidium thiocyanate-phénol-chloroforme, l’extraction hsp70 a diminué de 82,7 % par rapport aux témoins et de 92,3 % pour le ver au CT par rapport aux animaux de type sauvage (figure 3). Les résultats étaient comparables entre les deux méthodes et comparables aux rapportsprécédents 23. Ces résultats indiquent que la méthode Worm-to-CT est une alternative valable aux techniques de synthèse standard de l’ADN.
Validation de la plate-forme PCR nanofluidique utilisée pour amplifier les cibles d’ARNm
Pour tester la cohérence des résultats à l’aide d’un qPCR nanofluidique pour l’amplification des transcriptions, les résultats du PCR obtenus à partir de la méthode worm-to-CT en vrac ont été comparés à la fois sur un système qPCR standard(Tableau des matériaux)et un système qPCR nanofluidique à l’aide d’une puce multi-tableaux. Le changement de pli dans l’expression de trois gènes différents, sma-3 (Figure 4A), sma-10 (Figure 4B), et dnj-26, a été surveillé (Figure 4C) chez les animaux portant un allèle nul en dbl-1 (dbl-1 (nk3))25 par rapport à leurs homologues de type sauvage. Dbl-1 code le seul ligand de la voie de signalisation de la protéine morphogénétique osseuse (BMP). sma-3 et sma-10 sont des gènes codant orthologues SMAD, composants clés de la cascade de signalisation BMP. Dnj-26 code un chaperon moléculaire, une cible de signalisation BMP. Ces résultats montrent peu ou pas de différence dans le changement de pli comparant les résultats des deux méthodes, résultant en des valeurs P non significatives à 0,3113, 0,2635, et 0,3481 pour sma-3, sma-10, et dnj-26, respectivement. Dans l’ensemble, ces résultats montrent que la méthode Worm-to-CT appliquée aux échantillons en vrac est un moyen efficace et rapide d’extraire l’ARN de quelques vers et fournit des données fiables lorsqu’elle est couplée avec des systèmes PCR standard ou des plates-formes qPCR à haut débit basées sur les nanofluidiques.
Comparaison entre les niveaux d’expression obtenus par des échantillons en vrac et les moyennes obtenues à partir de vers simples
Les niveaux d’expression relatifs ont été calculés à l’aide soit de l’ADND obtenue à partir d’échantillons en vrac (25 vers) soit d’une moyenne de 36 échantillons de vers simples (figure 5). Les deux CDA ont été obtenus à l’aide de la méthode Worm-to-CT et amplifiés à l’aide de la technologie PCR nanofluidique. Comme on l’a observé à la figure 5A-C, pour tous les chaperons testés (c.-à-d. hsp16.1,F44E5.4, hsp-70),les méthodes ont détecté des niveaux d’expression comparables. Ces résultats indiquent que les paramètres obtenus à partir de vers simples sont fiables.
Application du Ver-to-CT couplée à la technologie nanofluidique pour estimer les paramètres d’expression des gènes d’un seul ver
Étant donné que la puce à tableau unique permet de surveiller jusqu’à 96 transcriptions cibles sur 96 échantillons individuels, elle est donc bien adaptée pour surveiller la variabilité individuelle de l’expression des transcriptions entre les vers individuels. La figure 6A présente un résultat représentatif montrant l’expression moyenne de transcriptions de plusieurs hsp à partir de vers simples à la suite d’un choc thermique court. Comme on l’a observé dans la figure, la variabilité dans l’expression des transcriptions différait considérablement d’un gène à l’autre (figure 6A). Pour mieux comprendre, le coefficient de variation (CV) a été calculé en divisant l’écart type par la moyenne des niveaux d’expression26 (figure 6B). Trois gènes dont les valeurs cv ont déjà été estimées par d’autres méthodes ont été suivis (données non publiées). Deux transcriptions stables (ife-1 et Y45F10D.4) et une variable (nlp-2927) ont montré leur variabilité attendue. Le graphique illustre également clairement la relation inverse bien connue entre les valeurs de variabilité et les niveaux d’expression26 (figure 6B).
Les répliques techniques sont d’une importance primordiale pour assurer la reproductibilité lors de l’utilisation d’échantillons en vrac. Cependant, ce n’est pas nécessairement le cas pour les expériences unicellcellules 14,15,28. Pour déterminer si l’utilisation de répliques techniques est nécessaire pour l’estimation des paramètres lors de l’utilisation d’échantillons de vers simples, 28 vers individuels ont été récoltés, à la suite d’un choc thermique court, et traités à l’aide de triplicates techniques. Les valeurs cv calculées à partir de données sur les vers simples obtenues en triplicate (points bleus de la figure 7,CV technique) par rapport à celles pour chaque transcription obtenue à partir de vers individuels (points rouges de la figure 7,variabilité biologique) ont été comparées. Pour chaque transcription testée, les CV techniques étaient inférieurs aux CV biologiques, ce qui indique que les triplicates techniques n’étaient pas nécessaires pour l’estimation des paramètres. Le fait que des répliques techniques ne soient pas nécessaires augmente le débit de l’expérience sans compromettre la qualité.

Figure 1 : Vue d’ensemble du Protocole vermifuge à CT.
Ce chiffre montre un bref aperçu des différentes étapes nécessaires pour exécuter les vers à travers le protocole Ver-to-CT. Deux méthodes optionnelles sont affichées pour l’étape de transcription inverse; ce sont des méthodes interchangeables pour l’un ou l’autre type de puce. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 2 : Aperçu de la préparation et de l’exécution du QPCR nanofluidique.
Cette figure représente les préparatifs pour l’exécution du système qPCR nanofluidique à l’aide d’une puce multi-tableaux et d’une puce à un seul tableau. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
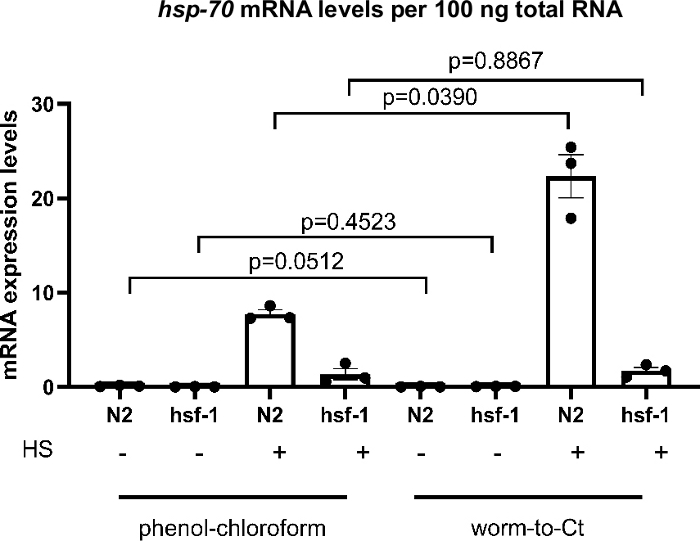
Figure 3 : Le protocole vermifuge à CT sur les échantillons en vrac a fourni des résultats fiables.
Comparaison du protocole ver-to-CT par rapport à l’extraction régulière de thiocyanate-phénol-chloroforme de guanidium22 sur des échantillons en vrac. Conformément aux résultats précédents, dans hsf-1 (sy441) mutants23, les niveaux de transcriptions hsp en réponse au choc thermique a diminué. Les histogrammes ci-dessus représentent l’induction de hsp-70 en l’absence de (-), ou suivant (+) un choc thermique court de 30 min à 34 °C. L’ADNC a été obtenue à l’aide de l’extraction de guanidium thiocyanate-phénol-chloroforme appliquée à 1 000 vers (à gauche) ou en utilisant la méthode ver-to-CT appliquée à 30 vers mis en commun (à droite). Les niveaux d’expression de hsp-70 pour 100 ng d’ARN total obtenus par chaque méthode ont été comparés. Comme prévu, dans hsf-1 (sy441) l’induction transcriptionnelle de hsp-70 en réponse au choc thermique a diminué de manière significative de 82,7% en utilisant le thiocyanate de guanidium-phénol-chloroforme et de 92,3% en utilisant la méthode ver-to-CT. Les niveaux d’ARNm des gènes cibles ont été normalisés par rapport à la moyenne des trois gènes d’entretien ménager cdc-42, pmp-3, et ire-1. Chaque point représente une réplique biologique. Les données ont été transformées en journal pour analyse statistique, car elles ne répondaient pas aux conventions requises pour l’analyse paramétrique. L’analyse statistique a été effectuée à l’aide d’un ANOVA RM-One-way à l’aide du test de comparaisons multiples de Sidak. Type sauvage = N2, hsf-1 = hsf-1 (sy441). Les barres dénotent l’erreur standard de la moyenne. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
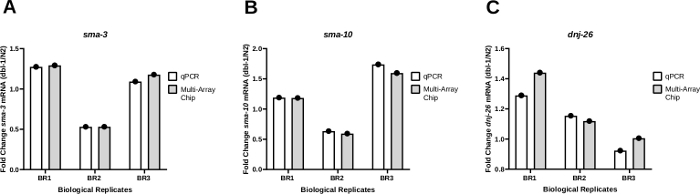
Figure 4 : Les modèles d’expression étaient cohérents entre les systèmes qPCR standard et nanofluidique qPCR.
(A) Le niveau d’expression de l’ARNm sma-3 (A), sma-10 (B) ou dnj-26 (C) a été déterminé par qPCR régulier et qPCR nanofluidique (puce multi-tableaux) à partir de trois répliques biologiques de l’ADNC générées par ver à CT à partir de la souche de type sauvage (N2) et de la souche dbl-1 (nk3) knockout 25. Des niveaux relatifs d’expression de l’ARNm ont été déterminés pour chaque souche à l’aide de la méthode Delta-Ct21. Le changement de pli a ensuite été déterminé en divisant les niveaux d’expression obtenus dans les vers dbl-1(nk3) par les niveaux correspondants d’ARNm dans la souche N2. Comme le montre le panneau A, les modèles étaient cohérents pour les deux méthodes dans chaque réplique biologique individuelle. (B) et (C) sont les mêmes que (A) pour les niveaux de sma-10 et dnj-26 ARNm, respectivement. Les niveaux cibles d’ARNm ont été normalisés par rapport aux gènes d’entretien ménager cdc-42 et pmp-3. L’analyse statistique a été calculée pour chaque gène à l’aide d’un t-test apparié comparant les résultats des trois répliques biologiques produites par qPCR standard et ceux générés par qPCR nanofluidique. Les valeurs P de ces comparaisons étaient respectivement de 0,3113, 0,2635 et 0,3481 pour le sma-3, le sma-10 et le dnj-26. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 5 : L’utilisation de la méthode du ver au CT sur des échantillons en vrac ou sur des vers simples a fourni des niveaux d’expression similaires lorsqu’ils sont normalisés par ver.
Les niveaux d’expression de (A) hsp-16.1/11, (B) F44E5.4, et (C) hsp-70 (C12C8.1) ont été analysés chez les jeunes animaux adultes en l’absence de choc thermique soit en effectuant Worm-to-CT sur une grande partie de 25 animaux, ou sur 36 individus individuels. Lorsque les données ont été normalisées par ver, il n’y avait pas de différence significative entre les niveaux obtenus par ver pour chaque transcription en utilisant les deux méthodes. Les niveaux d’ARNm des gènes cibles ont été normalisés par rapport à la moyenne des trois gènes d’entretien ménager cdc-42, pmp-3, et ire-1. Les barres représentent l’erreur standard de la moyenne. Statistiques = t-test apparié. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
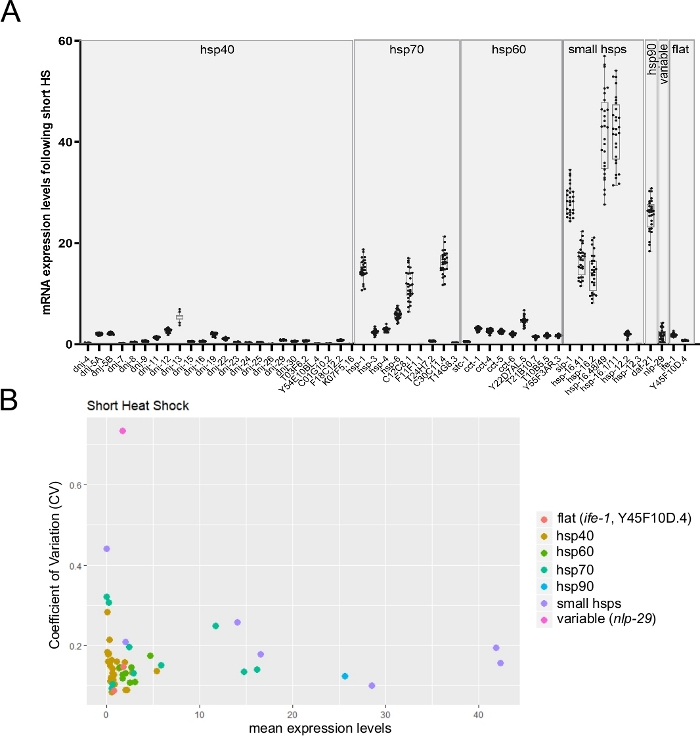
Figure 6 : Rt-qPCR à haut débit sur les vers individuels utilisant la méthode ver-to-ct pourrait surveiller la variabilité interinthique de l’expression des gènes.
(A) Les niveaux d’expression moyenne pour 53 transcriptions obtenues lors de l’exposition à un choc thermique court (30 min à 34 °C). Les boules de boîte représentent la distribution de l’expression moyenne de l’ARNm à partir de vers individuels (en moyenne, trois répliques techniques ont été utilisées par ver individuel). Les points représentent des niveaux d’expression chez 28 vers individuels. Les niveaux d’ARNm des gènes cibles ont été normalisés par rapport à la moyenne des trois gènes d’entretien ménager cdc-42, pmp-3, et ire-1. (B) Le coefficient de variation26 (CV) en fonction de l’expression moyenne de l’ARNm pour 53 transcriptions après l’exposition à un choc thermique court a été calculé à partir de 28 animaux individuels (données brutes indiquées dans le panneau B). L’ensemble des transcriptions comprend la transcription variable nlp-29 27 et deux transcriptions stables(ife-1 et Y45F10D.4; données non publiées). Le CV est le rapport entre l’écart type et la moyenne. Ce CV a été utilisé pour estimer la variabilité inter-individuelle dans l’expression de transcription entre les vers individuels. Comme prévu, la variabilité inter-individuelle s’est réduite avec une diminution des niveaux d’expression moyenne. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
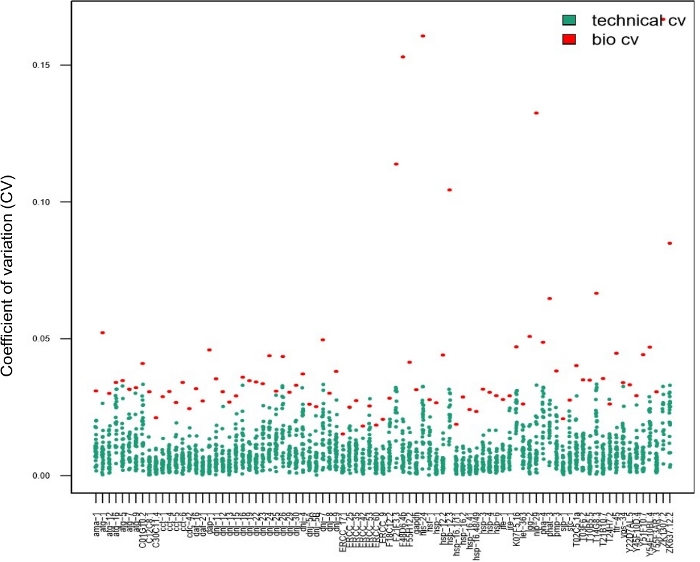
Figure 7 : Les répliques techniques n’étaient pas nécessaires lors de l’analyse de la variabilité inter-individuelle de l’expression des gènes à l’aide d’une puce nanofluidique.
Les données présentées dans ce graphique ont été obtenues chez 28 vers individuels à la suite d’un choc thermique court (30 min à 34 °C). Chaque point rouge représente le coefficient de variation (CV) des niveaux d’expression moyenne de transcription pour une transcription a été observée entre 28 vers individuels (bio CV). Chaque point bleu représente le CV des niveaux d’expression entre trois répliques techniques obtenues à partir d’un seul ver, selon la transcription assayed (CV technique). Ce graphique montre que la variabilité technique (entre les répliques techniques) était beaucoup plus faible que la variabilité biologique (entre les vers individuels), ce qui suggère qu’il n’est pas nécessaire d’effectuer des répliques techniques sur un tableau d’expression des gènes nanofluidiques lors de l’analyse de l’expression des gènes chez les vers simples, de la même manière que les études à cellulesindividuelles 14,15,28. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Tableau 1 : Planifier la mise en page d’une puce multi-tableaux. Le tableau ci-dessus affiche une mise en page simple qui peut être utilisée lors de la planification d’un jeu de puces multi-tableaux. Sur la gauche sont les espaces qui devraient être remplis avec les cibles d’amorce d’intérêt et sur la droite sont des espaces qui devraient être remplis avec les échantillons d’intérêt. Chaque analyse et tableau d’échantillons est jumelé au niveau du nombre à travers la puce. S’il vous plaît cliquez ici pour télécharger cette table.
Tableau 2 : Planifier la mise en page d’une puce à un seul tableau. Le tableau ci-dessus affiche une mise en page simple qui peut être utilisée lors de la planification d’une série de puces à un seul tableau. Sur la gauche sont des espaces qui devraient être remplis avec des cibles d’amorce d’intérêt et sur la droite sont des espaces qui devraient être remplis avec les échantillons d’intérêt. S’il vous plaît cliquez ici pour télécharger cette table.
Tableau 3 : Liste des amorces RT-qPCR utilisées dans cette étude. S’il vous plaît cliquez ici pour télécharger cette table.
Tableau supplémentaire 1 : Amorce de la base de données des amorces RT-qPCR. S’il vous plaît cliquez ici pour télécharger cette table.
Discussion
Dans cet article, le protocole Worm-to-CT s’est montré être une méthode rapide et efficace pour extraire l’ARN de vers simples ou d’un petit bassin de vers. Le débit élevé offert par le système nanofluidique le rend idéal pour la quantification des mesures inter-individuelles de variabilité. En outre, la sensibilité élevée de cette méthode permet la détection des gènes exprimés à de faibles niveaux qui tombent en dessous de la détection lors de l’utilisation de l’ARN-seq technologies à unseul ver 9.
Lors de l’examen du choix de la méthode pour préparer l’ADND à partir de vers simples. Ly et coll.29 ont optimisé un protocole qui repose sur la proteinase K pour la digestion des cuticules. La cuticule est un obstacle majeur pour l’isolement des molécules des vers et proteinase K fournit une méthode efficace pour la briser. Cependant, proteinase K doit être inactivé par la chaleur pour être en mesure d’utiliser des enzymes pour la transcription inverse. Bien que Ly et coll. aient utilisé une exposition de 10 min à 96 °C, cette étape a été évitée dans ce protocole parce que l’ARN est facilement dégradable. Au lieu d’utiliser la proteinase K, des cycles répétés de gel-dégel ont été utilisés pour briser la cuticule. Le gel-dégel est une méthode efficace pour briser la cuticule parce que plus d’ARN peut être isolé par ver. Ly et coll. rapportent que l’ARN total extrait par ver est de 35 ng à l’aide de proteinase K, alors que ce protocole obtient 51,75 ng ± 6,74 SEM d’ARN total par ver. L’évitement de l’exposition à la chaleur couplé à des étapes de préamplification élargit apparemment la gamme dynamique de détection de Worm-to-CT par rapport aux protocoles standard. Ly et coll. rapportent des valeurs ct absolues de 21,1 ± 0,15 pour hsp-16,2 et 22,8 ± 0,17 pour hsp-70 après le choc thermique. Utilisant les mêmes conditions de choc thermique (1 h à 30 °C), ce protocole obtient des valeurs ct absolues de 17,93 ± 0,57 pour hsp-16,2 et 21,13 ±0,33 pour hsp-70. Cela indique que la méthode de lyse gel-dégel offre des rendements plus élevés d’ARN et est plus appropriée pour les transcriptions faiblement exprimées.
Les systèmes nanofluidiques sont idéaux lorsqu’on étudie un ensemble donné de transcriptions cibles et l’utilisation d’échantillons plus petits (puces multi-réseaux) ou plus grands (puce à réseau unique) permettant une adaptation à l’échelle de l’expérience. Pour obtenir une image impartiale de toutes les transcriptions exprimées dans un seul ver, le choix évident est d’utiliser le séquençage de l’ARN. Si, toutefois, l’objectif de l’expérience est un ensemble plus petit mais encore relativement important de gènes cibles, il est plus rentable d’utiliser ce protocole, à condition que le chercheur ait accès à une machine PCR nanofluidique. Le coût des réaugmentés du système nanofluidique et d’une puce à réseau unique est estimé à environ 13 £ par ver, alors que les coûts des réaugmentés pour le séquençage d’un seul ver seraient d’environ £60 par ver, sans compter les coûts de séquençage.
Lors de l’examen de la plate-forme PCR à utiliser, la méthode Worm-to-CT couplée à qPCR nanofluidique offre des avantages en ce qui concerne le temps et le débit. Il est possible d’obtenir 9 216 résultats RT-qPCR en environ 2 jours de travail, alors que l’amplification du même nombre de cibles à l’aide d’une plate-forme qPCR standard prendrait environ 5 semaines de travail à l’aide de 96 analyses de plaques de puits, exécutant quatre plaques par jour. Toutefois, si le nombre de cibles à tester est plus faible, il est plus rentable d’utiliser Worm-to-CT couplé à une machine qPCR standard. Les puces mono-réseau ne peuvent pas être rediffusées, de sorte que l’exploitation de puits vides diminue le rapport coût-efficacité.
Une limitation de la méthode est la formation potentielle de dimers d’amorce-amorce pendant l’étape de multiplexage, mais ceci se produit dans moins de 1% des cas. Bien que le protocole Ver-to-CT soit efficace et donne des résultats fiables lorsqu’il est appliqué aux vers simples, il y a un taux d’échec d’environ 5 %, ce qui correspond probablement aux cas où le ver reste emprisonné dans le bouchon ou le haut du tube pendant l’étape de récolte.
Ensemble, cette méthode polyvalente et fiable offre un débit et une sensibilité accrus par rapport à des techniques plus standard. Cette méthode peut être très utile pour la validation d’écrans à haut débit et est un excellent choix pour surveiller ou valider les niveaux d’expression du gène mono-ver. Cette méthode peut être appliquée à d’autres techniques difficiles, telles que la quantitation de l’expression des gènes à partir de tissus isolés. Par exemple, l’isolement des tissus entiers, comme l’intestin, les 90 ou les cellules isolées par les CFC, fournit suffisamment de matériel pour effectuer des expériences de séquençage de l’ARN. Toutefois, des quantités limitées de documents mènent souvent à des relevés dupliqués, ce qui empêche la quantitation de transcriptions rares. Dans ce scénario, l’utilisation d’une technologie basée sur la nanofluidique devrait fournir une sensibilité accrue aux expériences et augmenter la rentabilité si les chercheurs n’ont besoin de surveiller qu’un sous-ensemble de toutes les transcriptions dans ces tissus ou cellules.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Sharlene Murdoch et les installations de l’Institut Babraham pour leur soutien. JLP a reçu l’appui du Wellcome Trust (093970/Z/10/Z) et OC est appuyé par ERC 638426 et BBSRC [BBS/E/B000C0426].
matériels
| Name | Company | Catalog Number | Comments |
| 100µM stock primers for genes of interest. | |||
| 20X DNA Binding Dye | Fluidigm | 100-7609 | for 96.96 chip (Single-Array Chip) |
| 2X Assay Loading Reagent | Fluidigm | 100-7611 | |
| 384 well plates | |||
| 8-strip PCR tubes | |||
| 96 well ice block | |||
| 96 well plates | |||
| 96.96 Dynamic Array IFC | Fluidigm | BMK-M-96.96 | Referenced throughout the manuscript as "Single-Array Chip" |
| 96-well sealing tape | |||
| BioMark & EP1 Software | Fluidigm | 101-6793 | Contains: Biomark HD Data Collection software, Real-Time PCR Analysis software, SNP Genotyping Analysis software, Digital PCR Analysis software, Melt Curve Analysis software |
| BioMark or BioMark HD system | Fluidigm | 100-2451 K1 | Referenced throughout the manuscript as "nanofluidics thermocycler" |
| Control Line Fluid Kit for 96.96 and FlexSix IFCs | Fluidigm | 89000021 | Referenced throughout manuscript as "control line fluid" |
| DNA Suspension buffer | TEKnova | T0021 | |
| domed PCR caps | |||
| Exonuclease I | New England BioLabs | M0293L | |
| Exonuclease I reaction buffer | New England BioLabs | B0293S | |
| FLEXsix DELTAgene Sample Reagent | Fluidigm | 100-7673 | referenced throughout manuscript as "sample reagent" |
| FLEXsix Gene Expression IFC | Fluidigm | 100-6308 | referenced throughout manuscript as "Multi-Array Chip" |
| Fluidigm Real-Time PCR Analysis User Guide | Fluidigm | 68000088 | This is the protocol used as a basis for section 2 of the protocol, referenced within the manuscript. |
| IFC Controller MX | Fluidigm | 68000112 I1 | Referenced throughout the manuscript as "nanofluidics PCR priming machine" |
| IFC Controllers SOFTWEAR | Fluidigm | 100-2297 | Contains all softwear and scripts required for running the IFC controller MX |
| liquid nitrogen in dewar | |||
| Microcentrifuge | StarLab | C1301B-230V | Used to spin down PCR tube strips in the protocol. |
| Nuclease Free Water | |||
| plate spinner | Labnet | K4050725 | mini plate spinner, mps 1000. referenced through the protocol as "tabletop plate spinner" |
| Power SYBR Green Cells-to-Ct kit | invitrogen | 4402955 | This kit is that which is adapted for use in nematodes in the Worm-to-CT protocol. Contense: Store Stop Solution, DNase I and 20X RT Enzyme Mix at -20°C. Store Lysis Solution, 2X SYBR RT Buffer (referenced throughout the manuscript as RT buffer), and Power SYBR®Green PCR Master Mix (refferenced througout the manuscript as PCR Master Mix). This Kit is for 400 reactions, kits also available for 40 and 100 reactions. |
| PreAmp Master Mix | Fluidigm | 100-5580, 100-5581 | |
| Reverse Transcription Master Mix—480 reactions | Fluidigm | 100-6299 | One tube also available for 96 reactions (PN100-6298) |
| Rnase Free water | |||
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad Laboratories | 172-5211 | referenced throughout the manuscript as "fluorescent probe supermix" |
| Standard 96 and 384-well Thermal Cycler | |||
| TE Buffer (10mM Tris, pH8.0, 1.0mM EDTA | TEKnova | T0224 | Referenced throughout the manuscript as Tris-EDTA buffer |
| ThermoMixer C | Eppendorf | 5382000031 | Referenced throughout the text as "thermal mixer" |
| Trizol | Thermo-Fisher | 15596026 | Referenced through the protocol as "guanidium thiocyanate-phenol-chloroform" |
| Warm water bath |
Références
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biology. 4 (10), 309 (2006).
- Semrau, S., van Oudenaarden, A. Study lineage decision-making in vitro: emerging concepts and novel tools. Annual Review of Cell Developmental Biology. 31, 317-345 (2015).
- Kelsey, G., Stegle, O., Reik, W. Single-cell epigenomics: Recording the past and predicting the future. Science. 358 (6359), 69-75 (2017).
- Eling, N., Morgan, M. D., Marioni, J. C. Challenges in measuring and understanding biological noise. Nature Review Genetics. 20 (9), 536-548 (2019).
- Kaufmann, B. B., van Oudenaarden, A. Stochastic gene expression: from single molecules to the proteome. Current Opinion in Genetics & Development. 17 (2), 107-112 (2007).
- Corsi, A. K., Wightman, B., Chalfie, M. A Transparent window into biology: A primer on Caenorhabditis elegans. WormBook. , (2015).
- Casanueva, M. O., Burga, A., Lehner, B. Fitness trade-offs and environmentally induced mutation buffering in isogenic C. elegans. Science. 335 (6064), 82-85 (2012).
- Raj, A., Rifkin, A. A., Andersen, E., van Oudenaarden, A. Variability in gene expression underlies incomplete penetrance. Nature. 463 (7283), 913-918 (2010).
- Perez, M. F., Francesconi, M., Hidalgo-Carcedo, C., Lehner, B. Maternal age generates phenotypic variation in Caenorhabditis elegans. Nature. 552 (7683), 106-109 (2017).
- Serra, L., et al. Adapting the Smart-seq2 Protocol for Robust Single Worm RNA-seq. Bio-protocol. 8 (4), 2729 (2018).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Biology. 14 (8), 1007559 (2018).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nature Review Genetics. 16 (3), 133-145 (2015).
- Ståhlberg, A., Kubista, M. The workflow of single-cell expression profiling using quantitative real-time PCR. Expert Review of Molecular Diagnostics. 14 (3), 323-331 (2014).
- Livak, K. J., et al. Methods for qPCR gene expression profiling applied to 1440 lymphoblastoid single cells. Methods. 59 (1), 71-79 (2013).
- Fluidigm. . Real-Time PCR Analysis User Guide. , (2018).
- Huggett, J. F., et al. The digital MIQE guidelines: Minimum Information for Publication of Quantitative Digital PCR Experiments. Clinical Chemistry. 59 (6), 892-902 (2013).
- . U. S. National Library of Reference. Primer Blast Available from: https://www.ncbi.nlm.nih.gov/tools/primer-blast/ (2020)
- Bustin, S. A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems. Journal of Molecular Endocrinology. 29 (1), 23-39 (2002).
- Nolan, T., Huggett, J., Sanchez, E. . Good Practice guide for the application of quantitative PCR (qPCR), LGC. , (2013).
- Yuan, J. S., Reed, A., Chen, F., Stewart, C. N. Statistical analysis of real-time PCR data. BMC Bioinformatics. 22 (7), 85 (2006).
- He, F. Total RNA Extraction from C. elegans. Bio-protocol. Bio-101. 47, (2011).
- Hajdu-Cronin, Y. M., Chen, W. J., Sternberg, P. W. The L-Type Cyclin CYL-1 and the Heat-Shock-Factor HSF-1 Are Required for Heat-Shock-Induced Protein Expression in Caenorhabditis elegans. Genetics. 168 (8), 1937-1949 (2004).
- Douglas, P. M., et al. Heterotypic Signals from Neural HSF-1 Separate Thermotolerance from Longevity. Cell Reports. 12 (7), 1196-1204 (2015).
- Zhang, X., Zhang, Y. Dbl-1 a TGF-beta is essential for C. elegans aversive olfactory learning. Proceedings of the National Academy of Sciences of the United States of America. 109 (42), 17081-17086 (2012).
- Sørensen, J. B. The Use and Misuse of the Coefficient of Variation in Organizational Demography Research. Sociological Methods & Research. 30 (4), 475-491 (2002).
- Pujol, N. Distinct innate immune responses to infection and wounding in the C. elegans epidermis. Current Biology. 18 (7), 481-489 (2008).
- Brennecke, P., et al. Accounting for technical noise in single-cell RNA-seq experiments. Nature Methods. 10 (11), 1093-1095 (2013).
- Ly, K., Reid, S. J., Snell, R. J. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 7 (2), 59-63 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon