Method Article
使用纳米流体技术的单体和散装 C. 电子样品 中的高通量定量 RT-PCR
* 这些作者具有相同的贡献
摘要
本文描述了一个高通量协议,用于快速、可靠地确定单个或批量 C.elegans 样本中的基因表达水平。此协议不需要 RNA 隔离,并且直接从样品中生成 cDNA。它可以与高通量多路复用纳米流体实时 qPCR 平台一起使用。
摘要
本文对快速、健壮、高度灵敏的 卡诺哈布迪炎患者 进行了高通量反向转录定量PCR(RT-qPCR)检测。此协议从单个蠕虫或批量样本中获取基因表达的精确测量。此处提交的协议提供了对现有补充DNA (cDNA) 制备方法的全新改编,并结合了纳米流体 RT-qPCR 平台。此协议的第一部分名为"蠕虫到CT",允许 cDNA 直接从线虫生产,而无需事先隔离 mRNA。它通过允许在 3.5 小时内从 96 种蠕虫中制备 cDNA 来增加实验吞吐量。协议的第二部分使用现有的纳米流体技术在 cDNA 上运行高通量 RT-qPCR。本文评估了两种不同的纳米流体芯片:第一种是运行96个样本和96个靶点,在大约1.5天的工作时间里产生9,216个反应。第二个芯片类型由六个 12 x 12 阵列组成,产生 864 个反应。在这里,蠕虫到CT的方法通过量化mRNA水平的基因编码热冲击蛋白从单一蠕虫和散装样品。提供的是一个广泛的引物列表,旨在放大 C.elegans 基因组中大多数编码基因的处理RNA。
引言
单细胞RNA测序和qPCR的优化表明,转录脉冲或爆发可导致每个细胞1的RNA分子数量发生巨大变化。此外,这些技术还发现了标准批量转录测量之前遗漏的大量细胞异质性。根据上下文,一些单细胞转录变异是由组织混合细胞组成引起的。然而,即使在同源细胞群生长在同一环境下,也有广泛的转录异质性2,3。这种"生物变异性"越来越被确定为细胞网络无处不在的特性,从细菌到人类。在某些情况下,它可能对发育、癌症进展、HIV延迟和化疗反应4、5产生表型后果。
线虫卡诺哈布迪炎是研究个体间生物变异的原因和后果的理想特征的独特模型生物体。这些线虫是一个由959个细胞组成的简单模型生物体,其透明的层基使其适合进行体内成像研究。雄性是一种主要通过自肥繁殖的草药物种:这导致了原生实验室菌株。尽管同源性和受控的培养条件,许多表型和成绩单是变化的个体,表明随机或微环境差异有助于异质性在个人7,8。基因表达的这种变异性具有多种健身后果,包括突变的渗透、生存、发育时机和生育能力7、8、9的变异性。由于这些特点,单虫研究为研究整个生物体的生物变异性提供了前所未有的机会。
该领域根本需要开发和优化技术,以准确检测单蠕虫级别的脚本。新技术,如单虫RNA测序10,RNA测序从分离组织11,单细胞测序12现在可用于C.elegans。然而,一个主要的挑战依然存在:在监测个人间时,表达微弱的基因往往低于可检测的13级。这对于从少量起始材料中分离出的稀有成绩单特别相关,因为平均表达和技术差异之间存在着既定的反向关系,往往导致罕见的成绩单低于统计截止点13。高通量多路复用qPCR技术的优化已被证明是有用的哺乳动物单细胞研究,特别是在研究罕见的成绩单14,15的表达。该技术还可用于其他单虫技术的基准和验证目的。
蠕虫到CT是一种快速、稳健的方法,它适应了细胞生物学研究中使用的工具包,用于单蠕虫cDNA制备。通过这种方法获得的cDNA加上多路复用纳米流体qPCR技术被选中,因为它提供了更高的实验吞吐量,更广阔的动态检测范围,并已验证为单细胞目的14,15。所述的 cDNA 制备也适用于标准 PCR 技术。产量在两个方面增加:首先,cDNA制剂比传统的硫酸钛酸-苯酚-氯仿提取更快、更可靠,因为蠕虫直接添加到裂解缓冲器中,跳过了容易降解RNA的直接分离。其次,利用纳米流体技术可显著增加可同时运行的样品和目标的数量。本文比较了两个芯片:单阵列芯片和多阵列芯片。单阵列芯片可以运行 96 个单蠕虫和 96 个引物集,每个实验产生 9,216 个反应。要使用标准 qPCR 技术实现类似的吞吐量,需要使用 96 个井板进行 96 个单独的 qPCR 实验。更小、更灵活的多阵列芯片由六个 12 x 12 阵列组成,可产生 864 个反应。该方法的卓越可靠性和灵敏度通过纳米流体技术和预放大步骤的引入而得到提高。本文中介绍的方法将与最先进的统计算法一起用于提取生物差异。本文介绍了单虫和批次蠕虫样品的快速cDNA制备和高通量qPCR协议:该算法将在其他地方发布。对于此协议,应在实验前准备每个芯片的组织。表 1和表 2分别显示了多阵列和单阵列芯片的这些计划示例。图1中还详细介绍了蠕虫到 CT 协议的概况,并在图2中运行多阵列和单阵列芯片。
研究方案
注:在整个协议 中,卡诺哈布迪炎 被称为"蠕虫"或"蠕虫"。各种 C.elegans 菌株可以通过在线数据库或直接联系使用模型生物体的实验室订购。此协议的第一部分(第 1+3 节)描述了通过蠕虫到 CT 协议的 cDNA 准备。本协议第二部分(第 4-13 节)描述了使用纳米流体运行高通量 RT-qPCR,该方案改编自流体16开发的协议。该协议适用于使用先前定义的两种类型的纳米流体芯片,即单阵列芯片,它可以将 96 个目标监控为 96 个样本(总计 9,216 个 RT-qPCR 反应),或多阵列芯片,其功能为 12 个目标 x 12 个样本的子单元。每个多阵列芯片包含六个独立的阵列,可以一起运行或单独运行。例如,使用整个多阵列芯片可以监控 72 个目标 x 12 个样本(反之亦然),或 36 个目标 x 24 个样本(反之亦然)。有关本协议中使用的任何材料的进一步信息,请参阅 材料表。
1. RT-qPCR 入门验证
注:实时入门设计基于最初由 MIQUE 指南17发布的推荐属性。为了使底漆特定于加工RNA,产品的设计使两个底漆绑定到至少一个拼接路口的两侧。对合适的引物的要求包括 20%~80% 的瓜宁和细胞氨酸含量、58~60°C 的熔化温度、≤0.5°C 的引物对和 70~120 bp 的产品长度之间的熔化温度差异。生成的引物序列可以在 补充表 1中找到。用于生成引物的脚本的开源代码可在 https://github.com/s-andrews/wormrtpcr 找到。与拼接站点的脚本的引物对被设计成,使他们位于两个exons侧翼一个因特龙,但不是设计为拼接变种特定,使用NCBI引物爆炸软件18。在这项研究中,引物组被炸到 C.elegans 基因组上,以测试任何偏离目标的互补性。
- 从 RT-qPCR 入门器数据库检索入门(补充表 1)。或者,使用在线工具(如 NCBI 引价爆炸18)设计 qPCR 入门对。
- 使用标准批量 qPCR 技术 19 和遵循 MIQUE 指南17、20来监控每对引头的特异性和 PCR 效率。
注:仅应使用 R2 = 0.98 和 PCR 效率在 85% 到 115% 之间的引入器对。本研究中使用的引漆的序列、PCR 效率和 R2详见表 3。
2. 蠕虫裂解通过蠕虫到CT
- 将蠕虫从细菌草坪上采摘到新鲜的非种子NGM板上,让蠕虫在盘子周围移动5分钟,通过移动从蠕虫身上去除大部分细菌。
注:细菌草坪和生长条件将因实验设计而异。这里提出的实验需要6厘米NGM板种子与OP50 大肠杆菌 与蠕虫的兴趣增长到阶段L4.9在20°C孵化器。 - 在无 RNase 罩中,准备一个主混合物,包括 12.5 μL 的 2 倍 RT 缓冲区、1.25 μL 的 20 倍 RT 酶缓冲区以及每个样品 0.25 μL 的无核水。从步骤 2.8 向每个样本的 11 μL 添加 14 μL 的主混合。
- 将 PCR 条的盖子倒置在解剖示波器的平台上,并在复合显微镜下将 10μL 的裂解组合添加到圆顶 PCR 管盖上。
注:只需一到两个样品,最好使用至少包含四根管子的 PCR 条,这样可以降低在随后的冻结步骤中打开盖子爆破的风险。或者,橡皮筋可用于将盖子放在原位。在这种情况下,确保每次转移管子时,盖子都正确关闭。 - 将含有裂解混合物的盖子中的蠕虫从盘子中挑入每个槽中,通过"铲"它们(即用拾取物捕捉下面的蠕虫),以避免细菌污染。关闭管子,用桌面微中心(材料表)将管子旋转5s,然后放入装满液氮的德瓦尔烧瓶中。
注:15至30种蠕虫应用于散装实验,1种蠕虫用于单虫测量。
注意:在处理液氮时佩戴冷冻手套以及防护眼镜并遵守标准服装规定,因为接触皮肤或眼睛可能会导致严重的冻伤。 - 通过在液氮和+40°C水浴之间转移PCR管,将PCR管冷冻10倍。将管子留在液氮中至少5s,以确保样品完全冷冻。将管子留在水浴中,直到样品解冻。不要离开更长的时间,因为这会导致RNA退化。
注意:管子可以长时间留在液氮中,因为样品被冷冻到-200°C左右,从而减少RNA降解。然而,这不应该是一个协议的停止点,因为液氮蒸发迅速。 - 将样品混合在热搅拌机(材料表)上,以 4 °C 为单位,以 1,800 rpm 的速度旋转 20~30 分钟。
- 当样品被混合时,解冻冰上的停止溶液。
- 使用桌面微中心(材料表)将样品旋转下来,并在每个管子中添加 1 μL 的停止溶液。
注:样品可在-80°C处留用长达1周,然后反向转录RNA(第3节)。
3. 反向转录
注:对于单个蠕虫的反向转录,此处显示的结果使用纳米流体芯片提供的试剂生成( 图 1中的选项 2)。 图 1 选项 2 中突出显示的试剂也用于倒转汇集样品。这两种方法可互换适用于不同的样本类型。
- 单个蠕虫的反向转录
- 在无 RNase 罩中,将 1.25 μL 的反向转录混合(材料表)添加到新鲜的 PCR 管中。
注意:如果有许多样品,可以使用 96 井 PCR 板和自动移液器。制造商的协议规定每个样品可使用 1 μL。 - 取 5 μL 的裂解溶液,从步骤 2.8 停止溶液混合,并将其添加到包含反向转录混合的新鲜 PCR 管中。
注:制造商的协议规定,每次反应可使用 1 μL 的RNA(2.5 pg/μL-250 ng/μL)。通过用 5 μL 的浸润样品和 1.25 μL 的无 RNAse 水替换反向转录混合,可以添加每个板的负 RT 控制。 - 在热车上使用以下反向转录程序运行样品:5 分钟 25 °C,30 分钟 42 °C,5 分钟 85 °C,∞ 4 °C。
注:生产的 cDNA 可存储在 -20 °C 下,然后使用高通量 qPCR 进行放大和数据收集。
- 在无 RNase 罩中,将 1.25 μL 的反向转录混合(材料表)添加到新鲜的 PCR 管中。
- 批量样品的反向转录(15+30 蠕虫)
- 在无 RNase 罩中,准备一个主混合物,包括 12.5 μL 的 2 倍 RT 缓冲区、1.25 μL 的 20 倍 RT 酶缓冲区以及每个样品 0.25 μL 的无核水。将主混合的 14 μL 添加到 11 μL 的裂解溶液中,并停止步骤 2.8 的溶液混合。
注:处理大量样品时,可在 96 个井板中执行。 - 使用以下反向转录程序通过热车运行样品:60 分钟 37 °C,5 分钟 95 °C,∞ 4 °C。
- 在无核水中稀释所生产的 cDNA 1:4。
注:一般来说,产品最终产量为 25 μL,在这种情况下应添加 75 μL。但是,由于凝结,最终音量可能会有所不同。因此,在最终解决方案中相应地进行调整,使比例为 1:4。如果在单个蠕虫上执行 qPCR,则此稀释步骤不适用。在使用高通量 qPCR 进行放大和数据收集之前,可将所生产的 cDNA 存储在 -20 °C 下。
- 在无 RNase 罩中,准备一个主混合物,包括 12.5 μL 的 2 倍 RT 缓冲区、1.25 μL 的 20 倍 RT 酶缓冲区以及每个样品 0.25 μL 的无核水。将主混合的 14 μL 添加到 11 μL 的裂解溶液中,并停止步骤 2.8 的溶液混合。
4. 准备多路复用引漆组合
- 在 50 μM 的最终浓度下,为每对引漆准备一个前/反向 (F/R) 入门库存。将相同体积的前进引漆和反向引漆混合在 100 μM 中。
- 结合 1 μL 的 50 μM F/R 入门库存,用于测试每个引漆对。添加DNA悬浮缓冲区,总体积可达100μL。
注:此处的库存引物浓度与制造商协议16中描述的浓度不同,但保留 500 nM 的相同最终浓度。
5. 目标特定的预置
- 准备包含 1 μL 的预增密大师混合(材料表)、0.5 μL 的池底漆混合(步骤 4.2)和 2.25 μL 的无核素水,每个反应的总剩余量为 10%。
- 在 96 井板中,主油井的 aliquot 3.75 μL 按照运行样品数量的要求混合到尽可能多的油井中。
- 在每个油井的第 3.1.3 步或 3.2.3 步生成的 cDNA 解决方案中添加 1.25 μL。
- 用 96 口密封胶带、短暂漩涡和离心机用台面板微调器盖住盘子。转移到热车并运行以下程序:2 分钟 95 °C,15 秒在 95 °C 下自然 15 周期,4 分钟在 60 °C 下退化/延长,∞ 4 °C。
注:制造商建议从10+20周期的预放大反应16。此协议根据目标基因的表达水平建议 10 或 15 个周期。
6. 外核酶I治疗
注:这是为了从预放大中去除未合并的引物。
- 准备一个外核酶I混合含有0.2μL的外核酶I反应缓冲器(材料表),0.4 μL的外核酶I在20 U/μL(材料表),和1.4 μL的无核糖酶每样品。将所有试剂都放在冰上,尤其是外核酶I。
- 从热车中取出 96 井板(步骤 5.4),用台面板微调器离心机,小心地取下密封件。将 2 μL 的外核酶混合到每个预增化反应中。重新塞,离心机,并使用以下程序将96井板放回热车:30分钟37°C,15分钟80°C,∞4°C。
- 将样品从热循环器中取出,通过添加 18 μL 的 1x 特里斯 EDTA 缓冲区(材料表),稀释 1:5。
注:可以将 cDNA 样本保持在 -20 °C 以供以后使用。制造商的协议建议在这个阶段16中潜在的稀释5倍,10倍,或20倍,这取决于感兴趣的目标的表达水平。
7. 准备检测混合物
注:Assay 混合物可在 384 个井板中制备,因为油井的间距与纳米流体芯片相同,因此加载更加容易。
- 为多阵列芯片准备检测混合
- 根据准备的计划,为每口井准备一个由2 μL的2倍检测装载试剂(材料表)和1.6μL的DNA悬浮缓冲液(材料表)组成的主混合物。阿里报价 3.6 μL 的这个主混合每口井成 384 井板。
- 根据准备的计划,将第 4.1 步准备的 50μM F/R 引物库存中的 0.4 μL 添加到适当的油井中。
注:这为每口油井提供总共 4 μL 的检测混合,盈余为 1 μL。
- 为单阵列芯片准备检测混合
- 根据准备的计划,为每口井准备一个由 3 μL 的 2 倍检测装载试剂和 2.4 μL 的 DNA 悬浮缓冲液组成的主组合。阿里引用 5.4 μL 的这个主混合每口井成标记的 384 油井板。
- 根据事先准备好的计划,将 50 μM F/R 引物库存中的 0.6 μL 添加到适当的油井中。
注:这为每口井提供总共 6 μL 的检测混合物,剩余量为 1 μL。
8. 准备样品混合物
注意:样品混合物可提前1天准备,并储存在4°C。
- 为多阵列芯片准备样品
- 准备一个样品主组合,包括2 μL的2倍荧光探头超级混合与低ROX(材料表)和0.2μL的样品试剂(材料表)每个样品。将此组合的 2.2 μL 分配到标记的 384 井板中。
注:制造商建议不要对样品试剂16进行旋涡。 - 根据事先准备好的计划,我处理的样品从步骤 3.2.3 到适当的油井中,每个预增的外核酶的移液器 1.8 μL。
注:这总共提供 4 μL,盈余为 1 μL。
- 准备一个样品主组合,包括2 μL的2倍荧光探头超级混合与低ROX(材料表)和0.2μL的样品试剂(材料表)每个样品。将此组合的 2.2 μL 分配到标记的 384 井板中。
- 为单阵列芯片准备样品
- 准备一个样品主组合,包括3 μL的2倍荧光探头超级混合与低ROX(材料表)和0.3 μL的20倍DNA结合染料样品加载试剂(材料表)每个样品。将此组合的 3.3 μL 分配到标记的 384 油井板中。
- 管道 2.7 μL 的每个预增和外核酶我处理样品从步骤 6.3 到适当的井根据准备的计划。
注:这总共提供 6 μL,盈余为 1 μL。如果有任何井运行没有样品,这些必须加载样品主混合和2.7 μL的水,而不是cDNA。这两种芯片类型都推荐这样做。该机器需要每个入口的低 ROX 才能检测芯片的网格。
9. 撬开纳米流体芯片
注意:多阵列芯片只需在第一次运行时准备。如果同一芯片随后运行,则可以跳过此阶段。这两种芯片类型的步骤都是相同的。
- 缓慢而小心地将包括注射器在内的控制线流体的全部 150 μL 注入芯片的蓄能器中。确保没有控制液体接触芯片,将芯片保持在 45° 角,并保持注射器的尖端,以避免溢出。
- 从芯片底部取出蓝色保护膜。
- 将芯片放入纳米流体PCR启动机(材料表),条形码朝外。运行"Prime(153x)"脚本,需要约15-20分钟。
注意:此时打开纳米流体热车(材料表),因为相机需要大约 10 分钟才能冷却到 0 °C 以下。
10. 装载纳米流体芯片
- 加载时按顺序卸下障碍插头。这减少了误装油井的机会。
- 根据准备的计划,将多阵列芯片的 3 μL 或 5 μL 的单阵列芯片传输到每个引物检测混合和样品混合的相应入口。确保不要引入气泡,这可能导致传输的总体积小于预期。
注意:如果有任何井将运行没有引物,重要的是这些加载主混合,用水代替引物体积。这适用于两种芯片类型。在这个阶段,将芯片放置在黑暗的表面上可能更容易,这样可以更容易地看到油井。
11. 运行纳米流体芯片
注:首次运行多阵列芯片,通过选择 工具 |设置跟踪文件 Flex 六使用情况跟踪,单击 "新",输入文件名称,然后在单击 "完成"之前选择一个位置。
- 打开数据收集软件。单击 "启动新运行"。将加载的芯片放入纳米流体热循环器中,条形码朝外。
- 如果适用,请选择项目设置,然后单击 "下一个 | 加载。如果加载多阵列芯片,请选择要运行的分区(阵列)。
- 选择应用 参考探头,然后将应用类型更改为 基因表达,并将被动引用更改为 ROX。选择 单个探头分析,将探头类型更改为 伊娃·格林,然后单击 "下一步"。
- 选择热循环协议 GE FLEX 六快速 PCR+Melt v1 来运行多阵列芯片,或选择 GE 96.96 快速 PCR+Melt v2 来运行单阵列芯片。
- 确认自动 曝光 已选中,然后单击 "开始运行"。
12. 芯片运行后
注意:此部分仅在不使用整个芯片时用于多阵列芯片。
- 将芯片从纳米流体热循环器中取出并加载到纳米流体 PCR 启动机中,并运行后运行 (153x) 脚本,该脚本持续 5 分钟。
- 标记用于个人参考的插头。
注:芯片现在可以在室温下存储,芯片上的其余阵列可在 2 个月内运行。
13. 数据清理和分析
- 打开"实时 PCR 分析"软件(材料表)中的数据。检查每个测试的引漆对的熔化峰值温度。消除显示多个熔化温度峰值的结果,用于给定引物对。
注:多个峰值仅偶尔出现,大概当引物对形成暗淡时,或从目标入门与其他引物在池入门组合中的交互。 - 将数据导出为"热图"电子表格文件,并消除失败的样本或引物。
- 使用标准的达美-Ct 方法21分析数据。对于统计评估,在相对表达水平上执行单向 ANOVA。
结果
蠕虫到CT的验证作为cDNA制备方法
为了测试蠕虫到CT协议是否是一种有效的cDNA提取方法,它被比作标准的硫酸钠-苯酚-氯仿提取方法。研究结果见 图3,其中cDNA是使用标准硫酸酸酯-苯酚-氯仿提取技术22 和使用蠕虫到CT方法的30种蠕虫平均约1,000只蠕虫中制备的。样品同时受到热冲击(30分钟在34°C)。在全球范围内 ,hsp-70 mRNA 表达水平每 100 ng 的总 RNA 是可比的,使用这两种方法。然而,在 hsp-70 表达率最高的情况下(即在热冲击后的 N2 中),使用蠕虫到 CT 方法的表达水平较高,表明灵敏度有所提高。
为了确定hsf-1(sy441)23中hsp表达的预期下降,是否可以复制分子伴奏23、24的主要转录调节器的突变,比较了短暂的热休克后转录陪护诱导。通过这两种方法,在hsf-1(sy441)动物中检测到hsp-70诱导的减少。这是预料之中的,因为突变的hsf-1(sy441)动物表现出由于HSF-1的转动域截断而诱导伴郎的能力下降。与野生类型动物相比,硫酸二甲酸酯-苯酚-氯仿提取hsp70比对照组下降82.7%,蠕虫-CT下降92.3%(图3)。结果在这两种方法之间是可比的,可与先前的报告23相媲美。这些结果表明,蠕虫到CT方法是标准cDNA合成技术的有效替代品。
用于放大mRNA目标的纳米流体PCR平台的验证
为了测试使用纳米流体 qPCR 进行成绩单放大的结果的一致性,在标准 qPCR 系统(材料表)和使用多阵列芯片的纳米流体 qPCR 系统上比较了从蠕虫到 CT 批量方法获得的 PCR 结果。与野生类型对应物相比,在携带dbl-1 (dbl-1 (nk3) 25 中携带空等位基因的动物中监测了三种不同基因的折叠变化,即 sma-3 (图 4A)、sma-10 (图4B)和dnj-26。 Dbl-1编码骨形态遗传蛋白 (BMP) 信号通路的唯一配体。sma-3和sma-10是编码 SMAD 正体的基因,是 BMP 信号级联的关键组件。Dnj-26编码分子伴奏,BMP 信号的目标。这些结果表明,与这两种方法的结果相比,折叠变化几乎没有差别,结果P值分别为0.3113、0.2635和0.3481,分别为sma-3、sma-10和dnj-26。 总的来说,这些结果表明,应用于散装样品的蠕虫到CT方法是一种从少数蠕虫中提取RNA的高效和快速的方法,并且与标准PCR系统或基于高通量纳米流体的qPCR平台相结合时提供了可靠的数据。
批量样本获得的表达水平与从单个蠕虫获得的平均值之间的比较
相对表达水平是使用从散装样本(25个蠕虫)或平均36个单个蠕虫样本(图5)获得的cDNA计算的。这两种 cDNA 都是使用蠕虫到 CT 方法获得的,并使用纳米流体 PCR 技术进行放大。如图5A-C所观察到的,对于所有测试的陪护人员(即hsp16.1、F44E5.4、hsp-70),这些方法检测到可比的表达水平。 这些结果表明,从单个蠕虫获得的参数是可靠的。
蠕虫到CT耦合纳米流体技术的应用,以估计单虫基因表达参数
由于单阵列芯片允许监测 96 个单个样本上多达 96 个目标成绩单,因此非常适合监测单个蠕虫之间成绩单表达的个体变异性。 图 6A 呈现一个具有代表性的结果,显示短热冲击后单个蠕虫的多个 hsp 成绩单的平均表达。如图所述,成绩单表达的变异性因不同基因(图6A)而显著差异。为了进一步深入了解,变异系数 (CV) 的计算方法是将标准偏差除以表达级别26 (图6B)的平均值。监测了三个基因,其CV值以前曾被替代方法估计过(未公布的数据)。两个稳定成绩单(ife-1 和 Y45F10D.4)和一个变量(nlp-2927)显示了它们的预期变异性。该图还清楚地描绘了变异值和表达级别26 (图6B)之间的已知反向关系。
技术复制对于确保使用批量样品时的可重复性至关重要。然而,单细胞实验14、15、28不一定如此。为了确定使用技术复制品在使用单虫样本时是否需要参数估计,在短暂的热冲击后收获了 28 种单个蠕虫,并使用技术三重处理。比较了从单个蠕虫获得的每份记录(图 7中的红点、生物变异性)中获得的单虫数据(图7中的蓝点、技术 CV)计算的 CV 值。对于每份测试成绩单,技术简历都低于生物简历,表明参数估计不需要技术三重。不需要技术复制,这一事实在不影响质量的情况下提高了实验的吞吐量。

图1:蠕虫到CT协议概述。
此图显示了通过蠕虫到 CT 协议运行蠕虫所需的不同步骤的简要概述。反向转录步骤显示两种可选方法:这些是任何一种芯片的可互换方法。 请点击这里查看此数字的较大版本。

图2:纳米流体qPCR的制备和运行概述。
此图描绘了使用多阵列芯片和单阵列芯片运行纳米流体 qPCR 系统的准备工作。 请点击这里查看此数字的较大版本。
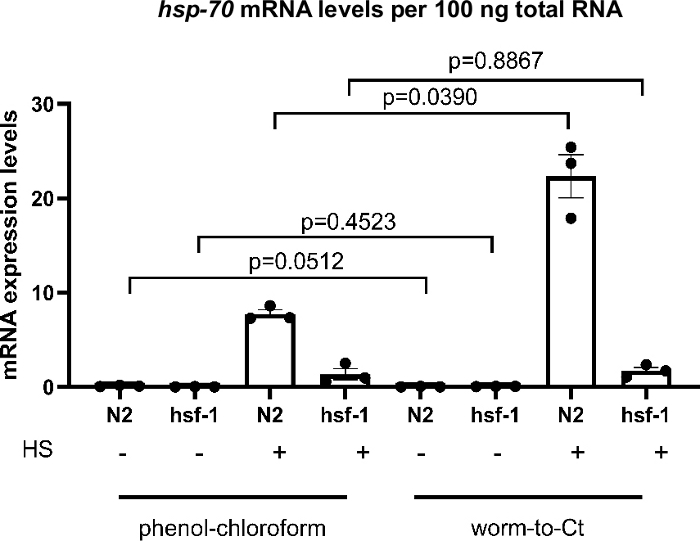
图3:散装样品上的蠕虫到CT协议提供了可靠的结果。
将蠕虫-CT协议与散装样品上的常规硫酸钛酸酯-苯酚-氯仿提取22进行比较。与先前的发现一致,在hsf-1(sy441)突变体23中,hsp记录以响应热冲击的水平下降。 以上直方图描绘了在没有(-)或跟随(+)在34°C下30分钟的短热冲击下hsp-70的感应。 cDNA 是使用适用于 1,000 种蠕虫(左)的硫酸二酸酯-苯酚-氯仿提取,或使用适用于 30 种集合蠕虫的蠕虫到 CT 方法(右)获得的。比较了每100克总RNA中每100克获得hsp-70的表达水平。不出所料,在hsf-1(sy441)中,hsp-70响应热冲击的转录诱导显著减少82.7%使用硫酸钠-苯酚-氯仿,使用蠕虫-CT方法减少92.3%。目标基因的mRNA水平与三个家政基因cdc-42、pmp-3和愤怒-1的平均值进行了正常化。 每个点代表生物复制。数据被转入日志进行统计分析,因为它们不符合参数分析所需的约定。使用 RM 单向 ANOVA 使用西达克的多重比较测试进行统计分析。野生类型 = N2, hsf - 1 = hsf - 1 (sy441)。条形表示平均值的标准错误。请点击这里查看此数字的较大版本。
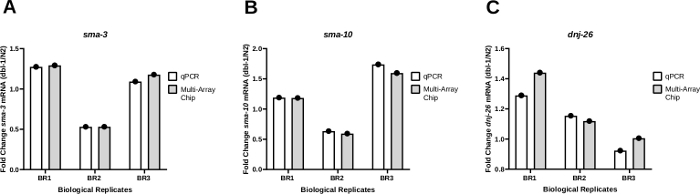
图4:标准qPCR和纳米流体qPCR系统之间的表达模式是一致的。
(A) sma-3 (A)、 sma-10 (B) 或dnj-26 (C) mRNA 的表达水平是通过常规 qPCR 和纳米流体 qPCR (多阵列芯片) 从野生类型菌株 (N2) 和dbl-1 (nk3)淘汰菌株25中通过蠕虫到 CT 生成的 cDNA 的三个生物复制品中确定的。使用 Delta-Ct 方法21确定每个菌株的相对 mRNA 表达水平。折叠变化然后通过将dbl-1 (nk3)蠕虫中获得的表达水平除以 N2 菌株中的相应 mRNA 级别来确定。如A组所示,每个生物复制方法的模式是一致的。(B) 和(C) 分别与sma-10和dnj-26 mRNA 级别的(A) 相同。目标mRNA水平正常化对内务基因cdc-42和pmp-3。使用配对T测试计算每个基因的统计分析,比较通过标准qPCR产生的三种生物复制品和通过纳米流体qPCR产生的复制品的结果。这些比较的P值分别为sma-3、sma-10和dnj-26的0.3113、0.2635和0.3481。 请点击这里查看此数字的较大版本。

图5:在散装样品或单个蠕虫上使用蠕虫到CT方法,当每个蠕虫正常化时,提供类似的表达水平。
在没有热休克的情况下,通过对25种动物中的大多数动物或36个单个个体进行蠕虫-CT,分析(A)hsp-16.1/11、(B)F44E5.4和(C)hsp-70(C)hsp-70(C12C8.1)的表达水平。 当每个蠕虫的数据规范化时,使用这两种方法每个记录获得的每个蠕虫的水平之间没有显著差异。目标基因的mRNA水平与三个家政基因cdc-42、pmp-3和愤怒-1的平均值进行了正常化。条表示平均值的标准错误。统计=配对t测试。请点击这里查看此数字的较大版本。
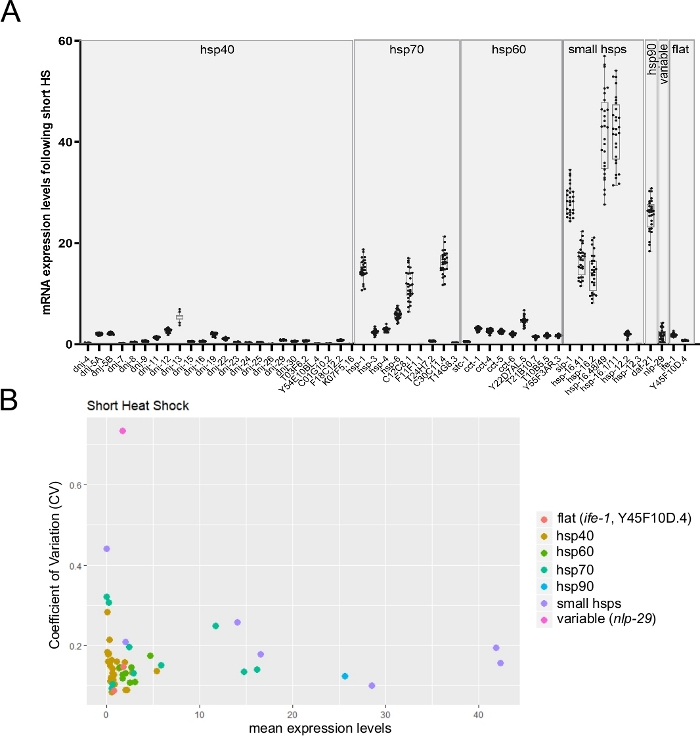
图6:使用蠕虫到CT方法的单个蠕虫的高通量RT-qPCR可以监测基因表达中的个体间变异性。
(A) 暴露在短热冲击下(30分钟,34°C)时获得的53份成绩单的平均表达水平。Boxplot 表示来自单个蠕虫的平均 mRNA 表达的分布(每个蠕虫平均使用三个技术副本)。这些点表示 28 种蠕虫中的表达水平。目标基因的mRNA水平与三个家政基因 cdc-42、pmp-3和 愤怒-1的平均值进行了正常化。(B) 变异26(CV) 作为暴露在短热冲击下53个成绩单的平均mRNA表达函数的系数是从28个个体动物 (B面板中显示的原始数据)计算出来的。一组成绩单包括可变 nlp-29 成绩单27 和两个稳定成绩单(ife-1 和 Y45F10D.4;未公布的数据)。CV 是标准偏差与平均值的比率。此 CV 用于估计单个蠕虫之间成绩单表达的个体间变异性。不出所料,个体间变异性随着平均表达水平的降低而缩小。 请点击这里查看此数字的较大版本。
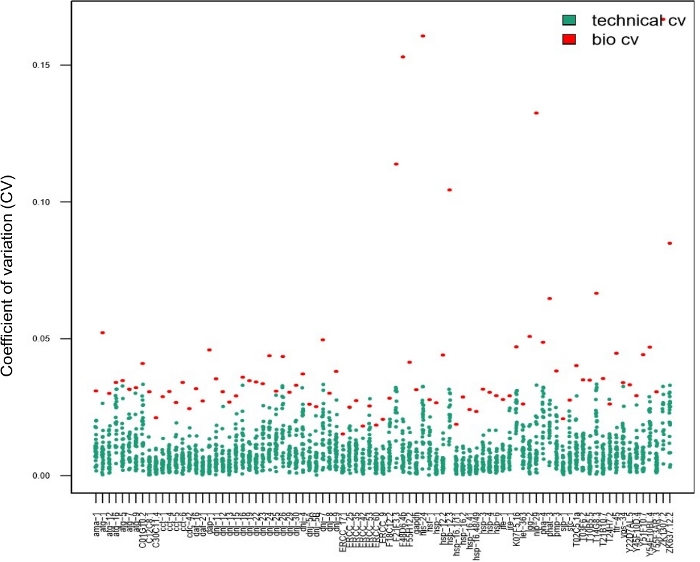
图7:使用纳米流体芯片分析基因表达中的个体间变异性时,不需要技术复制。
此图中显示的数据是在短热冲击(30 分钟在 34 °C 下)后在 28 种蠕虫中获得的。每个红点表示在 28 个单个蠕虫 (生物 CV) 之间检测的一个成绩单的平均成绩单表达水平的变化系数 (CV)。每个蓝点表示从单个蠕虫获得的三个技术副本之间的表达水平的 CV,每个成绩单检测(技术 CV)。该图显示,技术变异性(技术复制之间)远低于生物变异性(单个蠕虫之间),表明在单个蠕虫中测得基因表达时,没有必要在纳米流体基因表达阵列上进行技术复制,类似于单细胞研究14、15、28。请点击这里查看此数字的较大版本。
表1:规划多阵列芯片的布局。 上表显示了一个简单的布局,可在规划多阵列芯片运行时使用。左边是应该填充感兴趣的入门目标的空间,右边是应该填充感兴趣的样本的空间。每个检测和样本阵列都通过芯片进行数字配对。 请点击这里下载此表。
表2:规划单阵列芯片的布局。 上表显示了一个简单的布局,可在规划单阵列芯片运行时使用。左边是应该充满引水目标的空间,右边是应该充满兴趣样本的空间。 请点击这里下载此表。
表3:本研究中使用的RT-qPCR入门列表。请点击这里下载此表。
补充表1:RT-qPCR入门数据库的引言。请点击这里下载此表。
讨论
本文证明,蠕虫到CT协议是一种快速而高效的方法,可以从单个蠕虫或少量蠕虫中提取RNA。纳米流体系统提供的高通量使其成为单个变异度测量定量的理想之选。此外,这种方法的高灵敏度允许检测在低水平表达的基因,低于检测时,使用单蠕虫RNA-seq技术9。
当考虑从单个蠕虫中选择制备cDNA的方法时。Ly等人29 优化了一种依赖蛋白酶K进行层压消化的协议。层基是分子与蠕虫分离的主要障碍,蛋白酶K提供了一种有效的方法来打破它。然而,蛋白酶K必须热灭活,才能使用酶进行反向转录。虽然 Ly 等人使用 10 分钟暴露在 96 °C 下,但此协议中避免了这一步骤,因为 RNA 很容易降解。而不是使用蛋白酶K,重复的冷冻解冻周期被用来打破层。冻土是打破层层的有效方法,因为每个蠕虫可以分离出更多的RNA。Ly等人报告说,每只蠕虫提取的总RNA是35 ng使用蛋白酶K,而此协议获得51.75克±6.74 SEM的总RNA每蠕虫。与标准协议相比,避免热暴露加上预放大步骤显然扩大了蠕虫到CT的动态检测范围。Ly等人报告绝对 Ct 值为 21.1 ± 0.15 hsp - 16.2 和 22.8 ± 0.17 hsp - 70 在热冲击后。使用相同的热冲击条件(1 小时在 30 °C),此协议获得绝对 Ct 值 17.93 ± 0.57 hsp-16.2 和 21.13 ±0.33 hsp-70.这表明冷冻溶解方法提供更高的RNA产量,更适合低表达成绩单。
纳米流体系统是理想的,当调查一组给定的目标成绩单和使用更小(多阵列芯片)或更大的(单阵列芯片)数量的样本允许适应实验的规模。要获得在单个蠕虫中表达的所有成绩单的不偏不倚的图片,显而易见的选择是使用RNA测序。然而,如果实验的重点是一组较小但仍然相对较大的目标基因,则只要研究人员能够接触到纳米流体PCR机器,利用此方案就更具成本效益。纳米流体系统试剂和单阵列芯片的成本估计约为每只蠕虫13欧元,而单蠕虫测序试剂的成本约为每只蠕虫60欧元,不包括测序成本。
在考虑使用何种 PCR 平台时,与纳米流体 qPCR 相结合的蠕虫到 CT 方法在时间和吞吐量方面具有优势。在大约 2 天的工作时间内,可以获得 9,216 个 RT-qPCR 结果,而使用标准 qPCR 平台放大相同数量的目标大约需要 5 个工作周,使用 96 个井板检测,每天运行 4 个板。但是,如果要测试的目标数量较少,则使用蠕虫到 CT 以及标准 qPCR 机器更具成本效益。单阵列芯片无法重新运行,因此运行空井会降低成本效益。
该方法的一个局限性是在多路复用步骤中潜在地形成入门-入门调色器,但这发生在不到 1% 的情况下。虽然蠕虫到CT协议是有效的,并提供可靠的结果,当应用于单一蠕虫,有一个约5%的失败率,这可能相当于在收获步骤期间蠕虫仍然被困在盖或管顶部的情况。
与更标准的技术相比,这种多功能且可靠的方法共同提高了吞吐量和灵敏度。这种方法对于验证高通量屏幕非常有用,是监测或验证单虫基因表达水平的绝佳选择。这种方法可以应用于其他具有挑战性的技术,如从孤立的组织中量化基因表达。例如,全组织(如肠道、性腺或由 FAC 分离的细胞)的分离提供了足够的材料来进行RNA 测序实验。然而,数量有限的材料往往导致重复阅读,从而排除了对罕见成绩单的量化。在这种情况下,如果研究人员只需要监测这些组织或细胞中所有成绩单的子集,则使用基于纳米流体的技术应增加对实验的敏感性,并提高成本效益。
披露声明
作者没有什么可透露的。
致谢
我们感谢莎琳·默多克和巴布拉罕研究所设施的支持。JLP 得到威康信托 (093970/Z/10/Z) 的支持,OC 得到 ERC 638426 和 BBSRC [BBS/E/B/B000C0426] 的支持。
材料
| Name | Company | Catalog Number | Comments |
| 100µM stock primers for genes of interest. | |||
| 20X DNA Binding Dye | Fluidigm | 100-7609 | for 96.96 chip (Single-Array Chip) |
| 2X Assay Loading Reagent | Fluidigm | 100-7611 | |
| 384 well plates | |||
| 8-strip PCR tubes | |||
| 96 well ice block | |||
| 96 well plates | |||
| 96.96 Dynamic Array IFC | Fluidigm | BMK-M-96.96 | Referenced throughout the manuscript as "Single-Array Chip" |
| 96-well sealing tape | |||
| BioMark & EP1 Software | Fluidigm | 101-6793 | Contains: Biomark HD Data Collection software, Real-Time PCR Analysis software, SNP Genotyping Analysis software, Digital PCR Analysis software, Melt Curve Analysis software |
| BioMark or BioMark HD system | Fluidigm | 100-2451 K1 | Referenced throughout the manuscript as "nanofluidics thermocycler" |
| Control Line Fluid Kit for 96.96 and FlexSix IFCs | Fluidigm | 89000021 | Referenced throughout manuscript as "control line fluid" |
| DNA Suspension buffer | TEKnova | T0021 | |
| domed PCR caps | |||
| Exonuclease I | New England BioLabs | M0293L | |
| Exonuclease I reaction buffer | New England BioLabs | B0293S | |
| FLEXsix DELTAgene Sample Reagent | Fluidigm | 100-7673 | referenced throughout manuscript as "sample reagent" |
| FLEXsix Gene Expression IFC | Fluidigm | 100-6308 | referenced throughout manuscript as "Multi-Array Chip" |
| Fluidigm Real-Time PCR Analysis User Guide | Fluidigm | 68000088 | This is the protocol used as a basis for section 2 of the protocol, referenced within the manuscript. |
| IFC Controller MX | Fluidigm | 68000112 I1 | Referenced throughout the manuscript as "nanofluidics PCR priming machine" |
| IFC Controllers SOFTWEAR | Fluidigm | 100-2297 | Contains all softwear and scripts required for running the IFC controller MX |
| liquid nitrogen in dewar | |||
| Microcentrifuge | StarLab | C1301B-230V | Used to spin down PCR tube strips in the protocol. |
| Nuclease Free Water | |||
| plate spinner | Labnet | K4050725 | mini plate spinner, mps 1000. referenced through the protocol as "tabletop plate spinner" |
| Power SYBR Green Cells-to-Ct kit | invitrogen | 4402955 | This kit is that which is adapted for use in nematodes in the Worm-to-CT protocol. Contense: Store Stop Solution, DNase I and 20X RT Enzyme Mix at -20°C. Store Lysis Solution, 2X SYBR RT Buffer (referenced throughout the manuscript as RT buffer), and Power SYBR®Green PCR Master Mix (refferenced througout the manuscript as PCR Master Mix). This Kit is for 400 reactions, kits also available for 40 and 100 reactions. |
| PreAmp Master Mix | Fluidigm | 100-5580, 100-5581 | |
| Reverse Transcription Master Mix—480 reactions | Fluidigm | 100-6299 | One tube also available for 96 reactions (PN100-6298) |
| Rnase Free water | |||
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad Laboratories | 172-5211 | referenced throughout the manuscript as "fluorescent probe supermix" |
| Standard 96 and 384-well Thermal Cycler | |||
| TE Buffer (10mM Tris, pH8.0, 1.0mM EDTA | TEKnova | T0224 | Referenced throughout the manuscript as Tris-EDTA buffer |
| ThermoMixer C | Eppendorf | 5382000031 | Referenced throughout the text as "thermal mixer" |
| Trizol | Thermo-Fisher | 15596026 | Referenced through the protocol as "guanidium thiocyanate-phenol-chloroform" |
| Warm water bath |
参考文献
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biology. 4 (10), 309 (2006).
- Semrau, S., van Oudenaarden, A. Study lineage decision-making in vitro: emerging concepts and novel tools. Annual Review of Cell Developmental Biology. 31, 317-345 (2015).
- Kelsey, G., Stegle, O., Reik, W. Single-cell epigenomics: Recording the past and predicting the future. Science. 358 (6359), 69-75 (2017).
- Eling, N., Morgan, M. D., Marioni, J. C. Challenges in measuring and understanding biological noise. Nature Review Genetics. 20 (9), 536-548 (2019).
- Kaufmann, B. B., van Oudenaarden, A. Stochastic gene expression: from single molecules to the proteome. Current Opinion in Genetics & Development. 17 (2), 107-112 (2007).
- Corsi, A. K., Wightman, B., Chalfie, M. A Transparent window into biology: A primer on Caenorhabditis elegans. WormBook. , (2015).
- Casanueva, M. O., Burga, A., Lehner, B. Fitness trade-offs and environmentally induced mutation buffering in isogenic C. elegans. Science. 335 (6064), 82-85 (2012).
- Raj, A., Rifkin, A. A., Andersen, E., van Oudenaarden, A. Variability in gene expression underlies incomplete penetrance. Nature. 463 (7283), 913-918 (2010).
- Perez, M. F., Francesconi, M., Hidalgo-Carcedo, C., Lehner, B. Maternal age generates phenotypic variation in Caenorhabditis elegans. Nature. 552 (7683), 106-109 (2017).
- Serra, L., et al. Adapting the Smart-seq2 Protocol for Robust Single Worm RNA-seq. Bio-protocol. 8 (4), 2729 (2018).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Biology. 14 (8), 1007559 (2018).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nature Review Genetics. 16 (3), 133-145 (2015).
- Ståhlberg, A., Kubista, M. The workflow of single-cell expression profiling using quantitative real-time PCR. Expert Review of Molecular Diagnostics. 14 (3), 323-331 (2014).
- Livak, K. J., et al. Methods for qPCR gene expression profiling applied to 1440 lymphoblastoid single cells. Methods. 59 (1), 71-79 (2013).
- Fluidigm. . Real-Time PCR Analysis User Guide. , (2018).
- Huggett, J. F., et al. The digital MIQE guidelines: Minimum Information for Publication of Quantitative Digital PCR Experiments. Clinical Chemistry. 59 (6), 892-902 (2013).
- . U. S. National Library of Reference. Primer Blast Available from: https://www.ncbi.nlm.nih.gov/tools/primer-blast/ (2020)
- Bustin, S. A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems. Journal of Molecular Endocrinology. 29 (1), 23-39 (2002).
- Nolan, T., Huggett, J., Sanchez, E. . Good Practice guide for the application of quantitative PCR (qPCR), LGC. , (2013).
- Yuan, J. S., Reed, A., Chen, F., Stewart, C. N. Statistical analysis of real-time PCR data. BMC Bioinformatics. 22 (7), 85 (2006).
- He, F. Total RNA Extraction from C. elegans. Bio-protocol. Bio-101. 47, (2011).
- Hajdu-Cronin, Y. M., Chen, W. J., Sternberg, P. W. The L-Type Cyclin CYL-1 and the Heat-Shock-Factor HSF-1 Are Required for Heat-Shock-Induced Protein Expression in Caenorhabditis elegans. Genetics. 168 (8), 1937-1949 (2004).
- Douglas, P. M., et al. Heterotypic Signals from Neural HSF-1 Separate Thermotolerance from Longevity. Cell Reports. 12 (7), 1196-1204 (2015).
- Zhang, X., Zhang, Y. Dbl-1 a TGF-beta is essential for C. elegans aversive olfactory learning. Proceedings of the National Academy of Sciences of the United States of America. 109 (42), 17081-17086 (2012).
- Sørensen, J. B. The Use and Misuse of the Coefficient of Variation in Organizational Demography Research. Sociological Methods & Research. 30 (4), 475-491 (2002).
- Pujol, N. Distinct innate immune responses to infection and wounding in the C. elegans epidermis. Current Biology. 18 (7), 481-489 (2008).
- Brennecke, P., et al. Accounting for technical noise in single-cell RNA-seq experiments. Nature Methods. 10 (11), 1093-1095 (2013).
- Ly, K., Reid, S. J., Snell, R. J. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 7 (2), 59-63 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。