このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
シュワン細胞表現型仕様を評価するための細胞外マトリックス微小環境の調製
要約
この方法論は、基質剛性、タンパク質組成および細胞形態などの細胞外マトリックスの手掛かりがシュワン細胞(SC)表現型を調節するメカニズムを示すことを目的とする。
要約
外傷性末梢神経系(PNS)の傷害は現在、完全な機能的回復を取り戻すための適切な治療法を欠いている。シュワン細胞(SC)は、PNSの主要なグリア細胞として、損傷後の再生細胞表現型に脱分化することによってPNS再生を促進する上で重要な役割を果たす。しかし、SCの分化状態は、再生に必要な期間を通じて維持することが困難であり、周囲の細胞外マトリックス(ECM)の変化の影響を受けます。したがって、SCと異なるECMの間の複雑な相互作用を決定して、SCの再生電位の手掛かりを提供することが不可欠である。これに対処するために、異なるECMタンパク質を調整可能なポリジメチルシロキサン(PDMS)基質に吸着させ、剛性およびタンパク質組成を変調できるプラットフォームを提供する戦略が作成されました。SCをチューナブル基材に播種し、SC表現型のダイナミクスを表す重要な細胞機能を測定した。SCタンパク質発現と細胞形態との相互作用を例示するために、個別のマイクロコンタクト印刷細胞パターンに加えてSCの異なる播種密度が利用され、免疫蛍光染色およびウェスタンブロットによって特徴付けられていた。結果は、より小さな広がり領域を有し、細胞伸びの範囲が高い細胞がSC再生表現性マーカーのより高いレベルを促進することを示した。この方法論は、SCのECMと細胞機能の重要な関係を解明し始めるだけでなく、末梢神経修復における生体材料の将来の最適化のためのガイドラインを提供する。
概要
末梢神経系(PNS)傷害は、患者の生活の質を損ない、社会経済的要因11,22を通じて重大な影響を及ぼす医療における主要な臨床的課題であり続ける。シュワン細胞(SC)は、PNSの主要なグリア細胞として、PNS再生を誘導し、短いギャップ傷害における機能的回復を助けるために必要な分子および物理的手がかりを提供する。これは、SCがミエリン化またはレマク表現型3から「修復」細胞表現型に分解する顕著な能力によるものである。修復SCは、いくつかの点で特徴的な細胞表現型である。傷害後、SCは細胞周期に再入することによって増殖速度を増加させ、再活性化を容易にするためにいくつかの転写因子の発現を開始する。c-Junやp75 NTRなどのこれらの要因は、ミエリン塩基性タンパク質(MBP)などのSCマーカーを下方制御する一,方でアップレギュレートされる。さらに、SCは形態を変化させ、細長くなり、互いに整列して傷害部位6を横切ってブンナーバンドを形成する。これは、軸索が正しい遠位ターゲット7まで拡張するための物理的なガイダンスメカニズムを提供します。しかし、SCが短いギャップ傷害で神経再生を促進する能力を有しているにもかかわらず、機能的回復の結果は重度の傷害において依然として悪いままである。これは、細胞外マトリックス(ECM)誘導の喪失、ならびにSCが長期間にわたって回生表現型を維持できないことに起因する8。
神経の再生および回復プロセスは、損傷後の基底層層の状態と密接に結びついている。基底層ラミナは、指導を容易にし、損傷後に無傷のままである場合に軸索およびSCにECMバウンドキューを提供する神経の周りのECMの層である9。ECMの状態とセルに行列結合キューを提供する能力は非常に重要であり、以前は,10、11、12、13、14のさまざまな異なるコンテキストで探求されてきました。10,11,12,1314例えば、ECMの剛性が増殖や分化,11、15、16,15などの細胞機能を導くことができることが示されている。16ECMの組成はまた、明確な細胞応答を導き、細胞内シグナル伝達経路17、18,18を介した移動および分化などの細胞行動を調節することができる。さらに、細胞形態は、広域および細胞伸長を含む、機能を調節する上で大きな役割を果たし、ECM結合キュー19、20,20によって支配することができる。多くの以前の研究は、定義された系統に分化する幹細胞に焦点を当ててきましたが、SCは健康な神経内の恒常性の成人SCから、神経損傷55、2121に続くECMを再モデリングしながらタンパク質および成長因子を分泌することができる修復SCに表現型を変更する同様の能力を有する。したがって、神経再生のためにこの能力を最終的に活用するための洞察のために、先天的なSC再生能力とECMバウンドキューとの関係の根底にあるメカニズムを特定することが特に重要です。
これに対処するために、我々は、機械的剛性およびリガンドタイプが生理学的に関連する範囲で容易に調整できる細胞培養基質を生成するための詳細な方法論を開発した。ポリジメチルシロキサン(PDMS)は、ポリアクリルアミドゲルと比較して、その高度に調整可能な力学のために基材として選ばれました, 最大ヤング率は約12 kPaでPDMSと対比する約12 kPa22,,23,,24.これは、最近の研究では、発達中にウサギ坐骨神経のヤング率が50kPaを超えることができ、それによってPNS内の神経の剛性の範囲が以前に調べられたよりも広いことを示唆しているので、目の前の仕事に有益である。異なるタンパク質は、SC挙動上の力学とリガンドの組み合わせ調節を分析するためにPDMS基質に吸着することが可能です。これにより、PNS再生プロセスに存在する複数の微小環境手掛かりの調査と、基板25の剛性のみに焦点を当てた作業に対する高度なタンナビリティの比較を可能にする。また、これらの操作細胞培養基質は、免疫物質化学、ウェスタンブロット、定量ポリメラーゼ連鎖反応(q-PCR)などの多くの定量分析方法と互換性がある。
この工学的細胞培養プラットフォームは、各ECM結合信号の個々のタンナビリティの高いレベルに起因する機械経路の解析に非常に適しています。また、マイクロコンタクト印刷を含む細胞マイクロパターニングの一般的な方法は、他のECM結合キュー24に関連して細胞形状を解析する制御された細胞接着を可能にする基質上で達成することができる。これは、細胞集団の伸びを促進する線パターン化基質が、神経再生中にビュンナーバンド内の細長く再生的なSCを模倣し、研究するためのツールを提供するため、非常に重要です。また、細胞形態は複数の細胞機能の強力な調節因子であり、制御されない場合には交和実験結果を導入する可能性がある26,27。26,,ECM キュー28、 29、2930によって制御される SC 回生表現型を制御するメカニズムに、重要な注意が提供されています。これは、PNS神経再生の補助のための神経誘導導管として適用することができる生体材料の設計に関する洞察を提供するために不可欠です。これらの詳細なプロトコルは、最終的には、ECMバインドキューによって規制されるSCおよび他の細胞タイプの機能のメカニズムを解読する潜在的なツールとして適用することができる。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. チューナブル細胞培養基質の調製と特性評価
- 基板調製
- PDMSベースエラストマーと硬化剤を、10:1~60:1の比率でピペットチップを使用して、気泡が混合物内に均質に分散するまで激しく混合します。気泡が消散するまで真空乾燥を使用して気泡を除去します。
注:PDMS重合の間、硬化剤は、最終的なポリマー所望の機械的特性を提供するために、基エラストマーと架橋します。クロスリンク比を調整して、PDMS の剛性を変更できます。 - 22mm x 22 mmの正方形または円形のカバースリップに乾燥PDMS混合物を滴下(約0.2 mL)配置し、2500 rpmのスピンコート機でカバースリップを30s回転させます。
- PDMSを固めるために、60°Cのオーブンで1〜2時間、または室温でカバースリップをインキュベートします。
- 7分間のUV-オゾンクリーナー(UV波長:185 nmおよび254 nm)を使用してカバースリップを処理し、表面親水性を高めます。滅菌した6ウェルプレートに入れる。
- 細胞培養に使用する前に、少なくとも30分間、70%エタノールで基質をインキュベートする。
注意:UV-オゾンクリーナーは、人間に有害なオゾンを生成することができます。化学煙フードで、または換気のいくつかのフォームで動作します。 - 37°Cの無菌インキュベーターに60分間、タンパク質溶液(10μg/mLコラーゲンI、フィブロネクチン、またはラミニン)にカバースリップを浸します。
注:UV-オゾン処理後、PDMS表面は依然として疎水性である可能性があります。ウェルプレートを回転させて、各カバースリップがタンパク質溶液で覆われていることを確認します。 - タンパク質溶液を吸引し、リン酸緩衝生理食塩水(PBS)3xでカバースリップを洗浄する。
- 2.5%のトリプシンを使用して市販のEDTA溶液(1x)を使用して、2.5%のトリプシンを使用して、2.5%の細胞をマッサージし、ヘモサイトメーターを有する細胞を数えるので、RT4-D6P2Tシュワン細胞株(SC)を受け継ぎ皿から再中断します。必要なセル密度で調整可能な PDMS サーフェス上の SC をシードします。SCシード密度は、用途によって異なる場合があります。
- 細胞を所望の細胞培養パラメータ(湿度90%、CO2 5%、37°Cなど)で、実験の長さに維持します。210%のウシ胎児血清(FBS)と1%ペニシリンストレプトマイシンを添加したダルベックコの修飾イーグル培地(DMEM)を細胞培養培地として使用してください。
- PDMSベースエラストマーと硬化剤を、10:1~60:1の比率でピペットチップを使用して、気泡が混合物内に均質に分散するまで激しく混合します。気泡が消散するまで真空乾燥を使用して気泡を除去します。
- マイクロパターン化基板調製
- コンピュータ支援設計(CAD)ソフトウェアを使用して、希望の幾何学および細胞接着領域(900 μm2、1,600μm2および2,500 μm2)を描きます。商業サプライヤーからのパターンに基づいてクロムフォトマスクを作成します。
- クリーンルームやダストフリー環境では、シリコンウェーハを製造するために標準的なフォトリソグラフィ技術を使用します(プロトコルは他の場所で詳述されています31)。この特定のアプリケーションの重要なパラメータは次のとおりです: フォトレジスト: SU-8 2010;フォトレジストを分散させるスピンプロファイル:100rpm/sの加速度で10sの500rpm、300rpm/sの加速度で300rpm、UV光の露出エネルギー:130 mJ/cm2.
注:シリコンウェーハのパターンの高さは、これらのパラメータに従って約10 μmです。矩形または三角形パターンの外側のエッジの周りの潜在的な亀裂は、ステップ1.2.2の後に光顕微鏡を使用して見ることができます。シリコンウエハを190°Cで30分間焼き、亀裂をなくします。 - パターン化されたシリコンウェーハを直径150mmの直径x15mm高さのペトリ皿の中に入れ、ステップ1.1.1で作ったとおりに脱ガスPDMS(混合比10:1)をシリコンウェーハに注ぎます。
メモ:マイクロコンタクト印刷手順で取り扱いが容易になるように、PDMSの厚さが5mm以上であることを確認してください。 - シリコンウエハのPDMSをオーブンで一晩60°Cで固めます。PDMS を室温まで冷却します。外科用メスを使用してシリコンウエハーの正しいパターンを含む30mm x 30mmの正方形のスタンプを正確にカット。シリコンウエハを破損しないでください。
注:シリコンウェーハは、イソプロパノールで洗浄した後、より多くのスタンプを生成するために、この時点で何度も再利用することができます。 - PDMSスタンプと調整可能なカバーリップ(ステップ1.1.1~1.1.3で作成)を70%エタノールに30分間浸漬して滅菌します。
- マイクロコンタクト印刷後のPDMSスタンプによるマイクロパターンの有効性を確認するには、PDMSスタンプのパターン全体をカバーするために、濾過空気流およびピペット50μg/mL BSA(テキサスレッド共役)溶液を用いてPDMSスタンプの表面を乾燥させます。
- PDMS スタンプを BSA 溶液と共に 1 時間室温でインキュベートし、タンパク質吸着を可能にします。
- 濾過された空気流を使用して調整可能なカバーリップの表面を乾燥させ、ステップ1.1.5で説明したように表面の親水性を高める。
- PDMSスタンプを空気乾燥させて残りのBSA溶液を除去します。
メモ:残りの溶液はマイクロコンタクト印刷時にカバースリップにスタンプをスライドさせるので、BSA溶液がスタンプから完全に取り除かれるので注意してください。 - スタンプのパターン付き側を、カバースリップ表面のBSA吸着のための調整可能なカバースリップとの対角接触に持って来ます。スタンプをカバースリップに5分間軽く押し付けます。
メモ:スタンプが曲がり、スタンプとカバースリップの間に非特異的な接触を引き起こすので、スタンプに過度の力を加えないでください。マイクロコンタクト印刷を成功させるためには、スタンプに適用される適切な力が不可欠です。 - FITC(フルオレセイン・イソチオシアネート)フィルターを用いて蛍光顕微鏡を用いてマイクロパターンを調べます。
- 蛍光パターンではなく細胞接着領域をプリントするには、BSAタンパク質のラミニンを置換し、ステップ1.2.5~1.2.10を繰り返します。
- カバースリップからスタンプを取り除き、カバーリップを殺菌された6ウェルプレートに移します。カバースリップの表面を覆い、室温で1時間インキュベートするために、各ウェルに0.2%w/vプルロニックF-127溶液の2 mLを加えます。
注:Pluronic F-127はPDMS表面に吸着することができ、PDMS表面の疎水性を増加させ、接着から細胞をブロックすることができます。 - 吸引Pluronic F-127溶液を、細胞を播種する前にPBSで5倍、細胞培養培地で1xを洗浄した。SC の一般的なシード密度は 1,000 セル/cm2です。
- 45分後の細胞播種、細胞培養培地を取り除き、PBS 2xでカバーリップを洗浄し、複数のSCが同じパターンに付着するのを防ぎます。細胞培養環境で細胞を48時間保存してから定量化します。
- ラインパターン細胞培養基質を作成して整列した細胞を調べるには、ステップ1.2.1~1.2.4に従ってマイクロコンタクト印刷用スタンプを作成します。
注:スタンプの裏地パターンの溝/隆起の寸法は、輪郭を描いたセルに対して50 μm x 50 μmです。スタンプの合計寸法は10mm×10mmです。 - スタンプを目的の線パターンのみを含む寸法に切り取ります。
注:CADでスタンプを作成する場合、ラインパターンの周りのスタンプのパターンなしの領域は、マイクロコンタクト印刷後のセル接着領域に対応します。したがって、スタンプを切り取るときにパターン化されていない領域を除去し、表面にシードされたすべてのSCがパターンに従っていることを確認する必要があります。 - ステップ 1.1.1 から 1.1.3 に従って、2 つのペトリ皿をコーティングする調整可能な PDMS 表面を準備します。
注:これは、カバースリップではなく、ペトリ皿表面自体をカバーするPDMSになります。 - ステップ 1.2.5 から 1.2.10 に従ってマイクロコンタクト印刷を行い、PDMS コーティングされたペトリ皿の 1 つにラインパターンセル接着領域を印刷します。
注:60 mm x 15 mm のペトリ皿の表面積には、6 つの PDMS スタンプのライン パターン領域を含めることができます。 - スタンプを取り除き、ペトリ皿に0.2%w/vプルロニックF-127溶液の4 mLを充填し、1時間インキュベートします。
- マイクロコンタクト印刷後、PDMSスタンプの各側を70%エタノール3xでリンスし、空気で乾燥させます。PDMS スタンプを回転させ、ステップ 1.2.5 ~ 1.2.10 に従って、2 番目のペトリ皿のスタンプのパターン化されていない側を使用して、未パターンのセル接着領域を印刷します。ステップ 1.2.13 を繰り返します。
- 皿からF-127溶液を吸引し、PBSで3倍洗浄し、次いで新鮮な細胞培養培地を用いて1倍の洗浄を行った。皿にSCをシード。
注: ラインパターンの皿のシード密度は 5,000 セル/cm2で、パターン化されていない皿の場合は 10,000 セル/cm2です。 - SCを48時間所望の条件で維持し、プロトコルに従ってSCライセート32を準備する。
注:パターンなしの皿のセルシード密度は、ラインパターンの皿の場合よりも2倍高く、ラインパターンの皿にはパターン化されていない皿の細胞接着領域が半分しか含められていないためです。- ライセートを調製するには、十分な放射免疫沈降アッセイ(RIPA)バッファーを10 mL円錐形遠心分離管に移します。リパバッファーで1:100の比率でプロテアーゼとホスファターゼ阻害剤(100x)を希釈し、ピペット処理でよく混合します。
- 氷冷PBS(1x)で2分間洗浄し、ステップ1.2.23.1から調製した溶液を、各細胞接着領域(PDMSスタンプと吸着したタンパク質と接触した領域)に80μLを加えて、シャーレ内に入れなさい。氷ブロック上の溶液を15分間インキュベートします。
注:この溶液は、他の場所でのプルロニックF-127吸着の疎水性のために、細胞接着領域にのみ留まります。この機能により、ラインパターン SC のタンパク質抽出が成功し、十分に有効になります。 - 5分間のセルスクレーパーでSCを削り取り、1.5 mLマイクロ遠心チューブにリゼートを集める。
- マイクロ遠心分離液は、12,000 x gで4°Cで15分間にリセートする。1,000 μL ピペットで上清を収集し、クリーンマイクロ遠心分離チューブに移します。細胞のライセートを-20°Cで保存する。
- 基質特性評価
注:カバースリップ上のポリマーの力学を特徴付けるために、バルク圧縮試験11、33または33原子間力顕微鏡検査34を含む複数の方法が一般的に採用されています。このプロトコルは、一括圧縮テストの概要を示します。- 所望の混合比のPDMS前駆体(ステップ1.1)を30mmのペトリ皿に注ぎ、ペトリ皿内のPDMS層の厚さが少なくとも20mmであることを確認する。
- 1時間後に60°Cオーブンから固化PDMSでペトリ皿を取り出し、室温で冷却することができます。ポリマーを10mm×10mmの正方形に切ります。キャリパーを使用してPDMSの厚さを測定します。
- 圧縮力測定機のステージ上にPDMSを配置します。圧縮力センサー(モデル:112C)をセンサーポートに差し込み、センサーを試験機の軸に固定します。
- センサーの高さを、計測器の前面パネルの「ジョグ」コントロールを使用して、PDMSスタンプの約0.5cm上に調整します。
- [テストのセットアップ] ウィンドウを使用して関連するソフトウェアを開き、[サーボ プロファイル] を選択して、[セグメント] ウィンドウを開きます。[セグメント] ウィンドウで、テストに必要な "制御率" と "終了金額" を入力します。
注: 制御速度は、センサーが PDMS に向かって下がる速度を決定します。終了量は、センサーが移動する合計距離を決定します。 - ソフトウェアのコントロール パネルにある[Z]ボタンを使用して、この時点ですべての測定値をリセットします。
- 1-2ニュートン(N)がロードされるまで、PDMSに軽く接触するようにセンサーを下に移動します。センサーが移動する荷重と距離がソフトウェアに表示されます。
- 「Z」ボタンを利用した後、「再生」を使用して測定を実行し、ファイルの記録力と距離を保存します。
- PDMS の実験条件ごとに、ステップ 1.3.3 から 1.3.8 を繰り返します。
- ファイルを開き、次の数式を使用して、各比率の PDMS のヤング率 (E) を計算します。(F = 圧縮力、A = PDMS スタンプの面積、センサーの移動距離":"L = およびL0 = PDMS スタンプの元の厚さ)。
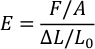
2. 調整可能な基材上の細胞特性の定量化
- 増殖アッセイ
- ステップ1.1.9から6ウェルプレートの5,000セル/cm2の密度で調製した基質上のSCをシード。標準的な細胞培養条件(37°Cおよび5%CO2)でSCが48時間インキュベートすることを可能にする。
- 10 mM ブロモデオキシウリジン(BrdU)ストック溶液を12 mLの37°C細胞培養培地に希釈し、ピペットとよく混合して10 μM BrdUラベリング溶液を作ります。
- 細胞培養培地を取り出し、SC2xをPBSで洗浄する。
- 各ウェルに2mLのBrdUラベリング溶液を加え、2時間の間にSCをインキュベートします。
注:BrdU標識溶液のインキュベーション時間は、特定の細胞増殖速度に依存します。RT4-D6P2T SC線は高い増殖速度を有するため、2時間のインキュベーション時間が使用された。 - BrdUラベル付け溶液を取り外し、PBSでSC 3xを洗います。各ウェルにPBSに3.7%ホルムアルデヒド1 mLを加え、細胞固定のために15分間室温でインキュベートします。
注意: ホルムアルデヒドはヒト発がん性物質です。したがって、適切な保護を備えた化学発煙フードの内部ですべての作業を行います。
注:PBSで洗浄する場合、ウェル内に細胞培養培地がなく、したがってPDMS表面は疎水性であってもよい。細胞の損傷を防ぐために、基板の表面を完全に乾燥させないように注意してください。 - 吸引ホルムアルデヒド溶液をPBSで3倍洗浄(各3分)PBSを取り除き、各ウェルに0.2%トリトンX-100を1mL加えて細胞膜を透過させます。トリトンX-100溶液を室温で20分間インキュベートします。
- トリトンX-100溶液を取り出し、SC 3xをPBSで洗浄します(各3分)。
- 各ウェルに1 mLを加え、氷上で10分間インキュベートし、1N HClを取り除き、各ウェルに2 N HClの1 mLを加え、室温で10分間インキュベートします。
- 0.2 mM Na2HPO4の 182 mL と 0.1 mM クエン酸の 18 mL を混合し、抗原検索用のリン酸/クエン酸バッファーを生成します。2 N HClを取り出し、各ウェルに1 mLリン酸/クエン酸バッファーを加え、室温で10分間インキュベートします。
- PBSで0.2%トリトンX-100でSC 3倍を洗浄します。PBSに3%のウシ血清アルブミン(BSA)の2mLを各ウェルに加え、抗体の非特異的結合をクロックするために室温で30分間インキュベートする。
- 希薄なBrdU一次抗体は、BrdU染色溶液に対して1:300の比率で3%BSA溶液でアレクサ・フルオール488と共役した。プレートがアルミホイルで覆われている間、室温で一晩染色液とSCをインキュベート。
- 増殖を定量化するために、蛍光顕微鏡のFITCおよびDAPIチャネルを用いて、それぞれBrdUと核を検出する画像SC。"nd.2" ファイルとして画像を保存します。
- 同じ空間位置で撮影した各画像の"nd.2"ファイルを開きます。
- 画像解析ソフトウェアを開きます。背景を右クリックして、 "分析コントロール" のセクションにあるウィンドウ "自動測定結果" と "自動測定" を開きます。
- [カウントと分類] メニューで、[カウント] を選択します。FITC画像では、緑色蛍光(BrdU陽性)を示す各核をクリックし、画像を右クリックします。
注: BrdU の正のセルの数は、「オートメーションと測定」のウィンドウに表示されます。 - DAPI イメージの場合、ステップ 2.1.14 を繰り返して、総核数をカウントします。この画像の BrdU 陽性セルの割合を計算します。
- 統計的な目的のために他の画像に対してステップ2.1.13から2.1.16を繰り返し、各基質条件のBrdU陽性細胞の平均パーセンテージを計算します。
- 免疫蛍光画像解析によるc-Jun発現の定量化
- ステップ1.1.9および1.2.23から6ウェルプレートの内部で調製された細胞は、前述の手順で固定され、透過化される(ステップ2.1.5-2.1.7)。
注:異なるECM条件の細胞間で蛍光強度の正確な比較を行うために、同じパラメータを持つすべてのサンプルを使用して、すべてのサンプルにカメラ設定を同一に適用します。 - イメージを ".nd2" ファイルとして保存します。
- 画像解析ソフトウェアを開きます。右クリックの背景は、ウィンドウ 「 分析制御」 のセクションにウィンドウ "自動測定結果" と "自動測定" を開きます。
- [自動測定結果] で 、[オブジェクト データ] を選択します。[測定の更新を継続する] ボタンを有効にします。
- ソフトウェアの上部のパネルで、[計測] を選択し、その後に "オブジェクト機能" を選択します。「測定用に選択された」のセクションに「平均強度」を追加します。
- c-Junと核のイメージを含む2つの".nd2"画像ファイルを開いてマージします。
- 上部のパネルで[ROI]を選択し、[長方形のROIを描画]を選択します。単一セルの核領域を含む矩形領域を描画します。
注:c-Jun発現は核35内に集中している。 - ソフトウェアの上部パネルで、「バイナリ」と「しきい値を定義」を選択すると、新しいウィンドウが表示され、c-Jun蛍光領域が正確に定義されます。
- 新しいウィンドウで、[フル イメージ/ROI を使用] をクリックして、プログラムをフル イメージ モデルから ROI モデルに切り替えます。ウィンドウの左側にあるルックアップ テーブルを調整して、四角形の ROI 内で強調表示された領域のサイズや形状を調整するには、"強度" を使用します。
注: ハイライトされた領域のサイズや形状が核と同じになるように注意してください。 - [OK]ボタンをクリックして、[自動測定結果] ウィンドウの平均 FITC 強度を取得し、[データの保存] をクリックします。
- "自動測定" のウィンドウで、赤い強調表示された領域を削除します。左側のパネルで、長方形の ROI を選択して削除するには、ポインティング ツールを使用します。
- ステップ 2.2.6 ~ 2.2.11 を繰り返して、追加の各セルの平均 FITC 強度を測定します。
- "自動測定結果" のウィンドウ領域で 、[保存済み] を選択すると、保存されているすべてのデータが表示されます。エクスポート機能を使用し、エクスポートされたスプレッドシートを保存し、追加の計算を実行するには、Excelにデータを選択します。
- ステップ1.1.9および1.2.23から6ウェルプレートの内部で調製された細胞は、前述の手順で固定され、透過化される(ステップ2.1.5-2.1.7)。
- 核伸長の定量化
- ステップ 1.2.22 から準備された SC を修正し、ステップ 2.1.5-2.1.7 に従ってパーメアビライズします。DAPI付き取り付け媒体を使用して核染色を行います。
- DAPIチャンネルと40倍の対物レンズを使用して、サンプルの画像を取得し、".nd2"ファイルとして保存します。
- 手順 2.2.3 および 2.2.4 に従って、画像解析ソフトウェアの [自動測定結果] および [自動測定] ウィンドウを開きます。
- [自動測定結果] で、"オプション" 関数の後に "オブジェクトの選択" を使用します。[機能] 列で 、[伸び] を選択し、[測定対象として選択]列に追加します。この機能を有効にするには、「測定の更新を継続」を使用してください。
- 核イメージを含む "nd.2" イメージ ファイルを開きます。[自動計測] ウィンドウで、[自動検出] 機能を選択し、核を選択します。画像を右クリックすると、測定された核アスペクト比が「自動測定結果」に表示されます。
- ステップ 2.3.5 を繰り返して、画像内の他の核の核アスペクト比を定量化します。[自動測定結果] のウィンドウで [データを保存] を選択します。
- 追加の画像については、2.3.5 から 2.3.6 を繰り返します。分析のために 2.2.13 で以前に行ったとおりに、データをスプレッドシート ファイルにエクスポートします。
- タンパク質発現を定量化するウェスタンブロット
- 他の場所で詳述されたウェスタンブロット分析のための標準的なプロトコルに従ってください32.研究で使用される抗体の希釈は以下に示されている: ウサギ抗 c-Jun 1:2,000;マウス抗β-アクチン 1:1,000;ウサギ抗p75NTR 1:1,000;ウサギ抗ミエリン塩基性タンパク質 1:1,000;抗マウス/ウサギIgG、HRP結合抗体1:10,000。
Access restricted. Please log in or start a trial to view this content.
結果
SC表現型上の基質剛性とタンパク質組成との相互作用を分析・定量化するために、調整可能なPDMS細胞培養基質を開発した(図1A)。異なる塩基におけるポリマーの圧縮試験:硬化剤比は、基材のヤング率(E)を定量するために利用された(図1B)。得られたモジュラス値の範囲は、生理学的に関連する基質条件を表す。基質の調製後、SCを培養し、チューナ?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
SCは、その表現型の変換と神経損傷後の再生可能性のために神経再生を促進することができます。しかし、ECMキューがこの再生能力をどのように調節するのかはほとんど不明であり、神経再生を促進することを目的とした生体材料の開発だけでなく、神経再生に関与するメカニズムの理解を妨げる可能性がある。この相互作用を検討するために、細胞培養基質は、剛性、タンパク質コーティ?...
Access restricted. Please log in or start a trial to view this content.
開示事項
潜在的な利益相反は著者によって報告されなかった。
謝辞
著者らは、シンシナティ大学からの資金援助を感謝して認めている。著者らはまた、シンシナティ大学先端材料特性評価研究所のロン・フレニケンの支援に感謝する。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Albumin from Bovine Serum (BSA), Texas Red conjugate | Thermo Fisher Scientific | A23017 | BSA staining to show micropatterns |
| Anti-mouse IgG, HRP-linked Antibody | Cell Signaling Technology | 7076S | Antibody used for western blot analysis |
| Anti-rabbit IgG, HRP-linked Antibody | Cell Signaling Technology | 7074S | Antibody used for western blot analysis |
| BrdU | Thermo Fisher Scientific | B23151 | Reagent used to measure cell proliferation |
| BrdU primary antibody conjugated with Alexa Fluor 488 | Thermo Fisher Scientific | B35130 | Used to visualize BrdU in cell proliferation assays |
| Collagen I | Thermo Fisher Scientific | A10483-01 | Protein used to coat coverslips |
| Compression force test machine | TestResources | Instrument to quantify mechanical properties of polymers | |
| Dulbecco's Modified Eagle Medium | Thermo Fisher Scientific | 11965092 | Cell culture medium |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | Cell culture medium supplemental |
| Fibronectin | Thermo Fisher Scientific | 33010-018 | Protein used to coat coverslips |
| Fluorescence microscope | Nikon | Eclipse Ti2 | Fluorescence microscope |
| Halt Protease and Phosphatase Inhibitor Cocktail (100X) | Thermo Fisher Scientific | 78440 | Protease and Phosphatase Inhibitor |
| Laminin | Thermo Fisher Scientific | 23017015 | Protein used to coat coverslips |
| Mounting medium with DAPI | Thermo Fisher Scientific | P36971 | Coverslip mountant and nuclei staining |
| Mouse c-Jun primary antibody | Thermo Fisher Scientific | 711202 | Primary antibody to visualize c-Jun protein |
| Mouse β-Actin primary antibody | Cell Signaling Technology | 3700S | Loading control for western blot experiments |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | Cell culture medium supplemental |
| Photoresist SU 2010 | KAYAKU | SU8-2010 | Photoresist |
| Pluronic F-127 | Sigma Aldrich | P-2443 | Block non-specific protein binding |
| Rabbit c-Jun primary antibody | Cell Signaling Technology | 9165S | Primary antibody for visualization of c-Jun protein |
| Rabbit myelin basic protein primary antibody | Abcam | ab40390 | Primary antibody for visualization of MBP |
| Rabbit p75NTR primary antibody | Cell Signaling Technology | 8238S | Primary antibody for visualization of p75NTR |
| Rhodamine phalloidin | Thermo Fisher Scientific | R415 | Visualization of cell cytoskeleton |
| RIPA buffer | Abcam | ab156034 | Cell lysis buffer |
| RT4-D6P2T Schwann cell line | ATCC | CRL-2768 | Cell line used in experiments |
| SYLGARD 184 PDMS base and curing agent | Sigma Aldrich | 761036 | Tunable polymer used to coat coverslips |
| Trypsin | Thermo Fisher Scientific | 15090-046 | Cell dissociation reagent |
| UV-Ozone cleaner | Novascan | Increase hydrophicility of PDMS | |
| Versene (1x) | Thermo Fisher Scientific | 15040066 | Cell dissociation reagent |
参考文献
- Taylor, C. A., Braza, D., Rice, J. B., Dillingham, T. The Incidence of Peripheral Nerve Injury in Extremity Trauma. American Journal of Physical Medicine & Rehabilitation. 87, 381-385 (2008).
- Noble, J., Munro, C. A., Prasad, V. S. S. V., Midha, R. Analysis of Upper and Lower Extremity Peripheral Nerve Injuries in a Population of Patients with Multiple Injuries. Journal of Trauma and Acute Care Surgery. 45, 116-122 (1998).
- Jessen, K. R., Mirsky, R. The repair Schwann cell and its function in regenerating nerves. Journal of Physiology. 594, 3521-3531 (2016).
- Arthur-Farraj, P. J., et al. c-Jun Reprograms Schwann Cells of Injured Nerves to Generate a Repair Cell Essential for Regeneration. Neuron. 75, 633-647 (2012).
- Jessen, K. R., Mirsky, R. The Success and Failure of the Schwann Cell Response to Nerve Injury. Frontiers in Cell Neurosciences. 13, 33(2019).
- Gomez-Sanchez, J. A., et al. After Nerve Injury, Lineage Tracing Shows That Myelin and Remak Schwann Cells Elongate Extensively and Branch to Form Repair Schwann Cells, Which Shorten Radically on Remyelination. Journal of Neuroscience. 37 (37), 9086-9099 (2017).
- Deumens, R., et al. Repairing injured peripheral nerves: Bridging the gap. Progress in Neurobiology. 92, 245-276 (2010).
- Höke, A., Gordon, T., Zochodne, D. W., Sulaiman, O. A. R. A decline in glial cell-line-derived neurotrophic factor expression is associated with impaired regeneration after long-term Schwann cell denervation. Experimental Neurology. 173, 77-85 (2002).
- Jones, S., Eisenberg, H. M., Jia, X. Advances and future applications of augmented peripheral nerve regeneration. International Journal of Molecular Sciences. 17, 1-17 (2016).
- Harris, G. M., et al. Nerve Guidance by a Decellularized Fibroblast Extracellular Matrix. Matrix Biology. 60-61, 176-189 (2017).
- Harris, G. M., Piroli, M. E., Jabbarzadeh, E. Deconstructing the Effects of Matrix Elasticity and Geometry in Mesenchymal Stem Cell Lineage Commitment. Advanced Function Mater. 24 (16), 2396-2403 (2014).
- Pryzhkova, M. V., Harris, G. M., Ma, S., Jabbarzadeh, E. Patterning pluripotent stem cells at a single cell level. Journal of Biomaterials and Tissue Engineering. 3 (4), 461-471 (2013).
- Engler, A. J., Sweeney, H. L., Discher, D. E., Schwarzbauer, J. E. Extracellular matrix elasticity directs stem cell differentiation. Journal of Musculoskeleton and Neuronal Interaction. 7 (4), 335(2007).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix Elasticity Directs Stem Cell Lineage Specification. Cell. 126, 677-689 (2006).
- Pickup, M. W., Mouw, J. K., Weaver, V. M. The extracellular matrix modulates the hallmarks of cancer. EMBO Reports. 15, 1243-1253 (2014).
- Chernousov, M. A., Carey, D. J. Schwann cell extracellular matrix molecules and their receptors. Histology and Histopathology. 15, 593-601 (2000).
- Shibata, S., et al. Selective Laminin-Directed Differentiation of Human Induced Pluripotent Stem Cells into Distinct Ocular Lineages. Cell Reports. 25 (6), 1668-1679 (2018).
- Mcbeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell Shape, Cytoskeletal tenstion and RhoA regulate stem cell lineage committment. Developmental Cell. 6, 483-495 (2004).
- Halder, G., Dupont, S., Piccolo, S. Transduction of mechanical and cytoskeletal cues by YAP and TAZ. Nature Reviews Molecular Cell Biology. 13, 591-600 (2012).
- Jessen, K. R., Mirsky, R. The repair Schwann cell and its function in regenerating nerves. Journal of Physiology. 594 (13), 3521-3531 (2016).
- Lopez-Fagundo, C., Bar-Kochba, E., Livi, L. L., Hoffman-Kim, D., Franck, C. Three-dimensional traction forces of Schwann cells on compliant substrates. Journal of The Royal Society Interface. 11, 20140247(2014).
- Gu, Y., et al. The influence of substrate stiffness on the behavior and functions of Schwann cells in culture. Biomaterials. 33, 6672-6681 (2012).
- Xu, Z. Y., Orkwis, J. A., DeVine, B. M., Harris, G. M. Extracellular matrix cues modulate Schwann cell morphology, proliferation, and protein expression. Journal of Tissue Engineering and Regenerative. , (2019).
- Urbanski, M. M., et al. Myelinating glia differentiation is regulated by extracellular matrix elasticity. Scientific Reports. 6, 1-12 (2016).
- Sun, Y., et al. Tunable stiffness of graphene oxide/polyacrylamide composite scaffolds regulates cytoskeleton assembly. Chemical Sciences. 9 (31), 6516-6522 (2018).
- Hwang, J. H., et al. Extracellular matrix stiffness regulates osteogenic differentiation through MAPK activation. PLoS One. 10, 1-16 (2015).
- Ryan, A. J., et al. A Physicochemically Optimized and Neuroconductive Biphasic Nerve Guidance Conduit for Peripheral Nerve Repair. Advanced Healthcare Materials. 6, 1-13 (2017).
- Du, J., et al. Prompt peripheral nerve regeneration induced by a hierarchically aligned fibrin nanofiber hydrogel. Acta Biomaterialia. 55, 296-309 (2017).
- Huang, L., et al. A compound scaffold with uniform longitudinally oriented guidance cues and a porous sheath promotes peripheral nerve regeneration in vivo. Acta Biomaterialia. 68, 223-236 (2018).
- Brower, K., White, A. K., Fordyce, P. M. Multi-step Variable Height Photolithography for Valved Multilayer Microfluidic Devices. Jouranl of Visualized Experiments. (119), e55276(2017).
- Gupta, R., et al. Shear stress alters the expression of myelin-associated glycoprotein (MAG) and myelin basic protein (MBP) in Schwann cells. Journal of Orthopaedic Research : Official Publication of the Orthopaedic Research Society. 23, 1232-1239 (2005).
- Harris, G. M., Shazly, T., Jabbarzadeh, E. Deciphering the combinatorial roles of geometric, mechanical, and adhesion cues in regulation of cell spreading. PLoS One. 8 (11), (2013).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Schreck, I., et al. C-Jun localizes to the nucleus independent of its phosphorylation by and interaction with JNK and vice versa promotes nuclear accumulation of JNK. Biochemical and Biophysical Research Communications. 407, 735-740 (2011).
- Shen, K., Qi, J., Kam, L. C. Microcontact printing of proteins for cell biology. Journal Visualized Experiments. (22), e1065(2008).
- Treter, J., et al. Washing-resistant surfactant coated surface is able to inhibit pathogenic bacteria adhesion. Applied Surface Science. 303, 147-154 (2014).
- Lutz, J. F. Polymerization of oligo(ethylene glycol) (meth)acrylates: Toward new generations of smart biocompatible materials. Journal of Polymer Science Part A: Polymer Chemistry. 46 (11), 3459-3470 (2008).
- Marcus, M., et al. Interactions of Neurons with Physical Environments. Advanced Healthcare Materials. 6, (2017).
- Pu, J. Golgi polarization in a strong electric field. Journal of Cell Science. 118, 1117-1128 (2005).
- Blaker, J. J., et al. Bioactive Silk-Based Nerve Guidance Conduits for Augmenting Peripheral Nerve Repair. Advanced Healthcare Materials. 7, 1800308(2018).
- Daly, W., Yao, L., Zeugolis, D., Windebank, A., Pandit, A. A biomaterials approach to peripheral nerve regeneration : bridging the peripheral nerve gap and enhancing functional recovery. Journal of the Royal Society of Interface. 9 (67), 202-221 (2012).
- Xia, H., et al. Directed neurite growth of rat dorsal root ganglion neurons and increased colocalization with Schwann cells on aligned poly(methyl methacrylate) electrospun nanofibers. Brain Research. 1565, 18-27 (2014).
- Wang, H. B., Mullins, M. E., Cregg, J. M., McCarthy, C. W., Gilbert, R. J. Varying the diameter of aligned electrospun fibers alters neurite outgrowth and Schwann cell migration. Acta Biomaterialia. 6, 2970-2978 (2010).
- Carvalho, C. R., Oliveira, J. M., Reis, R. L. Modern Trends for Peripheral Nerve Repair and Regeneration: Beyond the Hollow Nerve Guidance Conduit. Frontiers in Bioengineering and Biotechnology. 7, 337(2019).
- Yang, Y., Wang, K., Gu, X., Leong, K. W. Biophysical Regulation of Cell Behavior - Cross Talk between Substrate Stiffness and Nanotopography. Engineering. 3, 36-54 (2017).
- Tan, J. L., Liu, W., Nelson, C. M., Raghavan, S., Chen, C. S. Simple Approach to Micropattern Cells on Common Culture Substrates by Tuning Substrate Wettability. Tissue Engineering. 10, 865-872 (2004).
- Grove, M., et al. YAP/TAZ initiate and maintain schwann cell myelination. Elife. 6, 1-27 (2017).
- Poitelon, Y., et al. YAP and TAZ control peripheral myelination and the expression of laminin receptors in Schwann cells. Nature Neuroscience. 19, 879-887 (2016).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved