È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Preparazione di microambienti a matrice extracellulare tunable per valutare schwann Cell Phenotype Specification
In questo articolo
Riepilogo
Questa metodologia ha lo scopo di illustrare i meccanismi con cui i segnali di matrice extracellulare come la rigidità del substrato, la composizione delle proteine e la morfologia cellulare regolano il fenotipo delle cellule di Schwann (SC).
Abstract
Le lesioni del sistema nervoso periferico traumatico (PNS) attualmente non dispongono di trattamenti adeguati per recuperare il pieno recupero funzionale. Le cellule di Schwann (SC), come le principali cellule gliali del PNS, svolgono un ruolo vitale nel promuovere la rigenerazione del PNS dedifferenziando in un fenotipo cellulare rigenerativo a seguito di lesioni. Tuttavia, lo stato disdifferenziato delle SCs è difficile da mantenere durante il periodo di tempo necessario per la rigenerazione ed è influenzato dai cambiamenti nella matrice extracellulare circostante (ECM). Pertanto, è essenziale determinare la complessa interazione tra SC e ECM diverso per fornire indicazioni di potenziale rigenerativo delle SC. Per risolvere questo problema, è stata creata una strategia in cui diverse proteine ECM sono state assorbite su un substrato polidimethylsiloxane (PDMS) regolabile che ha fornito una piattaforma in cui la rigidità e la composizione delle proteine possono essere modulate. Le SC sono state seminate sui substrati regolabili e sono state misurate le funzioni cellulari critiche che rappresentano la dinamica del fenotipo SC. Per illustrare l'interazione tra l'espressione proteica SC e la morfologia cellulare, sono state utilizzate diverse densità di semità di SCs oltre ai singoli modelli cellulari stampati in microcontatto e caratterizzati da colorazione immunofluorescenza e macchie occidentali. I risultati hanno mostrato che le cellule con un'area di diffusione più piccola e una maggiore estensione dell'allungamento cellulare hanno promosso livelli più elevati di marcatori fenotipici rigenerativi SC. Questa metodologia non solo inizia a svelare la relazione significativa tra l'ECM e la funzione cellulare delle SC, ma fornisce anche linee guida per la futura ottimizzazione dei biomateriali nella riparazione dei nervi periferici.
Introduzione
Le lesioni del sistema nervoso periferico (PNS) rimangono una grande sfida clinica nel settore sanitario compromettendo la qualità della vita dei pazienti e creando un impatto significativo attraverso una moltitudine di fattori socioeconomici1,2. Le cellule di Schwann (SC), in quanto le principali cellule gliali del PNS, forniscono i necessari segnali molecolari e fisici per indurre la rigenerazione del PNS e aiutare nei recuperi funzionali in lesioni di breve variazione. Ciò è dovuto alla notevole capacità delle SCs di disdifferenziarsi in un fenotipo cellulare "riparare" da un mielinante o remak fenotipo3. La riparazione SC è un fenotipo cellulare distintivo in diversi modi. In seguito a lesioni, le SC aumentano il loro tasso di proliferazione rientrando nel ciclo cellulare e iniziano l'espressione di diversi fattori trascrizionali per facilitare la reinnervazione. Questi fattori, come c-Jun e p75 NTR, sono upregulated mentre i marcatori SC mielinanti, come la proteina di base della mielina (MBP), sono downregolati4,5. Inoltre, le SC cambiano morfologia per allungarsi e allinearsi l'una con l'altra per formare bande di Bànner in tutto il sito di infortunio6. Questo fornisce un meccanismo di guida fisica per gli assoni per estendere al bersaglio distale corretto7. Tuttavia, nonostante la capacità che le SC possiedono per promuovere la rigenerazione dei nervi in lesioni di breve gap, l'esito del recupero funzionale rimane scarso in gravi lesioni. Ciò è dovuto in parte alla perdita di segnali guida ecm (Extracellular Matrix), così come all'incapacità delle SCs di mantenere il fenotipo rigenerativo per lunghi periodi di tempo8.
La rigenerazione del nervo e il processo di recupero sono intimamente legati allo stato della lamina basale a seguito di lesioni. La lamina basale è uno strato di ECM intorno al nervo che facilita la guida e fornisce spunti eCM-bound per assoni e SC nei casi in cui rimane intatto dopo la lesione9. Lo stato dell'ECM e la sua capacità di fornire segnali legati a matrice alle celle è di vitale importanza ed è stato precedentemente esplorato in una varietà di contesti diversi10,11,12,13,14. Ad esempio, è stato dimostrato che la rigidità dell'ECM può guidare le funzioni cellulari come la proliferazione e la differenziazione11,15,16. La composizione dell'ECM può anche portare a una risposta cellulare distinta e regolare i comportamenti cellulari come la migrazione e la differenziazione attraverso le vie di segnalazione intracellulare17,18. Inoltre, la morfologia cellulare, compresa l'area di diffusione e l'allungamento cellulare, svolgono un ruolo importante nella regolazione della funzione e possono essere regolate da segnali legati all'ECM19,20. Molti studi precedenti si sono concentrati sulla differenziazione delle cellule staminali in linee definite, ma le SC possiedono una capacità simile di alterare il fenotipo da un SC omeostatico e adulto all'interno di un nervo sano, a una SC di riparazione in grado di secernere proteine e fattori di crescita rimodellando l'ECM a seguito di lesioni nervose5,21. Pertanto, è particolarmente importante identificare i meccanismi alla base della relazione tra la capacità rigenerativa SC innata e i segnali legati all'ECM per l'intuizione per sfruttare in ultima analisi questa capacità di rigenerazione dei nervi.
Per risolvere questo problema, abbiamo sviluppato una metodologia dettagliata per produrre un substrato di coltura cellulare in cui rigidità meccanica e tipo di ligando possono essere facilmente sintonizzati in gamme fisiologicamente rilevanti. Il siloxane polidimetile (PDMS) è stato scelto come substrato a causa della sua meccanica altamente regolabile rispetto al gel di poliacritilminismo, dove il modulus massimo di Young è di circa 12 kPa a contrasto con PDMS a circa 1000 kPa22,23,24. Questo è utile per il lavoro a portata di mano, come recenti studi hanno dimostrato il modulo del giovane di un nervo sciatico coniglio può superare 50 kPa durante lo sviluppo, suggerendo così che la gamma di rigidità dei nervi all'interno del PNS è più ampia di quanto precedentemente esaminato. Diverse proteine sono in grado di adsorgere su substrati PDMS per analizzare la regolazione combinatoria della meccanica e dei ligandi sul comportamento SC. Ciò consente di sforare molteplici segnali microambientali presenti nel processo di rigenerazione PNS e il confronto di un elevato grado di sintonizzabilità con il lavoro concentrandosi esclusivamente sulla rigidità del substrato25. Inoltre, questi substrati di coltura cellulare ingegnerizzati sono compatibili con una moltitudine di metodi di analisi quantitativa come l'immunohistochimica, la macchia occidentale e la reazione a catena quantitativa della polimerasi (q-PCR).
Questa piattaforma di coltura cellulare ingegnerizzata è altamente adatta per l'analisi dei percorsi meccanicistici a causa dell'elevato livello di sintonfiabilità individuale di ogni segnale legato a ECM. Inoltre, i metodi popolari per il micropatterning cellulare, compresa la stampa di microcontatto, possono essere ottenuti sui substrati per consentire l'adesione cellulare controllata per analizzare la forma cellulare in relazione ad altri segnali eCM legati24. Questo è fondamentale perché i substrati modellati a linee, che promuovono l'allungamento nelle popolazioni cellulari, forniscono uno strumento per imitare e studiare le SC allungate e rigenerative all'interno delle bande di Bàngner durante la rigenerazione dei nervi. Inoltre, la morfologia cellulare è un potente regolatore di molteplici funzioni cellulari e può potenzialmente introdurre risultati sperimentali confusi se non controllati26,27. Viene ora prestata particolare attenzione ai meccanismi che regolano il fenotipo rigenerativo SC come regolato da segnali ECM28,29,30. Questo è essenziale per fornire informazioni sulla progettazione di biomateriali che possono essere applicati come condotti di guida nervosa per aiutare nella rigenerazione dei nervi PNS. Questi protocolli dettagliati possono infine essere applicati come un potenziale strumento per decifrare i meccanismi di SC e altri tipi di cellule funzionano come regolato da segnali legati ECM.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparazione e caratterizzazione del substrato di coltura cellulare tunable
- Preparazione del substrato
- Mescolare vigorosamente l'elastomero di base PDMS e gli agenti di polimerazione utilizzando una punta di pipetta a un rapporto compreso tra 10:1 e 60:1 fino a quando le bolle sono disperse in modo omogeneo all'interno della miscela. Rimuovere le bolle utilizzando la disidratazione del vuoto fino a quando le bolle non vengono dissipate.
NOTA: durante la polimerizzazione PDMS, la polimerizzazione dell'agente di polimerizzazione con l'elastomero di base per fornire le proprietà meccaniche desiderate per il polimero finale. I rapporti di collegamento incrociato possono essere regolati per alterare la rigidità del PDMS. - Posizionare una goccia (0,2 mL) di miscela PDMS indipendente su un coperchio quadrato o circolare (ad esempio, 22 mm x 22 mm) e ruotare il coperchio su un rivestimento di spin a 2500 rpm per 30 s.
- Incubare il coperchio in un forno a 60 gradi centigradi per 1-2 h o temperatura ambiente durante la notte per la PDMS per solidificare.
- Trattare il coverslip utilizzando UV-Ozone cleaner per 7 min (lunghezza d'onda UV: 185 nm e 254 nm) per aumentare l'idrofilia superficiale. Mettetelo in una piastra sterilizzata a 6 pozze.
- Prima dell'uso per la coltura cellulare, incubare i substrati nel 70% di etanolo per almeno 30 min.
AVVISO: il detergente UV-ozono può generare ozono dannoso per l'uomo. Lavorare in una cappa di fumi chimici o con qualche forma di ventilazione. - Immergere i coprilabbra nella soluzione proteica (10 g/mL di collagene I, fibronectina o laminina) per 60 min in un'incubatrice sterile a 37 .
NOTA: dopo il trattamento UV-Ozone, la superficie del PDMS potrebbe essere ancora idrofobica. Ruotare la piastra del pozzo per garantire che ogni coverslip sia coperto con la soluzione proteica. - Aspirare la soluzione proteica e lavare il coperchio con fosfato tampone salina (PBS) 3x.
- Sospendere nuovamente la linea cellulare RT4-D6P2T Schwann (SCs) dal passaggio della parabola utilizzando la soluzione EDTA disponibile in commercio (1x) con 2,5% di metapina e contare le celle con emocitometro. Seme SC sulla superficie PDMS regolabile alla densità di cella desiderata. Densità di seeding SC possono variare per ogni applicazione diversa.
- Mantenere le cellule nei parametri di coltura cellulare desiderati (90% di umidità, 5% CO2, 37 gradi centigradi, ecc.) per la durata dell'esperimento. Utilizzare Il Dulbecco's Modified Eagle Medium (DMEM) integrato con 10% di siero bovino fetale (FBS) e 1% penicillina-streptomycin come mezzo di coltura cellulare.
- Mescolare vigorosamente l'elastomero di base PDMS e gli agenti di polimerazione utilizzando una punta di pipetta a un rapporto compreso tra 10:1 e 60:1 fino a quando le bolle sono disperse in modo omogeneo all'interno della miscela. Rimuovere le bolle utilizzando la disidratazione del vuoto fino a quando le bolle non vengono dissipate.
- Preparazione del substrato micromodellato
- Disegnare la geometria desiderata e le aree adesive delle celle (900 m2, 1.600 m2 e 2.500 m2)utilizzando un software CAD (Computer-Aided Design). Creare una maschera foto cromata basata su tali modelli di un fornitore commerciale.
- In una stanza pulita o in un ambiente privo di polvere, utilizzare tecniche di fotolitografia standard per fabbricare wafer di silicio (i protocolli sono dettagliati altrove31). I parametri critici per questa particolare applicazione sono i seguenti: Photoresist: SU-8 2010; Profilo di spin per disperdere il fotoresist: 500 rpm per 10 s con un'accelerazione di 100 rpm/s, quindi 3500 rpm per 30 s con un'accelerazione di 300 rpm/s; Energia di esposizione della luce UV: 130 mJ/cm2.
NOTA: L'altezza dei modelli sui wafer di silicio è di circa 10 m seguendo questi parametri. Potenziali crepe intorno al bordo esterno dei modelli rettangolari o triangolari possono essere viste utilizzando un microscopio luminoso dopo il passaggio 1.2.2. Cuocere il wafer di silicio a 190 gradi centigradi per 30 min aiuta ad eliminare le fessure. - Posizionare il wafer di silicio modellato all'interno di una parabola Petri di 150 mm di diametro circolare di 150 mm x 15 mm e versare il PDMS degassato (rapporto di miscelazione 10:1) come preparato nel passo 1.1.1 sul wafer di silicio.
NOTA: Assicurarsi che lo spessore del PDMS sia di almeno 5 mm per la facilità di gestione durante le fasi di stampa dei microcontatto. - Solidifica il PDMS su wafer di silicio in forno a 60 gradi durante la notte. Lasciare raffreddare PDMS a temperatura ambiente. Stampe tagliate con precisione di 30 mm x 30 mm quadrati contenenti i modelli corretti dal wafer di silicio utilizzando un bisturi chirurgico. Non danneggiare il wafer di silicio.
NOTA: i wafer di silicio possono essere riutilizzati più volte a questo punto per produrre più francobolli dopo la pulizia con isopropanolo. - Sterilizzare i francobolli PDMS e le copricapi regolabili (preparate al punto 1.1.1 a 1.1.3) immergendoli nel 70% di etanolo per 30 min.
- Per confermare l'efficacia del micropattern da parte dei francobolli PDMS dopo la stampa di microcontatto, asciugare la superficie dei francobolli PDMS utilizzando un flusso d'aria filtrato e una soluzione pipetta 50 sg/mL BSA (Texas Red coniugato) per coprire l'intero lato modellato del timbro PDMS.
- Incubare i francobolli PDMS con soluzione BSA per 1 h a temperatura ambiente per consentire l'assorbimento delle proteine.
- Asciugare la superficie dei coprilabbra regolabili utilizzando un flusso d'aria filtrato, aumentare l'idrofilia di superficie come descritto nel passaggio 1.1.5.
- Asciugare all'aria i francobolli PDMS per rimuovere la soluzione BSA rimanente.
NOTA: fare attenzione che la soluzione BSA sia completamente rimossa dal timbro perché qualsiasi soluzione rimanente farà scivolare i francobolli sul coperchio durante la stampa di microcontatto. - Portare il lato modellato del timbro a contatto conforme con la coverslip regolabile per l'adsorbimento BSA sulla superficie della coverslip. Premere delicatamente il timbro contro la coverslip per 5 min.
NOTA: Non applicare una forza eccessiva sul timbro poiché si piega e causa un contatto non specifico tra timbro e coverslip. L'appropriata quantità di forza applicata sul timbro è essenziale per una stampa di microcontatto di successo. - Esaminare il micromodello utilizzando il microscopio a fluorescenza con un filtro FITC (Fluorescein isothiocyanate).
- Per stampare aree adesive cellulari anziché modelli fluorescenti, sostituire la laminina con la proteina BSA e ripetere i passaggi da 1,2,5 a 1,2,10.
- Rimuovere i francobolli dai copriletti, trasferire i coperchi in una piastra sterilizzata a 6 pozze. Aggiungere 2 mL di 0,2% w/v soluzione Pluronic F-127 in ogni pozzo per coprire la superficie del coperchio e incubare per 1 h a temperatura ambiente.
NOTA: Pluronic F-127 può essere adsorbito alla superficie PDMS aumentando l'idrofobicità della superficie PDMS per bloccare le cellule dall'adesione. - Aspirate Pluronic F-127 soluzione e lavare 5x con PBS e 1x con il mezzo di coltura cellulare prima di seminare le cellule. Una densità di semina tipica per le SCs è di 1.000 celle/cm2.
- 45 min dopo il seeding cellulare, rimuovere il mezzo di coltura cellulare e lavare i coprilabbra con PBS 2x per evitare che più SC di aderire allo stesso modello. Mantenere le cellule nell'ambiente di coltura cellulare desiderato per 48 h prima della quantificazione.
- Per creare substrati di impostazioni cultura delle celle con motivi di linea per esaminare le celle allineate, seguire i passaggi da 1.2.1 a 1.2.4 per creare timbri per la stampa di microcontatti.
NOTA: Le dimensioni della scanalatura/cresta dei motivi foderati sul timbro sono pari a 50 m x 50 m per le celle delineate. Le dimensioni totali del timbro sono 10 mm x 10 mm. - Tagliare i timbri in quote contenenti solo i motivi di linea desiderati.
NOTA: Quando si creano timbri in CAD, l'area senza motivi del timbro intorno ai modelli di linea corrisponderà all'area adesiva cellulare dopo la stampa dei microcontatti. Pertanto, è necessario eliminare le aree non modellate quando si taglia il timbro per garantire che ogni semiSC sulla superficie segua i modelli. - Seguire i passaggi da 1.1.1 a 1.1.3 per preparare il rivestimento superficiale del PDMS regolatore con due piatti Petri.
NOTA: Questo sarà PDMS che copre la superficie del piatto Petri stessa e non su un coperchio. - Seguire i passaggi da 1.2.5 a 1.2.10 per eseguire la stampa di microcontatto per stampare aree adesive per celle con motivi di linea su uno dei piatti Petri rivestiti PDMS.
NOTA: la superficie di un piatto Petri da 60 mm x 15 mm può contenere aree con motivi di linea da 6 francobolli PDMS. - Togliere i francobolli e riempire la piastra Petri con 4 mL di 0,2% w/v soluzione Pluronic F-127 e incubare per 1 h.
- Dopo la stampa microcontatto, sciacquare ogni lato dei francobolli PDMS con 70% di etanolo 3x e asciugare con aria. Ruotare i timbri PDMS e seguire i passaggi da 1,2,5 a 1,2,10 per stampare un'area adesiva cellulare senza motivi utilizzando il lato non modellato del timbro sul secondo piatto Petri. Ripetere il passaggio 1.2.13.
- Aspirate soluzione F-127 da piatti, lavare 3x con PBS seguito da 1x lavaggio utilizzando il mezzo di coltura a cellule fresche. SC di semi su piatti.
NOTA: la densità di semina per un piatto con motivi di linea è 5.000 celle/cm2 e per un piatto non modellato è 10.000 celle/cm2. - Mantenere le SC alle condizioni desiderate per 48 h e seguire i protocolli per preparare le lisate SC32.
NOTA: La densità di semina delle cellule per i piatti non modellati è 2 volte superiore a quella per i piatti con motivi di linea, poiché il piatto con motivi di linea ha solo la metà dell'area adesiva della cella del piatto non modellato.- Per preparare i lismi, trasferire un buffer DI analisi radioimmunoprecipitazione (RIPA) adeguato in un tubo di centrifuga conica da 10 mL. Miscela di proteasi diluita e fosforasi (100x) con un rapporto di 1:100 nel buffer RIPA, si mescolano bene con il pipettaggio.
- Lavare le cellule con PBS ghiacciato (1x) per 2 min, aggiungere 80 -L della soluzione preparata dal punto 1.2.23.1 su ogni area adesiva cellulare (l'area che ha contattato con francobolli PDMS e proteine adsorbiti) all'interno di piatti petri. Incubare le cellule con la soluzione su un blocco di ghiaccio per 15 min.
NOTA: La soluzione rimarrà sull'area adesiva cellulare solo a causa dell'idrofobicità dell'adsorbizione Pluronic F-127 altrove. Questa funzione consente un'estrazione di proteine di successo e sufficiente per gli SC con motivi di linea. - SC scrape con un raschietto cellulare per 5 min. Raccogliere il lisato in un tubo di microcentrifuga da 1,5 ml etichettato.
- Microcentrifuga lisata a 12.000 x g per 15 min a 4 gradi centigradi. Raccogliere supernatali con una pipetta da 1.000 lun e trasferirlo in un tubo di microcentrifuga pulito. Conservare il lisato cellulare a -20 gradi centigradi.
- Caratterizzazione del substrato
NOTA: Per caratterizzare la meccanica del polimero sul coperchio, sono generalmente utilizzati più metodi tra cui test di compressione di massa11,33 o test di microscopia a forza atomica34. Questo protocollo descrive i test di compressione di massa.- Versare il precursore PDMS del rapporto di miscelazione desiderato (passaggio 1.1) in una parabola Petri da 30 mm, in modo che lo spessore dello strato PDMS all'interno della parabola Petri sia di almeno 20 mm.
- Togliere la piastra Petri con PDMS solidificata a partire dal forno a 60 gradi centigradi dopo 1 h e lasciar raffreddare a temperatura ambiente. Tagliare il polimero in quadrati da 10 mm x 10 mm. Misurare lo spessore del PDMS utilizzando le pinze.
- Posizionare PDMS sullo stage della macchina di misura della forza di compressione. Sensore di forza di compressione plug-in (modello: 112C) alla porta del sensore e fissaggio del sensore sull'asse della macchina di prova.
- Regolare l'altezza del sensore a circa 0,5 cm sopra il timbro PDMS utilizzando il controllo "jog" sul pannello anteriore dello strumento.
- Aprire il software associato utilizzando la finestra "Test Setup", selezionare "Servo profile" e aprire la finestra "Segmento". Nella finestra "Segmento", inserisci i "Control Rate" e "End Amount" desiderati per il test.
NOTA: La velocità di controllo determina la velocità di discesa del sensore verso il PDMS. La quantità finale determina la distanza totale percorsa dal sensore. - Utilizzare il pulsante """ situato sul pannello di controllo del software per reimpostare tutte le misurazioni a questo punto.
- Spostare il sensore verso il basso per contattare leggermente PDMS fino a quando non vengono caricati 1-2 newton (N). Il carico e la distanza percorsa dal sensore verranno visualizzati nel software.
- Dopo aver utilizzato il pulsante "s", eseguire la misurazione utilizzando "Play", e salvare la forza di registrazione del file e la distanza.
- Ripetere i passaggi da 1.3.3 a 1.3.8 per ogni condizione sperimentale di PDMS.
- Aprire il file e utilizzare la seguente formula per calcolare il modulo di Young (E) di PDMS per ogni rapporto. (F : forza di compressione, A e area del timbro PDMS,L - distanza di percorrenza del sensore e L0 - spessore originale del timbro PDMS).
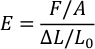
2. Quantificazione delle proprietà cellulari su substrati regolabili
- Saggio di proliferazione
- Seme SCs su substrati preparati dal passo 1.1.9 ad una densità di 5.000 cellule / cm2 in una piastra di 6 pozze. Consentire alle CC di incubare per 48 h in condizioni di coltura cellulare standard (37 e 5% di CO2).
- Diluire la soluzione di 12 ll di 10 mM Bromodeoxyuridine (BrdU) in 12 mL di 37 mL di 37 gradi centigradi, mescolare bene con la pipetta per realizzare una soluzione di etichettatura di 10 M BrdU.
- Rimuovere il mezzo di coltura cellulare e lavare i SCs 2x con PBS.
- Aggiungere 2 mL di soluzione di etichettatura BrdU in ogni pozzo e incubare sC per 2 h.
NOTA: il tempo di incubazione della soluzione di etichettatura BrdU dipende dal tasso specifico di proliferazione cellulare. La linea RT4-D6P2T SC ha un alto tasso di proliferazione, quindi sono stati utilizzati 2 h di tempo di incubazione. - Rimuovere la soluzione di etichettatura BrdU e lavare i SC 3x con PBS. Aggiungere 1 mL di 3,7% di formaldeide in PBS ad ogni pozzo e incubare a temperatura ambiente per 15 min per la fissazione cellulare.
DATIRISSE: La formaldeide è un cancerogeno umano; pertanto, eseguire tutto il lavoro all'interno di una cappa di fumi chimici con una protezione adeguata.
NOTA: Durante il lavaggio con PBS, non c'è nessun mezzo di coltura cellulare nel pozzo, e quindi la superficie PDMS può essere idrofobica. Prendere precauzioni per non asciugare completamente la superficie del substrato per evitare danni alle cellule. - Soluzione di formaldeide aspirata e lavare 3x con PBS (3 min ciascuno). Rimuovere PBS e aggiungere 1 mL di 0,2% Triton X-100 in PBS a ciascun pozzo per permeabilizzare la membrana cellulare. Incubare i CS con soluzione Triton X-100 per 20 min a temperatura ambiente.
- Rimuovere la soluzione Triton X-100 e lavare gli SCs 3x con PBS (3 min ciascuno).
- Aggiungere 1 mL di 1 N HCl in ogni pozzo e incubare sul ghiaccio per 10 min.
- Mescolare 182 mL di 0,2 mM Na2HPO4 e 18 mL di 0,1 mM di acido citrico per produrre un buffer di acido fosfato/acido citrico per il recupero dell'antigene. Rimuovere 2 N HCl e aggiungere 1 mL fosfato/tampone acido citrico in ogni pozzo e incubare a temperatura ambiente per 10 min.
- Lavare i SC 3x con 0,2% Triton X-100 in PBS. Aggiungere 2 mL di 3% di albumina di siero bovino (BSA) in PBS in ogni pozzo e incubare per 30 min a temperatura ambiente per orologiare il legame non specifico dell'anticorpo.
- Diluito anticorpo primario BrdU coniugato con Alexa Fluor 488 in 3% soluzione BSA ad un rapporto di 1:300 per la soluzione di colorazione BrdU. Incubare sCs con soluzione di colorazione durante la notte a temperatura ambiente, mentre la piastra è coperta in foglio di alluminio.
- Per quantificare la proliferazione, gli SC di immagine che utilizzano il canale FITC e DAPI di un microscopio fluorescente per rilevare rispettivamente BrdU e nuclei. Salvare le immagini come file "nd.2".
- Aprire i file "nd.2" per ogni immagine scattata in posizioni spaziali identiche.
- Aprire il software di analisi delle immagini. Fare clic con il pulsante destro del mouse sullo sfondo per aprire la finestra "Risultati misurazione automatizzata" e "Misurazione automatica" nella sezione di "Controllo analisi".
- Nel menu "Conteggio e tassonomia", selezionare "Conteggio". Sull'immagine FITC, fare clic su ogni nucleo che mostra la fluorescenza verde (BrdU positivo) e fare clic destro sull'immagine.
NOTA: il numero di celle BrdU positive viene visualizzato nella finestra di "Automazioni e misure". - Per le immagini DAPI, ripetere il passaggio 2.1.14 per contare il numero totale di nuclei. Calcolare la percentuale di celle positive BrdU per questa immagine.
- Ripetere i passaggi da 2.1.13 a 2.1.16 per altre immagini per scopi statistici e calcolare la percentuale media di celle positive BrdU per ogni condizione del substrato.
- Quantificazione dell'espressione c-Jun attraverso l'analisi immunofluorescente delle immagini
- Le cellule preparate all'interno di piastre a 6 pozzi dai gradi 1.1.9 e 1.2.23 sono fisse e permeabate con le procedure descritte in precedenza (passaggio 2.1.5-2.1.7).
NOTA: per eseguire confronti accurati dell'intensità fluorescente tra celle con diverse condizioni ECM, applicare le impostazioni della fotocamera in modo identico a tutti i campioni con tutti i campioni con gli stessi parametri. - Salvare le immagini come file ".nd2".
- Aprire il software di analisi delle immagini. Fare clic con il pulsante destro del mouse sullo sfondo per aprire la finestra "Risultati di misurazione automatizzata" e "Misurazione automatizzata" nella sezione di "Controllo analisi".
- In "Risultati misurazione automatizzata", selezionare "Dati oggetto". Attivare il pulsante "Mantieni misurazione aggiornamento".
- Nel pannello superiore del software, selezionare "Misura" seguito da "Caratteristiche oggetto". Aggiungete "Intensità media" alla sezione di "Selezionato per la misurazione".
- Aprire e unire due file di immagine ".nd2" che contengono immagini di c-Jun e nuclei.
- Nel pannello superiore, selezionare "ROI" e selezionare "Disegna ROI rettangolare". Disegnare un'area rettangolare contenente l'area nucleare di una singola cella.
NOTA: l'espressione c-jun è concentrata all'interno dei nuclei35. - Nel pannello superiore del software, selezionare "Binario" e "Definisci soglia", una nuova finestra apparirà per definire con precisione l'area fluorescente c-Jun.
- Nella nuova finestra, fare clic su "Full Image/Use ROI" per passare dal modello di immagine completo al modello ROI. Utilizzare "Intensità" per regolare la tabella di ricerca situata sul lato sinistro della finestra per la regolazione delle dimensioni/forma dell'area evidenziata all'interno del ROI rettangolare.
NOTA: fare attenzione a garantire che la dimensione/forma dell'area evidenziata sia identica al nucleo. - Fare clic sul pulsante "OK", per ottenere l'intensità media FITC nella finestra di "Risultati misurazione automatizzata" e quindi fare clic su "Dati negozio".
- Nella finestra di "Misurazione automatizzata", "Elimina oggetto" per rimuovere l'area evidenziata in rosso. Sul pannello laterale sinistro, utilizzare "Strumento di puntamento" per selezionare il ROI rettangolare ed eliminare.
- Ripetere i passaggi da 2.2.6 a 2.2.11 per misurare l'intensità fitC per ogni cella aggiuntiva.
- Nell'area finestra di "Risultati misurazione automatizzata", selezionare "Memorizzato" e tutti i dati memorizzati saranno presentati. Utilizzare la funzione "Esporta" e selezionare "Dati in Excel" per salvare il foglio di calcolo esportato ed eseguire calcoli aggiuntivi.
- Le cellule preparate all'interno di piastre a 6 pozzi dai gradi 1.1.9 e 1.2.23 sono fisse e permeabate con le procedure descritte in precedenza (passaggio 2.1.5-2.1.7).
- Quantificare l'allungamento nucleare
- Fissare e permeabilizzare i PC preparati dal passaggio 1.2.22 seguendo i passaggi 2.1.5-2.1.7. Eseguire la colorazione nucleare utilizzando il supporto di montaggio con DAPI.
- Utilizzando il canale DAPI e un obiettivo 40x, acquisire le immagini del campione e salvare come file ".nd2".
- Seguite i passaggi 2.2.3 e 2.2.4 per aprire la finestra "Risultati misurazione automatizzata" e "Misurazione automatica" nel software di analisi delle immagini.
- In "Risultati di misurazione automatizzata", utilizzare la funzione "Opzione" seguita da " Selezionafunzione oggetto". Nella colonna "Feature", selezionare "Elongation" e aggiungere alla colonna "Selected for Measurements". Utilizzare "Keep Updating Measurement" per attivare questa funzione.
- Aprire il file di immagine "nd.2" che contiene le immagini nucleari. Nella finestra "Misurazione automatizzata", selezionare la funzione "Rilevamento automatico" e selezionare un nucleo. Fare clic con il pulsante destro del mouse sull'immagine e le proporzioni nucleari misurate verranno mostrate in "Risultati misurazione automatizzata".
- Ripetere il passaggio 2.3.5 per quantificare le proporzioni nucleari per gli altri nuclei all'interno dell'immagine. Selezionare "Dati del negozio" nella finestra di " Risultatimisurazione automatizzata".
- Ripetere da 2.3.5 a 2.3.6 per altre immagini. Esportare i dati in un file di foglio di calcolo come precedentemente fatto in 2.2.13 per l'analisi.
- La macchia occidentale quantifica l'espressione proteica
- Seguire i protocolli standard per l'analisi delle macchie occidentali descritte altrove32. Le diluizioni degli anticorpi utilizzati nello studio sono mostrate di seguito: Coniglio anti-jun 1:2,000; Topo anti-atto di z-actin 1:1,000; Coniglio anti p75NTR 1:1.000; Proteina di base anti mieliina 1:1,000; Anti-topo/coniglio IgG, anticorpo HRP-linked 1:10,000.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per analizzare e quantificare l'interazione tra rigidità del substrato e composizione proteica sul fenotipo SC, è stato sviluppato un substrato di coltura cellulare PDMS regolabile (Figura 1A). Test di compressione del polimero in base diversa: sono stati utilizzati i rapporti degli agenti di polimerazione per quantificare il modulo del Giovane (E) del substrato (Figura 1B). L'intervallo risultante di valori del modulo rappresenta condizioni di substrato fisio...
Access restricted. Please log in or start a trial to view this content.
Discussione
Le SC possono promuovere la rigenerazione dei nervi a causa della loro trasformazione fenotipica e del potenziale rigenerativo in seguito a lesioni nervose. Tuttavia, il modo in cui i segnali ECM regolano questa capacità rigenerativa rimane per lo più poco chiaro, ostacolando potenzialmente non solo lo sviluppo di biomateriali che mirano a promuovere la rigenerazione dei nervi, ma anche la comprensione dei meccanismi coinvolti nella rigenerazione dei nervi. Per iniziare a esaminare questa interazione, sono stati creati...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno segnalato un potenziale conflitto di interessi.
Riconoscimenti
Gli autori riconoscono con gratitudine il sostegno dei finanziamenti dell'Università di Cincinnati. Gli autori ringraziano anche Ron Flenniken del laboratorio di caratterizzazione avanzata dei materiali dell'Università di Cincinnati per il supporto.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Albumin from Bovine Serum (BSA), Texas Red conjugate | Thermo Fisher Scientific | A23017 | BSA staining to show micropatterns |
| Anti-mouse IgG, HRP-linked Antibody | Cell Signaling Technology | 7076S | Antibody used for western blot analysis |
| Anti-rabbit IgG, HRP-linked Antibody | Cell Signaling Technology | 7074S | Antibody used for western blot analysis |
| BrdU | Thermo Fisher Scientific | B23151 | Reagent used to measure cell proliferation |
| BrdU primary antibody conjugated with Alexa Fluor 488 | Thermo Fisher Scientific | B35130 | Used to visualize BrdU in cell proliferation assays |
| Collagen I | Thermo Fisher Scientific | A10483-01 | Protein used to coat coverslips |
| Compression force test machine | TestResources | Instrument to quantify mechanical properties of polymers | |
| Dulbecco's Modified Eagle Medium | Thermo Fisher Scientific | 11965092 | Cell culture medium |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | Cell culture medium supplemental |
| Fibronectin | Thermo Fisher Scientific | 33010-018 | Protein used to coat coverslips |
| Fluorescence microscope | Nikon | Eclipse Ti2 | Fluorescence microscope |
| Halt Protease and Phosphatase Inhibitor Cocktail (100X) | Thermo Fisher Scientific | 78440 | Protease and Phosphatase Inhibitor |
| Laminin | Thermo Fisher Scientific | 23017015 | Protein used to coat coverslips |
| Mounting medium with DAPI | Thermo Fisher Scientific | P36971 | Coverslip mountant and nuclei staining |
| Mouse c-Jun primary antibody | Thermo Fisher Scientific | 711202 | Primary antibody to visualize c-Jun protein |
| Mouse β-Actin primary antibody | Cell Signaling Technology | 3700S | Loading control for western blot experiments |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | Cell culture medium supplemental |
| Photoresist SU 2010 | KAYAKU | SU8-2010 | Photoresist |
| Pluronic F-127 | Sigma Aldrich | P-2443 | Block non-specific protein binding |
| Rabbit c-Jun primary antibody | Cell Signaling Technology | 9165S | Primary antibody for visualization of c-Jun protein |
| Rabbit myelin basic protein primary antibody | Abcam | ab40390 | Primary antibody for visualization of MBP |
| Rabbit p75NTR primary antibody | Cell Signaling Technology | 8238S | Primary antibody for visualization of p75NTR |
| Rhodamine phalloidin | Thermo Fisher Scientific | R415 | Visualization of cell cytoskeleton |
| RIPA buffer | Abcam | ab156034 | Cell lysis buffer |
| RT4-D6P2T Schwann cell line | ATCC | CRL-2768 | Cell line used in experiments |
| SYLGARD 184 PDMS base and curing agent | Sigma Aldrich | 761036 | Tunable polymer used to coat coverslips |
| Trypsin | Thermo Fisher Scientific | 15090-046 | Cell dissociation reagent |
| UV-Ozone cleaner | Novascan | Increase hydrophicility of PDMS | |
| Versene (1x) | Thermo Fisher Scientific | 15040066 | Cell dissociation reagent |
Riferimenti
- Taylor, C. A., Braza, D., Rice, J. B., Dillingham, T. The Incidence of Peripheral Nerve Injury in Extremity Trauma. American Journal of Physical Medicine & Rehabilitation. 87, 381-385 (2008).
- Noble, J., Munro, C. A., Prasad, V. S. S. V., Midha, R. Analysis of Upper and Lower Extremity Peripheral Nerve Injuries in a Population of Patients with Multiple Injuries. Journal of Trauma and Acute Care Surgery. 45, 116-122 (1998).
- Jessen, K. R., Mirsky, R. The repair Schwann cell and its function in regenerating nerves. Journal of Physiology. 594, 3521-3531 (2016).
- Arthur-Farraj, P. J., et al. c-Jun Reprograms Schwann Cells of Injured Nerves to Generate a Repair Cell Essential for Regeneration. Neuron. 75, 633-647 (2012).
- Jessen, K. R., Mirsky, R. The Success and Failure of the Schwann Cell Response to Nerve Injury. Frontiers in Cell Neurosciences. 13, 33(2019).
- Gomez-Sanchez, J. A., et al. After Nerve Injury, Lineage Tracing Shows That Myelin and Remak Schwann Cells Elongate Extensively and Branch to Form Repair Schwann Cells, Which Shorten Radically on Remyelination. Journal of Neuroscience. 37 (37), 9086-9099 (2017).
- Deumens, R., et al. Repairing injured peripheral nerves: Bridging the gap. Progress in Neurobiology. 92, 245-276 (2010).
- Höke, A., Gordon, T., Zochodne, D. W., Sulaiman, O. A. R. A decline in glial cell-line-derived neurotrophic factor expression is associated with impaired regeneration after long-term Schwann cell denervation. Experimental Neurology. 173, 77-85 (2002).
- Jones, S., Eisenberg, H. M., Jia, X. Advances and future applications of augmented peripheral nerve regeneration. International Journal of Molecular Sciences. 17, 1-17 (2016).
- Harris, G. M., et al. Nerve Guidance by a Decellularized Fibroblast Extracellular Matrix. Matrix Biology. 60-61, 176-189 (2017).
- Harris, G. M., Piroli, M. E., Jabbarzadeh, E. Deconstructing the Effects of Matrix Elasticity and Geometry in Mesenchymal Stem Cell Lineage Commitment. Advanced Function Mater. 24 (16), 2396-2403 (2014).
- Pryzhkova, M. V., Harris, G. M., Ma, S., Jabbarzadeh, E. Patterning pluripotent stem cells at a single cell level. Journal of Biomaterials and Tissue Engineering. 3 (4), 461-471 (2013).
- Engler, A. J., Sweeney, H. L., Discher, D. E., Schwarzbauer, J. E. Extracellular matrix elasticity directs stem cell differentiation. Journal of Musculoskeleton and Neuronal Interaction. 7 (4), 335(2007).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix Elasticity Directs Stem Cell Lineage Specification. Cell. 126, 677-689 (2006).
- Pickup, M. W., Mouw, J. K., Weaver, V. M. The extracellular matrix modulates the hallmarks of cancer. EMBO Reports. 15, 1243-1253 (2014).
- Chernousov, M. A., Carey, D. J. Schwann cell extracellular matrix molecules and their receptors. Histology and Histopathology. 15, 593-601 (2000).
- Shibata, S., et al. Selective Laminin-Directed Differentiation of Human Induced Pluripotent Stem Cells into Distinct Ocular Lineages. Cell Reports. 25 (6), 1668-1679 (2018).
- Mcbeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell Shape, Cytoskeletal tenstion and RhoA regulate stem cell lineage committment. Developmental Cell. 6, 483-495 (2004).
- Halder, G., Dupont, S., Piccolo, S. Transduction of mechanical and cytoskeletal cues by YAP and TAZ. Nature Reviews Molecular Cell Biology. 13, 591-600 (2012).
- Jessen, K. R., Mirsky, R. The repair Schwann cell and its function in regenerating nerves. Journal of Physiology. 594 (13), 3521-3531 (2016).
- Lopez-Fagundo, C., Bar-Kochba, E., Livi, L. L., Hoffman-Kim, D., Franck, C. Three-dimensional traction forces of Schwann cells on compliant substrates. Journal of The Royal Society Interface. 11, 20140247(2014).
- Gu, Y., et al. The influence of substrate stiffness on the behavior and functions of Schwann cells in culture. Biomaterials. 33, 6672-6681 (2012).
- Xu, Z. Y., Orkwis, J. A., DeVine, B. M., Harris, G. M. Extracellular matrix cues modulate Schwann cell morphology, proliferation, and protein expression. Journal of Tissue Engineering and Regenerative. , (2019).
- Urbanski, M. M., et al. Myelinating glia differentiation is regulated by extracellular matrix elasticity. Scientific Reports. 6, 1-12 (2016).
- Sun, Y., et al. Tunable stiffness of graphene oxide/polyacrylamide composite scaffolds regulates cytoskeleton assembly. Chemical Sciences. 9 (31), 6516-6522 (2018).
- Hwang, J. H., et al. Extracellular matrix stiffness regulates osteogenic differentiation through MAPK activation. PLoS One. 10, 1-16 (2015).
- Ryan, A. J., et al. A Physicochemically Optimized and Neuroconductive Biphasic Nerve Guidance Conduit for Peripheral Nerve Repair. Advanced Healthcare Materials. 6, 1-13 (2017).
- Du, J., et al. Prompt peripheral nerve regeneration induced by a hierarchically aligned fibrin nanofiber hydrogel. Acta Biomaterialia. 55, 296-309 (2017).
- Huang, L., et al. A compound scaffold with uniform longitudinally oriented guidance cues and a porous sheath promotes peripheral nerve regeneration in vivo. Acta Biomaterialia. 68, 223-236 (2018).
- Brower, K., White, A. K., Fordyce, P. M. Multi-step Variable Height Photolithography for Valved Multilayer Microfluidic Devices. Jouranl of Visualized Experiments. (119), e55276(2017).
- Gupta, R., et al. Shear stress alters the expression of myelin-associated glycoprotein (MAG) and myelin basic protein (MBP) in Schwann cells. Journal of Orthopaedic Research : Official Publication of the Orthopaedic Research Society. 23, 1232-1239 (2005).
- Harris, G. M., Shazly, T., Jabbarzadeh, E. Deciphering the combinatorial roles of geometric, mechanical, and adhesion cues in regulation of cell spreading. PLoS One. 8 (11), (2013).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Schreck, I., et al. C-Jun localizes to the nucleus independent of its phosphorylation by and interaction with JNK and vice versa promotes nuclear accumulation of JNK. Biochemical and Biophysical Research Communications. 407, 735-740 (2011).
- Shen, K., Qi, J., Kam, L. C. Microcontact printing of proteins for cell biology. Journal Visualized Experiments. (22), e1065(2008).
- Treter, J., et al. Washing-resistant surfactant coated surface is able to inhibit pathogenic bacteria adhesion. Applied Surface Science. 303, 147-154 (2014).
- Lutz, J. F. Polymerization of oligo(ethylene glycol) (meth)acrylates: Toward new generations of smart biocompatible materials. Journal of Polymer Science Part A: Polymer Chemistry. 46 (11), 3459-3470 (2008).
- Marcus, M., et al. Interactions of Neurons with Physical Environments. Advanced Healthcare Materials. 6, (2017).
- Pu, J. Golgi polarization in a strong electric field. Journal of Cell Science. 118, 1117-1128 (2005).
- Blaker, J. J., et al. Bioactive Silk-Based Nerve Guidance Conduits for Augmenting Peripheral Nerve Repair. Advanced Healthcare Materials. 7, 1800308(2018).
- Daly, W., Yao, L., Zeugolis, D., Windebank, A., Pandit, A. A biomaterials approach to peripheral nerve regeneration : bridging the peripheral nerve gap and enhancing functional recovery. Journal of the Royal Society of Interface. 9 (67), 202-221 (2012).
- Xia, H., et al. Directed neurite growth of rat dorsal root ganglion neurons and increased colocalization with Schwann cells on aligned poly(methyl methacrylate) electrospun nanofibers. Brain Research. 1565, 18-27 (2014).
- Wang, H. B., Mullins, M. E., Cregg, J. M., McCarthy, C. W., Gilbert, R. J. Varying the diameter of aligned electrospun fibers alters neurite outgrowth and Schwann cell migration. Acta Biomaterialia. 6, 2970-2978 (2010).
- Carvalho, C. R., Oliveira, J. M., Reis, R. L. Modern Trends for Peripheral Nerve Repair and Regeneration: Beyond the Hollow Nerve Guidance Conduit. Frontiers in Bioengineering and Biotechnology. 7, 337(2019).
- Yang, Y., Wang, K., Gu, X., Leong, K. W. Biophysical Regulation of Cell Behavior - Cross Talk between Substrate Stiffness and Nanotopography. Engineering. 3, 36-54 (2017).
- Tan, J. L., Liu, W., Nelson, C. M., Raghavan, S., Chen, C. S. Simple Approach to Micropattern Cells on Common Culture Substrates by Tuning Substrate Wettability. Tissue Engineering. 10, 865-872 (2004).
- Grove, M., et al. YAP/TAZ initiate and maintain schwann cell myelination. Elife. 6, 1-27 (2017).
- Poitelon, Y., et al. YAP and TAZ control peripheral myelination and the expression of laminin receptors in Schwann cells. Nature Neuroscience. 19, 879-887 (2016).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon