É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Preparação de Microambientes de Matriz Extracelular Incapaz para avaliar especificação de fenótipo celular schwann
Neste Artigo
Resumo
Esta metodologia visa ilustrar os mecanismos pelos quais pistas de matriz extracelular, como rigidez do substrato, composição de proteínas e morfologia celular regulam o fenótipo de células schwann (SC).
Resumo
Atualmente, as lesões traumáticas do sistema nervoso periférico (PNS) não possuem tratamentos adequados para recuperar a recuperação funcional completa. As células schwann (SCs), como as principais células gliais da PNS, desempenham um papel vital na promoção da regeneração da PNS, dediferindo-se em um fenótipo celular regenerativo após a lesão. No entanto, o estado dediferenciado das SCs é desafiador para manter-se durante o período de tempo necessário para a regeneração e é impactado por mudanças na matriz extracelular circundante (ECM). Portanto, determinar a interação complexa entre SCs e ECM diferente para fornecer pistas de potencial regenerativo das SCs é essencial. Para lidar com isso, foi criada uma estratégia onde diferentes proteínas ECM foram adsorvidas em um substrato de polidimetilatilaxitano (PDMS) que forneceu uma plataforma onde a rigidez e a composição proteica podem ser moduladas. As SCs foram semeadas nos substratos tunable e foram medidas funções celulares críticas que representam a dinâmica do fenótipo de SC. Para ilustrar a interação entre a expressão proteica SC e a morfologia celular, foram utilizadas diferentes densidades de semeadura de SCs, além de padrões celulares impressos em microcontatos individuais e caracterizados pela coloração da imunofluorescência e mancha ocidental. Os resultados mostraram que as células com menor área de disseminação e maior extensão do alongamento celular promoveram níveis mais elevados de marcadores fenotípicos regenerativos de SC. Essa metodologia não só começa a desvendar a relação significativa entre o ECM e a função celular das SCs, mas também fornece diretrizes para a otimização futura de biomateriais na reparação do nervo periférico.
Introdução
As lesões do sistema nervoso periférico (PNS) continuam sendo um grande desafio clínico na saúde, comprometendo a qualidade de vida dos pacientes e criando um impacto significativo através de uma infinidade de fatores socioeconômicos1,2. As células schwann (SC), como as principais células gliais da PNS, fornecem sinais moleculares e físicos necessários para induzir a regeneração da PNS e auxiliar em recuperações funcionais em lesões de curto espaço. Isso se deve à notável capacidade dos SCs de dediferentes em um fenótipo celular de "reparação" de um fenótipo de mieliagem ou remak3. O reparo SC é um fenótipo celular distinto de várias maneiras. Após a lesão, as SCs aumentam sua taxa de proliferação reentrando no ciclo celular e iniciam a expressão de vários fatores transcricionais para facilitar a reinervação. Esses fatores, como c-Jun e p75 NTR, são regulados, enquanto os marcadores de SC de mielin, como a proteína básica de mielina (MBP), estão abaixo da regulação4,,5. Além disso, os SCs mudam a morfologia para se alongarem e se alinharem entre si para formar bandas de Büngner em todo o local da lesão6. Isso fornece um mecanismo de orientação física para que os axônios se estendam até o alvo distal correto7. No entanto, apesar da capacidade que as SCs possuem para promover a regeneração nervosa em lesões de curto espaço de espaço, o resultado da recuperação funcional permanece ruim em lesões graves. Isso se deve, em parte, à perda de pistas de orientação da matriz extracelular (ECM), bem como à incapacidade das SCs de manter o fenótipo regenerativo por longos períodos de tempo8.
O processo de regeneração nervosa e recuperação estão intimamente ligados ao estado da lamina basal após lesão. A lamina basal é uma camada de ECM em torno do nervo que facilita a orientação e fornece pistas vinculadas ao ECM para axônios e SCs nos casos em que permanece intacta após a lesão9. O estado do ECM e sua capacidade de entregar pistas ligadas à matriz às células é de vital importância e já foi explorado anteriormente em uma variedade de diferentes contextos10,11,,12,,13,14. Por exemplo, foi demonstrado que a rigidez do ECM pode orientar funções celulares como proliferação e diferenciação11,,15,,16. A composição do ECM também pode levar a uma resposta celular distinta e regular comportamentos celulares como migração e diferenciação através de vias de sinalização intracelular17,18. Além disso, a morfologia celular, incluindo a expansão da área e o alongamento celular, desempenham um papel importante na regulação da função e podem ser regidas pelas pistas vinculadas ao ECM19,,20. Muitos estudos anteriores se concentraram em células-tronco que se diferenciam em linhagens definidas, mas as SCs possuem uma capacidade semelhante de alterar o fenótipo de um HOMEOSTático, adulto SC dentro de um nervo saudável, para um SC reparador capaz de segregar proteínas e fatores de crescimento enquanto remodelam o ECM após lesão nervosa5,,21. Portanto, é especialmente crucial identificar mecanismos subjacentes à relação entre a capacidade regenerativa de SC inata e as pistas vinculadas ao ECM para que a percepção exaia essa capacidade de regeneração nervosa.
Para lidar com isso, desenvolvemos uma metodologia detalhada para produzir um substrato de cultura celular onde rigidez mecânica e tipo de ligante podem ser facilmente sintonizados em faixas fisiologicamente relevantes. O siloxano de polidimtil (PDMS) foi escolhido como substrato devido à sua mecânica altamente tíclica em comparação com o gel de poliacrilamida, onde o módulo máximo de Young é em torno de 12 kPa contrastado com PDMS em torno de 1000 kPa22,23,,24. Isso é benéfico para o trabalho em questão, pois estudos recentes mostraram que o módulo de um nervo ciático de coelho pode exceder 50 kPa durante o desenvolvimento, sugerindo assim que a gama de rigidez dos nervos dentro da PNS é maior do que o examinado anteriormente. Diferentes proteínas são capazes de adsorção em substratos PDMS para analisar a regulação combinatória de mecânica e ligantes sobre o comportamento de SC. Isso permite a investigação de múltiplas pistas microambientais presentes no processo de regeneração da PNS e comparação de um alto grau de sintonia com o trabalho com foco apenas na rigidez do substrato25. Além disso, esses substratos de cultura celular projetada são compatíveis com uma infinidade de métodos de análise quantitativa, como imunohistoquímica, mancha ocidental e reação quantitativa em cadeia de polimerase (q-PCR).
Esta plataforma de cultura celular projetada é altamente adequada para analisar caminhos mecanicistas devido ao alto nível de sintonia individual de cada sinal vinculado ao ECM. Além disso, métodos populares para micropatterning celular, incluindo impressão de microcontatos, podem ser alcançados nos substratos para permitir a adesão celular controlada para analisar a forma celular em relação a outras pistas ligadas ao ECM24. Isso é fundamental porque substratos padronizados de linha, que promovem o alongamento em populações celulares, fornecem uma ferramenta para imitar e estudar SCs alongados e regenerativos dentro de bandas Büngner durante a regeneração nervosa. Além disso, a morfologia celular é um potente regulador de múltiplas funções celulares e pode potencialmente introduzir resultados experimentais confusos se não for controlada26,27. Atenção significativa está sendo dada aos mecanismos que regem o fenótipo regenerativo de SC, conforme regulado pelas sugestões ECM28,,29,,30. Isso é essencial para fornecer uma visão sobre o desenho de biomateriais que podem ser aplicados como conduítes de orientação nervosa para auxiliar na regeneração nervosa da PNS. Esses protocolos detalhados podem, em última análise, ser aplicados como uma ferramenta potencial para decifrar os mecanismos de SC e outros tipos de células funcionam conforme regulado por pistas vinculadas ao ECM.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparação e caracterização da cultura celular incapaz
- Preparação de substrato
- Misture o elastômero base PDMS e os agentes de cura usando uma ponta de pipeta vigorosamente em uma relação entre 10:1 e 60:1 até que as bolhas sejam dispersas homogêneamente dentro da mistura. Remova bolhas usando a proficação do vácuo até que as bolhas sejam dissipadas.
NOTA: Durante a polimerização do PDMS, o agente de cura cruza com o elastômero base para fornecer propriedades mecânicas desejadas pelo polímero final. As razões do Crosslink podem ser ajustadas para alterar a rigidez do PDMS. - Coloque uma gota (~0,2 mL) de mistura PDMS dessecada em um deslizamento de cobertura quadrado ou circular (por exemplo, 22 mm x 22 mm) e gire a tampa em um revestimento de spin a 2500 rpm por 30 s.
- Incubar a tampa em um forno a 60 °C por 1-2 h ou temperatura ambiente durante a noite para pdms solidificar.
- Trate o deslizamento de cobertura usando limpador de UV-Ozônio por 7 min (comprimento de onda UV: 185 nm e 254 nm) para aumentar a hidrofilialidade da superfície. Coloque-o em uma placa esterilizada de 6 poços.
- Antes de usar para cultura celular, incubar substratos em 70% de etanol por pelo menos 30 min.
ATENÇÃO: O limpador de UV-Ozônio pode gerar ozônio que é prejudicial aos seres humanos. Trabalhe em um capô de fumaça química ou com alguma forma de ventilação. - Mergulhe as manchas na solução proteica (10 μg/mL colágeno I, fibronectina ou laminina) por 60 min em uma incubadora estéril a 37 °C.
NOTA: Após o tratamento UV-Ozônio, a superfície do PDMS ainda pode ser hidrofóbica. Gire a placa do poço para garantir que cada deslizamento de cobertura esteja coberto com a solução proteica. - Aspire a solução proteica e lave a tampa com salina tamponada de fosfato (PBS) 3x.
- Suspenda re suspendida a linha celular RT4-D6P2T Schwann (SCs) do prato de passagem usando a solução EDTA disponível comercialmente (1x) com trippsina de 2,5% e células de contagem com hemócitometro. SCs de sementes na superfície PDMS tunable na densidade celular desejada. As densidades de semeadura sc podem variar para cada aplicação diferente.
- Manter as células nos parâmetros de cultura celular desejados (90% de umidade, 5% de CO2,37 °C, etc.) para o comprimento do experimento. Use o DMEM (Modified Eagle Medium, meio de águia modificada) de Dulbecco, suplementado com 10% de soro bovino fetal (FBS) e 1% penicilina-estreptomicina como meio de cultura celular.
- Misture o elastômero base PDMS e os agentes de cura usando uma ponta de pipeta vigorosamente em uma relação entre 10:1 e 60:1 até que as bolhas sejam dispersas homogêneamente dentro da mistura. Remova bolhas usando a proficação do vácuo até que as bolhas sejam dissipadas.
- Preparação de substrato micropatterado
- Desenhe as áreas de geometria e adesivo de celular desejadas (900 μm2, 1.600 μm2 e 2.500 μm2) usando o software CAD (Computer-Aided Design, design aided design" . Crie uma fotomasmak cromada com base nesses padrões de um fornecedor comercial.
- Em uma sala limpa ou ambiente livre de poeira, use técnicas padrão de fotolitografia para fabricar wafers de silício (os protocolos são detalhados em outros lugares31). Os parâmetros críticos para esta aplicação em particular são os seguintes: Fotoresist: SU-8 2010; Perfil de rotação para dispersar o fotoresist: 500 rpm para 10 s com aceleração de 100 rpm/s, depois 3500 rpm para 30 s com aceleração de 300 rpm/s; Energia de exposição da luz UV: 130 mJ/cm2.
NOTA: A altura dos padrões nos wafers de silício é de aproximadamente 10 μm seguindo esses parâmetros. Rachaduras potenciais ao redor da borda fora dos padrões retangulares ou triangulares podem ser vistas usando um microscópio de luz após o passo 1.2.2. Assar o wafer de silício a 190 °C por 30 min ajuda a eliminar as rachaduras. - Coloque o wafer de silício estampado dentro de uma placa de Petri circular de 150 mm de diâmetro x 15 mm de altura e despeje PDMS desa gás (razão de mistura 10:1) conforme preparado na etapa 1.1.1 para o wafer de silício.
NOTA: Certifique-se de que a espessura do PDMS é de pelo menos 5 mm para facilitar o manuseio durante as etapas de impressão de microcontatos. - Solidificar PDMS no wafer de silício em um forno a 60 °C durante a noite. Deixe o PDMS esfriar à temperatura ambiente. Corte precisamente de selos de 30 mm x 30 mm quadrados contendo os padrões corretos do wafer de silício usando um bisturi cirúrgico. Não danifique o wafer de silício.
NOTA: Os wafers de silício podem ser reutilizados muitas vezes neste momento para produzir mais selos após a limpeza com isopropanol. - Esterilize os selos PDMS e as tampas tunable (preparadas na etapa 1.1.1 a 1.1.3) imergindo-os em 70% de etanol por 30 min.
- Para confirmar a eficácia da micropattern por selos PDMS após a impressão de microcontatos, seque a superfície dos selos PDMS usando uma solução filtrada de fluxo de ar e pipeta de 50 μg/mL BSA (Texas Red conjugado) para cobrir todo o lado padronizado do selo PDMS.
- Incubar selos PDMS com solução BSA por 1 h à temperatura ambiente para permitir a adsorção de proteínas.
- Seque a superfície de tampas tunable usando um fluxo de ar filtrado, aumente a hidrofilialidade superficial conforme descrito na etapa 1.1.5.
- A seco o ar dos selos PDMS para remover a solução BSA restante.
NOTA: Tome cuidado para que a solução BSA seja completamente removida do selo porque qualquer solução restante fará com que os selos deslizem no deslizamento de tampas durante a impressão de microcontatos. - Leve o lado padronizado do selo em contato conformal com o deslizamento de cobertura tunable para adsorção BSA na superfície do deslizamento de tampas. Pressione suavemente o carimbo contra o deslizamento por 5 minutos.
NOTA: Não aplique força excessiva no carimbo, pois ele irá dobrar e causar contato não específico entre carimbo e deslizamento de cobertura. A quantidade adequada de força aplicada no selo é essencial para a impressão de microcontato bem sucedida. - Examine o micropattern usando microscópio de fluorescência com um filtro FITC (Fluoresceína isothiocyanato).
- Para imprimir áreas adesivas celulares em vez de padrões fluorescentes, substitua a laminina pela proteína BSA e repita o passo 1.2.5 a 1.2.10.
- Remova os selos das tampas, transfira as tampas em uma placa esterilizada de 6 poços. Adicione 2 mL de solução Pluronic F-127 em cada poço para cobrir a superfície do deslizamento de tampas e incubar por 1h à temperatura ambiente.
NOTA: O F-127 plurônico pode ser adsorvido à superfície do PDMS aumentando a hidroofobidade da superfície PDMS para bloquear as células da adesão. - Aspirar solução Pluronic F-127 e lavar 5x com PBS e 1x com o meio de cultura celular antes de semeadura de células. Uma densidade de semeadura típica para SCs é de 1.000 células/cm2.
- 45 minutos após a semeadura celular, remova o meio de cultura celular e lave as tampas com PBS 2x para evitar que vários SCs aderam ao mesmo padrão. Manter células no ambiente de cultura celular desejada por 48 h antes da quantificação.
- Para criar substratos de cultura celular com padrão de linha para examinar células alinhadas, siga o passo 1.2.1 a 1.2.4 para criar selos para impressão de microcontatos.
NOTA: As dimensões do sulco/cume dos padrões forrados no selo são de 50 μm x 50 μm para as células delineadas. As dimensões totais do selo são de 10 mm x 10 mm. - Corte selos em dimensões que contenham apenas padrões de linha desejados.
NOTA: Ao criar selos em CAD, a área não padrão do selo em torno dos padrões de linha corresponderá à área adesiva celular após a impressão de microcontatos. Assim, é necessário eliminar áreas não respingadas ao cortar o selo para garantir que cada SC semeada na superfície siga os padrões. - Siga o passo 1.1.1 a 1.1.3 para preparar a superfície PDMS tunable revestindo duas placas de Petri.
NOTA: Este será pdms cobrindo a superfície da placa de Petri em si e não em um deslizamento de tampa. - Siga o passo 1.2.5 a 1.2.10 para realizar a impressão de microcontatos para imprimir áreas adesivas de células com padrão de linha em uma das placas de Petri revestidas do PDMS.
NOTA: A área de superfície de uma placa de Petri de 60 mm x 15 mm pode conter áreas padrão de linha a partir de 6 selos PDMS. - Retire os selos e encha a placa de Petri com 4 mL de solução plurônica F-127 e incubar por 1h.
- Após a impressão de microcontato, enxágue cada lado dos selos PDMS com 70% de etanol 3x e seque com ar. Gire os selos PDMS e siga o passo 1.2.5 a 1.2.10 para imprimir a área adesiva celular não respingada usando o lado não despachado do selo na segunda placa de Petri. Repita o passo 1.2.13.
- Aspirar solução F-127 a partir de pratos, lavar 3x com PBS seguido com lavagem 1x usando meio de cultura celular fresca. SCs de sementes em pratos.
NOTA: A densidade de semeadura para um prato com padrão de linha é de 5.000 células/cm2 e para um prato não padronizado é de 10.000 células/cm2. - Manter os SCs nas condições desejadas por 48 h e seguir protocolos para preparar os lysates SC32.
NOTA: A densidade de semeadura celular para pratos não padronizados é 2x maior do que a dos pratos com padrão de linha, já que o prato com padrão de linha tem apenas metade da área adesiva celular do prato não padronizado.- Para preparar os lises, transfira o buffer de ensaio de radioimunoprecipitação adequado (RIPA) para um tubo de centrífuga cônica de 10 mL. Diluir protease e inibidor de fosfatase (100x) a uma proporção de 1:100 em tampão RIPA, misture bem por pipetação.
- Lave as células com PBS gelado (1x) por 2 min, adicione 80 μL da solução preparada a partir da etapa 1.2.23.1 em cada área adesiva celular (área que entrou em contato com selos PDMS e proteína adsorvida) dentro de placas de petri. Incubar células com a solução em um bloco de gelo por 15 minutos.
NOTA: A solução só permanecerá na área adesiva celular devido à hidroofobidade da adsorção Pluronic F-127 em outros lugares. Este recurso permite uma extração de proteína bem-sucedida e suficiente para SCs padronizados em linha. - Raspar SCs com um raspador de células por 5 min. Coletar lysate em um tubo de microcentrifuge rotulado de 1,5 mL.
- Microcentrifuge lysate a 12.000 x g para 15 min a 4°C. Colete supernante com uma pipeta de 1.000 μL e transfira para um tubo de microcentrifuge limpo. Armazenar lysate celular a -20 °C.
- Caracterização de substrato
NOTA: Para caracterizar a mecânica do polímero no deslizamento de cobertura, vários métodos são geralmente empregados, incluindo testes de compressão a granel11,,33 ou teste de microscopia de força atômica34. Este protocolo irá delinear testes de compressão em massa.- Despeje o precursor PDMS da razão de mistura desejada (passo 1.1) em uma placa de Petri de 30 mm, garantindo que a espessura da camada PDMS dentro da placa de Petri seja de pelo menos 20 mm.
- Retire a placa de Petri com PDMS solidificado a partir do forno de 60 °C após 1h e deixe esfriar à temperatura ambiente. Corte o polímero em quadrados de 10 mm x 10 mm. Meça a espessura do PDMS usando pinças.
- Coloque pdms no estágio da máquina de medição da força de compressão. Conecte o sensor de força de compressão (modelo: 112C) à porta do sensor e fixe o sensor no eixo da máquina de teste.
- Ajuste a altura do sensor para aproximadamente 0,5 cm acima do selo PDMS usando controle "jog" no painel frontal do instrumento.
- Abra o software associado usando a janela "Configuração de teste", selecione "perfil Servo", e abra a janela "Segmento". Na janela "Segmento", entrada desejada "Taxa de Controle" e " ValorFinal" para o teste.
NOTA: A taxa de controle determina a taxa em que o sensor viaja para baixo em direção ao PDMS. A quantidade final determina a distância total que o sensor percorre. - Use o botão "Z" localizado no painel de controle do software para redefinir todas as medições neste momento.
- Mova o sensor para baixo para entrar em contato levemente com o PDMS até que 1-2 newtons (N) estejam carregados. O carregamento e a distância que o sensor percorre serão exibidos no software.
- Depois de utilizar o botão "Z", execute a medição usando "Reprodução", e salve a força de gravação de arquivo e distância.
- Repetir as etapas 1.3.3 a 1.3.8 para cada condição experimental do PDMS.
- Abra o arquivo e use a seguinte fórmula para calcular o módulo deScolado (E) do PDMS para cada razão. (F = força de compressão, A = área do carimbo PDMS, ◗L = distância de viagem do sensor, e L0 = espessura original do selo PDMS).
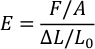
2. Quantificação de propriedades celulares em substratos tunable
- Ensaio de proliferação
- SCs de sementes em substratos preparados a partir da etapa 1.1.9 a uma densidade de 5.000 células/cm2 em uma placa de 6 poços. Permitir que os SCs incubam por 48 h em condições de cultura celular padrão (37 °C e 5% DE CO2).
- Diluir 12 μL de 10 mM Bromodeoxyuridine (BrdU) solução de estoque em 12 mL de meio de cultura celular de 37 °C, misture bem com pipeta para fazer uma solução de rotulagem brdU de 10 μM.
- Remova o meio de cultura celular e lave os SCs 2x com PBS.
- Adicione 2 mL de solução de rotulagem BrdU em cada poço e incubar SCs por 2 h.
NOTA: O tempo de incubação da solução de rotulagem brdU depende da taxa específica de proliferação celular. A linha RT4-D6P2T SC tem uma alta taxa de proliferação, por isso foi utilizado 2h de tempo de incubação. - Remova a solução de rotulagem BrdU e lave os SCs 3x com PBS. Adicione 1 mL de formaldeído de 3,7% em PBS a cada poço e incubar à temperatura ambiente por 15 minutos para fixação celular.
ATENÇÃO: O formaldeído é um cancerígeno humano; portanto, realize todo o trabalho dentro de um capô de fumaça química com proteção adequada.
NOTA: Ao lavar com PBS, não há meio de cultura celular no poço e, portanto, a superfície do PDMS pode ser hidrofóbica. Tome precauções para não secar completamente a superfície do substrato para evitar danos celulares. - Aspirar solução de formaldeído e lavar 3x com PBS (3 min cada). Remova o PBS e adicione 1 mL de 0,2% Triton X-100 em PBS a cada poço para permeabilize a membrana celular. Incubar SCs com solução Triton X-100 por 20 min a temperatura ambiente.
- Remova a solução Triton X-100 e lave os SCs 3x com PBS (3 min cada).
- Adicione 1 mL de 1 N HCl em cada poço e incubar no gelo por 10 min. Remova 1 N HCl e adicione 1 mL de 2 N HCl em cada poço e incubar à temperatura ambiente por 10 min. O tratamento de HCL é para hidrólise de DNA.
- Misture 182 mL de 0,2 mM Na2HPO4 e 18 mL de ácido cítrico de 0,1 mM para produzir um tampão de ácido fosfato/cítrico para recuperação de antígenos. Remova 2 N HCl e adicione 1 mL de fosfato/tampão de ácido cítrico em cada poço e incubar à temperatura ambiente por 10 minutos.
- Lave SCs 3x com Triton X-100 de 0,2% em PBS. Adicione 2 mL de 3% de gêrina bovina (BSA) em PBS em cada poço e incubar por 30 minutos à temperatura ambiente para marcar a ligação inespecífica do anticorpo.
- Diluir o anticorpo primário BrdU conjugado com alexa Fluor 488 em solução BSA de 3% a uma proporção de 1:300 para solução de coloração brdU. Incubar SCs com solução de coloração durante a noite à temperatura ambiente, enquanto a placa é coberta por papel alumínio.
- Para quantificar a proliferação, os SCs de imagem utilizam o canal FITC e DAPI de um microscópio fluorescente para detectar BrdU e núcleos, respectivamente. Salve imagens como arquivos "nd.2".
- Abra arquivos "nd.2" para cada imagem tirada em posições espaciais idênticas.
- Abra o software de análise de imagens. Clique com o botão direito do mouse no plano de fundo para abrir a janela "Resultados de Medição Automatizada" e " MediçãoAutomatizada" na seção " Controle deAnálise".
- No menu "Conde e Taxonomia", selecione "Conde". Na imagem FITC, clique em cada núcleo mostrando fluorescência verde (BrdU positivo) e clique com o botão direito do mouse na imagem.
NOTA: O número de células positivas de BrdU são mostrados na janela de "Automações e Medidas". - Para imagens DAPI, repita a etapa 2.1.14 para contar o número de núcleos totais. Calcule a porcentagem de células positivas de BrdU para esta imagem.
- Repita a etapa 2.1.13 a 2.1.16 para outras imagens para fins estatísticos e calcule o percentual médio de células positivas de R$ 1.000 para cada condição de substrato.
- Quantificação da expressão c-Jun através da análise de imagem imunofluorescente
- As células preparadas dentro de placas de 6 poços da etapa 1.1.9 e 1.2.23 são fixas e permeabilizadas com os procedimentos descritos anteriormente (etapa 2.1.5-2.1.7).
NOTA: Para realizar comparações precisas de intensidade fluorescente entre células de diferentes condições de ECM, aplique as configurações da câmera de forma idêntica em todas as amostras, com todas as amostras tendo os mesmos parâmetros. - Salvar imagens como arquivos ".nd2".
- Abra o software de análise de imagens. Clique com o botão direito do mouse para abrir a janela "Resultados de Medição Automatizada" e " MediçãoAutomatizada" na seção " Controle deAnálise".
- Em "Resultados de Medição Automatizada",selecione "Dados de Objeto". Ativar o botão "Manter atualizandoa medição ".
- No painel superior do software, selecione "Medida" seguido de "Recursos de objeto". Adicione "Intensidade Média" à seção de " Selecionadopara medição".
- Abra e mescle dois arquivos de imagem ".nd2" que contêm imagens de c-Jun e núcleos.
- No painel superior, selecione "ROI" e selecione "Desenhar ROI Retangular". Desenhe uma área retangular contendo a área nuclear de uma única célula.
NOTA: a expressão c-Jun está concentrada dentro dos núcleos35. - No painel superior do software, selecione "Binário" e "Definir Limiar", uma nova janela aparecerá para definir precisamente a área fluorescente c-Jun.
- Em nova janela, clique em "Full Image/Use ROI" para mudar o programa do modelo de imagem completo para o modelo ROI. Use "Intensidade" para ajustar a tabela de procuração localizada no lado esquerdo da janela para ajuste do tamanho/forma da área destacada dentro do ROI retangular.
NOTA: Tome cuidado para garantir que o tamanho/forma da área destacada seja idêntico ao núcleo. - Clique no botão "OK", para obter a intensidade fitc média na janela de " Resultados deMedição Automatizada" e, em seguida, clique em "Armazenar dados".
- Na janela de "Medição Automatizada", "Delete Object" para remover a área destacada vermelha. No painel do lado esquerdo, use "Ferramenta de Pontaria" para selecionar o ROI retangular e excluir.
- Repita a etapa 2.2.6 a 2.2.11 para medir a intensidade média fitc para cada célula adicional.
- Na área da janela de "Resultados de Medição Automatizada", selecione "Armazenado" e todos os dados armazenados serão apresentados. Use a função "Exportar" e selecione "Dados para Excel" para salvar a planilha exportada e realizar cálculos adicionais.
- As células preparadas dentro de placas de 6 poços da etapa 1.1.9 e 1.2.23 são fixas e permeabilizadas com os procedimentos descritos anteriormente (etapa 2.1.5-2.1.7).
- Quantificando o alongamento nuclear
- Fix and permeabilize SCs preparados a partir da etapa 1.2.22 seguindo as etapas 2.1.5-2.1.7. Realize a coloração nuclear utilizando meio de montagem com DAPI.
- Usando o canal DAPI e uma lente objetiva de 40x, adquira imagens da amostra e salve como arquivos ".nd2".
- Siga as etapas 2.2.3 e 2.2.4 para abrir "Resultados de Medição Automatizada" e " MediçãoAutomatizada" no software de análise de imagem.
- Em "Resultados de Medição Automatizada", use a função "Opção" seguida por " SelectObject Feature". Na coluna "Recurso", selecione "Alongamento" e adicione à coluna "Selecionado para Medidas". Use " Manter amedição de atualização" para ativar esta função.
- Abra o arquivo de imagem "nd.2" que contém as imagens nucleares. Na janela "Medição Automatizada", selecione "Auto Detect" e selecione um núcleo. Clique com o botão direito do mouse na imagem e na proporção nuclear medida será mostrado em "Resultados de Medição Automatizada".
- Repita o passo 2.3.5 para quantificar as proporções nucleares para outros núcleos dentro da imagem. Selecione "Armazenar dados" na janela de " Resultados deMedição Automatizada".
- Repita 2.3.5 a 2.3.6 para imagens adicionais. Exporte os dados para um arquivo de planilha como feito anteriormente em 2.2.13 para análise.
- Mancha ocidental para quantificar expressão proteica
- Siga os protocolos padrão para análise de manchas ocidentais detalhados em outros lugares32. As diluições dos anticorpos utilizados no estudo são mostradas abaixo: Coelho anti c-Jun 1:2.000; Mouse anti β-actin 1:1.000; Coelho anti p75NTR 1:1.000; Proteína básica anti-mielina de coelho 1:1.000; IgG anti-rato/coelho, anticorpo ligado ao HRP 1:10.000.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para analisar e quantificar a interação entre rigidez do substrato e composição proteica no fenótipo de SC, foi desenvolvido um substrato de cultura celular PDMS tunable(Figura 1A). Testes de compressão do polímero em base diferente: foram utilizadas relações de agente de cura para quantificar o módulo do Jovem (E) do substrato(Figura 1B). A faixa resultante de valores de módulo representa condições de substrato fisiologicamente relevantes. Após a ...
Access restricted. Please log in or start a trial to view this content.
Discussão
As SCs podem promover a regeneração nervosa devido à sua transformação fenotípica e potencial regenerativo após lesão nervosa. No entanto, a forma como as sugestões de ECM regulam essa capacidade regenerativa permanece em sua maioria incerta, potencialmente dificultando não apenas o desenvolvimento de biomateriais que visam promover a regeneração nervosa, mas também a compreensão dos mecanismos envolvidos na regeneração nervosa. Para começar a examinar essa interação, substratos de cultura celular fora...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Nenhum potencial conflito de interesses foi relatado pelos autores.
Agradecimentos
Os autores agradecem o apoio ao financiamento da Universidade de Cincinnati. Os autores também agradecem a Ron Flenniken, do laboratório de Caracterização de Materiais Avançados da Universidade de Cincinnati, pelo apoio.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Albumin from Bovine Serum (BSA), Texas Red conjugate | Thermo Fisher Scientific | A23017 | BSA staining to show micropatterns |
| Anti-mouse IgG, HRP-linked Antibody | Cell Signaling Technology | 7076S | Antibody used for western blot analysis |
| Anti-rabbit IgG, HRP-linked Antibody | Cell Signaling Technology | 7074S | Antibody used for western blot analysis |
| BrdU | Thermo Fisher Scientific | B23151 | Reagent used to measure cell proliferation |
| BrdU primary antibody conjugated with Alexa Fluor 488 | Thermo Fisher Scientific | B35130 | Used to visualize BrdU in cell proliferation assays |
| Collagen I | Thermo Fisher Scientific | A10483-01 | Protein used to coat coverslips |
| Compression force test machine | TestResources | Instrument to quantify mechanical properties of polymers | |
| Dulbecco's Modified Eagle Medium | Thermo Fisher Scientific | 11965092 | Cell culture medium |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | Cell culture medium supplemental |
| Fibronectin | Thermo Fisher Scientific | 33010-018 | Protein used to coat coverslips |
| Fluorescence microscope | Nikon | Eclipse Ti2 | Fluorescence microscope |
| Halt Protease and Phosphatase Inhibitor Cocktail (100X) | Thermo Fisher Scientific | 78440 | Protease and Phosphatase Inhibitor |
| Laminin | Thermo Fisher Scientific | 23017015 | Protein used to coat coverslips |
| Mounting medium with DAPI | Thermo Fisher Scientific | P36971 | Coverslip mountant and nuclei staining |
| Mouse c-Jun primary antibody | Thermo Fisher Scientific | 711202 | Primary antibody to visualize c-Jun protein |
| Mouse β-Actin primary antibody | Cell Signaling Technology | 3700S | Loading control for western blot experiments |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | Cell culture medium supplemental |
| Photoresist SU 2010 | KAYAKU | SU8-2010 | Photoresist |
| Pluronic F-127 | Sigma Aldrich | P-2443 | Block non-specific protein binding |
| Rabbit c-Jun primary antibody | Cell Signaling Technology | 9165S | Primary antibody for visualization of c-Jun protein |
| Rabbit myelin basic protein primary antibody | Abcam | ab40390 | Primary antibody for visualization of MBP |
| Rabbit p75NTR primary antibody | Cell Signaling Technology | 8238S | Primary antibody for visualization of p75NTR |
| Rhodamine phalloidin | Thermo Fisher Scientific | R415 | Visualization of cell cytoskeleton |
| RIPA buffer | Abcam | ab156034 | Cell lysis buffer |
| RT4-D6P2T Schwann cell line | ATCC | CRL-2768 | Cell line used in experiments |
| SYLGARD 184 PDMS base and curing agent | Sigma Aldrich | 761036 | Tunable polymer used to coat coverslips |
| Trypsin | Thermo Fisher Scientific | 15090-046 | Cell dissociation reagent |
| UV-Ozone cleaner | Novascan | Increase hydrophicility of PDMS | |
| Versene (1x) | Thermo Fisher Scientific | 15040066 | Cell dissociation reagent |
Referências
- Taylor, C. A., Braza, D., Rice, J. B., Dillingham, T. The Incidence of Peripheral Nerve Injury in Extremity Trauma. American Journal of Physical Medicine & Rehabilitation. 87, 381-385 (2008).
- Noble, J., Munro, C. A., Prasad, V. S. S. V., Midha, R. Analysis of Upper and Lower Extremity Peripheral Nerve Injuries in a Population of Patients with Multiple Injuries. Journal of Trauma and Acute Care Surgery. 45, 116-122 (1998).
- Jessen, K. R., Mirsky, R. The repair Schwann cell and its function in regenerating nerves. Journal of Physiology. 594, 3521-3531 (2016).
- Arthur-Farraj, P. J., et al. c-Jun Reprograms Schwann Cells of Injured Nerves to Generate a Repair Cell Essential for Regeneration. Neuron. 75, 633-647 (2012).
- Jessen, K. R., Mirsky, R. The Success and Failure of the Schwann Cell Response to Nerve Injury. Frontiers in Cell Neurosciences. 13, 33(2019).
- Gomez-Sanchez, J. A., et al. After Nerve Injury, Lineage Tracing Shows That Myelin and Remak Schwann Cells Elongate Extensively and Branch to Form Repair Schwann Cells, Which Shorten Radically on Remyelination. Journal of Neuroscience. 37 (37), 9086-9099 (2017).
- Deumens, R., et al. Repairing injured peripheral nerves: Bridging the gap. Progress in Neurobiology. 92, 245-276 (2010).
- Höke, A., Gordon, T., Zochodne, D. W., Sulaiman, O. A. R. A decline in glial cell-line-derived neurotrophic factor expression is associated with impaired regeneration after long-term Schwann cell denervation. Experimental Neurology. 173, 77-85 (2002).
- Jones, S., Eisenberg, H. M., Jia, X. Advances and future applications of augmented peripheral nerve regeneration. International Journal of Molecular Sciences. 17, 1-17 (2016).
- Harris, G. M., et al. Nerve Guidance by a Decellularized Fibroblast Extracellular Matrix. Matrix Biology. 60-61, 176-189 (2017).
- Harris, G. M., Piroli, M. E., Jabbarzadeh, E. Deconstructing the Effects of Matrix Elasticity and Geometry in Mesenchymal Stem Cell Lineage Commitment. Advanced Function Mater. 24 (16), 2396-2403 (2014).
- Pryzhkova, M. V., Harris, G. M., Ma, S., Jabbarzadeh, E. Patterning pluripotent stem cells at a single cell level. Journal of Biomaterials and Tissue Engineering. 3 (4), 461-471 (2013).
- Engler, A. J., Sweeney, H. L., Discher, D. E., Schwarzbauer, J. E. Extracellular matrix elasticity directs stem cell differentiation. Journal of Musculoskeleton and Neuronal Interaction. 7 (4), 335(2007).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix Elasticity Directs Stem Cell Lineage Specification. Cell. 126, 677-689 (2006).
- Pickup, M. W., Mouw, J. K., Weaver, V. M. The extracellular matrix modulates the hallmarks of cancer. EMBO Reports. 15, 1243-1253 (2014).
- Chernousov, M. A., Carey, D. J. Schwann cell extracellular matrix molecules and their receptors. Histology and Histopathology. 15, 593-601 (2000).
- Shibata, S., et al. Selective Laminin-Directed Differentiation of Human Induced Pluripotent Stem Cells into Distinct Ocular Lineages. Cell Reports. 25 (6), 1668-1679 (2018).
- Mcbeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell Shape, Cytoskeletal tenstion and RhoA regulate stem cell lineage committment. Developmental Cell. 6, 483-495 (2004).
- Halder, G., Dupont, S., Piccolo, S. Transduction of mechanical and cytoskeletal cues by YAP and TAZ. Nature Reviews Molecular Cell Biology. 13, 591-600 (2012).
- Jessen, K. R., Mirsky, R. The repair Schwann cell and its function in regenerating nerves. Journal of Physiology. 594 (13), 3521-3531 (2016).
- Lopez-Fagundo, C., Bar-Kochba, E., Livi, L. L., Hoffman-Kim, D., Franck, C. Three-dimensional traction forces of Schwann cells on compliant substrates. Journal of The Royal Society Interface. 11, 20140247(2014).
- Gu, Y., et al. The influence of substrate stiffness on the behavior and functions of Schwann cells in culture. Biomaterials. 33, 6672-6681 (2012).
- Xu, Z. Y., Orkwis, J. A., DeVine, B. M., Harris, G. M. Extracellular matrix cues modulate Schwann cell morphology, proliferation, and protein expression. Journal of Tissue Engineering and Regenerative. , (2019).
- Urbanski, M. M., et al. Myelinating glia differentiation is regulated by extracellular matrix elasticity. Scientific Reports. 6, 1-12 (2016).
- Sun, Y., et al. Tunable stiffness of graphene oxide/polyacrylamide composite scaffolds regulates cytoskeleton assembly. Chemical Sciences. 9 (31), 6516-6522 (2018).
- Hwang, J. H., et al. Extracellular matrix stiffness regulates osteogenic differentiation through MAPK activation. PLoS One. 10, 1-16 (2015).
- Ryan, A. J., et al. A Physicochemically Optimized and Neuroconductive Biphasic Nerve Guidance Conduit for Peripheral Nerve Repair. Advanced Healthcare Materials. 6, 1-13 (2017).
- Du, J., et al. Prompt peripheral nerve regeneration induced by a hierarchically aligned fibrin nanofiber hydrogel. Acta Biomaterialia. 55, 296-309 (2017).
- Huang, L., et al. A compound scaffold with uniform longitudinally oriented guidance cues and a porous sheath promotes peripheral nerve regeneration in vivo. Acta Biomaterialia. 68, 223-236 (2018).
- Brower, K., White, A. K., Fordyce, P. M. Multi-step Variable Height Photolithography for Valved Multilayer Microfluidic Devices. Jouranl of Visualized Experiments. (119), e55276(2017).
- Gupta, R., et al. Shear stress alters the expression of myelin-associated glycoprotein (MAG) and myelin basic protein (MBP) in Schwann cells. Journal of Orthopaedic Research : Official Publication of the Orthopaedic Research Society. 23, 1232-1239 (2005).
- Harris, G. M., Shazly, T., Jabbarzadeh, E. Deciphering the combinatorial roles of geometric, mechanical, and adhesion cues in regulation of cell spreading. PLoS One. 8 (11), (2013).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Schreck, I., et al. C-Jun localizes to the nucleus independent of its phosphorylation by and interaction with JNK and vice versa promotes nuclear accumulation of JNK. Biochemical and Biophysical Research Communications. 407, 735-740 (2011).
- Shen, K., Qi, J., Kam, L. C. Microcontact printing of proteins for cell biology. Journal Visualized Experiments. (22), e1065(2008).
- Treter, J., et al. Washing-resistant surfactant coated surface is able to inhibit pathogenic bacteria adhesion. Applied Surface Science. 303, 147-154 (2014).
- Lutz, J. F. Polymerization of oligo(ethylene glycol) (meth)acrylates: Toward new generations of smart biocompatible materials. Journal of Polymer Science Part A: Polymer Chemistry. 46 (11), 3459-3470 (2008).
- Marcus, M., et al. Interactions of Neurons with Physical Environments. Advanced Healthcare Materials. 6, (2017).
- Pu, J. Golgi polarization in a strong electric field. Journal of Cell Science. 118, 1117-1128 (2005).
- Blaker, J. J., et al. Bioactive Silk-Based Nerve Guidance Conduits for Augmenting Peripheral Nerve Repair. Advanced Healthcare Materials. 7, 1800308(2018).
- Daly, W., Yao, L., Zeugolis, D., Windebank, A., Pandit, A. A biomaterials approach to peripheral nerve regeneration : bridging the peripheral nerve gap and enhancing functional recovery. Journal of the Royal Society of Interface. 9 (67), 202-221 (2012).
- Xia, H., et al. Directed neurite growth of rat dorsal root ganglion neurons and increased colocalization with Schwann cells on aligned poly(methyl methacrylate) electrospun nanofibers. Brain Research. 1565, 18-27 (2014).
- Wang, H. B., Mullins, M. E., Cregg, J. M., McCarthy, C. W., Gilbert, R. J. Varying the diameter of aligned electrospun fibers alters neurite outgrowth and Schwann cell migration. Acta Biomaterialia. 6, 2970-2978 (2010).
- Carvalho, C. R., Oliveira, J. M., Reis, R. L. Modern Trends for Peripheral Nerve Repair and Regeneration: Beyond the Hollow Nerve Guidance Conduit. Frontiers in Bioengineering and Biotechnology. 7, 337(2019).
- Yang, Y., Wang, K., Gu, X., Leong, K. W. Biophysical Regulation of Cell Behavior - Cross Talk between Substrate Stiffness and Nanotopography. Engineering. 3, 36-54 (2017).
- Tan, J. L., Liu, W., Nelson, C. M., Raghavan, S., Chen, C. S. Simple Approach to Micropattern Cells on Common Culture Substrates by Tuning Substrate Wettability. Tissue Engineering. 10, 865-872 (2004).
- Grove, M., et al. YAP/TAZ initiate and maintain schwann cell myelination. Elife. 6, 1-27 (2017).
- Poitelon, Y., et al. YAP and TAZ control peripheral myelination and the expression of laminin receptors in Schwann cells. Nature Neuroscience. 19, 879-887 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados