このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
細胞および組織サンプルからのヒト乳がん幹細胞の単離と機能評価
要約
この実験プロトコルでは、乳がん細胞および組織サンプルからのBCSCの単離、ならびにBCSCの表現型と機能を評価するために使用できるin vitro および in vivo アッセイについて説明します。
要約
乳がん幹細胞(BCSC)は、遺伝性または後天性の幹細胞のような特徴を持つがん細胞です。頻度が低いにもかかわらず、乳がんの開始、再発、転移、治療抵抗性の主な原因です。乳がんを治療するための新しい治療標的を特定するためには、乳がん幹細胞の生物学を理解することが不可欠です。乳がん幹細胞は、CD44、CD24などのユニークな細胞表面マーカーの発現およびアルデヒドデヒドロゲナーゼ(ALDH)の酵素活性に基づいて単離および特徴付けられます。これらのALDH高CD44+CD24- 細胞はBCSC集団を構成し、下流の機能研究のために蛍光活性化セルソーティング(FACS)によって単離することができます。科学的問題に応じて、異なるインビ トロ および インビボ の方法を使用して、BCSCの機能的特性を評価することができます。ここでは、乳がん細胞の不均一な集団と乳がん患者から得られた原発腫瘍組織の両方からヒトBCSCを単離するための詳細な実験プロトコルを提供します。さらに、BCSC機能の評価に使用できるコロニー形成アッセイ、マンモスフェアアッセイ、3D培養モデル、腫瘍異種移植アッセイなど、下流のin vitro および in vivo 機能アッセイを強調しています。
概要
ヒト乳がん幹細胞(BCSC)の細胞的および分子的メカニズムを理解することは、乳がん治療で直面する課題に取り組むために重要です。BCSCの概念の出現は21世紀初頭にさかのぼり、CD44 + CD24-/低乳がん細胞の小さな集団がマウスで不均一な腫瘍を生成できることが判明しました1,2。その後、アルデヒドデヒドロゲナーゼの酵素活性が高い(ALDHhigh)ヒト乳がん細胞も同様の幹細胞様特性を示すことが観察されました3。これらのBCSCは、自己複製および分化が可能な細胞の小さな集団を表し、バルク腫瘍の不均一な性質に寄与する1、2、3。進化的に保存されたシグナル伝達経路の変化がBCSCの生存と維持を促進することを示唆する証拠の蓄積4,5,6,7,8,9,10,11,12,13,14 .さらに、細胞外因性微小環境は、異なるBCSC機能を指示する上で極めて重要な役割を果たすことが示されている15、16、17。これらの分子経路とBCSC機能を調節する外的要因は、乳がんの再発、転移18、および治療に対する耐性の発達に寄与しており19,20,21、治療後のBCSCの残存存在は、乳がん患者の全生存に大きな課題をもたらします22,23。.したがって、これらの因子の前臨床評価は、乳がん患者のより良い治療結果と全生存期間の改善を達成するために有益である可能性のあるBCSC標的療法を特定するために非常に重要です。
いくつかのインビトロヒト乳癌細胞株モデルおよびインビボヒト異種移植片モデルが、BCSCを特徴付けるために使用されています24、25、26、27、28、29。連続継代のたびに細胞株が継続的に再増殖する能力により、これらはオミクスベースおよび薬理ゲノム研究を実施するための理想的なモデルシステムになります。しかし、細胞株は、患者サンプルで観察された不均一性を再現できないことがよくあります。したがって、細胞株データを患者由来のサンプルで補完することが重要です。BCSCを最も純粋な形で単離することは、BCSCの詳細な特性評価を可能にするために重要です。 この純度を達成することは、BCSCに特異的な表現型マーカーの選択に依存します。 現在、ALDH高CD44 + CD24-細胞表現型は、最大純度の蛍光活性化細胞選別(FACS)を使用して、バルク乳がん細胞集団からヒトBCSCを区別および分離するために最も一般的に使用されています1、3,26。さらに、自己複製、増殖、分化などの単離されたBCSCの特性は、in vitroおよびin vivo技術を使用して評価できます。
例えば、インビトロコロニー形成アッセイは、異なる処理条件30の存在下で50個以上のコロニーを形成するために単一細胞が自己複製する能力を評価するために使用することができる。マンモスフェアアッセイは、足場非依存条件下での乳がん細胞の自己複製の可能性を評価するためにも使用できます。このアッセイは、無血清非接着培養条件における各連続継代において、単一細胞がスフェア(BCSCと非BCSCの混合物)として生成および増殖する能力を測定する31。さらに、3次元(3D)培養モデルを使用して、 in vivo 微小環境を厳密に再現し、潜在的なBCSC標的療法の活性の調査を可能にする細胞-細胞間および細胞-マトリックス相互作用を含むBCSC機能を評価できます32。in vitroモデルの多様なアプリケーションにもかかわらず、in vitroアッセイのみを使用して in vivo 条件の複雑さをモデル化することは困難です。この課題は、マウス異種移植片モデルを使用して in vivoでのBCSC挙動を評価することで克服できます。特に、このようなモデルは、乳癌転移33を評価し、疾患進行中の微小環境との相互作用を調査し34、 in vivo イメージング35、および抗腫瘍剤34の患者特異的毒性および有効性を予測するための理想的なシステムとして役立つ。
このプロトコルは、不均一な乳がん細胞のバルク集団から最大純度でヒトALDH高CD44 + CD24-BCSCを単離するための詳細な説明を提供します。また、3つのin vitro技術(コロニー形成アッセイ、マンモスフェアアッセイ、および3D培養モデル)と、BCSCのさまざまな機能を評価するために使用できるin vivo腫瘍異種移植アッセイの詳細な説明も提供します。これらの方法は、BCSC生物学の理解および/または新規BCSC標的療法の調査を目的として、ヒト乳がん細胞株または初代患者由来の乳がん細胞および腫瘍組織からのBCSCの単離および特性評価に関心のある研究者による使用に適しています。
プロトコル
同意した乳がん患者から直接患者由来の外科的または生検サンプルを収集することは、施設倫理委員会によって承認された承認された人間の倫理プロトコルの下で実施されました。患者由来の異種移植片モデルを生成するために使用されたすべてのマウスは、施設が承認した動物施設で維持および収容された。マウスを用いた患者由来の異種移植片モデルからの腫瘍組織は、施設動物管理委員会によって承認された承認された倫理プロトコルに従って生成された。
1. 細胞株の作製
- バイオセーフティキャビネット内の無菌条件下ですべての細胞培養および染色手順を実行します。滅菌済みの細胞培養皿/フラスコおよび試薬を使用してください。
- ヒト乳がん細胞を37°Cに維持し、ウシ胎児血清(FBS)および各細胞株に特異的に必要な成長因子を添加した定義培地に5%CO2 を入れます。
- マウスNIH3T3線維芽細胞培養物(コロニー形成アッセイに使用)を、10%FBSを添加したダルベッコ改変イーグル培地(DMEM)中の5%CO2 を用いて37°Cに維持します。
- すべての文化について、2〜3日ごとに古いメディアに新しいメディアを補充します。培養物が75〜80%のコンフルエントに達したら、複数の滅菌細胞培養フラスコに継代培養する。
2.乳がん腫瘍組織の作製
- 施設倫理委員会によって承認された人間の倫理プロトコルの下で、同意した乳がん患者から直接患者由来の外科的または生検サンプルを収集します。
- 続いて、施設動物管理委員会によって承認された動物倫理プロトコルの下で、マウスを使用して患者由来の異種移植片モデルから腫瘍組織を収集して生成します。
- 30 mL DMEM:F12培地を含む50 mL滅菌コニカルチューブに無菌条件下ですべての腫瘍組織を採取し、氷上に保ち、収集から2時間以内に以下に説明するようにサンプルを処理します。
3. 乳癌細胞の単一細胞懸濁液の生成
- 60〜80%コンフルエントである乳癌細胞の単層(選択細胞株)を含むフラスコからの吸引培地。1xリン酸緩衝生理食塩水(PBS)で細胞を洗浄します。PBSを吸引し、適切な細胞解離溶液(例えば、トリプシン:EDTA;細胞の単層を覆うのにちょうど十分)を加え、室温(推奨)または37°Cで5分間インキュベートします。
- 5 mLの培地を添加して、細胞解離溶液の活性を中和します。
- 得られた解離した細胞溶液を50 mLのコニカルチューブに移し、1000 x g で5分間遠心分離します。
- 上清を捨て、細胞ペレットを5 mLの1x PBSに再懸濁しました。血球計算盤と顕微鏡を使用して細胞を数えます。
注:血球計算盤で細胞の凝集を観察します。単一細胞懸濁液が形成されていない場合は細胞解離工程を繰り返す。 - 細胞計数後、細胞懸濁液を1000 x g で5分間再遠心分離し、上清を廃棄し、細胞ペレットを1 x 106 細胞/mLの濃度のALDH基質バッファーに再懸濁します。
4. 組織サンプルからのシングルセル懸濁液の生成
- 十字法を使用して外科用ブレードで腫瘍組織を細かく刻み、サイズが約1mmの小さな断片を取得します。組織片を、10 mLの解離バッファー(DMEM:F12の1Xコラゲナーゼ)を含む新しい50 mLコニカルチューブに移します。コニカルチューブをパラフィルムで密封し、シェーカーインキュベーター内で37°Cで40分間インキュベートします。
注意: シェーカーインキュベーターがない場合は、チューブを37°Cの水浴に入れ、5〜10分ごとにボルテックスしてチューブを混合します。 - サンプルを530 x g で5分間遠心分離することにより、消化された組織をペレット化します。上清を捨て、5mLのトリプシンを加える。1 mLピペット(750 μLマークに設定)を使用してピペットを上下にピペットし、ペレットを破壊し、37°Cの水浴中で5分間インキュベートします。インキュベーション後、ピペットで上下に激しく動かして単一細胞を放出します。
- DMEM:F12培地でチューブ内の全容量を25 mLに補充し、1000 x g で5分間遠心分離します。上清を捨て、ペレットを1 mLのディスパーゼ-DNase溶液に再懸濁します。37°Cの水浴中で5分間インキュベートします。
- チューブ内の総容量をPBSで10 mLまで補充します。上下にピペッティングして混合し、得られた細胞懸濁液を新しい50 mLコニカルチューブに取り付けられた40 μmセルストレーナーに通します。1000 x g で5分間遠心分離します。
- 上清を捨て、細胞ペレットを5 mLの1x PBSに再懸濁します。細胞をカウントし、ステップ3.4および3.5に記載されるように細胞懸濁液の調製を完了する。
5.乳がん幹細胞(BCSC)の分離
- 非染色コントロール用のラベルフローチューブ、シングルセル染色コントロール(DEABコントロール、ALDH、CD44-PE、CD24-PE-Cy7、7AAD)、ネガティブコントロールチューブ(DEAB、CD44-PE、CD24-PE-Cy7および7AADで染色)、蛍光マイナス1(FMO)コントロール、および「ソート」チューブ(ALDH、CD44、CD24および7AADで染色)。
- ステップ3.5またはステップ4.5の細胞懸濁液500 μL(0.5 x 106 細胞)を、細胞のみ、CD44、CD24、および7AADとラベル付けされた各チューブに移します。使用するまでチューブを氷の上に置きます。
- 2 mLのサンプル(2 x 106 セル)をそれぞれの「ALDH」チューブに移します。5 μLのDEABを「DEABコントロール」および「ネガティブコントロール」チューブに加え、しっかりと蓋をします。10 μLのALDH基質を「ALDH」チューブに加え、ボルテックスでよく混合し、すぐに500 μLを対応する「DEABコントロール」および「ネガティブコントロール」チューブに移します。「DEABコントロール」、「ネガティブコントロール」、および「ALDHチューブ」を要約し、37°Cで30〜60分間インキュベートします(60分を超えないでください)。
注:最適なインキュベーション時間は、細胞株によっては最適化が必要な場合があります。ALDH基板と染色された細胞を含むチューブを常に光から保護してください。 - インキュベーション後、すべてのサンプルを250 x gで5分間遠心分離します。細胞を500 μLのALDH基質バッファーに再懸濁します。メーカー推奨またはユーザー最適化濃度の抗CD44-PEおよび抗CD24-PE-Cy7抗体カクテルを加え、4°Cで30分間インキュベートします。抗CD44-PEおよび抗CD24-PE-Cy7抗体をそれぞれの「CD44」および「CD24」標識チューブに追加します。
- インキュベーション後、すべてのサンプルを250 x g で5分間遠心分離します。細胞を500 μLのALDH基質バッファーに再懸濁します。「ネガティブコントロール」チューブ、「ソートチューブ」、および「7ADD」チューブを7AAD(推奨濃度:0.25 μg/1 x 106 細胞)で氷上で10分間インキュベートします。
注:ALDH活性は緑色の蛍光チャネルで検出されるため、互換性のある発光スペクトルが異なる蛍光色素を使用する必要があります。マルチパラメーターフローサイトメトリー中にスペクトルのオーバーラップが観察される場合は、蛍光色素間の補正を可能にするためのガイドとして、蛍光シグナルの他のチャンネルへのこぼれを最小限に抑える必要があります。 - サンプル分析の準備として、FACS機器の分析プロトコルを設定します。散布図を作成します(前方散乱対側散乱、前方散乱対蛍光チャネル)。
- 無染色コントロールを使用して、光電子増倍管を調整して全細胞集団から破片を分離し、蛍光電圧を調整して、細胞集団全体を最初の対数スケール(101)の周りに移動します。DEABコントロールを使用して、緑色の蛍光電圧チャネルを調整することにより、細胞集団全体を第2の対数スケール(102)内に移動します。
- 最初にすべての単一染色コントロール(ALDH、CD44-PE、CD24-PE-Cy7)および7AADおよびFMOコントロールを分析し、電圧を調整して染色されていない細胞から染色細胞を分離し、他のチャネルへの蛍光シグナルのこぼれを最小限に抑えます。
- 染色された各細胞サンプルの陽性集団のゲート。陰性対照管を用いて、生存率(7AAD陰性)のためのゲート、ALDH低 およびALDH高 細胞集団( 図1Bに示す代表的なゲーティング戦略)。
- 目的のマルチパラメータ染色サンプルを分析して、BCSCを単離します。 生存可能なALDH低ゲートおよびALDH高ゲートを使用して、CD44+CD24-(BCSC)およびCD44-CD24+(非BCSC)細胞集団をそれぞれ選択します(図1B)。
- 滅菌収集チューブ( 図2A&Bに示す2つの代表的な細胞株からの集団)の収集培地で生存可能なBCSCおよび非BCSCを収集する。以下に説明するように、下流のin vitro および in vivo アッセイに選別された細胞を使用してください。
注:下記のインビ トロ および インビボ アッセイに加えて、BCSCは、標準的なイムノブロッティング技術を介してSOX2、OCT4、NANOGなどの多能性マーカーの発現を測定することによって検証できます。
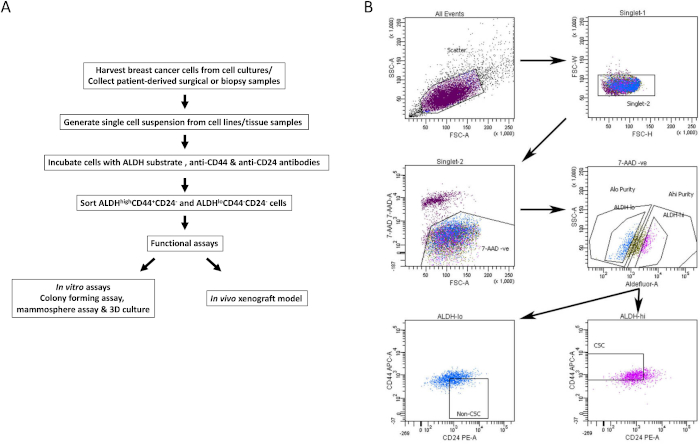
図1:乳がん細胞株および組織サンプルからBCSCを単離するためのFACSゲーティング戦略。 (A)BCSC分離の手順を説明するフローチャート。(B)不均一な細胞プールから生存可能なBCSCおよび非BCSCを単離するために使用されるソート戦略を示す代表的なFACSプロット。MDA-MB-231ヒト乳癌細胞は、7-AAD、CD44-APC、CD24-PEおよびALDH基質と同時に標識される。細胞サブセットは、FACSマシン上で4色プロトコルを使用して分離されました。細胞は、予想される光散乱に基づいて選択され、次にシングレットについて、および生存率は7-AAD除外に基づいて選択されます。次に、細胞をALDH活性について分析し、最も陽性の高い上位20%をALDH高集団として選択し、ALDH活性が最も低い下位20%の細胞をALDH低集団と見なした。最後に、ALDH低細胞の50%がCD44低/-CD24+表現型に基づいてさらに選択され、ALDH高細胞の50%がCD44+CD24-表現型に基づいて選択される。この図はChuら17から改作されたものである。この図の拡大版を表示するには、ここをクリックしてください。
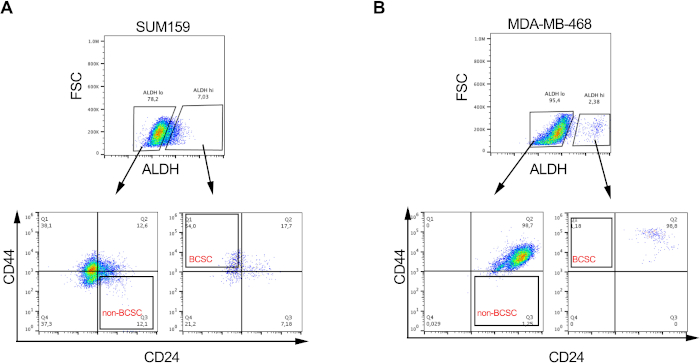
図2:BCSCの割合は、乳がん細胞株によって変動します。 図1に記載されるように標識および選別に続く(A)SUM159および(B)MDA−MD−468トリプルネガティブ乳癌細胞株におけるBCSCsおよび非BCSCsの差別割合を示す代表的な画像。この図の拡大版を表示するには、ここをクリックしてください。
6. コロニー形成アッセイ
- 目的の細胞(ステップ5.11のソート済みセル、またはステップ3.5または4.5のソートされていないセル)を完全な培地に再懸濁します。
- 1 x 10 2、2 x 10 2、および5 x 102セルの3つのフローチューブにラベルを付けます。2 mLの完全培地を加え、適切な細胞番号(ステップ5.11でソートされた細胞、またはステップ3.5または4.5でソートされていない細胞)をそれぞれのチューブに移します。細胞溶液を5回上下にピペッティングして完全に混合します。
- 細胞を6ウェルプレートにプレートし、プレートを穏やかに回転させて細胞懸濁液を分配し、細胞の均一な分布を得ます。
- コロニーが現れるまで、プレートを37°C、5%CO2 インキュベーター内でインキュベートする(ここで、コロニー=コロニー当たり≥50細胞)。コロニー形成を妨げることなく、週に2回慎重に培地を補充してください。
- 培地を吸引し、1 mL PBSで1回洗浄します。0.5 mLの0.05%クリスタルバイオレット溶液を各ウェルに加え、プレートを30分間インキュベートします。2mLの水で洗って余分なクリスタルバイオレットの汚れを取り除きます。背景の汚れが除去されるまで、洗浄手順を繰り返します。
- 4倍および10倍の倍率で顕微鏡を用いて、生成されたコロニーの総数をカウントおよび記録する( 図3Aに示す代表的な画像)。
- コロニー形成の頻度を次のように計算します:頻度(%)=(形成されたコロニーの#/播種された細胞数)x 100。たとえば、1 x 102 個の細胞から25個のコロニーが生成される場合、コロニー形成の頻度は、頻度=(25/100)x100 = 25%です。
- あるいは、ステップ6.1から6.4を、必要な成長因子と生存因子の産生を通じてBCSCの微小環境サポートを提供する線維芽細胞との共培養を含む代替方法に置き換えます。
- I型ウシコラーゲン(3 mg/mLコラーゲンの30分の1希釈)を含むプレコート細胞60 mm培養皿。コラーゲンを37°Cのインキュベーターで30分間重合させます。未重合コラーゲンを吸引し、1x PBSでプレートを2回洗浄します。コラーゲンコーティングプレートを1 mLのPBSで覆い、使用するまで室温で放置します。
- 1 x 10 3、5 x 10 3、および1 x 104セルの3つのフローチューブにラベルを付けます。4 mLのコロニー形成アッセイ培地を加え、適切な数の細胞(ステップ5.11から選別された細胞、またはステップ3.5または4.5から選別されていない細胞)をそれぞれのチューブに移します。照射したマウスNIH3T3線維芽細胞(4 x 104細胞/mL培地)を追加します。細胞溶液を5回上下にピペッティングして完全に混合します。
- ステップ6.1のコラーゲンコーティング培養皿からPBSを吸引し、ステップ6.3に記載されているように、細胞混合物を各細胞培養プレートにプレートします。
- プレートを37°C、5%CO2 インキュベーターでインキュベートし、培地を補充せずに、7〜10日間、またはコロニーが形成されるまで放置します。ステップ6.6および6.7の説明に従って、生成されたコロニーの総数をカウントして記録します。
7.マンモスフェアアッセイ
- 目的の細胞(ステップ5.11の選別された細胞またはステップ3.5または4.5の選別されていない細胞)を、96ウェル超低接着細胞培養プレートに5 x 102 細胞/cm2 の領域を播種密度で完全なマンモスフェア培地およびプレート細胞に再懸濁します。
注:細胞播種密度は、異なる細胞株に対して最適化する必要があります。 - 培養プレートを5%CO2を含む37°Cのインキュベーター内で5〜10日間インキュベートします。マンモスフェアの形成を妨げることなく、週に2回慎重にメディアを補充してください。
- インキュベーション後、顕微鏡を使用して各ウェルで生成されたマンモスフェアの数を数えます。ここで、マンモスフェアは、直径100μmを超える乳がん細胞クラスターとして定義されます( 図3Bに示す代表的な画像)。
- マンモスフェア形成効率(MFE)を次のように計算します:MFE(%)=(ウェルあたりのマンモスフェアの数)/(ウェルあたりに播種された細胞数)x 100(つまり、ウェル内の1 x 102 細胞によって5つのマンモスフェアが生成される場合、MFE =(5/100)x 100 = 5%)。
- マンモスフィアを継代培養するには、マンモスフェアの内容物を含む培地を新しい50 mLコニカルチューブと遠心分離機培地に1000 x g で5分間慎重に移します。上清を注意深く除去し、細胞ペレットを500 μLのトリプシンに再懸濁し、室温で5分間インキュベートします。
- 上清を廃棄し、ペレットを1 mLの完全なマンモスフェア培地に再懸濁します。血球計算盤を使用して細胞をカウントし、ステップ7.1に記載されているように、超低付着細胞培養プレートで細胞を再プレートします。
注:継代培養に加えて、マンモスフェア由来の細胞をFACSによってさらに分析して、BCSC表現型を評価したり、他のダウンストリームアッセイ用のBCSCの純粋な集団を取得したりすることもできます。 - 細胞集団に含まれるマンモスフェア開始細胞の数を決定するには、球体限界希釈分析(SLDA)を含む別の方法を使用します。プレート細胞は、96ウェル超低接着細胞培養プレートで高細胞数から低細胞数の段階希釈を行い、最も高い希釈率で1ウェルあたり1細胞未満になります。
- 培養プレートを5%CO2 を含む37°Cのインキュベーターで10〜14日間インキュベートし、細胞の凝集を避けるために邪魔しないように放置します。
- インキュベーション後、顕微鏡を使用して各ウェルで生成されたマンモスフェアの数を数えます。ここで、マンモスフェアは、直径100μmを超える乳がん細胞クラスターとして定義されます。極限希釈分析(ELDA)オンラインソフトウェア(http://bioinf.wehi.edu.au/software/elda/)を使用して、球開始頻度と有意性を計算します。
8.3D培養モデル
- 実験の質問に応じて、成長因子の有無にかかわらず基底膜抽出物(BME)を使用します(減少)。癌細胞に対する個々の成長因子の効果を評価するために、BMEを低下させた成長因子を使用する。また、BMEに存在する内因性成長因子の非特異的影響を最小限に抑えるのにも役立ちます。
注:BMEは10°C以上で固化します。 解凍ステップの間でさえ、常にBMEを氷の上に置いてください。 - 気泡を発生させずに96ウェルプレートにウェルあたり50 μLのBMEを慎重に添加し、37°Cで1時間重合させます。10分間のインキュベーション後、ゲル層の乾燥を避けるために100 μL PBSを追加します。
- ステップ5.11の選別された細胞、またはステップ3.5または4.5の選別されていない細胞を、5 x 103 〜5 x 104/200 μLの濃度で3D培地に再懸濁します。
- BMEが重合したら、PBSを除去し、200 μLの細胞懸濁液を各ウェルに加え、5%CO2を含む37°Cのインキュベーターでインキュベートします。培地の蒸発を防ぐために、周囲のウェルにPBSを追加します。
注:実験の設定前に、めっきに最適な細胞数を決定する必要があります。実験の質問に応じて、BCSCは単独で、または他の細胞タイプ(線維芽細胞/内皮/免疫細胞など)と一緒に培養できます。 - 週に2回、培養プレートに新鮮な培地を追加します。オルガノイドの形成を分析する前に、培養物を10〜14日間維持します( 図3Cに示す代表的な画像)。
- 継代培養の場合は、培地を注意深く吸引し、細胞を含む各ウェルに200 μLのディスパーゼを加えます。プレートを37°Cのインキュベーターで1時間インキュベートします。インキュベーション期間の途中(30分)でプレートを取り出し、ディスパーゼ溶液を5回静かにピペットで上下させ、さらに30分間インキュベーターに戻します。
- 1時間後、解離した細胞溶液をフローチューブに移す。2%FBS(fPBS)を含む1x PBSでウェルを洗浄し、フローチューブに移します。チューブを1000 x g で5分間遠心分離します。上清を注意深く吸引し、500 μLのトリプシンを加え、37°Cで5分間インキュベートします。等量のfPBSを添加してトリプシンを不活性化し、1000 x g で5分間遠心分離します。
- 上清を廃棄し、ペレットを1 mLの3D培地に再懸濁します。細胞をカウントし、ステップ8.2〜8.4のようにBME内の必要な数のセルを再プレートします。
注:複数のウェルをプールして、目的の細胞集団をさらに分析または分類することができます。
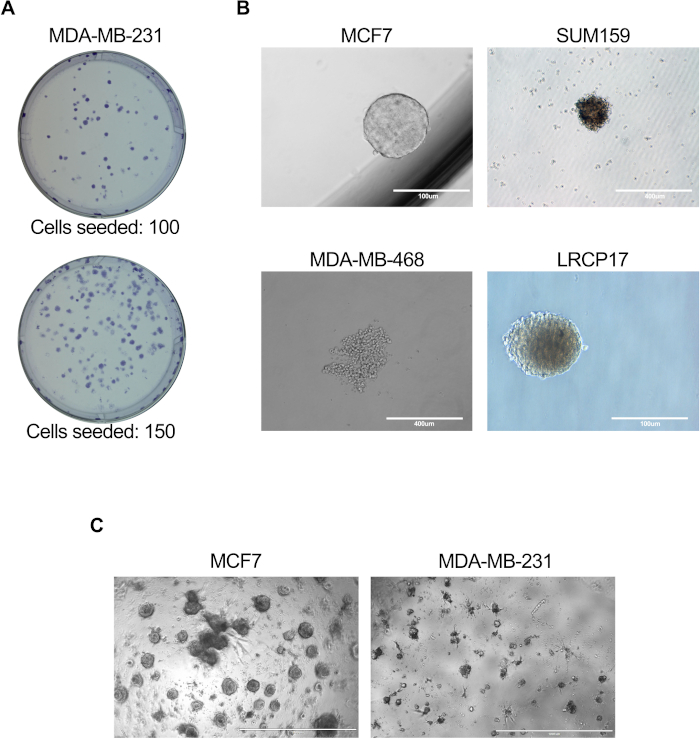
図3:BCSC細胞機能を評価するための インビトロ アッセイ。インビトロ アッセイは、プロトコルセクション6.1〜6.5(A)、7.1〜7.4(B)、または81に記載のとおりに実施した。を 8.4 + 8.6 (C) に設定します。(a)MDA-MB-231ヒト乳癌細胞により生成されたコロニーを示す代表的な画像;(B)MCF7、SUM159、またはMDA-MB-468ヒト細胞株および患者由来LRCP17乳癌細胞によるマンモスフェア形成を示す代表的な画像。(C)3D培養モデルにおけるMCF7およびMDA-MB-231乳癌細胞によって形成された3D構造を示す代表的な画像。 この図の拡大版を表示するには、ここをクリックしてください。
注:施設動物管理委員会によって承認された動物倫理プロトコルに基づいて動物実験を実施してください。
9. 生体内 異種移植モデル
- 乳がん幹細胞の腫瘍開始能力を決定するために、限界希釈アプローチを使用して細胞(ステップ5.7からの選別集団またはステップ3.5または4.5からの選別されていない集団)を調製する。1〜5つの異なる希釈グループを使用してPBSで細胞を連続希釈し、0.01〜0.2 x10 2 細胞/ 100 μLから1 x 106 細胞/ 100 μLの用量で連続的に希釈します。
注:ソートされていない/全体の母集団セルをコントロールとして使用できます。使用する希釈グループの数は、望ましい科学的結果によって異なります(たとえば、腫瘍原性のみを検査する場合は、より高い細胞数の1つのグループを使用できますが、腫瘍開始能を計算する場合は、5つの限界希釈用量をテストするのが最適です)。 - ヒト乳がん細胞から異種移植片モデルを生成するには、免疫不全の雌マウス(胸腺ヌード[nu/nu]、非肥満糖尿病/重症複合免疫不全[NOD/SCID]またはNOD/SCID IL2γ[NGS]株)を使用します。
注:グループごとに最低4匹の動物を使用できますが、特に限界希釈分析で堅牢な結果を得るには、グループあたり8〜12匹の動物をお勧めします。 - バイオセーフティキャビネット内の無菌条件下で、各細胞調製物100 μL/マウスを使用して標準的な乳脂肪パッド(MFP)注射を実行します。
注:最適な乳房腫瘍の成長と遠隔臓器への自発的転移のために、胸部MFPが推奨されます。あるいは、鼠径MFPも使用することができる。 - 注射後、注射部位の一般的な健康状態と腫瘍の成長について、マウスを毎日監視します。触知可能な腫瘍が検出されたら、2つの垂直寸法のノギスによる腫瘍サイズの測定を開始し、エンドポイントまで毎週記録します。
注:実験のエンドポイントは、制度的動物倫理プロトコルに定められた規制に基づいて決定されます。典型的には、安楽死によるエンドポイントは、通常、腫瘍体積が1500mm3に達すると必要とされる。BCSC集団および/またはより高い細胞用量(例:. >1 x 104細胞)、このエンドポイントはMFP注射から4〜8週間以内に到達する可能性があります。非常に低い細胞用量および/または非BCSC細胞集団の場合、腫瘍増殖は注射後最大8か月間進行させる必要があります。. - これらの測定値から、次の式を使用して腫瘍体積を計算します:mm3 単位の体積= 0.52 x(幅)2 x長さ。限界希釈アプローチを使用する場合は、ELDAオンラインソフトウェアを使用して腫瘍開始能力と有意性を計算します(http://bioinf.wehi.edu.au/software/elda/)。
- あるいは、エンドポイントを人道的に延長するために、原発腫瘍を外科的に切除し、マウスの健康および/または遠隔臓器における自然転移の発症を監視し続けます。連続異種移植の生成のために切除された腫瘍組織を使用してください。
- エンドポイントでは、原発腫瘍および遠隔臓器(リンパ節、肺、肝臓、脳、骨)から組織を採取し、組織病理学的および/または免疫組織化学的分析を実施するか、腫瘍組織を解離して、セクション6〜8に記載されているin vitroアッセイで使用します。
結果
記載されたプロトコルは、細胞株または解離した腫瘍組織のいずれかからの乳癌細胞の不均一な集団からのヒトBCSCの単離を可能にする。特定の細胞株または組織サンプルについて、汚染された非BCSC集団が変動する可能性があるため、BCSCを最大純度で単離するための均一な単一細胞懸濁液を生成することが重要です。 厳格な選別戦略を適用すると、汚染された非BCSCの存在が最小限に抑えら?...
ディスカッション
乳がんの転移と治療に対する抵抗性は、世界中の女性の死亡の主な原因となっています。乳がん幹細胞(BCSC)の亜集団の存在は、転移の増強に寄与する26、43、44、45、46および治療抵抗性21、47、48に寄与する。
開示事項
著者は開示するものは何もありません。
謝辞
研究室のメンバーの有益な議論とサポートに感謝します。乳がん幹細胞と腫瘍微小環境に関する私たちの研究は、カナダがん研究協会研究所および米国陸軍国防総省乳がんプログラム(Grant # BC160912)からの助成金によって資金提供されています。V.B.はウエスタン・ポスドク・フェローシップ(ウエスタン大学)の支援を受けており、A.L.A.とV.B.はどちらもカナダ乳がん協会の支援を受けています。CLは、カナダ政府からのバニエカナダ大学院奨学金によってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| 7-Aminoactinomycin D (7AAD) | BD | 51-68981E | suggested: 0.25 µg/1x106 cells |
| Acetone | Fisher | A18-1 | |
| Aldehyde dehydrogenase (ALDH) substrate | Stemcell Technologies | 1700 | Sold commerically as part of the ALDEFLOUR Assay kit; follow manufacturer's instructions for ALDH substrate preparation |
| Basement membrane extract (BME) | Corning | 354234 | Sold under the commercial name Matrigel |
| Cell culture plates: 6 well | Corning | 877218 | |
| Cell culture plates: 60mm | Corning | 353002 | |
| Cell culture plates: 96-well ultra low attachment | Corning | 3474 | |
| Cell strainer: 40 micron | BD | 352340 | |
| Collagen | Stemcell Technologies | 7001 | Prepare 1:30 dilution of 3 mg/mL collagen in PBS |
| Collagenase | Sigma | 11088807001 | 1x |
| Conical tubes: 50 mL | Fisher scientific | 05-539-7 | |
| Crystal violet | Sigma | C6158 | Use 0.05% crystal violet solution in water for staining |
| Dispase | Stemcell Technologies | 7913 | 5U/mL |
| DMEM:F12 | Gibco | 11330-032 | 1x, With L-glutamine and 15 mM HEPES |
| DNAse | Sigma | D5052 | 0.1 mg/mL final concentration |
| FBS | Avantor Seradigm Lifescience | 97068-085 | |
| Flow tubes: 5ml | BD | 352063 | Polypropylene round-bottom tubes |
| Methanol | Fisher | 84124 | |
| mouse anti-Human CD24 antibody | BD | 561646 | R-phycoerythrin and Cyanine dye conjugated Clone: ML5 |
| mouse anti-Human CD44 antibody | BD | 555479 | R-phycoerythrin conjugated, Clone: G44-26 |
| N,N-diethylaminobenzaldehyde (DEAB) | Stemcell Technologies | 1700 | Sold commerically as part of the ALDEFLOUR Assay kit; follow manufacturer's instructions DEAB preparation |
| PBS | Wisent Inc | 311-425-CL | 1x, Without calcium and magnesium |
| Trypsin-EDTA | Gibco | 25200-056 | |
| Mammosphere Media Composition | |||
| B27 | Gibco | 17504-44 | 1x |
| bFGF | Sigma | F2006 | 10 ng/mL |
| BSA | Bioshop | ALB003 | 04% |
| DMEM:F12 | Gibco | 11330-032 | 1x, With L-glutamine and 15 mM HEPES |
| EGF | Sigma | E9644 | 20 ng/mL |
| Insulin | Sigma | 16634 | 5 µg/mL |
| 3D Organoid Media Composition | |||
| A8301 | Tocris | 2939 | 500 nM |
| B27 | Gibco | 17504-44 | 1x |
| DMEM:F12 | Gibco | 11330-032 | 1x, With L-glutamine and 15 mM HEPES |
| EGF | Sigma | E9644 | 5 ng/mL |
| FGF10 | Peprotech | 100-26 | 20 ng/mL |
| FGF7 | Peprotech | 100-19 | 5 ng/mL |
| GlutaMax | Invitrogen | 35050-061 | 1x |
| HEPES | Gibco | 15630-080 | 10 mM |
| N-acetylcysteine | Sigma | A9165 | 1.25 mM |
| Neuregulin β1 | Peprotech | 100-03 | 5 nM |
| Nicotinamide | Sigma | N0636 | 5 mM |
| Noggin | Peprotech | 120-10C | 100 ng/mL |
| R-spondin3 | R&D | 3500 | 250 ng/mL |
| SB202190 | Sigma | S7067 | 500 nM |
| Y-27632 | Tocris | 1254 | 5 µM |
参考文献
- Al-Hajj, M., Wicha, M. S., Benito-Hernandez, A., Morrison, S. J., Clarke, M. F. Prospective identification of tumorigenic breast cancer cells. Proceedings of the National Academy of Sciences of the United States of America. 100 (7), 3983-3988 (2003).
- Shipitsin, M., et al. Molecular definition of breast tumor heterogeneity. Cancer Cell. 11 (3), 259-273 (2007).
- Ginestier, C., et al. ALDH1 is a marker of normal and malignant human mammary stem cells and a predictor of poor clinical outcome. Cell Stem Cell. 1 (5), 555-567 (2007).
- Sulaiman, A., et al. Dual inhibition of Wnt and Yes-associated protein signaling retards the growth of triple-negative breast cancer in both mesenchymal and epithelial states. Molecular Oncology. 12 (4), 423-440 (2018).
- Debeb, B. G., et al. Histone deacetylase inhibitors stimulate dedifferentiation of human breast cancer cells through WNT/β-catenin signaling. Stem Cells. 30 (11), 2366-2377 (2012).
- Klutzny, S., et al. PDE5 inhibition eliminates cancer stem cells via induction of PKA signaling. Cell Death & Disease. 9 (2), 192 (2018).
- DiMeo, T. A., et al. A novel lung metastasis signature links Wnt signaling with cancer cell self-renewal and epithelial-mesenchymal transition in basal-like breast cancer. Cancer Research. 69 (13), 5364-5373 (2009).
- Liu, C. C., Prior, J., Piwnica-Worms, D., Bu, G. LRP6 overexpression defines a class of breast cancer subtype and is a target for therapy. Proceedings of the National Academy of Sciences of the United States of America. 107 (11), 5136-5141 (2010).
- Miller-Kleinhenz, J., et al. Dual-targeting Wnt and uPA receptors using peptide conjugated ultra-small nanoparticle drug carriers inhibited cancer stem-cell phenotype in chemo-resistant breast cancer. Biomaterials. 152, 47-62 (2018).
- Mamaeva, V., et al. Inhibiting Notch Activity in Breast Cancer Stem Cells by Glucose Functionalized Nanoparticles Carrying γ-secretase Inhibitors. Molecular Therapy. 24 (5), 926-936 (2016).
- Ithimakin, S., et al. HER2 drives luminal breast cancer stem cells in the absence of HER2 amplification: implications for efficacy of adjuvant trastuzumab. Cancer Research. 73 (5), 1635-1646 (2013).
- Koike, Y., et al. Anti-cell growth and anti-cancer stem cell activities of the non-canonical hedgehog inhibitor GANT61 in triple-negative breast cancer cells. Breast Cancer. 24 (5), 683-693 (2017).
- Sun, Y., et al. Estrogen promotes stemness and invasiveness of ER-positive breast cancer cells through Gli1 activation. Molecular Cancer. 13, 137 (2014).
- Colavito, S. A., Zou, M. R., Yan, Q., Nguyen, D. X., Stern, D. F. Significance of glioma-associated oncogene homolog 1 (GLI1) expression in claudin-low breast cancer and crosstalk with the nuclear factor kappa-light-chain-enhancer of activated B cells (NFκB) pathway. Breast Cancer Research. 16 (5), 444 (2014).
- Bhat, V., Allan, A. L., Raouf, A. Role of the Microenvironment in Regulating Normal and Cancer Stem Cell Activity: Implications for Breast Cancer Progression and Therapy Response. Cancers. 11 (9), (2019).
- Pio, G. M., Xia, Y., Piaseczny, M. M., Chu, J. E., Allan, A. L. Soluble bone-derived osteopontin promotes migration and stem-like behavior of breast cancer cells. PloS One. 12 (5), 0177640 (2017).
- Chu, J. E., et al. Lung-derived factors mediate breast cancer cell migration through CD44 receptor-ligand interactions in a novel ex vivo system for analysis of organ-specific soluble proteins. Neoplasia. 16 (2), 180-191 (2014).
- McGowan, P. M., et al. Notch1 inhibition alters the CD44hi/CD24lo population and reduces the formation of brain metastases from breast cancer. Molecular Cancer Research. 9 (7), 834-844 (2011).
- Mao, J., et al. ShRNA targeting Notch1 sensitizes breast cancer stem cell to paclitaxel. International Journal of Biochemistry and Cell Biology. 45 (6), 1064-1073 (2013).
- Duru, N., et al. HER2-associated radioresistance of breast cancer stem cells isolated from HER2-negative breast cancer cells. Clinical Cancer Research. 18 (24), 6634-6647 (2012).
- Croker, A. K., Allan, A. L. Inhibition of aldehyde dehydrogenase (ALDH) activity reduces chemotherapy and radiation resistance of stem-like ALDHhiCD44+ human breast cancer cells. Breast Cancer Research and Treatment. 133 (1), 75-87 (2012).
- Creighton, C. J., et al. Residual breast cancers after conventional therapy display mesenchymal as well as tumor-initiating features. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13820-13825 (2009).
- Calcagno, A. M., et al. Prolonged drug selection of breast cancer cells and enrichment of cancer stem cell characteristics. Journal of the National Cancer Institute. 102 (21), 1637-1652 (2010).
- Feng, Y., et al. Breast cancer development and progression: Risk factors, cancer stem cells, signaling pathways, genomics, and molecular pathogenesis. Genes Dis. 5 (2), 77-106 (2018).
- Samanta, D., Gilkes, D. M., Chaturvedi, P., Xiang, L., Semenza, G. L. Hypoxia-inducible factors are required for chemotherapy resistance of breast cancer stem cells. Proceedings of the National Academy of Sciences of the United States of America. 111 (50), 5429-5438 (2014).
- Croker, A. K., et al. High aldehyde dehydrogenase and expression of cancer stem cell markers selects for breast cancer cells with enhanced malignant and metastatic ability. Journal of Cellular and Molecular Medicine. 13 (8), 2236-2252 (2009).
- Morel, A. P., et al. Generation of breast cancer stem cells through epithelial-mesenchymal transition. PloS One. 3 (8), 2888 (2008).
- Muntimadugu, E., Kumar, R., Saladi, S., Rafeeqi, T. A., Khan, W. CD44 targeted chemotherapy for co-eradication of breast cancer stem cells and cancer cells using polymeric nanoparticles of salinomycin and paclitaxel. Colloids Surf B Biointerfaces. 143, 532-546 (2016).
- Liu, S., et al. Breast cancer stem cells transition between epithelial and mesenchymal states reflective of their normal counterparts. Stem Cell Reports. 2 (1), 78-91 (2014).
- Munshi, A., Hobbs, M., Meyn, R. E. Clonogenic cell survival assay. Methods in Molecular Medicine. 110, 21-28 (2005).
- Shaw, F. L., et al. A detailed mammosphere assay protocol for the quantification of breast stem cell activity. Journal of Mammary Gland Biology and Neoplasia. 17 (2), 111-117 (2012).
- Shin, C. S., Kwak, B., Han, B., Park, K. Development of an in vitro 3D tumor model to study therapeutic efficiency of an anticancer drug. Molecular Pharmaceutics. 10 (6), 2167-2175 (2013).
- Khanna, C., Hunter, K. Modeling metastasis in vivo. Carcinogenesis. 26 (3), 513-523 (2005).
- Cheon, D. J., Orsulic, S. Mouse models of cancer. Annual Review of Pathology. 6, 95-119 (2011).
- Lyons, S. K. Advances in imaging mouse tumour models in vivo. Journal of Pathology. 205 (2), 194-205 (2005).
- Margaryan, N. V., et al. The Stem Cell Phenotype of Aggressive Breast Cancer Cells. Cancers. 11 (3), (2019).
- Ma, F., et al. Enriched CD44(+)/CD24(-) population drives the aggressive phenotypes presented in triple-negative breast cancer (TNBC). Cancer Letters. 353 (2), 153-159 (2014).
- Chatterjee, S., et al. Paracrine Crosstalk between Fibroblasts and ER(+) Breast Cancer Cells Creates an IL1β-Enriched Niche that Promotes Tumor Growth. iScience. 19, 388-401 (2019).
- Phan-Lai, V., et al. Three-dimensional scaffolds to evaluate tumor associated fibroblast-mediated suppression of breast tumor specific T cells. Biomacromolecules. 14 (5), 1330-1337 (2013).
- O'Brien, C. A., Kreso, A., Jamieson, C. H. Cancer stem cells and self-renewal. Clinical Cancer Research. 16 (12), 3113-3120 (2010).
- Hu, Y., Smyth, G. K. ELDA: extreme limiting dilution analysis for comparing depleted and enriched populations in stem cell and other assays. Journal of Immunological Methods. 347 (1-2), 70-78 (2009).
- Stewart, J. M., et al. Phenotypic heterogeneity and instability of human ovarian tumor-initiating cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (16), 6468-6473 (2011).
- Abraham, B. K., et al. Prevalence of CD44+/CD24-/low cells in breast cancer may not be associated with clinical outcome but may favor distant metastasis. Clinical Cancer Research. 11 (3), 1154-1159 (2005).
- Balic, M., et al. Most early disseminated cancer cells detected in bone marrow of breast cancer patients have a putative breast cancer stem cell phenotype. Clinical Cancer Research. 12 (19), 5615-5621 (2006).
- Charafe-Jauffret, E., et al. Aldehyde dehydrogenase 1-positive cancer stem cells mediate metastasis and poor clinical outcome in inflammatory breast cancer. Clinical Cancer Research. 16 (1), 45-55 (2010).
- Marcato, P., et al. Aldehyde dehydrogenase activity of breast cancer stem cells is primarily due to isoform ALDH1A3 and its expression is predictive of metastasis. Stem Cells. 29 (1), 32-45 (2011).
- Lacerda, L., Pusztai, L., Woodward, W. A. The role of tumor initiating cells in drug resistance of breast cancer: Implications for future therapeutic approaches. Drug Resist Updat. 13 (4-5), 99-108 (2010).
- Liu, S., Wicha, M. S. Targeting breast cancer stem cells. Journal of Clinical Oncology. 28 (25), 4006-4012 (2010).
- D'Angelo, R. C., et al. Notch reporter activity in breast cancer cell lines identifies a subset of cells with stem cell activity. Molecular Cancer Therapeutics. 14 (3), 779-787 (2015).
- Neve, R. M., et al. A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes. Cancer Cell. 10 (6), 515-527 (2006).
- Forozan, F., et al. Comparative genomic hybridization analysis of 38 breast cancer cell lines: a basis for interpreting complementary DNA microarray data. Cancer Research. 60 (16), 4519-4525 (2000).
- Lanier, L. L. Just the FACS. Journal of Immunology. 193 (5), 2043-2044 (2014).
- Ibrahim, S. F., van den Engh, G. Flow cytometry and cell sorting. Advances in Biochemical Engineering/Biotechnology. 106, 19-39 (2007).
- Shapiro, H. M. Flow Cytometry: The Glass Is Half Full. Methods in Molecular Biology. 1678, 1-10 (2018).
- Tsuji, K., et al. Effects of Different Cell-Detaching Methods on the Viability and Cell Surface Antigen Expression of Synovial Mesenchymal Stem Cells. Cell Transplantation. 26 (6), 1089-1102 (2017).
- Sun, C., et al. Immunomagnetic separation of tumor initiating cells by screening two surface markers. Scientific Reports. 7, 40632 (2017).
- Rodríguez, C. E., et al. Breast cancer stem cells are involved in Trastuzumab resistance through the HER2 modulation in 3D culture. Journal of Cellular Biochemistry. 119 (2), 1381-1391 (2018).
- Kim, D. W., Cho, J. Y. NQO1 is Required for β-Lapachone-Mediated Downregulation of Breast-Cancer Stem-Cell Activity. International Journal of Molecular Sciences. 19 (12), (2018).
- Xu, L. Z., et al. p62/SQSTM1 enhances breast cancer stem-like properties by stabilizing MYC mRNA. Oncogene. 36 (3), 304-317 (2017).
- Huang, X., et al. Breast cancer stem cell selectivity of synthetic nanomolar-active salinomycin analogs. BMC Cancer. 16, 145 (2016).
- Liu, T. J., et al. CD133+ cells with cancer stem cell characteristics associates with vasculogenic mimicry in triple-negative breast cancer. Oncogene. 32 (5), 544-553 (2013).
- Ponti, D., et al. Isolation and in vitro propagation of tumorigenic breast cancer cells with stem/progenitor cell properties. Cancer Research. 65 (13), 5506-5511 (2005).
- Velasco-Velázquez, M. A., Popov, V. M., Lisanti, M. P., Pestell, R. G. The role of breast cancer stem cells in metastasis and therapeutic implications. American Journal of Pathology. 179 (1), 2-11 (2011).
- Palomeras, S., Ruiz-Martínez, S., Puig, T. Targeting Breast Cancer Stem Cells to Overcome Treatment Resistance. Molecules. 23 (9), (2018).
- McClements, L., et al. Targeting treatment-resistant breast cancer stem cells with FKBPL and its peptide derivative, AD-01, via the CD44 pathway. Clinical Cancer Research. 19 (14), 3881-3893 (2013).
- Berger, D. P., Henss, H., Winterhalter, B. R., Fiebig, H. H. The clonogenic assay with human tumor xenografts: evaluation, predictive value and application for drug screening. Annals of Oncology. 1 (5), 333-341 (1990).
- Tian, J., et al. Dasatinib sensitises triple negative breast cancer cells to chemotherapy by targeting breast cancer stem cells. British Journal of Cancer. 119 (12), 1495-1507 (2018).
- Samoszuk, M., Tan, J., Chorn, G. Clonogenic growth of human breast cancer cells co-cultured in direct contact with serum-activated fibroblasts. Breast Cancer Research. 7 (3), 274-283 (2005).
- Linnemann, J. R., et al. Quantification of regenerative potential in primary human mammary epithelial cells. Development. 142 (18), 3239-3251 (2015).
- Xu, Y., Hu, Y. D., Zhou, J., Zhang, M. H. Establishing a lung cancer stem cell culture using autologous intratumoral fibroblasts as feeder cells. Cell Biology International. 35 (5), 509-517 (2011).
- Palmieri, C., et al. Fibroblast growth factor 7, secreted by breast fibroblasts, is an interleukin-1beta-induced paracrine growth factor for human breast cells. Journal of Endocrinology. 177 (1), 65-81 (2003).
- Bourguignon, L. Y., Peyrollier, K., Xia, W., Gilad, E. Hyaluronan-CD44 interaction activates stem cell marker Nanog, Stat-3-mediated MDR1 gene expression, and ankyrin-regulated multidrug efflux in breast and ovarian tumor cells. Journal of Biological Chemistry. 283 (25), 17635-17651 (2008).
- Yin, X., et al. Engineering Stem Cell Organoids. Cell Stem Cell. 18 (1), 25-38 (2016).
- Sachs, N., et al. A Living Biobank of Breast Cancer Organoids Captures Disease Heterogeneity. Cell. 172 (1-2), 373-386 (2018).
- Kim, M., et al. Patient-derived lung cancer organoids as in vitro cancer models for therapeutic screening. Nature Communications. 10 (1), 3991 (2019).
- Okano, M., et al. Orthotopic Implantation Achieves Better Engraftment and Faster Growth Than Subcutaneous Implantation in Breast Cancer Patient-Derived Xenografts. Journal of Mammary Gland Biology and Neoplasia. 25 (1), 27-36 (2020).
- Zhang, Y., et al. Establishment of a murine breast tumor model by subcutaneous or orthotopic implantation. Oncology Letters. 15 (5), 6233-6240 (2018).
- Zhang, W., et al. Comparative Study of Subcutaneous and Orthotopic Mouse Models of Prostate Cancer: Vascular Perfusion, Vasculature Density, Hypoxic Burden and BB2r-Targeting Efficacy. Scientific Reports. 9 (1), 11117 (2019).
- Kim, R., Emi, M., Tanabe, K. Cancer immunoediting from immune surveillance to immune escape. Immunology. 121 (1), 1-14 (2007).
- Rosato, R. R., et al. Evaluation of anti-PD-1-based therapy against triple-negative breast cancer patient-derived xenograft tumors engrafted in humanized mouse models. Breast Cancer Research. 20 (1), 108 (2018).
- Choi, Y., et al. Studying cancer immunotherapy using patient-derived xenografts (PDXs) in humanized mice. Experimental and Molecular Medicine. 50 (8), 99 (2018).
- Meraz, I. M., et al. An Improved Patient-Derived Xenograft Humanized Mouse Model for Evaluation of Lung Cancer Immune Responses. Cancer Immunol Res. 7 (8), 1267-1279 (2019).
- Wege, A. K. Humanized Mouse Models for the Preclinical Assessment of Cancer Immunotherapy. Biodrugs. 32 (3), 245-266 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved