Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement et évaluation fonctionnelle des cellules souches du cancer du sein humain à partir d’échantillons de cellules et de tissus
Dans cet article
Résumé
Ce protocole expérimental décrit l’isolement des CSCA à partir d’échantillons de cellules et de tissus cancéreux du sein ainsi que les tests in vitro et in vivo qui peuvent être utilisés pour évaluer le phénotype et la fonction des CSBC.
Résumé
Les cellules souches du cancer du sein (CSBC) sont des cellules cancéreuses dont les caractéristiques héréditaires ou acquises ressemblent à des cellules souches. Malgré leur faible fréquence, elles contribuent grandement à l’initiation du cancer du sein, aux rechutes, aux métastases et à la résistance au traitement. Il est impératif de comprendre la biologie des cellules souches du cancer du sein afin d’identifier de nouvelles cibles thérapeutiques pour traiter le cancer du sein. Les cellules souches du cancer du sein sont isolées et caractérisées en fonction de l’expression de marqueurs de surface cellulaire uniques tels que CD44, CD24 et l’activité enzymatique de l’aldéhyde déshydrogénase (ALDH). Ces cellules ALDHà CD44+CD24- élevé constituent la population BCSC et peuvent être isolées par tri cellulaire activé par fluorescence (FACS) pour des études fonctionnelles en aval. Selon la question scientifique, différentes méthodes in vitro et in vivo peuvent être utilisées pour évaluer les caractéristiques fonctionnelles des BCSC. Ici, nous fournissons un protocole expérimental détaillé pour l’isolement des BCSC humains à partir de populations hétérogènes de cellules cancéreuses du sein ainsi que de tissus tumoraux primaires obtenus à partir de patientes atteintes d’un cancer du sein. En outre, nous mettons en évidence des tests fonctionnels in vitro et in vivo en aval, y compris des tests de formation de colonies, des tests de mammosphère, des modèles de culture 3D et des tests de xénogreffe tumorale qui peuvent être utilisés pour évaluer la fonction BCSC.
Introduction
Comprendre les mécanismes cellulaires et moléculaires des cellules souches du cancer du sein humain (CSCA) est crucial pour relever les défis rencontrés dans le traitement du cancer du sein. L’émergence du concept BCSC remonte au début du 21esiècle, où une petite population de cellules cancéreuses du sein CD44 + CD24 / faible s’est avérée capable de générer des tumeurs hétérogènes chez la souris 1,2. Par la suite, il a été observé que les cellules cancéreuses du sein humain ayant une activité enzymatique élevée de l’aldéhyde déshydrogénase (ALDHélevée) présentaient également des propriétés similaires à celles des cellules souches3. Ces BCSC représentent une petite population de cellules capables de s’auto-renouveler et de se différencier, contribuant à la nature hétérogène des tumeurs en vrac 1,2,3. L’accumulation de preuves suggère que les altérations des voies de signalisation conservées au cours de l’évolution déterminent la survie et le maintiende la BCSC 4,5,6,7,8,9,10,11,12,13,14 . En outre, il a été démontré que le microenvironnement extrinsèque cellulaire joue un rôle central dans la dictée des différentes fonctions BCSC15,16,17. Ces voies moléculaires et les facteurs externes régulant la fonction BCSC contribuent à la rechute du cancer du sein, aux métastases18 et au développement d’une résistance aux thérapies 19,20,21, l’existence résiduelle de BCSC post-traitement posant un défi majeur pour la survie globale des patientes atteintes d’un cancer du sein22,23 . L’évaluation préclinique de ces facteurs est donc très importante pour identifier les thérapies ciblant le BCSC qui pourraient être bénéfiques pour obtenir de meilleurs résultats de traitement et améliorer la survie globale chez les patientes atteintes d’un cancer du sein.
Plusieurs modèles in vitro de lignées cellulaires de cancer du sein humain et modèles in vivo de xénogreffes humaines ont été utilisés pour caractériser les CSCA 24,25,26,27,28,29. La capacité des lignées cellulaires à se repeupler continuellement après chaque passage successif en fait un système modèle idéal pour effectuer des études omiques et pharmacogénomiques. Cependant, les lignées cellulaires ne parviennent souvent pas à récapituler l’hétérogénéité observée dans les échantillons de patients. Par conséquent, il est important de compléter les données de lignées cellulaires avec des échantillons dérivés de patients. L’isolement des CSCA dans leur forme la plus pure est important pour permettre une caractérisation détaillée des CSCA. L’atteinte de cette pureté dépend de la sélection de marqueurs phénotypiques spécifiques aux CSBC. Actuellement, le phénotypeALDH à CD44+CD24- élevé est le plus souvent utilisé pour distinguer et isoler les BCSC humaines des populations de cellules cancéreuses du sein en vrac en utilisant le tri cellulaire activé par fluorescence (FACS) pour une pureté maximale1, 3,26. De plus, les propriétés des BCSC isolées, telles que l’auto-renouvellement, la prolifération et la différenciation, peuvent être évaluées à l’aide de techniques in vitro et in vivo.
Par exemple, les essais in vitro formant des colonies peuvent être utilisés pour évaluer la capacité d’une seule cellule à s’auto-renouveler pour former une colonie de 50 cellules ou plus en présence de différentes conditions de traitement30. Les tests de mammosphère peuvent également être utilisés pour évaluer le potentiel d’auto-renouvellement des cellules cancéreuses du sein dans des conditions indépendantes de l’ancrage. Ce test mesure la capacité de cellules individuelles à générer et à se développer sous forme de sphères (mélange de BCSC et de non-BCSC) à chaque passage successif dans des conditions de culture non adhérentes sans sérum31. De plus, des modèles de culture en 3 dimensions (3D) peuvent être utilisés pour évaluer la fonction BCSC, y compris les interactions cellule-cellule et cellule-matrice qui récapitulent étroitement le microenvironnement in vivo et permettent d’étudier l’activité des thérapies potentielles ciblant le BCSC32. Malgré les diverses applications des modèles in vitro, il est difficile de modéliser la complexité des conditions in vivo en utilisant uniquement des essais in vitro. Ce défi peut être surmonté en utilisant des modèles de xénogreffes de souris pour évaluer le comportement de BCSC in vivo. En particulier, ces modèles constituent un système idéal pour évaluer les métastases du cancer du sein33, étudier les interactions avec le microenvironnement au cours de la progression de la maladie 34, l’imagerie in vivo 35 et prédire la toxicité et l’efficacité spécifiques au patient des agents antitumoraux 34.
Ce protocole fournit une description détaillée de l’isolement des BCSC humains à CD44+CD24- élevés en ALDHà unepureté maximale à partir de populations en vrac de cellules hétérogènes du cancer du sein. Nous fournissons également une description détaillée de trois techniques in vitro (test de formation de colonies, test de mammosphère et modèle de culture 3D) et un test de xénogreffe tumorale in vivo qui peut être utilisé pour évaluer différentes fonctions des BCSC. Ces méthodes seraient appropriées pour les chercheurs intéressés à isoler et à caractériser les CSCA à partir de lignées cellulaires de cancer du sein humain ou de cellules cancéreuses du sein et de tissus tumoraux dérivés de patients primaires dans le but de comprendre la biologie du BCSC et/ou d’étudier de nouvelles thérapies ciblant le BCSC.
Protocole
Le prélèvement d’échantillons chirurgicaux ou de biopsie provenant directement de patientes consentantes atteintes d’un cancer du sein a été effectué conformément à un protocole d’éthique humaine approuvé et approuvé par le conseil d’éthique de l’établissement. Toutes les souris utilisées pour générer des modèles de xénogreffes dérivées de patients ont été conservées et hébergées dans une animalerie approuvée par l’établissement. Le tissu tumoral provenant de modèles de xénogreffes dérivées de patients utilisant des souris a été généré conformément au protocole d’éthique approuvé approuvé par le comité de soins aux animaux de l’établissement.
1. Préparation des lignées cellulaires
- Effectuer toutes les procédures de culture cellulaire et de coloration dans des conditions stériles dans une enceinte de biosécurité. Utiliser des plats/flacons et des réactifs de culture cellulaire stérile.
- Maintenir les cellules cancéreuses du sein humain à 37 °C avec 5% de CO2 dans des milieux définis complétés par du sérum fœtal bovin (FBS) et les facteurs de croissance nécessaires spécifiques à chaque lignée cellulaire.
- Maintenir les cultures de cellules de fibroblastes NIH3T3 de souris (pour une utilisation dans les essais formant des colonies) à 37 °C avec 5% de CO2 dans le milieu d’aigle modifié de Dulbecco (DMEM) complété par 10% FBS.
- Pour toutes les cultures, réapprovisionnez les anciens médias tous les 2-3 jours avec de nouveaux médias. Une fois que les cultures atteignent 75-80% de confluence, sous-culture dans plusieurs flacons de culture cellulaire stérile.
2. Préparation du tissu tumoral du cancer du sein
- Prélever les échantillons chirurgicaux ou biopsiques provenant directement des patientes consentantes atteintes d’un cancer du sein dans le cadre d’un protocole d’éthique humaine approuvé par le comité d’éthique de l’établissement.
- Par la suite, prélever et générer du tissu tumoral à partir de modèles de xénogreffes dérivés de patients en utilisant des souris dans le cadre d’un protocole d’éthique animale approuvé par le comité institutionnel de soins aux animaux.
- Recueillir tous les tissus tumoraux dans des conditions stériles dans un tube conique stérile de 50 ml contenant 30 ml de milieu DMEM:F12, garder sur la glace et traiter les échantillons comme décrit ci-dessous dans les 2 heures suivant le prélèvement.
3. Génération de suspensions unicellulaires de cellules cancéreuses du sein
- Aspirer le milieu du flacon contenant une monocouche de cellules cancéreuses du sein confluentes à 60-80% (lignées cellulaires de choix). Lavez les cellules avec 1x solution saline tamponnée au phosphate (PBS). Aspirer le PBS et ajouter une solution de dissociation cellulaire appropriée (p. ex., trypsine:EDTA; juste assez pour recouvrir la monocouche de cellules) et incuber pendant 5 minutes à température ambiante (recommandé) ou à 37 °C.
- Ajouter 5 mL de milieu de culture pour neutraliser l’activité de la solution de dissociation cellulaire.
- Transférer la solution cellulaire dissociée obtenue dans un tube conique de 50 ml et centrifuger à 1000 x g pendant 5 min.
- Jeter le surnageant et remettre en suspension la pastille de cellule dans 5 mL de 1x PBS. Comptez les cellules à l’aide d’un hémocytomètre et d’un microscope.
REMARQUE: Observez l’agglutination cellulaire dans l’hémocytomètre. Répéter l’étape de dissociation cellulaire si la suspension unicellulaire ne s’est pas formée. - Après comptage cellulaire, recentrifuger la suspension cellulaire à 1000 x g pendant 5 min, jeter le surnageant et remettre en suspension la pastille cellulaire dans un tampon de substrat ALDH à une concentration de 1 x 106 cellules/mL.
4. Production d’une suspension unicellulaire à partir d’échantillons de tissus
- Hachez le tissu tumoral avec des lames chirurgicales en utilisant une technique entrecroisée pour obtenir des morceaux plus petits d’environ 1 mm. Transférer les morceaux de tissu dans un tube conique frais de 50 mL contenant 10 mL de tampon de dissociation (1X collagénase dans DMEM:F12). Sceller le tube conique avec un parafilm et incuber à 37 °C dans un incubateur agitateur pendant 40 min.
REMARQUE: S’il n’y a pas d’incubateur vibreur, placez le tube dans un bain-marie à 37 °C et mélangez le tube en tourbillonnant toutes les 5-10 minutes. - Enduire en granuler le tissu digéré en centrifugeant l’échantillon à 530 x g pendant 5 min. Jeter le surnageant et ajouter 5 mL de trypsine. Pipeter de haut en bas à l’aide d’une pipette de 1 mL (réglée à 750 μL) pour perturber la pastille et incuber dans un bain-marie à 37 °C pendant 5 min. Après l’incubation, pipeter vigoureusement de haut en bas pour libérer les cellules individuelles.
- Complétez le volume total dans le tube à 25 mL avec un média DMEM:F12 et une centrifugeuse à 1000 x g pendant 5 min. Jeter le surnageant et remettre en suspension la pastille dans 1 mL de solution de dispase-DNase. Incuber au bain-marie à 37 °C pendant 5 min.
- Complétez le volume total dans le tube à 10 ml avec du PBS. Mélanger en pipetant de haut en bas, faire passer la suspension cellulaire résultante à travers une crépine cellulaire de 40 μm fixée à un tube conique frais de 50 mL. Centrifuger à 1000 x g pendant 5 min.
- Jeter le surnageant et remettre en suspension la pastille de cellule dans 5 mL de 1x PBS. Compter les cellules et terminer la préparation de la suspension cellulaire comme décrit aux étapes 3.4 et 3.5.
5. Isolement des cellules souches du cancer du sein (CSCSB)
- Tubes d’écoulement étiquetés pour le témoin non coloré, les témoins de coloration unicellulaire (contrôle DEAB, ALDH, CD44-PE, CD24-PE-Cy7, 7AAD), le tube témoin négatif (coloré avec DEAB, CD44-PE, CD24-PE-Cy7 et 7AAD), le témoin fluorescent moins un (FMO) et le tube « tri » (coloré avec ALDH, CD44, CD24 et 7AAD).
- Transfère 500 μL (0,5 x 106 cellules) des suspensions cellulaires de l’étape 3.5 ou 4.5 à chaque tube marqué uniquement des cellules CD44, CD24 et 7AAD. Placer les tubes sur de la glace jusqu’à utilisation.
- Transférer 2 mL d’échantillon (2 x 106 cellules) dans le tube 'ALDH' respectif. Ajoutez 5 μL de DEAB aux tubes de contrôle DEAB et de contrôle négatif et coiffez-le hermétiquement. Ajouter 10 μL de substrat ALDH dans le tube « ALDH », bien mélanger par vortex et transférer immédiatement 500 μL dans les tubes « contrôle DEAB » et « témoin négatif » correspondants. Récapituler les « tubes témoins DEAB », « témoin négatif » et « ALDH » et incuber à 37 °C pendant 30 à 60 min (ne pas dépasser 60 min).
REMARQUE: Le temps d’incubation optimal peut nécessiter une optimisation en fonction de la lignée cellulaire. Protégez toujours du substrat ALDH et les tubes contenant les cellules colorées de la lumière. - Après l’incubation, centrifuger tous les échantillons pendant 5 min à 250 x g. Remettez les cellules en suspension dans 500 μL de tampon substrat ALDH. Ajouter la concentration recommandée par le fabricant ou optimisée pour l’utilisateur du cocktail d’anticorps anti-CD44-PE et anti-CD24-PE-Cy7 et incuber à 4 °C pendant 30 min. Ajoutez des anticorps anti-CD44-PE et anti-CD24-PE-Cy7 aux tubes marqués 'CD44' et 'CD24' respectifs.
- Après l’incubation, centrifuger tous les échantillons à 250 x g pendant 5 min. Remettez les cellules en suspension dans 500 μL de tampon substrat ALDH. Incuber le tube « témoin négatif », le tube « Sort tube » et le tube « 7ADD » avec 7AAD (concentration suggérée : 0,25 μg/1 x 106 cellules) pendant 10 min sur glace.
NOTE: L’activité ALDH est détectée dans le canal fluorescent vert, donc un fluorochrome avec un spectre d’émission compatible différent doit être utilisé. Lorsqu’un chevauchement spectral est observé pendant la cytométrie en flux multiparamétrique, des contrôles monochromes et un contrôle FMO devraient être utilisés comme guide pour permettre la compensation entre les fluorochromes afin de minimiser le débordement du signal fluorescent dans d’autres canaux. - Mettre en place le protocole d’analyse sur l’instrument FACS en préparation de l’analyse des échantillons. Créez des nuages de points (diffusion avant vs diffusion latérale, diffusion avant vs canaux fluorescents).
- À l’aide de la commande non tachée, ajuster le photomultiplicateur pour séparer les débris de la population cellulaire entière et ajuster la tension fluorescente pour déplacer la population cellulaire entière autour de la première échelle logarithmique (101). À l’aide du contrôle DEAB, déplacez la population cellulaire entière dans la deuxième échelle logarithmique (102) en ajustant le canal de tension fluorescent vert.
- Analysez d’abord tous les contrôles de coloration simples (ALDH, CD44-PE, CD24-PE-Cy7) et 7AAD et FMO, en ajustant la tension pour séparer les cellules colorées des cellules non colorées et minimiser le déversement de signaux fluorescents dans d’autres canaux.
- Porte sur la population positive pour chaque échantillon de cellules colorées. Utilisation du tube témoin négatif, porte de viabilité (7AAD négatif), ALDHfaibles etALDH populations cellulaires élevées (stratégie de déclenchement représentative illustrée à la figure 1B).
- Analyser des échantillons colorés multiparamètres d’intérêt pour isoler les CSCA. À l’aidedes portes hautes et basses ALDH viables, sélectionner la population cellulaire CD44+CD24- (BCSC) et CD44-CD24+ (non-BCSC) respectivement (Figure 1B).
- Recueillir des CSCA viables et des non-CSCA dans des milieux de collecte dans des tubes de prélèvement stériles (populations provenant de deux lignées cellulaires représentatives illustrées à la figure 2A et B). Utiliser des cellules triées pour les essais in vitro et in vivo en aval, comme décrit ci-dessous.
REMARQUE : En plus des essais in vitro et in vivo décrits ci-dessous, les BCSC peuvent être validés en mesurant l’expression de marqueurs pluripotents tels que SOX2, OCT4 et NANOG au moyen de techniques standard d’immunobuvardage.
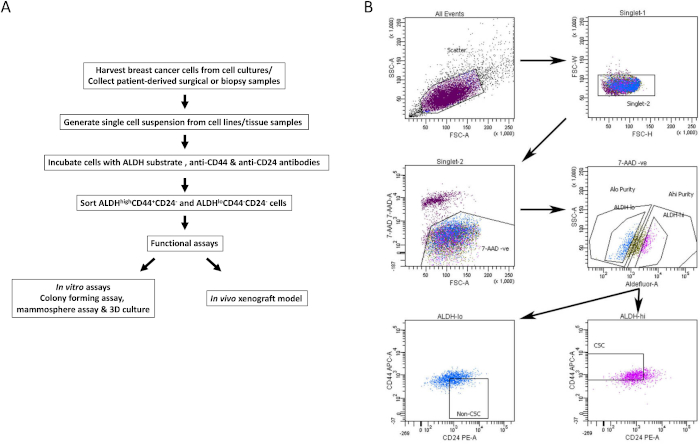
Figure 1 : Stratégie de contrôle FACS pour l’isolement des CSCA à partir de lignées cellulaires et d’échantillons de tissus cancéreux du sein. (A) Organigramme décrivant la procédure d’isolement de la BCSC. (B) Graphiques FACS représentatifs montrant la stratégie de tri utilisée pour isoler les CSCA viables et les non-CSCA d’un pool hétérogène de cellules. Les cellules cancéreuses du sein humain MDA-MB-231 sont marquées simultanément avec le 7-AAD, le CD44-APC, le CD24-PE et le substrat ALDH. Les sous-ensembles de cellules ont été isolés à l’aide d’un protocole à quatre couleurs sur une machine FACS. Les cellules sont sélectionnées en fonction de la diffusion de la lumière attendue, puis pour les singulettes, et la viabilité est basée sur l’exclusion 7-AAD. Les cellules sont ensuite analysées pour l’activité de l’ALDH et les 20% les plus positifs sont sélectionnés comme la populationélevée de l’ALDH, tandis que les 20% inférieurs des cellules ayant la plus faible activité ALDH ont été jugés comme étantALDH faible. Enfin, 50 % des cellules ALDHbasses sont sélectionnées en fonction d’un phénotype CD44faible/CD24+, et 50 % des cellulesALDH élevées sont sélectionnées en fonction du phénotype CD44+CD24-. Cette figure a été adaptée de Chu et al.17. Veuillez cliquer ici pour voir une version agrandie de cette figure.
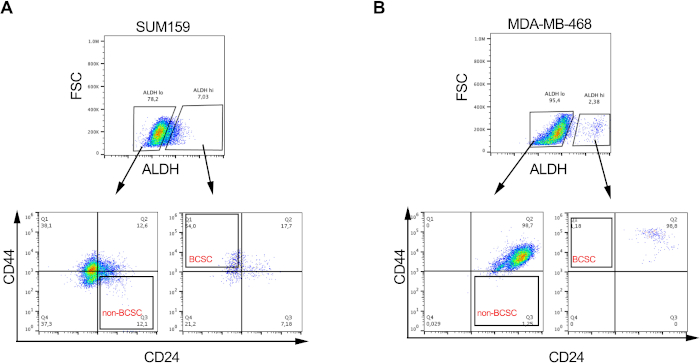
Figure 2 : Les proportions de CSCA varient selon les lignées cellulaires de cancer du sein. Image représentative montrant la proportion différentielle de CSCA et de non-CSCA dans (A) les lignées cellulaires de cancer du sein triple négatif (A) SUM159 et (B) MDA-MD-468 après marquage et tri, comme décrit à la figure 1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
6. Essai de formation de colonies
- Remettez en suspension les cellules d’intérêt (cellules triées de l’étape 5.11 ou cellules non triées des étapes 3.5 ou 4.5) dans un milieu complet.
- Étiquetez trois tubes d’écoulement pour 1 x 10 2, 2 x 10 2 et 5 x 102 cellules. Ajouter 2 mL de média complet et transférer le numéro de cellule approprié (trié à partir de l’étape 5.11 ou cellules non triées à partir des étapes 3.5 ou 4.5) dans les tubes respectifs. Bien mélanger les solutions cellulaires en les pipetant de haut en bas 5 fois.
- Plaquez les cellules dans une plaque à 6 puits et distribuez la suspension cellulaire en faisant tourner doucement les plaques pour obtenir une distribution uniforme des cellules.
- Incuber les plaques dans un incubateur à 37 °C, 5% de CO2 jusqu’à l’apparition des colonies (où colonies = ≥50 cellules par colonie). Reconstituer soigneusement le milieu deux fois par semaine sans perturber la formation des colonies.
- Aspirer le média et laver une fois avec 1 mL de PBS. Ajouter 0,5 mL de solution de cristal violet à 0,05 % dans chaque puits et incuber la plaque pendant 30 minutes. Enlevez l’excès de tache de violet cristallin en lavant avec 2 ml d’eau. Répétez l’étape de lavage jusqu’à ce que la tache de fond ait été éliminée.
- À l’aide d’un microscope à un grossissement de 4x et 10x, compter et enregistrer le nombre total de colonies générées (images représentatives illustrées à la figure 3A).
- Calculer la fréquence de formation des colonies comme suit : Fréquence (%) = (# de colonies formées/nombre de cellules ensemencées) x 100. Par exemple, si 25 colonies sont générées à partir de 1 x 102 cellules, alors la fréquence de formation des colonies est, Fréquence = (25/100) x100 = 25%.
- Sinon, remplacez les étapes 6.1 à 6.4 par une autre méthode impliquant la co-culture avec des fibroblastes, qui fournit un soutien microenvironnemental aux CSCA grâce à la production des facteurs de croissance et de survie nécessaires.
- Boîtes de culture de 60 mm de cellules pré-enrobées de collagène bovin de type I (dilution de 1 dans 30 de collagène à 3 mg/mL). Laisser le collagène polymériser pendant 30 min dans un incubateur à 37 °C. Aspirez le collagène non polymérisé et lavez la plaque deux fois avec 1x PBS. Couvrir la plaque enduite de collagène avec 1 mL de PBS et la mettre de côté à température ambiante jusqu’à utilisation.
- Étiquetez trois tubes d’écoulement pour 1 x 10 3, 5 x 103 et 1 x 104 cellules. Ajouter 4 mL de milieu de dosage formant colonie et transférer le nombre approprié de cellules (triées à partir de l’étape 5.11 ou cellules non triées à partir des étapes 3.5 ou 4.5) dans les tubes respectifs. Ajouter des fibroblastes NIH3T3 irradiés de souris (4 x 104 cellules/ml de milieu). Bien mélanger les solutions cellulaires en les pipetant de haut en bas 5 fois.
- Aspirer le PBS de la boîte de culture enrobée de collagène à l’étape 6.1 et plaquer le mélange cellulaire sur chacune des plaques de culture cellulaire comme décrit à l’étape 6.3.
- Incuber les plaques dans un incubateur à 37 °C, 5% de CO2 et les laisser intactes pendant 7 à 10 jours ou jusqu’à la formation de colonies, sans reconstituer le milieu. Compter et consigner le nombre total de colonies générées comme décrit aux étapes 6.6 et 6.7.
7. Dosage de la mammosphère
- Resuspendre les cellules d’intérêt (cellules triées de l’étape 5.11 ou cellules non triées des étapes 3.5 ou 4.5) dans des milieux mammosphères complets et des cellules en plaques à une densité d’ensemencement de 5 x 10 2 cellules/cm2 dans une plaque de culture cellulaire ultra-basse de 96 puits.
REMARQUE : La densité d’ensemencement cellulaire doit être optimisée pour différentes lignées cellulaires. - Incuber les plaques de culture pendant 5-10 jours dans un incubateur à 37 °C avec 5% de CO2. Reconstituer soigneusement le milieu deux fois par semaine sans perturber la formation de mammosphères.
- Après l’incubation, compter le nombre de mammosphères générées dans chaque puits à l’aide d’un microscope; où les mammosphères sont définies comme des amas de cellules cancéreuses du sein de plus de 100 μm de diamètre (images représentatives illustrées à la figure 3B).
- Calculer l’efficacité de formation des mammosphères (EMM) comme suit : EMF (%) = (nombre de mammosphères par puits)/ (nombre de cellules ensemencées par puits) x 100 (c.-à-d. si 5 mammosphères sont générées par 1 x 102 cellules dans un puits, alors MFE = (5/100) x 100 = 5 %).
- Pour les mammosphères de sous-culture, transférer soigneusement les milieux contenant le contenu des mammosphères dans un tube conique frais de 50 ml et un milieu centrifuge à 1000 x g pendant 5 min. Retirer délicatement le surnageant, remettre en suspension la pastille cellulaire dans 500 μL de trypsine et incuber pendant 5 min à température ambiante.
- Jeter le surnageant et remettre en suspension la pastille dans 1 mL de milieu mammosphère complet. Compter les cellules à l’aide d’un hémocytomètre et replaquer les cellules dans une plaque de culture de cellules à très faible attache, comme décrit à l’étape 7.1.
REMARQUE : En plus de la sous-culture, les cellules dérivées de la mammosphère peuvent également être analysées plus en détail par FACS pour évaluer le phénotype BCSC et/ou obtenir des populations pures de BCSC pour d’autres tests en aval. - Pour déterminer le nombre de cellules initiatrices de mammosphères contenues dans vos populations cellulaires, utilisez une autre méthode impliquant une analyse de dilution limite de sphère (SLDA). Cellules en plaques dans des dilutions en série d’un nombre élevé à faible de cellules dans une plaque de culture de cellules à très faible attachement de 96 puits, la dilution la plus élevée donnant moins d’une cellule par puits.
- Incuber la plaque de culture pendant 10-14 jours dans un incubateur à 37 °C avec 5% de CO2 et les laisser intactes pour éviter l’agrégation cellulaire.
- Après l’incubation, compter le nombre de mammosphères générées dans chaque puits à l’aide d’un microscope; où les mammosphères sont définies comme des amas de cellules cancéreuses du sein de plus de 100 μm de diamètre. Calculez la fréquence et la signification d’initiation de la sphère à l’aide du logiciel en ligne ELDA (Extreme Limiting Dilution Analysis) (http://bioinf.wehi.edu.au/software/elda/).
8.3D modèle de culture
- Selon la question expérimentale, utiliser l’extrait membranaire basal (EMB) avec ou sans facteurs de croissance (réduit). Afin d’évaluer l’effet du facteur de croissance individuel sur les cellules cancéreuses, utilisez le facteur de croissance réduit BME. Il aide également à minimiser les effets non spécifiques des facteurs de croissance endogènes présents dans les EMB.
NOTA: BME se solidifie au-dessus de 10 ° C. Gardez toujours BME sur la glace même pendant l’étape de décongélation. - Ajouter délicatement 50 μL de BME par puits dans une plaque de 96 puits sans créer de bulles d’air et laisser polymériser à 37 °C pendant 1 h. Après 10 min d’incubation, ajouter 100 μL de PBS pour éviter le dessèchement de la couche de gel.
- Resuspendre les cellules triées de l’étape 5.11 ou les cellules non triées des étapes 3.5 ou 4.5 à une concentration de 5 x 103 à 5 x 104/200 μL dans un milieu de culture 3D.
- Une fois le BME polymérisé, enlever le PBS, ajouter 200 μL de suspension cellulaire à chaque puits et incuber dans un incubateur à 37 °C avec 5% de CO2. Ajouter du PBS aux puits environnants pour éviter l’évaporation du média.
REMARQUE : Le nombre optimal de cellules pour le placage doit être déterminé avant le réglage de l’expérience. Selon la question expérimentale, les BCSC peuvent être cultivées seules ou avec d’autres types de cellules (fibroblastes/cellules endothéliales/immunitaires, etc.). - Ajouter des supports frais aux assiettes de culture deux fois par semaine. Maintenir les cultures pendant 10 à 14 jours avant d’analyser la formation des organoïdes (images représentatives illustrées à la figure 3C).
- Pour la sous-culture, aspirez soigneusement le milieu et ajoutez 200 μL de dispase à chaque puits contenant des cellules. Incuber la plaque dans un incubateur à 37 °C pendant 1 h. À mi-chemin de la période d’incubation (30 min), sortez la plaque, pipeter doucement la solution de dispase de haut en bas 5 fois et remettez-la dans l’incubateur pendant 30 minutes supplémentaires.
- Après 1 h, transférer la solution cellulaire dissociée dans un tube d’écoulement. Lavez le puits avec 1x PBS contenant 2% de FBS (fPBS) et transférez-le dans le tube d’écoulement. Centrifuger le tube à 1000 x g pendant 5 min. Aspirer soigneusement le surnageant et ajouter 500 μL de trypsine, incuber à 37 °C pendant 5 min. Inactiver la trypsine en ajoutant une quantité égale de fPBS et centrifuger à 1000 x g pendant 5 min.
- Jeter le surnageant et remettre en suspension le granulé dans 1 mL de milieu de culture 3D. Comptez les cellules et replaquez le nombre requis de cellules dans le BME comme aux étapes 8.2 à 8.4.
REMARQUE: Plusieurs puits peuvent être regroupés pour analyser ou trier plus en détail la population cellulaire d’intérêt.
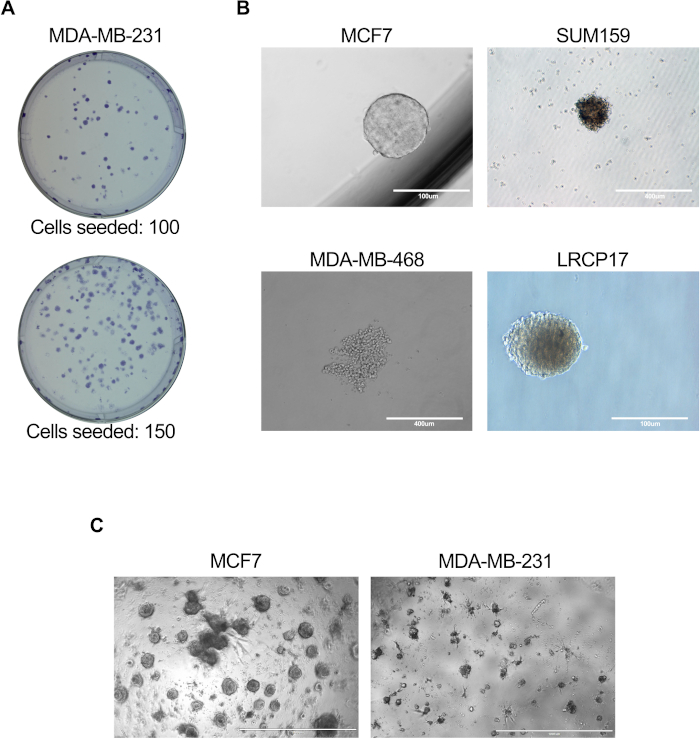
Figure 3 : Essais in vitro pour évaluer la fonction des cellules BCSC. Des essais in vitro ont été effectués comme décrit dans les sections 6.1 à 6.5 (A), 7.1 à 7.4 (B) ou 81 du protocole. à 8,4 + 8,6 (C). (A) Image représentative montrant les colonies générées par les cellules cancéreuses du sein humain MDA-MB-231; (B) Images représentatives montrant la formation de mammosphères par des lignées cellulaires humaines MCF7, SUM159 ou MDA-MB-468 ainsi que par des cellules cancéreuses du sein LRCP17 dérivées de patientes. (C) Images représentatives montrant les structures 3D formées par les cellules cancéreuses du sein MCF7 et MDA-MB-231 dans des modèles de cultures 3D. Veuillez cliquer ici pour voir une version agrandie de cette figure.
REMARQUE : Effectuer des expériences sur les animaux en vertu d’un protocole d’éthique animale approuvé par le comité de protection des animaux de l’établissement.
9. Modèle de xénogreffe in vivo
- Afin de déterminer la capacité d’initiation tumorale des cellules souches du cancer du sein, préparer des cellules (population triée de l’étape 5.7 ou populations non triées des étapes 3.5 ou 4.5) en utilisant une approche de dilution limitante. Diluer en série les cellules dans le PBS en utilisant entre 1 et 5 groupes de dilution différents, avec des doses aussi faibles que 0,01-0,2 x 102 cellules/100 μL et aussi élevées que 1 x 106 cellules/100 μL.
REMARQUE : Les cellules de population non triées/entières peuvent être utilisées comme contrôle. Le nombre de groupes de dilution utilisés dépendra du résultat scientifique souhaité (par exemple, si seulement tester la tumorgénicité, alors qu’un groupe à un nombre de cellules plus élevé peut être utilisé, alors que lors du calcul de la capacité d’initiation de la tumeur, il est optimal de tester 5 doses de dilution limitantes). - Pour générer des modèles de xénogreffes à partir de cellules cancéreuses du sein humain, utilisez des souris femelles immunodéprimées (souches athymiques nues [nu/nu], diabétiques non obèses/immunodéficientes combinées sévères [NOD/SCID] ou NOD/SCID IL2γ [NGS]).
NOTE: Bien qu’un minimum de 4 animaux par groupe puisse être utilisé, 8 à 12 animaux par groupe sont recommandés pour obtenir des résultats robustes, en particulier pour limiter l’analyse de dilution. - Effectuer des injections standard de coussinets adipeux mammaires (MFP) en utilisant 100 μL/souris de chaque préparation cellulaire, dans des conditions stériles dans une enceinte de biosécurité.
REMARQUE: Pour une croissance optimale de la tumeur du sein et des métastases spontanées aux organes distants, la MFP thoracique est recommandée. Alternativement, le MFP inguinal peut également être utilisé. - Après l’injection, surveiller quotidiennement les souris pour l’état de santé général et la croissance tumorale au site d’injection. Lors de la détection d’une tumeur palpable, commencez à mesurer la taille de la tumeur par des étriers en deux dimensions perpendiculaires et enregistrez chaque semaine jusqu’à la fin.
REMARQUE : Le critère d’évaluation expérimental est déterminé en fonction des règlements établis dans le protocole institutionnel d’éthique animale; Typiquement, le critère d’évaluation par euthanasie est généralement requis une fois que les volumes tumoraux atteignent 1500 mm3. Pour les populations BCSC et/ou des doses cellulaires plus élevées (p. ex. >1 x 104 cellules), ce paramètre sera probablement atteint dans les 4 à 8 semaines suivant l’injection de la PMF. Pour les très faibles doses cellulaires et / ou les populations de cellules non-BCSC, la croissance tumorale doit être autorisée à progresser jusqu’à 8 mois après l’injection. - À partir de ces mesures, calculez le volume tumoral à l’aide de la formule suivante: Volume en mm3 = 0,52 x (largeur)2 x longueur. Si vous utilisez une approche de dilution limitante, calculez la capacité et la signification de l’initiation de la tumeur à l’aide du logiciel en ligne ELDA (http://bioinf.wehi.edu.au/software/elda/).
- Alternativement, pour étendre humainement le critère d’évaluation, enlever chirurgicalement les tumeurs primaires et continuer à surveiller les souris pour la santé et / ou le développement de métastases spontanées dans des organes éloignés. Utiliser du tissu tumoral réséqué pour la génération de xénotransplants en série.
- Au point final, prélever des tissus sur des tumeurs primaires et des organes distants (ganglions lymphatiques, poumons, foie, cerveau, os) et effectuer des analyses histopathologiques et/ou immunohistochimiques ou dissocier le tissu tumoral et utiliser dans les essais in vitro décrits aux rubriques 6 à 8.
Résultats
Le protocole décrit permet d’isoler des CSCA humaines à partir d’une population hétérogène de cellules cancéreuses du sein, soit à partir de lignées cellulaires, soit à partir de tissus tumoraux dissociés. Pour une lignée cellulaire ou un échantillon de tissu donné, il est crucial de générer une suspension unicellulaire uniforme pour isoler les CSCA avec une pureté maximale, car la contamination des populations non BCSC pourrait entraîner des réponses cellulaires variables, en particulier si le but ...
Discussion
Les métastases du cancer du sein et la résistance au traitement sont devenues des causes majeures de mortalité chez les femmes dans le monde entier. L’existence d’une sous-population de cellules souches du cancer du sein (CSBC) contribue à l’augmentation des métastases 26,43,44,45,46 et à la résistance au traitement21,47,48.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions les membres de notre laboratoire pour leurs discussions utiles et leur soutien. Notre recherche sur les cellules souches du cancer du sein et le microenvironnement tumoral est financée par des subventions de l’Institut de recherche de la Société canadienne de recherche sur le cancer et du U.S. Army Department of Defense Breast Cancer Program (subvention # BC160912). V.B. est soutenu par une bourse postdoctorale Western (Université Western), et A.L.A. et V.B. sont soutenus par la Société du cancer du sein du Canada. C.L. est soutenu par une bourse d’études supérieures du Canada Vanier du gouvernement du Canada.
matériels
| Name | Company | Catalog Number | Comments |
| 7-Aminoactinomycin D (7AAD) | BD | 51-68981E | suggested: 0.25 µg/1x106 cells |
| Acetone | Fisher | A18-1 | |
| Aldehyde dehydrogenase (ALDH) substrate | Stemcell Technologies | 1700 | Sold commerically as part of the ALDEFLOUR Assay kit; follow manufacturer's instructions for ALDH substrate preparation |
| Basement membrane extract (BME) | Corning | 354234 | Sold under the commercial name Matrigel |
| Cell culture plates: 6 well | Corning | 877218 | |
| Cell culture plates: 60mm | Corning | 353002 | |
| Cell culture plates: 96-well ultra low attachment | Corning | 3474 | |
| Cell strainer: 40 micron | BD | 352340 | |
| Collagen | Stemcell Technologies | 7001 | Prepare 1:30 dilution of 3 mg/mL collagen in PBS |
| Collagenase | Sigma | 11088807001 | 1x |
| Conical tubes: 50 mL | Fisher scientific | 05-539-7 | |
| Crystal violet | Sigma | C6158 | Use 0.05% crystal violet solution in water for staining |
| Dispase | Stemcell Technologies | 7913 | 5U/mL |
| DMEM:F12 | Gibco | 11330-032 | 1x, With L-glutamine and 15 mM HEPES |
| DNAse | Sigma | D5052 | 0.1 mg/mL final concentration |
| FBS | Avantor Seradigm Lifescience | 97068-085 | |
| Flow tubes: 5ml | BD | 352063 | Polypropylene round-bottom tubes |
| Methanol | Fisher | 84124 | |
| mouse anti-Human CD24 antibody | BD | 561646 | R-phycoerythrin and Cyanine dye conjugated Clone: ML5 |
| mouse anti-Human CD44 antibody | BD | 555479 | R-phycoerythrin conjugated, Clone: G44-26 |
| N,N-diethylaminobenzaldehyde (DEAB) | Stemcell Technologies | 1700 | Sold commerically as part of the ALDEFLOUR Assay kit; follow manufacturer's instructions DEAB preparation |
| PBS | Wisent Inc | 311-425-CL | 1x, Without calcium and magnesium |
| Trypsin-EDTA | Gibco | 25200-056 | |
| Mammosphere Media Composition | |||
| B27 | Gibco | 17504-44 | 1x |
| bFGF | Sigma | F2006 | 10 ng/mL |
| BSA | Bioshop | ALB003 | 04% |
| DMEM:F12 | Gibco | 11330-032 | 1x, With L-glutamine and 15 mM HEPES |
| EGF | Sigma | E9644 | 20 ng/mL |
| Insulin | Sigma | 16634 | 5 µg/mL |
| 3D Organoid Media Composition | |||
| A8301 | Tocris | 2939 | 500 nM |
| B27 | Gibco | 17504-44 | 1x |
| DMEM:F12 | Gibco | 11330-032 | 1x, With L-glutamine and 15 mM HEPES |
| EGF | Sigma | E9644 | 5 ng/mL |
| FGF10 | Peprotech | 100-26 | 20 ng/mL |
| FGF7 | Peprotech | 100-19 | 5 ng/mL |
| GlutaMax | Invitrogen | 35050-061 | 1x |
| HEPES | Gibco | 15630-080 | 10 mM |
| N-acetylcysteine | Sigma | A9165 | 1.25 mM |
| Neuregulin β1 | Peprotech | 100-03 | 5 nM |
| Nicotinamide | Sigma | N0636 | 5 mM |
| Noggin | Peprotech | 120-10C | 100 ng/mL |
| R-spondin3 | R&D | 3500 | 250 ng/mL |
| SB202190 | Sigma | S7067 | 500 nM |
| Y-27632 | Tocris | 1254 | 5 µM |
Références
- Al-Hajj, M., Wicha, M. S., Benito-Hernandez, A., Morrison, S. J., Clarke, M. F. Prospective identification of tumorigenic breast cancer cells. Proceedings of the National Academy of Sciences of the United States of America. 100 (7), 3983-3988 (2003).
- Shipitsin, M., et al. Molecular definition of breast tumor heterogeneity. Cancer Cell. 11 (3), 259-273 (2007).
- Ginestier, C., et al. ALDH1 is a marker of normal and malignant human mammary stem cells and a predictor of poor clinical outcome. Cell Stem Cell. 1 (5), 555-567 (2007).
- Sulaiman, A., et al. Dual inhibition of Wnt and Yes-associated protein signaling retards the growth of triple-negative breast cancer in both mesenchymal and epithelial states. Molecular Oncology. 12 (4), 423-440 (2018).
- Debeb, B. G., et al. Histone deacetylase inhibitors stimulate dedifferentiation of human breast cancer cells through WNT/β-catenin signaling. Stem Cells. 30 (11), 2366-2377 (2012).
- Klutzny, S., et al. PDE5 inhibition eliminates cancer stem cells via induction of PKA signaling. Cell Death & Disease. 9 (2), 192 (2018).
- DiMeo, T. A., et al. A novel lung metastasis signature links Wnt signaling with cancer cell self-renewal and epithelial-mesenchymal transition in basal-like breast cancer. Cancer Research. 69 (13), 5364-5373 (2009).
- Liu, C. C., Prior, J., Piwnica-Worms, D., Bu, G. LRP6 overexpression defines a class of breast cancer subtype and is a target for therapy. Proceedings of the National Academy of Sciences of the United States of America. 107 (11), 5136-5141 (2010).
- Miller-Kleinhenz, J., et al. Dual-targeting Wnt and uPA receptors using peptide conjugated ultra-small nanoparticle drug carriers inhibited cancer stem-cell phenotype in chemo-resistant breast cancer. Biomaterials. 152, 47-62 (2018).
- Mamaeva, V., et al. Inhibiting Notch Activity in Breast Cancer Stem Cells by Glucose Functionalized Nanoparticles Carrying γ-secretase Inhibitors. Molecular Therapy. 24 (5), 926-936 (2016).
- Ithimakin, S., et al. HER2 drives luminal breast cancer stem cells in the absence of HER2 amplification: implications for efficacy of adjuvant trastuzumab. Cancer Research. 73 (5), 1635-1646 (2013).
- Koike, Y., et al. Anti-cell growth and anti-cancer stem cell activities of the non-canonical hedgehog inhibitor GANT61 in triple-negative breast cancer cells. Breast Cancer. 24 (5), 683-693 (2017).
- Sun, Y., et al. Estrogen promotes stemness and invasiveness of ER-positive breast cancer cells through Gli1 activation. Molecular Cancer. 13, 137 (2014).
- Colavito, S. A., Zou, M. R., Yan, Q., Nguyen, D. X., Stern, D. F. Significance of glioma-associated oncogene homolog 1 (GLI1) expression in claudin-low breast cancer and crosstalk with the nuclear factor kappa-light-chain-enhancer of activated B cells (NFκB) pathway. Breast Cancer Research. 16 (5), 444 (2014).
- Bhat, V., Allan, A. L., Raouf, A. Role of the Microenvironment in Regulating Normal and Cancer Stem Cell Activity: Implications for Breast Cancer Progression and Therapy Response. Cancers. 11 (9), (2019).
- Pio, G. M., Xia, Y., Piaseczny, M. M., Chu, J. E., Allan, A. L. Soluble bone-derived osteopontin promotes migration and stem-like behavior of breast cancer cells. PloS One. 12 (5), 0177640 (2017).
- Chu, J. E., et al. Lung-derived factors mediate breast cancer cell migration through CD44 receptor-ligand interactions in a novel ex vivo system for analysis of organ-specific soluble proteins. Neoplasia. 16 (2), 180-191 (2014).
- McGowan, P. M., et al. Notch1 inhibition alters the CD44hi/CD24lo population and reduces the formation of brain metastases from breast cancer. Molecular Cancer Research. 9 (7), 834-844 (2011).
- Mao, J., et al. ShRNA targeting Notch1 sensitizes breast cancer stem cell to paclitaxel. International Journal of Biochemistry and Cell Biology. 45 (6), 1064-1073 (2013).
- Duru, N., et al. HER2-associated radioresistance of breast cancer stem cells isolated from HER2-negative breast cancer cells. Clinical Cancer Research. 18 (24), 6634-6647 (2012).
- Croker, A. K., Allan, A. L. Inhibition of aldehyde dehydrogenase (ALDH) activity reduces chemotherapy and radiation resistance of stem-like ALDHhiCD44+ human breast cancer cells. Breast Cancer Research and Treatment. 133 (1), 75-87 (2012).
- Creighton, C. J., et al. Residual breast cancers after conventional therapy display mesenchymal as well as tumor-initiating features. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13820-13825 (2009).
- Calcagno, A. M., et al. Prolonged drug selection of breast cancer cells and enrichment of cancer stem cell characteristics. Journal of the National Cancer Institute. 102 (21), 1637-1652 (2010).
- Feng, Y., et al. Breast cancer development and progression: Risk factors, cancer stem cells, signaling pathways, genomics, and molecular pathogenesis. Genes Dis. 5 (2), 77-106 (2018).
- Samanta, D., Gilkes, D. M., Chaturvedi, P., Xiang, L., Semenza, G. L. Hypoxia-inducible factors are required for chemotherapy resistance of breast cancer stem cells. Proceedings of the National Academy of Sciences of the United States of America. 111 (50), 5429-5438 (2014).
- Croker, A. K., et al. High aldehyde dehydrogenase and expression of cancer stem cell markers selects for breast cancer cells with enhanced malignant and metastatic ability. Journal of Cellular and Molecular Medicine. 13 (8), 2236-2252 (2009).
- Morel, A. P., et al. Generation of breast cancer stem cells through epithelial-mesenchymal transition. PloS One. 3 (8), 2888 (2008).
- Muntimadugu, E., Kumar, R., Saladi, S., Rafeeqi, T. A., Khan, W. CD44 targeted chemotherapy for co-eradication of breast cancer stem cells and cancer cells using polymeric nanoparticles of salinomycin and paclitaxel. Colloids Surf B Biointerfaces. 143, 532-546 (2016).
- Liu, S., et al. Breast cancer stem cells transition between epithelial and mesenchymal states reflective of their normal counterparts. Stem Cell Reports. 2 (1), 78-91 (2014).
- Munshi, A., Hobbs, M., Meyn, R. E. Clonogenic cell survival assay. Methods in Molecular Medicine. 110, 21-28 (2005).
- Shaw, F. L., et al. A detailed mammosphere assay protocol for the quantification of breast stem cell activity. Journal of Mammary Gland Biology and Neoplasia. 17 (2), 111-117 (2012).
- Shin, C. S., Kwak, B., Han, B., Park, K. Development of an in vitro 3D tumor model to study therapeutic efficiency of an anticancer drug. Molecular Pharmaceutics. 10 (6), 2167-2175 (2013).
- Khanna, C., Hunter, K. Modeling metastasis in vivo. Carcinogenesis. 26 (3), 513-523 (2005).
- Cheon, D. J., Orsulic, S. Mouse models of cancer. Annual Review of Pathology. 6, 95-119 (2011).
- Lyons, S. K. Advances in imaging mouse tumour models in vivo. Journal of Pathology. 205 (2), 194-205 (2005).
- Margaryan, N. V., et al. The Stem Cell Phenotype of Aggressive Breast Cancer Cells. Cancers. 11 (3), (2019).
- Ma, F., et al. Enriched CD44(+)/CD24(-) population drives the aggressive phenotypes presented in triple-negative breast cancer (TNBC). Cancer Letters. 353 (2), 153-159 (2014).
- Chatterjee, S., et al. Paracrine Crosstalk between Fibroblasts and ER(+) Breast Cancer Cells Creates an IL1β-Enriched Niche that Promotes Tumor Growth. iScience. 19, 388-401 (2019).
- Phan-Lai, V., et al. Three-dimensional scaffolds to evaluate tumor associated fibroblast-mediated suppression of breast tumor specific T cells. Biomacromolecules. 14 (5), 1330-1337 (2013).
- O'Brien, C. A., Kreso, A., Jamieson, C. H. Cancer stem cells and self-renewal. Clinical Cancer Research. 16 (12), 3113-3120 (2010).
- Hu, Y., Smyth, G. K. ELDA: extreme limiting dilution analysis for comparing depleted and enriched populations in stem cell and other assays. Journal of Immunological Methods. 347 (1-2), 70-78 (2009).
- Stewart, J. M., et al. Phenotypic heterogeneity and instability of human ovarian tumor-initiating cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (16), 6468-6473 (2011).
- Abraham, B. K., et al. Prevalence of CD44+/CD24-/low cells in breast cancer may not be associated with clinical outcome but may favor distant metastasis. Clinical Cancer Research. 11 (3), 1154-1159 (2005).
- Balic, M., et al. Most early disseminated cancer cells detected in bone marrow of breast cancer patients have a putative breast cancer stem cell phenotype. Clinical Cancer Research. 12 (19), 5615-5621 (2006).
- Charafe-Jauffret, E., et al. Aldehyde dehydrogenase 1-positive cancer stem cells mediate metastasis and poor clinical outcome in inflammatory breast cancer. Clinical Cancer Research. 16 (1), 45-55 (2010).
- Marcato, P., et al. Aldehyde dehydrogenase activity of breast cancer stem cells is primarily due to isoform ALDH1A3 and its expression is predictive of metastasis. Stem Cells. 29 (1), 32-45 (2011).
- Lacerda, L., Pusztai, L., Woodward, W. A. The role of tumor initiating cells in drug resistance of breast cancer: Implications for future therapeutic approaches. Drug Resist Updat. 13 (4-5), 99-108 (2010).
- Liu, S., Wicha, M. S. Targeting breast cancer stem cells. Journal of Clinical Oncology. 28 (25), 4006-4012 (2010).
- D'Angelo, R. C., et al. Notch reporter activity in breast cancer cell lines identifies a subset of cells with stem cell activity. Molecular Cancer Therapeutics. 14 (3), 779-787 (2015).
- Neve, R. M., et al. A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes. Cancer Cell. 10 (6), 515-527 (2006).
- Forozan, F., et al. Comparative genomic hybridization analysis of 38 breast cancer cell lines: a basis for interpreting complementary DNA microarray data. Cancer Research. 60 (16), 4519-4525 (2000).
- Lanier, L. L. Just the FACS. Journal of Immunology. 193 (5), 2043-2044 (2014).
- Ibrahim, S. F., van den Engh, G. Flow cytometry and cell sorting. Advances in Biochemical Engineering/Biotechnology. 106, 19-39 (2007).
- Shapiro, H. M. Flow Cytometry: The Glass Is Half Full. Methods in Molecular Biology. 1678, 1-10 (2018).
- Tsuji, K., et al. Effects of Different Cell-Detaching Methods on the Viability and Cell Surface Antigen Expression of Synovial Mesenchymal Stem Cells. Cell Transplantation. 26 (6), 1089-1102 (2017).
- Sun, C., et al. Immunomagnetic separation of tumor initiating cells by screening two surface markers. Scientific Reports. 7, 40632 (2017).
- Rodríguez, C. E., et al. Breast cancer stem cells are involved in Trastuzumab resistance through the HER2 modulation in 3D culture. Journal of Cellular Biochemistry. 119 (2), 1381-1391 (2018).
- Kim, D. W., Cho, J. Y. NQO1 is Required for β-Lapachone-Mediated Downregulation of Breast-Cancer Stem-Cell Activity. International Journal of Molecular Sciences. 19 (12), (2018).
- Xu, L. Z., et al. p62/SQSTM1 enhances breast cancer stem-like properties by stabilizing MYC mRNA. Oncogene. 36 (3), 304-317 (2017).
- Huang, X., et al. Breast cancer stem cell selectivity of synthetic nanomolar-active salinomycin analogs. BMC Cancer. 16, 145 (2016).
- Liu, T. J., et al. CD133+ cells with cancer stem cell characteristics associates with vasculogenic mimicry in triple-negative breast cancer. Oncogene. 32 (5), 544-553 (2013).
- Ponti, D., et al. Isolation and in vitro propagation of tumorigenic breast cancer cells with stem/progenitor cell properties. Cancer Research. 65 (13), 5506-5511 (2005).
- Velasco-Velázquez, M. A., Popov, V. M., Lisanti, M. P., Pestell, R. G. The role of breast cancer stem cells in metastasis and therapeutic implications. American Journal of Pathology. 179 (1), 2-11 (2011).
- Palomeras, S., Ruiz-Martínez, S., Puig, T. Targeting Breast Cancer Stem Cells to Overcome Treatment Resistance. Molecules. 23 (9), (2018).
- McClements, L., et al. Targeting treatment-resistant breast cancer stem cells with FKBPL and its peptide derivative, AD-01, via the CD44 pathway. Clinical Cancer Research. 19 (14), 3881-3893 (2013).
- Berger, D. P., Henss, H., Winterhalter, B. R., Fiebig, H. H. The clonogenic assay with human tumor xenografts: evaluation, predictive value and application for drug screening. Annals of Oncology. 1 (5), 333-341 (1990).
- Tian, J., et al. Dasatinib sensitises triple negative breast cancer cells to chemotherapy by targeting breast cancer stem cells. British Journal of Cancer. 119 (12), 1495-1507 (2018).
- Samoszuk, M., Tan, J., Chorn, G. Clonogenic growth of human breast cancer cells co-cultured in direct contact with serum-activated fibroblasts. Breast Cancer Research. 7 (3), 274-283 (2005).
- Linnemann, J. R., et al. Quantification of regenerative potential in primary human mammary epithelial cells. Development. 142 (18), 3239-3251 (2015).
- Xu, Y., Hu, Y. D., Zhou, J., Zhang, M. H. Establishing a lung cancer stem cell culture using autologous intratumoral fibroblasts as feeder cells. Cell Biology International. 35 (5), 509-517 (2011).
- Palmieri, C., et al. Fibroblast growth factor 7, secreted by breast fibroblasts, is an interleukin-1beta-induced paracrine growth factor for human breast cells. Journal of Endocrinology. 177 (1), 65-81 (2003).
- Bourguignon, L. Y., Peyrollier, K., Xia, W., Gilad, E. Hyaluronan-CD44 interaction activates stem cell marker Nanog, Stat-3-mediated MDR1 gene expression, and ankyrin-regulated multidrug efflux in breast and ovarian tumor cells. Journal of Biological Chemistry. 283 (25), 17635-17651 (2008).
- Yin, X., et al. Engineering Stem Cell Organoids. Cell Stem Cell. 18 (1), 25-38 (2016).
- Sachs, N., et al. A Living Biobank of Breast Cancer Organoids Captures Disease Heterogeneity. Cell. 172 (1-2), 373-386 (2018).
- Kim, M., et al. Patient-derived lung cancer organoids as in vitro cancer models for therapeutic screening. Nature Communications. 10 (1), 3991 (2019).
- Okano, M., et al. Orthotopic Implantation Achieves Better Engraftment and Faster Growth Than Subcutaneous Implantation in Breast Cancer Patient-Derived Xenografts. Journal of Mammary Gland Biology and Neoplasia. 25 (1), 27-36 (2020).
- Zhang, Y., et al. Establishment of a murine breast tumor model by subcutaneous or orthotopic implantation. Oncology Letters. 15 (5), 6233-6240 (2018).
- Zhang, W., et al. Comparative Study of Subcutaneous and Orthotopic Mouse Models of Prostate Cancer: Vascular Perfusion, Vasculature Density, Hypoxic Burden and BB2r-Targeting Efficacy. Scientific Reports. 9 (1), 11117 (2019).
- Kim, R., Emi, M., Tanabe, K. Cancer immunoediting from immune surveillance to immune escape. Immunology. 121 (1), 1-14 (2007).
- Rosato, R. R., et al. Evaluation of anti-PD-1-based therapy against triple-negative breast cancer patient-derived xenograft tumors engrafted in humanized mouse models. Breast Cancer Research. 20 (1), 108 (2018).
- Choi, Y., et al. Studying cancer immunotherapy using patient-derived xenografts (PDXs) in humanized mice. Experimental and Molecular Medicine. 50 (8), 99 (2018).
- Meraz, I. M., et al. An Improved Patient-Derived Xenograft Humanized Mouse Model for Evaluation of Lung Cancer Immune Responses. Cancer Immunol Res. 7 (8), 1267-1279 (2019).
- Wege, A. K. Humanized Mouse Models for the Preclinical Assessment of Cancer Immunotherapy. Biodrugs. 32 (3), 245-266 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon