このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
モルフォリノアンチセンスオリゴヌクレオチドとmRNAのゼブラフィッシュ胚へのマイクロインジェクションによる心臓発生における特異的遺伝子機能の解明
要約
本プロトコルは、代表的な心臓遺伝子に対する翻訳ブロッキングモルフォリノの設計、調製、およびマイクロインジェクションについて記載しています。Heart And Neural Crest Derivatives Expressioned2 (hand2) を新たに受精したゼブラフィッシュの胚の卵黄に注入し、遺伝子機能をノックダウンします。また、この遺伝子産物をコードするmRNAの同時注入によるこれらの「モルファント」の一過性の救助も示しています。
要約
モルフォリノオリゴマーベースのノックダウンシステムは、さまざまな遺伝子産物の喪失や発現低下による機能を特定するために使用されてきました。 モルフォリノス(MO)は、酵素分解を受けにくいため、DNAオリゴよりも生物学的安定性に優れています。 最適な効果を得るために、MOは1〜4細胞期の胚に注入されます。 ノックダウンの時間的有効性はさまざまですが、MOは最終的に希釈により効果を失うと考えられています。モルフォリノの希釈量と注入量は、オンターゲット効果を維持しながらオフターゲット効果の発生を最小限に抑えるために厳密に制御する必要があります。.CRISPR/Cas9などの追加の補完的ツールを目的の標的遺伝子に対して実行して、変異株を生成し、これらの系統でモーファント表現型を確認する必要があります。この記事では、hand2に対する翻訳阻害モルフォリノを1-4細胞期のゼブラフィッシュ胚の卵黄にマイクロインジェクションして、 hand2 の機能をノックダウンし、対応するcDNAをコードするmRNAを同時注入することでこれらの「モルファント」をレスキューする方法を紹介いたします。その後、モルホリンマイクロインジェクションの有効性は、最初に卵黄中のモルホリノの存在(フェノールレッドと同時注入)を確認し、次に表現型分析によって評価されます。また、ノックダウン効果を検証するための心臓機能解析についても検討します。最後に、モルフォリン誘発性遺伝子翻訳のウェスタンブロッティングによるブロックの影響を評価することについて説明する。
概要
ゼブラフィッシュを心血管疾患の研究モデルとして利用すると、遺伝子機能の高い保存、光透過性、急速な心血管発達、従来のin vivoモデルと比較して低コストなど、さまざまな利点があります1。モルフォリノオリゴヌクレオチド(MO)は、ゼブラフィッシュモデルに最も一般的に使用されるアンチセンス遺伝子ノックダウンツールです。MOは、表現型を決定したり、遺伝子機能を調べたりするために頻繁に使用されます。ジェームズ・サマートン博士は当初、ヒトの発生障害の治療法を開発する試みとして、mRNA翻訳のin vivo阻害のためのモルフォリノ送達システムを開発しました2,3。MOは、in vitroおよびin vivoモデル生物が遺伝子をノックダウンし、このノックダウンが表現型に及ぼす影響を調査するために使用されてきました。これは、特定の臓器、例えば心臓の発達の変化を観察することによって行われます。WTゼブラフィッシュの胚における心臓特異的遺伝子のノックダウンは、適切な心拍の失敗につながり、これらの遺伝子が心臓の発達に不可欠な機能であることを証明しています4,5。これらの表現型は、特定の遺伝子に対するmRNAの同時注入によって救出されました。心筋トロポニンT(Tnnt2)を用いた研究では、完全長tnnt2 mRNAの発現がモルフォリノノックダウンによる肉腫表現型を救うことができることが示されました6。別の研究では、cmcl2モーファントにおける調節性ミオシン軽鎖オルソログ(cmlc2)mRNAの過剰発現により、AバンドとZディスクの完全性が回復できることが明らかになった7。
MOは、プレmRNAスプライシングを標的とするか、翻訳をブロックすることにより、遺伝子発現をノックダウンするために一般的に使用されます。スプライスブロッキングMOは、スプライソームを阻害することにより、pre-mRNAに結合し、阻害します。翻訳ブロッキングは、MOが相補的mRNAの5'非翻訳領域に結合してリボソームの集合を阻害するときに発生します。MOは、 in vivo モデルの遺伝子発現をノックダウンするために最も広く使用されている遺伝子特異的な方法です。また、細胞培養に使用される最も効率的なmRNAブロッキング剤でもあります。モルフォリノ自体は、典型的にはモルフォリノサブユニット塩基の短鎖(約25)からなる。各MOサブユニットには、核酸塩基、モルホリン環、および非イオン性ホスホロジアミデートが含まれます。2種類のMOの作用機序が異なるため、ノックダウンの有効性を検証するために異なる試験が必要です。翻訳ブロッキングMOの場合、ATG翻訳開始部位の閉塞により目的のタンパク質が産生されないため、ウェスタンブロット分析が最も信頼性の高い有効性の試験です。MOは標的mRNAを直接分解しません。代わりに、それらは特定の領域に結合し、自然に分解されるまで発現を阻害します。しかし、スプライスブロッキングMOは、スプライス修飾を誘導することでpre-mRNAを修飾し、逆転写酵素ポリメラーゼ連鎖反応(RT-PCR)とゲル電気泳動によってアッセイすることができます。
MOスクリーニングプロセスの3つの重要な部分を標準化する必要があります:(i)MO用量曲線は、表現型の認識に合わせて調整する必要があります。線量曲線は、表現型の「シグナル」とオフターゲットの「ノイズ」を最適化する能力を改善するためにテストされた各MOの致死量50(LD50:注入された胚の50%が死亡する線量)も示しています3。(ii)適応された表現型命名法は、十分に文書化されるべきである。正確で理解しやすい表現型の説明は、既存の文献と研究者の経験に基づいて広範な説明を提供し、胚を直接検査しなかった人々の間での情報共有を促進するために重要です。(iii) 明確に定義された言語を持つことで、Morpholino Database8からデータを一元的に収集することが容易になります。
心筋疾患のノックダウン研究では、MOノックダウン実験が心血管系の機能に及ぼす影響を判断するために、動物の心臓活動と血流動態をモニタリングする必要があります。このような解析には、心血管系を高解像度でリアルタイムに可視化する必要があります。ゼブラフィッシュの皮膚は、開発の最初の1週間は透明であるため、顕微鏡で心臓や血液の循環を可視化することができます。心機能の評価のために、最も計算された生理学的パラメータは、心拍数と心拍出量、フラクショナル短縮、フラクショナルエリア変化、駆出率です。血流速度は、移動する赤血球を追跡することで測定でき、これらの測定値は、内皮細胞の重要な機械生物学的因子であるせん断応力レベルを決定するために使用されます。このような評価には、ハイスピードカメラを搭載した倒立顕微鏡や実体顕微鏡で、心臓の鼓動や血液の流れをタイムラプス動画で記録する必要があります。
この論文では、受精したばかりのゼブラフィッシュ胚の卵黄に、目的の遺伝子に対する翻訳阻害モルフォリノを設計、調製、およびマイクロインジェクションして遺伝子機能をノックダウンする方法を示します。また、この遺伝子をコードするmRNAの同時注入によってこれらの「モルファント」を救うことも示します。次に、表現型の特性評価と心臓の構造的および機能的分析を通じて、モルフォリン微量注射の有効性を分析します。このアプローチは、広く研究されている心臓遺伝子hand2で実証されます。
プロトコル
すべての実験は、QUのIACUCの規制の下で、人道的な動物管理の受け入れられた基準に従って実施されました。動物は、カタール大学生物医学研究センター(QU-BRC)のゼブラフィッシュ施設で飼育されました。これらの実験的研究で使用されたすべての動物は、受精後3日未満(dpf)でした。
注:各実験グループについて、統計的な厳密さのために少なくとも30個の胚を使用することをお勧めします。実験グループは以下の通りです。
コントロールグループ: このグループには、注射なしで卵水で培養された胚が含まれます。ここでの結果が制御ベースラインを形成します。
ネガティブコントロールグループ: このグループには、スクランブルMOを注入した卵水で培養した胚が含まれます。
注入されたグループ: このグループには、表現型を救うために hand2 MOのみを注入した胚と hand2 MOをhand2 mRNAとともに注入した胚が含まれます。ここでの結果は、観察された表現型が注射されたMOによって現れたことを裏付けるものです。実験群の比較により、 hand2 の抑制と救助が心機能に及ぼす影響を正確に評価することができます。
1.手のためのモルフォリノデザイン 2。
注:MO配列は、文献9,10,11から適合させることができます。あるいは、これらのオリゴはGene-toolsによってオンラインで設計することもできます。Gene-toolsは、無料で高速なオンラインデザインサービスを提供しており、Webサイト12からアクセスできます。カスタムMOは、配列情報やアクセッション番号など、目的の遺伝子に関する情報を提供することにより、容易に設計できます。次の具体的な手順は、ゼブラフィッシュの hand2 に対する MO の設計方法をまとめたものです。
- まず、NCBIのデータベースから目的の遺伝子の詳細を検索します。 hand2の場合、ゼブラフィッシュ13の場合。
- NCBI14からGenBank mRNA転写産物IDを取得します。
- NCBI15からmRNA配列を取得します。
注:ゼブラフィッシュhand2遺伝子のモルフォリノ(MO)修飾アンチセンスオリゴヌクレオチドが利用可能であり、以前に発表されました。ハンド2MO配列9,10,11:5'-CCTCCAACTAAACTCATGGCGACAG-3'、研究で使用されたMO配列は、材料の表に記載されています。 - 遺伝子ツール16から、ミスマッチまたはスクランブルされたMO(ランダムな塩基対の変化を伴う類似の配列)のいずれかからネガティブコントロールを得る。これは、特定のMO注射で観察された表現型の特異性を証明し、観察された影響が注射手順のアーティファクトによるものであるリスクを最小限に抑えるのに役立ちます。.
2. モルフォリノ注射剤の調製
- ストックの準備
- MOストックをddH2Oで溶解します。ジエチルピロカーボネート(DEPC)は、MOを損傷し、胚に毒性を示す可能性があります。したがって、DEPCフリーのddH2Oを使用してください。注文後、MOは300 nMの濃度でバイアルに入れて配送されます。
注:Gene Toolsは1 mMのストック溶液(~8 ng/nL)を推奨していますが、特にモルフォリノが高用量を必要とする場合や他のMOと混合される場合は、低すぎる可能性があります。したがって、さまざまな濃度のMOストック溶液(2または3 mM)の作成に最適です。 - 0.2 mMのワーキング溶液を調製するには、ワーキング溶液をサブ希釈します(つまり、2 mMストックの場合は1:10希釈)。ダニオー溶液(7 μL)、ストックモルフォリノ(1 μL)、およびフェノールレッド(10xストックから2 μL)を加えて、10 μLの作業溶液を調製します。フェノールレッドは、溶液に濃い赤色を与え、胚に注入された材料を追跡するのに役立ちます。
- MOは、蒸発を防ぐために密閉容器に室温(RT)で保管してください。溶液が沈殿する可能性があるため、MOをベンチトップの氷の上に置いたままにしないでください。RTの取り扱いが最適です。
注:Gene Toolsは、MOsに対して2つの保存方法のいずれかを推奨しています:作業溶液は-20°C(数ヶ月から数年)に保持するか、密閉チューブ内で長期保存するためにRTすることができます。-20°Cに保った場合は、オリゴヌクレオチド溶液を65°Cで10分間加熱し、ボルテックスしてモルフォリノを完全に溶解してから使用してください(常温に保温した場合は温める必要はありません)。
- MOストックをddH2Oで溶解します。ジエチルピロカーボネート(DEPC)は、MOを損傷し、胚に毒性を示す可能性があります。したがって、DEPCフリーのddH2Oを使用してください。注文後、MOは300 nMの濃度でバイアルに入れて配送されます。
- mRNAのin vitro転写
注: in vitro 転写は、モルフォリノを使用してゼブラフィッシュ hand2 をノックダウンすることによる表現型を救うために、ゼブラフィッシュ胚にマイクロインジェクションされるプラスミドHAND2(NM_021973)ヒトタグ付きORFクローンからHand2のmRNAを生成するために使用され、この研究で使用されたプラスミドの詳細は 、材料の表に記載されています。- 適切な制限酵素でプラスミドDNAを直鎖化し、DNA精製キット(材料表)を使用して精製します。精製した直鎖状化DNAを in vitro 転写のテンプレートとして使用し、製造元の指示に従って注射用のmRNAを生成します(材料表)。
- 最後に、目的の遺伝子に250 pgのヒトmRNAを注入して表現型のレスキューを行い、表現型の欠如が本当にMOによるゼブラフィッシュ遺伝子の喪失によるものかどうかをテストします。
- 注入の準備
- マイクロピペットプーラー(材料表)を使用して、手順2.3.2〜2.3.6に従って針を引っ張ります。
- フィラメント付きの1mmキャピラリーチューブとマイクロピペットプーラーを使用します。マシンの電源を入れ、蓋を開けます
- モード選択つまみを NO.2 ヒーターに回します。 NO.2 ヒーターを使用して、つまみを調整して熱を68°Cにします
- モード選択ノブを ステップ1 の位置に戻します。ウエイトは4種類(ライト2種/ヘビー2種)を使用
- キャピラリーをホルダーに置き、蓋を閉めます。 スタート ボタンを押して、引っ張られた針を取ります。
注:これにより、長いテーパーを持つ毛細血管針が生成されます - 鉗子を使用して、針の先端を短く切ります。引っ張った針の先端をゼブラフィッシュの胚に浸透しやすいようにキャピラリーベベルで約30秒間研ぎ、先端の直径が約15〜25μmになります。
- 手順2.3.8-2.3.13に従って注射量を推定するために針を校正します。
- mRNA/モルフォリノ溶液を針にロードする前に短時間混合し、針を詰まらせる可能性のある粒子を完全に溶解します。
- マイクロニムチップのピペットチップを使用して、注射針の後ろ部分に~3〜4μLのMO溶液を加えます。注射針には、毛細血管の埋め戻しを容易にするための小さなトレンチが装備されています。
- 注入針に気泡がないことを確認します。針をマイクロインジェクターに取り付けます。
- 針の先端が詰まっていないか、注射ペダルを踏むことでMO溶液が針から流れ出せるか確認してください。
- 液滴サイズを測定して、注入量を校正します。針の先をマイクロメータスライドに鉱物油で入れます。インジェクションペダルを踏み、噴射(Eject)圧力(psi)および/または注入時間(ms)を調整して、噴出する液滴の直径が0.5 nLに対応する0.1 mmになるまで調整します。
- 注入量が約0.5〜1nLであることを確認してください。注入(イジェクト)、圧力(psi)、または時間(ms)を変更して、容量を調整します。
- ピコリットルインジェクターのセットアッププロトコルについては、手順2.3.15-2.3.16に従ってください
- ゼブラフィッシュの注射にはピコリットルインジェクターを使用してください。インジェクターのフィードポンプをオンにします。
- 以下に示す要件に従ってインジェクターを準備します。
- Pバランス: Pバランスが0前後であることを確認してください。卵黄が針に逆流し、針のMO溶液が希釈されるのを防ぐために、わずかにマイナスであることを確認してください。逆に、背圧が高すぎると、ペダルに圧力がかからなくてもMOが絶えず針から流れ出し、注入された用量と観察された表現型にばらつきと不整合が生じます。
- Pインジェクション: 20〜25psiが理想的です。これを変更して射出量を調整します。これは 10 から 30 psi の範囲ですが、~20 psi から始めて、目的のボリュームが供給されるかどうかを確認します。
- インジェクション時間: 注入時間を300 msに短縮してください。注入量を調整する前に、注入時間を調整してください。
- モルフォリノ注射の場合は、手順2.3.18-2.3.19に従います。
- 次のように注入チャンバーを準備します:0.6 gのアガロースと30 mLのE3培地から2%アガロースを作ります。アガロースをペトリ皿に注ぎ、TU-1射出成形金型を使用してアガロースに射出成形金型を置きます。冷凍庫で冷却すると、胚を配置および安定化するための溝が形成されます(図1A)。
注:60x E3Mストック溶液レシピは、5.0 mM NaCl、0.17 mM KCl、0.16 mM MgSO4·7H2O、0.4 mM CaCl 2.2H2O、1 L ddH2O、最終pH 7.6で構成されています。 - 採取した胚をトランスファーピペットを使用して溝に移し、胚の浮遊を防ぐために胚の周りの余分なE3培地をすべて取り除きます(図1B)。
3. 卵黄へのMOおよびmRNA溶液の注入
- 針を絨毛膜に挿入し、細胞/卵黄の境界近くから胚側に注入し、注入ペダルを押します(図1C)。
注:注入されたMO溶液/フェノールレッドは、卵黄内ですぐには拡散しません。卵黄に赤い斑点が観察され、徐々に消えていきます。注入された材料は、胚サイズの10%を超えてはなりません。MOは、新たに産まれたゼブラフィッシュの卵の卵黄に最大8細胞段階でマイクロインジェクションすることができる、なぜなら、MOは、発生中の細胞17,18によって卵黄嚢の細胞質の流れに容易に取り込まれるからである。プラスミドまたはキャップ付きmRNA注入では、1細胞期胚の単一割球に注入する必要があります。 - 胚をE3培地を入れたシャーレに胚を移し、E3培地を毎日補充しながら、3 dpfまで28°Cでインキュベートします。その段階で、顕微鏡を使用して表現型評価を行います。
注:MO溶液は、特定の表現型を誘導するために必要な最低濃度を確立するために、調査ごとに慎重に希釈する必要があります。各MOの胚致死率も決定する必要があります。高濃度(MOに応じて4〜10 ng以上)では、MOは脳死や一般的な細胞死などの非特異的な影響を誘発する傾向があります。 - 同様に、ヒト hand2のmRNA250 pgにMOを同時注入し、ゼブラフィッシュ hand2 遺伝子の欠損によって誘発された表現型がMOによって救出されることを確認しました。
4. モルフォリノノックダウンの成功を確認するためのウェスタンブロット
- 特定の時点(48 hpf、72 hpf)で胚を採取し、必要に応じてPronase酵素を使用してデコリオネートします。
- 胚を脱ヨークして、以前に知られていた方法論19を使用してビテロゲニンを除去します。
注:ビテロゲニンは、成長する胚の栄養源として機能するリン脂質糖タンパク質です。高分解能の2Dゲル電気泳動と大幅に改善されたウェスタンブロッティングは、胚を脱ヨークし、ビテロゲニンを除去することによって可能になります。 - 前述したようにウェスタンブロッティングを行う20。
5.心臓の構造と機能の評価:
- ゼブラフィッシュのライブイメージング:心室のイメージング
注:ゼブラフィッシュ胚21,22の鼓動する心臓を視覚化することにより、いくつかの心臓機能/血行動態パラメータを計算できます。これらのパラメータには、心拍出量(CO)、駆出率(EF)、一回換気量(SV)、フラクショナル短縮(FS)、およびフラクショナルエリアチェンジ(FAC)が含まれます(ゼブラフィッシュ胚の心機能評価に関する詳細なプロトコルを含む以前の論文を参照してください22,23)。次の手順では、ゼブラフィッシュの胚の~3 dpf(これらの生物の皮膚が透明な段階)でこれらのパラメータを計算する方法を簡単に説明し、明視野顕微鏡による可視化を可能にします。- 3%メチルセルロース溶液(RT)を凹型ウェルイメージングスライドに滴下します。
- 適切なプラスチック製スポイトを使用して、ゼブラフィッシュの胚をウェルに配置します(図2A、B)
注意: 井戸をいっぱいにしすぎると、魚が井戸から移動する可能性があります。 - 3%メチルセルロース滴をE3培地と穏やかに混合して、孵化した胚を安定させます。
注:3%メチルセルロース溶液を調製するには、3 gのメチルセルロース粉末を100 mLのPBSまたは他の封入剤にフラスコで溶解します。混合物のフラスコに攪拌磁石を置き、フラスコを磁気攪拌プレートの上に置きます。速度を「低速」に設定し、4°Cで~1日間保ち、すべての塊を溶解します。メチルセルロースが完全に溶解したら、小さなチューブに分注し、-20°Cで保存します。 - 魚を左側に配置し、右側を上にして前面を左に向けて、心室の明確なイメージングを容易にします(図2C)。
- 顕微鏡で、胚の心臓を100倍の倍率で拡大し、~5秒間記録を開始します。心室の境界がイメージングウィンドウの内側にあることを確認します(図2D)。
- 高速度カメラと実体顕微鏡を用いて、全胚(図2C)、心室を鼓動させる(図2D)、背側大動脈や後枢機卿静脈(図2E)などの主要血管内の赤血球(RBC)を約100フレーム/秒(fps)でタイムラプス動画を撮影し、心機能解析を行います。
- ムービーを AVI ムービー形式または TIFF(または JPEG)イメージ シーケンス形式で保存します。
- 心拍数を計算する
- 2 つの連続したフレームを記録するのに必要な時間を計算します。100 fps の場合、時間間隔は 0.01 秒に等しくなります。
- 記録された心周期(すなわち、拡張末期または収縮期末)から既知のポイントを選択します。サイクルを繰り返すために必要なフレーム数を計算します。
- ステップ 5.2.1 の時間間隔にステップ 5.2.2 のフレーム数を掛けます。結果は、1 つのハートビートの期間 (秒単位) です。
- 1分あたりのビート数を計算するには、60を前の手順で取得した数値で割ります。正常な3 dpf胚の場合、これは約150 bpm(2.5 Hz)である必要があります。
注意: 多くのソフトウェアアプリケーションは、ViewPoint24 やDanioVision25などの心拍記録から心拍数を自動的に計算できます。 - 約100fpsのタイムラプス動画で心臓の構造を解析し、心臓全体を調べることで、心浮腫などの構造上の欠陥がないか確認します。
注: 図3A は正常な3dpf胚心臓を示し、 図3B は心浮腫と心臓ループ欠損(細長い心室)を持つ3dpf心臓を示しています。
- 小数面積変化(FAC)を計算します。
注: フラクショナル エリア チェンジ (FAC) は、心室拡張末期領域と収縮末期領域を比較して、心室の収縮性を評価するために使用されるパラメーターです。- 約 100 fps のタイムラプス ムービーを録画して、1 つの心周期を表すフレームを決定します。収縮期末期と拡張期末の両方の終わりを示すフレームを抽出します。
- ImageJまたは同様の画像解析ソフトウェアを使用して、拡張末期領域(EDA)と収縮末期領域(ESA)の両方を計算します。
- 次の式を使用して、FAC 22,26,27 を計算します。
FACの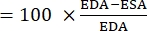
- 分数短縮 (FS) を計算します。
注:フラクショナルショートニング(FS)は、心室の収縮性を評価するために使用される別のパラメーターです。FSを決定するには、心室の拡張末期および収縮末期の直径を測定する必要があります21。- 手順5.3.1を繰り返します
- ImageJまたはその他の同等の画像解析プログラムを使用して、拡張末期Dd および収縮期末Ds 点の心室壁の直径を測定します。ほとんどの場合、短軸の直径を使用して分数平滑性(FS)を決定します(図4)。
- 次の式を使用して、FS22,27を計算します。
FSの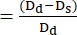
- ストローク容積(SV)の計算:
注:各心拍について、SVは心室から送り出される血液の量であり、これは心室の拡張末期および収縮期末の容積から容易に計算できる28。- 手順 5.3.1 と 5.3.2 を繰り返します
- 図4に示すように、拡張末期(DL)と収縮期末(DS)の直径を測定します。
注:ゼブラフィッシュの心臓の心室が拉致した球状体21,22の形状をしていると仮定すると、心室容積は次の式を使用して計算されます。
容積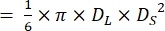
SVは、以下の式27に従って計算できます。正常なSV範囲は、2-6dpf胚29で0.15-0.3 nLである。ここで、EDVは拡張末期容積、ESVは収縮末期容積です。
SV = (EDV - ESV)
- 駆出率 (EF) を計算します。
注:EFは、各心拍28で心室から排出される血液の割合として定義されます。- 上記の式から、魚のEDVとESVを抽出します。
- EF を次のように計算します22,27:
EF (%)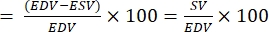
- 心拍出量(CO)を計算します。
注:COは、心臓28によって送り出される血液の量です。COは、2〜6dpfの胚29に対して10〜55 nL / minの値を持っています。- 前のサブセクション5.5および5.2で説明したように、SVとHRを計算します。
- 次の式を使用して、CO22,27を計算します。
CO(nL/分)= SV(nL/拍)×HR(拍/分)。
- RBCの速度を測定します。
注:細胞速度の決定は、流量、血管径を評価し、血管の内皮細胞にかかるせん断応力を計算するために不可欠です(つまり、せん断応力は、一方、血液の移動によって内皮細胞に課せられる摩擦力です)。2〜5 dpf胚の場合、平均赤血球(RBC)速度は約300〜750μm /s29です。RBCの速度を測定するには:- 顕微鏡下で、動物の尾を100倍の倍率で拡大し、この倍率で赤血球の動きが見えるはずです(図5A、B)。
- 約8秒間録音を開始します。最も重要な2つの軸動脈である背側大動脈(DA)と後方枢機卿静脈(PCV)がイメージングウィンドウ内で100〜120fpsであることを確認します。シーケンシャルフレームから個々のセルを追跡します(図5A、B)。
- ImageJまたは他の同様の画像解析ソフトウェアを使用して、複数のフレームを抽出します。
- 個々の赤血球が移動する距離の差(Δx)を計算します。
- 連続するフレーム間の時間差 (Δt) を求めます。
- RBC 速度22,27 には、次の式を使用します。
RBC速度(μm / s)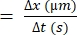
注:最大速度および平均速度は、個々のセル21の連続フレームの繰り返しから抽出することができる。RBCの速度は、血流の速度も表します。あるいは、さまざまな市販のソフトウェアアプリケーションを使用して、RBCの速度を自動的に測定することもできます。ViewPoint24 と DanioVision25 には、このようなアプリケーションがあります (図 5C)。さらに、TrackMate30 やMTrackJ31など、100fps以上で録画された動画に対応したImageJ搭載のプラグインはほとんどありません。
- 測定されたセル速度からのせん断応力の計算
血管内で測定されたRBC速度も血流速度を表します(図5C)。これらの測定値から、Poiseuille Flow の仮定22,27 を使用して、対象の容器内のせん断応力 τ を次のように計算します。
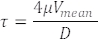
ここで、Vは平均血流速度(μm/s)、μは血液粘度(dynes/cm2)、Dは血管径(μm)です。3 dpf胚の場合、DA 22,26,27のせん断応力は約4ダイン/cm 2です。
結果
図6のグラフは、HAND2特異的MOおよびコントロールスクランブルMO注入胚の両方について、24、48、および72hpfで生存する胚の平均割合を示しています。1 mM(8 ng/μL)および0.8 mM(6.4 ng/μL)のMO注入胚は、対照のスクランブルMO注入胚と比較して生存率の有意な低下を示しました。これは、致死性または奇形が観察された各測定時点で観察されました。そ?...
ディスカッション
モルフォリノ(MO)技術は、ゼブラフィッシュ、ゼノープス、ウニ、そして最近では細胞培養モデルシステムで広く使用されています。ほとんどの方法では、利点とともに、実験者が注意すべき落とし穴もあります。MO技術の主要な落とし穴の1つは、MO媒介遺伝子ノックダウンアプローチによって観察される表現型効果が、一次遺伝子産物に関連する機能の喪失によるも...
開示事項
著者は、金銭的利益またはその他の利益相反を宣言します。
謝辞
この記事の公開は、BARZAN HOLDINGSの寛大な支援を受けて行われました。RRは、Department of Pediatrics and Children's HospitalからのR61HL154254と資金によって部分的にサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| Acrylamide 40% | Sigma | Sigma, cat. no. C977M88 | |
| Agarose | Sigma-Aldrich | Sigma-Aldrich cat. no A9539-250G | |
| All Prep DNA/RNA Mini Kit | Qiagen | Qiagen cat. no. 80204. | |
| alpha Tubulin | Abcam | Abcam- ab4074 | Rabbit polyclonal to alpha Tubulin lot GR3 180877-1 (50 kDa) |
| Ammonium persulfate molecular grade | Sigma | Sigma, cat. no C991U65 | |
| BV10 capillary beveller | Sutter Instruments Product | Sutter Instruments Product Catalog # BV10 | |
| Chemiluminescence Imaging Gene Gnome | SYNGENE | SYNGENE | |
| Cleaver Scientific Blotting | CVS10D_OmniPAGEMini | CVS10D_OmniPAGEMini | |
| Coomassie | Thermo Fisher | Thermo Fisher cat. no C861C44 | |
| Electrochemiluminescence (ECL) kit | Abcam Biochemicals | Abcam Biochemicals cat. no ab65623 | |
| Glycine | Sigma | Sigma, cat. no C988U91 | |
| Goat anti Rabbit | Abcam | Abcam- ab6721 | Goat Anti-Rabbit IgG H&L (HRP) 2nd antibodies lot GR3179871-1 |
| HAND2 | Gene tools | Custom made for HAND2 (NM_021973) | 5'-CCTCCAACTAAACTCATGGCGAC AG-3' |
| Hand2 | Abcam | Abcam- ab10131 | Rabbit polyclonal Anti-HAND2 antibody lot GR143200-9 (24- 26 kDa) |
| HAND2 (NM_021973) Human Tagged ORF Clone | OriGene Technologies, Inc | RC224436L3 | Vector: pLenti-C-Myc-DDK-P2A-Puro (PS100092) |
| IBI DNA/RNA/Protein Extraction Kit | IBI Scientific | IBI Scientific cat. no -r IB47702 | |
| Imaging System | iBright | iBright CL1000 Imaging System | |
| Isopropanol | Sigma-Aldrich | Sigma-Aldrich cat. no 278475-2L | |
| Laemmli sample loading buffer (4x) | Sigma-Aldrich | Sigma-Aldrich cat. no 70607 | |
| Mercaptoethanol | Sigma | Sigma, cat. no M6250-1L | |
| Microplate Spectrophotometer with the Gen5 Data Analysis software interface | Epoch | Epoch | |
| Microscope | Ziess SteREO Lumar V12 Flourescence Microscope | Ziess SteREO Lumar V12 Flourescence Microscope | |
| Mineral oil | Fisher Scientific | Fisher Scientific cat. no 0121-1 | |
| mMESSAGE mMACHINE T7/T3/SP6 Transcription Kit | Thermo Fisher | Thermo Fisher cat. no.AM1340 | for mRNA generation |
| Nuclease-free water | New England Biolabs | New England Biolabs cat. no B1500L | |
| PC-100 Micropipette Puller | NARISHIGE GROUP Product | NARISHIGE GROUP Product Catalog # PC-100 | |
| Phenol red | Sigma | Sigma, cat. no. P-0290 | |
| Picolitre Injector | Harvard Apparatus | Harvard Apparatus cataloge # PLI-90A | |
| Pierce Bicinchoninic acid assay (BCA) Protein Assay kit | Thermo Fisher | Thermo Fisher cat. no 23227 | |
| PMSF, Protease inhibitor as protease inhibitors | Thermo Fisher | Thermo Fisher cat. no 36978 | |
| Ponceau S | Sigma-Aldrich | Sigma-Aldrich cat. no 10165921001 | |
| Protease Inhibitor Cocktail | Thermo Fisher | Thermo Fisher cat. no 88668 | |
| Protein ladder | SMOBiO | SMOBiO cat. no PM2500 | |
| Radioimmunoprecipitation Assay (RIPA) | Thermo Fisher | Thermo Fisher cat. no 89900 | |
| Ringer’s solution | Thermofisher | Catalog No.S25513 | |
| SDS | Sigma | Sigma, cat. no 436143 | |
| Standard Control | Gene tools | SKU: PCO-StandardControl-100 | 5'-CCTCTTACCTCAGTTACAATTTAT A-3'- that targets a human beta-globin intron mutation |
| Stripping buffer | Sigma-Aldrich | Sigma-Aldrich cat. 21059 | |
| Temed | IBI scientific | IBI scientific cat. no C000A52 | |
| Tris Base | Thermo Fisher | Thermo Fisher cat. no BP-152-500 | |
| Tween | sigma life science | sigma life science cat. no P2287 | |
| Zebra Box Revolution-Danio Track system chamber with the EthoVision XT 11.5 software | Noldus Information Technology, NL | Noldus Information Technology, NL | |
| Zeiss Axiocam ERc 5s | Zeiss | Stemi 508 Zeiss | |
| Zeiss Stemi 2000-C | Zeiss | Stemi 2000-C |
参考文献
- Bakkers, J. Zebrafish as a model to study cardiac development and human cardiac disease. Cardiovascular Research. 91 (2), 279-288 (2011).
- Bill, B. R., Petzold, A. M., Clark, K. J., Schimmenti, L. A., Ekker, S. C. A primer for morpholino use in zebrafish. Zebrafish. 6 (1), 69-77 (2009).
- Stainier, D. Y., et al. Guidelines for morpholino use in zebrafish. PLoS Genetics. 13 (10), 1007000 (2017).
- Zhou, B., et al. Epicardial progenitors contribute to the cardiomyocyte lineage in the developing heart. Nature. 454 (7200), 109-113 (2008).
- Eve, A. M., Place, E. S., Smith, J. C. Comparison of Zebrafish tmem88a mutant and morpholino knockdown phenotypes. PLoS One. 12 (2), 0172227 (2017).
- Huang, W., Zhang, R., Xu, X. Myofibrillogenesis in the developing zebrafish heart: A functional study of tnnt2. Developmental Biology. 331 (2), 237-249 (2009).
- Chen, Z., et al. Depletion of zebrafish essential and regulatory myosin light chains reduces cardiac function through distinct mechanisms. Cardiovascular Research. 79 (1), 97-108 (2008).
- Bedell, V. M., Westcot, S. E., Ekker, S. C. Lessons from morpholino-based screening in zebrafish. Briefings in Functional Genomics. 10 (4), 181-188 (2011).
- Reichenbach, B., et al. Endoderm-derived Sonic hedgehog and mesoderm Hand2 expression are required for enteric nervous system development in zebrafish. Developmental Biology. 318 (1), 52-64 (2008).
- Maves, L., Tyler, A., Moens, C. B., Tapscott, S. J. J. Pbx acts with Hand2 in early myocardial differentiation. Developmental Biology. 333 (2), 409-418 (2009).
- Hinits, Y., et al. Zebrafish Mef2ca and Mef2cb are essential for both first and second heart field cardiomyocyte differentiation. Developmental Biology. 369 (2), 199-210 (2012).
- . Gene Tools, custom-sequence Morpholinos Available from: https://oligodesign.gene-tools.com/ (2011)
- . hand2 heart and neural crest derivatives expressed 2 [ Danio rerio (zebrafish) Available from: https://www.ncbi.nlm.nih.gov/gene/58150 (2021)
- . Danio rerio heart and neural crest derivatives expressed 2 (hand2), mRNA Available from: https://www.ncbi.nlm.nih.gov/nuccore/NM_131626.3 (2021)
- . NCBI reference sequence -Danio rerio heart and neural crest derivatives expressed 2 (hand2), mRNA FASTA Available from: https://www.ncbi.nlm.nih.gov/nuccore/NM_131626.3?report=fast (2021)
- . Prepared control oligos Available from: https://store.gene-tools.com/prepared-control-oligos (2021)
- Schubert, S., Keddig, N., Hanel, R., Kammann, U. Microinjection into zebrafish embryos (Danio rerio)-a useful tool in aquatic toxicity testing. Environmental Sciences Europe. 26 (1), 32 (2014).
- Moulton, J. D. Guide for morpholino users: toward therapeutics. Journal of Drug Discovery, Development, and Delivery. 3 (2), 1023 (2016).
- Link, V., Shevchenko, A., Heisenberg, C. -. P. Proteomics of early zebrafish embryos. BMC Developmental Biology. 6 (1), 1 (2006).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. Journal of Visualized Experiments: JoVE. (44), e2359 (2010).
- Shin, J. T., Pomerantsev, E. V., Mably, J. D., MacRae, C. A. High-resolution cardiovascular function confirms functional orthology of myocardial contractility pathways in zebrafish. Physiological Genomics. 42 (2), 300-309 (2010).
- Yalcin, H. C., Amindari, A., Butcher, J. T., Althani, A., Yacoub, M. Heart function and hemodynamic analysis for zebrafish embryos. Developmental Dynamics: An Officical Publication of the American Association of Anatomists. 246 (11), 868-880 (2017).
- Zakaria, Z. Z., et al. Using zebrafish for investigating the molecular mechanisms of drug-induced cardiotoxicity. Biomed Research International. 2018, 1642684 (2018).
- Parker, T., et al. A multi-endpoint in vivo larval zebrafish (Danio rerio) model for the assessment of integrated cardiovascular function. Journal of Pharmacological and Toxicological Methods. 69 (1), 30-38 (2014).
- Grone, B. P., et al. Epilepsy, behavioral abnormalities, and physiological comorbidities in syntaxin-binding protein 1 (STXBP1) mutant zebrafish. PLoS One. 11 (3), 0151148 (2016).
- Salman, H. E., Yalcin, H. C. Advanced blood flow assessment in Zebrafish via experimental digital particle image velocimetry and computational fluid dynamics modeling. Micron. 130, 102801 (2020).
- Benslimane, F. M., et al. Cardiac function and blood flow hemodynamics assessment of zebrafish (Danio rerio) using high-speed video microscopy. Micron. 136, 102876 (2020).
- DeGroff, C. G. Doppler echocardiography. Pediatric Cardiology. 23 (3), 307-333 (2002).
- Bagatto, B., Burggren, W. A three-dimensional functional assessment of heart and vessel development in the larva of the zebrafish (Danio rerio). Physiological and Biochemical Zoology. 79 (1), 194-201 (2005).
- Tinevez, J. -. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods (San Diego, Calif.). 115, 80-90 (2017).
- Meijering, E., Dzyubachyk, O., Smal, I. Methods for cell and particle tracking. Methods in Enzymology. 504, 183-200 (2012).
- Kok, F. O., et al. Reverse genetic screening reveals poor correlation between morpholino-induced and mutant phenotypes in zebrafish. Developmental Cell. 32 (1), 97-108 (2015).
- Sumanas, S. Inducible inhibition of gene function with photomorpholinos. Methods in Molecular Biology. 1565, 51-57 (2017).
- Kyritsi, K., et al. Knockdown of hepatic gonadotropin-releasing hormone by vivo-morpholino decreases liver fibrosis in multidrug resistance gene 2 knockout mice by down-regulation of miR-200b. The American Journal of Pathology. 187 (7), 1551-1565 (2017).
- Flynt, A. S., Rao, M., Patton, J. G. Blocking zebrafish microRNAs with morpholinos. Methods in Molecular Biology. 1565, 59-78 (2017).
- Schoenebeck, J. J., Keegan, B. R., Yelon, D. Vessel and blood specification override cardiac potential in anterior mesoderm. Developmental Cell. 13 (2), 254-267 (2007).
- Lu, F., Langenbacher, A., Chen, J. -. N. Transcriptional regulation of heart development in zebrafish. Journal of Cardiovascular Development and Disease. 3 (2), 14 (2016).
- Laurent, F., et al. HAND2 target gene regulatory networks control atrioventricular canal and cardiac valve development. Cell Reports. 19 (8), 1602-1613 (2017).
- Miura, G. I., Yelon, D. A guide to analysis of cardiac phenotypes in the zebrafish embryo. Methods in Cell Biology. 101, 161-180 (2011).
- De Luca, E., et al. ZebraBeat: a flexible platform for the analysis of the cardiac rate in zebrafish embryos. Scientific Reports. 4, 4898 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved