É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Projeto e microinjeção de oligonucleotídeos morfolinos antisense e mRNA em embriões de peixe-zebra para elucidar a função gênica específica no desenvolvimento do coração
Neste Artigo
Resumo
O presente protocolo descreve o projeto, preparação e microinjeção de um morfolino bloqueador translacional contra um gene cardíaco representativo; Derivados do coração e da crista neural Expressos2 (mão2) na gema de embriões de peixe-zebra recém-fertilizados para derrubar a função do gene. Também mostra um resgate transitório desses "morfos" por co-injeção de mRNA que codifica esse produto gênico.
Resumo
O sistema de knockdown baseado em oligômero morfolino tem sido usado para identificar a função de vários produtos gênicos por meio de perda ou expressão reduzida. Os morfolinos (MOs) têm a vantagem na estabilidade biológica sobre os oligos de DNA porque não são suscetíveis à degradação enzimática. Para uma eficácia ideal, os MOs são injetados em embriões de 1 a 4 células. A eficácia temporal do knockdown é variável, mas acredita-se que os MOs percam seus efeitos devido à diluição eventualmente. A diluição do morfolino e a quantidade de injeção devem ser controladas de perto para minimizar a ocorrência de efeitos fora do alvo, mantendo a eficácia no alvo. Ferramentas complementares adicionais, como CRISPR/Cas9, devem ser realizadas contra o gene alvo de interesse para gerar linhagens mutantes e confirmar o fenótipo do morfante com essas linhagens. Este artigo demonstrará como projetar, preparar e microinjetar um morfolino bloqueador de tradução contra hand2 na gema de embriões de peixe-zebra em estágio de 1-4 células para derrubar a função hand2 e resgatar esses "morfantes" por co-injeção de mRNA que codifica o cDNA correspondente. Posteriormente, a eficácia das microinjeções de morfolino é avaliada verificando primeiro a presença de morfolino na gema (co-injetada com vermelho de fenol) e depois por análise fenotípica. Além disso, a análise funcional cardíaca para testar a eficácia do knockdown será discutida. Finalmente, será explicada a avaliação do efeito do bloqueio da tradução gênica induzido por morfolino via western blotting.
Introdução
A utilização do peixe-zebra como modelo para o estudo do desenvolvimento cardiovascular e doenças oferece uma variedade de vantagens, incluindo alta conservação da função gênica, transparência óptica, rápido desenvolvimento cardiovascular e custo mais barato quando comparado aos modelos tradicionais in vivo 1. Os oligonucleotídeos morfolinos (MOs) são as ferramentas de knockdown de genes antisense mais comumente usadas para o modelo de peixe-zebra. Os MOs são freqüentemente usados para determinar um fenótipo ou para sondar a função do gene. O Dr. James Summerton desenvolveu inicialmente o sistema de entrega de morfolino para a inibição in vivo da tradução de mRNA como uma tentativa de desenvolver terapêuticas para defeitos de desenvolvimento humano 2,3. MOs têm sido usados para organismos modelo in vitro e in vivo para derrubar genes e investigar a consequência desse knockdown no fenótipo. Isso é feito observando alterações no desenvolvimento de órgãos específicos, por exemplo, o coração. O knockdown de genes específicos do coração em embriões de peixe-zebra WT levou à falha de um batimento cardíaco adequado, atestando a função indispensável desses genes para o desenvolvimento do coração 4,5. Esses fenótipos foram resgatados por co-injeção de mRNAs para os genes específicos. Um estudo envolvendo troponina T cardíaca (Tnnt2) mostrou que a expressão de mRNA tnnt2 de comprimento total poderia resgatar fenótipos sarcoméricos causados pelo knockdown demorfolino 6. Outro estudo revelou que a integridade das bandas A e dos discos Z pode ser restaurada pela superexpressão do mRNA do ortólogo de cadeia leve da miosina reguladora (cmlc2) em mutantes cmcl2 7.
Os MOs são comumente usados para derrubar a expressão gênica, visando o splicing pré-mRNA ou bloqueando a tradução. Os MOs bloqueadores de splice se ligam e inibem o pré-mRNA inibindo o splicesomema. O bloqueio translacional ocorre quando o MO se liga à região 5'-não traduzida do mRNA complementar para impedir a montagem do ribossomo. Os MOs são o método específico de gene mais amplamente utilizado para derrubar a expressão gênica em modelos in vivo ; eles também são os agentes bloqueadores de mRNA mais eficientes usados em culturas de células. O morfolino em si normalmente consiste em uma cadeia curta (cerca de 25) de bases de subunidades morfolino. Cada subunidade de MO inclui uma base de ácido nucleico, um anel de morfolina e um fosforodiamidato não iônico. Os diferentes mecanismos de ação para os dois tipos de MOs requerem testes diferentes para verificar a eficácia do knockdown. Para MOs bloqueadores de tradução, uma análise de western blot é o teste de eficácia mais confiável, pois a proteína de interesse não deve ser produzida devido ao bloqueio do local de início da tradução do ATG. Os MOs não degradam diretamente seu mRNA alvo; em vez disso, eles se ligam a regiões específicas e inibem a expressão até serem naturalmente degradados. No entanto, os MOs bloqueadores de splice modificam o pré-mRNA induzindo a modificação do splice, que pode ser testada por reação em cadeia da polimerase via transcriptase reversa (RT-PCR) e eletroforese em gel.
Três partes cruciais do processo de triagem de MOs devem ser padronizadas: (i) A curva de dose de MO deve ser ajustada para reconhecimento fenotípico. A curva de dose também mostra a dose letal 50 (LD50: a dose na qual 50% dos embriões injetados morrem) para cada MO testado para melhorar a capacidade de otimizar o 'sinal' fenotípico versus o 'ruído' fora do alvo3. (ii) A nomenclatura fenotípica que foi adaptada deve ser bem documentada; Uma descrição fenotípica precisa e facilmente compreensível é fundamental para fornecer explicações extensas com base na literatura existente e na experiência do investigador para facilitar o compartilhamento de informações entre aqueles que não examinaram diretamente os embriões. (iii) Ter uma linguagem bem definida facilita a coleta de dados centralmente do Morpholino Database8.
Em estudos de knockdown de MO para genes cardíacos, a atividade cardíaca e a dinâmica do fluxo sanguíneo dos animais devem ser monitoradas para determinar o impacto dos experimentos de knockdown de MO na função do sistema cardiovascular. Tais análises requerem visualização em tempo real do sistema cardiovascular em alta resolução. A pele do peixe-zebra é transparente na primeira semana de desenvolvimento, permitindo a visualização do coração e da circulação sanguínea por microscopia. Para avaliação da função cardíaca, os parâmetros fisiológicos mais calculados são frequência cardíaca e débito cardíaco, bem como encurtamento fracionário, alteração de área fracionária e fração de ejeção. As velocidades do fluxo sanguíneo podem ser medidas rastreando as hemácias em movimento, e essas medições são usadas para determinar os níveis de tensão de cisalhamento, um fator mecanobiológico crucial nas células endoteliais. Tal avaliação requer a gravação de filmes em time-lapse para batimentos cardíacos e fluxo de sangue por meio de um microscópio invertido ou estereomicroscópio equipado com uma câmera de alta velocidade.
Este artigo mostra como projetar, preparar e microinjetar um morfolino bloqueador translacional contra um gene de interesse na gema de embriões de peixe-zebra recém-fertilizados para derrubar a função do gene. Também mostrará o resgate desses "morfantes" por co-injeção de mRNA que codifica esse gene. Em seguida, analisaremos a eficácia das microinjeções de morfolino por meio de caracterizações fenotípicas, bem como análises estruturais e funcionais cardíacas. Essa abordagem será demonstrada em um gene cardíaco amplamente estudado, hand2.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos os experimentos foram realizados de acordo com os padrões aceitos de cuidados humanitários com animais sob a regulamentação da IACUC na QU; os animais foram mantidos nas instalações de peixe-zebra do Centro de Pesquisa Biomédica da Universidade do Catar (QU-BRC). Todos os animais usados nesses estudos experimentais estavam abaixo de 3 dias após a fertilização (dpf).
NOTA: Para cada grupo experimental, é aconselhável usar pelo menos 30 embriões para rigor estatístico. Os grupos experimentais são os seguintes:
Grupo controle: Este grupo inclui embriões cultivados em água de ovo sem injeções. Os resultados aqui formarão a linha de base de controle.
Grupo de controle negativo: Este grupo inclui embriões cultivados em água de ovo injetada com MOs mexidos.
Grupo injetado: Este grupo inclui embriões injetados com hand2 MO sozinho e hand2 MO com hand2 mRNA para resgatar o fenótipo. Os resultados aqui confirmarão que os fenótipos observados apareceram devido a MOs injetados. A comparação de grupos experimentais permitirá avaliar com precisão a influência da inibição e resgate da mão2 na função cardíaca.
1. Projetos de Morpholino para hand2.
NOTA: As sequências de MO podem ser adaptadas da literatura 9,10,11. Alternativamente, esses oligos podem ser projetados online pela Gene-tools. A Gene-tools oferece um serviço de design online gratuito e rápido, que pode ser acessado através de seu site12. Um MO personalizado pode ser prontamente projetado fornecendo informações sobre os genes de interesse, como informações de sequência ou números de acesso. As etapas específicas a seguir resumem como projetar MOs em relação à mão2 no peixe-zebra:
- Primeiro, pesquise detalhes do gene de interesse no banco de dados do NCBI. Para a mão2, no peixe-zebra13.
- Obtenha o ID de transcrição de mRNA do GenBank do NCBI14.
- Obtenha a sequência de mRNA do NCBI15.
NOTA: Oligonucleotídeos antisense modificados com morfolino (MO) para o gene da mão do peixe-zebra2 estão disponíveis e foram publicados anteriormente. mão 2 Sequência MO 9,10,11: 5'-CCTCCAACTAAACTCATGGCGACAG-3', as sequências MO usadas no estudo estão listadas na Tabela de Materiais. - Obtenha um controle negativo de um MO incompatível ou embaralhado (sequências semelhantes com mudanças aleatórias de pares de bases) das ferramentas genéticas16 a serem injetadas. Isso ajudaria a atestar a especificidade do(s) fenótipo(s) observado(s) em injeções específicas de MO e minimizaria o risco de que os efeitos observados sejam de um artefato do procedimento de injeção.
2. Preparação da injeção de morfolino
- Preparação de estoque
- Dissolver os estoques de MO com ddH2O; O dietilpirocarbonato (DEPC) pode danificar MOs e ser tóxico para os embriões. Portanto, use ddH2O sem DEPC. Após o pedido, os MOs são entregues em um frasco na concentração de 300 nM.
NOTA: Embora a Gene Tools recomende soluções estoque de 1 mM (~ 8 ng / nL), isso pode ser muito baixo, especialmente se o morfolino exigir uma dose alta ou for misturado com outros MOs. Portanto, é ideal para fazer várias concentrações de soluções estoque de MO (2 ou 3 mM). - Preparar uma solução de trabalho a 0,2 mM subdiluindo a solução de trabalho (ou seja, para massa a 2 mM, diluição a 1:10). Prepare 10 μL de solução de trabalho adicionando solução de Danieaux (7 μL), morfolino estoque (1 μL) e vermelho de fenol (2 μL de estoque 10x). O vermelho de fenol dá à solução uma cor vermelha escura para ajudar a rastrear o material injetado no embrião.
- Mantenha os MOs em temperatura ambiente (RT) em recipientes herméticos para evitar a evaporação. Não mantenha os MOs congelados na bancada, pois a solução pode precipitar. O manuseio de RT é mais apropriado.
NOTA: A Gene Tools recomenda um dos dois métodos de armazenamento para MOs: A solução de trabalho pode ser mantida a -20 ° C (por muitos meses a anos) ou RT em um tubo selado para armazenamento de longo prazo. Se mantido a -20 °C, aqueça a solução de oligonucleótidos a 65 °C durante 10 min e vortex para dissolver completamente o morfolino antes da utilização (não é necessário aquecê-lo se for mantido em RT).
- Dissolver os estoques de MO com ddH2O; O dietilpirocarbonato (DEPC) pode danificar MOs e ser tóxico para os embriões. Portanto, use ddH2O sem DEPC. Após o pedido, os MOs são entregues em um frasco na concentração de 300 nM.
- Transcrição in vitro de mRNA
NOTA: A transcrição in vitro foi usada para gerar mRNA de Hand2 a partir do clone ORF marcado com plasma HAND2 (NM_021973) humano para ser microinjetado em embriões de peixe-zebra para resgatar o fenótipo devido à derrubada da mão de peixe-zebra2 usando morfolinos, os detalhes do plasmídeo usados no estudo estão listados na Tabela de Materiais.- Linearizar o DNA do plasmídeo por meio de uma enzima de restrição apropriada e purificar usando um kit de purificação de DNA (Tabela de Materiais). Use o DNA linearizado purificado como modelo para transcrição in vitro para gerar mRNA para injeção de acordo com as instruções do fabricante (Tabela de Materiais).
- Finalmente, injete 250 pg de mRNA humano para o gene de interesse para realizar o resgate do fenótipo para testar se a falta de um fenótipo é de fato da perda do gene do peixe-zebra pelo MO.
- Preparando-se para injetar
- Puxe uma agulha usando um extrator de micropipeta (Tabela de Materiais), seguindo as etapas 2.3.2-2.3.6.
- Use tubos capilares de 1 mm com filamentos e um extrator de micropipeta. Ligue a máquina e abra a tampa
- Gire o botão de seleção de modo para NO.2 Heater. Usando o aquecedor NO.2 , ajuste o botão para levar o calor a 68 °C
- Gire o botão de seleção de modo de volta para a posição Etapa 1 . Use quatro pesos (2 Tipo Leve / 2 Tipo Pesado)
- Coloque o capilar no suporte e feche a tampa. Pressione o botão Iniciar para obter a agulha puxada.
NOTA: Isso gerará agulhas capilares com um cone longo - Corte a ponta das agulhas brevemente, usando uma pinça. Afie a ponta da agulha puxada para facilitar a penetração em embriões de peixe-zebra usando um chanfrador capilar por cerca de 30 s, resultando em pontas com diâmetros de cerca de 15-25 μm.
- Calibre a agulha para estimar a quantidade de injeção seguindo as etapas 2.3.8 a 2.3.13.
- Misture brevemente a solução de mRNA/morfolino antes de carregá-la na agulha para dissolver totalmente as partículas que podem entupir totalmente a agulha.
- Adicione ~ 3-4 μL de solução de MO na parte de trás da agulha de injeção usando uma ponta de pipeta com ponta de mícron. As agulhas de injeção são equipadas com uma pequena vala para facilitar o enchimento capilar.
- Confirme se não há bolhas na agulha de injeção. Monte a agulha no microinjetor.
- Verifique se a ponta da agulha não está entupida e se a solução de MO pode fluir para fora da agulha pressionando o pedal de injeção.
- Calibre o volume de injeção medindo o tamanho da gota. Coloque a ponta da agulha em óleo mineral em uma lâmina micrométrica. Pressione o pedal de injeção e ajuste a pressão de injeção (ejetar) (psi) e/ou o tempo de injeção (ms) até que o diâmetro da gota ejetada seja de 0.1 mm, correspondendo a 0.5 nL.
- Certifique-se de que o volume de injeção esteja em torno de 0,5-1 nL. Ajuste o volume alterando a pressão de injeção (ejetar) (psi) ou o tempo (ms).
- Para o protocolo de configuração do injetor picoliter, siga as etapas 2.3.15-2.3.16
- Use o injetor de picolitros para injeções de peixe-zebra. Ligue a bomba de alimentação do injetor.
- Prepare o injetor de acordo com os requisitos mostrados abaixo:
- Equilíbrio P: Certifique-se de que o equilíbrio de P esteja em torno de 0. Certifique-se de que seja ligeiramente negativo para evitar que a gema flua de volta para a agulha, diluindo a solução de MO na agulha. Por outro lado, se a contrapressão for muito alta, o MO fluirá constantemente para fora da agulha, mesmo sem o esforço de pressão no pedal, resultando em variabilidade e inconsistências na dose injetada e nos fenótipos observados.
- P-injetar: 20-25 psi é o ideal; Altere isso para ajustar o volume de injeção. Isso pode variar de 10 a 30 psi, mas comece com ~ 20 psi para verificar se ele fornece o volume desejado.
- Tempo de injeção: Certifique-se de reduzir o tempo de injeção para 300 ms. Antes de ajustar o volume da injeção, faça ajustes no tempo da injeção.
- Para injeção de morfolino, siga as etapas 2.3.18-2.3.19.
- Prepare a câmara de injeção da seguinte forma: Fazer 2% de agarose a partir de 0,6 g de agarose e 30 ml de meio E3. Despeje a agarose em uma placa de Petri e coloque um molde de injeção na agarose usando um molde de injeção TU-1. Uma vez resfriados em um freezer, sulcos se formarão para colocar e estabilizar os embriões (Figura 1A).
NOTA: A receita da solução estoque 60x E3M consiste em 5,0 mM de NaCl, 0,17 mM de KCl, 0,16 mM de MgSO4·7H2O, 0,4 mM de CaCl2·2H2O em 1 L ddH2O com um pH final de 7,6. - Transferir os embriões recolhidos para os sulcos utilizando uma pipeta de transferência, remover todo o excesso de meio E3 à volta dos embriões para evitar a flutuação dos embriões (Figura 1B).
3. Injeção de MO e solução de mRNA na gema
- Introduza a agulha através do córion, injete perto do limite da célula/gema no lado do embrião e pressione o pedal de injeção (Figura 1C).
NOTA: A solução de MO injetada / vermelho de fenol não se difundirá imediatamente dentro da gema. Uma mancha vermelha será observada na gema que se dissipa gradualmente. O material injetado não deve exceder mais de 10% do tamanho do embrião. Os MOs podem ser microinjetados na gema de ovos de peixe-zebra recém-postos até o estágio de 8 células, porque os MOs podem ser facilmente absorvidos no fluxo citoplasmático do saco vitelino pelas células em desenvolvimento17,18. Em injeções de plasmídeo ou mRNA tampado, a injeção deve ser realizada em um único blastômero de um embrião em estágio de uma célula. - Transferir os embriões para uma placa de Petri com meio E3 e incubá-los a 28 °C até 3 dpf, com reposição diária do meio E3. Nessa fase, realizar a avaliação fenotípica usando um microscópio.
NOTA: As soluções de MO devem ser cuidadosamente diluídas para cada investigação para estabelecer a concentração mais baixa necessária para induzir um fenótipo específico. A letalidade embrionária para cada OM também deve ser determinada. Em concentrações mais altas (acima de 4-10 ng, dependendo do MO), os MOs tendem a provocar efeitos inespecíficos, como morte cerebral ou celular geral. - Co-injetar MO com 250 pg de mRNA da mão humana2 da mesma forma para confirmar o resgate do fenótipo induzido pela perda do gene da mão do peixe-zebra2 por MO.
4. Western blot para verificar o sucesso do knockdown do morfolino
- Colete os embriões em pontos de tempo específicos (48 hpf, 72 hpf) e descorione, se necessário, usando a enzima Pronase.
- Deyolk os embriões para remover a vitelogenina usando uma metodologia previamente conhecida19.
NOTA: A vitelogenina é uma fosfolipoglicoproteína que serve como fonte de nutrientes para o embrião em crescimento. A eletroforese em gel 2D de alta resolução e o western blotting amplamente aprimorado são possíveis com a remoção dos embriões e a remoção da vitelogenina. - Realize western blotting conforme descrito anteriormente20.
5. Avaliação da estrutura e função cardíaca:
- Imagens ao vivo do peixe-zebra: imagens do ventrículo
NOTA: Vários parâmetros de função cardíaca/hemodinâmica podem ser calculados visualizando o coração batendo do embrião de peixe-zebra21,22. Esses parâmetros incluem débito cardíaco (DC), fração de ejeção (FE), volume sistólico (VS), encurtamento fracionário (FS) e alteração de área fracionária (FAC) (consulte artigos anteriores envolvendo protocolos detalhados para avaliação da função cardíaca para embriões de peixe-zebra22,23). As etapas a seguir explicam brevemente como calcular esses parâmetros para embriões de peixe-zebra a ~ 3 dpf, um estágio em que a pele desses organismos é transparente, permitindo a visualização com microscopia de campo claro.- Coloque uma gota de solução de metilcelulose a 3% (RT) na lâmina côncava de imagem do poço.
- Posicione o embrião de peixe-zebra no poço usando um conta-gotas de plástico adequado (Figura 2A, B)
NOTA: Encher demais o poço pode causar deslocamento de peixes para fora do poço. - Misture suavemente a gota de metilcelulose a 3% com o meio E3 para estabilizar o embrião eclodido.
NOTA: Para preparar a solução de metilcelulose a 3%, dissolva 3 g de pó de metilcelulose em 100 mL de PBS, ou outro meio de montagem, em um frasco. Colocar um íman de agitação no balão da mistura e colocá-lo numa placa de agitação magnética. Defina a velocidade para "baixa" e mantenha-a a 4 °C por ~ 1 dia para dissolver todos os aglomerados. Uma vez que a metilcelulose esteja completamente dissolvida, alíquota em pequenos tubos e armazenar a -20 °C. - Posicione o peixe à esquerda, com o lado direito voltado para cima e o ponto anterior para a esquerda para facilitar a imagem inequívoca do ventrículo (Figura 2C).
- Sob o microscópio, amplie o coração do embrião com ampliação de 100x e comece a gravar por ~ 5 s. Certifique-se de que as bordas do ventrículo estejam dentro da janela de imagem (Figura 2D).
- Grave filmes com lapso de tempo usando uma câmera de alta velocidade e estereomicroscópio a cerca de 100 quadros por segundo (fps) de todo o embrião (Figura 2C), ventrículo batendo (Figura 2D) e movendo glóbulos vermelhos (RBC) nos principais vasos, como a aorta dorsal ou a veia cardinal posterior (Figura 2E), para análise da função cardíaca.
- Salve o filme no formato de filme AVI ou no formato de sequências de imagens TIFF (ou JPEG).
- Calcule a frequência cardíaca
- Calcule o tempo necessário para gravar dois quadros consecutivos. Para 100 fps, o intervalo de tempo é igual a 0,01 s.
- Escolha um ponto conhecido de qualquer ciclo cardíaco registrado (ou seja, diástole final ou sístole final). Calcule o número de quadros necessários para repetir o ciclo.
- Multiplique o intervalo de tempo da etapa 5.2.1 pelo número de quadros da etapa 5.2.2. O resultado é a duração do tempo (em segundos) para uma pulsação.
- Para calcular batimentos por minuto, divida 60 pelo número obtido na etapa anterior. Para embriões normais de 3 dpf, isso deve ser em torno de 150 bpm (2,5 Hz).
NOTA: Muitos aplicativos de software podem calcular a frequência cardíaca automaticamente a partir de gravações de batimentos cardíacos, como ViewPoint24 e DanioVision25. - Analise a estrutura cardíaca usando um filme de lapso de tempo gravado a cerca de 100 fps para verificar todo o coração para detectar a presença de edema cardíaco ou qualquer outro defeito estrutural.
NOTA: A Figura 3A mostra um coração embrionário normal de 3 dpf e a Figura 3B mostra um coração de 3 dpf com edema cardíaco e defeito de looping cardíaco (ventrículo alongado).
- Calcule a variação da área fracionária (FAC):
NOTA: A alteração da área fracionária (FAC) é um parâmetro usado para comparar as áreas da diástole final e da sístole ventricular para avaliar a contratilidade do ventrículo.- Utilize um filme time-lapse gravado a cerca de 100 fps para determinar os fotogramas que representam um ciclo cardíaco. Extraia os quadros que mostram o fim da sístole final e da diástole.
- Calcule a área de diástole final (EDA) e a área de sístole final (ESA) usando o ImageJ ou um software de análise de imagem semelhante.
- Use a seguinte fórmula para calcular FAC 22,26,27:
FAC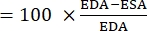
- Calcule o encurtamento fracionário (FS):
NOTA: O encurtamento fracionado (FS) é outro parâmetro utilizado para avaliar a contratilidade do ventrículo; para determinar a SF, os diâmetros diastólico final e sistólico final dos ventrículos devem ser medidos21.- Repita a etapa 5.3.1
- Use o ImageJ ou qualquer outro programa de análise de imagem equivalente para medir os diâmetros das paredes ventriculares nos pontos Dd d e Ds da diástole final. Na maioria dos casos, diâmetros de eixo curto são empregados para determinar a suavidade fracionária (FS) (Figura 4).
- Use a seguinte fórmula para calcular FS22,27:
FS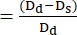
- Cálculo do volume sistólico (VS):
NOTA: Para cada batimento cardíaco, o VS é a quantidade de sangue bombeada do ventrículo, que é facilmente calculada a partir dos volumes final da diástole e final da sístole dos ventrículos28.- Repita as etapas 5.3.1 e 5.3.2
- Meça os diâmetros da diástole final (DL) e da sístole final (DS) conforme mostrado na Figura 4.
NOTA: Supondo que os ventrículos dos corações do peixe-zebra tenham a forma de um esferoidal prolato21,22, o volume do ventrículo é calculado usando a seguinte fórmula.
Volume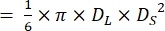
SV pode ser calculado seguindo a fórmula abaixo27. A faixa normal de SV é de 0,15-0,3 nL para embriões de 2-6 dpf29. Aqui, o EDV é o volume final da diástole e o ESV é o volume final da sístole.
SV = (EDV - ESV)
- Calcular a fração de ejeção (EF):
NOTA: A FE é definida como a fração de sangue ejetada do ventrículo a cada batimento cardíaco28.- A partir das fórmulas acima, extraia EDV e ESV para o peixe.
- Calcular EF da seguinteforma 22,27:
FE (%)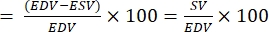
- Calcule o débito cardíaco (DC):
NOTA: CO é o volume de sangue que está sendo bombeado pelo coração28. O CO tem um valor de 10-55 nL/min para embriões de 2-6 dpf29.- Calcule o VS e a FC conforme mencionado nas subseções 5.5 e 5.2 anteriores.
- Utilizar a seguinte fórmula para calcular o CO 22,27:
CO (nL/min) = VS (nL/batimento) x FC (batimentos/min).
- Meça as velocidades das hemácias:
NOTA: A determinação da velocidade das células é vital para avaliar a taxa de fluxo, o diâmetro do vaso e calcular a tensão de cisalhamento exercida nas células endoteliais do vaso (ou seja, a tensão de cisalhamento, por outro lado, é a força de atrito imposta às células endoteliais pelo movimento do sangue). Para embriões de 2-5 dpf, a velocidade média dos glóbulos vermelhos (RBC) é de cerca de 300-750 μm/s29. Para medir as velocidades das hemácias:- Sob o microscópio, amplie a cauda do animal com uma ampliação de 100x. O movimento das hemácias deve ser visível nesta ampliação (Figura 5A, B).
- Comece a gravar por cerca de 8 s. Certifique-se de que a aorta dorsal (DA) e a veia cardinal posterior (PCV), as duas artérias axiais mais importantes, estejam 100-120 fps dentro da janela de imagem. Rastreie células individuais a partir de quadros sequenciais (Figura 5A, B).
- Extraia vários quadros usando o ImageJ ou outro software de análise de imagem semelhante.
- Calcule a diferença na distância que um eritrócitos individual se move (Δx).
- Determine a diferença de tempo entre os quadros consecutivos (Δt).
- Use a seguinte fórmula para a velocidade das hemácias22,27:
Velocidade das hemácias (μm/s)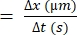
NOTA: A velocidade máxima e média pode ser extraída da repetição de quadros consecutivos para células individuais21. A velocidade das hemácias também representa a velocidade do fluxo sanguíneo. Como alternativa, uma variedade de aplicativos de software disponíveis comercialmente pode ser usada para medir a velocidade das hemácias automaticamente. O ViewPoint24, assim como o DanioVision25, têm esses aplicativos (Figura 5C). Além disso, existem poucos plugins disponíveis com ImageJ, como TrackMate30 e MTrackJ31, compatíveis com filmes gravados acima de 100 fps.
- Cálculo da tensão de cisalhamento a partir das velocidades das células medidas
As velocidades de hemácias medidas nos vasos sanguíneos também representam as velocidades do fluxo sanguíneo (Figura 5C). A partir dessas medições, calcule a tensão de cisalhamento τ em um vaso de interesse da seguinte forma com a suposição de fluxo de Poiseuille22,27:
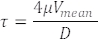
Onde V é a velocidade média do sangue (μm/s), μ é a viscosidade do sangue (dinas/cm2) e D é o diâmetro do vaso (μm). Para embriões de 3 dpf, a tensão de cisalhamento em DA 22,26,27 é de cerca de 4 dinas / cm 2.
Access restricted. Please log in or start a trial to view this content.
Resultados
O gráfico na Figura 6 ilustra a porcentagem média de embriões que sobrevivem a 24, 48 e 72 hpf para MO específico para HAND2 e embriões injetados com MO mexidos de controle. Os embriões injetados com MO de 1 mM (8 ng/μL) e 0,8 mM (6,4 ng/μL) mostraram uma redução significativa na porcentagem de sobrevivência em comparação com os embriões injetados com MO mexidos. Isso foi observado em cada ponto de tempo medido em que letalidade ou malformaçã...
Access restricted. Please log in or start a trial to view this content.
Discussão
A tecnologia Morpholino (MO) tem sido amplamente utilizada em peixe-zebra, xenopus, ouriços-do-mar e, mais recentemente, em sistemas de modelo de cultura de células. Com a maioria dos métodos, juntamente com os benefícios, também existem armadilhas das quais o experimentador deve estar ciente. Uma das principais armadilhas da tecnologia MO inclui a preocupação de que os efeitos fenotípicos observados pela abordagem de knockdown do gene mediada por MO não sejam devidos à perda d...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram não ter interesses financeiros ou outros conflitos de interesse.
Agradecimentos
A publicação deste artigo foi coberta com um generoso apoio da BARZAN HOLDINGS. O RR é parcialmente apoiado por R61HL154254 e fundos do Departamento de Pediatria e do Hospital Infantil.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Acrylamide 40% | Sigma | Sigma, cat. no. C977M88 | |
| Agarose | Sigma-Aldrich | Sigma-Aldrich cat. no A9539-250G | |
| All Prep DNA/RNA Mini Kit | Qiagen | Qiagen cat. no. 80204. | |
| alpha Tubulin | Abcam | Abcam- ab4074 | Rabbit polyclonal to alpha Tubulin lot GR3 180877-1 (50 kDa) |
| Ammonium persulfate molecular grade | Sigma | Sigma, cat. no C991U65 | |
| BV10 capillary beveller | Sutter Instruments Product | Sutter Instruments Product Catalog # BV10 | |
| Chemiluminescence Imaging Gene Gnome | SYNGENE | SYNGENE | |
| Cleaver Scientific Blotting | CVS10D_OmniPAGEMini | CVS10D_OmniPAGEMini | |
| Coomassie | Thermo Fisher | Thermo Fisher cat. no C861C44 | |
| Electrochemiluminescence (ECL) kit | Abcam Biochemicals | Abcam Biochemicals cat. no ab65623 | |
| Glycine | Sigma | Sigma, cat. no C988U91 | |
| Goat anti Rabbit | Abcam | Abcam- ab6721 | Goat Anti-Rabbit IgG H&L (HRP) 2nd antibodies lot GR3179871-1 |
| HAND2 | Gene tools | Custom made for HAND2 (NM_021973) | 5'-CCTCCAACTAAACTCATGGCGAC AG-3' |
| Hand2 | Abcam | Abcam- ab10131 | Rabbit polyclonal Anti-HAND2 antibody lot GR143200-9 (24- 26 kDa) |
| HAND2 (NM_021973) Human Tagged ORF Clone | OriGene Technologies, Inc | RC224436L3 | Vector: pLenti-C-Myc-DDK-P2A-Puro (PS100092) |
| IBI DNA/RNA/Protein Extraction Kit | IBI Scientific | IBI Scientific cat. no -r IB47702 | |
| Imaging System | iBright | iBright CL1000 Imaging System | |
| Isopropanol | Sigma-Aldrich | Sigma-Aldrich cat. no 278475-2L | |
| Laemmli sample loading buffer (4x) | Sigma-Aldrich | Sigma-Aldrich cat. no 70607 | |
| Mercaptoethanol | Sigma | Sigma, cat. no M6250-1L | |
| Microplate Spectrophotometer with the Gen5 Data Analysis software interface | Epoch | Epoch | |
| Microscope | Ziess SteREO Lumar V12 Flourescence Microscope | Ziess SteREO Lumar V12 Flourescence Microscope | |
| Mineral oil | Fisher Scientific | Fisher Scientific cat. no 0121-1 | |
| mMESSAGE mMACHINE T7/T3/SP6 Transcription Kit | Thermo Fisher | Thermo Fisher cat. no.AM1340 | for mRNA generation |
| Nuclease-free water | New England Biolabs | New England Biolabs cat. no B1500L | |
| PC-100 Micropipette Puller | NARISHIGE GROUP Product | NARISHIGE GROUP Product Catalog # PC-100 | |
| Phenol red | Sigma | Sigma, cat. no. P-0290 | |
| Picolitre Injector | Harvard Apparatus | Harvard Apparatus cataloge # PLI-90A | |
| Pierce Bicinchoninic acid assay (BCA) Protein Assay kit | Thermo Fisher | Thermo Fisher cat. no 23227 | |
| PMSF, Protease inhibitor as protease inhibitors | Thermo Fisher | Thermo Fisher cat. no 36978 | |
| Ponceau S | Sigma-Aldrich | Sigma-Aldrich cat. no 10165921001 | |
| Protease Inhibitor Cocktail | Thermo Fisher | Thermo Fisher cat. no 88668 | |
| Protein ladder | SMOBiO | SMOBiO cat. no PM2500 | |
| Radioimmunoprecipitation Assay (RIPA) | Thermo Fisher | Thermo Fisher cat. no 89900 | |
| Ringer’s solution | Thermofisher | Catalog No.S25513 | |
| SDS | Sigma | Sigma, cat. no 436143 | |
| Standard Control | Gene tools | SKU: PCO-StandardControl-100 | 5'-CCTCTTACCTCAGTTACAATTTAT A-3'- that targets a human beta-globin intron mutation |
| Stripping buffer | Sigma-Aldrich | Sigma-Aldrich cat. 21059 | |
| Temed | IBI scientific | IBI scientific cat. no C000A52 | |
| Tris Base | Thermo Fisher | Thermo Fisher cat. no BP-152-500 | |
| Tween | sigma life science | sigma life science cat. no P2287 | |
| Zebra Box Revolution-Danio Track system chamber with the EthoVision XT 11.5 software | Noldus Information Technology, NL | Noldus Information Technology, NL | |
| Zeiss Axiocam ERc 5s | Zeiss | Stemi 508 Zeiss | |
| Zeiss Stemi 2000-C | Zeiss | Stemi 2000-C |
Referências
- Bakkers, J. Zebrafish as a model to study cardiac development and human cardiac disease. Cardiovascular Research. 91 (2), 279-288 (2011).
- Bill, B. R., Petzold, A. M., Clark, K. J., Schimmenti, L. A., Ekker, S. C. A primer for morpholino use in zebrafish. Zebrafish. 6 (1), 69-77 (2009).
- Stainier, D. Y., et al. Guidelines for morpholino use in zebrafish. PLoS Genetics. 13 (10), 1007000(2017).
- Zhou, B., et al. Epicardial progenitors contribute to the cardiomyocyte lineage in the developing heart. Nature. 454 (7200), 109-113 (2008).
- Eve, A. M., Place, E. S., Smith, J. C. Comparison of Zebrafish tmem88a mutant and morpholino knockdown phenotypes. PLoS One. 12 (2), 0172227(2017).
- Huang, W., Zhang, R., Xu, X. Myofibrillogenesis in the developing zebrafish heart: A functional study of tnnt2. Developmental Biology. 331 (2), 237-249 (2009).
- Chen, Z., et al. Depletion of zebrafish essential and regulatory myosin light chains reduces cardiac function through distinct mechanisms. Cardiovascular Research. 79 (1), 97-108 (2008).
- Bedell, V. M., Westcot, S. E., Ekker, S. C. Lessons from morpholino-based screening in zebrafish. Briefings in Functional Genomics. 10 (4), 181-188 (2011).
- Reichenbach, B., et al. Endoderm-derived Sonic hedgehog and mesoderm Hand2 expression are required for enteric nervous system development in zebrafish. Developmental Biology. 318 (1), 52-64 (2008).
- Maves, L., Tyler, A., Moens, C. B., Tapscott, S. J. J. Pbx acts with Hand2 in early myocardial differentiation. Developmental Biology. 333 (2), 409-418 (2009).
- Hinits, Y., et al. Zebrafish Mef2ca and Mef2cb are essential for both first and second heart field cardiomyocyte differentiation. Developmental Biology. 369 (2), 199-210 (2012).
- Gene Tools, custom-sequence Morpholinos. , Available from: https://oligodesign.gene-tools.com/ (2011).
- hand2 heart and neural crest derivatives expressed 2 [ Danio rerio (zebrafish). , Available from: https://www.ncbi.nlm.nih.gov/gene/58150 (2021).
- Danio rerio heart and neural crest derivatives expressed 2 (hand2), mRNA. , Available from: https://www.ncbi.nlm.nih.gov/nuccore/NM_131626.3 (2021).
- NCBI reference sequence -Danio rerio heart and neural crest derivatives expressed 2 (hand2), mRNA FASTA. , hand2), mRNA FASTA at (2021). Prepared control oligos at (2021).
- Prepared control oligos. , Available from: https://store.gene-tools.com/prepared-control-oligos (2021).
- Schubert, S., Keddig, N., Hanel, R., Kammann, U. Microinjection into zebrafish embryos (Danio rerio)-a useful tool in aquatic toxicity testing. Environmental Sciences Europe. 26 (1), 32(2014).
- Moulton, J. D. Guide for morpholino users: toward therapeutics. Journal of Drug Discovery, Development, and Delivery. 3 (2), 1023(2016).
- Link, V., Shevchenko, A., Heisenberg, C. -P. Proteomics of early zebrafish embryos. BMC Developmental Biology. 6 (1), 1(2006).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. Journal of Visualized Experiments: JoVE. (44), e2359(2010).
- Shin, J. T., Pomerantsev, E. V., Mably, J. D., MacRae, C. A. High-resolution cardiovascular function confirms functional orthology of myocardial contractility pathways in zebrafish. Physiological Genomics. 42 (2), 300-309 (2010).
- Yalcin, H. C., Amindari, A., Butcher, J. T., Althani, A., Yacoub, M. Heart function and hemodynamic analysis for zebrafish embryos. Developmental Dynamics: An Officical Publication of the American Association of Anatomists. 246 (11), 868-880 (2017).
- Zakaria, Z. Z., et al. Using zebrafish for investigating the molecular mechanisms of drug-induced cardiotoxicity. Biomed Research International. 2018, 1642684(2018).
- Parker, T., et al. A multi-endpoint in vivo larval zebrafish (Danio rerio) model for the assessment of integrated cardiovascular function. Journal of Pharmacological and Toxicological Methods. 69 (1), 30-38 (2014).
- Grone, B. P., et al. Epilepsy, behavioral abnormalities, and physiological comorbidities in syntaxin-binding protein 1 (STXBP1) mutant zebrafish. PLoS One. 11 (3), 0151148(2016).
- Salman, H. E., Yalcin, H. C. Advanced blood flow assessment in Zebrafish via experimental digital particle image velocimetry and computational fluid dynamics modeling. Micron. 130, 102801(2020).
- Benslimane, F. M., et al. Cardiac function and blood flow hemodynamics assessment of zebrafish (Danio rerio) using high-speed video microscopy. Micron. 136, 102876(2020).
- DeGroff, C. G. Doppler echocardiography. Pediatric Cardiology. 23 (3), 307-333 (2002).
- Bagatto, B., Burggren, W. A three-dimensional functional assessment of heart and vessel development in the larva of the zebrafish (Danio rerio). Physiological and Biochemical Zoology. 79 (1), 194-201 (2005).
- Tinevez, J. -Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods (San Diego, Calif.). 115, 80-90 (2017).
- Meijering, E., Dzyubachyk, O., Smal, I. Methods for cell and particle tracking. Methods in Enzymology. 504, 183-200 (2012).
- Kok, F. O., et al. Reverse genetic screening reveals poor correlation between morpholino-induced and mutant phenotypes in zebrafish. Developmental Cell. 32 (1), 97-108 (2015).
- Sumanas, S. Inducible inhibition of gene function with photomorpholinos. Methods in Molecular Biology. 1565, Clifton, N.J. 51-57 (2017).
- Kyritsi, K., et al. Knockdown of hepatic gonadotropin-releasing hormone by vivo-morpholino decreases liver fibrosis in multidrug resistance gene 2 knockout mice by down-regulation of miR-200b. The American Journal of Pathology. 187 (7), 1551-1565 (2017).
- Flynt, A. S., Rao, M., Patton, J. G. Blocking zebrafish microRNAs with morpholinos. Methods in Molecular Biology. 1565, Clifton, N.J. 59-78 (2017).
- Schoenebeck, J. J., Keegan, B. R., Yelon, D. Vessel and blood specification override cardiac potential in anterior mesoderm. Developmental Cell. 13 (2), 254-267 (2007).
- Lu, F., Langenbacher, A., Chen, J. -N. Transcriptional regulation of heart development in zebrafish. Journal of Cardiovascular Development and Disease. 3 (2), 14(2016).
- Laurent, F., et al. HAND2 target gene regulatory networks control atrioventricular canal and cardiac valve development. Cell Reports. 19 (8), 1602-1613 (2017).
- Miura, G. I., Yelon, D. A guide to analysis of cardiac phenotypes in the zebrafish embryo. Methods in Cell Biology. 101, 161-180 (2011).
- De Luca, E., et al. ZebraBeat: a flexible platform for the analysis of the cardiac rate in zebrafish embryos. Scientific Reports. 4, 4898(2014).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados