Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Design und Mikroinjektion von Morpholino-Antisense-Oligonukleotiden und mRNA in Zebrafischembryonen zur Aufklärung spezifischer Genfunktionen in der Herzentwicklung
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt das Design, die Vorbereitung und die Mikroinjektion eines translational blockierenden Morpholinos gegen ein repräsentatives kardiales Gen; Herz- und Neuralleistenderivate2 (Hand2) werden in das Eigelb von frisch befruchteten Zebrafischembryonen exprimiert, um die Genfunktion zu beeinträchtigen. Es zeigt auch eine vorübergehende Rettung dieser "Morphanten" durch Co-Injektion von mRNA, die für dieses Genprodukt kodiert.
Zusammenfassung
Das auf Morpholino-Oligomeren basierende Knockdown-System wurde verwendet, um die Funktion verschiedener Genprodukte durch Verlust oder verminderte Expression zu identifizieren. Morpholinos (MOs) haben gegenüber DNA-Oligos den Vorteil in ihrer biologischen Stabilität, da sie nicht anfällig für enzymatischen Abbau sind. Für eine optimale Wirksamkeit werden MOs in Embryonen im 1-4-Zellstadium injiziert. Die zeitliche Wirksamkeit des Knockdowns ist variabel, aber es wird angenommen, dass MOs ihre Wirkung aufgrund der Verdünnung schließlich verlieren. Die Verdünnung von Morpholino und die Injektionsmenge sollten genau kontrolliert werden, um das Auftreten von Off-Target-Effekten zu minimieren und gleichzeitig die On-Target-Wirksamkeit zu erhalten. Zusätzliche komplementäre Werkzeuge, wie z.B. CRISPR/Cas9, sollten gegen das Zielgen von Interesse durchgeführt werden, um mutierte Linien zu generieren und den morphanten Phänotyp mit diesen Linien zu bestätigen. In diesem Artikel wird gezeigt, wie ein translationsblockierendes Morpholino gegen Hand2 in das Eigelb von Zebrafischembryonen im Zellstadium 1-4 entworfen, vorbereitet und mikroinjiziert wird, um die Hand2-Funktion zu unterbrechen und diese "Morphanten" durch Co-Injektion von mRNA, die für die entsprechende cDNA kodiert, zu retten. Anschließend wird die Wirksamkeit der Morpholino-Mikroinjektionen bewertet, indem zunächst das Vorhandensein von Morpholino im Eigelb (co-injiziert mit Phenolrot) und dann durch phänotypische Analyse überprüft wird. Darüber hinaus wird die kardiale Funktionsanalyse zur Überprüfung der Knockdown-Wirksamkeit diskutiert. Schließlich wird die Bewertung des Effekts der Morpholino-induzierten Blockierung der Gentranslation durch Western Blotting erläutert.
Einleitung
Die Verwendung des Zebrafisches als Modell für die Untersuchung der kardiovaskulären Entwicklung und Krankheit bietet eine Vielzahl von Vorteilen, darunter eine hohe Erhaltung der Genfunktion, optische Transparenz, eine schnelle kardiovaskuläre Entwicklung und günstigere Kosten im Vergleich zu herkömmlichen In-vivo-Modellen 1. Morpholino-Oligonukleotide (MOs) sind die am häufigsten verwendeten Antisense-Gen-Knockdown-Werkzeuge für das Zebrafischmodell. MOs werden häufig verwendet, um einen Phänotyp zu bestimmen oder die Genfunktion zu untersuchen. Dr. James Summerton entwickelte ursprünglich das Morpholino-Verabreichungssystem zur Inhibition der mRNA-Translation in vivo als Versuch, Therapeutika für menschliche Entwicklungsdefekte zu entwickeln 2,3. MOs wurden für in vitro und in vivo Modellorganismen verwendet, um Gene zu knockdown und die Auswirkungen dieses Knockdowns auf den Phänotyp zu untersuchen. Dies geschieht durch die Beobachtung von Veränderungen in der Entwicklung bestimmter Organe, zum Beispiel des Herzens. Der Knockdown von herzspezifischen Genen in WT-Zebrafischembryonen führte zum Versagen eines korrekten Herzschlags, was die unverzichtbare Funktion dieser Gene für die Herzentwicklung belegt 4,5. Diese Phänotypen wurden durch Co-Injektion von mRNAs für die spezifischen Gene gerettet. Eine Studie mit kardialem Troponin T (Tnnt2) zeigte, dass die Expression von tnnt2-mRNA in voller Länge sarkomerische Phänotypen retten kann, die durch Morpholino-Knockdownverursacht werden 6. Eine weitere Studie zeigte, dass die Integrität von A-Banden und Z-Scheiben durch Überexpression der regulatorischen Myosin-Leichtketten-Ortholog (cmlc2) mRNA in cmcl2-Morphanten wiederhergestellt werden konnte7.
MOs werden häufig verwendet, um die Genexpression zu unterbinden, indem sie auf das Prä-mRNA-Spleißen abzielen oder die Translation blockieren. Spleißblockierende MOs binden und hemmen prä-mRNA, indem sie das Spleißsom hemmen. Eine translationale Blockierung tritt auf, wenn das MO an die 5'-untranslatierte Region der komplementären mRNA bindet, um die Ribosomenassemblierung zu behindern. MOs sind die am weitesten verbreitete genspezifische Methode zur Unterdrückung der Genexpression für in vivo-Modelle ; Sie sind auch die effizientesten mRNA-Blocker, die in Zellkulturen verwendet werden. Das Morpholino selbst besteht typischerweise aus einer kurzkettigen (etwa 25) von Morpholino-Untereinheitsbasen. Jede MO-Untereinheit umfasst eine Nukleinsäurebase, einen Morpholinring und ein nichtionisches Phosphordiamidat. Die unterschiedlichen Wirkmechanismen für die beiden Arten von MOs erfordern unterschiedliche Tests, um die Wirksamkeit des Knockdowns zu überprüfen. Für translationsblockierende MOs ist eine Western-Blot-Analyse der zuverlässigste Wirksamkeitstest, da das interessierende Protein aufgrund einer Blockade der ATG-Translationsstartstelle nicht produziert werden sollte. MOs bauen ihre Ziel-mRNA nicht direkt ab; Stattdessen binden sie an bestimmte Regionen und hemmen die Expression, bis sie auf natürliche Weise abgebaut werden. Die Spleiß-blockierenden MOs modifizieren jedoch die Prä-mRNA, indem sie eine Spleißmodifikation induzieren, die durch Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-PCR) und Gelelektrophorese getestet werden kann.
Drei entscheidende Teile des MOs-Screening-Prozesses müssen standardisiert werden: (i) Die MO-Dosiskurve muss auf die phänotypische Erkennung abgestimmt werden. Die Dosiskurve zeigt auch die letale Dosis 50 (LD50: die Dosis, bei der 50 % der injizierten Embryonen sterben) für jedes getestete MO, um die Fähigkeit zu verbessern, das phänotypische "Signal" im Vergleich zum Off-Target-"Rauschen" zu optimieren3. (ii) Die angepasste Nomenklatur der Phänotypisierung sollte gut dokumentiert sein; Eine präzise und leicht verständliche phänotypische Beschreibung ist entscheidend, um umfassende Erklärungen auf der Grundlage der vorhandenen Literatur und der Erfahrung der Prüfärzte zu liefern und den Informationsaustausch zwischen denjenigen zu erleichtern, die die Embryonen nicht direkt untersucht haben. (iii) Eine klar definierte Sprache macht es einfach, Daten zentral aus der Morpholino Database8 zu sammeln.
In MO-Knockdown-Studien für kardiale Gene müssen die Herzaktivität und die Blutflussdynamik der Tiere überwacht werden, um den Einfluss von MO-Knockdown-Experimenten auf die Funktion des Herz-Kreislauf-Systems zu bestimmen. Solche Analysen erfordern eine Echtzeit-Visualisierung des Herz-Kreislauf-Systems mit hoher Auflösung. Die Haut des Zebrafisches ist in der ersten Entwicklungswoche transparent und ermöglicht die Visualisierung des Herzens und des Blutkreislaufs durch Mikroskopie. Für die Beurteilung der Herzfunktion sind die am häufigsten berechneten physiologischen Parameter die Herzfrequenz und das Herzzeitvolumen sowie die fraktionierte Verkürzung, die fraktionierte Flächenveränderung und die Ejektionsfraktion. Blutflussgeschwindigkeiten können durch Verfolgung von sich bewegenden Erythrozyten gemessen werden, und diese Messungen werden verwendet, um das Scherspannungsniveau zu bestimmen, ein entscheidender mechanobiologischer Faktor auf Endothelzellen. Eine solche Beurteilung erfordert die Aufnahme von Zeitrafferfilmen für das schlagende Herz und das fließende Blut mit einem inversen oder Stereomikroskop, das mit einer Hochgeschwindigkeitskamera ausgestattet ist.
Diese Arbeit zeigt, wie man ein translationsblockierendes Morpholino gegen ein interessierendes Gen in das Eigelb frisch befruchteter Zebrafischembryonen entwirft, vorbereitet und mikroinjiziert, um die Genfunktion zu beeinträchtigen. Es wird auch gezeigt, wie diese "Morphanten" durch Co-Injektion von mRNA, die für dieses Gen kodiert, gerettet werden können. Anschließend werden wir die Wirksamkeit der Morpholino-Mikroinjektionen durch phänotypische Charakterisierungen sowie kardiale Struktur- und Funktionsanalysen analysieren. Dieser Ansatz wird an einem viel untersuchten kardiologischen Gen, der Hand2, demonstriert.
Protokoll
Alle Versuche wurden in Übereinstimmung mit den anerkannten Standards der humanen Tierhaltung gemäß den Vorschriften der IACUC an der QU durchgeführt; Die Tiere wurden in der Zebrafischanlage des Qatar University Biomedical Research Center (QU-BRC) gehalten. Alle Tiere, die in diesen experimentellen Studien verwendet wurden, waren weniger als 3 Tage nach der Befruchtung (dpf).
HINWEIS: Für jede Versuchsgruppe ist es ratsam, mindestens 30 Embryonen zu verwenden, um die statistische Genauigkeit zu gewährleisten. Die Versuchsgruppen sind wie folgt:
Kontrollgruppe: Zu dieser Gruppe gehören Embryonen, die ohne Injektionen in Eiwasser kultiviert wurden. Die Ergebnisse hier bilden die Kontrollbasis.
Negativkontrollgruppe: Zu dieser Gruppe gehören Embryonen, die in Eiwasser kultiviert und mit Rührmilch injiziert wurden.
Injizierte Gruppe: Zu dieser Gruppe gehören Embryonen, denen nur hand2 MO und hand2 MO hand2 mRNA injiziert wurde, um den Phänotyp zu retten. Die Ergebnisse bestätigen, dass die beobachteten Phänotypen aufgrund von injizierten MOs auftraten. Der Vergleich der Versuchsgruppen wird es ermöglichen, den Einfluss der Hemmung und Rettung der Hand2 auf die Herzfunktion genau zu beurteilen.
1. Morpholino-Designs für Hand2.
HINWEIS: MO-Sequenzen können aus der Literatur angepasst werden 9,10,11. Alternativ können diese Oligos online mit Gen-Tools entworfen werden. Gene-tools bietet einen kostenlosen und schnellen Online-Design-Service an, auf den über die Website12 zugegriffen werden kann. Ein benutzerdefiniertes MO kann leicht entworfen werden, indem Informationen über die interessierenden Gene bereitgestellt werden, wie z. B. Sequenzinformationen oder Zugangsnummern. Die folgenden spezifischen Schritte fassen zusammen, wie MOs gegen hand2 im Zebrafisch entworfen werden:
- Suchen Sie zunächst in der NCBI-Datenbank nach Details des interessierenden Gens. Für Hand2, im Zebrafisch13.
- Holen Sie sich die GenBank mRNA-Transkript-ID von NCBI14.
- Beziehen Sie die mRNA-Sequenz von NCBI15.
HINWEIS: Morpholino (MO)-modifizierte Antisense-Oligonukleotide für das Zebrafisch-Hand2-Gen sind verfügbar und wurden bereits veröffentlicht. Hand2 MO-Sequenz 9,10,11: 5'-CCTCCAACTAAACTCATGGCGACAG-3', MO-Sequenzen, die in der Studie verwendet wurden, sind in der Materialtabelle aufgeführt. - Erhalten Sie eine Negativkontrolle entweder von einem nicht übereinstimmenden oder verzerrten MO (ähnliche Sequenzen mit zufälligen Basenpaaränderungen) von den zu injizierenden Genwerkzeugen16. Dies würde dazu beitragen, die Spezifität des Phänotyps bzw. der Phänotypen, die bei spezifischen MO-Injektionen beobachtet werden, zu bestätigen und das Risiko zu minimieren, dass die beobachteten Wirkungen auf ein Artefakt des Injektionsverfahrens zurückzuführen sind.
2. Vorbereitung der Morpholino-Injektion
- Vorbereitung des Stoffes
- MO-Bestände mit ddH2O auflösen; Diethylpyrocarbonat (DEPC) kann MOs schädigen und für Embryonen giftig sein. Verwenden Sie daher DEPC-freies ddH2O. Bei der Bestellung werden MOs in einem Fläschchen mit einer Konzentration von 300 nM geliefert.
HINWEIS: Obwohl Gene Tools 1 mM Stammlösungen (~8 ng/nL) empfehlen, kann dies zu niedrig sein, insbesondere wenn das Morpholino eine hohe Dosis erfordert oder mit anderen MOs gemischt wird. Daher ist es ideal für die Herstellung verschiedener Konzentrationen von MO-Stammlösungen (2 oder 3 mM). - Bereiten Sie eine Arbeitslösung von 0,2 mM vor, indem Sie die Arbeitslösung unterverdünnen (d. h. für 2 mM Material, 1:10 Verdünnung). Bereiten Sie 10 μl Arbeitslösung vor, indem Sie Danieaux-Lösung (7 μl), Stammmorpholino (1 μl) und Phenolrot (2 μl aus 10x Stamm) hinzufügen. Phenolrot verleiht der Lösung eine dunkelrote Farbe, um die Verfolgung des injizierten Materials im Embryo zu erleichtern.
- Bewahren Sie MOs bei Raumtemperatur (RT) in luftdichten Behältern auf, um eine Verdunstung zu verhindern. Bewahren Sie MOs nicht auf Eis auf der Arbeitsplatte auf, da die Lösung ausfallen kann. Die RT-Handhabung ist am besten geeignet.
HINWEIS: Gene Tools empfiehlt eine von zwei Lagerungsmethoden für MOs: Die Arbeitslösung kann bei -20 °C (für viele Monate bis Jahre) oder RT in einem verschlossenen Röhrchen für die Langzeitlagerung aufbewahrt werden. Bei einer Temperatur von -20 °C die Oligonukleotidlösung 10 Minuten lang auf 65 °C erhitzen und vor der Verwendung einen Vortex durchführen, um das Morpholino vollständig aufzulösen (bei RT muss es nicht erwärmt werden).
- MO-Bestände mit ddH2O auflösen; Diethylpyrocarbonat (DEPC) kann MOs schädigen und für Embryonen giftig sein. Verwenden Sie daher DEPC-freies ddH2O. Bei der Bestellung werden MOs in einem Fläschchen mit einer Konzentration von 300 nM geliefert.
- In-vitro-Transkription von mRNA
HINWEIS: Die In-vitro-Transkription wurde verwendet, um mRNA von Hand2 aus dem Plasmid HAND2 (NM_021973) Human Tagged ORF-Klon zu generieren, der in Zebrafischembryonen mikroinjiziert wurde, um den Phänotyp aufgrund des Knockdowns von Zebrafisch Hand2 mit Morpholinos zu retten.- Linearisieren Sie die Plasmid-DNA durch ein geeignetes Restriktionsenzym und reinigen Sie sie mit einem DNA-Aufreinigungskit (Table of Materials). Verwenden Sie die gereinigte linearisierte DNA als Vorlage für die In-vitro-Transkription , um mRNA für die Injektion gemäß den Anweisungen des Herstellers zu generieren (Materialtabelle).
- Injizieren Sie schließlich 250 pg menschliche mRNA für das interessierende Gen, um die Rettung des Phänotyps durchzuführen und zu testen, ob das Fehlen eines Phänotyps tatsächlich auf den Verlust des Zebrafisch-Gens durch MO zurückzuführen ist.
- Vorbereitung der Injektion
- Ziehen Sie eine Nadel mit einem Mikropipettenzieher (Materialtabelle) und befolgen Sie die Schritte 2.3.2 bis 2.3.6.
- Verwenden Sie 1 mm Kapillarröhrchen mit Filamenten und einem Mikropipettenabzieher. Schalten Sie die Maschine ein und öffnen Sie den Deckel
- Drehen Sie den Moduswahlknopf auf NO.2 Heizung. Stellen Sie mit der NO.2-Heizung den Knopf ein, um die Hitze auf 68 °C zu bringen
- Drehen Sie den Moduswahlknopf zurück in Schritt 1 . Verwenden Sie vier Gewichte (2 Type Light / 2 Type Heavy)
- Setzen Sie die Kapillare auf die Halterung und schließen Sie den Deckel. Drücken Sie die Start-Taste , um die gezogene Nadel zu erhalten.
HINWEIS: Dadurch werden Kapillarnadeln mit einer langen Verjüngung erzeugt - Schneiden Sie die Spitze der Nadeln mit einer Pinzette kurz ab. Schärfen Sie die Spitze der gezogenen Nadel für ein leichtes Eindringen in die Zebrafischembryonen mit einem Kapillaranfaser für ca. 30 s, wodurch Spitzen mit einem Durchmesser von ca. 15-25 μm entstehen.
- Kalibrieren Sie die Nadel, um die Injektionsmenge gemäß den Schritten 2.3.8-2.3.13 zu schätzen.
- Mischen Sie mRNA/Morpholino-Lösung kurz, bevor Sie sie in die Nadel laden, um die Partikel aufzulösen, die die Nadel vollständig verstopfen könnten.
- Geben Sie ~3-4 μl MO-Lösung mit einer Pipettenspitze mit Mikrometerspitze in den hinteren Teil der Injektionsnadel. Die Injektionsnadeln sind mit einem kleinen Graben ausgestattet, um die Kapillarverfüllung zu erleichtern.
- Vergewissern Sie sich, dass sich keine Blasen in der Injektionsnadel befinden. Montieren Sie die Nadel am Mikroinjektor.
- Prüfen Sie, ob die Spitze der Nadel nicht verstopft ist und dass MO-Lösung aus der Nadel fließen kann, indem Sie das Einspritzpedal betätigen.
- Kalibrieren Sie das Injektionsvolumen, indem Sie die Tröpfchengröße messen. Legen Sie die Nadelspitze in Mineralöl auf einen Mikrometerschieber. Drücken Sie das Einspritzpedal und stellen Sie den Einspritzdruck (psi) und/oder die Einspritzzeit (ms) ein, bis der Durchmesser des ausgestoßenen Tropfens 0,1 mm beträgt, was 0,5 nL entspricht.
- Stellen Sie sicher, dass das Injektionsvolumen etwa 0,5-1 nL beträgt. Passen Sie das Volumen an, indem Sie den Einspritzdruck (Auswurf), den Druck (psi) oder die Zeit (ms) ändern.
- Führen Sie für das Protokoll zur Einrichtung des Pikoliter-Injektors die Schritte 2.3.15-2.3.16 aus
- Verwenden Sie einen Pikoliter-Injektor für Zebrafisch-Injektionen. Schalten Sie die Förderpumpe des Injektors ein.
- Bereiten Sie den Injektor gemäß den unten gezeigten Anforderungen vor:
- P-Balance: Stellen Sie sicher, dass der P-Saldo bei 0 liegt. Stellen Sie sicher, dass es leicht negativ ist, um zu verhindern, dass Eigelb in die Nadel zurückfließt, und verdünnen Sie die MO-Lösung in der Nadel. Ist der Gegendruck hingegen zu hoch, fließt der MO auch ohne Druck auf das Pedal ständig aus der Nadel, was zu Variabilität und Inkonsistenzen in der injizierten Dosis und den beobachteten Phänotypen führt.
- P-Injektion: 20-25 psi ist ideal; Ändern Sie diese Option, um die Injektionsmenge anzupassen. Dies kann zwischen 10 und 30 psi liegen, aber beginnen Sie mit ~20 psi, um zu überprüfen, ob es das gewünschte Volumen liefert.
- Einspritzzeit: Stellen Sie sicher, dass die Injektionszeit auf 300 ms reduziert wird. Bevor Sie das Injektionsvolumen einstellen, passen Sie den Zeitpunkt der Injektion an.
- Für die Morpholino-Injektion befolgen Sie die Schritte 2.3.18-2.3.19.
- Bereiten Sie die Injektionskammer wie folgt vor: Stellen Sie 2% Agarose aus 0,6 g Agarose und 30 ml E3-Medium her. Gießen Sie die Agarose in eine Petrischale und platzieren Sie eine Spritzgussform mit einer TU-1-Spritzgussform auf die Agarose. Nach dem Abkühlen im Gefrierschrank bilden sich Furchen, um die Embryonen zu platzieren und zu stabilisieren (Abbildung 1A).
HINWEIS: Die Rezeptur der 60x E3M-Stammlösung besteht aus 5,0 mM NaCl, 0,17 mM KCl, 0,16 mM MgSO4,7H 2O, 0,4 mM CaCl2,2H 2O in 1 L ddH2O mit einem endgültigen pH-Wert von 7,6. - Übertragen Sie die entnommenen Embryonen mit einer Transferpipette in die Furchen, entfernen Sie das überschüssige E3-Medium um die Embryonen herum, um das Schwimmen der Embryonen zu verhindern (Abbildung 1B).
3. Injektion von MO und mRNA-Lösung in das Eigelb
- Führen Sie die Nadel durch das Chorion ein, injizieren Sie nahe der Grenze von Zelle/Eigelb zur Embryoseite und drücken Sie das Injektionspedal (Abbildung 1C).
HINWEIS: Die injizierte MO-Lösung/Phenolrot diffundiert nicht sofort im Eigelb. Im Eigelb ist ein roter Fleck zu beobachten, der sich allmählich auflöst. Das injizierte Material sollte nicht mehr als 10 % der Embryogröße überschreiten. MOs können bis zum 8-Zell-Stadium in das Eigelb von frisch gelegten Zebrafischeiern mikroinjiziert werden, da die MOs von den sich entwickelnden Zellen leicht im zytoplasmatischen Strom des Dottersacks aufgenommen werden können 17,18. Bei Plasmid- oder gedeckelten mRNA-Injektionen sollte die Injektion in ein einzelnes Blastomer eines Embryos im Einzelzellstadium durchgeführt werden. - Die Embryonen werden in eine Petrischale mit E3-Medium überführt und bei 28 °C bis 3 dpf inkubiert, wobei täglich E3-Medium nachgefüllt wird. Führen Sie in diesem Stadium die phänotypische Beurteilung mit einem Mikroskop durch.
HINWEIS: MO-Lösungen müssen bei jeder Untersuchung sorgfältig verdünnt werden, um die niedrigste Konzentration zu ermitteln, die zur Induktion eines bestimmten Phänotyps erforderlich ist. Die embryonale Letalität für jeden MO muss ebenfalls bestimmt werden. Bei höheren Konzentrationen (über 4-10 ng, je nach MO) neigen MOs dazu, unspezifische Effekte wie Gehirn- oder allgemeinen Zelltod hervorzurufen. - Co-Injektion von MO mit 250 pg mRNA der menschlichen Hand2 auf die gleiche Weise, um die Rettung des Phänotyps zu bestätigen, der durch den Verlust des Zebrafisch-Hand2-Gens durch MO induziert wurde.
4. Western-Blot zur Verifizierung des Erfolgs des Morpholino-Knockdowns
- Entnehmen Sie die Embryonen zu bestimmten Zeitpunkten (48 hpf, 72 hpf) und dechorionieren Sie bei Bedarf mit dem Pronase-Enzym.
- Entyoldung der Embryonen, um Vitellogenin unter Verwendung einer bereits bekannten Methodezu entfernen 19.
HINWEIS: Vitellogenin ist ein Phospholipo-Glykoprotein, das als Nährstoffquelle für den wachsenden Embryo dient. Die hochauflösende 2D-Gelelektrophorese und das stark verbesserte Western Blotting werden durch das Deyolking der Embryonen und die Entfernung von Vitellogenin ermöglicht. - Führen Sie Western Blotting wie zuvor beschriebendurch 20.
5. Beurteilung der Herzstruktur und -funktion:
- Live-Bildgebung des Zebrafisches: Bildgebung des Ventrikels
HINWEIS: Mehrere Herzfunktions-/hämodynamische Parameter können berechnet werden, indem das schlagende Herz des Zebrafischembryos21,22 visualisiert wird. Zu diesen Parametern gehören das Herzzeitvolumen (CO), die Ejektionsfraktion (EF), das Schlagvolumen (SV), die fraktionierte Verkürzung (FS) und die fraktionierte Flächenveränderung (FAC) (siehe frühere Arbeiten mit detaillierten Protokollen zur Beurteilung der Herzfunktion für Zebrafischembryonen22,23). Die folgenden Schritte erklären kurz, wie diese Parameter für Zebrafischembryonen bei ~3 dpf berechnet werden, einem Stadium, in dem die Haut dieser Organismen transparent ist, was eine Visualisierung mit Hellfeldmikroskopie ermöglicht.- Geben Sie einen Tropfen 3%ige Methylcelluloselösung (RT) in den Objektträger für die konkave Vertiefung.
- Positionieren Sie den Zebrafischembryo mit einer geeigneten Kunststoffpipette in der Vertiefung (Abbildung 2A, B)
HINWEIS: Eine Überfüllung des Brunnens kann dazu führen, dass Fische aus dem Brunnen verdrängt werden. - Mischen Sie den 3%igen Methylcellulosetropfen vorsichtig mit dem E3-Medium, um den geschlüpften Embryo zu stabilisieren.
HINWEIS: Zur Herstellung einer 3%igen Methylcelluloselösung lösen Sie 3 g Methylcellulosepulver in 100 ml PBS oder einem anderen Einbettmedium in einem Kolben auf. Setzen Sie einen Rührmagneten in den Kolben der Mischung ein und stellen Sie den Kolben auf eine magnetische Rührplatte. Stellen Sie die Geschwindigkeit auf "niedrig" und halten Sie sie ~1 Tag lang bei 4 °C, um alle Klumpen aufzulösen. Sobald die Methylcellulose vollständig aufgelöst ist, aliquotiert sie in kleine Röhrchen und lagert sie bei -20 °C. - Positionieren Sie den Fisch auf der linken Seite, mit der rechten Seite nach oben und der vorderen Spitze nach links, um eine eindeutige Abbildung des Ventrikels zu ermöglichen (Abbildung 2C).
- Vergrößern Sie unter dem Mikroskop das Herz des Embryos mit 100-facher Vergrößerung und beginnen Sie mit der Aufnahme für ~5 s. Stellen Sie sicher, dass sich die Ventrikelränder innerhalb des Bildgebungsfensters befinden (Abbildung 2D).
- Nehmen Sie Zeitrafferfilme mit einer Hochgeschwindigkeitskamera und einem Stereomikroskop mit etwa 100 Bildern pro Sekunde (fps) des gesamten Embryos (Abbildung 2C), des schlagenden Ventrikels (Abbildung 2D) und der Bewegung roter Blutkörperchen (RBC) in wichtigen Gefäßen wie der dorsalen Aorta oder der hinteren Kardinalvene (Abbildung 2E) für die Analyse der Herzfunktion auf.
- Speichern Sie den Film entweder im AVI-Filmformat oder im TIFF- (oder JPEG-) Format für Bildsequenzen.
- Berechnen Sie die Herzfrequenz
- Berechnen Sie die Zeit, die benötigt wird, um zwei aufeinanderfolgende Frames aufzunehmen. Für 100 fps beträgt das Zeitintervall 0,01 s.
- Wählen Sie einen bekannten Punkt aus einem aufgezeichneten Herzzyklus (z. B. Enddiastole oder Endsystole). Berechnen Sie die Anzahl der Frames, die benötigt werden, um den Zyklus zu wiederholen.
- Multiplizieren Sie das Zeitintervall aus Schritt 5.2.1 mit der Anzahl der Frames aus Schritt 5.2.2. Das Ergebnis ist die Zeitdauer (in Sekunden) für einen Heartbeat.
- Um die Schläge pro Minute zu berechnen, dividieren Sie 60 durch die Zahl, die Sie im vorherigen Schritt erhalten haben. Bei normalen 3-dph-Embryonen sollte diese bei etwa 150 Schlägen pro Minute (2,5 Hz) liegen.
HINWEIS: Viele Softwareanwendungen können die Herzfrequenz automatisch aus den Aufzeichnungen des schlagenden Herzens berechnen, z. B. ViewPoint24 und DanioVision25. - Analysieren Sie die Herzstruktur mit einem aufgezeichneten Zeitrafferfilm mit etwa 100 Bildern pro Sekunde, um das gesamte Herz auf das Vorhandensein eines Herzödems oder eines anderen strukturellen Defekts zu überprüfen.
HINWEIS: Abbildung 3A zeigt ein normales Embryoherz mit 3 dpf und Abbildung 3B ein Herz mit 3 dpf mit Herzödem und Herzschleifendefekt (verlängerter Ventrikel).
- Berechnen Sie die fraktionierte Flächenänderung (FAC):
HINWEIS: Die fraktionierte Flächenveränderung (FAC) ist ein Parameter, der zum Vergleich von ventrikulären Enddiastolen- und Endsystolenbereichen verwendet wird, um die Kontraktilität des Ventrikels zu beurteilen.- Verwenden Sie einen aufgezeichneten Zeitrafferfilm mit etwa 100 Bildern pro Sekunde, um Bilder zu bestimmen, die einen Herzzyklus darstellen. Extrahieren Sie die Frames, die das Ende sowohl der Endsystole als auch der Enddiastole anzeigen.
- Berechnen Sie sowohl den Enddiastolenbereich (EDA) als auch den Endsystolenbereich (ESA) mit ImageJ oder einer ähnlichen Bildanalysesoftware.
- Verwenden Sie die folgende Formel, um FAC 22,26,27 zu berechnen:
FAC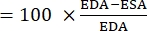
- Berechnen Sie die fraktionelle Verkürzung (FS):
HINWEIS: Die fraktionierte Verkürzung (FS) ist ein weiterer Parameter, der zur Bewertung der Kontraktilität des Ventrikels verwendet wird. Zur Bestimmung des FS müssen die enddiastolischen und endsystolischen Durchmesser der Ventrikel gemessen werden21.- Wiederholen Sie Schritt 5.3.1
- Verwenden Sie ImageJ oder ein anderes gleichwertiges Bildanalyseprogramm, um die Durchmesser der Ventrikelwände an den Punkten der Enddiastole Dd und der Endsystole Ds zu messen. In den meisten Fällen werden Kurzachsendurchmesser verwendet, um die fraktionale Glätte (FS) zu bestimmen (Abbildung 4).
- Verwenden Sie die folgende Formel, um FS 22,27 zu berechnen:
FS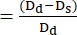
- Berechnung des Hubvolumens (SV):
HINWEIS: Für jeden Herzschlag ist die SV die Menge an Blut, die aus dem Ventrikel gepumpt wird, die leicht aus dem Enddiastolen- und Endsystolenvolumen der Ventrikelberechnet werden kann 28.- Wiederholen Sie die Schritte 5.3.1 und 5.3.2
- Messen Sie die Durchmesser der Enddiastole (D L) und der Endsystole (DS) wie in Abbildung 4 gezeigt.
HINWEIS: Unter der Annahme, dass die Ventrikel von Zebrafischherzen die Form einer prolaten sphäroidalen21,22 haben, wird das Ventrikelvolumen nach der folgenden Formel berechnet.
Volumen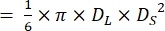
SV kann nach der folgenden Formelberechnet werden 27. Der normale SV-Bereich liegt bei 0,15-0,3 nL für 2-6 dpf Embryonen29. Hier ist EDV das Enddiastolenvolumen und ESV das Endsystolenvolumen.
SV = (EDV - ESV)
- Berechnen Sie die Ejektionsfraktion (EF):
HINWEIS: EF ist definiert als der Anteil des Blutes, der mit jedem Herzschlag28 aus dem Ventrikel ausgestoßen wird.- Extrahieren Sie aus den obigen Formeln EDV und ESV für den Fisch.
- Berechnen Sie EF wie folgt22,27:
EF (%)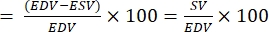
- Berechnen Sie das Herzzeitvolumen (CO):
HINWEIS: CO ist das Blutvolumen, das vom Herzen gepumptwird 28. CO hat einen Wert von 10-55 nL/min für 2-6 dpf Embryonen29.- Berechnen Sie SV und HR wie in den vorstehenden Unterabschnitten 5.5 und 5.2 erwähnt.
- Verwenden Sie die folgende Formel, um CO 22,27 zu berechnen:
CO (nL/min) = SV (nL/Beat) x HR (Beats/min).
- Messen Sie die Geschwindigkeiten der Erythrozyten:
HINWEIS: Die Bestimmung der Zellgeschwindigkeit ist entscheidend für die Bewertung der Flussrate, des Gefäßdurchmessers und die Berechnung der Scherspannung, die auf die Endothelzellen des Gefäßes ausgeübt wird (d. h. die Scherspannung hingegen ist die Reibungskraft, die durch bewegtes Blut auf die Endothelzellen ausgeübt wird). Bei Embryonen mit 2-5 dpf beträgt die durchschnittliche Geschwindigkeit der roten Blutkörperchen (RBC) etwa 300-750 μm/s29. So messen Sie die Geschwindigkeiten der Erythrozyten:- Vergrößern Sie unter dem Mikroskop den Schwanz des Tieres mit einer 100-fachen Vergrößerung. Die Bewegung der Erythrozyten sollte bei dieser Vergrößerung sichtbar sein (Abbildung 5A, B).
- Starten Sie die Aufnahme für ca. 8 s. Stellen Sie sicher, dass die dorsale Aorta (DA) und die hintere Kardinalvene (PCV), die beiden wichtigsten axialen Arterien, 100-120 fps innerhalb des Bildgebungsfensters haben. Verfolgen Sie einzelne Zellen aus sequenziellen Frames (Abbildung 5A, B).
- Extrahieren Sie mehrere Frames mit ImageJ oder einer ähnlichen Bildanalysesoftware.
- Berechnen Sie die Differenz in der Entfernung, die ein einzelner RBC bewegt (Δx).
- Bestimmen Sie die Zeitdifferenz zwischen den aufeinanderfolgenden Bildern (Δt).
- Verwenden Sie die folgende Formel für die RBC-Geschwindigkeit22,27:
RBC-Geschwindigkeit (μm/s)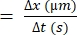
HINWEIS: Die maximale und durchschnittliche Geschwindigkeit können aus der Wiederholung aufeinanderfolgender Frames für einzelne Zellen21 extrahiert werden. Die Geschwindigkeit der Erythrozyten stellt auch die Geschwindigkeit des Blutflusses dar. Alternativ kann eine Vielzahl von kommerziell erhältlichen Softwareanwendungen verwendet werden, um die Geschwindigkeit von RBCs automatisch zu messen. Sowohl ViewPoint24 als auch DanioVision25 verfügen über solche Anwendungen (Abbildung 5C). Darüber hinaus gibt es einige verfügbare Plugins mit ImageJ, wie z. B. TrackMate30 und MTrackJ31, die mit Filmen kompatibel sind, die mit mehr als 100 fps aufgenommen wurden.
- Berechnung der Scherspannung aus gemessenen Zellgeschwindigkeiten
Die gemessenen Erythrozytengeschwindigkeiten in Blutgefäßen stellen auch die Blutflussgeschwindigkeiten dar (Abbildung 5C). Aus diesen Messungen wird die Schubspannung τ in einem Gefäß von Interesse wie folgt berechnet, wobei die Poiseuille-Strömungsannahme22,27 angenommen wird:
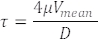
Dabei ist V die durchschnittliche Blutgeschwindigkeit (μm/s), μ die Blutviskosität (dynes/cm2) und D der Gefäßdurchmesser (μm). Bei 3 dpf Embryonen beträgt die Scherspannung in DA 22,26,27 etwa 4 dynes/cm 2.
Ergebnisse
Die Grafik in Abbildung 6 zeigt den durchschnittlichen Prozentsatz der Embryonen, die mit 24, 48 und 72 hpf überleben, sowohl für HAND2-spezifische MO- als auch für Kontroll-Crambled-MO-injizierte Embryonen. Die 1 mM (8 ng/μL) und 0,8 mM (6,4 ng/μL) MO-injizierten Embryonen zeigten eine signifikante Verringerung des Überlebensprozentsatzes im Vergleich zu Kontrollembryonen mit Crambled MO-injiziert. Dies wurde zu jedem gemessenen Zeitpunkt beobachtet, ...
Diskussion
Die Morpholino (MO)-Technologie wurde in großem Umfang in Zebrafischen, Xenopus, Seeigeln und in jüngerer Zeit in Zellkulturmodellsystemen eingesetzt. Bei den meisten Methoden gibt es neben den Vorteilen auch Fallstricke, die der Experimentator kennen sollte. Eine der größten Fallstricke bei der MO-Technologie ist die Befürchtung, dass phänotypische Effekte, die durch den MO-vermittelten Gen-Knockdown-Ansatz beobachtet werden, nicht auf den Funktionsverlust zurückzuführen sind, d...
Offenlegungen
Die Autoren erklären, dass keine finanziellen Interessen oder andere Interessenkonflikte bestehen.
Danksagungen
Die Veröffentlichung dieses Artikels erfolgte mit freundlicher Unterstützung von BARZAN HOLDINGS. RR wird teilweise durch R61HL154254 und Mittel der Abteilung für Pädiatrie und des Kinderkrankenhauses unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Acrylamide 40% | Sigma | Sigma, cat. no. C977M88 | |
| Agarose | Sigma-Aldrich | Sigma-Aldrich cat. no A9539-250G | |
| All Prep DNA/RNA Mini Kit | Qiagen | Qiagen cat. no. 80204. | |
| alpha Tubulin | Abcam | Abcam- ab4074 | Rabbit polyclonal to alpha Tubulin lot GR3 180877-1 (50 kDa) |
| Ammonium persulfate molecular grade | Sigma | Sigma, cat. no C991U65 | |
| BV10 capillary beveller | Sutter Instruments Product | Sutter Instruments Product Catalog # BV10 | |
| Chemiluminescence Imaging Gene Gnome | SYNGENE | SYNGENE | |
| Cleaver Scientific Blotting | CVS10D_OmniPAGEMini | CVS10D_OmniPAGEMini | |
| Coomassie | Thermo Fisher | Thermo Fisher cat. no C861C44 | |
| Electrochemiluminescence (ECL) kit | Abcam Biochemicals | Abcam Biochemicals cat. no ab65623 | |
| Glycine | Sigma | Sigma, cat. no C988U91 | |
| Goat anti Rabbit | Abcam | Abcam- ab6721 | Goat Anti-Rabbit IgG H&L (HRP) 2nd antibodies lot GR3179871-1 |
| HAND2 | Gene tools | Custom made for HAND2 (NM_021973) | 5'-CCTCCAACTAAACTCATGGCGAC AG-3' |
| Hand2 | Abcam | Abcam- ab10131 | Rabbit polyclonal Anti-HAND2 antibody lot GR143200-9 (24- 26 kDa) |
| HAND2 (NM_021973) Human Tagged ORF Clone | OriGene Technologies, Inc | RC224436L3 | Vector: pLenti-C-Myc-DDK-P2A-Puro (PS100092) |
| IBI DNA/RNA/Protein Extraction Kit | IBI Scientific | IBI Scientific cat. no -r IB47702 | |
| Imaging System | iBright | iBright CL1000 Imaging System | |
| Isopropanol | Sigma-Aldrich | Sigma-Aldrich cat. no 278475-2L | |
| Laemmli sample loading buffer (4x) | Sigma-Aldrich | Sigma-Aldrich cat. no 70607 | |
| Mercaptoethanol | Sigma | Sigma, cat. no M6250-1L | |
| Microplate Spectrophotometer with the Gen5 Data Analysis software interface | Epoch | Epoch | |
| Microscope | Ziess SteREO Lumar V12 Flourescence Microscope | Ziess SteREO Lumar V12 Flourescence Microscope | |
| Mineral oil | Fisher Scientific | Fisher Scientific cat. no 0121-1 | |
| mMESSAGE mMACHINE T7/T3/SP6 Transcription Kit | Thermo Fisher | Thermo Fisher cat. no.AM1340 | for mRNA generation |
| Nuclease-free water | New England Biolabs | New England Biolabs cat. no B1500L | |
| PC-100 Micropipette Puller | NARISHIGE GROUP Product | NARISHIGE GROUP Product Catalog # PC-100 | |
| Phenol red | Sigma | Sigma, cat. no. P-0290 | |
| Picolitre Injector | Harvard Apparatus | Harvard Apparatus cataloge # PLI-90A | |
| Pierce Bicinchoninic acid assay (BCA) Protein Assay kit | Thermo Fisher | Thermo Fisher cat. no 23227 | |
| PMSF, Protease inhibitor as protease inhibitors | Thermo Fisher | Thermo Fisher cat. no 36978 | |
| Ponceau S | Sigma-Aldrich | Sigma-Aldrich cat. no 10165921001 | |
| Protease Inhibitor Cocktail | Thermo Fisher | Thermo Fisher cat. no 88668 | |
| Protein ladder | SMOBiO | SMOBiO cat. no PM2500 | |
| Radioimmunoprecipitation Assay (RIPA) | Thermo Fisher | Thermo Fisher cat. no 89900 | |
| Ringer’s solution | Thermofisher | Catalog No.S25513 | |
| SDS | Sigma | Sigma, cat. no 436143 | |
| Standard Control | Gene tools | SKU: PCO-StandardControl-100 | 5'-CCTCTTACCTCAGTTACAATTTAT A-3'- that targets a human beta-globin intron mutation |
| Stripping buffer | Sigma-Aldrich | Sigma-Aldrich cat. 21059 | |
| Temed | IBI scientific | IBI scientific cat. no C000A52 | |
| Tris Base | Thermo Fisher | Thermo Fisher cat. no BP-152-500 | |
| Tween | sigma life science | sigma life science cat. no P2287 | |
| Zebra Box Revolution-Danio Track system chamber with the EthoVision XT 11.5 software | Noldus Information Technology, NL | Noldus Information Technology, NL | |
| Zeiss Axiocam ERc 5s | Zeiss | Stemi 508 Zeiss | |
| Zeiss Stemi 2000-C | Zeiss | Stemi 2000-C |
Referenzen
- Bakkers, J. Zebrafish as a model to study cardiac development and human cardiac disease. Cardiovascular Research. 91 (2), 279-288 (2011).
- Bill, B. R., Petzold, A. M., Clark, K. J., Schimmenti, L. A., Ekker, S. C. A primer for morpholino use in zebrafish. Zebrafish. 6 (1), 69-77 (2009).
- Stainier, D. Y., et al. Guidelines for morpholino use in zebrafish. PLoS Genetics. 13 (10), 1007000 (2017).
- Zhou, B., et al. Epicardial progenitors contribute to the cardiomyocyte lineage in the developing heart. Nature. 454 (7200), 109-113 (2008).
- Eve, A. M., Place, E. S., Smith, J. C. Comparison of Zebrafish tmem88a mutant and morpholino knockdown phenotypes. PLoS One. 12 (2), 0172227 (2017).
- Huang, W., Zhang, R., Xu, X. Myofibrillogenesis in the developing zebrafish heart: A functional study of tnnt2. Developmental Biology. 331 (2), 237-249 (2009).
- Chen, Z., et al. Depletion of zebrafish essential and regulatory myosin light chains reduces cardiac function through distinct mechanisms. Cardiovascular Research. 79 (1), 97-108 (2008).
- Bedell, V. M., Westcot, S. E., Ekker, S. C. Lessons from morpholino-based screening in zebrafish. Briefings in Functional Genomics. 10 (4), 181-188 (2011).
- Reichenbach, B., et al. Endoderm-derived Sonic hedgehog and mesoderm Hand2 expression are required for enteric nervous system development in zebrafish. Developmental Biology. 318 (1), 52-64 (2008).
- Maves, L., Tyler, A., Moens, C. B., Tapscott, S. J. J. Pbx acts with Hand2 in early myocardial differentiation. Developmental Biology. 333 (2), 409-418 (2009).
- Hinits, Y., et al. Zebrafish Mef2ca and Mef2cb are essential for both first and second heart field cardiomyocyte differentiation. Developmental Biology. 369 (2), 199-210 (2012).
- . Gene Tools, custom-sequence Morpholinos Available from: https://oligodesign.gene-tools.com/ (2011)
- . hand2 heart and neural crest derivatives expressed 2 [ Danio rerio (zebrafish) Available from: https://www.ncbi.nlm.nih.gov/gene/58150 (2021)
- . Danio rerio heart and neural crest derivatives expressed 2 (hand2), mRNA Available from: https://www.ncbi.nlm.nih.gov/nuccore/NM_131626.3 (2021)
- . NCBI reference sequence -Danio rerio heart and neural crest derivatives expressed 2 (hand2), mRNA FASTA Available from: https://www.ncbi.nlm.nih.gov/nuccore/NM_131626.3?report=fast (2021)
- . Prepared control oligos Available from: https://store.gene-tools.com/prepared-control-oligos (2021)
- Schubert, S., Keddig, N., Hanel, R., Kammann, U. Microinjection into zebrafish embryos (Danio rerio)-a useful tool in aquatic toxicity testing. Environmental Sciences Europe. 26 (1), 32 (2014).
- Moulton, J. D. Guide for morpholino users: toward therapeutics. Journal of Drug Discovery, Development, and Delivery. 3 (2), 1023 (2016).
- Link, V., Shevchenko, A., Heisenberg, C. -. P. Proteomics of early zebrafish embryos. BMC Developmental Biology. 6 (1), 1 (2006).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. Journal of Visualized Experiments: JoVE. (44), e2359 (2010).
- Shin, J. T., Pomerantsev, E. V., Mably, J. D., MacRae, C. A. High-resolution cardiovascular function confirms functional orthology of myocardial contractility pathways in zebrafish. Physiological Genomics. 42 (2), 300-309 (2010).
- Yalcin, H. C., Amindari, A., Butcher, J. T., Althani, A., Yacoub, M. Heart function and hemodynamic analysis for zebrafish embryos. Developmental Dynamics: An Officical Publication of the American Association of Anatomists. 246 (11), 868-880 (2017).
- Zakaria, Z. Z., et al. Using zebrafish for investigating the molecular mechanisms of drug-induced cardiotoxicity. Biomed Research International. 2018, 1642684 (2018).
- Parker, T., et al. A multi-endpoint in vivo larval zebrafish (Danio rerio) model for the assessment of integrated cardiovascular function. Journal of Pharmacological and Toxicological Methods. 69 (1), 30-38 (2014).
- Grone, B. P., et al. Epilepsy, behavioral abnormalities, and physiological comorbidities in syntaxin-binding protein 1 (STXBP1) mutant zebrafish. PLoS One. 11 (3), 0151148 (2016).
- Salman, H. E., Yalcin, H. C. Advanced blood flow assessment in Zebrafish via experimental digital particle image velocimetry and computational fluid dynamics modeling. Micron. 130, 102801 (2020).
- Benslimane, F. M., et al. Cardiac function and blood flow hemodynamics assessment of zebrafish (Danio rerio) using high-speed video microscopy. Micron. 136, 102876 (2020).
- DeGroff, C. G. Doppler echocardiography. Pediatric Cardiology. 23 (3), 307-333 (2002).
- Bagatto, B., Burggren, W. A three-dimensional functional assessment of heart and vessel development in the larva of the zebrafish (Danio rerio). Physiological and Biochemical Zoology. 79 (1), 194-201 (2005).
- Tinevez, J. -. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods (San Diego, Calif.). 115, 80-90 (2017).
- Meijering, E., Dzyubachyk, O., Smal, I. Methods for cell and particle tracking. Methods in Enzymology. 504, 183-200 (2012).
- Kok, F. O., et al. Reverse genetic screening reveals poor correlation between morpholino-induced and mutant phenotypes in zebrafish. Developmental Cell. 32 (1), 97-108 (2015).
- Sumanas, S. Inducible inhibition of gene function with photomorpholinos. Methods in Molecular Biology. 1565, 51-57 (2017).
- Kyritsi, K., et al. Knockdown of hepatic gonadotropin-releasing hormone by vivo-morpholino decreases liver fibrosis in multidrug resistance gene 2 knockout mice by down-regulation of miR-200b. The American Journal of Pathology. 187 (7), 1551-1565 (2017).
- Flynt, A. S., Rao, M., Patton, J. G. Blocking zebrafish microRNAs with morpholinos. Methods in Molecular Biology. 1565, 59-78 (2017).
- Schoenebeck, J. J., Keegan, B. R., Yelon, D. Vessel and blood specification override cardiac potential in anterior mesoderm. Developmental Cell. 13 (2), 254-267 (2007).
- Lu, F., Langenbacher, A., Chen, J. -. N. Transcriptional regulation of heart development in zebrafish. Journal of Cardiovascular Development and Disease. 3 (2), 14 (2016).
- Laurent, F., et al. HAND2 target gene regulatory networks control atrioventricular canal and cardiac valve development. Cell Reports. 19 (8), 1602-1613 (2017).
- Miura, G. I., Yelon, D. A guide to analysis of cardiac phenotypes in the zebrafish embryo. Methods in Cell Biology. 101, 161-180 (2011).
- De Luca, E., et al. ZebraBeat: a flexible platform for the analysis of the cardiac rate in zebrafish embryos. Scientific Reports. 4, 4898 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten