Method Article
オープンソースピペッティングロボットを活用した効率的なSARS-CoV-2定量逆転写酵素PCR唾液診断戦略
* これらの著者は同等に貢献しました
要約
このプロトコルは、オープンソースの自動化を利用して唾液サンプルのRT-qPCR分子検査を実行するSARS-CoV-2診断方法を記述しています。このスケーラブルなアプローチは、臨床公衆衛生サーベイランスや小規模な大学研究所の能力向上にも適用できます。
要約
最近のSARS-CoV-2グローバルヘルス危機の出現は、疫学研究と臨床試験に重要な課題をもたらしました。COVID-19のパンデミックは、高い感染率と低い死亡率を特徴とする、特に住宅大学などの閉鎖的な集団において、正確で効率的な診断検査を必要としました。上咽頭スワブのような核酸検査の初期の利用可能性は、サプライチェーンの圧力のために制限され、検査結果の報告も遅れました。唾液ベースの逆転写酵素定量ポリメラーゼ連鎖反応(RT-qPCR)試験は、感度および特異性において他の試験方法に匹敵することが示されており、唾液採取は参加者に対する物理的侵襲性が低い。その結果、クレムソン大学とその周辺コミュニティの集団サーベイランスのためのマルチプレックスRT-qPCR診断アッセイを開発しました。このアッセイでは、複雑な臨床自動化システムの代わりにオープンソースの液体処理ロボットとサーモサイクラーを使用して、ワークフローとシステムの柔軟性を最適化しました。唾液ベースのRT-qPCRの自動化により、大規模および小規模の両方の試験要求に対して、幅広いウイルスRNA濃度を迅速かつ正確に検出できます。自動システムの平均ターンアラウンドは、サンプルの95%で9時間<、サンプルの99%で24時間<でした。1回の試験のコストは、すべての試薬を一括購入した場合、2.80ドルでした。
概要
新型コロナウイルスである重症急性呼吸器症候群関連コロナウイルス-2(SARS-CoV-2)は、2019年後半に出現し、世界人口全体に急速に広がりました1。SARS-CoV-2感染は、潜在的に重度の呼吸器および炎症症状を伴う伝染性の高い疾患であるコロナウイルス病2019(COVID-19)を引き起こす。高い伝染性と低い死亡率は、ウイルスが集団を通じて急速に広がり、診断検査の増加が必要であることを示しました2,3。公衆衛生上の勧告は、症例を隔離し、その後感染率を低下させるための大規模な集団スクリーニングを奨励した4,5,6。さらに、集団サーベイランスのモデルから、検査頻度の増加と報告時間の短縮は、検査感度の向上よりも感染伝播の減少に大きな効果があることが明らかになりました7。これは、感染者が早期に隔離され、感染の連鎖が断ち切られる可能性があるためです。
元の核酸増幅試験(NAAT)標準は、RT-qPCR8によって処理された鼻咽頭(NP)綿棒であった。しかし、この形態の検査では、相対コストの増加やサプライチェーンの圧力の悪化など、非常に大規模な集団に対するこの形式の検査で複雑さが生じます9,10。さらに、一般的なNAAT法(NPスワブ、中咽頭スワブ、中甲介スワブ、および鼻スワブを含む)の検体収集と処理の両方が、特殊な機器、試薬、および医療従事者に依存しています9,10。
NPスワブRT-qPCR検査の適切な代替品は、SARS-CoV-2検出のための正確な診断ツールである唾液ベースの検査です11,12,13,14。唾液サンプルに対してRT-qPCRを直接実行すると、NPスワブと同様の感度と特異性が得られます15。唾液検査がNP綿棒検査よりも優れている大きな利点の1つは、検体の自己収集を可能にすることです16。これにより、医療従事者の必要性が最小限に抑えられ、NP綿棒よりも侵襲性が低いため、患者のサンプル収集の容易さが最大化されます。さらに、唾液サンプルは綿棒からサンプルを除去するために緩衝液を必要としないため(NPサンプルの場合のように)、唾液ベースの検査では熱ベースのリボ核酸(RNA)抽出を直接利用することができ、追加のバッファー、輸送媒体、および/またはRNA抽出試薬の必要性を排除することで検査コストを削減できます14,17。
クレムソン大学の疾患診断と介入の研究と教育(REDDI)ラボは、COVID-19の検査とサーベイランスに対する大学のニーズに対処するために設立されました。大学を含む閉鎖的な集団では、頻繁なサーベイランス検査と社会的距離が相まって、疾患罹患率の疫学的モデルにおいて最も好ましい結果がもたらされました18。統合されたCDC 2019-nCOV RT-qPCR19およびSalivaDirect14プロトコルが適応され、自動化が臨床ワークフローで利用され、コストを削減し、ターンアラウンドタイムを改善しました。これまでのグループは、SARS-CoV-2 RNA抽出ステップ20,21にオープンソースの液体処理ロボットを使用していましたが、テストプレートの作製と試料のロードにロボットを最大限に活用しました22。ここでは、適応されたプロトコルとオープンソースの液体処理システム(図1)の利用により、迅速かつ正確な唾液ベースのRT-qPCRが可能になり、大規模な公衆衛生サーベイランスに効果的な戦略であることを示しています。
プロトコル
すべての研究は、クレムソン大学およびPrisma Health Institutional Review Boards(Prisma Health IRB # Pro00099491、July 1, 2020)に準拠して実施されました。
1. オープンソースの液体処理ロボットのセットアップ
- メーカーの指示に従って、高効率微粒子空気(HEPA)フィルターモジュール( 材料表を参照)を各液体処理ロボットの上部に取り付けます。
- メーカーの指示に従って、マスターミックスプレート調製ロボットの左マウントに8チャンネルP20ピペットを取り付けます。
- メーカーの指示に従って、サンプルローディングロボットの右側のマウントにP20ピペットを取り付けます。
- カスタム Python スクリプト (補足ファイル 1 および 補足ファイル 2) を適切なコンピューターにダウンロードします。
- TigerSaliva Full 384 Loading.py をデスクトップアプリケーションで開き、サンプルローディングロボットコンピュータで使用します。「キャリブレーション」をクリックし、ソフトウェアの指示に従ってピペットとプログラムの両方を設定します。
- マスターミックスロボットコンピューターのデスクトップアプリケーションで 12フル Plates.py を開きます。 「キャリブレーション 」をクリックし、ソフトウェアの指示に従ってピペットとプログラムの両方を設定します。
- コンピュータ支援設計(CAD)ファイル(https://www.myminifactory.com/object/3d-print-141363)を使用して、溶融堆積モデリング3次元(3D)プリンタでカスタムサンプルラックを印刷します。サンプルローディングロボットあたり16ラックを印刷し、2セットの8ラックに対して印刷します。
2. 20倍マルチプレックスN1+P1プローブ/プライマーミックスの調製
- 合成SARS-CoV-2 RNAまたは患者検体から離れた滅菌環境で、50 mL 円錐管内に 20 x マルチプレックス N1+P1 プローブ/プライマーミックス (総容量 20 mL、 表 1) のバッチを調製します。
- 血清学的ピペットを用いて混合物1.6 mLを滅菌2.0 mL遠沈管にアリコートし、適切に標識した。
- アリコートは、使用準備が整うまで-20°Cの冷凍庫に保管してください。
3. ポジティブコントロールミックスの調製
注: ポジティブコントロールミックスは、プローブ/プライマーミックスまたは他のマスターミックス成分と同じ滅菌環境で作成しないでください。ヌクレアーゼを含まない水の別の容器を使用する必要があります。
- 990 μLのヌクレアーゼフリー水に10 μLのストック溶液を加えることにより、SARS-CoV-2合成RNA(N1)を1,000,000遺伝子コピー/μL(cpμ)から10,000cpμに希釈する。滅菌 0.2 mL チューブに 10,000 cpμ の 25 μL をアリコートし、適切に標識します。未使用のアリコートを-80°Cで保管してください。
注: 合成 RNA は、分解を防ぐために -80 °C で保存する必要があります。 - Hs_RPP30合成DNA(P1)を950 μLのヌクレアーゼフリー水に50 μLの原液を加えて200,000 cpμから10,000 cpμに希釈する。滅菌 0.2 mL チューブに 10,000 cpμ の 25 μL をアリコートし、適切に標識します。未使用のアリコートを-80°Cで保管してください。
- 960 μLのヌクレアーゼ非含有水にSARS-CoV-2ストックとHs_RPP30 10,000 cpμストックの両方を20 μL加え、合計1000 μLにして、各成分を終濃度200 cpμに希釈します。アリコートは、使用できる状態になるまで-80°Cで保管してください。
4. マスターミックスプレートの作製
注:合成SARS-CoV-2 RNAまたは患者検体から離れた滅菌環境でマスターミックスを作成します。すべての成分は、混合物に添加する前に完全に解凍されなければならない。適切な解凍を行わないと、濃度が正しくない可能性があります。不十分な解凍は、氷の存在または試薬の不均一な色によって示される。混合物を調製しながら冷凍庫ブロックに保管する。
- マルチプレックスマスターミックス(総容量48mL、 表2)のバッチを50mL円錐管に調製する。
- チューブを3倍に回して混合物を均質化します。上下にピペッティングしたり、ボルテックスをかけたりして混合すると酵素が損傷するため、混ぜないでください。
- 滅菌 96 ウェル深部ウェルリザーバーのカラム 1 ~ 4 を充填するには、1.48 mL のマスターミックスを各ウェルに移します。1つの50mL円錐形が4つのカラムを満たし、12プレートに十分です。
- 深い井戸リザーバをホイルシールで覆い、専用のマスターミックス液体処理ロボットに入れます。充填された深井戸リザーバをデッキ10に置き、6つの空の384ウェルプレートをデッキ1〜6に置き、P20チップボックスをデッキ11に配置します。深い井戸プレートとチップボックスを発見し、ロボットを閉じます。
- カスタム Python 操作プロトコルを初期化するには、ロボットデスクトップアプリケーションで [実行の開始] をクリックします。
- 40 分後、実行は一時停止します。充填された384ウェルプレートをロボットに保持している間にホイルシールで覆い、ローラーで押し下げて接着性を確保します。各プレートの端にバッチ識別子のラベルを付けます。
- 6 つの新しい空の 384 ウェル プレートをデッキ 1 ~ 6 に配置し、[実行の再開] をクリックして操作プロトコル を再開します。ランが完了したら、384ウェルプレートの最終セットをホイルシールで覆います。
- 残りのマスターのピペット 2 μL を、バッチ品質管理用の空の 384 ウェルプレートのカラム 1 ~ 3 および 22 ~ 24 に混合します。光学的に透明なシールでシールし、サーモサイクラーで走ります(セクション10.1-10.2)。いずれかのウェルにN1閾値サイクル(Ct)値がある場合、または10を超えるウェルがP1 Ct値を有する場合、バッチは汚染されており、使用できません。
- 調製したマスターミックスプレートを4°Cで保管し、調製後7日以内に使用する。
5. サンプル採取、取水、熱処理
- 唾液採取の30分前に、食事、飲酒、喫煙、または歯科衛生の実施を避けるよう参加者に指示する。参加者に、口の中に自然に溜まる少なくとも1 mLの唾液を収集し、防腐剤を含まない滅菌50 mL円錐形チューブに堆積させ、チューブにキャップをするように指示する(補足ファイル3)。
- 唾液収集チューブの外側を70%エタノールまたは消毒用ワイプで除染し、検査室に移して試験します。
- 各サンプルバーコードをスキャンして、毎日の摂取スプレッドシート(補足ファイル4)にサンプルの到着を記録します。
- スキャンしたサンプルを95°Cのオーブンで30分間熱処理します。耐熱手袋を着用したままサンプルを取り出します。
注:未処理のサンプルは、室温(23°C)で最大72時間安定しています。熱処理後、サンプルはすぐに処理されない場合は、4°Cで保存する必要があります。
6. サンプル割当
- サンプルアサインメントステーションのコンピューターで、各サンプルローディングロボット(補足ファイル5)の毎日のサンプルローディングスプレッドシートを開きます。
- サンプル 1 ~ 48 は第 1 四半期、サンプル 49 ~ 96 は第 2 四半期、サンプル 97 ~ 144 は第 3 四半期、サンプル 145 ~ 188 は第 4 四半期と見なされます。サンプルは重複して分析されます。
- プレート名、日付、および四半期番号が記載されたラベルトレイ。サンプルを順番にスキャンして、サンプル読み込みスプレッドシートに入れます。
メモ: 図 3 で定義されているように、以前のプレートからのサンプルは手動で実行する必要があります。手動サンプルの割り当てとロード手順については、セクション 8.1 ~ 8.3 を参照してください。
7. サンプルローディングロボットの操作
- サンプルローディングステーションでは、ロボットのデッキ配置に対応する8つの3Dプリントラックの2つのフルセットを並べます。
- 4分の 1 チューブのキャップを外し、ラック 1 の位置 A1 から始めて、3D プリントされたラックに入れます。各ラックを左から右、上から下まで満たします。このロード・パターンをラック 2 で続行してから、ラック 4 および 5 に進みます ( 図 2C を参照)。
メモ: ラックには、ロボットプログラムパラメータの都合上、連続した番号が付けられていません。 - ロードされたクォーター 1 サンプル・ラックをデッキ 1、2、4、5、7、8、10、11 に置きます。P20のヒントをデッキ3と9に置きます。セットアッププロセスを簡素化するには、材料を背面から前面までロボットにロードします。
- 4°Cから既製のマスターミックスプレートを取り出し、プレート名でラベルを付け、鋭利な刃を使用してコントロールウェル(N23/24、O23/24、およびP23/24)の周りのホイルの線を切断します。
- マスターミックスプレートをデッキ6に置き、ホイルカバーを剥がして、コントロールウェルを覆う小さな長方形を残します。先端ボックスを発見し、ロボットを閉じます。
- カスタム Python 操作プロトコルを初期化するには、ロボット デスクトップ アプリケーションで [実行の開始] をクリックします。各四半期はプレートに積み込むのに24.5分かかります。タイマーをリマインダーとして設定します。
- ロボットの実行中に、セクション7.2で説明されているように、4分の1サンプルチューブのキャップを外して、3Dプリントされたラックの2番目のセットにロードします。
- ロボットが一時停止したら、第 1 四半期ラックを取り外し、第 2 四半期ラックと交換します。デスクトップ・アプリケーションで 「実行の再開 」をクリックします。
- 4分の1サンプルチューブを要約し、結果を待っている間に4°Cの冷蔵庫に保管してください。
この積載プロセスを第 3 四半期と第 4 四半期に繰り返します。 - 装填されたプレートをバイオセーフティキャビネットに移す。汚染を最小限に抑えるために、転写中はプレートを覆ったままにしてください。
8. 手動サンプルローディング
メモ: ロボットの読み込みが不十分な場合は、繰り返しサンプルに対して 1 回の手動実行 (N1 Rerun または Rerun、 図 3 を参照) を実行します。
- 繰り返しサンプルを収集し、第4四半期の最後のサンプルとして割り当てます(セクション6.2を参照)。サンプルに番号を付け、バーコードをスキャンし、元のサンプルの場所と結果をサンプルローディングスプレッドシートに入力します。
- 繰り返しサンプルをバイオセーフティキャビネットに移します。繰り返しサンプルチューブをロボットローディングラックに積み込まないでください。
- 各反復サンプルのピペット2 μLをプレートレイアウト図(補足ファイル6)に従って正しいウェルに。患者サンプルを追加するには、指定されたピペットを使用してください。汚染を最小限に抑えるためにサンプルを追加しながら、ホイルで覆われた制御井戸を維持します。
9. テストプレートへのコントロールの追加
- 鉗子を使用して制御井戸の上のホイルカバーを剥がします。
- N23-N24ウェルにヌクレアーゼフリー水(テンプレートコントロールなし)2 μLをピペットし、200 cpμ混合ポジティブコントロール(セクション3を参照)の2 μLをウェルO23-O24にピペットします。確認された陽性患者サンプルコントロールのピペット2 μLを、追加のコントロールとしてウェルM23−M24に入れた。ウェルP23-P24を空のままにして、マスターミックスバッチの品質を監視します。
- プレートを光学的に透明なシールで覆い、アプリケーターローラーを使用してすべてのウェルにシールを接着します。プレートを2500rpmで30秒間渦巻き、徹底的に混合します。プレートを500 x g で1分間遠心分離します。
10. RT-qPCR の実行
- サーモサイクラーソフトウェアで、記載された条件に従ってプロトコルプログラムを作成します(表3)。将来のプレートのためにプロトコルを保存します。密閉プレートをサーモサイクラーに置き、プロトコルを実行します。
- Ct 値を .xslx ファイルとしてエクスポートし、その値をサンプル読み込みスプレッドシート (補足ファイル 5) にコピーします。
メモ: これらのシートは、製造元のソフトウェアからの Ct 出力ファイル用にカスタム設計されており、他の形式を受け入れるために変更が必要な場合があります。
11. プレートの有効性の決定
- 陽性対照および/または既知の陽性サンプルと陰性対照の両方を検証して、プレートの結果を有効と見なします。以下の基準で対照ウェルを評価した。
- 陽性コントロールの場合は、少なくとも1つのポジティブコントロールウェル(O23/O24)がP1プローブとN1プローブの両方で22〜28のCt値を生成するかどうかを確認します。あるいは、既知の陽性サンプルウェル(M23/M24)は、P1およびN1プローブでP1およびN1 Ct値<33を生成します。
- 陰性対照の場合、2つの陰性対照ウェル(N23/N24)のいずれにもN1またはP1 Ct値がないことを確認してください。プレートを無効にする前に、Ct値に有効な増幅曲線があることを確認してください。
12. サンプル結果の解釈
- 図(図3)に従って患者の結果を決定し、解決されたサンプルを報告します。
- P1 の結果を VALID または INVALID として評価します。P1 が Ct <33 の結果を生成する場合、ウェルは VALID と考え、N1 の結果に進みます。P1 が Ct >=33 の結果を生成する場合、または Ct 値がない場合、ウェルは無効であると考えてください。
- N1 の結果を YES、NO、または NO* として評価します。N1 が Ct <33 の結果を生成する場合、ウェルは YES です。N1 が Ct 値を生成しない場合、ウェルは NO です。N1 が Ct >=33 を生成する場合、ウェルは NO* です。すべてのN1 Ct値が実際の増幅曲線に関連付けられていることを確認します。N1 の Ct 値に増幅曲線がない場合、ウェルは NO です。
- 繰り返しサンプルを識別し(N1再実行または再実行)、内部サンプル番号とサンプルタイプでラベルを付け、ローディングワークフローに返します(セクション8.1-8.3)。
- P1 の結果を VALID または INVALID として評価します。P1 が Ct <33 の結果を生成する場合、ウェルは VALID と考え、N1 の結果に進みます。P1 が Ct >=33 の結果を生成する場合、または Ct 値がない場合、ウェルは無効であると考えてください。
13. 研究室の清掃
- リキッドハンドリングロボット
- 中級レベルの消毒剤ですべての側面を清掃します。エタノールはプラスチックを劣化させるので使用しないでください。
- ピペットチップの端とごみ箱をアルコール(70%エタノールまたは100%イソプロパノール)で優しく拭き取ります。キーボードとマウスを拭き取ります。
- バイオセーフティキャビネット
- 中級レベルの消毒剤ですべての表面を清掃します。UVライトを15分間オンにします。
結果
SARS-CoV-2(N1)およびHs_RPP30(P1)の両方の合成核酸含有量について、RT-qPCRプローブおよびプライマーの検出範囲を決定しました。既知の濃度の合成SARS-CoV-2 RNAおよび合成Hs_RPP30 DNAを水中で10倍段階希釈した。分子量を遺伝子コピー数に変換するために以下の式を使用した。
遺伝子コピー数 = (ng * 6.0221 x 1023)/((塩基対の長さ* 660 g/mole) *1 x 109 ng/g)
RT-qPCRを行った。RT-qPCRを実施した後、N1検出(図4A)およびP1検出(図4B)の線形曲線は、広範囲の遺伝子コピー濃度(それぞれR2=0.9975およびR2=0.9884)にわたって良好な相関係数を示した。この結果は、プライマーとプローブセットの組み合わせが阻害性がなく、1つの遺伝子コピー/μL(Cq=33)でSARS-CoV-2 RNAを正確に検出できることを示しています。1つの遺伝子コピーは、1つのウイルスコピーにほぼ等しい。しかし、RT-qPCRの半定量的性質のために、唾液中の定量的ウイルスコピー数は決定されなかった。我々は、既知の濃度の合成SARS-CoV-2 RNAをウイルスを含まない唾液(熱処理および非熱処理の両方)にスパイクすることによって陽性唾液サンプルをシミュレートすることを試みたが、低濃度のRNAでN1増幅を生成することができなかった(データは示さず)。これは、RNaseの分解または他の交絡因子に起因する可能性がある。
自動サンプルローディング法と手動サンプルローディング法の間のアッセイ間およびアッセイ内変動性も評価しました。アッセイ間の変動性を評価するために、マニュアル(セクション8.1~8.3に記載)および自動(セクション7.1~7.11に記載)を使用して、20個の固有の陽性サンプルをロードしました。N1 Ct値を比較して、液体処理ロボットと手動サンプルローディングが同等の結果をもたらしたかどうかを判断しました(図5A)。手動法と自動法の線形関係により、高い相関係数(R2= 0.9088)が得られ、両方の方法が機能的に同等であることを示しています。N1 Ct値が増加するにつれて、Ct値の変動性も増加した。この傾向は、唾液内のウイルス粒子の不均一な分布に起因する可能性が高く、これは、より少ない粒子が存在する場合により顕著である。アッセイ内変動性を評価するために、両方のサンプルローディング方法を用いて、固有の唾液サンプルの複製ウェルからのN1 Ct値間の比較が行われた(図5B)。自動サンプルローディング(R2= 0.9622)の反復間の線形関係は、手動ローディング(R2= 0.9589)のそれよりもわずかに高い相関係数を生成し、両方のローディング法でSARS-CoV-2検出の再現性が高いことを示しています。
最後に、熱処理方法に対する唾液粘度低下の評価を行った(図6)。唾液は、サンプルの変動を排除するために単一の供給源から得られた。1つの熱処理方法におけるP1 Ct値の変動が大きいことは、粘性のある唾液を正確に吸引および分配することができないため、サンプル粘度が高いことを示している可能性があります。30分熱処理法と60分熱処理法の両方で、処理対照なし(それぞれp = 0.0006およびp = 0.0429)と比較して、サンプル変動性が有意に減少しました。30分と60分の治療の間に有意差はなかった(p = 0.2245)。そこで、30分の熱処理方式を採用し、処理時間を短縮しました。
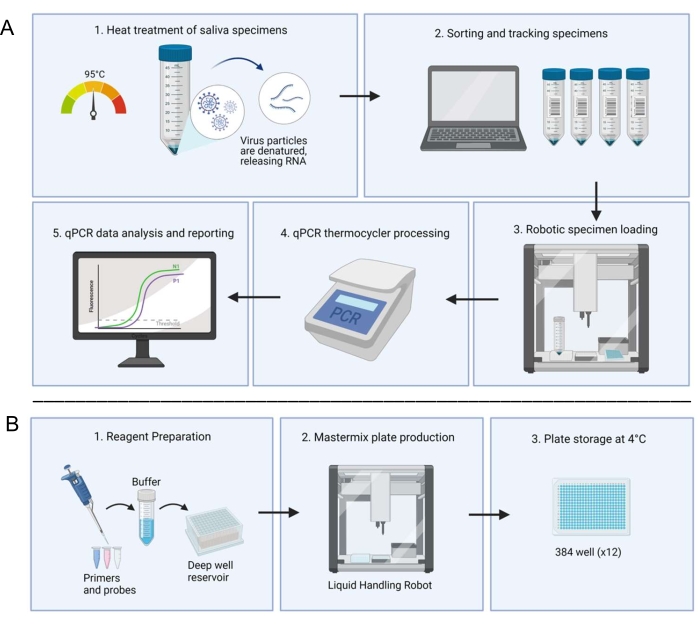
図1:唾液ベースのRT-qPCR診断システムを利用したラボワークフロー 。(A)サンプルを収集し、95°Cで30分間熱処理します。処理されたサンプルは、社内のスプレッドシートシステムを介して患者情報とともにソートおよび追跡されます。液体処理ロボットは、サンプルを調製されたマスターミックスプレートの複製ウェルにロードします。技術者が手動でコントロールをロードし、プレートを密封し、プレートをサーモサイクラーに入れて処理します。結果は、自動化されたコンピュータシステムを介して分析され、技術者によって検証されます。(B)技術者は、滅菌バイオセーフティキャビネット内の深い井戸リザーバに添加されるマスターミックス用の試薬を準備する。充填された深い井戸貯水池は、専用の液体処理ロボットにロードされます。完成したプレートはホイルで密封され、ラベルが貼られ、4 °C で保管 されます。この図の拡大版を表示するには、ここをクリックしてください。
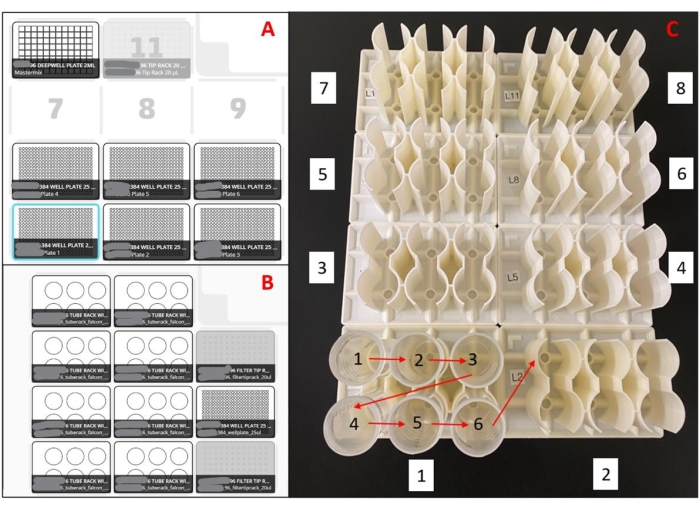
図2:液体ハンドリングロボットに使用されるレイアウト 。(A)マスターミックスプレート準備ロボットのデッキレイアウト。8チャンネルのピペットにより、ロボットはピペットチップを拾い上げ、96ウェルの深いウェルリザーバからマスターミックスを吸引し、マスターミックスを空の384ウェルプレートに分配し、ピペットチップをゴミ箱に排出するようにプログラムされています。これは、1 回の実行につき 6 つのプレートに対して繰り返されます。(B) サンプルローディングロボットのデッキ設定シングルチャンネルピペットでは、ロボットはピペットチップを拾い上げ、唾液サンプルを吸引し、唾液サンプルを384ウェルマスターミックスプレートの二重ウェルに分配し、ピペットチップをゴミ箱に排出するようにプログラムされています。これは、実行ごとに48サンプルに対して繰り返されます。(C) 3Dプリントラックのサンプルチューブローディング順序。赤い矢印はラック内のロード順序を示し、白いボックスの数字はラックのセット全体のロード順序を示します。セットアップ全体では、188サンプルを384ウェルプレートに二重にロードします。 この図の拡大版を表示するには、ここをクリックしてください。
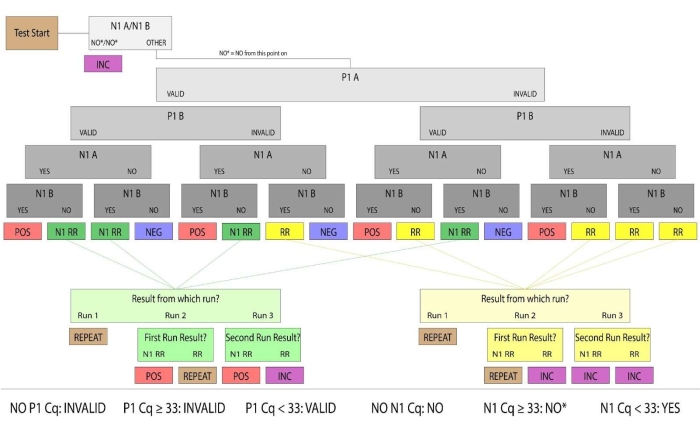
図 3: サンプルの結果のフローチャート。 有効なP1および陽性N1を有する試料は、SARS−CoV−2に対して陽性のヒト唾液試料であると決定された。有効および陽性/陰性のサンプル結果は決定的であると考えられた。最初の実行で決定的な結果をもたらさなかったサンプルは、再実行 (RR と表記) または N1 再実行 (N1 RR と表記) に分類されました。Rerunサンプルには有効なP1増幅はなく、N1Rerunサンプルは1回の反復でN1増幅が陽性でした。その後の手動実行で有効なP1増幅が得られない場合、または両方の反復でN1 Ct値が正の閾値(Ct>33)を超える場合、サンプル結果は決定的ではないと考えられました。臨床目的のために、実験室に到着しなかった患者サンプル、ピペットへの唾液の量が不十分であった、または損傷を受けた患者サンプルは無効とみなされた。 この図の拡大版を表示するには、ここをクリックしてください。
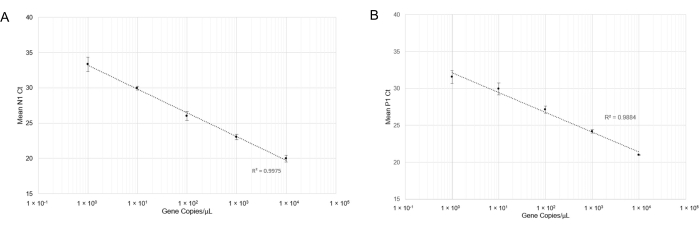
図4:N1(SARS-CoV-2)合成RNAおよびP1(Hs_RPP 30)合成DNAのRT-qPCR検出。標準曲線を標準偏差でプロットし、このプローブ/プライマーの組み合わせを使用して正確な検出の範囲を決定しました。(a)それぞれの希釈液で得られた平均Ct値(n=4)を、推定合成RNA量(10μLのRT-qPCR反応における1x100~1x104のRNAコピー)に対してプロットした。(b)各希釈液で得られた平均Ct値(n=3)を、推定合成DNA量(RT-qPCR反応10μLで1 x 100~1 x 104遺伝子コピー)に対してプロットした。この図の拡大版を表示するには、ここをクリックしてください。
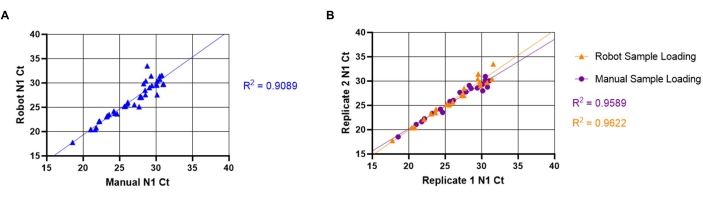
図5:手動と自動の唾液伝達SARS-CoV-2(N1)Ct値の比較。既知のSARS-CoV-2陽性唾液サンプル(n = 20)を、液体ハンドリングロボットによってRT-qPCRマスターミックスプレートに二重にロードした。サンプルの Ct 値は、N1 に対して 18 ~ 32 の範囲です。その後、同じサンプルを別のプレート位置の重複ウェルに手動でロードしました。(A)ロボットと手動サンプルローディングの両方を使用して固有のサンプルから得られたN1 Ct値を転置して、手動ローディングとロボットローディングの間のアッセイ間変動性を決定した。(B)アッセイ内変動性は、ロボットおよび手動サンプルローディングの両方から得られたN1 Ct値の転置反復を用いても決定した。この図の拡大版を表示するには、ここをクリックしてください。
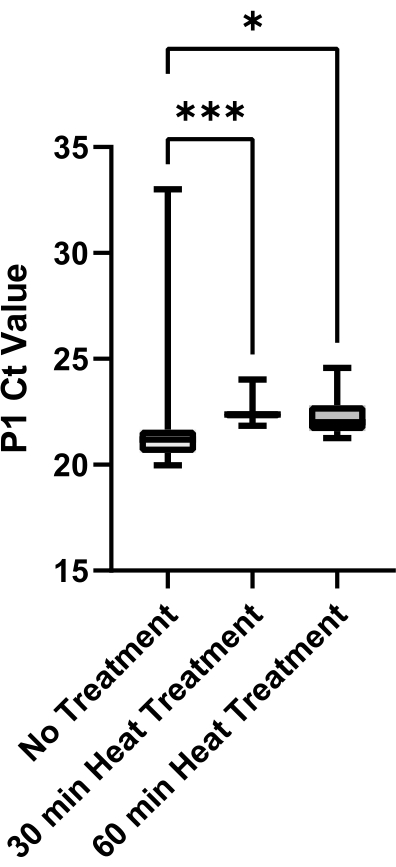
図6:唾液の粘度低下に対する熱処理方法の評価。 SARS-CoV-2陰性唾液を単一供給源から採取し、アリコートを95°Cで0分、30分、または60分間熱処理した。 各条件の技術的反復(n=12)からのP1 Ct値をプロットし、治療方法間の変動性を決定した。群間のペアワイズ比較は、非対応t検定で評価した(***はp<0.001を示し、*はp<0.05)を示す)。 この図の拡大版を表示するには、ここをクリックしてください。
補足図1:低P1 Ct唾液サンプルにおけるN1 Ctの比較。 P1 Ctの低い陽性サンプルを選択し、N1 Ctと比較した(n = 106)。N1 Ct値は14〜33の範囲であり、アッセイが標準曲線に匹敵する唾液サンプル中のダイナミックレンジを有することを示す。 このファイルをダウンロードするには、ここをクリックしてください。
| コンポーネント | シーケンス (5'→3') | 在庫濃度 | 容積 | ||
| 2019-nCoV-N1プローブ | /5FAM/ACCCCGCAT/ZEN/TACGTTTGGTGGACC/3IABkFQ | 50 μM | 500 μL | ||
| 2019-nCoV-N1-フォー | GACCCCAAAATCAGCGAAAT | 100 μM | 2000 μL | ||
| 2019-nCoV-N1-Rev | TCTGGTTACTGCCAGTTGAATCTG | 100 μM | 2000 μL | ||
| Hs RPP30 Cy5プローブ | /5Cy5/TTCTGACCT/ZEN/GAAGGCTCTGCGCG/3IABkFQ | 50 μM | 500 μL | ||
| Hs-RPP30-For | AGATTTGGACCTGCGAGCG | 100 μM | 2000 μL | ||
| Hs-RPP30-Rev | GAGCGGCTGTCTCCACAAGT | 100 μM | 2000 μL | ||
| 水 | - | - | 11000 μL | ||
表1:N1+P1プローブ/プライマーミックスの成分
| コンポーネント | 在庫濃度 | 反応あたりの体積 | 最終濃度 | バッチボリューム | ||
| ルナウォームスタートRT酵素ミックス | 20倍速 | 0.5 μL | 1倍速 | 3 ミリリットル | ||
| ルナバッファー反応ミックス | 2倍速 | 5.0 μL | 1倍速 | 30キロリットル | ||
| N1 + P1プライマー/プローブミックス | nCoV N1 F: 10 μM | 0.5 μL | 500 nM | 3 ミリリットル | ||
| nCoV N1 R: 10 μM | 500 nM | |||||
| プローブ nCoV N1: 2.5 μM | 125 nM | |||||
| RPP_30 P1 F: 10 μM | 500 nM | |||||
| RPP_30 P1 R: 10 μM | 500 nM | |||||
| プローブRPP_30 P1:2.5μM | 125 nM | |||||
| ヌクレアーゼフリーウォーター | --- | 2 μL | --- | 12キロリットル | ||
| 小計 | --- | 8 μL | --- | 48キロリットル | ||
| テンプレート | 2 μL | |||||
表2:マルチプレックスSARS-CoV-2マスターミックスの成分
| 舞台 | 温度(°C) | 期間 | サイクル数 |
| 逆転写 | 55 | 10分 | 1 |
| 初期変性 | 95 | 1分 | 1 |
| タッチダウン | 95 | 10秒 | 3 |
| 72 | 30秒 | ||
| 95 | 10秒 | 3 | |
| 69 | 30秒 | ||
| 95 | 10秒 | 3 | |
| 66 | 30秒 | ||
| 主な増幅 | 95 | 10秒 | 40 |
| 65 | 30秒 |
表 3: タッチダウン RT-qPCR プロトコルワンステップRT-qPCR SARS-CoV-2診断アッセイのための熱サイクル条件。
| タッチダウンステップ | タッチダウンステップなし | |||
| 平均 N1 Ct | 平均P1 Ct | 平均 N1 Ct | 平均P1 Ct | |
| サンプル1 | 19.65 | 22.7 | 27.8 | 28.3 |
| サンプル2 | 22.24 | 24.9 | 28.77 | 30.5 |
| サンプル3 | 18.85 | 19.2 | 24.65 | 25.9 |
| サンプル4 | 25.56 | 22.8 | 31.93 | 29.2 |
| サンプル5 | 22.34 | 24.8 | 38.48 | 40.0 (検出の失敗) |
表 4: 5 つの陽性サンプルのタッチダウン Ct 値と、タッチダウンなしの Ct 値の比較。
| 見本 | タイガー唾液 | 市販の唾液ベースのSARS-CoV-2アッセイ | ||
| N1 Ct | P1 Ct | Covid-19の価値 | RNaseP 値 | |
| D11 · | 16.4 | 18.1 | 20.86 | 23.4 |
| E11 · | 18.9 | 19.1 | 25.6 | 21.2 |
| F11 キー | 19.5 | 18.4 | 22.8 | 22.2 |
| G11 · | 22.2 | 19.1 | 23.7 | 22.9 |
| H11 · | 26.4 | 21.3 | 32.2 | 26.7 |
| A12 · | 14.8 | 16.5 | 29.15 | 19 |
| B12 · | 24 | 19.6 | 31.05 | 21.35 |
| C12 · | 14.9 | 17.5 | 20.84 | 18.9 |
表5:TigerSaliva Ctの結果と市販の唾液ベースのSARS-CoV-2アッセイ結果との比較。両方のアッセイを、同じ唾液サンプル(n=8)について実施した。
補足ファイル 1: ロボット マスター ミックス プレート作成用のカスタム スクリプト。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 2: サンプルローディングロボットの唾液処理用のカスタムスクリプト。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル3:参加者からの高品質の唾液サンプルの自己収集のための指示。 詳細については、https://www.clemson.edu/centers-institutes/reddilab/index.html で入手可能なテストプロセスの短いビデオ説明を参照してください。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 4: サンプル摂取量スプレッドシート。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 5: スプレッドシートの読み込みサンプル。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 6: サンプル 384 ウェルプレートレイアウト図。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
プロトコールに記載されたアッセイを、独立した検証研究によって評価した。同時に採取した対になった上咽頭綿棒に対して評価した場合、アッセイは98.9%の特異性(1.1%偽陽性)および90.0%の感度(10.0%偽陰性)を有することが判明した(n = 837;817陰性、20陽性)。重要なことに、TigerSalivaで陽性、鼻咽頭スワブで陰性であった3人の参加者が、48時間後に綿棒で再検査され、陽性の結果が返され、TigerSalivaが病気の経過の早い段階でSARS-CoV-2感染を検出できる可能性があることが示された。
我々は、既知の濃度のSARS-CoV-2合成RNAでウイルスフリーの唾液(熱処理および非熱処理の両方)をスパイクすることによって陽性の唾液サンプルをシミュレートし、10倍希釈を行って唾液中のCt限界を決定した。N1遺伝子は、シミュレートされた陽性サンプルにおいて10,000遺伝子コピー(約Ct=28)未満では検出できなかった。これはRNaseの分解やその他の交絡因子によるものと思われます。しかし、唾液RNaseと裸の合成RNAとの相互作用は、熱によって変性した後でも、ウイルス粒子との相互作用とは異なる可能性が高い。陽性の唾液サンプルはCt>30で同定されており、外部ラボはこれらのサンプルからSARS-CoV-2遺伝子配列データを取得しました。我々は、ウイルスタンパク質が患者の唾液サンプル中のRNA分解からの保護を提供すると推測している。
プロトコルの最も重要なステップは、マスターミックス調製と唾液サンプル処理(それぞれセクション4と7)の自動化の実装です。これにより、タスクプロセスが重複し、ターンアラウンドタイムが大幅に短縮されます。もう一つの重要なステップは、臨床結果の解釈である(セクション11および12)。中間結果カテゴリ (再実行および N1 再実行) を確立することで、決定的でないテスト結果の発生も最小限に抑えられました。
我々は、手動と自動の唾液サンプルローディング方法のばらつきはごくわずかであり(図5A)、自動化がSARS-CoV-2検出の再現性を向上させる可能性があることを実証しました(図5B)。臨床ラボの設計と拡張時にテストを容易にするために、自動化が優先されるべきです25。ラボのワークフローは、ロボットによる自動タスクの実装により改善されました26。リキッドハンドリングロボットのオープンソース機能により、プロトコル設計のためのカスタムスクリプトの実装が可能になります。これにより、液体処理ロボットは、従来の臨床自動化方法と比較して安価で高度に変更可能なシステムになります。また、反復性の高いラボタスクを実行するための理想的な戦略です。システムの高度なカスタマイズ性は、不足した場合にラボウェア(収集チューブ、ピペットチップ、384ウェルプレートなど)を自由に変更できることを意味します。したがって、液体処理ロボットを使用した自動化は、大規模および小規模の両方の監視および研究に実行可能です。
この検査戦略の主な利点は、他の臨床ラボと比較してターンアラウンドタイムがはるかに短いことです。自動液体処理ロボットの利用は、ターンアラウンドタイムの短縮に重要な役割を果たしますが、ロボットとサーモサイクラーの同時使用もテスト効率を最大化するのに役立ちます。1台のロボットとサーモサイクラーをペアで操作し、両方の機械を並行して使用して、中断のないサンプルローディングとサンプル結果分析を行う必要があります。割り当てられたサンプルの安定した流れが確立されると、すべての機械ペアを同時に操作できます。ロボットとサーモサイクラーを常時同時に使用すると、テスト能力と効率が大幅に向上し、大量のテストに対応するために不可欠です。
他の確立されたSARS-CoV-2 RT-qPCRプロトコルとは対照的に、サーモサイクラープロトコルにタッチダウンステップを組み込んで、標的遺伝子へのプローブとプライマーセットのアニーリングを改善し27、増幅失敗のリスクを低減しました。結果は、タッチダウンが特異的プライマー結合の喪失を危険にさらすことなく陽性サンプルの検出を改善したことを実証した(表 4)。我々は、広範囲のSARS-CoV-2 RNAコピー(図4A)とHs_RPP30 DNAコピー(図4B)の両方をRT-qPCRアッセイによって同時に検出できることを決定しました。
液体処理ロボットの限界の1つは、唾液移送中に陽性サンプルからの交差汚染の可能性です。唾液は粘弾性流体28であり、ピペットチップから分配された後、隣接するウェルを横切ってひもでつながれてもよい。さらに、唾液29の不均一性は、サンプル全体にわたるウイルス粒子の不均一な分布を引き起こし得る。これにより、偽陽性と陰性の両方が発生する可能性が高くなり、N1 再実行サンプルと再実行サンプルの指定が必要になります。しかし、最初にN1 Rerunとして指定されたサンプルの14.1%がSARS-CoV-2に対して陽性と判定され、Rerunサンプルよりも再検査後に陽性として解決する可能性が30倍以上高かった。その結果、RerunとN1 Rerunを区別することで(図3)、潜在的に陽性のサンプルをより正確に分離することができ、診断アッセイの感度と特異性が向上しました。診断唾液検査のための他の結果パラメータは、この区別をしなかった12,14,24,30,31。
唾液検体は、不均一性と粘度のためにピペットを流すのが難しい場合があります32。熱処理は、唾液バイオマトリックス中のタンパク質を適切に変性させ、粘度を低下させ、パンデミックの初期には不足していたRNA抽出試薬9の必要性を排除します10。延長熱処理はまた、現在のウイルスを不活性化し33、より低いバイオセーフティレベルでの実験室処理を可能にする。その結果、タンパク質の変性によって粘度を低下させるために、熱ベースのRNA抽出(セクション5.4で説明)が実施されました(図6)。この結果に基づいて、我々は、熱処理がタンパク質バイオマトリックスを変性させることに加えて、唾液サンプルを均質化する可能性があると仮定する。他のグループは、均質性を高めるために熱処理とプロテイナーゼK処理を組み合わせ9、14、34。ウイルスRNAを熱分解にさらす速度でビリオンタンパク質を変性させる可能性があるため、このステップを実施しないことを選択しました35。さらに、プロテイナーゼKによるサンプル希釈は、より少ないウイルス粒子を含む陽性サンプルをマスクし、したがって感度を低下させる可能性がある。さらに、アッセイ結果を、磁気ビーズRNA抽出を使用する市販の唾液ベースのSARS-CoV-2アッセイ(Logix Smart COVID-19)と比較した(表5)。現在のアッセイは、市販のアッセイと比較して、弱い陽性サンプルを検出するのにより適していることがわかりました。
qPCRは半定量的であるため、RT-qPCRのみを用いて唾液中のウイルスコピー数を定量することは困難である。Ct値の間には、技術的な制限に起因する固有のばらつきがあります。遺伝子コピー数は、Ct値(図4)から求めることができ、ウイルスコピー数とほぼ同等である。唾液サンプル中のウイルスコピー数を決定するための1つの可能な解決策はddPCRであり、これは反応中の遺伝子コピーの定量化を困難にする。しかし、臨床医に定性的な結果を提供することは適切であり、相対的なウイルス含有量は、当社の方法で処理されたサンプル間で比較することができると考えています。
唾液を使用する場合に生じるいくつかの制限にもかかわらず、唾液ベースのRT-qPCRによるSARS-CoV-2アッセイは、あらゆる規模の試験で迅速かつ信頼性の高いウイルスRNA検出のための効果的な方法であることが証明されています。これは、オープンソースの液体処理システムの利用と組み合わせると特に当てはまります。この検査アプローチは、感染症原体、疾患マーカー、または他のウイルスなど、診断に関連する他の核酸配列を検出するように変更することができる。これにより、アッセイは臨床および研究の両方の診断努力に適用可能になります。
開示事項
著者らは開示するものは何もありません。プロトコールに記載されているアッセイは、イェール大学公衆衛生大学院が提出したSalivaDirect EUAに該当します。
謝辞
著者らは、SARS-CoV-2検査の実施と管理を支援したREDDI Labのクレムソンの行政、医療スタッフ、臨床検査員に感謝する。サウスカロライナ大学のフィリップ・バックハウツ博士とキャロリン・バニスター博士に、初期のプロジェクトコンサルティングと機器調達に関する業界との連絡に感謝します。多くの学生、教授、スタッフの皆様のサンプル収集のご協力に感謝いたします。クリエイティブ・インクワイアメントの学生に、標準曲線データ収集のご協力をお願いいたします。この研究のための資金は、国立衛生研究所助成金P20GM121342(DDおよびLGPに授与)、クレムソン運動部、クレムソン大学の研究担当副学長、およびサウスカロライナ州知事および共同債券審査委員会から受領されました。
資料
| Name | Company | Catalog Number | Comments |
| 100% EtOH | Fisher scientific | 22-032-601 | |
| 20 uL Filtered Pipette Tips | Opentrons | 20uL tips | |
| 2mL Microcentrifuge Tubes | Fisher Scientific | 14-666-313 | Alternate product may be used |
| Armadillo PCR Plate, 384-well, clear, white wells | Thermo Scientific | AB3384 | Alternate product may be used |
| Celltreat 2mL 96 Deep Well Plates | Fisher Scientific | 50-828-743 | For mastermix preparation |
| Clear PCR Sealing Sheets | Thermo Scientific | AB0558 | Alternate product may be used |
| DPEC Treated Water | Ambion (Thermo Scientific) | AM9916 | |
| Flip Cap 50 mL Conical Tubes | VWR | 75845-210 | For sample collection |
| Foil PCR Sealing Sheets | Thermo Scientific | AB0626 | For storage of mastermix plates, Alternate product may be used |
| HS_RPP30 Synthetic DNA | Integrated DNA Technologies | 299788131 | P1 positive control |
| Luna Buffer Probe One-Step Reaction | New England Biolabs | M3006B | |
| Luna WarmStart RT Enzyme Mix | New England Biolabs | M3002B | |
| nCOV_N1 Forward Primer, 100 nmol | Integrated DNA Technologies | 10006830 | |
| nCOV_N1 Probe Aliquot, 50 nmol | Integrated DNA Technologies | 10006832 | Probe can be synthesized by other vendors with SYBR or FAM fluophores |
| nCOV_N1 Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006831 | |
| Opentron HEPA Filter Module | Opentrons | N/A | Not required, but useful to reduce contamination |
| Opentron Multichannel Attachment, P20 | Opentrons | 999-00005 | For mastermix preparation |
| Opentron OT-2 Liquid Handling Robot | Opentrons | OT-2 | |
| Opentron Pipette Attachment, P20 | Opentrons | 999-0000215 | For sample loading |
| Oven | Memmert | UF450 PLUS 208V-3PH | |
| PCR Tubes (rnase, dnase free) | Fisher Scientific | 14-230-225 | For aliquots of positive and neg controls |
| PolarSafe Aluminum Cooling Block, 15-Well (1.5/2.0 mL Tubes) | VWR | 10808-952 | |
| PolarSaf Aluminum Cooling Block, 24-Well (0.5mL tubes) | VWR | 10808-956 | |
| RNAse P (ATTO 647) Probe, 50 nmol | Integrated DNA Technologies | 10007062 | Probe can be synthesized by other vendors with Cy5 fluorophore |
| RNAse P Forward Primer, 100nmol | Integrated DNA Technologies | 10006836 | |
| RNAse P Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006837 | |
| Sars-CoV-2 Synthetic RNA Control 2 | Twist Biosciences | 102024 / 103907 / 103909 | N1 Positive Control |
| Scanners | Code | CR1500 | Only required when scaling up |
| Small HEPA Filtered Hood | Erlab | Captair Bio 321 | For mastermix preparation |
| Thermocycler CFX384 Touch | Biorad | CFX384 Touch | Alternate models can be used, e.g. CFX384 Opus |
| X-acto Knife Set | Staples | N/A | To cut foil for keeping control wells covered |
参考文献
- Zhu, N., et al. A novel coronavirus from patients with pneumonia in China, 2019. New England Journal Medicine. 382 (8), 727-733 (2020).
- Chen, J. Pathogenicity and transmissibility of 2019-nCoV-A quick overview and comparison with other emerging viruses. Microbes and Infection. 22 (2), 69-71 (2020).
- Petersen, E., et al. Comparing SARS-CoV-2 with SARS-CoV and influenza pandemics. The Lancet. Infectious Diseases. 20 (9), 238-244 (2020).
- Honein, M. A., et al. Summary of Guidance for Public Health Strategies to Address High Levels of Community Transmission of SARS-CoV-2 and Related Deaths, December 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (49), 1860-1867 (2020).
- World Health Organization. Surveillance strategies for COVID-19 human infection: interim guidance. World Health Organization. , Available from: https://apps.who.int/iris/handle/10665/332051 (2020).
- Screening testing for early detection of SARS-CoV-2 infection. Division of viral diseases. Centers for Disease Control. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/hcp/tsting-overview.html#PublicHealthSurveillance (2020).
- Larremore, D. B., et al. Test sensitivity is secondary to frequency and turnaround time for COVID-19 screening. Science Advances. 7 (1), 5393(2021).
- Sethuraman, N., Jeremiah, S. S., Ryo, A. Interpreting Diagnostic Tests for SARS-CoV-2. JAMA. 323 (22), 2249-2251 (2020).
- Chu, A. W., et al. Evaluation of simple nucleic acid extraction methods for the detection of SARS-CoV-2 in nasopharyngeal and saliva specimens during global shortage of extraction kits. Journal of clinical virology: the official publication of the Pan American Society for Clinical Virology. 129, 104519(2020).
- Guan, D., et al. Global supply-chain effects of COVID-19 control measures. Nature Human Behaviour. 4 (6), 577-587 (2020).
- Bastos, M. L., Perlman-Arrow, S., Menzies, D., Campbell, J. R. The Sensitivity and Costs of Testing for SARS-CoV-2 Infection With Saliva Versus Nasopharyngeal Swabs: A Systematic Review and Meta-analysis. Annals of Internal Medicine. 174 (4), 501-510 (2021).
- Pasomsub, E., et al. Saliva sample as a non-invasive specimen for the diagnosis of coronavirus disease 2019: a cross-sectional study. Clinical Microbiology and Infection: The Official Publication of The European Society of Clinical Microbiology and Infectious Diseases. 27 (2), (2021).
- To, K. K., et al. Consistent Detection of 2019 Novel Coronavirus in Saliva. Clinical Infectious Diseases: An Official Publication of The Infectious Diseases Society of America. 71 (15), 841-843 (2020).
- Vogels, C. B., et al. SalivaDirect: A simplified and flexible platform to enhance SARS-CoV-2 testing capacity. Med (New York, N.Y.). 2 (3), 263-280 (2021).
- Griesemer, S. B., et al. Evaluation of Specimen Types and Saliva Stabilization Solutions for SARS-CoV-2 Testing. Journal of Clinical Microbiology. 59 (5), 1418-1420 (2021).
- Wehrhahn, M. C., et al. Self-collection: An appropriate alternative during the SARS-CoV-2 pandemic. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 128, 104417(2020).
- Barza, R., Patel, P., Sabatini, L., Singh, K. Use of a simplified sample processing step without RNA extraction for direct SARS-CoV-2 RT-PCR detection. Journal of Clinical Virology: The Official Publication of the Pan American Society for Clinical Virology. 132, 104587(2020).
- Paltiel, A. D., Zheng, A., Walensky, R. P. Assessment of SARS-CoV-2 Screening Strategies to Permit the Safe Reopening of College Campuses in the United States. JAMA Network Open. 3 (7), 2016818(2020).
- Centers for Disease Control. 2019-Novel Coronavirus (2019-nCoV) Real-Time RT-PCR Primers and Probes. U.S. Department of Health and Human Services. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/lab/rt-pcr-panel-primer-probes.html (2020).
- Cresswell, K., Ramalingam, S., Sheikh, A. Can Robots Improve Testing Capacity for SARS-CoV-2. Journal of Medical Internet Research. 22 (8), 20169(2020).
- Villanueva-Cañas, J. L., et al. Implementation of an open-source robotic platform for SARS-CoV-2 testing by real-time RT-PCR. PLoS One. 16 (7), 0252509(2021).
- Robot Boosts COVID-19 Testing Efficiency. University of South Carolina College of Pharmacy. , Available from: https://sc.edu/study/colleges_schools/pharmacy/about/news/2020/robot-boosts-testing-effort.php (2020).
- Matic, N., et al. Practical challenges to the clinical implementation of saliva for SARS-CoV-2 detection. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of The European Society of Clinical Microbiology. 40 (2), 447-450 (2021).
- Sahajpal, N. S., et al. SalivaSTAT: Direct-PCR and Pooling of Saliva Samples Collected in Healthcare and Community Setting for SARS-CoV-2 Mass Surveillance. Diagnostics. 11 (5), Basel, Switzerland. 904(2021).
- Genzen, J. R., et al. Challenges and Opportunities in Implementing Total Laboratory Automation. Clinical Chemistry. 64 (2), 259-264 (2018).
- Archetti, C., Montanelli, A., Finazzi, D., Caimi, L., Garrafa, E. Clinical Laboratory Automation: A Case Study. Journal of Public Health Research. 6 (1), 881(2017).
- Hecker, K. H., Roux, K. H. High and low annealing temperatures increase both specificity and yield in touchdown and stepdown PCR. BioTechniques. 20 (3), 478-485 (1996).
- Bhat, P. P., et al. Formation of beads-on-a-string structures during break-up of viscoelastic filaments. Nature Physics. 6, 625-631 (2010).
- Miller, C. S., et al. Current developments in salivary diagnostics. Biomarkers in Medicine. 4 (1), 171-189 (2010).
- Moreno-Contreras, J., et al. Saliva Sampling and Its Direct Lysis, an Excellent Option To Increase the Number of SARS-CoV-2 Diagnostic Tests in Settings with Supply Shortages. Journal of Clinical Microbiology. 58 (10), 01659(2020).
- Wang, W., et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA. 323 (18), 1843-1844 (2020).
- Landry, M. L., Criscuolo, J., Peaper, D. R. Challenges in use of saliva for detection of SARS CoV-2 RNA in symptomatic outpatients. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 130, 104567(2020).
- Lista, M. J., et al. Resilient SARS-CoV-2 diagnostics workflows including viral heat inactivation. PLoS One. 16 (9), 0256813(2021).
- Brotons, P., et al. Validation and implementation of a direct RT-qPCR method for rapid screening of SARS-CoV-2 infection by using non-invasive saliva samples. International Journal of Infectious Diseases: IJID: Official Publication of The International Society for Infectious Diseases. 110, 363-370 (2021).
- Batéjat, C., Grassin, Q., Manuguerra, J. C., Leclercr, I. Heat inactivation of the severe acute respiratory syndrome coronavirus 2. Journal of Biosafety and Biosecurity. 3 (1), 1-3 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved