Method Article
Эффективная sars-CoV-2 количественная обратная транскриптаза ПЦР стратегия диагностики слюны с использованием роботов для пипетирования с открытым исходным кодом
* Эти авторы внесли равный вклад
В этой статье
Резюме
Протокол описывает диагностический метод SARS-CoV-2, который использует автоматизацию с открытым исходным кодом для выполнения молекулярного тестирования образцов слюны RT-qPCR. Этот масштабируемый подход может быть применен к клиническому надзору за общественным здравоохранением, а также для увеличения потенциала небольших университетских лабораторий.
Аннотация
Появление недавнего глобального кризиса в области здравоохранения SARS-CoV-2 создало ключевые проблемы для эпидемиологических исследований и клинических испытаний. Пандемия COVID-19, характеризующаяся высоким уровнем передачи и низкой смертностью, потребовала точного и эффективного диагностического тестирования, особенно в закрытых группах населения, таких как университеты-интернаты. Первоначальная доступность тестирования нуклеиновых кислот, таких как мазки из носоглотки, была ограничена из-за давления в цепочке поставок, которое также задерживало представление результатов испытаний. Тестирование количественной полимеразной цепной реакции (RT-qPCR) на основе слюны показало, что оно сопоставимо по чувствительности и специфичности с другими методами тестирования, а сбор слюны менее физически инвазивный для участников. Следовательно, мы разработали мультиплексный диагностический анализ RT-qPCR для наблюдения за населением Университета Клемсона и окружающего сообщества. В анализе использовались роботы с открытым исходным кодом для обработки жидкостей и термоциклеры вместо сложных систем клинической автоматизации для оптимизации рабочего процесса и гибкости системы. Автоматизация RT-qPCR на основе слюны позволяет быстро и точно обнаруживать широкий диапазон концентраций вирусных РНК как для крупномасштабных, так и для небольших испытаний. Средний оборот автоматизированной системы составил < 9 ч для 95% образцов и < 24 ч для 99% образцов. Стоимость одного теста составляла 2,80 доллара, когда все реагенты закупались в больших количествах.
Введение
Тяжелый острый респираторный синдром,ассоциированный коронавирус-2 (SARS-CoV-2), новый коронавирус, появился в конце 2019 года и быстро распространился среди населения мира1. Инфекция SARS-CoV-2 вызывает коронавирусную болезнь 2019 года (COVID-19), очень заразное заболевание с потенциально тяжелыми респираторными и воспалительными симптомами. Высокая трансмиссивность в сочетании с низкой смертностью указывает на то, что вирус будет быстро распространяться среди населения и потребует расширения диагностического тестирования2,3. Рекомендации общественного здравоохранения поощряли широкомасштабный скрининг населения для выявления случаев заболевания и последующего снижения показателей передачи4,5,6. Кроме того, модели популяционного эпиднадзора показали, что увеличение частоты тестирования и сокращение времени отчетности оказывают большее влияние на снижение передачи, чем повышение чувствительности тестов7. Вероятно, это связано с тем, что инфицированные люди могут быть помещены в карантин раньше, тем самым разрывая цепочки инфекции.
Первоначальным стандартом тестирования амплификации нуклеиновых кислот (NAAT) были мазки из носоглотки (NP), обработанные RT-qPCR8. Однако с этой формой тестирования для очень больших групп населения возникают осложнения, такие как увеличение относительной стоимости и обострение давления в цепочке поставок9,10. Кроме того, как сбор образцов, так и обработка общих методов NAAT (включая тампоны NP, ротоглоточные тампоны, мазки средней раковины и мазки из носа) зависят от специализированного оборудования, реагентов и медицинского персонала9,10.
Адекватной заменой тестированию NP-мазка RT-qPCR является тестирование на основе слюны, которое является точным диагностическим инструментом для обнаружения SARS-CoV-211,12,13,14. Непосредственное выполнение RT-qPCR на образцах слюны дает ту же чувствительность и специфичность, что и NP-мазки15. Одним из основных преимуществ тестирования слюны по сравнению с тестированием мазка NP является то, что оно позволяет самостоятельно собирать образцы16. Это сводит к минимуму потребность в медицинском персонале и максимизирует простоту сбора образцов для пациентов, будучи менее инвазивным, чем мазки NP. Кроме того, поскольку образцы слюны не требуют буферов для извлечения образца из тампона (как в случае образцов NP), тесты на основе слюны могут напрямую использовать экстракцию рибонуклеиновой кислоты (РНК) на тепловой основе, что снижает затраты на тестирование за счет устранения необходимости в дополнительных буферах, транспортных средах и / или реагентах экстракции РНК14,17.
Научно-исследовательская и образовательная лаборатория Университета Клемсона в области диагностики и вмешательства в болезни (REDDI) была создана для удовлетворения потребностей университета в тестировании и эпиднадзоре за COVID-19. В закрытых группах населения, включая университеты, частое тестирование эпиднадзора в сочетании с социальным дистанцированием дало наиболее благоприятные результаты в эпидемиологических моделях распространенности заболевания18. Консолидированные протоколы CDC 2019-nCOV RT-qPCR19 и SalivaDirect14 были адаптированы, а автоматизация была использована в клиническом рабочем процессе для снижения затрат и сокращения времени выполнения работ. Предыдущие группы использовали роботов с открытым исходным кодом для обработки жидкостей для этапов экстракции РНК SARS-CoV-220,21, но мы максимизировали использование роботов для подготовки тестовых пластин и образцов нагрузки22. Здесь мы показываем, что адаптированный протокол и использование систем обработки жидкостей с открытым исходным кодом (рисунок 1) позволяют быстро и точно проводить RT-qPCR на основе слюны и являются эффективной стратегией для крупномасштабного эпиднадзора за общественным здравоохранением.
протокол
Все исследования проводились в соответствии с Clemson University и Prisma Health Institutional Review Boards (Prisma Health IRB # Pro00099491, 1 июля 2020 г.).
1. Настройка робота с открытым исходным кодом для обработки жидкостей
- Установите высокоэффективные фильтрующие модули для твердых частиц (HEPA) (см. Таблицу материалов) в верхней части каждого робота для обработки жидкостей в соответствии с инструкциями производителя.
- Прикрепите 8-канальную пипетку P20 к левому креплению робота (роботов) для подготовки основной смеси в соответствии с инструкциями производителя.
- Прикрепите пипетку P20 к правому креплению робота (роботов) для загрузки образцов в соответствии с инструкциями производителя.
- Загрузите пользовательские скрипты Python (Дополнительный файл 1 и Дополнительный файл 2) на соответствующие компьютеры.
- Откройте TigerSaliva Full 384 Loading.py в настольном приложении на примере загрузки компьютеров-роботов. Нажмите « Калибровать» и настройте пипетку и программу в соответствии с указаниями программного обеспечения.
- Откройте 12 Full Plates.py в настольном приложении на компьютере master mix robot. Нажмите « Калибровать» и настройте пипетку и программу в соответствии с указаниями программного обеспечения.
- Печатайте пользовательские стойки для образцов с помощью трехмерного (3D) принтера для моделирования плавленого осаждения с помощью файла автоматизированного проектирования (CAD) (https://www.myminifactory.com/object/3d-print-141363). Печать 16 стоек на каждого робота по загрузке образцов для двух комплектов по восемь стоек.
2. Приготовление 20-кратной мультиплексной смеси зонд/грунтовки N1+P1
- Готовят партию из 20-кратной мультиплексной смеси зонда/праймера N1+P1 (общий объем 20 мл, таблица 1) в конической трубке объемом 50 мл в стерильной среде вдали от синтетической РНК SARS-CoV-2 или образцов пациента.
- Аликвот 1,6 мл смеси с использованием серологической пипетки в стерильные 2,0 мл центрифужных пробирок и соответствующим образом маркируют.
- Храните аликвоты в морозильной камере при температуре -20 °C до готовности к использованию.
3. Приготовление положительного контрольного микса
ПРИМЕЧАНИЕ: Положительная контрольная смесь не должна производиться в той же стерильной среде, что и смесь зонда/грунтовки или другие компоненты основной смеси. Следует использовать отдельную емкость с водой без нуклеаз.
- Разбавляют синтетическую РНК SARS-CoV-2 (N1) от 1 000 000 копий генов/мкл (cpμ) до 10 000 cpμ путем добавления 10 мкл исходного раствора к 990 мкл воды без нуклеазы. Аликвот 25 мкл 10 000 cpμ в стерильные пробирки 0,2 мл и соответствующим образом маркировать. Храните неиспользованные аликвоты при -80°C.
ПРИМЕЧАНИЕ: Синтетическая РНК должна храниться при -80°C для предотвращения деградации. - Разбавляйте Hs_RPP30 синтетической ДНК (P1) от 200 000 cpμ до 10 000 cpμ путем добавления 50 мкл исходного раствора в воду без нуклеазы 950 мкл. Аликвот 25 мкл 10 000 cpμ в стерильные пробирки 0,2 мл и соответствующим образом маркировать. Храните неиспользованные аликвоты при -80°C.
- Разбавляют каждый компонент до конечной концентрации 200 cpμ путем добавления 20 мкл как SARS-CoV-2, так и Hs_RPP30 10 000 cpμ запасов до 960 мкл воды без нуклеазы, в общей сложности 1000 мкл. Аликвот 20 мкл смешанного положительного контроля 200 cpμ в трубки 0,2 мл и метят соответствующим образом. Хранить аликвоты при -80°C до готовности к использованию.
4. Подготовка пластин мастер-микса
ПРИМЕЧАНИЕ: Делайте мастер-микс в стерильной среде вдали от синтетической РНК SARS-CoV-2 или образцов пациента. Все компоненты необходимо полностью разморозить перед добавлением в смесь; без надлежащего размораживания концентрации могут быть неправильными. О недостаточном оттаивании свидетельствует наличие льда или неравномерный цвет реагентов. Хранить на морозильной камере во время приготовления смеси.
- Приготовьте партию мультиплексной мастер-смеси (общий объем 48 мл, таблица 2) в конической трубке объемом 50 мл.
- Гомогенизируйте смесь, повернув трубку в 3 раза. Не смешивайте путем пипетирования вверх и вниз или вихря, так как это повредит фермент.
- Заполните колонны 1-4 стерильного 96-луночного глубинного ствола скважины путем переноса 1,48 мл мастер-смеси в каждую скважину. Одна коническая колонна объемом 50 мл заполняет четыре колонки, которых хватит на 12 пластин.
- Накройте резервуар глубокой скважины фольгированным уплотнением и поместите его в специальный робот для обработки жидкости master mix. Поместите заполненный резервуар глубокой скважины на палубу 10, поместите шесть пустых плит из 384 скважин на палубы 1-6 и наконечник P20 на палубу 11. Раскройте глубокую пластину скважины и наконечники коробок и закройте робота.
- Инициализируйте пользовательский операционный протокол Python, нажав кнопку Начать запуск в настольном приложении робота.
- Через 40 минут запуск будет приостановлен. Накройте заполненные 384 пластины скважин фольгой, пока они остаются в роботе, и прижмите роликом, чтобы обеспечить сцепление. Пометьте край каждой пластины идентификатором партии.
- Поместите шесть новых пустых плит из 384 скважин на палубы 1-6 и возобновите работу по протоколу, щелкнув Возобновить запуск. После завершения пробега накройте окончательный комплект из 384 плит скважин фольгой.
- Пипетка 2 мкл оставшейся основной смеси в колонны 1-3 и 22-24 пустой 384-луночной пластины для контроля качества партии. Уплотнение оптически прозрачным уплотнением и запуск на термоциклере (раздел 10.1-10.2). Если какие-либо скважины имеют значения порогового цикла N1 (Ct) или если более 10 скважин имеют значения P1 Ct, партия загрязнена и не может быть использована.
- Храните подготовленные пластины мастер-микса при температуре 4 °C и используйте их в течение 7 дней после приготовления.
5. Отбор проб, прием и термическая обработка
- Проинструктируйте участников избегать еды, питья, курения или проведения гигиены полости рта за 30 минут до сбора слюны. Проинструктируйте участников собрать не менее 1 мл слюны, которая естественным образом накапливается во рту, и поместить ее в стерильную коническую трубку объемом 50 мл без консервантов, а затем закрыть трубку (дополнительный файл 3).
- Обеззараживайте внешнюю часть пробирок для сбора слюны 70% этанолом или дезинфицирующими салфетками и передавайте их в лабораторию для тестирования.
- Запишите поступление образца, отсканировав каждый образец штрих-кода в электронную таблицу ежедневного приема (Дополнительный файл 4).
- Термически обработайте отсканированные образцы в течение 30 минут в духовке с температурой 95 °C. Извлекайте образцы, надевая термостойкие перчатки.
ПРИМЕЧАНИЕ: Необработанные образцы стабильны при комнатной температуре (23 °C) в течение 72 ч. После термической обработки образцы должны храниться при температуре 4 °C, если они не обрабатываются немедленно.
6. Образец назначения
- Откройте таблицы ежедневной загрузки образцов для каждого робота-загрузчика образца (Дополнительный файл 5) на компьютере на станции назначения образцов.
- На каждую 384-луночную пластину назначьте 188 образцов следующим образом: Образцы 1-48 считаются четвертью 1, образцы 49-96 считаются четвертью 2, образцы 97-144 считаются четвертью 3, а образцы 145-188 считаются четвертью 4. Образцы анализируются в двух экземплярах.
- Лотки для этикеток с названием пластины, датой и номером квартала. Отсканируйте образцы по порядку в электронную таблицу загрузки образцов.
ПРИМЕЧАНИЕ: Образцы с более ранних пластин, возможно, потребуется запустить вручную, как определено на рисунке 3. В разделах 8.1-8.3 приведены инструкции по ручному назначению образцов и загрузке.
7. Работающие роботы для загрузки образцов
- На станции загрузки образцов выстройте два полных комплекта из восьми 3D-печатных стеллажей, соответствующих размещению палубы в роботе.
- Снимите крышку с трубки четвертью 1 и поместите в 3D-печатные стойки, начиная с позиции A1 в стойке 1. Заполняйте каждую стойку слева направо и сверху вниз. Продолжите эту схему загрузки в стойке 2, затем перейдите к стойкам 4 и 5 (см. рисунок 2C).
ПРИМЕЧАНИЕ: Стойки не нумеруются последовательно из-за параметров программы робота. - Разместите загруженные четверти 1 пробные стеллажи на палубах 1, 2, 4, 5, 7, 8, 10, 11. Разместите наконечники P20 на палубах 3 и 9. Чтобы упростить процесс настройки, загрузите материалы сзади наперед в робот.
- Возьмите готовую пластину мастер-микса с температурой 4 °C, пометьте ее именем пластины и используйте острое лезвие, чтобы разрезать линию в фольге вокруг контрольных скважин (N23/24, O23/24 и P23/24).
- Поместите пластину мастер-микса на палубу 6 и снимите крышку из фольги, оставив за собой небольшой прямоугольник, покрывающий контрольные колодцы. Откройте коробки с наконечниками и закройте робота.
- Инициализируйте пользовательский операционный протокол Python, нажав кнопку Начать запуск через настольное приложение робота. Каждая четверть занимает 24,5 мин для загрузки на пластину; установите таймер в качестве напоминания.
- Пока робот работает, открутите и загрузите четверть 2 пробобой во второй набор 3D-печатных стоек, как описано в разделе 7.2.
- Когда робот сделает паузу, снимите стойки quarter 1 и замените их стойками quarter 2. Нажмите кнопку Возобновить запуск в классическом приложении.
- Сделайте обзор пробирок с образцами на четверть 1 и храните их в холодильнике с температурой 4 °C в ожидании результатов.
Повторите этот процесс загрузки для четвертей 3 и 4. - Перенесите загруженную пластину в шкаф биобезопасности. Чтобы свести к минимуму загрязнение, держите пластину закрытой во время переноса.
8. Ручная загрузка образца
ПРИМЕЧАНИЕ: Выполните один ручной запуск на повторных образцах (N1 Rerun или Rerun, см. Рисунок 3) в случае недостаточной роботизированной нагрузки.
- Соберите любые повторные образцы и назначьте их в качестве последних образцов в четверти 4 (см. раздел 6.2). Пронумеруйте образцы, отсканируйте штрих-коды и введите исходное местоположение образца и результат в электронную таблицу загрузки образца.
- Перенесите повторные образцы в шкаф биобезопасности. Не загружайте повторные пробоотборники в загрузочные стойки робота.
- Пипетка 2 мкл каждого повторного образца в правильные скважины в соответствии со схемой компоновки пластин (Дополнительный файл 6). Используйте специальную пипетку для добавления образцов пациента. Держите контрольные колодцы покрытыми фольгой, добавляя образцы, чтобы свести к минимуму загрязнение.
9. Добавление элементов управления к испытательным табличкам
- Снимите крышку из фольги над контрольными колодцами с помощью щипцов.
- Пипетка 2 мкл безнуклеазной воды (без шаблонного контроля) к скважинам N23-N24 и 2 мкл 200 cpμ смешанного положительного контроля (см. раздел 3) к скважинам O23-O24. Пипетка 2 мкл подтвержденного положительного контроля пробы пациента в скважины М23-М24 в качестве дополнительного контроля. Оставьте скважины P23-P24 пустыми, чтобы контролировать качество партии основной смеси.
- Накройте пластину оптически прозрачным уплотнением и используйте аппликаторный ролик, чтобы прикрепить уплотнение ко всем скважинам. Вихрьте пластину при 2500 об/мин в течение 30 секунд, чтобы тщательно перемешать. Центрифугировать пластину при 500 х г в течение 1 мин.
10. Выполнение RT-qPCR
- Создайте программу протокола в программном обеспечении термоциклера в соответствии с описанными условиями (таблица 3). Сохраните протокол для будущих пластин. Поместите герметичную пластину в термоциклер и запустите протокол.
- Экспортируйте значения Ct в виде XSLX-файла и скопируйте их в пример загрузочной электронной таблицы (дополнительный файл 5).
ПРИМЕЧАНИЕ: Эти листы были специально разработаны для выходных файлов Ct из программного обеспечения производителя и могут нуждаться в модификации для принятия других форматов.
11. Определение действительности пластины
- Проверьте как положительные контрольные и/или известные положительные образцы, так и отрицательный контроль, чтобы считать результаты пластины действительными. Оцените контрольные скважины по следующим критериям.
- Для положительного контроля проверьте, дает ли по крайней мере одна положительная контрольная скважина (O23/O24) значения Ct между 22-28 для датчиков P1 и N1. В качестве альтернативы, известные положительные пробные скважины (M23/M24) дают значения P1 и N1 Ct <33 на зондах P1 и N1.
- Для отрицательного контроля проверьте, что ни в одной из двух отрицательных контрольных ям (N23/N24) нет значений N1 или P1 Ct. Убедитесь, что значения Ct имеют допустимые кривые усиления, прежде чем аннулировать пластину.
12. Интерпретация результатов выборки
- Определите результат пациента по схеме (рисунок 3) и сообщите о разрешенных образцах.
- Оцените результат P1 как ДЕЙСТВИТЕЛЬНЫЙ или НЕДОПУСТИМЫЙ. Если P1 дает результат Ct <33, считайте скважину ДЕЙСТВИТЕЛЬНОЙ и переходите к результату N1. Если P1 дает результат Ct > =33 или нет значения Ct, считайте скважину НЕДОПУСТИМОЙ.
- Оцените результат N1 как ДА, НЕТ или НЕТ*. Если N1 дает результат Ct <33, то скважина ДА. Если N1 не дает значения Ct, скважина равна NO. Если N1 дает Ct >=33, то скважина равна NO*. Убедитесь, что все значения N1 Ct связаны с реальной кривой усиления. Если значение Ct для N1 не имеет кривой усиления, то скважина равна NO.
- Определите повторные образцы (N1 Rerun или Rerun), пометьте их внутренним номером образца и типом образца и верните их в рабочий процесс загрузки (раздел 8.1-8.3).
- Оцените результат P1 как ДЕЙСТВИТЕЛЬНЫЙ или НЕДОПУСТИМЫЙ. Если P1 дает результат Ct <33, считайте скважину ДЕЙСТВИТЕЛЬНОЙ и переходите к результату N1. Если P1 дает результат Ct > =33 или нет значения Ct, считайте скважину НЕДОПУСТИМОЙ.
13. Очистка лаборатории
- Роботы для обработки жидкостей
- Очистите все стороны дезинфицирующим средством среднего уровня. Не используйте этанол, так как он разрушит пластик.
- Аккуратно протрите конец наконечника пипетки и мусорное ведро спиртом (70% этанола или 100% изопропанола). Протрите клавиатуру и мышь.
- Кабинет биобезопасности
- Очистите все поверхности дезинфицирующим средством среднего уровня. Включите ультрафиолетовый свет на 15 минут.
Результаты
Определен диапазон обнаружения для зондов RT-qPCR и праймеров по содержанию синтетических нуклеиновых кислот как для SARS-CoV-2 (N1), так и для Hs_RPP30 (P1). Проведено 10-кратное серийное разведение известных концентраций комбинированной синтетической РНК SARS-CoV-2 и синтетической Hs_RPP30 ДНК в воде. Следующая формула была использована для преобразования молекулярной массы в число копий генов
Номер копии гена = (нг * 6,0221 x 1023)/((длина в парах оснований*660 г/моль) *1 x 109 нг/г)
и была проведена RT-qPCR. После проведения RT-qPCR линейные кривые для обнаружения N1 (рисунок 4A) и обнаружения P1 (рисунок 4B) показали хорошие коэффициенты корреляции в широком диапазоне концентраций копий генов (R2 = 0,9975 и R2 = 0,9884 соответственно). Этот результат указывает на то, что комбинация наборов праймера и зонда не является тормозной и может точно обнаруживать РНК SARS-CoV-2 при одной копии гена / мкл (Cq = 33). Одна копия гена примерно эквивалентна одной вирусной копии; однако мы не определили количественные цифры вирусных копий в слюне из-за полуколичественной природы RT-qPCR. Мы попытались смоделировать положительные образцы слюны путем добавления синтетической РНК SARS-CoV-2 известных концентраций в слюну без вирусов (как термически обработанную, так и нетермическую), но не смогли произвести амплификацию N1 при низких концентрациях РНК (данные не показаны). Это может быть связано с деградацией РНКазы или другими мешающими факторами.
Была также проведена оценка меж- и внутрипробирной изменчивости между автоматизированными и ручными методами загрузки проб. Для оценки межпробирной изменчивости 20 уникальных положительных образцов были загружены с использованием ручных (описанных в разделах 8.1-8.3) и автоматизированных (описанных в разделах 7.1-7.11) методов. Значения N1 Ct сравнивались, чтобы определить, дают ли роботы для обработки жидкостей и ручную загрузку образцов эквивалентные результаты (рисунок 5A). Линейная зависимость между ручными и автоматизированными методами дала высокий коэффициент корреляции (R2 = 0,9088), что указывает на то, что оба метода функционально эквивалентны. По мере увеличения значений N1 Ct вариабельность значений Ct также увеличивалась. Эта тенденция, вероятно, связана с гетерогенным распределением вирусных частиц внутри слюны, которое более выражено, когда присутствует меньше частиц. Для оценки вариабельности внутри анализа было проведено сравнение значений N1 Ct из реплицированных скважин уникальных образцов слюны с использованием обоих методов загрузки образцов (рисунок 5B). Линейная зависимость между репликами автоматизированной загрузки образца (R2 = 0,9622) дала несколько более высокий коэффициент корреляции, чем при ручной загрузке (R2 = 0,9589), что указывает на высокую воспроизводимость обнаружения SARS-CoV-2 для обоих методов загрузки.
Наконец, была проведена оценка снижения вязкости слюны по отношению к методам термической обработки (рисунок 6). Слюну получали из одного источника для устранения изменчивости образца. Большая вариабельность значений P1 Ct в рамках одного метода термообработки может свидетельствовать о более высокой вязкости образца, поскольку вязкая слюна не может быть аспирирована и дозирована точно. Оба метода термической обработки в течение 30 и 60 минут приводили к значительному снижению изменчивости образца по сравнению с отсутствием контроля обработки (p = 0,0006 и p = 0,0429 соответственно). Не было выявлено существенной разницы между 30 мин и 60 мин процедур (p = 0,2245); поэтому для сокращения времени обработки был внедрен 30-минутный метод термообработки.
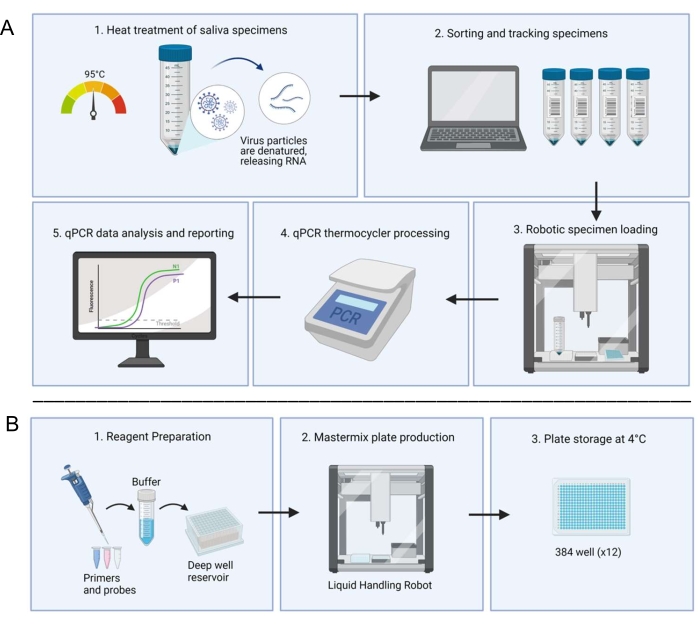
Рисунок 1: Лабораторный рабочий процесс с использованием диагностической системы RT-qPCR на основе слюны. (A) Образцы собираются и подвергаются термической обработке при 95 °C в течение 30 мин. Обработанные образцы сортируются и отслеживаются с информацией о пациенте через внутреннюю систему электронных таблиц. Робот для обработки жидкостей загружает образцы в дублирующиеся колодцы подготовленных пластин мастер-микса. Техник вручную загружает органы управления, герметизирует пластину и помещает пластину в термоциклер для обработки. Результаты анализируются с помощью автоматизированной компьютерной системы и проверяются техническим специалистом. (B) Техник готовит реагенты для основной смеси, которые добавляются в глубокий резервуар скважины в стерильном шкафу биобезопасности. Заполненные резервуары глубоких скважин загружаются в специальный робот для обработки жидкостей. Готовые пластины запечатаны фольгой, маркированы и хранятся при температуре 4 °C. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
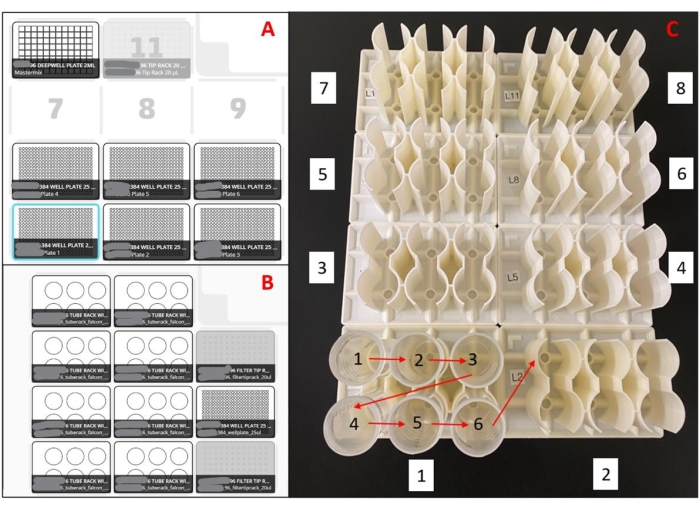
Рисунок 2: Макеты, используемые для робота по обработке жидкости. (A) Макет палубы для робота (роботов) для подготовки основной смеси. С помощью восьмиканальной пипетки робот запрограммирован на то, чтобы подбирать наконечники пипеток, аспирировать мастер-микс из резервуара скважины глубиной 96 скважин, дозировать мастер-микс в пустые пластины из 384 скважин и выбрасывать наконечники пипеток в мусорное ведро. Это повторяется для шести пластин за прогон. (B) Установка палубы для робота (роботов) загрузки образцов. С помощью одноканальной пипетки робот запрограммирован на то, чтобы взять наконечник пипетки, аспирировать образец слюны, дозировать образец слюны в дубликаты колодцев из 384-луночной пластины для смеси и выбросить наконечник пипетки в мусорное ведро. Это повторяется для 48 сэмплов за прогон. (C) Заказ на загрузку пробоотборников для 3D-печатных стоек. Красные стрелки обозначают порядок загрузки внутри стойки, а белые номера в коробке обозначают порядок загрузки всего набора стоек. Вся установка загрузит 188 образцов в двух экземплярах в плиту из 384 скважин. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
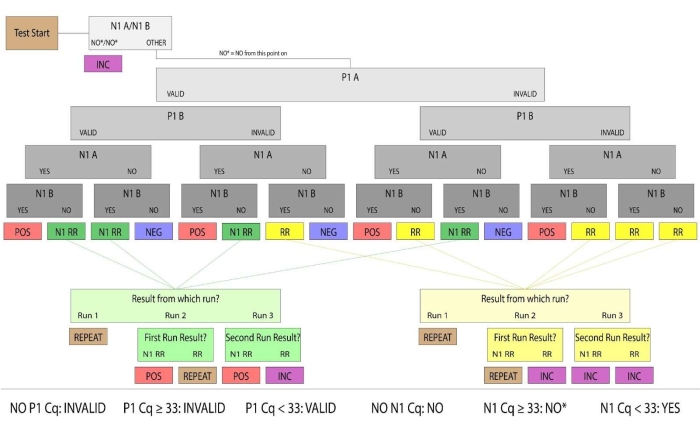
Рисунок 3: Образец результирующей блок-схемы. Образцы с действительным P1 и положительным N1 были определены как образцы слюны человека, положительные на SARS-CoV-2. Действительные и положительные/отрицательные результаты выборки были признаны окончательными. Образцы, которые не дали окончательных результатов в первом запуске, были классифицированы как Повторный запуск (обозначаемый RR) или N1 Повторный запуск (обозначенный N1 RR). Повторные образцы не имели действительного усиления P1, а образцы повторного запуска N1 имели положительную амплификацию N1 в одной реплике. Если в результате последующего ручного запуска не было получено действительное усиление P1 или оба реплики имели значения N1 Ct выше положительного порога (Ct >33), результаты выборки считались неубедительными. В клинических целях образцы пациентов, которые не прибыли в лабораторию, имели недостаточное количество слюны для пипетки или были повреждены, считались недействительными. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
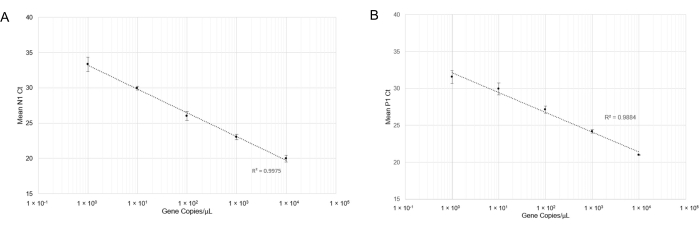
Рисунок 4: Обнаружение RT-qPCR синтетической РНК N1 (SARS-CoV-2) и синтетической ДНК P1 (Hs_RPP 30). Стандартные кривые были построены со стандартными отклонениями для определения диапазона точного обнаружения с использованием этой комбинации зонд/грунтовка. (A) Средние значения Ct (n =4), полученные в соответствующих разведениях, были построены по отношению к расчетному количеству синтетической РНК (от 1x100 до 1x104 копий РНК в 10 мкл реакции RT-qPCR). (B) Средние значения Ct (n = 3), полученные в соответствующих разведениях, были построены по отношению к расчетному количеству синтетической ДНК (от 1 х 100 до 1 х 104 копий гена в 10 мкл реакции ОТ-qPCR). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
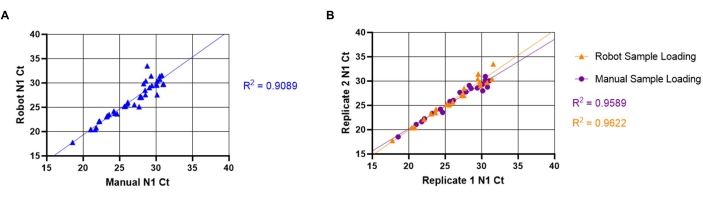
Рисунок 5: Сравнение ручных и автоматических значений ПЕРЕНОСА слюны SARS-CoV-2 (N1) Ct. Известные образцы положительной слюны SARS-CoV-2 (n = 20) были загружены в дубликаты в основную микшерную пластину RT-qPCR роботом по обработке жидкости. Образцы имеют значение Ct в диапазоне от 18 до 32 для N1. Затем одни и те же образцы вручную загружались в дублирующиеся скважины в другом месте пластины. (A) Значения N1 Ct, полученные из уникальных образцов с использованием как роботизированной, так и ручной загрузки образцов, были перенесены для определения межпробирной изменчивости между ручной и роботизированной загрузкой. (B) Вариабельность внутри анализа также определялась с помощью транспонированной реплики значений N1 Ct, полученных как при загрузке образцов роботом, так и вручную. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
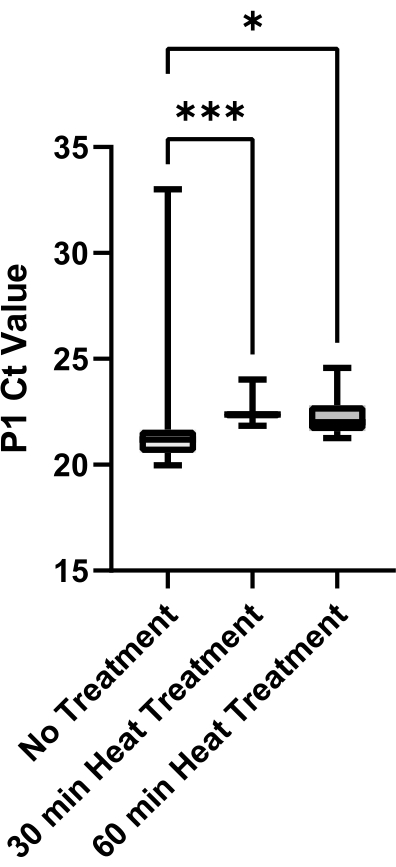
Рисунок 6: Оценка методов термообработки для снижения вязкости слюны. Отрицательную слюну SARS-CoV-2 собирали из одного источника, а аликвоты подвергали термической обработке в течение 0 мин, 30 мин или 60 мин при 95 °C. Значения P1 Ct из технических реплик (n = 12) каждого условия были построены для определения изменчивости между методами лечения. Попарные сравнения между группами оценивались с помощью непарного t-теста (*** указывает на p <0,001, * указывает на p <0,05). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительный рисунок 1: Сравнение N1 Ct в образцах слюны с низким содержанием P1 Ct. Положительные образцы с низким P1 Ct были отобраны и сопоставлены с N1 Ct (n = 106). Значения N1 Ct варьировались от 14 до 33, что указывает на то, что анализ имеет динамический диапазон в образцах слюны, который сопоставим со стандартной кривой. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
| Компонент | Последовательность (5'→3') | Концентрация запасов | Том | ||
| Зонд 2019-nCoV-N1 | /5FAM/ACCCCGCAT/ZEN/TACGTTTGGTGGACC/3IABkFQ | 50 мкМ | 500 мкл | ||
| 2019-nCoV-N1-For | GACCCCAAAATCAGCGAAAT | 100 мкМ | 2000 мкл | ||
| 2019-nCoV-N1-Rev | TCTGGTTACTGCCAGTTGAATCTG | 100 мкМ | 2000 мкл | ||
| Hs RPP30 Cy5 Зонд | /5Cy5/TTCTGACCT/ZEN/GAAGGCTCTGCGCG/3IABkFQ | 50 мкМ | 500 мкл | ||
| Hs-RPP30-For | AGATTTGGACCTGCGAGCG | 100 мкМ | 2000 мкл | ||
| Hs-RPP30-Rev | GAGCGGCTGTCTCCACAAGT | 100 мкМ | 2000 мкл | ||
| Вода | - | - | 11000 мкл | ||
Таблица 1: Компоненты смеси зонда/грунтовки N1+P1.
| Компонент | Концентрация запасов | Объем на реакцию | Конечная концентрация | Объем партии | ||
| Луна ТеплыйСтарт RT Фермент Микс | в 20 раз | 0.5 мкл | в 1 раз | 3 мл | ||
| Буферная реакционная смесь Luna | в 2 раза | 5.0 мкл | в 1 раз | 30 мл | ||
| N1 + P1 Грунтовка / Зонд Смесь | нКоВ N1 F: 10 мкМ | 0.5 мкл | 500 нМ | 3 мл | ||
| нКоВ N1 R: 10 мкМ | 500 нМ | |||||
| Зонд nCoV N1: 2,5 мкМ | 125 нМ | |||||
| RPP_30 P1 F: 10 мкМ | 500 нМ | |||||
| RPP_30 P1 R: 10 мкМ | 500 нМ | |||||
| RPP_30 зонда P1: 2,5 мкМ | 125 нМ | |||||
| Вода без нуклеаз | --- | 2 мкл | --- | 12 мл | ||
| Промежуточная сумма | --- | 8 мкл | --- | 48 мл | ||
| Шаблон | 2 мкл | |||||
Таблица 2: Компоненты мультиплексного мастер-микса SARS-CoV-2.
| Сцена | Температура (°C) | Длительность | Количество циклов |
| Обратная транскрипция | 55 | 10 мин | 1 |
| Начальная денатурация | 95 | 1 мин | 1 |
| Приземление | 95 | 10 сек | 3 |
| 72 | 30 сек | ||
| 95 | 10 сек | 3 | |
| 69 | 30 сек | ||
| 95 | 10 сек | 3 | |
| 66 | 30 сек | ||
| Основное усиление | 95 | 10 сек | 40 |
| 65 | 30 сек |
Таблица 3: Протокол ПРИЗЕМЛЕНИЯ RT-qPCR. Условия термоциклирования для одноэтапного диагностического анализа RT-qPCR SARS-CoV-2.
| Шаг приземления | Без шага приземления | |||
| Среднее N1 Ct | Среднее значение P1 Ct | Среднее N1 Ct | Среднее значение P1 Ct | |
| Пример 1 | 19.65 | 22.7 | 27.8 | 28.3 |
| Пример 2 | 22.24 | 24.9 | 28.77 | 30.5 |
| Пример 3 | 18.85 | 19.2 | 24.65 | 25.9 |
| Пример 4 | 25.56 | 22.8 | 31.93 | 29.2 |
| Пример 5 | 22.34 | 24.8 | 38.48 | 40.0 (Неудачное обнаружение) |
Таблица 4: Сравнение значений Ct приземления для пяти положительных образцов с значениями Ct без приземления.
| Образец | ТайгерСалива | Коммерчески доступный анализ SARS-CoV-2 на основе слюны | ||
| Н1 Кт | П1 Кт | Значение Covid-19 | Значение RNaseP | |
| Д11 | 16.4 | 18.1 | 20.86 | 23.4 |
| Е11 | 18.9 | 19.1 | 25.6 | 21.2 |
| Ф11 | 19.5 | 18.4 | 22.8 | 22.2 |
| Г11 | 22.2 | 19.1 | 23.7 | 22.9 |
| Н11 | 26.4 | 21.3 | 32.2 | 26.7 |
| А12 | 14.8 | 16.5 | 29.15 | 19 |
| В12 | 24 | 19.6 | 31.05 | 21.35 |
| С12 | 14.9 | 17.5 | 20.84 | 18.9 |
Таблица 5: Сравнение результатов TigerSaliva Ct и коммерчески доступных результатов анализа SARS-CoV-2 на основе слюны. Оба анализа проводились на одних и тех же образцах слюны (n = 8).
Дополнительный файл 1: Пользовательский скрипт для создания мастер-микса робота. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 2: Пользовательский скрипт для обработки слюны на роботах для загрузки образцов. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 3: Инструкции по самостоятельному сбору высококачественных образцов слюны у участников. Более подробную информацию можно найти в кратком видео-описании процесса тестирования, доступном на https://www.clemson.edu/centers-institutes/reddilab/index.html. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 4: Образец электронной таблицы приема. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 5: Пример загрузки электронной таблицы. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 6: Образец схемы компоновки плит 384 скважины. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Анализ, описанный в протоколе, был оценен независимым валидационным исследованием. Было обнаружено, что анализ имел 98,9% специфичности (1,1% ложноположительных) и 90,0% чувствительности (10,0% ложноотрицательных) при оценке по парным мазкам из носоглотки, взятым в то же время (n = 837; 817 отрицательных, 20 положительных). Важно отметить, что три участника, которые дали положительный результат с TigerSaliva и отрицательный с мазком из носоглотки, были повторно протестированы с мазками через 48 часов и вернули положительные результаты, указывающие на то, что TigerSaliva может быть в состоянии обнаружить инфекции SARS-CoV-2 ранее в ходе болезни.
Мы смоделировали положительные образцы слюны путем всплеска невирусной слюны (как термически обработанной, так и нетермически обработанной) с известными концентрациями синтетической РНК SARS-CoV-2 и выполнили 10-кратное разведение для определения предела Ct в слюне. Ген N1 не был обнаружен ниже 10 000 копий генов (приблизительно Ct = 28) в смоделированных положительных образцах. Мы подозреваем, что это связано с деградацией РНКазы или другими мешающими факторами. Однако взаимодействие РНаз слюны с голой синтетической РНК, вероятно, отличается от взаимодействия с вирусными частицами, даже после того, как они были денатурированы теплом. Положительные образцы слюны были идентифицированы с помощью Ct >30, и внешние лаборатории получили данные генетической последовательности SARS-CoV-2 из этих образцов. Мы предполагаем, что вирусные белки обеспечивают защиту от деградации РНК в образцах слюны пациентов.
Наиболее важным этапом в протоколе является внедрение автоматизации подготовки мастер-микса и обработки проб слюны (разделы 4 и 7 соответственно). Это позволяет перекрывать процессы задач, что значительно сокращает время выполнения работ. Другим важным этапом является интерпретация клинических результатов (разделы 11 и 12). Установление промежуточных категорий результатов (повторный запуск и повторный запуск N1) также сводило к минимуму возникновение неубедительных результатов испытаний.
Мы продемонстрировали, что различия между ручными и автоматизированными методами загрузки образцов слюны незначительны (рисунок 5A) и что автоматизация может улучшить воспроизводимость обнаружения SARS-CoV-2 (рисунок 5B). Следует отдавать предпочтение автоматизации для облегчения тестирования при проектировании и расширении клинических лабораторий25. Улучшается лабораторный документооборот с реализацией роботизированных автоматизированных задач26. Возможности роботов с открытым исходным кодом для обработки жидкостей позволяют реализовать пользовательские сценарии для разработки протоколов. Это делает роботов для обработки жидкостей недорогой и легко модифицируемой системой по сравнению с традиционными методами клинической автоматизации. Это также идеальная стратегия для выполнения повторяющихся лабораторных задач. Высокий уровень настраиваемости системы приводит к свободе изменять лабораторную посуду (например, коллекторные трубки, наконечники пипеток или пластины на 384 скважины) в случае нехватки. Таким образом, автоматизация с использованием роботов для обработки жидкостей жизнеспособна как для крупномасштабного, так и для мелкомасштабного наблюдения и исследований.
Основным преимуществом этой стратегии тестирования является гораздо более короткое время обработки по сравнению с другими клиническими лабораториями. Использование автоматизированных роботов для обработки жидкостей играет ключевую роль в сокращении времени обработки, но одновременное использование роботов и термоциклеров также играет важную роль в максимизации эффективности тестирования. Один робот и термоциклер должны работать как пара, где обе машины используются в тандеме для непрерывной загрузки образцов и анализа результатов проб. Как только постоянный поток назначенных образцов установлен, все пары машин могут работать одновременно. Постоянное одновременное использование роботов и термоциклеров резко увеличивает производительность и эффективность тестирования, что имеет решающее значение для размещения большого объема испытаний.
В отличие от других установленных протоколов SARS-CoV-2 RT-qPCR, мы включили шаг приземления в протокол термоциклера для улучшения отжига набора зонда и праймера к генам-мишеням27, снижая риск неудачной амплификации. Результаты показали, что приземление улучшило обнаружение положительных образцов без риска потери специфического связывания грунтовки (таблица 4). Мы определили, что широкий спектр как копий РНК SARS-CoV-2 (рисунок 4A), так и Hs_RPP30 копий ДНК (рисунок 4B) может быть одновременно обнаружен с помощью анализа RT-qPCR.
Одним из ограничений роботов для обработки жидкостей является возможность перекрестного загрязнения от положительных образцов во время переноса слюны. Слюна представляет собой вязкоупругую жидкость28 и может нанизываться на соседние колодцы после дозирования из кончика пипетки. Кроме того, неоднородность слюны29 может привести к неравномерному распределению вирусных частиц по всему образцу. Это увеличивает вероятность как ложноположительных, так и отрицательных результатов, что требует обозначения образцов N1 Rerun и Rerun. Тем не менее, 14,1% образцов, первоначально обозначенных как N1 Rerun, разрешились как положительные для SARS-CoV-2 и более чем в 30 раз чаще, чем повторные образцы, разрешались как положительные после повторного тестирования. Следовательно, дифференциация Rerun от N1 Rerun (рисунок 3) позволила более точно разделить потенциально положительные образцы, повысив чувствительность и специфичность нашего диагностического анализа. Другие результирующие параметры для диагностического тестирования слюны не сделали этого различия12,14,24,30,31.
Образцы слюны может быть трудно пипеткой из-за неоднородности и вязкости32. Термическая обработка адекватно денатурирует белки в биоматрице слюны, снижая вязкость и устраняя необходимость в реагентах для экстракции РНК9, которых не хватало на ранних стадиях пандемии10. Расширенная термическая обработка также инактивирует присутствующие вирусы33, что позволяет проводить лабораторную обработку при более низких уровнях биобезопасности. Следовательно, экстракция РНК на тепловой основе (описанная в разделе 5.4) была реализована для снижения вязкости за счет денатурации белка (рисунок 6). Основываясь на результатах, мы постулируем, что термическая обработка может также гомогенизировать образцы слюны в дополнение к денатурации белковой биоматрицы. Другие группы комбинировали термическую обработку и обработку протеиназой К для повышения однородности9,14,34. Мы решили не реализовывать этот шаг, поскольку он может денатурировать белки вириона со скоростью, которая оставляет вирусную РНК подверженной тепловой деградации35. Кроме того, разбавление проб протеиназой К может маскировать положительные образцы, содержащие меньше вирусных частиц, тем самым снижая чувствительность. Кроме того, результаты анализа сравнивались с коммерчески доступным анализом SARS-CoV-2 на основе слюны (Logix Smart COVID-19), в котором используется магнитная экстракция РНК (таблица 5). Было обнаружено, что текущий анализ лучше подходит для обнаружения слабых положительных образцов по сравнению с коммерчески доступным анализом.
Трудно количественно оценить количество копий вируса в слюне, используя только RT-qPCR, потому что qPCR является полуколичественным. Существуют внутренние различия между значениями Ct, которые проистекают из технических ограничений. Номер копии гена может быть определен по значениям Ct (рисунок 4) и примерно эквивалентен номеру вирусной копии. Одним из возможных решений для определения числа вирусных копий в образцах слюны является ddPCR, который обеспечивает жесткую количественную оценку копий генов в реакции. Тем не менее, мы считаем, что достаточно предоставить качественные результаты клиницистам, и относительное содержание вируса может быть сравнено с образцами, обработанными нашими методами.
Несмотря на некоторые ограничения, возникающие при использовании слюны, анализ SARS-CoV-2 методом RT-qPCR на основе слюны оказывается эффективным методом быстрого и надежного обнаружения вирусной РНК в любом масштабе тестирования. Это особенно верно в сочетании с использованием систем обработки жидкостей с открытым исходным кодом. Этот подход к тестированию может быть модифицирован для обнаружения других последовательностей нуклеиновых кислот, имеющих отношение к диагностике, таких как агенты инфекционных заболеваний, маркеры заболеваний или другие вирусы. Это делает анализ применимым как для клинических, так и для исследовательских диагностических усилий.
Раскрытие информации
Авторам нечего раскрывать. Анализ, описанный в протоколе, подпадает под действие SalivaDirect EUA, поданного Йельской школой общественного здравоохранения.
Благодарности
Авторы благодарят администрацию Клемсона, медицинский персонал и сотрудников клинической лаборатории в лаборатории REDDI, которые помогли внедрить и управлять тестированием SARS-CoV-2. Мы благодарим д-ра Филиппа Бакхолтса и д-ра Кэролин Баннистер из Университета Южной Каролины за первоначальный консалтинг по проектам и отраслевые контакты по закупкам оборудования. Мы благодарим многих студентов, преподавателей и сотрудников за помощь в сборе образцов. Спасибо студентам Creative Inquiry за стандартный сбор данных кривых. Финансирование этого исследования было получено от гранта Национального института здравоохранения P20GM121342 (присуждается DD и LGP), Спортивного департамента Клемсона, вице-президента по исследованиям Университета Клемсона и Комитета губернатора Южной Каролины и Объединенного комитета по рассмотрению облигаций.
Материалы
| Name | Company | Catalog Number | Comments |
| 100% EtOH | Fisher scientific | 22-032-601 | |
| 20 uL Filtered Pipette Tips | Opentrons | 20uL tips | |
| 2mL Microcentrifuge Tubes | Fisher Scientific | 14-666-313 | Alternate product may be used |
| Armadillo PCR Plate, 384-well, clear, white wells | Thermo Scientific | AB3384 | Alternate product may be used |
| Celltreat 2mL 96 Deep Well Plates | Fisher Scientific | 50-828-743 | For mastermix preparation |
| Clear PCR Sealing Sheets | Thermo Scientific | AB0558 | Alternate product may be used |
| DPEC Treated Water | Ambion (Thermo Scientific) | AM9916 | |
| Flip Cap 50 mL Conical Tubes | VWR | 75845-210 | For sample collection |
| Foil PCR Sealing Sheets | Thermo Scientific | AB0626 | For storage of mastermix plates, Alternate product may be used |
| HS_RPP30 Synthetic DNA | Integrated DNA Technologies | 299788131 | P1 positive control |
| Luna Buffer Probe One-Step Reaction | New England Biolabs | M3006B | |
| Luna WarmStart RT Enzyme Mix | New England Biolabs | M3002B | |
| nCOV_N1 Forward Primer, 100 nmol | Integrated DNA Technologies | 10006830 | |
| nCOV_N1 Probe Aliquot, 50 nmol | Integrated DNA Technologies | 10006832 | Probe can be synthesized by other vendors with SYBR or FAM fluophores |
| nCOV_N1 Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006831 | |
| Opentron HEPA Filter Module | Opentrons | N/A | Not required, but useful to reduce contamination |
| Opentron Multichannel Attachment, P20 | Opentrons | 999-00005 | For mastermix preparation |
| Opentron OT-2 Liquid Handling Robot | Opentrons | OT-2 | |
| Opentron Pipette Attachment, P20 | Opentrons | 999-0000215 | For sample loading |
| Oven | Memmert | UF450 PLUS 208V-3PH | |
| PCR Tubes (rnase, dnase free) | Fisher Scientific | 14-230-225 | For aliquots of positive and neg controls |
| PolarSafe Aluminum Cooling Block, 15-Well (1.5/2.0 mL Tubes) | VWR | 10808-952 | |
| PolarSaf Aluminum Cooling Block, 24-Well (0.5mL tubes) | VWR | 10808-956 | |
| RNAse P (ATTO 647) Probe, 50 nmol | Integrated DNA Technologies | 10007062 | Probe can be synthesized by other vendors with Cy5 fluorophore |
| RNAse P Forward Primer, 100nmol | Integrated DNA Technologies | 10006836 | |
| RNAse P Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006837 | |
| Sars-CoV-2 Synthetic RNA Control 2 | Twist Biosciences | 102024 / 103907 / 103909 | N1 Positive Control |
| Scanners | Code | CR1500 | Only required when scaling up |
| Small HEPA Filtered Hood | Erlab | Captair Bio 321 | For mastermix preparation |
| Thermocycler CFX384 Touch | Biorad | CFX384 Touch | Alternate models can be used, e.g. CFX384 Opus |
| X-acto Knife Set | Staples | N/A | To cut foil for keeping control wells covered |
Ссылки
- Zhu, N., et al. A novel coronavirus from patients with pneumonia in China, 2019. New England Journal Medicine. 382 (8), 727-733 (2020).
- Chen, J. Pathogenicity and transmissibility of 2019-nCoV-A quick overview and comparison with other emerging viruses. Microbes and Infection. 22 (2), 69-71 (2020).
- Petersen, E., et al. Comparing SARS-CoV-2 with SARS-CoV and influenza pandemics. The Lancet. Infectious Diseases. 20 (9), 238-244 (2020).
- Honein, M. A., et al. Summary of Guidance for Public Health Strategies to Address High Levels of Community Transmission of SARS-CoV-2 and Related Deaths, December 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (49), 1860-1867 (2020).
- World Health Organization. Surveillance strategies for COVID-19 human infection: interim guidance. World Health Organization. , Available from: https://apps.who.int/iris/handle/10665/332051 (2020).
- Screening testing for early detection of SARS-CoV-2 infection. Division of viral diseases. Centers for Disease Control. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/hcp/tsting-overview.html#PublicHealthSurveillance (2020).
- Larremore, D. B., et al. Test sensitivity is secondary to frequency and turnaround time for COVID-19 screening. Science Advances. 7 (1), 5393(2021).
- Sethuraman, N., Jeremiah, S. S., Ryo, A. Interpreting Diagnostic Tests for SARS-CoV-2. JAMA. 323 (22), 2249-2251 (2020).
- Chu, A. W., et al. Evaluation of simple nucleic acid extraction methods for the detection of SARS-CoV-2 in nasopharyngeal and saliva specimens during global shortage of extraction kits. Journal of clinical virology: the official publication of the Pan American Society for Clinical Virology. 129, 104519(2020).
- Guan, D., et al. Global supply-chain effects of COVID-19 control measures. Nature Human Behaviour. 4 (6), 577-587 (2020).
- Bastos, M. L., Perlman-Arrow, S., Menzies, D., Campbell, J. R. The Sensitivity and Costs of Testing for SARS-CoV-2 Infection With Saliva Versus Nasopharyngeal Swabs: A Systematic Review and Meta-analysis. Annals of Internal Medicine. 174 (4), 501-510 (2021).
- Pasomsub, E., et al. Saliva sample as a non-invasive specimen for the diagnosis of coronavirus disease 2019: a cross-sectional study. Clinical Microbiology and Infection: The Official Publication of The European Society of Clinical Microbiology and Infectious Diseases. 27 (2), (2021).
- To, K. K., et al. Consistent Detection of 2019 Novel Coronavirus in Saliva. Clinical Infectious Diseases: An Official Publication of The Infectious Diseases Society of America. 71 (15), 841-843 (2020).
- Vogels, C. B., et al. SalivaDirect: A simplified and flexible platform to enhance SARS-CoV-2 testing capacity. Med (New York, N.Y.). 2 (3), 263-280 (2021).
- Griesemer, S. B., et al. Evaluation of Specimen Types and Saliva Stabilization Solutions for SARS-CoV-2 Testing. Journal of Clinical Microbiology. 59 (5), 1418-1420 (2021).
- Wehrhahn, M. C., et al. Self-collection: An appropriate alternative during the SARS-CoV-2 pandemic. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 128, 104417(2020).
- Barza, R., Patel, P., Sabatini, L., Singh, K. Use of a simplified sample processing step without RNA extraction for direct SARS-CoV-2 RT-PCR detection. Journal of Clinical Virology: The Official Publication of the Pan American Society for Clinical Virology. 132, 104587(2020).
- Paltiel, A. D., Zheng, A., Walensky, R. P. Assessment of SARS-CoV-2 Screening Strategies to Permit the Safe Reopening of College Campuses in the United States. JAMA Network Open. 3 (7), 2016818(2020).
- Centers for Disease Control. 2019-Novel Coronavirus (2019-nCoV) Real-Time RT-PCR Primers and Probes. U.S. Department of Health and Human Services. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/lab/rt-pcr-panel-primer-probes.html (2020).
- Cresswell, K., Ramalingam, S., Sheikh, A. Can Robots Improve Testing Capacity for SARS-CoV-2. Journal of Medical Internet Research. 22 (8), 20169(2020).
- Villanueva-Cañas, J. L., et al. Implementation of an open-source robotic platform for SARS-CoV-2 testing by real-time RT-PCR. PLoS One. 16 (7), 0252509(2021).
- Robot Boosts COVID-19 Testing Efficiency. University of South Carolina College of Pharmacy. , Available from: https://sc.edu/study/colleges_schools/pharmacy/about/news/2020/robot-boosts-testing-effort.php (2020).
- Matic, N., et al. Practical challenges to the clinical implementation of saliva for SARS-CoV-2 detection. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of The European Society of Clinical Microbiology. 40 (2), 447-450 (2021).
- Sahajpal, N. S., et al. SalivaSTAT: Direct-PCR and Pooling of Saliva Samples Collected in Healthcare and Community Setting for SARS-CoV-2 Mass Surveillance. Diagnostics. 11 (5), Basel, Switzerland. 904(2021).
- Genzen, J. R., et al. Challenges and Opportunities in Implementing Total Laboratory Automation. Clinical Chemistry. 64 (2), 259-264 (2018).
- Archetti, C., Montanelli, A., Finazzi, D., Caimi, L., Garrafa, E. Clinical Laboratory Automation: A Case Study. Journal of Public Health Research. 6 (1), 881(2017).
- Hecker, K. H., Roux, K. H. High and low annealing temperatures increase both specificity and yield in touchdown and stepdown PCR. BioTechniques. 20 (3), 478-485 (1996).
- Bhat, P. P., et al. Formation of beads-on-a-string structures during break-up of viscoelastic filaments. Nature Physics. 6, 625-631 (2010).
- Miller, C. S., et al. Current developments in salivary diagnostics. Biomarkers in Medicine. 4 (1), 171-189 (2010).
- Moreno-Contreras, J., et al. Saliva Sampling and Its Direct Lysis, an Excellent Option To Increase the Number of SARS-CoV-2 Diagnostic Tests in Settings with Supply Shortages. Journal of Clinical Microbiology. 58 (10), 01659(2020).
- Wang, W., et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA. 323 (18), 1843-1844 (2020).
- Landry, M. L., Criscuolo, J., Peaper, D. R. Challenges in use of saliva for detection of SARS CoV-2 RNA in symptomatic outpatients. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 130, 104567(2020).
- Lista, M. J., et al. Resilient SARS-CoV-2 diagnostics workflows including viral heat inactivation. PLoS One. 16 (9), 0256813(2021).
- Brotons, P., et al. Validation and implementation of a direct RT-qPCR method for rapid screening of SARS-CoV-2 infection by using non-invasive saliva samples. International Journal of Infectious Diseases: IJID: Official Publication of The International Society for Infectious Diseases. 110, 363-370 (2021).
- Batéjat, C., Grassin, Q., Manuguerra, J. C., Leclercr, I. Heat inactivation of the severe acute respiratory syndrome coronavirus 2. Journal of Biosafety and Biosecurity. 3 (1), 1-3 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены