Method Article
Stratégie de diagnostic de la salive par PCR à transcriptase inverse quantitative efficace du SRAS-CoV-2 utilisant des robots de pipetage open source
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le protocole décrit une méthode de diagnostic du SRAS-CoV-2 qui utilise l’automatisation open source pour effectuer des tests moléculaires RT-qPCR sur des échantillons de salive. Cette approche évolutive peut être appliquée à la surveillance clinique de la santé publique ainsi qu’à l’augmentation de la capacité des petits laboratoires universitaires.
Résumé
L’émergence de la récente crise sanitaire mondiale du SRAS-CoV-2 a posé des défis majeurs pour la recherche épidémiologique et les essais cliniques. Caractérisée par un taux élevé de transmission et une faible mortalité, la pandémie de COVID-19 a nécessité des tests diagnostiques précis et efficaces, en particulier dans les populations fermées telles que les universités résidentielles. La disponibilité initiale des tests d’acides nucléiques, comme les écouvillons nasopharyngés, était limitée en raison de la pression de la chaîne d’approvisionnement qui a également retardé la communication des résultats des tests. Les tests de réaction en chaîne quantitative par polymérase inverse (RT-qPCR) à base de salivaire se sont révélés comparables en sensibilité et en spécificité à d’autres méthodes de test, et la collecte de la salive est moins invasive physiquement pour les participants. Par conséquent, nous avons développé un test de diagnostic multiplex RT-qPCR pour la surveillance de la population de l’Université Clemson et de la communauté environnante. Le test a utilisé des robots de manipulation de liquides et des thermocycleurs open source au lieu de systèmes d’automatisation clinique complexes pour optimiser le flux de travail et la flexibilité du système. L’automatisation de la RT-qPCR à base de salive permet une détection rapide et précise d’un large éventail de concentrations d’ARN viral pour les demandes de tests à grande et à petite échelle. Le délai d’exécution moyen du système automatisé était de < 9 h pour 95 % des échantillons et < 24 h pour 99 % des échantillons. Le coût d’un seul test était de 2,80 $ lorsque tous les réactifs étaient achetés en vrac.
Introduction
Le coronavirus 2 associé au syndrome respiratoire aigu sévère (SARS-CoV-2), un nouveau coronavirus, est apparu à la fin de 2019 et s’est rapidement propagé dans les populations mondiales1. L’infection par le SRAS-CoV-2 provoque la maladie à coronavirus 2019 (COVID-19), une maladie très contagieuse avec des symptômes respiratoires et inflammatoires potentiellement graves. Une transmissibilité élevée associée à une faible mortalité indiquait que le virus se propagerait rapidement dans les populations et nécessiterait des tests diagnostiques accrus2,3. Les recommandations de santé publique encourageaient le dépistage à grande échelle de la population afin d’isoler les cas et de réduire par la suite les taux de transmission4,5,6. De plus, les modèles de surveillance de la population ont révélé que l’augmentation de la fréquence des tests et la diminution du temps de déclaration avaient un effet plus important sur la réduction de la transmission que sur l’augmentation de la sensibilité des tests7. Cela est probablement dû au fait que les personnes infectées pourraient être mises en quarantaine plus tôt, brisant ainsi les chaînes d’infection.
La norme originale de test d’amplification des acides nucléiques (NAAT) était des écouvillons nasopharyngés (NP) traités par RT-qPCR8. Cependant, cette forme de test entraîne des complications pour de très grandes populations, telles que l’augmentation du coût relatif et l’exacerbation de la pression sur la chaîne d’approvisionnement9,10. De plus, la collecte d’échantillons et le traitement des méthodes courantes de LAA (y compris les écouvillons NP, les écouvillons oropharyngés, les écouvillons à cornets moyens et les écouvillons nasaux) dépendent de l’équipement spécialisé, des réactifs et du personnel médical9,10.
Un substitut adéquat au test RT-qPCR par écouvillonnage NP est le test à base de salive, qui est un outil de diagnostic précis pour la détection du SARS-CoV-211,12,13,14. L’exécution directe de la RT-qPCR sur des échantillons de salive donne une sensibilité et une spécificité similaires à celles des écouvillons NP15. L’un des principaux avantages des tests salivaires par rapport aux tests par écouvillonnage NP est qu’ils permettent l’auto-prélèvement d’échantillons16. Cela minimise le besoin de personnel médical et maximise la facilité de prélèvement d’échantillons pour les patients en étant moins invasif que les écouvillons NP. De plus, étant donné que les échantillons de salive ne nécessitent pas de tampons pour retirer l’échantillon d’un écouvillon (comme dans le cas des échantillons de NP), les tests à base de salive peuvent utiliser directement l’extraction de l’acide ribonucléique (ARN) à base de chaleur, ce qui réduit les coûts de test en éliminant le besoin de tampons supplémentaires, de milieux de transport et / ou de réactifs d’extraction d’ARN14,17.
Le laboratoire de recherche et d’éducation en diagnostic et intervention des maladies (REDDI) de l’Université Clemson a été créé pour répondre aux besoins de l’université en matière de dépistage et de surveillance de la COVID-19. Dans les populations fermées, y compris les universités, les tests de surveillance fréquents associés à la distanciation sociale ont produit les résultats les plus favorables dans les modèles épidémiologiques de prévalence de la maladie18. Les protocoles consolidés CDC 2019-nCOV RT-qPCR19 et SalivaDirect14 ont été adaptés et l’automatisation a été utilisée dans le flux de travail clinique pour réduire les coûts et améliorer les délais d’exécution. Les groupes précédents avaient utilisé des robots de manipulation de liquides open source pour les étapes d’extraction de l’ARN SARS-CoV-220,21, mais nous avons maximisé l’utilisation des robots pour préparer des plaques d’essai et charger des échantillons22. Ici, nous montrons que le protocole adapté et l’utilisation de systèmes de manipulation de liquides open source (Figure 1) permettent une RT-qPCR rapide et précise à base de salive et constituent une stratégie efficace pour la surveillance de la santé publique à grande échelle.
Protocole
Toutes les recherches ont été effectuées conformément aux conseils d’examen institutionnel de l’Université Clemson et de Prisma Health (Prisma Health IRB # Pro00099491, 1er juillet 2020).
1. Configuration d’un robot de manipulation de liquides open source
- Installez des modules de filtre à air particulaire à haut rendement (HEPA) (voir tableau des matériaux) sur le dessus de chaque robot de manipulation de liquides conformément aux instructions du fabricant.
- Fixez une pipette P20 à 8 canaux sur le support gauche du ou des robots de préparation de la plaque de mélange principale conformément aux instructions du fabricant.
- Fixez une pipette P20 sur le support droit du ou des robots de chargement d’échantillons conformément aux instructions du fabricant.
- Téléchargez les scripts Python personnalisés (fichier supplémentaire 1 et fichier supplémentaire 2) sur les ordinateurs appropriés.
- Ouvrez TigerSaliva Full 384 Loading.py dans l’application de bureau sur l’exemple d’ordinateurs robots de chargement. Cliquez sur Calibrer et configurez la pipette et le programme conformément aux instructions du logiciel.
- Ouvrez 12 Full Plates.py dans l’application de bureau sur l’ordinateur du robot master mix. Cliquez sur Calibrer et configurez la pipette et le programme conformément aux instructions du logiciel.
- Imprimez des racks d’échantillons personnalisés avec une imprimante tridimensionnelle (3D) de modélisation de dépôt fondu à l’aide d’un fichier de conception assistée par ordinateur (CAO) (https://www.myminifactory.com/object/3d-print-141363). Imprimez 16 racks par robot de chargement d’échantillons, pour deux ensembles de huit racks.
2. Préparation d’un mélange sonde/apprêt multiplex 20x N1+P1
- Préparer un lot de 20x multiplex mélange sonde/amorce N1+P1 (volume total de 20 mL, tableau 1) dans un tube conique de 50 mL dans un environnement stérile à l’écart de l’ARN synthétique du SRAS-CoV-2 ou des échantillons de patients.
- Aliquoter 1,6 mL du mélange à l’aide d’une pipette sérologique dans des tubes centrifuges stériles de 2,0 mL et étiqueter de manière appropriée.
- Conserver les aliquotes dans un congélateur à -20 °C jusqu’à ce qu’elles soient prêtes à l’emploi.
3. Préparation du mélange témoin positif
REMARQUE : Le mélange témoin positif ne doit pas être fabriqué dans le même environnement stérile que le mélange sonde/apprêt ou d’autres composants du mélange maître. Un récipient séparé d’eau sans nucléase doit être utilisé.
- Diluer l’ARN synthétique (N1) du SARS-CoV-2 de 1 000 000 de copies de gènes/μL (cpμ) à 10 000 cpμ en ajoutant 10 μL de solution mère à 990 μL d’eau sans nucléase. Aliquote 25 μL de 10 000 cpμ dans des tubes stériles de 0,2 mL et étiqueter de manière appropriée. Conserver les aliquotes inutilisées à -80°C.
REMARQUE: L’ARN synthétique doit être stocké à -80 ° C pour éviter la dégradation. - Diluer Hs_RPP30'ADN synthétique (P1) de 200 000 cpμ à 10 000 cpμ en ajoutant 50 μL de solution mère à 950 μL d’eau sans nucléase. Aliquote 25 μL de 10 000 cpμ dans des tubes stériles de 0,2 mL et étiqueter de manière appropriée. Conserver les aliquotes inutilisées à -80°C.
- Diluer chaque composant à une concentration finale de 200 cpμ en ajoutant 20 μL de SARS-CoV-2 et de Hs_RPP30 10 000 cpμ stocks à 960 μL d’eau sans nucléase, pour un total de 1000 μL. Aliquote 20 μL de témoin positif mélangé de 200 cpμ dans des tubes de 0,2 mL et étiqueter de manière appropriée. Conserver les aliquotes à -80°C jusqu’à ce qu’elles soient prêtes à l’emploi.
4. Préparation des plaques de mélange maître
REMARQUE: Faites le mélange principal dans un environnement stérile loin de l’ARN synthétique du SRAS-CoV-2 ou des échantillons de patients. Tous les composants doivent être complètement décongelés avant d’être ajoutés au mélange; sans décongélation appropriée, les concentrations peuvent être incorrectes. Un dégel inadéquat est indiqué par la présence de glace ou la couleur inégale des réactifs. Conserver sur un bloc congélateur pendant la préparation du mélange.
- Préparer un lot de mélange maître multiplex (volume total de 48 mL, tableau 2) dans un tube conique de 50 mL.
- Homogénéiser le mélange en tournant le tube sur 3x. Ne mélangez pas en pipetant de haut en bas ou en vortexant car cela endommagerait l’enzyme.
- Remplir les colonnes 1 à 4 d’un réservoir de puits stérile de 96 puits en transférant 1,48 mL de mélange principal dans chaque puits. Un conique de 50 mL remplit quatre colonnes, assez pour 12 plaques.
- Couvrez le réservoir du puits profond avec un joint en aluminium et placez-le dans le robot de manipulation de liquide de mélange maître dédié. Placez le réservoir de puits profond rempli sur le pont 10, placez six plaques vides de 384 puits sur les ponts 1 à 6 et la boîte à embouts P20 sur le pont 11. Découvrez la plaque de puits profond et les boîtes à embouts et fermez le robot.
- Initialisez le protocole d’exploitation Python personnalisé en cliquant sur Démarrer l’exécution dans l’application de bureau du robot.
- Après 40 min, la course s’interrompt. Couvrez les 384 plaques de puits remplies avec des joints en aluminium pendant qu’elles restent dans le robot et appuyez avec le rouleau pour assurer l’adhérence. Étiquetez le bord de chaque plaque avec un identificateur de lot.
- Placez six nouvelles plaques vides de 384 puits sur les ponts 1 à 6 et reprenez le protocole d’exploitation en cliquant sur Reprendre l’exécution. Une fois la course terminée, couvrez le dernier ensemble de 384 plaques de puits avec des joints en aluminium.
- Pipetter 2 μL du mélange maître restant dans les colonnes 1-3 et 22-24 d’une plaque vide de 384 puits pour le contrôle de la qualité des lots. Sceller avec un joint optiquement transparent et courir sur le thermocycleur (section 10.1-10.2). Si des puits ont des valeurs de cycle seuil (Ct) N1 ou si plus de 10 puits ont des valeurs P1 Ct, le lot est contaminé et ne peut pas être utilisé.
- Conservez les plaques de mélange principal préparées à 4 °C et utilisez-les dans les 7 jours suivant la préparation.
5. Prélèvement d’échantillons, admission et traitement thermique
- Demandez aux participants d’éviter de manger, de boire, de fumer ou de faire de l’hygiène dentaire 30 minutes avant la collecte de salive. Demandez aux participants de prélever au moins 1 mL de salive qui s’accumule naturellement dans la bouche et de la déposer dans un tube conique stérile de 50 mL sans agent de conservation, puis de boucher le tube (dossier supplémentaire 3).
- Décontaminer l’extérieur des tubes de collecte de salive avec de l’éthanol à 70% ou des lingettes désinfectantes et les transférer au laboratoire pour analyse.
- Enregistrez l’arrivée de l’échantillon en scannant chaque code-barres de l’échantillon dans la feuille de calcul de l’apport quotidien (fichier supplémentaire 4).
- Traiter thermiquement les échantillons scannés pendant 30 min dans un four à 95 °C. Retirez les échantillons tout en portant des gants résistants à la chaleur.
REMARQUE: Les échantillons non traités sont stables à température ambiante (23 °C) jusqu’à 72 h. Une fois traités thermiquement, les échantillons doivent être conservés à 4 °C, s’ils ne sont pas traités immédiatement.
6. Affectation d’échantillons
- Ouvrez les feuilles de calcul de chargement d’échantillons quotidiens pour chaque robot de chargement d’échantillons (fichier supplémentaire 5) sur l’ordinateur de la station d’affectation d’échantillons.
- Attribuez 188 échantillons à chaque plaque de 384 puits comme suit : Les échantillons 1 à 48 sont considérés comme le trimestre 1, les échantillons 49 à 96 sont considérés comme le trimestre 2, les échantillons 97 à 144 sont considérés comme le trimestre 3 et les échantillons 145 à 188 sont considérés comme le trimestre 4. Les échantillons sont analysés en double.
- Bacs d’étiquettes avec le nom de la plaque, la date et le numéro de trimestre. Analysez les échantillons dans l’ordre dans la feuille de calcul de chargement des exemples.
REMARQUE : Les échantillons de plaques antérieures peuvent devoir être exécutés manuellement, comme défini à la figure 3. Reportez-vous aux sections 8.1 à 8.3 pour obtenir des instructions manuelles d’affectation et de chargement des échantillons.
7. Utilisation de robots de chargement d’échantillons
- À la station de chargement des échantillons, alignez deux ensembles complets de huit racks imprimés en 3D correspondant à l’emplacement du pont dans le robot.
- Décapsulez les tubes quart 1 et placez-les dans des racks imprimés en 3D, en commençant par la position A1 dans le rack 1. Remplissez chaque rack de gauche à droite et de haut en bas. Poursuivez ce schéma de chargement dans le rack 2, puis passez aux racks 4 et 5 (reportez-vous à la Figure 2C).
REMARQUE: Les racks ne sont pas numérotés consécutivement en raison des paramètres du programme du robot. - Placez les porte-échantillons quart 1 chargés sur les ponts 1, 2, 4, 5, 7, 8, 10, 11. Placez les embouts P20 sur les ponts 3 et 9. Pour simplifier le processus de configuration, chargez les matériaux de l’arrière vers l’avant dans le robot.
- Prenez une plaque de mélange maître préfabriquée à partir des 4 °C, étiquetez-la avec le nom de la plaque et utilisez une lame tranchante pour couper une ligne dans la feuille autour des puits de contrôle (N23/24, O23/24 et P23/24).
- Placez la plaque de mélange principale sur le pont 6 et décollez le couvercle en aluminium, en laissant derrière lui le petit rectangle recouvrant les puits de contrôle. Découvrez les boîtes à embouts et fermez le robot.
- Initialisez le protocole d’exploitation Python personnalisé en cliquant sur Démarrer exécuter via l’application de bureau du robot. Chaque trimestre prend 24,5 minutes à charger sur la plaque; réglez une minuterie comme rappel.
- Pendant que le robot fonctionne, décapez et chargez les tubes d’échantillonnage du quart 2 dans le deuxième ensemble de racks imprimés en 3D, comme décrit à la section 7.2.
- Lorsque le robot s’arrête, retirez les racks du quart 1 et remplacez-les par des racks du quart 2. Cliquez sur Reprendre l’exécution dans l’application de bureau.
- Récapitulez les tubes d’échantillon du quart 1 et conservez-les dans un réfrigérateur à 4 °C en attendant les résultats.
Répétez ce processus de chargement pour les trimestres 3 et 4. - Transférez la plaque chargée dans une armoire de biosécurité. Pour minimiser la contamination, gardez la plaque couverte pendant le transfert.
8. Chargement manuel des échantillons
REMARQUE: Effectuez une seule exécution manuelle sur des échantillons répétés (N1 Rerun ou Rerun, voir Figure 3) en cas de chargement robotique inadéquat.
- Recueillez tous les échantillons répétés et attribuez-les comme derniers échantillons au trimestre 4 (voir rubrique 6.2). Numérotez les échantillons, scannez les codes-barres, puis entrez l’emplacement et le résultat de l’échantillon d’origine dans la feuille de calcul de chargement de l’échantillon.
- Transférer les échantillons répétés dans l’armoire de biosécurité. Ne chargez pas les tubes d’échantillonnage répétés dans les racks de chargement du robot.
- Pipette 2 μL de chaque échantillon répété vers les puits corrects en suivant le schéma de disposition des plaques (fichier supplémentaire 6). Utilisez une pipette désignée pour ajouter des échantillons de patients. Gardez les puits de contrôle recouverts de papier d’aluminium tout en ajoutant des échantillons pour minimiser la contamination.
9. Ajout de commandes aux plaques d’essai
- Enlevez la couverture de papier d’aluminium sur les puits de contrôle à l’aide de pinces.
- Pipette 2 μL d’eau sans nucléase (pas de contrôle de gabarit) vers les puits N23-N24 et 2 μL de 200 cpμ de témoin positif mixte (voir rubrique 3) vers les puits O23-O24. Pipette 2 μL d’un témoin d’échantillon de patient positif confirmé dans les puits M23-M24 comme témoin supplémentaire. Laissez les puits P23-P24 vides pour surveiller la qualité des lots du mélange maître.
- Couvrez la plaque avec un joint optiquement transparent et utilisez le rouleau applicateur pour adhérer à tous les puits. Vortex la plaque à 2500 tr/min pendant 30 secondes pour bien mélanger. Centrifuger la plaque à 500 x g pendant 1 min.
10. Exécution de rt-qPCR
- Créez un programme de protocole dans le logiciel thermocycleur selon les conditions décrites (Tableau 3). Enregistrez le protocole pour les futures plaques. Placez la plaque scellée dans le thermocycleur et exécutez le protocole.
- Exportez les valeurs Ct sous forme de fichier .xslx et copiez les valeurs dans l’exemple de feuille de calcul de chargement (fichier supplémentaire 5).
REMARQUE: Ces feuilles ont été conçues sur mesure pour les fichiers de sortie Ct du logiciel du fabricant et peuvent nécessiter une modification pour accepter d’autres formats.
11. Détermination de la validité de la plaque
- Valider à la fois le témoin positif et/ou les échantillons positifs connus et le témoin négatif pour considérer les résultats de la plaque comme valides. Évaluez les puits témoins à l’aide des critères suivants.
- Pour le contrôle positif, vérifiez si au moins un puits témoin positif (O23/O24) produit des valeurs Ct comprises entre 22 et 28 pour les sondes P1 et N1. Alternativement, les puits d’échantillon positifs connus (M23/M24) produisent des valeurs Ct P1 et N1 <33 sur les sondes P1 et N1.
- Pour le contrôle négatif, vérifiez qu’il n’y a pas de valeurs N1 ou P1 Ct dans l’un des deux puits témoins négatifs (N23/N24). Vérifiez que les valeurs Ct ont des courbes d’amplification valides avant d’invalider la plaque.
12. Interprétation des résultats de l’échantillon
- Déterminez le résultat du patient en suivant le diagramme (Figure 3) et rapportez les échantillons résolus.
- Évaluez le résultat P1 comme VALIDE ou NON VALIDE. Si P1 produit un résultat de Ct <33, considérez le puits VALIDE et procédez au résultat de N1. Si P1 produit un résultat de Ct >=33 ou aucune valeur Ct, considérez le puits INVALID.
- Évaluez le résultat N1 comme OUI, NON ou NON*. Si N1 produit un résultat de Ct <33, le puits est OUI. Si N1 ne produit pas de valeur Ct, le puits est NO. Si N1 produit un Ct >=33, le puits est NO*. Confirmez que toutes les valeurs N1 Ct sont associées à une courbe d’amplification réelle. Si une valeur Ct pour N1 n’a pas de courbe d’amplification, le puits est NO.
- Identifiez les échantillons répétés (N1 Rerun ou Rerun), étiquetez-les avec un numéro d’échantillon interne et un type d’échantillon et retournez-les au flux de travail de chargement (section 8.1-8.3).
- Évaluez le résultat P1 comme VALIDE ou NON VALIDE. Si P1 produit un résultat de Ct <33, considérez le puits VALIDE et procédez au résultat de N1. Si P1 produit un résultat de Ct >=33 ou aucune valeur Ct, considérez le puits INVALID.
13. Nettoyage du laboratoire
- Robots de manutention de liquides
- Nettoyez tous les côtés avec un désinfectant de niveau intermédiaire. N’utilisez pas d’éthanol car il dégraderait le plastique.
- Essuyez délicatement l’extrémité de la pipette et la poubelle avec une lingette à l’alcool (70 % d’éthanol ou 100 % d’isopropanol). Essuyez le clavier et la souris.
- Armoire de biosécurité
- Nettoyez toutes les surfaces avec un désinfectant de niveau intermédiaire. Allumez la lumière UV pendant 15 min.
Résultats
Nous avons déterminé la plage de détection des sondes RT-qPCR et des amorces pour la teneur en acides nucléiques synthétiques pour le SARS-CoV-2 (N1) et Hs_RPP30 (P1). Une dilution en série de 10 fois des concentrations connues d’ARN synthétique combiné du SRAS-CoV-2 et d’ADN synthétique Hs_RPP30 dans l’eau a été effectuée. La formule suivante a été utilisée pour convertir le poids moléculaire en nombre de copies de gènes
Nombre de copies de gènes = (ng * 6,0221 x 1023)/((longueur en paires de bases*660 g/mole) *1 x 109 ng/g)
et la RT-qPCR a été effectuée. Après avoir effectué la RT-qPCR, les courbes linéaires pour la détection N1 (Figure 4A) et la détection P1 (Figure 4B) ont montré de bons coefficients de corrélation sur un large éventail de concentrations de copies de gènes (R2 = 0,9975 et R2 = 0,9884, respectivement). Ce résultat indique que la combinaison des ensembles d’amorces et de sondes n’est pas inhibitrice et peut détecter avec précision l’ARN du SRAS-CoV-2 à une copie de gène/μL (Cq = 33). Une copie de gène équivaut à peu près à une copie virale; cependant, nous n’avons pas déterminé le nombre quantitatif de copies virales dans la salive en raison de la nature semi-quantitative de la RT-qPCR. Nous avons tenté de simuler des échantillons de salive positifs en injectant de l’ARN synthétique du SRAS-CoV-2 de concentrations connues dans de la salive exempte de virus (traitée thermiquement et non traitée thermiquement), mais nous n’avons pas été en mesure de produire une amplification N1 à de faibles concentrations d’ARN (données non présentées). Cela peut être dû à la dégradation de la RNase ou à d’autres facteurs de confusion.
La variabilité entre les essais et les méthodes de chargement manuel des échantillons a également été évaluée. Pour évaluer la variabilité entre les essais, 20 échantillons positifs uniques ont été chargés à l’aide des méthodes manuelles (décrites aux sections 8.1 à 8.3) et automatisées (décrites aux sections 7.1 à 7.11). Les valeurs N1 Ct ont été comparées pour déterminer si les robots de manutention des liquides et le chargement manuel des échantillons produisaient des résultats équivalents (figure 5A). La relation linéaire entre les méthodes manuelles et automatisées a produit un coefficient de corrélation élevé (R2 = 0,9088), ce qui indique que les deux méthodes sont fonctionnellement équivalentes. À mesure que les valeurs N1 Ct augmentaient, la variabilité des valeurs Ct augmentait également. Cette tendance est probablement due à la distribution hétérogène des particules virales dans la salive, qui est plus prononcée lorsque moins de particules sont présentes. Pour évaluer la variabilité intra-essai, une comparaison entre les valeurs N1 Ct des puits répliqués d’échantillons de salive uniques à l’aide des deux méthodes de chargement des échantillons a été effectuée (figure 5B). La relation linéaire entre les répétitions du chargement automatisé des échantillons (R2 = 0,9622) a produit un coefficient de corrélation légèrement plus élevé que celui du chargement manuel (R2 = 0,9589), indiquant une reproductibilité élevée de la détection du SARS-CoV-2 pour les deux méthodes de chargement.
Enfin, une évaluation de la réduction de la viscosité de la salive par rapport aux méthodes de traitement thermique a été effectuée (figure 6). La salive a été obtenue à partir d’une seule source pour éliminer la variabilité de l’échantillon. Une plus grande variabilité des valeurs de P1 Ct au sein d’une méthode de traitement thermique peut indiquer une viscosité plus élevée de l’échantillon, car la salive visqueuse ne peut pas être aspirée et distribuée avec précision. Les méthodes de traitement thermique de 30 min et 60 min ont produit une diminution significative de la variabilité de l’échantillon par rapport à l’absence de contrôle du traitement (p = 0,0006 et p = 0,0429, respectivement). Il n’y avait pas de différence significative entre les traitements de 30 min et 60 min (p = 0,2245); par conséquent, la méthode de traitement thermique de 30 minutes a été mise en œuvre pour réduire le temps de traitement.
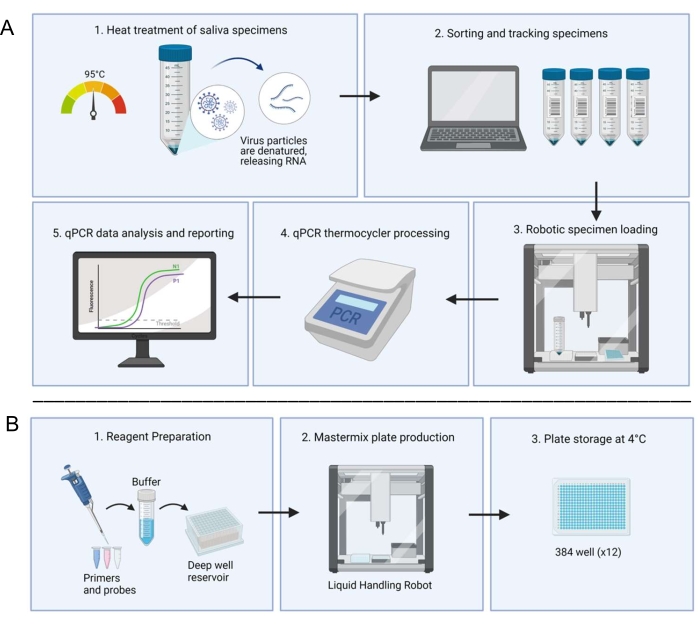
Figure 1 : Flux de travail en laboratoire utilisant le système de diagnostic RT-qPCR à base de salive. (A) Les échantillons sont prélevés et traités thermiquement à 95 °C pendant 30 min. Les échantillons traités sont triés et suivis avec les informations sur les patients via un système de tableur interne. Un robot de manutention de liquides charge des échantillons dans des puits en double de plaques de mélange principales préparées. Un technicien charge manuellement les commandes, scelle la plaque et place la plaque dans un thermocycleur pour le traitement. Les résultats sont analysés à l’aide d’un système informatique automatisé et vérifiés par un technicien. (B) Un technicien prépare des réactifs pour le mélange maître qui sont ajoutés à un réservoir de puits profond dans une armoire de biosécurité stérile. Les réservoirs de puits profonds remplis sont chargés dans un robot de manutention de liquide dédié. Les plaques terminées sont scellées avec du papier d’aluminium, étiquetées et stockées à 4 °C. Veuillez cliquer ici pour voir une version plus grande de cette figure.
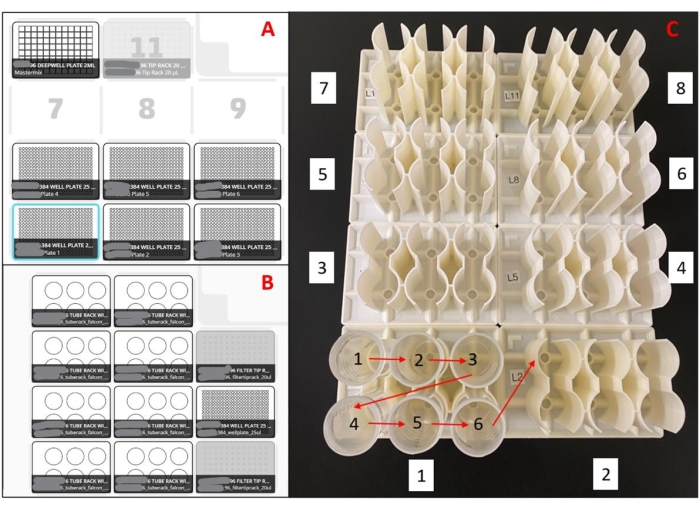
Figure 2: Dispositions utilisées pour le robot de manutention des liquides. (A) Disposition du pont pour le(s) robot(s) de préparation des plaques de mélange principales. Avec une pipette à huit canaux, le robot est programmé pour ramasser les embouts de pipette, aspirer le mélange maître d’un réservoir de puits de 96 puits de profondeur, distribuer le mélange principal dans des plaques vides de 384 puits et éjecter les embouts de pipette dans une poubelle. Ceci est répété pour six plaques par course. (B) Configuration du pont pour le(s) robot(s) de chargement d’échantillons. Avec une pipette à canal unique, le robot est programmé pour ramasser une pointe de pipette, aspirer un échantillon de salive, distribuer un échantillon de salive dans des puits en double d’une plaque de mélange principale de 384 puits et éjecter l’extrémité de la pipette dans une poubelle. Ceci est répété pour 48 échantillons par exécution. (C) Ordre de chargement des tubes d’échantillonnage pour les racks imprimés en 3D. Les flèches rouges indiquent l’ordre de chargement dans un rack et les numéros en boîte blanche indiquent l’ordre de chargement de l’ensemble des racks. L’ensemble de la configuration chargera 188 échantillons en double dans une plaque de 384 puits. Veuillez cliquer ici pour voir une version agrandie de cette figure.
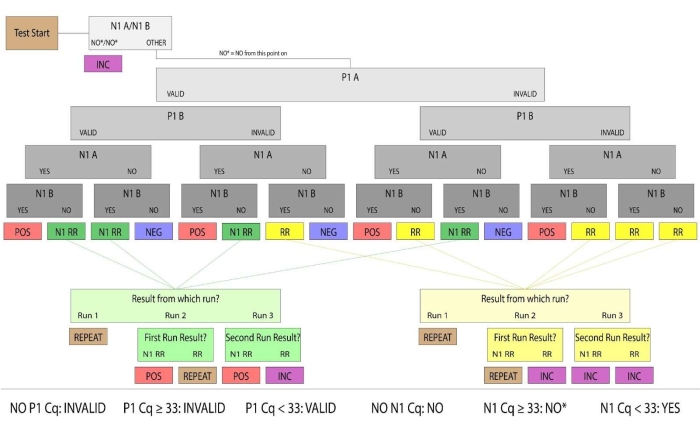
Figure 3 : Organigramme résultant de l’échantillon. Les échantillons avec P1 valide et N1 positif ont été déterminés comme étant des échantillons de salive humaine positifs pour le SRAS-CoV-2. Les résultats valides et positifs/négatifs de l’échantillon ont été considérés comme concluants. Les échantillons qui n’ont pas produit de résultats concluants lors de la première exécution ont été classés comme Réexécution (noté RR) ou N1 Réexécution (noté N1 RR). Les échantillons réexécutés n’avaient pas d’amplification P1 valide, et les échantillons N1 Rerun avaient une amplification N1 positive dans une seule réplique. Si aucune amplification P1 valide n’a pu être produite par une exécution manuelle ultérieure, ou si les deux répétitions avaient des valeurs N1 Ct supérieures au seuil positif (Ct >33), les résultats de l’échantillon ont été considérés comme non concluants. À des fins cliniques, les échantillons de patients qui n’arrivaient pas au laboratoire, qui avaient une quantité insuffisante de salive à pipette ou qui étaient endommagés étaient considérés comme invalides. Veuillez cliquer ici pour voir une version agrandie de cette figure.
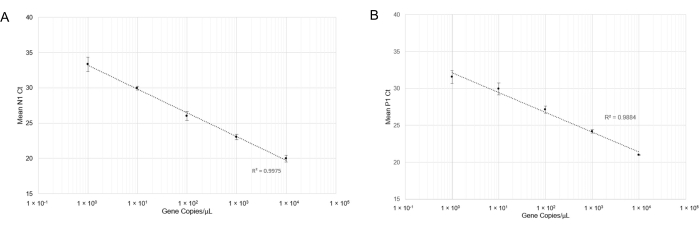
Figure 4 : Détection RT-qPCR de l’ARN synthétique N1 (SARS-CoV-2) et de l’ADN synthétique P1 (Hs_RPP 30). Les courbes standard ont été tracées avec des écarts-types pour déterminer la plage de détection précise à l’aide de cette combinaison sonde/amorce. (A) Les valeurs moyennes de Ct (n = 4) obtenues dans les dilutions respectives ont été tracées par rapport à la quantité estimée d’ARN synthétique (1x100 à 1x104 copies d’ARN dans 10 μL de réaction RT-qPCR). (B) Les valeurs moyennes de Ct (n = 3) obtenues dans les dilutions respectives ont été tracées par rapport à la quantité estimée d’ADN synthétique (1 x 100 à 1 x 104 copies de gènes dans 10 μL de réaction RT-qPCR). Veuillez cliquer ici pour voir une version agrandie de cette figure.
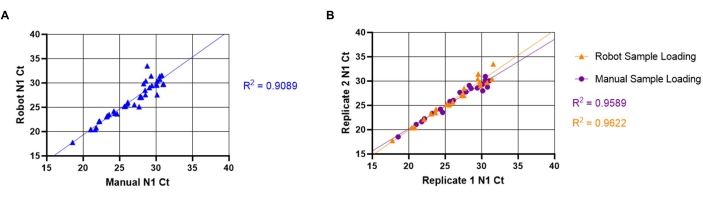
Figure 5 : Comparaison entre les valeurs manuelles et automatisées du SRAS-CoV-2 (N1) Ct de transfert de salive. Les échantillons de salive positifs connus pour le SRAS-CoV-2 (n = 20) ont été chargés en double exemplaire dans une plaque de mélange maître RT-qPCR par un robot de manipulation de liquides. Les échantillons ont une valeur Ct allant de 18 à 32 pour N1. Les mêmes échantillons ont ensuite été chargés manuellement dans des puits en double dans un emplacement de plaque différent. (A) Les valeurs N1 Ct obtenues à partir d’échantillons uniques utilisant à la fois le robot et le chargement manuel de l’échantillon ont été transposées pour déterminer la variabilité entre les essais entre le chargement manuel et le chargement robotisé. (B) La variabilité intra-essai a également été déterminée en utilisant une répétition transposée des valeurs N1 Ct obtenues à partir du chargement robotisé et manuel de l’échantillon. Veuillez cliquer ici pour voir une version agrandie de cette figure.
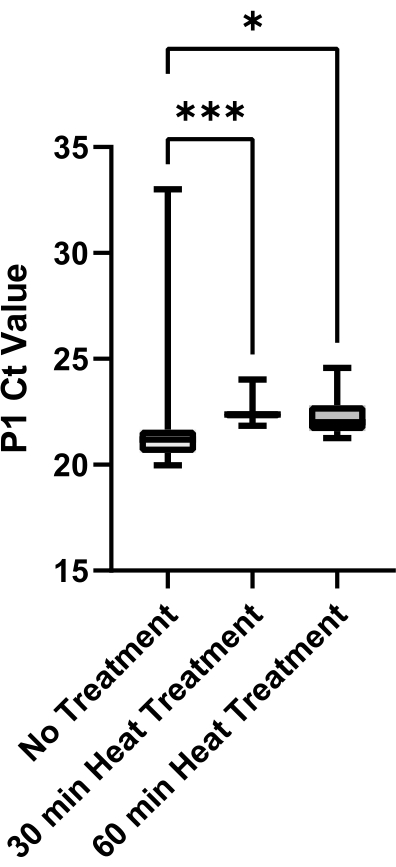
Figure 6 : Évaluation des méthodes de traitement thermique pour la réduction de la viscosité dans la salive. La salive négative du SRAS-CoV-2 a été recueillie à partir d’une seule source et les aliquotes ont été traitées thermiquement pendant 0 min, 30 min ou 60 min à 95 °C. Les valeurs Ct P1 des répétitions techniques (n = 12) de chaque condition ont été tracées pour déterminer la variabilité entre les méthodes de traitement. Les comparaisons par paires entre les groupes ont été évaluées à l’aide d’un test t non apparié (*** indique p <0,001, * indique p <0,05). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Comparaison de N1 Ct dans des échantillons de salive à faible teneur en P1 Ct. Les échantillons positifs avec un faible Ct P1 ont été sélectionnés et comparés au N1 Ct (n = 106). Les valeurs N1 Ct variaient de 14 à 33, ce qui indique que le test a une plage dynamique dans les échantillons de salive comparable à la courbe standard. Veuillez cliquer ici pour télécharger ce fichier.
| Composant | Séquence (5'→3') | Concentration des stocks | Volume | ||
| Sonde 2019-nCoV-N1 | /5FAM/ACCCCGCAT/ZEN/TACGTTTGGTGGACC/3IABkFQ | 50 μM | 500 μL | ||
| 2019-nCoV-N1-Pour | GACCCCAAAATCAGCGAAAT | 100 μM | 2000 μL | ||
| 2019-nCoV-N1-Rev | TCTGGTTACTGCCAGTTGAATCTG | 100 μM | 2000 μL | ||
| Sonde Hs RPP30 Cy5 | /5Cy5/TTCTGACCT/ZEN/GAAGGCTCTGCGCG/3IABkFQ | 50 μM | 500 μL | ||
| Hs-RPP30-Pour | AGATTTGGACCTGCGAGCG | 100 μM | 2000 μL | ||
| Hs-RPP30-Rev | GAGCGGCTGTCTCCACAAGT | 100 μM | 2000 μL | ||
| Eau | - | - | 11000 μL | ||
Tableau 1 : Composants du mélange sonde/apprêt N1+P1.
| Composant | Concentration des stocks | Volume par réaction | Concentration finale | Volume de lot | ||
| Mélange d’enzymes Luna WarmStart RT | 20X | 0,5 μL | 1X | 3 mL | ||
| Mélange de réaction de tampon Luna | 2X | 5,0 μL | 1X | 30 mL | ||
| Mélange d’amorce/sonde N1+P1 | nCoV N1 F : 10 μM | 0,5 μL | 500 nM | 3 mL | ||
| nCoV N1 R : 10 μM | 500 nM | |||||
| Sonde nCoV N1 : 2,5 μM | 125 nM | |||||
| RPP_30 P1 F : 10 μM | 500 nM | |||||
| RPP_30 P1 R : 10 μM | 500 nM | |||||
| Sonde RPP_30 P1 : 2,5 μM | 125 nM | |||||
| Eau sans nucléase | --- | 2 μL | --- | 12 mL | ||
| Sous-total | --- | 8 μL | --- | 48 mL | ||
| Modèle | 2 μL | |||||
Tableau 2 : Composants du mélange maître multiplex SARS-CoV-2.
| Étape | Température (°C) | Durée | Nombre de cycles |
| Transcription inverse | 55 | 10 min | 1 |
| Dénaturation initiale | 95 | 1 min | 1 |
| Touché | 95 | 10 secondes | 3 |
| 72 | 30 secondes | ||
| 95 | 10 secondes | 3 | |
| 69 | 30 secondes | ||
| 95 | 10 secondes | 3 | |
| 66 | 30 secondes | ||
| Amplification principale | 95 | 10 secondes | 40 |
| 65 | 30 secondes |
Tableau 3 : Protocole RT-qPCR d’atterrissage. Conditions de thermocyclage pour le test de diagnostic RT-qPCR SARS-CoV-2 en une seule étape.
| Étape d’atterrissage | Pas d’étape d’atterrissage | |||
| Moyenne N1 Ct | Moyenne P1 Ct | Moyenne N1 Ct | Moyenne P1 Ct | |
| Exemple 1 | 19.65 | 22.7 | 27.8 | 28.3 |
| Exemple 2 | 22.24 | 24.9 | 28.77 | 30.5 |
| Exemple 3 | 18.85 | 19.2 | 24.65 | 25.9 |
| Exemple 4 | 25.56 | 22.8 | 31.93 | 29.2 |
| Exemple 5 | 22.34 | 24.8 | 38.48 | 40.0 (Échec de la détection) |
Tableau 4 : Comparaison des valeurs de Ct d’atterrissage pour cinq échantillons positifs par rapport aux valeurs de Ct sans toucher.
| Échantillon | TigerSaliva | Dosage du SRAS-CoV-2 à base de salive disponible dans le commerce | ||
| N1 Ct | P1 Ct | Valeur Covid-19 | Valeur RNaseP | |
| D11 | 16.4 | 18.1 | 20.86 | 23.4 |
| E11 | 18.9 | 19.1 | 25.6 | 21.2 |
| F11 | 19.5 | 18.4 | 22.8 | 22.2 |
| G11 | 22.2 | 19.1 | 23.7 | 22.9 |
| H11 | 26.4 | 21.3 | 32.2 | 26.7 |
| A12 | 14.8 | 16.5 | 29.15 | 19 |
| B12 | 24 | 19.6 | 31.05 | 21.35 |
| C12 | 14.9 | 17.5 | 20.84 | 18.9 |
Tableau 5 : Comparaison des résultats de TigerSaliva Ct et des résultats du test SARS-CoV-2 à base de salive disponibles dans le commerce. Les deux tests ont été effectués sur les mêmes échantillons de salive (n = 8).
Fichier supplémentaire 1 : Script personnalisé pour la création de plaques de mixage maître de robot. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 2: Script personnalisé pour le traitement de la salive sur des robots de chargement d’échantillons. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 3 : Instructions pour l’auto-prélèvement d’échantillons de salive de haute qualité auprès des participants. Vous trouverez plus de détails dans la courte description vidéo du processus de test disponible sur https://www.clemson.edu/centers-institutes/reddilab/index.html. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 4 : Exemple de feuille de calcul d’admission. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 5 : Exemple de feuille de calcul de chargement. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 6 : Exemple de schéma de mise en page de plaques de 384 puits. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le test décrit dans le protocole a été évalué par une étude de validation indépendante. Il a été constaté que le test avait une spécificité de 98,9 % (1,1 % de faux positifs) et une sensibilité de 90,0 % (10,0 % de faux négatifs) lorsqu’ils étaient évalués par rapport à des écouvillons nasopharyngés appariés prélevés en même temps (n = 837; 817 négatifs, 20 positifs). Fait important, trois participants testés positifs avec TigerSaliva et négatifs avec un écouvillon nasopharyngé ont été testés à nouveau avec des écouvillons 48 heures plus tard et ont donné des résultats positifs, indiquant que TigerSaliva pourrait être en mesure de détecter les infections à SARS-CoV-2 plus tôt au cours de la maladie.
Nous avons simulé des échantillons de salive positifs en piquant de la salive sans virus (traitée thermiquement et non traitée thermiquement) avec des concentrations connues d’ARN synthétique du SRAS-CoV-2 et avons effectué une dilution de 10 fois pour déterminer la limite de Ct dans la salive. Le gène N1 n’était pas détectable en dessous de 10 000 copies de gènes (environ Ct = 28) dans des échantillons positifs simulés. Nous soupçonnons que cela est dû à la dégradation de la RNase ou à d’autres facteurs de confusion. Cependant, l’interaction des RNases de salive avec l’ARN synthétique nu est probablement différente de l’interaction avec les particules virales, même après qu’elles aient été dénaturées par la chaleur. Des échantillons de salive positifs ont été identifiés avec Ct >30 et des laboratoires externes ont obtenu des données sur la séquence génétique du SRAS-CoV-2 à partir de ces échantillons. Nous supposons que les protéines virales offrent une protection contre la dégradation de l’ARN dans les échantillons de salive des patients.
L’étape la plus critique du protocole est la mise en œuvre de l’automatisation pour la préparation du mélange maître et le traitement des échantillons de salive (sections 4 et 7 respectivement). Cela permet de chevaucher les processus de tâche, ce qui réduit considérablement le délai d’exécution. Une autre étape critique est l’interprétation des résultats cliniques (sections 11 et 12). L’établissement de catégories de résultats intermédiaires (Réexécution et Réexécution N1) a également minimisé l’occurrence de résultats d’essais non concluants.
Nous avons démontré que la variation entre les méthodes manuelles et automatisées de chargement des échantillons de salive est négligeable (figure 5A) et que l’automatisation peut améliorer la reproductibilité de la détection du SARS-CoV-2 (figure 5B). L’automatisation devrait être privilégiée pour faciliter les tests lors de la conception et de l’expansion des laboratoires cliniques25. Le flux de travail du laboratoire est amélioré avec la mise en œuvre de tâches automatisées par robot26. Les capacités open source des robots de manipulation de liquides permettent la mise en œuvre de scripts personnalisés pour la conception de protocoles. Cela fait des robots de manutention de liquides un système peu coûteux et hautement modifiable par rapport aux méthodes d’automatisation clinique traditionnelles. C’est également une stratégie idéale pour exécuter des tâches de laboratoire très répétitives. Le haut niveau de personnalisation du système se traduit par la liberté de modifier le matériel de laboratoire (par exemple, les tubes de collecte, les embouts de pipette ou les plaques à 384 puits) en cas de pénurie. Par conséquent, l’automatisation à l’aide de robots de manipulation de liquides est viable pour la surveillance et la recherche à grande et à petite échelle.
Un avantage majeur de cette stratégie de test est un délai d’exécution beaucoup plus court par rapport aux autres laboratoires cliniques. L’utilisation de robots automatisés de manutention de liquides joue un rôle clé dans la réduction des délais d’exécution, mais l’utilisation simultanée de robots et de thermocycleurs est également essentielle pour maximiser l’efficacité des tests. Un robot et un thermocycleur doivent être utilisés en paire, où les deux machines sont utilisées en tandem pour le chargement ininterrompu des échantillons et l’analyse des résultats des échantillons. Une fois qu’un flux régulier d’échantillons assignés est établi, toutes les paires de machines peuvent être utilisées simultanément. L’utilisation simultanée constante de robots et de thermocycleurs augmente considérablement la capacité et l’efficacité des tests, ce qui est crucial pour s’adapter au volume élevé de tests.
Contrairement à d’autres protocoles RT-qPCR SARS-CoV-2 établis, nous avons inclus une étape d’atterrissage dans le protocole thermocycleur pour améliorer le recuit de la sonde et des ensembles d’amorces aux gènes cibles27, réduisant ainsi le risque d’échec de l’amplification. Les résultats ont démontré que l’atterrissage améliorait la détection des échantillons positifs sans risquer la perte d’une liaison d’amorce spécifique (tableau 4). Nous avons déterminé qu’un large éventail de copies d’ARN du SRAS-CoV-2 (figure 4A) et de copies d’ADN Hs_RPP30 (figure 4B) peuvent être détectées simultanément par le test RT-qPCR.
L’une des limites des robots de manipulation de liquides est la possibilité d’une contamination croisée à partir d’échantillons positifs lors du transfert de salive. La salive est un fluide viscoélastique28 et peut s’enfiler dans les puits adjacents après avoir été distribuée à partir de la pointe de la pipette. De plus, l’hétérogénéité de la salive29 peut entraîner une répartition inégale des particules virales dans l’ensemble de l’échantillon. Cela augmente la possibilité de faux positifs et de négatifs, ce qui nécessite la désignation d’échantillons N1 Rerun et Rerun. Cependant, 14,1 % des échantillons initialement désignés comme N1 Rerun se sont révélés positifs pour le SARS-CoV-2 et étaient plus de 30 fois plus susceptibles que les échantillons De Rerun de se résoudre comme positifs après un nouveau test. Par conséquent, la différenciation de Rerun de N1 Rerun (Figure 3) a permis une séparation plus précise des échantillons potentiellement positifs, augmentant ainsi la sensibilité et la spécificité de notre test de diagnostic. D’autres paramètres résultants pour les tests de salive diagnostiques n’ont pas fait cette distinction12,14,24,30,31.
Les échantillons de salive peuvent être difficiles à pipeter en raison de l’hétérogénéité et de la viscosité32. Le traitement thermique dénature adéquatement les protéines de la biomatrice de la salive, réduisant ainsi la viscosité et éliminant le besoin de réactifs d’extraction de l’ARN9, qui étaient rares aux premiers stades de la pandémie10. Le traitement thermique prolongé inactive également les virus présents33, ce qui permet un traitement en laboratoire à des niveaux de biosécurité inférieurs. Par conséquent, une extraction d’ARN à base de chaleur (décrite à la section 5.4) a été mise en œuvre pour diminuer la viscosité par dénaturation des protéines (figure 6). Sur la base des résultats, nous postulons que le traitement thermique peut également homogénéiser les échantillons de salive en plus de dénaturer la biomatrice protéique. D’autres groupes ont combiné le traitement thermique et le traitement à la protéinase K pour augmenter l’homogénéité9,14,34. Nous avons choisi de ne pas mettre en œuvre cette étape car elle pourrait dénaturer les protéines virion à un rythme qui laisse l’ARN viral exposé à la dégradation thermique35. De plus, la dilution de l’échantillon avec la protéinase K peut masquer des échantillons positifs contenant moins de particules virales, diminuant ainsi la sensibilité. De plus, les résultats du test ont été comparés à un test SARS-CoV-2 à base de salive disponible dans le commerce (Logix Smart COVID-19) qui utilise l’extraction magnétique de l’ARN des billes (tableau 5). Il a été constaté que le test actuel était mieux adapté à la détection d’échantillons positifs faibles par rapport au test disponible dans le commerce.
Il est difficile de quantifier le nombre de copies virales dans la salive en utilisant uniquement la RT-qPCR, car la qPCR est semi-quantitative. Il existe une variation inhérente entre les valeurs Ct qui provient de limitations techniques. Le nombre de copies de gènes peut être déterminé à partir des valeurs Ct (Figure 4) et est à peu près équivalent au nombre de copies virales. Une solution possible pour déterminer le nombre de copies virales dans les échantillons de salive est la ddPCR, qui fournit une quantification stricte des copies de gènes dans la réaction. Cependant, nous pensons qu’il est adéquat de fournir des résultats qualitatifs aux cliniciens et que le contenu viral relatif peut être comparé à travers les échantillons traités avec nos méthodes.
Malgré certaines limitations qui surviennent lors de l’utilisation de la salive, le test SARS-CoV-2 par RT-qPCR à base de salive s’avère être une méthode efficace pour la détection rapide et fiable de l’ARN viral à n’importe quelle échelle de test. Cela est particulièrement vrai lorsqu’il est associé à l’utilisation de systèmes de manipulation de liquides open source. Cette approche de test peut être modifiée pour détecter d’autres séquences d’acides nucléiques pertinentes pour le diagnostic, telles que les agents de maladies infectieuses, les marqueurs de maladies ou d’autres virus. Cela rend le test applicable aux efforts de diagnostic clinique et de recherche.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer. Le test décrit dans le protocole relève de l’EUA SalivaDirect déposée par la Yale School of Public Health.
Remerciements
Les auteurs remercient l’administration de Clemson, le personnel médical et les employés du laboratoire clinique du laboratoire REDDI qui ont aidé à mettre en œuvre et à gérer les tests du SRAS-CoV-2. Nous remercions le Dr Phillip Buckhaults et la Dre Carolyn Bannister de l’Université de Caroline du Sud pour leurs premiers conseils sur le projet et leurs contacts avec l’industrie pour l’achat d’équipement. Nous remercions de nombreux étudiants, professeurs et membres du personnel pour leur aide dans la collecte d’échantillons. Merci aux étudiants de Creative Inquiry pour la collecte de données de courbe standard. Le financement de cette étude a été reçu de la subvention P20GM121342 des National Institutes of Health (accordée à DD et LGP), du département des sports de Clemson, du vice-président de la recherche de l’Université de Clemson et du comité d’examen conjoint du gouverneur de Caroline du Sud et des obligations.
matériels
| Name | Company | Catalog Number | Comments |
| 100% EtOH | Fisher scientific | 22-032-601 | |
| 20 uL Filtered Pipette Tips | Opentrons | 20uL tips | |
| 2mL Microcentrifuge Tubes | Fisher Scientific | 14-666-313 | Alternate product may be used |
| Armadillo PCR Plate, 384-well, clear, white wells | Thermo Scientific | AB3384 | Alternate product may be used |
| Celltreat 2mL 96 Deep Well Plates | Fisher Scientific | 50-828-743 | For mastermix preparation |
| Clear PCR Sealing Sheets | Thermo Scientific | AB0558 | Alternate product may be used |
| DPEC Treated Water | Ambion (Thermo Scientific) | AM9916 | |
| Flip Cap 50 mL Conical Tubes | VWR | 75845-210 | For sample collection |
| Foil PCR Sealing Sheets | Thermo Scientific | AB0626 | For storage of mastermix plates, Alternate product may be used |
| HS_RPP30 Synthetic DNA | Integrated DNA Technologies | 299788131 | P1 positive control |
| Luna Buffer Probe One-Step Reaction | New England Biolabs | M3006B | |
| Luna WarmStart RT Enzyme Mix | New England Biolabs | M3002B | |
| nCOV_N1 Forward Primer, 100 nmol | Integrated DNA Technologies | 10006830 | |
| nCOV_N1 Probe Aliquot, 50 nmol | Integrated DNA Technologies | 10006832 | Probe can be synthesized by other vendors with SYBR or FAM fluophores |
| nCOV_N1 Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006831 | |
| Opentron HEPA Filter Module | Opentrons | N/A | Not required, but useful to reduce contamination |
| Opentron Multichannel Attachment, P20 | Opentrons | 999-00005 | For mastermix preparation |
| Opentron OT-2 Liquid Handling Robot | Opentrons | OT-2 | |
| Opentron Pipette Attachment, P20 | Opentrons | 999-0000215 | For sample loading |
| Oven | Memmert | UF450 PLUS 208V-3PH | |
| PCR Tubes (rnase, dnase free) | Fisher Scientific | 14-230-225 | For aliquots of positive and neg controls |
| PolarSafe Aluminum Cooling Block, 15-Well (1.5/2.0 mL Tubes) | VWR | 10808-952 | |
| PolarSaf Aluminum Cooling Block, 24-Well (0.5mL tubes) | VWR | 10808-956 | |
| RNAse P (ATTO 647) Probe, 50 nmol | Integrated DNA Technologies | 10007062 | Probe can be synthesized by other vendors with Cy5 fluorophore |
| RNAse P Forward Primer, 100nmol | Integrated DNA Technologies | 10006836 | |
| RNAse P Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006837 | |
| Sars-CoV-2 Synthetic RNA Control 2 | Twist Biosciences | 102024 / 103907 / 103909 | N1 Positive Control |
| Scanners | Code | CR1500 | Only required when scaling up |
| Small HEPA Filtered Hood | Erlab | Captair Bio 321 | For mastermix preparation |
| Thermocycler CFX384 Touch | Biorad | CFX384 Touch | Alternate models can be used, e.g. CFX384 Opus |
| X-acto Knife Set | Staples | N/A | To cut foil for keeping control wells covered |
Références
- Zhu, N., et al. A novel coronavirus from patients with pneumonia in China, 2019. New England Journal Medicine. 382 (8), 727-733 (2020).
- Chen, J. Pathogenicity and transmissibility of 2019-nCoV-A quick overview and comparison with other emerging viruses. Microbes and Infection. 22 (2), 69-71 (2020).
- Petersen, E., et al. Comparing SARS-CoV-2 with SARS-CoV and influenza pandemics. The Lancet. Infectious Diseases. 20 (9), 238-244 (2020).
- Honein, M. A., et al. Summary of Guidance for Public Health Strategies to Address High Levels of Community Transmission of SARS-CoV-2 and Related Deaths, December 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (49), 1860-1867 (2020).
- World Health Organization. Surveillance strategies for COVID-19 human infection: interim guidance. World Health Organization. , Available from: https://apps.who.int/iris/handle/10665/332051 (2020).
- Screening testing for early detection of SARS-CoV-2 infection. Division of viral diseases. Centers for Disease Control. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/hcp/tsting-overview.html#PublicHealthSurveillance (2020).
- Larremore, D. B., et al. Test sensitivity is secondary to frequency and turnaround time for COVID-19 screening. Science Advances. 7 (1), 5393(2021).
- Sethuraman, N., Jeremiah, S. S., Ryo, A. Interpreting Diagnostic Tests for SARS-CoV-2. JAMA. 323 (22), 2249-2251 (2020).
- Chu, A. W., et al. Evaluation of simple nucleic acid extraction methods for the detection of SARS-CoV-2 in nasopharyngeal and saliva specimens during global shortage of extraction kits. Journal of clinical virology: the official publication of the Pan American Society for Clinical Virology. 129, 104519(2020).
- Guan, D., et al. Global supply-chain effects of COVID-19 control measures. Nature Human Behaviour. 4 (6), 577-587 (2020).
- Bastos, M. L., Perlman-Arrow, S., Menzies, D., Campbell, J. R. The Sensitivity and Costs of Testing for SARS-CoV-2 Infection With Saliva Versus Nasopharyngeal Swabs: A Systematic Review and Meta-analysis. Annals of Internal Medicine. 174 (4), 501-510 (2021).
- Pasomsub, E., et al. Saliva sample as a non-invasive specimen for the diagnosis of coronavirus disease 2019: a cross-sectional study. Clinical Microbiology and Infection: The Official Publication of The European Society of Clinical Microbiology and Infectious Diseases. 27 (2), (2021).
- To, K. K., et al. Consistent Detection of 2019 Novel Coronavirus in Saliva. Clinical Infectious Diseases: An Official Publication of The Infectious Diseases Society of America. 71 (15), 841-843 (2020).
- Vogels, C. B., et al. SalivaDirect: A simplified and flexible platform to enhance SARS-CoV-2 testing capacity. Med (New York, N.Y.). 2 (3), 263-280 (2021).
- Griesemer, S. B., et al. Evaluation of Specimen Types and Saliva Stabilization Solutions for SARS-CoV-2 Testing. Journal of Clinical Microbiology. 59 (5), 1418-1420 (2021).
- Wehrhahn, M. C., et al. Self-collection: An appropriate alternative during the SARS-CoV-2 pandemic. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 128, 104417(2020).
- Barza, R., Patel, P., Sabatini, L., Singh, K. Use of a simplified sample processing step without RNA extraction for direct SARS-CoV-2 RT-PCR detection. Journal of Clinical Virology: The Official Publication of the Pan American Society for Clinical Virology. 132, 104587(2020).
- Paltiel, A. D., Zheng, A., Walensky, R. P. Assessment of SARS-CoV-2 Screening Strategies to Permit the Safe Reopening of College Campuses in the United States. JAMA Network Open. 3 (7), 2016818(2020).
- Centers for Disease Control. 2019-Novel Coronavirus (2019-nCoV) Real-Time RT-PCR Primers and Probes. U.S. Department of Health and Human Services. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/lab/rt-pcr-panel-primer-probes.html (2020).
- Cresswell, K., Ramalingam, S., Sheikh, A. Can Robots Improve Testing Capacity for SARS-CoV-2. Journal of Medical Internet Research. 22 (8), 20169(2020).
- Villanueva-Cañas, J. L., et al. Implementation of an open-source robotic platform for SARS-CoV-2 testing by real-time RT-PCR. PLoS One. 16 (7), 0252509(2021).
- Robot Boosts COVID-19 Testing Efficiency. University of South Carolina College of Pharmacy. , Available from: https://sc.edu/study/colleges_schools/pharmacy/about/news/2020/robot-boosts-testing-effort.php (2020).
- Matic, N., et al. Practical challenges to the clinical implementation of saliva for SARS-CoV-2 detection. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of The European Society of Clinical Microbiology. 40 (2), 447-450 (2021).
- Sahajpal, N. S., et al. SalivaSTAT: Direct-PCR and Pooling of Saliva Samples Collected in Healthcare and Community Setting for SARS-CoV-2 Mass Surveillance. Diagnostics. 11 (5), Basel, Switzerland. 904(2021).
- Genzen, J. R., et al. Challenges and Opportunities in Implementing Total Laboratory Automation. Clinical Chemistry. 64 (2), 259-264 (2018).
- Archetti, C., Montanelli, A., Finazzi, D., Caimi, L., Garrafa, E. Clinical Laboratory Automation: A Case Study. Journal of Public Health Research. 6 (1), 881(2017).
- Hecker, K. H., Roux, K. H. High and low annealing temperatures increase both specificity and yield in touchdown and stepdown PCR. BioTechniques. 20 (3), 478-485 (1996).
- Bhat, P. P., et al. Formation of beads-on-a-string structures during break-up of viscoelastic filaments. Nature Physics. 6, 625-631 (2010).
- Miller, C. S., et al. Current developments in salivary diagnostics. Biomarkers in Medicine. 4 (1), 171-189 (2010).
- Moreno-Contreras, J., et al. Saliva Sampling and Its Direct Lysis, an Excellent Option To Increase the Number of SARS-CoV-2 Diagnostic Tests in Settings with Supply Shortages. Journal of Clinical Microbiology. 58 (10), 01659(2020).
- Wang, W., et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA. 323 (18), 1843-1844 (2020).
- Landry, M. L., Criscuolo, J., Peaper, D. R. Challenges in use of saliva for detection of SARS CoV-2 RNA in symptomatic outpatients. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 130, 104567(2020).
- Lista, M. J., et al. Resilient SARS-CoV-2 diagnostics workflows including viral heat inactivation. PLoS One. 16 (9), 0256813(2021).
- Brotons, P., et al. Validation and implementation of a direct RT-qPCR method for rapid screening of SARS-CoV-2 infection by using non-invasive saliva samples. International Journal of Infectious Diseases: IJID: Official Publication of The International Society for Infectious Diseases. 110, 363-370 (2021).
- Batéjat, C., Grassin, Q., Manuguerra, J. C., Leclercr, I. Heat inactivation of the severe acute respiratory syndrome coronavirus 2. Journal of Biosafety and Biosecurity. 3 (1), 1-3 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon