Method Article
Eficiente SARS-CoV-2 Quantitative Reverse Transcriptase PCR Saliva Diagnostic Strategy utilizando robôs de pipetação de código aberto
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
O protocolo descreve um método de diagnóstico SARS-CoV-2 que utiliza a automação de código aberto para realizar testes moleculares RT-qPCR de amostras de saliva. Essa abordagem escalável pode ser aplicada à vigilância clínica em saúde pública, bem como ao aumento da capacidade de laboratórios universitários menores.
Resumo
O surgimento da recente crise global de saúde SARS-CoV-2 introduziu desafios fundamentais para a pesquisa epidemiológica e testes clínicos. Caracterizada por uma alta taxa de transmissão e baixa mortalidade, a pandemia COVID-19 necessitava de testes diagnósticos precisos e eficientes, particularmente em populações fechadas, como universidades residenciais. A disponibilidade inicial de testes de ácido nucleico, como cotonetes nasofaríngeos, foi limitada devido à pressão da cadeia de suprimentos, o que também atrasou o relato dos resultados dos testes. O teste de reação em cadeia de polimerase quantitativa baseado em saliva (RT-qPCR) mostrou-se comparável em sensibilidade e especificidade a outros métodos de teste, e a coleta de saliva é menos fisicamente invasiva aos participantes. Consequentemente, desenvolvemos um ensaio diagnóstico multiplex RT-qPCR para vigilância populacional da Universidade Clemson e da comunidade circundante. O ensaio utilizou robôs de manuseio líquido de código aberto e termociclistas em vez de sistemas complexos de automação clínica para otimizar o fluxo de trabalho e a flexibilidade do sistema. A automação do RT-qPCR baseado em saliva permite a detecção rápida e precisa de uma ampla gama de concentrações de RNA virais para demandas de testes em grande e pequena escala. A média de retorno para o sistema automatizado foi de < 9h para 95% das amostras e < 24h para 99% das amostras. O custo para um único teste foi de US$ 2,80 quando todos os reagentes foram comprados em quantidades a granel.
Introdução
Coronavírus-2 associado à síndrome respiratória aguda grave (SARS-CoV-2), um novo coronavírus, surgiu no final de 2019 e rapidamente se espalhou por toda a população global1. A infecção pelo SARS-CoV-2 causa a doença coronavírus 2019 (COVID-19), doença altamente contagiosa com sintomas respiratórios e inflamatórios potencialmente graves. A alta transmissibilidade aliada à baixa mortalidade indicou que o vírus se espalharia rapidamente pelas populações e exigiria um aumento dos testes diagnósticos2,3. As recomendações de saúde pública incentivaram a triagem populacional em larga escala para isolar os casos e, posteriormente, reduzir as taxas de transmissão4,5,6. Além disso, modelos de vigilância populacional revelaram que o aumento da frequência de testes e a diminuição do tempo de notificação tiveram um efeito maior na redução da transmissão do que no aumento da sensibilidade ao teste7. Isso é provável porque indivíduos infectados poderiam ser colocados em quarentena mais cedo, quebrando assim cadeias de infecção.
O padrão original de teste de amplificação de ácido nucleico (NAAT) era cotonetes nasofaríngeos (NP) processados por RT-qPCR8. No entanto, complicações surgem com essa forma de testes para populações muito grandes, como aumento do custo relativo e pressão exacerbada da cadeia de suprimentos9,10. Além disso, tanto a coleta de espécimes quanto o processamento de métodos comuns de NAAT (incluindo cotonetes NP, cotonetes orofaríngeos, cotonetes de turbinas médias e cotonetes nasais) dependem de equipamentos especializados, reagentes e pessoal médico9,10.
Um substituto adequado para testes de NP swab RT-qPCR é o teste baseado em saliva, que é uma ferramenta de diagnóstico precisa para detecção SARS-CoV-211,12,13,14. A realização direta de RT-qPCR em amostras de saliva produz sensibilidade e especificidade semelhantes aos cotonetes NP15. Uma grande vantagem que o teste de saliva tem sobre o teste de swab NP é que ele permite a auto-coleta de amostras16. Isso minimiza a necessidade de pessoal médico e maximiza a facilidade de coleta de amostras para os pacientes por serem menos invasivos que os cotonetes de PN. Além disso, uma vez que as amostras de saliva não requerem buffers para remover a amostra de um cotonete (como no caso de amostras de NP), os testes à base de saliva podem utilizar diretamente a extração de ácido ribonucleico baseado no calor (RNA), o que diminui os custos de teste removendo a necessidade de buffers adicionais, mídia de transporte e/ou reagentes de extração de RNA14,17.
O Laboratório de Pesquisa e Educação em Diagnóstico e Intervenção de Doenças da Universidade Clemson (REDDI) foi criado para atender às necessidades da universidade para testes e vigilância covid-19. Em populações fechadas, incluindo universidades, testes frequentes de vigilância aliados ao distanciamento social produziram os desfechos mais favoráveis nos modelos epidemiológicos da prevalência da doença18. Os protocolos CONSOLIDADOS CDC 2019-nCOV RT-qPCR19 e SalivaDirect14 foram adaptados, e a automação foi utilizada no fluxo de trabalho clínico para diminuir o custo e melhorar o tempo de retorno. Grupos anteriores haviam usado robôs de manuseio líquido de código aberto para etapas de extração de RNA SARS-CoV-2 2, mas maximizamos o uso dos robôs para preparar placas de teste e carregar espécimes22. Aqui, mostramos que o protocolo adaptado e a utilização de sistemas de manuseio líquido de código aberto (Figura 1) permitem a RT-qPCR rápida e precisa baseada em saliva e é uma estratégia eficaz para a vigilância em saúde pública em larga escala.
Protocolo
Todas as pesquisas foram realizadas em conformidade com os Conselhos de Revisão Institucional da Universidade Clemson e prisma (Prisma Health IRB # Pro00099491, 1º de julho de 2020).
1. Configuração de robô de manuseio líquido de código aberto
- Instale módulos de filtro de ar particulado de alta eficiência (HEPA) (ver Tabela de Materiais) na parte superior de cada robô de manuseio líquido de acordo com as instruções do fabricante.
- Conecte uma pipeta P20 de 8 canais ao suporte esquerdo do robô de preparação da placa de mixagem mestre, de acordo com as instruções do fabricante.
- Conecte uma pipeta P20 ao suporte direito do robô de carregamento de amostras conforme as instruções do fabricante.
- Baixe os scripts python personalizados (Arquivo Suplementar 1 e Arquivo Suplementar 2) nos computadores apropriados.
- Abra o TigerSaliva Full 384 Loading.py no aplicativo de desktop no computador robô de carregamento de amostras. Clique em Calibrar e configure tanto a pipeta quanto o programa de acordo com as instruções do software.
- Abra 12 Plates.py completos no aplicativo de desktop no computador robô master mix. Clique em Calibrar e configure tanto a pipeta quanto o programa de acordo com as instruções do software.
- Imprima racks de amostra personalizados com uma impressora tridimensional de modelagem de deposição fundida (3D) usando um arquivo CAD (Computer-aided Design, design aided design, design aided) (https://www.myminifactory.com/object/3d-print-141363). Imprima 16 racks por robô de carregamento de amostras, para dois conjuntos de oito racks.
2. Preparação de 20x multiplex N1+P1 sonda/primer mix
- Prepare um lote de 20x multiplex N1+P1 probe/primer mix (volume total de 20 mL, Tabela 1) em um tubo cônico de 50 mL em um ambiente estéril longe de amostras sintéticas sars-cov-2 ou pacientes.
- Alíquota de 1,6 mL da mistura usando uma pipeta sorológica em tubos de centrífugas estéreis de 2,0 mL e etiquetar adequadamente.
- Armazene alíquotas em um congelador de -20 °C até estar pronto para usar.
3. Preparação de mistura de controle positiva
NOTA: A mistura de controle positiva não deve ser feita no mesmo ambiente estéril que a mistura de sonda/primer ou outros componentes de mixagem mestre. Deve-se utilizar um recipiente separado de água sem nuclease.
- Diluir SARS-CoV-2 RNA sintético (N1) de 1.000.000 cópias genéticas/μL (cpμ) a 10.000 cpμ adicionando 10 μL de solução de estoque a 990 μL de água sem nuclease. Alíquota de 25 μL de 10.000 cpμ em tubos estéreis de 0,2 mL e etiqueta apropriadamente. Armazene alíquotas nãousadas a -80°C.
NOTA: O RNA sintético deve ser armazenado a -80°C para evitar a degradação. - Diluir Hs_RPP30 DNA sintético (P1) de 200.000 cpμ a 10.000 cpμ adicionando 50 μL de solução de estoque a 950 μL de água sem nuclease. Alíquota de 25 μL de 10.000 cpμ em tubos estéreis de 0,2 mL e etiqueta apropriadamente. Armazene alíquotas nãousadas a -80°C.
- Diluir cada componente a uma concentração final de 200 cpμ adicionando 20 μL de SARS-CoV-2 e Hs_RPP30 estoques de 10.000 cpμ para 960 μL de água sem nuclease, para um total de 1000 μL. Aliquot 20 μL de controle positivo misto de 200 cpμ em tubos de 0,2 mL e etiqueta apropriadamente. Armazene alíquotas a -80°C até ficar pronto para uso.
4. Preparação de placas de mixagem mestre
NOTA: Faça mistura mestre em um ambiente estéril longe de amostras sintéticas de RNA SARS-CoV-2 ou pacientes. Todos os componentes devem ser completamente descongelados antes de adicionar à mistura; sem o degelo adequado, as concentrações podem estar incorretas. O descongelamento inadequado é indicado pela presença de gelo ou cor irregular de reagentes. Guarde em um bloco congelador enquanto prepara a mistura.
- Prepare um lote de mix mestre multiplex (volume total de 48 mL, Tabela 2) em um tubo cônico de 50 mL.
- Homogeneize a mistura girando o tubo em 3x. Não misture por tubulação para cima e para baixo ou vórtice, pois isso danificará a enzima.
- Encha as colunas 1-4 de um reservatório de poços estéreis de 96 poços profundos, transferindo 1,48 mL de mistura mestre em cada poço. Um cônico de 50 mL preenche quatro colunas, o suficiente para 12 placas.
- Cubra o reservatório do poço profundo com um selo de papel alumínio e coloque-o no dedicado robô de manuseio líquido master mix. Coloque o reservatório de poços profundos cheios no deck 10, coloque seis placas vazias de 384 poços nos decks 1-6, e a caixa de ponta P20 no deck 11. Descubra a placa do poço profundo e caixas de ponta e feche o robô.
- Inicialize o protocolo operacional Python personalizado clicando em Iniciar Run no aplicativo de desktop robô.
- Depois de 40 min, a corrida fará uma pausa. Cubra as 384 placas de poço preenchidas com vedações de papel alumínio enquanto elas permanecem no robô e pressione para baixo com o rolo para garantir a adesão. Rotule a borda de cada placa com um identificador de lote.
- Coloque seis novas placas vazias de 384 poços nos decks 1-6 e retome o protocolo de funcionamento clicando em Retomar Run. Após a execução ser concluída, cubra o conjunto final de 384 placas com selos de papel alumínio.
- Pipeta 2 μL da mistura principal restante em colunas 1-3 e 22-24 de uma placa vazia de 384 poços para controle de qualidade em lote. Vedação com uma vedação opticamente clara e executada no termociclador (seção 10.1-10.2). Se algum poço tiver valores de ciclo de limiar N1 (Ct) ou se mais de 10 poços tiverem valores P1 Ct, o lote está contaminado e não pode ser utilizado.
- Armazene as placas de mixagem mestre preparadas a 4 °C e use-as dentro de 7 dias após a preparação.
5. Coleta de amostras, ingestão e tratamento térmico
- Instrua os participantes a evitar comer, beber, fumar ou conduzir a higiene dental 30 minutos antes da coleta de saliva. Instrua os participantes a coletar pelo menos 1 mL de saliva que naturalmente se agrupa na boca e depositá-la em um tubo cônico estéril de 50 mL sem conservantes, em seguida, tampar o tubo (Arquivo Suplementar 3).
- Descontaminar o exterior dos tubos de coleta de saliva com 70% de etanol ou desinfetar lenços umedecidos e transferi-los para o laboratório para testes.
- Registo a chegada da amostra escaneando cada código de barras da amostra na planilha de admissão diária (Arquivo Suplementar 4).
- Trate as amostras escaneadas por 30 minutos em um forno de 95 °C. Remova amostras usando luvas resistentes ao calor.
NOTA: As amostras não tratadas são estáveis à temperatura ambiente (23 °C) por até 72 h. Uma vez tratadas pelo calor, as amostras devem ser armazenadas a 4 °C, se não forem processadas imediatamente.
6. Atribuição da amostra
- Abra as planilhas diárias de carregamento de amostras para cada robô de carregamento de amostras (Arquivo Suplementar 5) no computador na estação de atribuição de amostras.
- Atribuir 188 amostras a cada placa de 384 poços da seguinte forma: Amostras 1-48 são consideradas no quarto trimestre, amostras 49-96 são consideradas quarto 2, amostras 97-144 são consideradas 3º trimestre e amostras 145-188 são consideradas quarto 4. A amostra é analisada em duplicata.
- Rotular bandejas com nome da placa, data e número do trimestre. Escaneie as amostras para entrar na planilha de carregamento da amostra.
NOTA: As amostras das placas anteriores podem precisar ser executadas manualmente, conforme definido na Figura 3. Consulte as seções 8.1-8.3 para atribuição manual da amostra e instruções de carregamento.
7. Robôs de carregamento de amostras operacionais
- Na estação de carregamento de amostras, forque dois conjuntos completos de oito racks impressos em 3D correspondentes à colocação do convés no robô.
- Uncap quarter 1 tubos e coloque em racks impressos em 3D, começando com a posição A1 no rack 1. Encha cada rack da esquerda para a direita e de cima para baixo. Continue este padrão de carregamento no rack 2 e, em seguida, prossiga para racks 4 e 5 (consulte a Figura 2C).
NOTA: Os racks não são numerados consecutivamente devido aos parâmetros do programa robô. - Coloque racks de amostra carregados no 1, 2, 4, 5, 7, 8, 10, 11. Coloque dicas P20 nos decks 3 e 9. Para simplificar o processo de configuração, carregue materiais de trás para frente no robô.
- Pegue uma placa de mistura mestre pré-feita a partir dos 4 °C, rotule-a com o nome da placa e use uma lâmina afiada para cortar uma linha na folha ao redor dos poços de controle (N23/24, O23/24 e P23/24).
- Coloque a placa de mistura mestre no deck 6 e retire a tampa da folha, deixando para trás o pequeno retângulo cobrindo os poços de controle. Descubra as caixas de ponta e feche o robô.
- Inicialize o protocolo operacional Python personalizado clicando em Iniciar Run através do aplicativo de desktop robô. Cada trimestre leva 24,5 min para carregar na placa; definir um temporizador como um lembrete.
- Enquanto o robô está em execução, tubos de amostra de uncap e load quarter 2 no segundo conjunto de racks impressos em 3D, conforme descrito na seção 7.2.
- Quando o robô pausar, remova os racks do quarto 1 e substitua-os por racks de trimestre 2. Clique em Retomar o executa no aplicativo de desktop.
- Recapitulando tubos de amostra do trimestre 1 e armazene-os em uma geladeira de 4 °C enquanto aguarda resultados.
Repita este processo de carregamento para os trimestres 3 e 4. - Transfira a placa carregada para um armário de biossegurança. Para minimizar a contaminação, mantenha a placa coberta durante a transferência.
8. Carregamento manual de amostras
NOTA: Realize uma única execução manual em amostras repetidas (N1 Rerun ou Rerun, consulte Figura 3) em caso de carregamento robótico inadequado.
- Reúna quaisquer amostras repetidas e atribua-as como as últimas amostras no quarto trimestre (ver seção 6.2). Amostras numé somadas, digitalizam os códigos de barras e digitam o local original da amostra e resultam na planilha de carregamento da amostra.
- Transfira amostras repetidas para o armário de biossegurança. Não carregue tubos de amostra repetidos nos racks de carregamento do robô.
- Pipeta 2 μL de cada amostra repetida para os poços corretos após o diagrama de layout da placa (Arquivo Suplementar 6). Use uma pipeta designada para adicionar amostras de pacientes. Mantenha os poços de controle cobertos com papel alumínio, adicionando amostras para minimizar a contaminação.
9. Adição de controles às placas de teste
- Retire a tampa da folha sobre os poços de controle usando fórceps.
- Pipeta 2 μL de água sem nuclease (sem controle de modelo) para poços N23-N24 e 2 μL de controle positivo misto de 200 cpμ (consulte a seção 3) aos poços O23-O24. Pipeta 2 μL de um controle amostral positivo confirmado em poços M23-M24 como um controle adicional. Deixe os poços P23-P24 vazios para monitorar a qualidade do lote master mix.
- Cubra a placa com uma vedação opticamente clara e use o rolo aplicador para aderir ao selo a todos os poços. Vórtice a placa a 2500 rpm por 30 segundos para misturar bem. Centrifugar a placa a 500 x g por 1 min.
10. Executando RT-qPCR
- Crie um programa de protocolo no software termocicr de acordo com as condições descritas (Tabela 3). Guarde o protocolo para placas futuras. Coloque a placa selada no termociclador e execute o protocolo.
- Exportar valores ct como um arquivo .xslx e copiar os valores na planilha de carregamento de amostra (Arquivo Suplementar 5).
NOTA: Estas folhas foram projetadas sob medida para arquivos de saída ct do software do fabricante e podem precisar de modificações para aceitar outros formatos.
11. Determinando a validade da placa
- Validar tanto o controle positivo quanto/ou amostras positivas conhecidas e o controle negativo para considerar os resultados da placa como válidos. Avalie os poços de controle com os seguintes critérios.
- Para um controle positivo, verifique se pelo menos um poço de controle positivo (O23/O24) produz valores ct entre 22-28 para as sondas P1 e N1. Alternativamente, os conhecidos poços de amostra positiva (M23/M24) produzem valores P1 e N1 Ct <33 nas sondas P1 e N1.
- Para controle negativo, verifique se não há valores de N1 ou P1 Ct em nenhum dos dois poços de controle negativo (N23/N24). Confirme se os valores da TC têm curvas de amplificação válidas antes de invalidar a placa.
12. Interpretação dos resultados amostrais
- Determine o resultado do paciente após o diagrama (Figura 3) e informe amostras resolvidas.
- Avalie o resultado P1 como VÁLIDO ou INVÁLIDO. Se a P1 produzir um resultado de Ct <33, considere o bem VÁLIDO e prossiga para o resultado de N1. Se p1 produzir um resultado de Ct >=33 ou nenhum valor de TC, considere o bem INVÁLIDO.
- Avalie o Resultado N1 como SIM, NÃO ou NÃO*. Se n1 produz um resultado de Ct <33, o poço é SIM. Se n1 não produzir um valor ct, o poço é NÃO. Se o N1 produzir um Ct >=33, o poço é NO*. Confirme se todos os valores do N1 Ct estão associados a uma curva de amplificação real. Se um valor ct para N1 não tem curva de amplificação, o poço é NÃO.
- Identifique amostras repetidas (N1 Rerun ou Rerun), rotule-as com um número de amostra interna e tipo de amostra e devolva-as ao fluxo de trabalho de carregamento (seção 8.1-8.3).
- Avalie o resultado P1 como VÁLIDO ou INVÁLIDO. Se a P1 produzir um resultado de Ct <33, considere o bem VÁLIDO e prossiga para o resultado de N1. Se p1 produzir um resultado de Ct >=33 ou nenhum valor de TC, considere o bem INVÁLIDO.
13. Limpeza laboratorial
- Robôs de manuseio líquido
- Limpe todos os lados com desinfetante de nível intermediário. Não use etanol, pois vai degradar o plástico.
- Limpe suavemente a ponta da pipeta e a lixeira com um álcool (70% de etanol ou 100% isopropanol) limpe. Limpe o teclado e o mouse.
- Armário de biossegurança
- Limpe todas as superfícies com desinfetante de nível intermediário. Ligue a luz UV por 15 minutos.
Resultados
Determinamos o intervalo de detecção de sondas RT-qPCR e primers para conteúdo de ácido nucleico sintético tanto para SARS-CoV-2 (N1) quanto para Hs_RPP30 (P1). Uma diluição serial de 10 vezes de concentrações conhecidas de RNA sars-cov-2 sintético combinado e DNA sintético Hs_RPP30 na água foi feita. A fórmula a seguir foi usada para converter peso molecular em número de cópia genética
Número de cópia genética = (ng * 6.0221 x 1023)/((comprimento em pares de base* 660 g/mole) *1 x 109 ng/g)
e o RT-qPCR foi realizado. Após a realização do RT-qPCR, as curvas lineares para detecção de N1 (Figura 4A) e P1 (Figura 4B) apresentaram bons coeficientes de correlação em uma ampla gama de concentrações de cópia genética (R2= 0,9975 e R2= 0,9884, respectivamente). Este resultado indica que a combinação de conjuntos de primer e sonda não é inibitória e pode detectar com precisão SARS-CoV-2 RNA em uma cópia genética/μL (Cq=33). Uma cópia genética é aproximadamente equivalente a uma cópia viral; no entanto, não determinamos números quantitativos de cópias virais na saliva devido à natureza semi-quantitativa do RT-qPCR. Tentamos simular amostras positivas de saliva, espiando o RNA sars-cov-2 sintético de concentrações conhecidas em saliva livre de vírus (tratadas pelo calor e não tratadas pelo calor), mas não foram capazes de produzir amplificação N1 em baixas concentrações de RNA (Dados não mostrados). Isso pode ser devido à degradação do RNase ou outros fatores de confusão.
Também foi avaliada a variabilidade entre métodos automatizados e manuais de carregamento de amostras. Para avaliar a variabilidade entre ensaios, foram carregadas 20 amostras positivas únicas utilizando-se os métodos manuais (descritos na seção 8.1-8.3) e automatizados (descritos na seção 7.1-7.11). Os valores de N1 Ct foram comparados para determinar se robôs de manuseio líquido e carregamento manual de amostras produziram resultados equivalentes (Figura 5A). A relação linear entre métodos manuais e automatizados produziu um alto coeficiente de correlação (R2= 0,9088), indicando que ambos os métodos são funcionalmente equivalentes. À medida que os valores do CT N1 aumentaram, a variabilidade dos valores do CT também aumentou. Essa tendência é provavelmente devido à distribuição heterogênea de partículas virais dentro da saliva, que é mais pronunciada quando menos partículas estão presentes. Para avaliar a variabilidade intra-ensaio, foi feita uma comparação entre os valores do N1 Ct a partir de poços de replicação de amostras de saliva únicas utilizando ambos os métodos de carregamento amostral (Figura 5B). A relação linear entre as réplicas do carregamento automatizado de amostras (R2= 0,9622) produziu um coeficiente de correlação ligeiramente maior do que o do carregamento manual (R2= 0,9589), indicando alta reprodutibilidade da detecção sars-cov-2 para ambos os métodos de carregamento.
Por fim, foi feita uma avaliação da redução da viscosidade da saliva em relação aos métodos de tratamento térmico (Figura 6). A saliva foi obtida a partir de uma única fonte para eliminar a variabilidade da amostra. Maior variabilidade nos valores de P1 Ct dentro de um método de tratamento térmico pode ser indicativo de maior viscosidade amostral, pois a saliva viscosa não pode ser aspirada e dispensada com precisão. Ambos os métodos de tratamento térmico de 30 minutos e 60 min produziram uma redução significativa da variabilidade amostral quando comparados ao não controle de tratamento (p = 0,0006 e p = 0,0429, respectivamente). Não houve diferença significativa entre tratamentos de 30 min e 60 min (p = 0,2245); por isso, implementou-se o método de tratamento térmico de 30 minutos para reduzir o tempo de processamento.
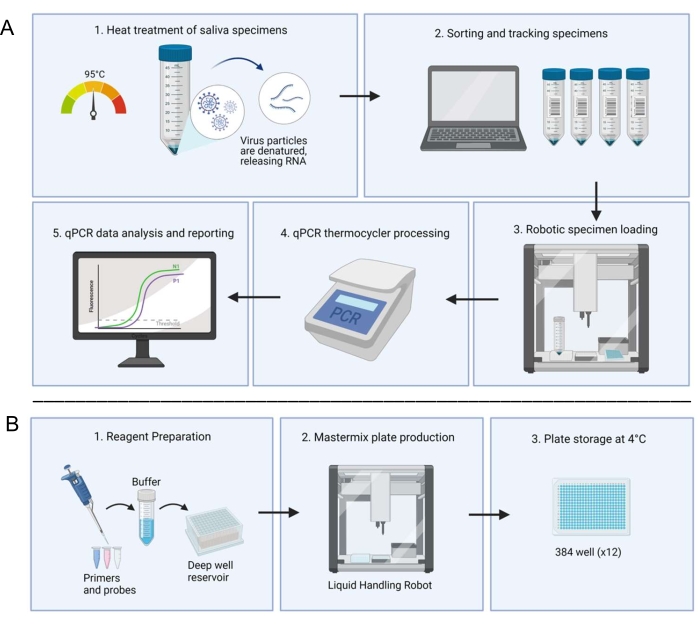
Figura 1: Fluxo de trabalho laboratorial utilizando o sistema de diagnóstico RT-qPCR baseado em saliva. (A) As amostras são coletadas e tratadas a calor a 95 °C por 30 minutos. As amostras tratadas são classificadas e rastreadas com informações do paciente através de um sistema interno de planilhas. Um robô de manuseio líquido carrega amostras em poços duplicados de placas de mistura mestre preparadas. Um técnico carrega manualmente os controles, sela a placa e coloca a placa em um termociclador para processamento. Os resultados são analisados através de um sistema de computador automatizado e verificados por um técnico. (B) Um técnico prepara reagentes para a mistura mestre que são adicionados a um reservatório de poços profundos em um armário de biossegurança estéril. Os reservatórios de poços profundos cheios são carregados em um robô de manuseio líquido dedicado. As placas completas são seladas com papel alumínio, rotuladas e armazenadas a 4 °C. Clique aqui para ver uma versão maior desta figura.
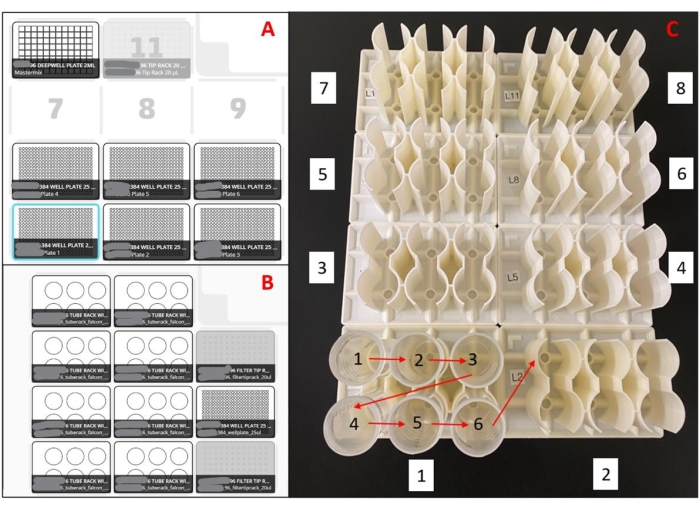
Figura 2: Layouts usados para o robô de manuseio líquido. (A) Layout de deck para robô de preparação de placas de mixagem mestre(s). Com uma pipeta de oito canais, o robô é programado para pegar pontas de pipeta, aspirar mistura mestre de um reservatório de poços de 96 poços profundos, dispensar mistura mestre em placas vazias de 384 poços e ejetar as pontas da pipeta em uma lixeira. Isso é repetido para seis placas por corrida. (B) Configuração do deck para robôs de carregamento de amostras. Com uma pipeta de canal único, o robô é programado para pegar uma ponta de pipeta, aspirar uma amostra de saliva, distribuir uma amostra de saliva em poços duplicados de uma placa de mistura mestre de 384 poços e ejetar a ponta pipeta em uma lixeira. Isso é repetido para 48 amostras por execução. (C) Ordem de carregamento do tubo de amostra para racks impressos em 3D. As setas vermelhas indicam a ordem de carregamento dentro de um rack, e os números brancos encaixotados indicam a ordem de carregamento de todo o conjunto de racks. Toda a configuração carregará 188 amostras em duplicata em uma placa de 384 poços. Clique aqui para ver uma versão maior desta figura.
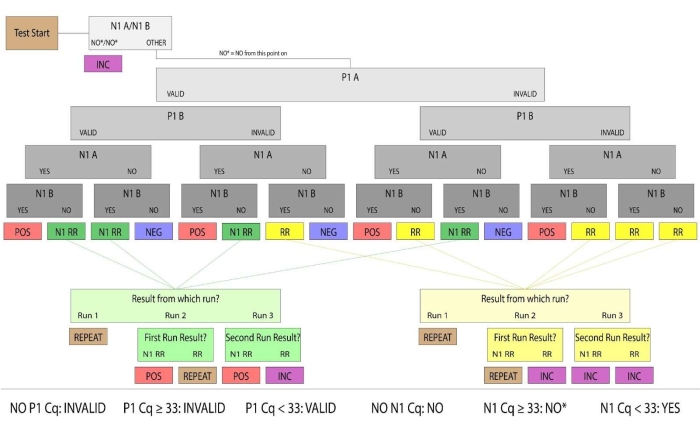
Figura 3: Fluxograma resultante da amostra. Amostras com P1 válido e N1 positivo foram determinadas como amostras de saliva humana positivas para SARS-CoV-2. Os resultados amostrais válidos e positivos/negativos foram considerados conclusivos. Amostras que não produziram resultados conclusivos no primeiro turno foram categorizadas como Rerun (denotada RR) ou N1 Rerun (denotada N1 RR). As amostras de reexeção não apresentaram amplificação P1 válida, e as amostras de reprise N1 apresentaram amplificação N1 positiva em uma única réplica. Se nenhuma amplificação P1 válida pudesse ser produzida por uma execução manual subsequente, ou ambas as réplicas tivessem valores de TC N1 acima do limiar positivo (Ct >33), os resultados da amostra foram considerados inconclusivos. Para fins clínicos, amostras de pacientes que não chegaram ao laboratório, apresentaram quantidade insuficiente de saliva para pipeta ou foram danificadas foram consideradas inválidas. Clique aqui para ver uma versão maior desta figura.
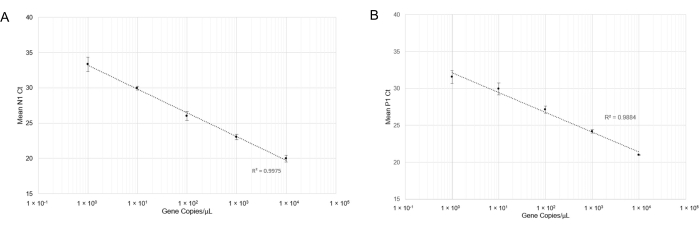
Figura 4: Detecção rt-qPCR de N1 (SARS-CoV-2) DNA sintético e DNA sintético P1 (Hs_RPP 30). As curvas padrão foram traçadas com desvios padrão para determinar o alcance da detecção precisa usando esta combinação de sonda/primer. (A) Os valores médios do TC (n =4) obtidos em respectivas diluições foram plotados contra a quantidade estimada de RNA sintético (1x100 a 1x104 cópias de RNA em 10 μL de reação RT-qPCR). (B) Os valores médios do TC (n =3) obtidos em respectivas diluições foram traçados contra a quantidade estimada de DNA sintético (1 x 100 a 1 x 104 cópias genéticas em 10 μL de reação RT-qPCR). Clique aqui para ver uma versão maior desta figura.
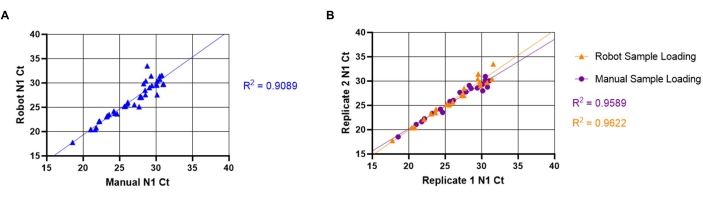
Figura 5: Comparação entre os valores de tomografia sars-cov-2 (N1) de transferência manual e automatizada de saliva. As conhecidas amostras de saliva positiva SARS-CoV-2 (n =20) foram carregadas em duplicata em uma placa de mistura mestre RT-qPCR por um robô de manuseio líquido. As amostras têm um valor de Tomografia que varia de 18 a 32 para N1. As mesmas amostras foram então carregadas manualmente em poços duplicados em uma localização de placa diferente. (A) Os valores de N1 Ct obtidos a partir de amostras únicas usando o robô e o carregamento manual de amostras foram transpostos para determinar a variabilidade entre o carregamento manual e o robô. (B) A variabilidade intra-assay também foi determinada pelo uso da réplica transposta dos valores de N1 Ct obtidos tanto do robô quanto do carregamento manual da amostra. Clique aqui para ver uma versão maior desta figura.
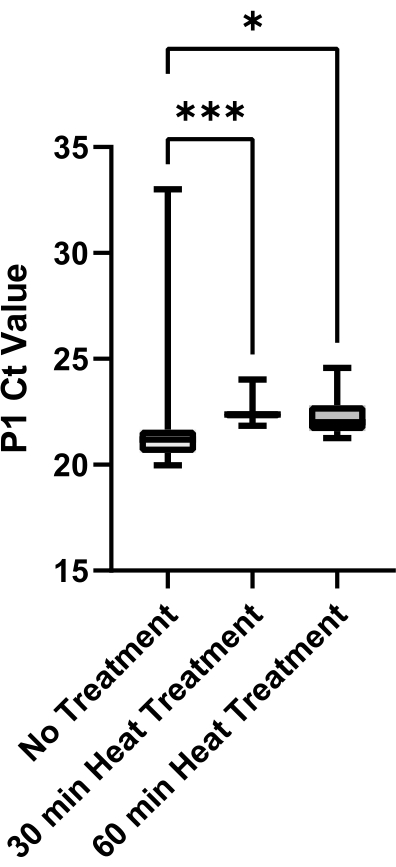
Figura 6: Avaliação dos métodos de tratamento térmico para redução da viscosidade na saliva. A saliva negativa SARS-CoV-2 foi coletada de uma única fonte e as alíquotas foram tratadas pelo calor por 0 min, 30 min ou 60 min a 95 °C. Os valores p1 ct de réplicas técnicas (n =12) de cada condição foram traçados para determinar a variabilidade entre os métodos de tratamento. Comparações parientas entre os grupos foram avaliadas com um teste t não realizado (*** indica p <0.001, * indica p <0,05). Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Comparação de N1 Ct em amostras baixas de saliva P1 Ct. Foram selecionadas as amostras positivas com P1 Ct baixo e comparadas com o N1 Ct (n =106). Os valores do N1 Ct variaram de 14 a 33, indicando que o ensaio tem uma faixa dinâmica em amostras de saliva comparáveis à curva padrão. Clique aqui para baixar este Arquivo.
| Componente | Sequência (5'→3') | Concentração de Estoque | Volume | ||
| Sonda 2019-nCoV-N1 | /5FAM/ACCCCGCAT/ZEN/TACGTTTGGTGGACC/3IABkFQ | 50 μM | 500 μL | ||
| 2019-nCoV-N1-For | GACCCCAAAAATCAGCGAAAT | 100 μM | 2000 μL | ||
| 2019-nCoV-N1-Rev | TCTGGTTACTGCCAGTTGAATCTG | 100 μM | 2000 μL | ||
| Sonda Hs RPP30 Cy5 | /5Cy5/TTCTGACCT/ZEN/GAAGGCTCTGCGCG/3IABkFQ | 50 μM | 500 μL | ||
| Hs-RPP30-Para | AGATTTGGACCTGCGAGCG | 100 μM | 2000 μL | ||
| Hs-RPP30-Rev | GAGCGGCTGTCTCCACAAGT | 100 μM | 2000 μL | ||
| Água | - | - | 11000 μL | ||
Tabela 1: Componentes da mistura de sonda/primer N1+P1.
| Componente | Concentração de Estoque | Volume por reação | Concentração Final | Volume de lote | ||
| Luna WarmStart RT Enzima Mix | 20X | 0,5 μL | 1X | 3 mL | ||
| Mistura de reação do buffer de Luna | 2X | 5,0 μL | 1X | 30 mL | ||
| Mistura de primer/sonda N1+P1 | nCoV N1 F: 10 μM | 0,5 μL | 500 nM | 3 mL | ||
| nCoV N1 R: 10 μM | 500 nM | |||||
| Sonda nCoV N1: 2,5 μM | 125 nM | |||||
| RPP_30 P1 F: 10 μM | 500 nM | |||||
| RPP_30 P1 R: 10 μM | 500 nM | |||||
| Sonda RPP_30 P1: 2,5 μM | 125 nM | |||||
| Água livre de nuclease | --- | 2 μL | --- | 12 mL | ||
| Subtotal | --- | 8 μL | --- | 48 mL | ||
| Modelo | 2 μL | |||||
Tabela 2: Componentes do multiplex SARS-CoV-2 master mix.
| Palco | Temperatura (°C) | Duração | Número de ciclos |
| Transcrição reversa | 55 | 10 min. | 1 |
| Denaturação Inicial | 95 | 1 min. | 1 |
| Touchdown | 95 | 10 segundos | 3 |
| 72 | 30 segundos | ||
| 95 | 10 segundos | 3 | |
| 69 | 30 segundos | ||
| 95 | 10 segundos | 3 | |
| 66 | 30 segundos | ||
| Amplificação principal | 95 | 10 segundos | 40 |
| 65 | 30 segundos |
Tabela 3: Protocolo de TOUCHDOWN RT-qPCR. Condições de termociclamento para um passo rt-qPCR SARS-CoV-2 ensaio diagnóstico.
| Passo de touchdown | Sem passo de touchdown | |||
| Média N1 Ct | P1 Médio CT | Média N1 Ct | P1 Médio CT | |
| Amostra 1 | 19.65 | 22.7 | 27.8 | 28.3 |
| Amostra 2 | 22.24 | 24.9 | 28.77 | 30.5 |
| Amostra 3 | 18.85 | 19.2 | 24.65 | 25.9 |
| Amostra 4 | 25.56 | 22.8 | 31.93 | 29.2 |
| Amostra 5 | 22.34 | 24.8 | 38.48 | 40.0 (Detecção de falha) |
Tabela 4: Comparação dos valores do touchdown para cinco amostras positivas contra valores de touchdown ct.
| Amostra | TigerSaliva | Ensaio SARS-CoV-2 à base de saliva comercialmente disponível | ||
| N1 Ct | P1 Ct | Valor Covid-19 | Valor RNaseP | |
| D11 | 16.4 | 18.1 | 20.86 | 23.4 |
| E11 | 18.9 | 19.1 | 25.6 | 21.2 |
| F11 | 19.5 | 18.4 | 22.8 | 22.2 |
| G11 | 22.2 | 19.1 | 23.7 | 22.9 |
| H11 | 26.4 | 21.3 | 32.2 | 26.7 |
| A12 | 14.8 | 16.5 | 29.15 | 19 |
| B12 | 24 | 19.6 | 31.05 | 21.35 |
| C12 | 14.9 | 17.5 | 20.84 | 18.9 |
Tabela 5: Comparação dos resultados do TigerSaliva Ct e resultados de ensaio sars-cov-2 disponíveis comercialmente. Ambos os ensaios foram realizados nas mesmas amostras de saliva (n =8).
Arquivo Suplementar 1: Script personalizado para criação de placa de mixagem mestre robô. Por favor, clique aqui para baixar este Arquivo.
Arquivo Suplementar 2: Script personalizado para processamento de saliva em robôs de carregamento de amostras. Por favor, clique aqui para baixar este Arquivo.
Arquivo Suplementar 3: Instruções para auto-coleta de amostras de saliva de alta qualidade dos participantes. Mais detalhes podem ser encontrados na breve descrição do vídeo do processo de teste disponível em https://www.clemson.edu/centers-institutes/reddilab/index.html. Clique aqui para baixar este Arquivo.
Arquivo suplementar 4: Folha de entrada de amostra. Clique aqui para baixar este Arquivo.
Arquivo suplementar 5: Folha de carregamento de amostras. Clique aqui para baixar este Arquivo.
Arquivo suplementar 6: Prove o diagrama de layout da placa de 384 poços. Clique aqui para baixar este Arquivo.
Discussão
O ensaio descrito no protocolo foi avaliado por um estudo de validação independente. Verificou-se que o ensaio tinha especificidade de 98,9% (1,1% falso positivo) e sensibilidade de 90,0% (10,0% falso negativo) quando avaliados contra cotonetes nasofaríngeos emparelhados tomados ao mesmo tempo (n =837; 817 negativos, 20 positivos). É importante ressaltar que três participantes que testaram positivo com TigerSaliva e negativo com cotonete nasofaríngeo foram retestados com cotonetes 48 h depois e retornaram resultados positivos, indicando que o TigerSaliva pode ser capaz de detectar infecções pelo SARS-CoV-2 mais cedo no curso da doença.
Simulamos amostras positivas de saliva por meio da saliva livre de vírus (tratada com calor e não tratada pelo calor) com concentrações conhecidas de RNA sintético SARS-CoV-2 e realizamos uma diluição de 10 vezes para determinar o limite de tomografia computadorizada na saliva. O gene N1 não foi detectável abaixo de 10.000 cópias genéticas (aproximadamente Ct = 28) em amostras simuladas positivas. Suspeitamos que isso seja devido à degradação da RNase ou outros fatores de confusão. No entanto, a interação de RNases saliva com RNA sintético nu é provavelmente diferente da interação com partículas virais, mesmo depois de terem sido desnaturadas pelo calor. Amostras positivas de saliva foram identificadas com o Ct >30 e laboratórios externos obtiveram dados de sequência genética SARS-CoV-2 dessas amostras. Especulamos que as proteínas virais fornecem proteção contra a degradação do RNA em amostras de saliva de pacientes.
O passo mais crítico no protocolo é a implementação da automação para preparação de mix master e processamento de amostras de saliva (seções 4 e 7, respectivamente). Isso permite a sobreposição de processos de tarefas, o que reduz drasticamente o tempo de retorno. Outro passo crítico é a interpretação do resultado clínico (seções 11 e 12). A criação de categorias intermediárias de resultados (Reprise e Reprise N1) também minimizou a ocorrência de resultados inconclusivos.
Demonstramos que a variação entre métodos manuais e automatizados de carregamento de amostras de saliva é insignificante (Figura 5A) e que a automação pode melhorar a reprodutibilidade da detecção sars-cov-2 (Figura 5B). A automação deve ser favorecida para facilitar os testes ao projetar e expandir laboratórios clínicos25. O fluxo de trabalho de laboratório é melhorado com a implementação de tarefas automatizadas por robôs26. Os recursos de código aberto dos robôs de manuseio líquido permitem a implementação de scripting personalizado para design de protocolo. Isso faz dos robôs de manuseio líquido um sistema barato e altamente modificável em comparação com os métodos tradicionais de automação clínica. É também uma estratégia ideal para executar tarefas laboratoriais altamente repetitivas. O alto nível de personalização do sistema traduz-se na liberdade de alterar labware (por exemplo, tubos de coleta, pontas de pipeta ou placas de 384 poços) em caso de escassez. Portanto, a automação usando robôs de manuseio líquido é viável tanto para vigilância e pesquisa em larga escala quanto em pequena escala.
Uma grande vantagem dessa estratégia de teste é um tempo de reviravolta muito menor em relação a outros laboratórios clínicos. A utilização de robôs automatizados de manuseio de líquidos desempenha um papel fundamental na redução do tempo de retorno, mas o uso simultâneo de robôs e termociclos também é fundamental para maximizar a eficiência dos testes. Um robô e um termociclador devem ser operados como um par, onde ambas as máquinas são usadas em conjunto para carregamento ininterrupto de amostras e análise de resultados de amostras. Uma vez estabelecido um fluxo constante de amostras atribuídas, todos os pares de máquinas podem ser operados simultaneamente. O uso constante e simultâneo de robôs e termociclistas aumenta drasticamente a capacidade e a eficiência dos testes, o que é crucial para acomodar o alto volume de testes.
Em contraste com outros protocolos SARS-CoV-2 RT-qPCR estabelecidos, incluímos um passo de touchdown no protocolo termociclador para melhorar o ressarcimento dos conjuntos de sonda e primer para os genes-alvo27, reduzindo o risco de amplificação falha. Os resultados demonstraram que o touchdown melhorou a detecção de amostras positivas sem correr o risco de perda de ligação específica de primer (Tabela 4). Determinamos que uma ampla gama de cópias de RNA SARS-CoV-2 (Figura 4A) e Hs_RPP30 cópias de DNA (Figura 4B) podem ser detectadas simultaneamente pelo ensaio RT-qPCR.
Uma limitação dos robôs de manuseio líquido é a possibilidade de contaminação cruzada de amostras positivas durante a transferência de saliva. A saliva é um fluido viscoelástico28 e pode amarrar poços adjacentes após ser dispensado da ponta da pipeta. Além disso, a heterogeneidade da saliva29 pode causar distribuição desigual de partículas virais ao longo da amostra. Isso aumenta a possibilidade de falsos positivos e negativos, exigindo a designação de amostras de N1 Rerun e Rerun. No entanto, 14,1% das amostras inicialmente designadas como N1 Rerun foram resolvidas como positivas para SARS-CoV-2 e eram mais de 30 vezes mais propensas do que as amostras de rerun para resolver como positivas após o novo teste. Consequentemente, diferenciar a reprise de N1 (Figura 3) permitiu uma separação mais precisa de amostras potencialmente positivas, aumentando a sensibilidade e a especificidade do nosso ensaio diagnóstico. Outros parâmetros resultantes para o teste de saliva diagnóstica não fizeram essa distinção12,14,24,30,31.
Amostras de saliva podem ser difíceis de pipeta devido à heterogeneidade e viscosidade32. O tratamento térmico desnatura adequadamente as proteínas na biomatrix saliva, reduzindo a viscosidade e eliminando a necessidade de reagentes de extração de RNA9, que eram escassos durante os estágios iniciais da pandemia10. O tratamento térmico prolongado também inativa os vírus33 que permitem o processamento laboratorial em níveis mais baixos de biossegurança. Consequentemente, foi implementada uma extração de RNA baseada no calor (descrita na seção 5.4) para diminuir a viscosidade por meio da desinaturação da proteína (Figura 6). Com base nos resultados, postulamos que o tratamento térmico também pode homogeneizar amostras de saliva, além de desnaturar a biomatrix proteica. Outros grupos combinaram tratamento térmico e proteinase K para aumentar a homogeneidade9,14,34. Optamos por não implementar esta etapa, pois pode desnaturar proteínas virion a uma taxa que deixa o RNA viral exposto à degradação do calor35. Além disso, a diluição amostral com proteinase K pode mascarar amostras positivas contendo menos partículas virais, diminuindo assim a sensibilidade. Além disso, os resultados do ensaio foram comparados a um ensaio SARS-CoV-2 baseado em saliva comercialmente disponível (Logix Smart COVID-19) que usa extração de RNA de contas magnéticas (Tabela 5). Verificou-se que o ensaio atual foi mais adequado para detectar amostras positivas fracas em comparação com o ensaio comercialmente disponível.
É difícil quantificar o número de cópia do vírus na saliva usando apenas RT-qPCR, porque o qPCR é semi-quantitativo. Há variação inerente entre os valores da TC que se origina de limitações técnicas. O número da cópia genética pode ser determinado a partir de valores ct (Figura 4) e é aproximadamente equivalente ao número de cópia viral. Uma solução possível para determinar o número de cópia viral em amostras de saliva é o ddPCR, que fornece quantificação dura de cópias genéticas na reação. No entanto, acreditamos que é adequado fornecer resultados qualitativos aos médicos e o conteúdo relativo viral pode ser comparado entre amostras processadas com nossos métodos.
Apesar de algumas limitações que surgem ao usar saliva, o ensaio SARS-CoV-2 por RT-qPCR baseado em saliva prova ser um método eficaz para detecção rápida e confiável de RNA viral em qualquer escala de testes. Isso é especialmente verdade quando juntamente com a utilização de sistemas de manuseio líquido de código aberto. Essa abordagem de teste pode ser modificada para detectar outras sequências de ácido nucleico relevantes para diagnósticos, como agentes de doenças infecciosas, marcadores de doenças ou outros vírus. Isso torna o ensaio aplicável tanto para os esforços clínicos quanto de diagnóstico de pesquisa.
Divulgações
Os autores não têm nada a revelar. O ensaio descrito no protocolo está no SalivaDirect EUA arquivado pela Yale School of Public Health.
Agradecimentos
Os autores agradecem à administração, equipe médica e funcionários do laboratório clínico da Clemson no Reddi Lab, que ajudaram a implementar e gerenciar os testes sars-cov-2. Agradecemos ao Dr. Phillip Buckhaults e à Dra. Agradecemos a muitos alunos, professores e funcionários pela assistência na coleta de amostras. Obrigado aos alunos do Creative Inquiry pela coleta de dados de curva padrão. O financiamento deste estudo foi recebido do subsídio dos Institutos Nacionais de Saúde P20GM121342 (concedido à DD e à LGP), ao Departamento Atlético de Clemson, ao Vice-Presidente de Pesquisa da Universidade Clemson e ao Comitê de Revisão de Títulos Conjuntos da Carolina do Sul.
Materiais
| Name | Company | Catalog Number | Comments |
| 100% EtOH | Fisher scientific | 22-032-601 | |
| 20 uL Filtered Pipette Tips | Opentrons | 20uL tips | |
| 2mL Microcentrifuge Tubes | Fisher Scientific | 14-666-313 | Alternate product may be used |
| Armadillo PCR Plate, 384-well, clear, white wells | Thermo Scientific | AB3384 | Alternate product may be used |
| Celltreat 2mL 96 Deep Well Plates | Fisher Scientific | 50-828-743 | For mastermix preparation |
| Clear PCR Sealing Sheets | Thermo Scientific | AB0558 | Alternate product may be used |
| DPEC Treated Water | Ambion (Thermo Scientific) | AM9916 | |
| Flip Cap 50 mL Conical Tubes | VWR | 75845-210 | For sample collection |
| Foil PCR Sealing Sheets | Thermo Scientific | AB0626 | For storage of mastermix plates, Alternate product may be used |
| HS_RPP30 Synthetic DNA | Integrated DNA Technologies | 299788131 | P1 positive control |
| Luna Buffer Probe One-Step Reaction | New England Biolabs | M3006B | |
| Luna WarmStart RT Enzyme Mix | New England Biolabs | M3002B | |
| nCOV_N1 Forward Primer, 100 nmol | Integrated DNA Technologies | 10006830 | |
| nCOV_N1 Probe Aliquot, 50 nmol | Integrated DNA Technologies | 10006832 | Probe can be synthesized by other vendors with SYBR or FAM fluophores |
| nCOV_N1 Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006831 | |
| Opentron HEPA Filter Module | Opentrons | N/A | Not required, but useful to reduce contamination |
| Opentron Multichannel Attachment, P20 | Opentrons | 999-00005 | For mastermix preparation |
| Opentron OT-2 Liquid Handling Robot | Opentrons | OT-2 | |
| Opentron Pipette Attachment, P20 | Opentrons | 999-0000215 | For sample loading |
| Oven | Memmert | UF450 PLUS 208V-3PH | |
| PCR Tubes (rnase, dnase free) | Fisher Scientific | 14-230-225 | For aliquots of positive and neg controls |
| PolarSafe Aluminum Cooling Block, 15-Well (1.5/2.0 mL Tubes) | VWR | 10808-952 | |
| PolarSaf Aluminum Cooling Block, 24-Well (0.5mL tubes) | VWR | 10808-956 | |
| RNAse P (ATTO 647) Probe, 50 nmol | Integrated DNA Technologies | 10007062 | Probe can be synthesized by other vendors with Cy5 fluorophore |
| RNAse P Forward Primer, 100nmol | Integrated DNA Technologies | 10006836 | |
| RNAse P Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006837 | |
| Sars-CoV-2 Synthetic RNA Control 2 | Twist Biosciences | 102024 / 103907 / 103909 | N1 Positive Control |
| Scanners | Code | CR1500 | Only required when scaling up |
| Small HEPA Filtered Hood | Erlab | Captair Bio 321 | For mastermix preparation |
| Thermocycler CFX384 Touch | Biorad | CFX384 Touch | Alternate models can be used, e.g. CFX384 Opus |
| X-acto Knife Set | Staples | N/A | To cut foil for keeping control wells covered |
Referências
- Zhu, N., et al. A novel coronavirus from patients with pneumonia in China, 2019. New England Journal Medicine. 382 (8), 727-733 (2020).
- Chen, J. Pathogenicity and transmissibility of 2019-nCoV-A quick overview and comparison with other emerging viruses. Microbes and Infection. 22 (2), 69-71 (2020).
- Petersen, E., et al. Comparing SARS-CoV-2 with SARS-CoV and influenza pandemics. The Lancet. Infectious Diseases. 20 (9), 238-244 (2020).
- Honein, M. A., et al. Summary of Guidance for Public Health Strategies to Address High Levels of Community Transmission of SARS-CoV-2 and Related Deaths, December 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (49), 1860-1867 (2020).
- World Health Organization. Surveillance strategies for COVID-19 human infection: interim guidance. World Health Organization. , Available from: https://apps.who.int/iris/handle/10665/332051 (2020).
- Screening testing for early detection of SARS-CoV-2 infection. Division of viral diseases. Centers for Disease Control. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/hcp/tsting-overview.html#PublicHealthSurveillance (2020).
- Larremore, D. B., et al. Test sensitivity is secondary to frequency and turnaround time for COVID-19 screening. Science Advances. 7 (1), 5393(2021).
- Sethuraman, N., Jeremiah, S. S., Ryo, A. Interpreting Diagnostic Tests for SARS-CoV-2. JAMA. 323 (22), 2249-2251 (2020).
- Chu, A. W., et al. Evaluation of simple nucleic acid extraction methods for the detection of SARS-CoV-2 in nasopharyngeal and saliva specimens during global shortage of extraction kits. Journal of clinical virology: the official publication of the Pan American Society for Clinical Virology. 129, 104519(2020).
- Guan, D., et al. Global supply-chain effects of COVID-19 control measures. Nature Human Behaviour. 4 (6), 577-587 (2020).
- Bastos, M. L., Perlman-Arrow, S., Menzies, D., Campbell, J. R. The Sensitivity and Costs of Testing for SARS-CoV-2 Infection With Saliva Versus Nasopharyngeal Swabs: A Systematic Review and Meta-analysis. Annals of Internal Medicine. 174 (4), 501-510 (2021).
- Pasomsub, E., et al. Saliva sample as a non-invasive specimen for the diagnosis of coronavirus disease 2019: a cross-sectional study. Clinical Microbiology and Infection: The Official Publication of The European Society of Clinical Microbiology and Infectious Diseases. 27 (2), (2021).
- To, K. K., et al. Consistent Detection of 2019 Novel Coronavirus in Saliva. Clinical Infectious Diseases: An Official Publication of The Infectious Diseases Society of America. 71 (15), 841-843 (2020).
- Vogels, C. B., et al. SalivaDirect: A simplified and flexible platform to enhance SARS-CoV-2 testing capacity. Med (New York, N.Y.). 2 (3), 263-280 (2021).
- Griesemer, S. B., et al. Evaluation of Specimen Types and Saliva Stabilization Solutions for SARS-CoV-2 Testing. Journal of Clinical Microbiology. 59 (5), 1418-1420 (2021).
- Wehrhahn, M. C., et al. Self-collection: An appropriate alternative during the SARS-CoV-2 pandemic. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 128, 104417(2020).
- Barza, R., Patel, P., Sabatini, L., Singh, K. Use of a simplified sample processing step without RNA extraction for direct SARS-CoV-2 RT-PCR detection. Journal of Clinical Virology: The Official Publication of the Pan American Society for Clinical Virology. 132, 104587(2020).
- Paltiel, A. D., Zheng, A., Walensky, R. P. Assessment of SARS-CoV-2 Screening Strategies to Permit the Safe Reopening of College Campuses in the United States. JAMA Network Open. 3 (7), 2016818(2020).
- Centers for Disease Control. 2019-Novel Coronavirus (2019-nCoV) Real-Time RT-PCR Primers and Probes. U.S. Department of Health and Human Services. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/lab/rt-pcr-panel-primer-probes.html (2020).
- Cresswell, K., Ramalingam, S., Sheikh, A. Can Robots Improve Testing Capacity for SARS-CoV-2. Journal of Medical Internet Research. 22 (8), 20169(2020).
- Villanueva-Cañas, J. L., et al. Implementation of an open-source robotic platform for SARS-CoV-2 testing by real-time RT-PCR. PLoS One. 16 (7), 0252509(2021).
- Robot Boosts COVID-19 Testing Efficiency. University of South Carolina College of Pharmacy. , Available from: https://sc.edu/study/colleges_schools/pharmacy/about/news/2020/robot-boosts-testing-effort.php (2020).
- Matic, N., et al. Practical challenges to the clinical implementation of saliva for SARS-CoV-2 detection. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of The European Society of Clinical Microbiology. 40 (2), 447-450 (2021).
- Sahajpal, N. S., et al. SalivaSTAT: Direct-PCR and Pooling of Saliva Samples Collected in Healthcare and Community Setting for SARS-CoV-2 Mass Surveillance. Diagnostics. 11 (5), Basel, Switzerland. 904(2021).
- Genzen, J. R., et al. Challenges and Opportunities in Implementing Total Laboratory Automation. Clinical Chemistry. 64 (2), 259-264 (2018).
- Archetti, C., Montanelli, A., Finazzi, D., Caimi, L., Garrafa, E. Clinical Laboratory Automation: A Case Study. Journal of Public Health Research. 6 (1), 881(2017).
- Hecker, K. H., Roux, K. H. High and low annealing temperatures increase both specificity and yield in touchdown and stepdown PCR. BioTechniques. 20 (3), 478-485 (1996).
- Bhat, P. P., et al. Formation of beads-on-a-string structures during break-up of viscoelastic filaments. Nature Physics. 6, 625-631 (2010).
- Miller, C. S., et al. Current developments in salivary diagnostics. Biomarkers in Medicine. 4 (1), 171-189 (2010).
- Moreno-Contreras, J., et al. Saliva Sampling and Its Direct Lysis, an Excellent Option To Increase the Number of SARS-CoV-2 Diagnostic Tests in Settings with Supply Shortages. Journal of Clinical Microbiology. 58 (10), 01659(2020).
- Wang, W., et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA. 323 (18), 1843-1844 (2020).
- Landry, M. L., Criscuolo, J., Peaper, D. R. Challenges in use of saliva for detection of SARS CoV-2 RNA in symptomatic outpatients. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 130, 104567(2020).
- Lista, M. J., et al. Resilient SARS-CoV-2 diagnostics workflows including viral heat inactivation. PLoS One. 16 (9), 0256813(2021).
- Brotons, P., et al. Validation and implementation of a direct RT-qPCR method for rapid screening of SARS-CoV-2 infection by using non-invasive saliva samples. International Journal of Infectious Diseases: IJID: Official Publication of The International Society for Infectious Diseases. 110, 363-370 (2021).
- Batéjat, C., Grassin, Q., Manuguerra, J. C., Leclercr, I. Heat inactivation of the severe acute respiratory syndrome coronavirus 2. Journal of Biosafety and Biosecurity. 3 (1), 1-3 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados