このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
抗がんアプタマーの選択性と特異性を評価するためのフローサイトメトリーベースの細胞表面タンパク質結合アッセイ
要約
抗がんアプタマー開発に必要なステップは、標的への結合をテストすることです。この結合を研究するためのフローサイトメトリーベースのアッセイを実証し、ネガティブコントロールのアプタマーとその特定のタンパク質に対して陽性または陰性の癌細胞を含めることの重要性を強調しています。
要約
抗がんアプタマーの開発における重要な課題は、開発したアプタマーの標的タンパク質に対する選択性と特異性を効率的に決定することです。モノクローナル抗体に比べていくつかの利点があるため、アプタマーの開発は癌研究者の間で絶大な人気を得ています。指数関数的濃縮(SELEX)によるリガンドの系統的進化は、目的のタンパク質に特異的なアプタマーを開発する最も一般的な方法です。SELEXに続いて、迅速かつ効率的な結合アッセイにより同定プロセスが加速され、アプタマーの選択性と特異性が確認されます。
この論文では、上皮細胞接着分子(EpCAM)に特異的なアプタマーの段階的なフローサイトメトリーベースの結合アッセイについて説明します。膜貫通糖タンパク質EpCAMは、ほとんどの癌腫で過剰発現しており、癌の開始、進行、および転移に役割を果たしています。したがって、腫瘍への標的薬物送達の貴重な候補です。膜結合型EpCAMに対するアプタマーの選択性および特異性を評価するには、EpCAM陽性および陰性細胞が必要である。さらに、EpCAM結合アプタマーと同様の長さおよび2次元(2D)構造を有する非結合EpCAMアプタマーが必要である。結合アッセイには、異なるバッファー(ブロッキングバッファー、洗浄バッファー、インキュベーションバッファー、およびFACSバッファー)およびインキュベーションステップが含まれます。
アプタマーは細胞株と共にインキュベートされる。インキュベーションおよび洗浄ステップに続いて、細胞は高感度フローサイトメトリーアッセイを使用して評価されます。結果の分析は、EpCAM特異的アプタマーのEpCAM陰性細胞ではなくEpCAM陽性細胞への結合を示す。EpCAM陽性細胞では、これは、非結合アプタマー対照と比較して、EpCAMアプタマーの右への結合におけるバンドシフトとして描かれる。EpCAM陰性細胞では、EpCAM結合アプタマーおよび非結合アプタマーの対応するバンドが重複する。これは、EpCAMアプタマーの選択性と特異性を示しています。このプロトコルはEpCAMアプタマーに焦点を当てていますが、このプロトコルは他の公開されているアプタマーにも適用できます。
概要
がんは依然として世界中の主要な死亡原因の1つです1。ここ数十年で癌治療が大幅に改善されたにもかかわらず、抗癌剤の開発は依然として非常に議論されているトピックです。これは、がん治療の主力である化学療法には、患者の治療へのコンプライアンスを制限する重篤な副作用が伴うためです。さらに、化学療法誘発性癌治療に対する耐性は、医学的介入の唯一の選択肢としてのその適用を制限している。モノクローナル抗体(mAb)の適用により、がん治療に対する反応が増強されました2。モノクローナル抗体を使用する理論的根拠は、化学療法薬の有効性を改善し、その副作用を最小限に抑えることでした。.しかし、モノクローナル抗体の投与も課題となりました。これは、mAbによる免疫反応だけでなく、動物に依存して高価な製造コストと困難な保管条件によるものでした3。1990年代のアプタマーの導入4 は、アプタマーの適用がモノクローナル抗体に関連する課題に対処できるため、癌治療に新たな希望をもたらしました。
アプタマーは、特定の標的に対して特異的に産生される短い核酸配列です。指数関数的濃縮(SELEX)によるリガンドの系統的進化は、アプタマー生産における一般的な方法です。SELEXでは、目的のタンパク質をランダムなヌクレオチド配列のライブラリとともにインキュベートし、一連の反復サイクルを経て、そのタンパク質に特異的なアプタマーを精製します。アプタマーはモノクローナル抗体と同様の標的選択性・特異性を有しており、この分野の医薬品開発は有望な応用が期待されています。がんバイオマーカーに特異的なアプタマーは、単剤およびがん診断ツールとして適用できます5、6、7。ナノサイズの構造により、これらのアプタマーは、細胞傷害性物質を腫瘍に特異的に送達するための薬物キャリアとしても作用する可能性があります8。これにより、標的薬物送達の有効性が高まり、化学療法に関連するオフターゲットの副作用が減少します。.さらに、これらのナノ医薬品は組織浸透率が高いため、深部腫瘍の薬物送達および治療の望ましい候補となります。アプタマーは、血液脳関門(BBB)上に発現するトランスポーターを標的にして、脳腫瘍への薬物送達を改善するように設計することもできる9。このようなアプタマーの良い例は、トランスフェリン受容体(TfR)10 を標的にしてBBB全体の薬物送達を増強し、細胞傷害性薬物ペイロードを腫瘍細胞に送達する二機能性アプタマーである11。
アプタマーのすべての利点にもかかわらず、この分野での医薬品開発はまだ市販され、成功した抗がん剤を生み出していません。この理由の1つは、この分野の研究者が世界的に従うことができる標準的で再現可能な方法の欠如である可能性があります。この論文では、細胞表面に発現する天然タンパク質に結合するアプタマーの段階的なプロトコルが実証されています。このプロトコルは、抗がんアプタマーの前臨床評価における前提条件ステップです。アッセイは、選択性および特異性の確認のためにSELEXまたは公表されたアプタマー配列から収集された精製アプタマーの選択性および特異性を示すために行われる。このフローサイトメトリーベースのアッセイは、アプタマーの選択性と特異性を正確に示す迅速で信頼性の高い高感度アッセイであり、アプタマーは細胞表面のタンパク質に対してテストされています12、13、14。この方法は、この論文15に示すEpCAMに特異的なアプタマーの結合を用いて実証される。膜貫通糖タンパク質としてのEpCAMは、腫瘍細胞のシグナル伝達、進行、遊走、および転移において役割を果たします16,17。このアプタマーの選択性と特異性を示すために、EpCAM陽性および陰性の癌細胞を使用した。以前に開発されたEpCAM特異的アプタマーであるTEPP(5′-GC GCG GTAC CGC GC TAACG GA GGTTGCG TCC GT-3′)および陰性対照アプタマーであるTENN(5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3)を、それぞれEpCAM結合性アプタマーおよび非結合性アプタマーとして用いた10。TEPPおよびTENNの両方の3'末端をTYE665フルオロフォアで標識した。
TEPPは、一方の端からEpCAMを、もう一方の端からTfRを標的とする二機能性アプタマーです。これにより、TEPPはEpCAM+ 脳腫瘍への薬物送達に適した候補となっています。TEPPは、そのTfR特異的末端を使用して血液脳関門を横断し、EpCAM特異的末端を使用して腫瘍を発見し、その貨物(細胞傷害性薬物など)を腫瘍に送達する。TENNはTEPPと同様の長さと2D構造を持っていますが、EpCAMやTfRに親和性がないため、適切なネガティブコントロールアプタマーです。TEPPとTENNを用いて、フローサイトメトリーを用いてアプタマーの標的タンパク質への結合を試験する試験を本論文に示します。このプロトコルは、細胞特異的アプタマーの開発に適用されます。また、文献で入手可能なアプタマー配列のさらなる相補的および確認的解析にも適用可能である。このプロトコルは、以前に公開されたアプタマーを研究開発(R&D)目的で使用することを検討しているアプタマー分野の初心者にも使用できます。この論文では、文献で利用可能な2つのアプタマー配列を研究しています。
プロトコル
注意: 実験を開始する前に、白衣、手袋、ゴーグルなどの個人用保護具を着用してください。このプロトコルで使用される材料、試薬、機器、およびソフトウェアの詳細については、 材料の表 を参照してください。
1. アッセイに必要なバッファー
- この実験に必要なバッファーであるアプタマーフォールディングに必要なSELEXバッファー、ブロッキングバッファー(BB)、ウォッシュバッファー(WB)、および結合バッファー(BiB)(表1)を実験当日に新たに準備し、氷上または4°Cに保ちます。
注:各アプタマーには固有の折り畳み条件が必要です。これには、SELEXバッファーと折り畳み温度条件が含まれます。アプタマー10を記載した元の論文の方法を完全に再現するように注意する必要があります。この実験では、すべてのバッファーをダルベッコのリン酸緩衝生理食塩水(DPBS)で調製します。各実験に必要なバッファー量は、試験する細胞株の数、反復の数、およびアプタマー濃度の数によって異なります。
| ざいりょう | 必要なボリューム | ||
| アイテム | 濃度 | ||
| セレックスバッファ | マグネシウム2 | 5ミリメートル | サンプルあたり50 μL + 10%ピペッティング誤差 |
| ブロッキングバッファ | マグネシウム2 | 5ミリメートル | 細胞株あたり500 μL |
| BSA a | 1ミリグラム/ミリリットル | ||
| tRNA b | 0.1 ミリグラム/ミリリットル | ||
| FBS c | 10% (V/V) | ||
| 洗浄バッファー | マグネシウム2 | 5ミリメートル | 最初の洗浄で1 mL + 試験サンプルあたり100 μL + 10%ピペッティング誤差 |
| バインド バッファー | マグネシウム2 | 5ミリメートル | サンプルあたり50 μL + 10%ピペッティング誤差 |
| ティッカー | 2ミリグラム/ミリリットル | ||
| ティッカー | 0.2ミリグラム/ミリリットル | ||
| ティッカー | 20% (v/v) | ||
表1:結合アッセイに必要なバッファー。 あるウシ血清アルブミン、 bトランスファーリボ核酸、 cウシ胎児血清。
2. アプタマーの調製
注:アッセイで使用されるアプタマーは蛍光レポーター分子でタグ付けされているため、光から保護するように注意する必要があります。
- 実験に先立ち、パイロジェンおよびRNaseフリーの超純水を使用して、100 μMのストック(ストックA)の試験および対照アプタマーを調製します(図1)。
注意: 長期保存のために、ストックAは-20°Cの冷凍庫に保管する必要があります。 - SELEXバッファーを用いてストックAを希釈することによりアプタマーの使用濃度としてストックBを調製する(表1)。このプロトコルに従うには、ストックAを1,000nMストックに希釈してストックBを調製します(図1)。
- アプタマーを3次元(3D)構造の形成に備えるために、250 μLチューブ中でストックBをSELEXバッファーで希釈し、折り畳みに必要な容量と濃度のアプタマーを調製します。
注:折りたたまれたアプタマーは、等量の細胞に曝露されます。したがって、折り畳み用に設定されたアプタマーの濃度は、所望の最終濃度よりも2倍高濃度でなければならない。式(1)を使用して、必要な体積と濃度を計算します。ピペッティングエラーのために追加の10%容量を考慮することを忘れないでください。
濃縮在庫 A × 容積在庫A = 濃縮在庫 B × 容積在庫 B (1)
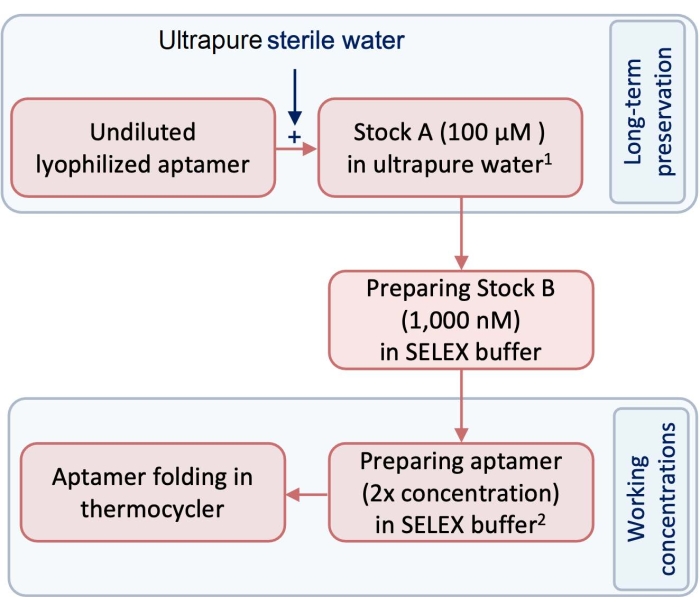
図1:アプタマーの調製における工程を示す図である。 1 ストック 1は長期保存のために-20°Cで保存されます。 2使用濃度はSELEXバッファーで調製され、保存されません。 この図の拡大版を表示するには、ここをクリックしてください。
3.がん細胞の維持
注:研究を開始する前に、細胞が初期の通過数にあり、典型的な形態学的特徴を示し、マイコプラズマがないことを確認してください。アプタマーの選択性と特異性を試験するには、目的のタンパク質の高、中、低/陰性の発現因子である細胞株が理想的です。
- 適切な培養条件を使用して、T75培養フラスコに細胞を播種します。5%CO2 加湿インキュベーターで37°Cで栽培します。
注:この研究では、10%ウシ胎児血清(完全培地)を添加したダルベッコの改変イーグル培地(DMEM)を使用しました。 - 細胞が~80%のコンフルエントに達したら、新鮮な完全培地を含む新しいフラスコに細胞を継代します。
注:目的のタンパク質と細胞株によっては、80%のコンフルエントが結合アッセイに適した細胞集団を提供する可能性があります。本実験における細胞株に対しては、MDA-MB-231およびHEK 293Tが、80%コンフルエントであることが適している。この段階で、セクション4の結合アッセイに進みます。目的のタンパク質の発現を常に確認し、そのタンパク質に特異的なモノクローナル抗体を使用してください。
4. 結合アッセイ
注: 図2 は、接着細胞における結合アッセイに必要なステップをまとめたものです。
- クラスIIバイオセーフティキャビネットで、次のように各フラスコの細胞をチューブに集めます。
- フラスコ内の培地を集めて廃棄し、2 mLのPBSを加えて細胞全体に広げ、PBSを集めて廃棄します。この手順をさらに2回繰り返して、トリプシンを不活性化する可能性のある培地の痕跡をすべて取り除きます。各フラスコに0.25%のトリプシン/EDTAを1 mL加え、37 oCで5〜10分間インキュベートします。 顕微鏡下で細胞の剥離を可視化します。
- 1 mLの完全培地を細胞に加え、細胞を上下にピペットで動かして単一細胞懸濁液を作ります。細胞を適切なチューブにピペットで入れ、200 × g で5分間遠心分離します。
注意: 非接着性細胞の場合は、細胞をチューブに集め、遠心分離(200 × g、5分)して、手順4.1.3に進みます。 - 上清を廃棄し、細胞を1 mLの新しい培地に再懸濁します。トリパンブルー染色を用いて細胞を計数し、一定量の細胞懸濁液をトリパンブルーで希釈する。~15 μLの混合物を血球計算盤とカバーガラスの間に分配します。式(2)を使用して、前述の18のようにセルをカウントします。
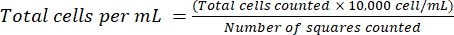 (2)
(2)
注:可能な限り最小量の細胞懸濁液を使用し、希釈係数に注意してください。例えば、等量の細胞懸濁液と0.04%トリパンブルーを混合すると、希釈係数は2になります。先に進む前に、ほとんどの接着細胞株で~90%の高い生存率(生細胞/全細胞×100)を確認してください。死細胞は非特異的にアプタマーを取り込み、結果を変化させる19。セルカウンターを用いるなどの他の細胞計数技術を用いることができる。 - 必要な数の細胞を収集し、テストサンプルごとに10×104 セルがあることを確認します。ピペッティングエラーのために追加の10%容量を検討してください。
注:実験と反復の間で常に同じ細胞数を維持することが重要です。 - 細胞を37°Cで2時間インキュベートして、酵素剥離後の細胞膜上の目的のタンパク質の安定化を可能にします。
注:このインキュベーション期間は、目的のタンパク質によって異なる場合があります。
- この2時間のインキュベーション中:
- 遠心分離機の温度を4°Cに設定します。 tRNAとアプタマーストックを室温または氷上に放置して解凍します。蛍光レポーター分子を保護するには、アプタマーチューブを光から保護します。
注:tRNAの役割は、核酸結合部位をブロックすることです。 - SELEXバッファー、BB、WB、およびBiB(セクション1を参照)を準備し、それらをすべて氷上または4°Cに保ちます。 サーモサイクラーマシンを空のサイクルに設定します。96ウェルブラックプレートとフローサイトメトリーチューブを氷上に置きます。
注意: サーモサイクラーを空のサイクルに設定すると、冷却および加熱システムが準備され、より再現性の高い結果が得られます。
- 遠心分離機の温度を4°Cに設定します。 tRNAとアプタマーストックを室温または氷上に放置して解凍します。蛍光レポーター分子を保護するには、アプタマーチューブを光から保護します。
- 2時間のインキュベーションの後、細胞を500 × g で5分間遠心分離します。上清を廃棄し、細胞を500 μLのBBに再懸濁します。細胞を4°Cで30分間、断続的に混合しながらインキュベートします。
- この30分間のインキュベーション中に、以下のようにアプタマーフォールディングを行います。
- 2倍の濃度のアプタマーを構成し(セクション2を参照)、必要な折り畳み条件に従って、サーモサイクラーマシンでアプタマーを混合してインキュベートします。このEpCAMアプタマーに対して、95°C、5分間、続いて22°C、10分間、および37°C、15分間の以下の折り畳み条件を使用する。
注:常にネガティブコントロール(すなわち、アプタマーを含まないSELEXバッファー)を含めてください。
- 2倍の濃度のアプタマーを構成し(セクション2を参照)、必要な折り畳み条件に従って、サーモサイクラーマシンでアプタマーを混合してインキュベートします。このEpCAMアプタマーに対して、95°C、5分間、続いて22°C、10分間、および37°C、15分間の以下の折り畳み条件を使用する。
- 30分間のインキュベーションの後、細胞を遠心分離し(500 × g、5分間、4°C)、上清を除去し、1 mLのWBを加え、細胞を再度遠心分離する(500 × g、5分、4°C)。上清を除去し、細胞を適切な容量のBiBに再懸濁する。
- 再懸濁した細胞50 μLを、氷冷した96ウェルブラックプレートの各ウェルにピペットで入れます。細胞を氷上に保ち、目的のタンパク質のインターナリゼーションを阻害します。
- 50 μLのアプタマーを50 μL容量の細胞にピペットで移動させ、暗所で4°Cで30分間インキュベートします。プレートを500× g、5分、4°Cで遠心分離し、上清を注意深く除去する。
- ペレットをWBに注意深く再懸濁し、500 × g で5分間遠心分離します。洗浄ステップ(4.7)を2回繰り返し、フローサイトメトリー分析のために100 μLのWBに再懸濁します。
注:アプタマーと細胞間の相互作用の図については、 図3 を参照してください。

図2:アプタマータンパク質結合アッセイを実行するステップを示す図である。 略語:SELEX = エクスポネンシャルエンリッチメントによる配位子の系統的進化;BB = ブロッキングバッファ;WB = 洗浄バッファー;BiB = バインド バッファー。 この図の拡大版を表示するには、ここをクリックしてください。

図3:アプタマー結合アッセイを行うのに必要な異なる種類の細胞およびアプタマーを示す図である。 略称:EpCAM =上皮細胞接着分子。この図は Biorender.com を使用して作成されました。この図の拡大版を表示するには、ここをクリックしてください。
5. フローサイトメトリーとデータ解析
注意: フローサイトメーターをオンにする前に、シャットダウン溶液、洗浄液、およびシース液(0.9%NaCl)のメンブレンフィルターユニットに「気泡」がないことを確認してください。カプセルに気泡がある場合は、気泡を「ブリードアウト」します。廃棄物容器が空で、シース液、水、超純水1%漂白剤の容器がいっぱいになっていることを確認してください。
- フローサイトメーターの電源を入れてから、コンピューターの電源を入れます。
注意: ここで説明するフローサイトメーターの実行の詳細は、ビデオで示されているマシンとソフトウェアに固有のものです( 材料の表を参照)。他のソフトウェアを使用するには、適切なトレーニングが必要です。 - フローサイトメトリー解析ソフトウェアを開き、プログラムにログインして、[ サイトメーター ]タブで Fluidics Startを実行します。
- 新しい実験を作成するには、[ 実験 ] タブで [新しいフォルダー] をクリックし、 フォルダー /実験に適切な名前を付けます。
- [新しいフォルダー] をクリックして強調表示し、[実験] タブでもう一度 [新しい実験] をクリックし、実験に適切な名前を付けます。
- 最初のサンプル/検体を追加するには、[実験]タブで[新規検体]をクリックし、この検体に適切な名前を付けます(細胞株/コントロールサンプル/実験サンプルの名前)。
- チューブサンプルを追加するには、検体(グループ)をハイライト表示し、 実験 タブで新規 チューブをクリックします。適切な数のチューブと名前を追加します。
- 必要なグラフを準備するには、ワークシートタブで新しい ワークシート を開きます。新しいワークシートウィンドウがポップアップしたら、ワークシート画面を使用して以下を開きます(ロゴ/画像の上にマウスを置くと名前が見つかります)。
- 前方散乱(FSC)対側方散乱(SCC)の ドットブロット グラフを準備して、対象の母集団を選択します。前方散乱および側方散乱密度プロットで対象の母集団(P1)を特定して選択することにより、最初のゲートを定義します。前方散乱信号が最も低い母集団を構成する破片を除外します。
注: FSC パラメータはサイズに基づいてセルまたはイベントを検出し、SCC は粒度20 に基づいてそれらを区別します。 - FSC面積(FSC-A)とFSC高さ(FSC-H)の ドットブロット グラフを準備して、単一細胞集団を選択します。ダブレット細胞は結果と結論に大きな影響を与えるため、ダブレット細胞集団を除外して2番目のゲートを定義します。FSC-H密度プロットとFSC-A密度プロットを使用してダブレットを除外し、同じサイズのセルが同様の面積と高さを示します。したがって、シングレットは斜めにクラスター化され、ダブレットから分離されます。
注:FSCはセルサイズにほぼ比例します。電圧パルスは、信号の強度であるFSC-H、セルサイズと信号の持続時間を反映するFSC幅、およびH×WであるFSC-Aとして定義されます。したがって、シングレットのゲーティングは、ダブレットによって引き起こされるH、W、およびA間の不均衡の検出に基づいています。 - 目的の蛍光色素に対するイベント数の ヒストグラム を準備します。
- 前方散乱(FSC)対側方散乱(SCC)の ドットブロット グラフを準備して、対象の母集団を選択します。前方散乱および側方散乱密度プロットで対象の母集団(P1)を特定して選択することにより、最初のゲートを定義します。前方散乱信号が最も低い母集団を構成する破片を除外します。
- フローサイトメトリーを開始する前に、サンプル取得を制御するための取得ダッシュボード、電圧パラメータを調整するためのインスペクター、サイトメーター、およびすべてのグラフを含むワークシートが開いていることを確認してください。
注:分析を実行するには、フローサイトメトリーチューブ内の10 ×10 4細胞懸濁液を少なくとも100 μL必要です。特に生存率が低い場合には、ヨウ化プロピジウム染色を行って生細胞集団を選択することができる21,22。 - 最初のサンプルを実行するには、画面の左側で、チューブを指す矢印が緑色であることを確認します。この矢印が緑色でない場合は、矢印をクリックして緑色にします。
- ピペットを使用して、各サンプルを96ウェルブラックプレートからフローサイトメトリーチューブに移します。未処理、未染色のコントロールサンプルを低速で実行します。
- 集録ダッシュボードで、記録する適切なイベント数(30,000)を選択し、 流量 を 低に変更して、[ データ集録]をクリックします。
- FSCおよびSCCパラメータの電圧を調整します。目的の細胞が失われないように、細胞集団がドットプロット内で集中し、グラフのどちらの軸にも接触していないことを確認します。
- 取得速度を中速または高に上げてサンプルをより速く分析しますが、200イベント/秒を超えないようにしてください。次に、[ データの記録] をクリックします。
- P1(図4A)と単一細胞集団(図4B)のゲーティングを実行します。使用した蛍光色素に対するイベントのヒストグラムを作成し、データ(この場合はアロフィコシアニン(APC))に基づいてP1を選択します(図4C)。
- 電圧を調整し、データをゲーティングして記録した後、サンプルを取り出し、[ 次のチューブ]をクリックします。
- 次のサンプルを挿入し、すべてのコントロールサンプルとテストサンプルのデータ記録を繰り返します(図3)。
- すべてのデータが収集されたら、50%漂白剤、FACSリンス、超純水の3本のチューブをそれぞれ高流量で5分間流して、フローサイトメーターを洗浄します。
- 次に、[ サイトメーター ]ドロップダウンメニューから、[ フルイディクスシャットダウン]をクリックします。
- ソフトウェアを閉じてマシンとコンピューターの電源を切る前に、次のように結果を.fcsファイルとしてUSBドライブにエクスポートして転送および分析します。
- 分析ソフトウェアで、 NEW ボタンを押して、分析を処理するための新しいドキュメントとウィンドウを作成します。サンプル ファイルを新しいウィンドウにドラッグします。
- ダブルクリックして、染色されていないサンプルを開きます。P1母集団を選択し、P1母集団をダブルクリックしてFSC-H対FSC-Aグラフを作成し、単一セル母集団をゲートします。
- ゲートされた単一セルをダブルクリックして、使用されている蛍光色素に対するイベントのヒストグラムを作成します。
- 元のウィンドウで、 P1 と 単一セル を選択し、[ すべてのサンプル ] にドラッグして、すべてのサンプルに同じゲーティングが含まれるようにします。
- [レイアウトエディタ]ボタンをクリックして、[レイアウト]ウィンドウを開きます。2つのサンプル(コントロールとテスト)を互いにドラッグして、オーバーレイヒストグラムを作成します。
結果
新薬の発見と開発の重要な側面は、医薬品候補の選択性と特異性を保証することです。これは、薬剤候補が異なる細胞を識別でき、目的の細胞集団にのみ影響を与えることができる必要があることを意味します(選択性)。選択性は、目的のタンパク質の発現に関して異なる細胞株を用いて研究される。この研究では、MDA-MB-231およびHEK 293T細胞株をEpCAM陽性および陰性細胞として選択した。特異...
ディスカッション
新しいアプタマーを開発する際の重要な課題は、このプロセスのさまざまなステップに適用される標準的なガイドラインがないことです。McKeagueらは最近、関連する課題のいくつかを実証し、出版物でのデータの不明確な提示と研究の複製の失敗につながりました。彼らは、アプタマー19を特徴づける際に考慮するために必要な基本的なガイドラインを提案した。アプタマー...
開示事項
著者は開示する利益相反を持っていません。
謝辞
著者らは、心身の健康と臨床翻訳研究所(IMPACT)SEEDの資金提供、ディーキン大学の「アルフレッドディーキンポスドク研究フェローシップ」プログラム、および「オーストラリア政府研究トレーニングプログラム奨学金」を認めています。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes with attached lid | Sigma-Aldrich | T6649 | |
| 15 mL CellStar blue screw cap, conical bottom tube | Greiner Bio One | 188271 | |
| 5 mL serological pipettes | Greiner Bio One | 606180 | |
| BD FACSCanto II Flow Becton Dickinson Cytometer | Becton Dickinson | N/A | |
| BD FACSDiva V9.0 | BD Biosciences | N/A | |
| Bovine Serum Albumin (BSA), Lyophilized powder | Sigma-AldrichTM | A7906-50G | |
| Bright-line Hemocytometer | Sigma-Aldrich | Z359629 | |
| Dulbecco’s Modified Eagle Medium (DMEM) High Glucose Media Powder | Life Technologies | 12100046 | |
| Dulbecco’s Phosphate- Buffered Saline (DPBS) | Life Technologies | 21300025 | |
| FlowJo, LLC 10.8.1 | BD Biosciences | N/A | |
| Foetal Bovine Serum (FBS) | Bovogen | SFBS-F | |
| HEK293T | American Type Culture Collection | ACS-4500 | |
| Heracell 150i CO2 Incubator | Thermo Fisher Scientific | N/A | |
| Heraeus Megafuge 16R Centrifuge | Thermo Fisher Scientific | N/A | |
| Magnesium Chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| MDA-MB-231 | American Type Culture Collection | CRM-HTB-26 | |
| Microplate, PS, 96 well, F-bottom (Chimney well), Black | Greiner Bio One | 655076 | |
| MiniAmp Thermal Cycler | Thermo Fisher Scientific | A37834 | |
| Phosphate-Buffered Saline (PBS) tablets | Life Technologies | 18912014 | |
| Pyrogen- and RNase-free ultrapure water | Milli-Q | ||
| T75 Cell Culture flask | Cellstar | 658170 | |
| TENN | Integrated DNA Technologies | N/A | 5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3 |
| TEPP | Integrated DNA Technologies | N/A | 5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′ |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8508-5X1ML | |
| Trypan Blue Solution | Life Technologies | 15250061 | |
| Trypsin-EDTA | Gibco | 15400054 |
参考文献
- Cancer. World Health Organization Available from: https://www.who.int/news-room/fact-sheets/detail/cancer#:~:text=Cancer%20is%20a%20leading%20cause.and%20rectum%20and%20prostate%20cancers (2022)
- Liu, J. K. H. The history of monoclonal antibody development - Progress, remaining challenges and future innovations. Annals of Medicine and Surgery. 3 (4), 113-116 (2014).
- Nakhjavani, M., Shigdar, S. Future of PD-1/PD-L1 axis modulation for the treatment of triple-negative breast cancer. Pharmacological Research. 175, 106019 (2022).
- Bukari, B., Samarasinghe, R. M., Noibanchong, J., Shigdar, S. L. Non-invasive delivery of therapeutics into the brain: the potential of aptamers for targeted delivery. Biomedicines. 8 (5), 120 (2020).
- Wu, X., Chen, J., Wu, M., Zhao, J. X. Aptamers: active targeting ligands for cancer diagnosis and therapy. Theranostics. 5 (4), 322 (2015).
- Feng, X., et al. The aptamer functionalized nanocomposite used for prostate cancer diagnosis and therapy. Journal of Nanomaterials. 2022, (2022).
- Huang, J., et al. Advances in aptamer-based biomarker discovery. Frontiers in Cell and Developmental Biology. 9, 571 (2021).
- Ashrafuzzaman, M. Aptamers as both drugs and drug-carriers. BioMed Research International. 2014, (2014).
- Nakhjavani, M., Samarasinghe, R. M., Shigdar, S. Triple-negative breast cancer brain metastasis: an update on druggable targets, current clinical trials, and future treatment options. Drug Discovery Today. , (2022).
- Macdonald, J., et al. Development of a bifunctional aptamer targeting the transferrin receptor and epithelial cell adhesion molecule (EpCAM) for the treatment of brain cancer metastases. ACS Chemical Neuroscience. 8 (4), 777-784 (2017).
- Macdonald, J., et al. Bifunctional aptamer-doxorubicin conjugate crosses the blood-brain barrier and selectively delivers its payload to EpCAM-positive tumor cells. Nucleic Acid Therapeutics. 30 (2), 117-128 (2020).
- Shigdar, S., Agnello, L., Fedele, M., Camorani, S., Cerchia, L. Profiling cancer cells by cell-SELEX: use of aptamers for discovery of actionable biomarkers and therapeutic applications thereof. Pharmaceutics. 14 (1), 28 (2021).
- Rahimizadeh, K., et al. Development of cell-specific aptamers: recent advances and insight into the selection procedures. Molecules. 22 (12), 2070 (2017).
- Chen, M., et al. Development of cell-SELEX technology and its application in cancer diagnosis and therapy. International Journal of Molecular Sciences. 17 (12), 2079 (2016).
- Shigdar, S., et al. The use of sensitive chemical antibodies for diagnosis: detection of low levels of EpCAM in breast cancer. PLoS One. 8 (2), 57613 (2013).
- Ni, J., et al. Role of the EpCAM (CD326) in prostate cancer metastasis and progression. Cancer and Metastasis Reviews. 31 (3), 779-791 (2012).
- Ni, J., et al. Epithelial cell adhesion molecule (EpCAM) is associated with prostate cancer metastasis and chemo/radioresistance via the PI3K/Akt/mTOR signaling pathway. The International Journal of Biochemistry & Cell Biology. 45 (12), 2736-2748 (2013).
- McKeague, M., Kruse, P. F., Patterson, M. K., et al. . Tissue Culture. , 395-397 (1973).
- McKeague, M., et al. The minimum aptamer publication standards (MAPS guidelines) for de novo aptamer selection. Aptamers. 6, 10-18 (2022).
- Schoofs, G., Van Hout, A., D'huys, T., Schols, D., Van Loy, T. A flow cytometry-based assay to identify compounds that disrupt binding of fluorescently-labeled CXC Chemokine ligand 12 to CXC Chemokine receptor 4. Journal of Visualized Experiments. (133), e57271 (2018).
- McKinnon, K. M. Flow cytometry: an overview. Current Protocols in Immunology. 120 (1), 1-11 (2018).
- Kamiloglu, S., Sari, G., Ozdal, T., Capanoglu, E. Guidelines for cell viability assays. Food Frontiers. 1 (3), 332-349 (2020).
- Ruscito, A., DeRosa, M. C. Small-molecule binding aptamers: Selection strategies, characterization, and applications. Frontiers in Chemistry. 4, 14 (2016).
- McKeague, M., et al. Comprehensive analytical comparison of strategies used for small molecule aptamer evaluation. Analytical Chemistry. 87 (17), 8608-8612 (2015).
- Henri, J., Bayat, N., Macdonald, J., Shigdar, S. A guide to using nucleic acid aptamers in cell based assays. Aptamers. 23, (2019).
- Mao, H., et al. The mechanism and regularity of quenching the effect of bases on fluorophores: the base-quenched probe method. Analyst. 143 (14), 3292-3301 (2018).
- McKeague, M., et al. Analysis of in vitro aptamer selection parameters. Journal of Molecular Evolution. 81 (5), 150-161 (2015).
- Chen, B., et al. Targeting negative surface charges of cancer cells by multifunctional nanoprobes. Theranostics. 6 (11), 1887 (2016).
- Shigdar, S., et al. RNA aptamers targeting cancer stem cell marker CD133. Cancer Letters. 330 (1), 84-95 (2013).
- Amraee, M., Oloomi, M., Yavari, A., Bouzari, S. DNA aptamer identification and characterization for E. coli O157 detection using cell based SELEX method. Analytical Biochemistry. 536, 36-44 (2017).
- Yu, X. -. X., et al. Selection and characterization of a novel DNA aptamer, Apt-07S specific to hepatocellular carcinoma cells. Drug Design, Development and Therapy. 14, 1535 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved