É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um ensaio de ligação de proteínas de superfície celular baseado em citometria de fluxo para avaliar a seletividade e a especificidade de um Aptamer anticancerígeno
Neste Artigo
Resumo
Um passo necessário no desenvolvimento do aptâmero anticâncer é testar sua ligação ao alvo. Demonstramos um ensaio baseado em citometria de fluxo para estudar essa ligação, enfatizando a importância de incluir um aptâmero de controle negativo e células cancerígenas que são positivas ou negativas para essa proteína em particular.
Resumo
Um dos principais desafios no desenvolvimento de um aptâmero anticâncer é determinar eficientemente a seletividade e a especificidade do aptâmero desenvolvido para a proteína alvo. Devido às suas várias vantagens sobre os anticorpos monoclonais, o desenvolvimento do aptâmero ganhou enorme popularidade entre os pesquisadores do câncer. A evolução sistemática de ligantes por enriquecimento exponencial (SELEX) é o método mais comum de desenvolvimento de aptâmeros específicos para proteínas de interesse. Após o SELEX, um ensaio de ligação rápido e eficiente acelera o processo de identificação, confirmando a seletividade e especificidade do aptâmero.
Este artigo explica um ensaio de ligação baseado em citometria de fluxo passo-a-passo de um aptâmero específico para molécula de adesão celular epitelial (EpCAM). A glicoproteína transmembrana EpCAM é superexpressa na maioria dos carcinomas e desempenha papéis no início, progressão e metástase do câncer. Portanto, é um candidato valioso para a entrega de medicamentos direcionados aos tumores. Para avaliar a seletividade e a especificidade do aptâmero para a EpCAM ligada à membrana, são necessárias células positivas e negativas para EpCAM. Além disso, é necessário um aptâmero EpCAM não vinculativo com um comprimento e estrutura 2-dimensional (2D) semelhantes ao aptâmero de ligação ao EpCAM. O ensaio de ligação inclui diferentes buffers (buffer de bloqueio, buffer de lavagem, buffer de incubação e buffer FACS) e etapas de incubação.
O aptâmero é incubado com as linhas celulares. Após as etapas de incubação e lavagem, as células serão avaliadas por meio de um ensaio de citometria de fluxo sensível. A análise dos resultados mostra a ligação do aptâmero específico do EpCAM às células positivas para EpCAM e não às células negativas para EpCAM. Em células positivas para EpCAM, isso é descrito como um deslocamento de banda na ligação do aptâmero EpCAM à direita em comparação com o controle do aptâmero não ligante. Em células EpCAM-negativas, as bandas correspondentes de aptâmeros de ligação e não ligação a EpCAM se sobrepõem. Isso demonstra a seletividade e a especificidade do aptamer EpCAM. Enquanto este protocolo é focado no aptâmero EpCAM, o protocolo é aplicável a outros aptâmeros publicados.
Introdução
O câncer ainda é uma das principais causas de mortalidade em todo o mundo1. Apesar da melhoria significativa no tratamento do câncer nas últimas décadas, o desenvolvimento de medicamentos anticâncer ainda é um tópico altamente debatido. Isso ocorre porque a quimioterapia, como a base do tratamento do câncer, é acompanhada por efeitos colaterais graves que limitam a adesão do paciente ao tratamento. Além disso, a resistência ao tratamento do câncer induzida pela quimioterapia restringiu sua aplicação como a única escolha de intervenção médica. A aplicação de anticorpos monoclonais (mAbs) introduziu uma resposta aprimorada aos tratamentos contra o câncer2. A lógica do uso de mAbs foi melhorar a eficácia dos quimioterápicos e minimizar suas reações adversas. No entanto, a administração de mAbs também se tornou um desafio. Isso não ocorreu apenas por causa das reações imunológicas induzidas por mAb, mas também devido aos custos de produção caros e dependentes de animais e às difíceis condições de armazenamento3. A introdução dos aptâmerosna década de 19904 levantou novas esperanças no tratamento do câncer, já que a aplicação de aptâmeros poderia enfrentar os desafios associados ao mAbs.
Aptamers são sequências curtas de ácidos nucleicos que são produzidas especificamente para um determinado alvo. A evolução sistemática de ligantes por enriquecimento exponencial (SELEX) é um método comum na produção de aptâmeros. No SELEX, a proteína de interesse é incubada com uma biblioteca de sequências aleatórias de nucleotídeos e, através de uma série de ciclos iterativos, o aptâmero específico para essa proteína é purificado. Os aptâmeros têm seletividade e especificidade de alvo semelhantes aos mAbs e, portanto, o desenvolvimento de medicamentos neste campo mostra aplicações futuras promissoras. Aptamers específicos para biomarcadores de câncer poderiam ser aplicados como medicamentos únicos e ferramentas de diagnóstico de câncer 5,6,7. Devido à sua estrutura nanométrica, esses aptâmeros também poderiam atuar como portadores de fármacos para fornecer agentes citotóxicos especificamente ao tumor8. Isso aumentaria a eficácia da administração direcionada de medicamentos e diminuiria as reações adversas fora do alvo associadas à quimioterapia. Além disso, esses nanomedicamentos têm uma alta penetração tecidual, o que os torna um candidato desejável para a entrega e tratamento de medicamentos de tumores profundos. Os aptâmeros também podem ser projetados para atingir os transportadores expressos na barreira hematoencefálica (BBB) para melhorar a entrega de drogas a tumores cerebrais9. Um bom exemplo de tal aptâmero são os aptâmeros bifuncionais, visando o receptor de transferrina (TfR)10 para melhorar a entrega de drogas em todo o BBB e entregando uma carga útil de drogas citotóxicas às células tumorais11.
Apesar de todas as vantagens dos aptâmeros, o desenvolvimento de medicamentos neste campo ainda não produziu um medicamento anticâncer comercializado e bem-sucedido. Uma razão para isso pode ser a falta de métodos padrão e reprodutíveis que poderiam ser seguidos globalmente por pesquisadores da área. Neste trabalho, um protocolo passo-a-passo de um aptâmero se ligando a uma proteína nativa expressa na superfície celular é demonstrado. Este protocolo é uma etapa pré-requisito na avaliação pré-clínica de aptâmeros anticancerígenos. O ensaio é realizado para mostrar a seletividade e especificidade do aptâmero purificado coletado do SELEX ou uma sequência de aptâmeros publicada para confirmação da seletividade e especificidade. Este ensaio baseado em citometria de fluxo é um ensaio rápido, confiável e sensível que mostra com precisão a seletividade e a especificidade do aptâmero, onde o aptâmero está sendo testado contra proteínas na superfície celular12,13,14. Este método é demonstrado usando a ligação de um aptâmero específico para EpCAM mostrado neste trabalho15. A EpCAM, como glicoproteína transmembrana, desempenha papéis na sinalização, progressão, migração e metástase de células tumorais16,17. Para mostrar a seletividade e especificidade deste aptâmero, foram utilizadas células cancerígenas positivas e negativas para EpCAM. O aptâmero específico de EpCAM previamente desenvolvido, TEPP (5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′), e um aptâmero controle negativo, TENN (5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3), foram utilizados como aptâmeros ligantes e não ligantes de EpCAM, respectivamente10. A extremidade 3' do TEPP e do TENN foi marcada com um fluoróforo TYE665.
O TEPP é um aptâmero bifuncional que tem como alvo o EpCAM de uma extremidade e o TfR do outro. Isso tornou o TEPP um candidato adequado para a administração de medicamentos para tumores cerebrais EpCAM +. Usando sua extremidade específica da TfR, a TEPP atravessa a barreira hematoencefálica e, usando a extremidade específica da EpCAM, encontra o tumor e entrega sua carga (por exemplo, drogas citotóxicas) ao tumor. O TENN tem um comprimento e uma estrutura 2D semelhantes ao TEPP, mas não tem afinidade com o EpCAM ou TfR e, portanto, é um aptâmero de controle negativo adequado. Usando TEPP e TENN, testar a ligação de um aptâmero à proteína-alvo usando citometria de fluxo é mostrado neste artigo. Este protocolo aplica-se ao desenvolvimento de aptâmeros específicos de células. Também é aplicável a outras análises complementares e de confirmação das sequências de aptâmeros disponíveis na literatura. O protocolo também pode ser usado por aqueles novos no campo do aptâmero que estão procurando usar um aptâmero publicado anteriormente para seus fins de pesquisa e desenvolvimento (P & D). Neste trabalho, são estudadas duas sequências de aptâmeros disponíveis na literatura.
Protocolo
NOTA: Antes de iniciar o experimento, use equipamentos de proteção individual, incluindo um jaleco, luvas e óculos. Consulte a Tabela de Materiais para obter detalhes sobre materiais, reagentes, equipamentos e software usados neste protocolo.
1. Buffers necessários para o ensaio
- Preparar os buffers necessários para este experimento - o buffer SELEX necessário para dobrar aptâmeros, Blocking Buffer (BB), Wash Buffer (WB) e Binding Buffer (BiB) (Tabela 1) - recentemente no dia do experimento e mantê-los no gelo ou a 4 °C.
NOTA: Cada aptâmero requer uma condição de dobragem única. Isso inclui o buffer SELEX e as condições de temperatura dobrável. Deve-se ter o cuidado de replicar completamente os métodos do artigo original que descreve o aptâmero10. Neste experimento, todos os tampões são preparados na solução salina tamponada com fosfato (DPBS) da Dulbecco. O volume tampão necessário em cada experimento depende do número de linhagens celulares, do número de replicações e do número de concentrações de aptâmeros que são testadas.
| Ingredientes | Volume necessário | ||
| Item | Concentração | ||
| Buffer SELEX | MgCl2 | 5 mM | 50 μL por amostra + erro de pipetagem de 10% |
| Bloqueando o buffer | MgCl2 | 5 mM | 500 μL por linhagem celular |
| BSA a | 1 mg/mL | ||
| RNAt b | 0,1 mg/ml | ||
| FBS c | 10% (v/v) | ||
| Buffer de lavagem | MgCl2 | 5 mM | 1 mL para a primeira lavagem + 100 μL por amostra de teste + 10% de erro de pipetagem |
| Buffer de ligação | MgCl2 | 5 mM | 50 μL por amostra + erro de pipetagem de 10% |
| BSA | 2 mg/mL | ||
| Trna | 0,2 mg/ml | ||
| FBS | 20% (v/v) | ||
Tabela 1: Buffers necessários para o ensaio de ligação. um Albumina Sérica Bovina, bTransferência de Ácido Ribonucleico, cSoro Fetal Bovino.
2. Preparação de aptâmeros
NOTA: Os aptâmeros usados no ensaio são marcados com uma molécula repórter de fluorescência e, portanto, deve-se tomar cuidado para protegê-los da luz.
- Antes do experimento, preparar um estoque de 100 μM (estoque A) de aptâmeros de teste e controle usando água ultrapura livre de pirogênio e RNase (Figura 1).
NOTA: Para a preservação a longo prazo, a unidade populacional A deve ser mantida no congelador a -20 °C. - Preparar a unidade populacional B como concentração de trabalho dos aptâmeros diluindo a reserva A utilizando o tampão SELEX (quadro 1). Para seguir esse protocolo, dilua o estoque A para um estoque de 1.000 nM para preparar o estoque B (Figura 1).
- Para preparar o aptâmero para a formação da estrutura tridimensional (3D), num tubo de 250 μL, dilua o reservatório B com tampão SELEX para preparar o volume e a concentração necessários do aptâmero para dobragem.
NOTA: Os aptâmeros dobrados serão expostos a um volume igual de células. Portanto, a concentração do aptâmero que é ajustado para dobramento deve ser 2x mais concentrada do que a concentração final desejada. Use a equação (1) para calcular os volumes e concentrações necessários. Lembre-se de considerar um volume extra de 10% para o erro de pipetagem.
Existênciasde concentração A × Existências de volume A = Existências de concentração B ×Existências de volumeB (1)
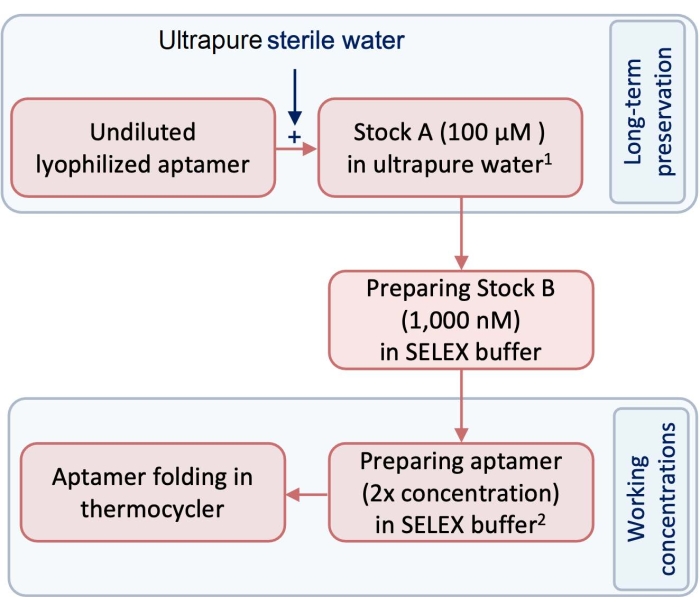
Figura 1: Um diagrama mostrando as etapas na preparação dos aptâmeros. 1 O estoque 1é armazenado a -20 °C para preservação a longo prazo. algarismo As concentrações de trabalho são preparadas em tampão SELEX e não são armazenadas. Por favor, clique aqui para ver uma versão maior desta figura.
3. Manutenção das células cancerosas
NOTA: Antes do início do estudo, certifique-se de que as células estão em seus números de passagem iniciais, mostram suas características morfológicas típicas e estão livres de micoplasma. Para testar a seletividade e a especificidade do aptâmero, linhagens celulares que sejam expressores altos, moderados e baixos/negativos da proteína de interesse são idealmente necessários.
- Semeia as células num balão de cultura T75, utilizando condições de cultura adequadas. Cultivá-los em uma incubadora umidificada de CO2 a 5%, a 37 °C.
NOTA: Neste estudo, foi utilizado o Dulbecco's Modified Eagle Medium (DMEM) suplementado com 10% de soro fetal bovino (meio completo). - Quando as células atingirem ~80% de confluência, passá-las para um novo frasco contendo meio fresco completo.
NOTA: Dependendo da proteína de interesse e da linhagem celular, a confluência de 80% pode fornecer uma população celular adequada para o ensaio de ligação. Para as linhagens celulares deste experimento, MDA-MB-231 e HEK 293T, a confluência de 80% é adequada. Nesta fase, prossiga para a secção 4, o ensaio vinculativo. Verifique sempre a expressão da proteína de interesse, usando mAbs específicos para essa proteína.
4. Ensaio vinculativo
NOTA: A Figura 2 resume as etapas necessárias no ensaio de vinculação em células aderentes.
- Num armário de biossegurança da classe II, recolher as células de cada balão em tubos do seguinte modo:
- Recolher e deitar fora o meio no balão, adicionar 2 ml de PBS, espalhá-lo sobre as células e, em seguida, recolher e descartar o PBS. Repita esta etapa mais duas vezes para remover todos os vestígios de mídia que podem inativar a tripsina. Adicionar 1 ml de 0,25% de tripsina/EDTA a cada balão e incubar durante 5-10 min a 37 o C. Visualize odescolamento das células ao microscópio.
- Adicione 1 mL de meio completo às células e pipete as células para cima e para baixo para fazer uma suspensão de célula única. Pipetar as células para um tubo adequado e centrifugar a 200 × g durante 5 minutos.
NOTA: Para as células não aderentes, recolher as células num tubo, centrifugar (200 × g, 5 min) e passar ao passo 4.1.3. - Descarte o sobrenadante e ressuspenda as células em 1 mL de meio fresco. Conte as células usando a coloração azul de tripano, diluindo um certo volume de suspensão celular com azul de tripano. Distribua ~15 μL da mistura entre um hemocitômetro e um copo de cobertura. Conte as células conforme descrito anteriormente18, usando a equação (2):
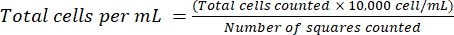 (2)
(2)
NOTA: Utilize o volume mínimo possível de suspensão celular e tome nota do factor de diluição. Por exemplo, misturar volumes iguais de suspensão celular e azul de tripano a 0,04% dá um fator de diluição de 2. Garantir alta viabilidade (células vivas / células totais × 100) de ~ 90% para a maioria das linhagens celulares aderentes antes de prosseguir. Células mortas não especificamente absorvem aptâmeros e alteram os resultados19. É possível usar outras técnicas de contagem de células, como usar um contador de células. - Colete o número necessário de células, certificando-se de ter 10 × 104 células por amostra de teste. Considere um volume extra de 10% para erro de pipetagem.
NOTA: É importante manter sempre a mesma contagem de células entre experimentos e replicações. - Incubar as células a 37 °C durante 2 h para permitir a estabilização da proteína de interesse na membrana celular após descolamento enzimático.
NOTA: Este período de incubação pode diferir de acordo com a proteína de interesse.
- Durante esta incubação de 2 h:
- Ajustar a temperatura da centrífuga para 4 °C. Deixe o RNAt e os estoques de aptâmero à temperatura ambiente ou no gelo para descongelar. Para proteger a molécula repórter de fluorescência, proteja os tubos de aptâmero da luz.
NOTA: O papel do RNAt é bloquear os locais de ligação do ácido nucleico. - Preparar o tampão SELEX, BB, WB e BiB (ver secção 1), mantendo-os todos no gelo ou a 4 °C. Coloque a máquina termocicladora em um ciclo vazio. Coloque uma placa preta de 96 poços e tubos de citometria de fluxo no gelo.
NOTA: Colocar o termociclador em um ciclo vazio prepara o sistema de resfriamento e aquecimento e ajuda a gerar resultados mais reprodutíveis.
- Ajustar a temperatura da centrífuga para 4 °C. Deixe o RNAt e os estoques de aptâmero à temperatura ambiente ou no gelo para descongelar. Para proteger a molécula repórter de fluorescência, proteja os tubos de aptâmero da luz.
- Após a incubação de 2 h, centrifugar as células a 500 × g por 5 min. Descarte o sobrenadante e ressuspenda as células em 500 μL de BB. Incubar as células a 4 °C durante 30 minutos com mistura intermitente.
- Durante esta incubação de 30 minutos, realize a dobragem do aptâmero da seguinte forma:
- Completar as concentrações de 2x de aptâmeros (ver secção 2) e, em seguida, misturar e incubar os aptâmeros na máquina termocicladora, de acordo com as condições de dobragem exigidas. Para este aptâmero EpCAM, use as seguintes condições de dobragem de 95 °C, 5 min, seguido por 22 °C, 10 min, e 37 °C, 15 min.
NOTA: Inclua sempre um controlo negativo (ou seja, tampão SELEX sem aptâmeros).
- Completar as concentrações de 2x de aptâmeros (ver secção 2) e, em seguida, misturar e incubar os aptâmeros na máquina termocicladora, de acordo com as condições de dobragem exigidas. Para este aptâmero EpCAM, use as seguintes condições de dobragem de 95 °C, 5 min, seguido por 22 °C, 10 min, e 37 °C, 15 min.
- Após a incubação de 30 min, centrifugar as células (500 × g, 5 min, 4 °C), remover o sobrenadante, adicionar 1 mL de WB e centrifugar as células novamente (500 × g, 5 min, 4 °C). Remova o sobrenadante e ressuspenda as células em um volume adequado do BiB.
- Pipetar 50 μL das células ressuspensas em cada poço de uma placa preta gelada de 96 poços. Mantenha as células no gelo para inibir a internalização da proteína de interesse.
- Pipetar 50 μL dos aptâmeros para um volume de 50 μL de células, misturar e incubar no escuro a 4 °C durante 30 min. Centrifugar a placa a 500 × g, 5 min, 4 °C e remover cuidadosamente o sobrenadante.
- Ressuscite cuidadosamente o pellet em WB e centrifugar a 500 × g durante 5 min. Repetir a etapa de lavagem (4.7) 2x e ressuspender em 100 μL de WB para análise citométrica de fluxo.
NOTA: Consulte a Figura 3 para obter um diagrama de interações entre aptâmeros e células.

Figura 2: Um diagrama que descreve as etapas na execução de um ensaio de ligação à proteína do aptâmero. Abreviaturas: SELEX = Evolução Sistemática de Ligantes por Enriquecimento EXPONENCIAL; BB = Buffer de Bloqueio; WB = Tampão de Lavagem; BiB = buffer de ligação. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 3: Um diagrama mostrando os diferentes tipos de células e aptâmeros necessários para executar o ensaio de ligação do aptâmero. Abreviação: EpCAM = molécula de adesão celular epitelial. Esta figura foi criada usando Biorender.com. Por favor, clique aqui para ver uma versão maior desta figura.
5. Citometria de fluxo e análise dos dados
NOTA: Antes de ligar o citômetro de fluxo, certifique-se de que não há "bolhas" nas unidades de filtro de membrana para a solução de desligamento, solução de limpeza e fluido da bainha (NaCl a 0,9%). "Sangra" bolhas se houver bolhas nas cápsulas. Certifique-se de que o recipiente de resíduos está vazio e os recipientes de fluido da bainha, água e 1% de água sanitária em água ultrapura estão cheios.
- Ligue o citômetro de fluxo e, em seguida, o computador.
NOTA: Os detalhes da execução do citômetro de fluxo explicados aqui são específicos da máquina e do software demonstrados no vídeo (consulte Tabela de materiais). Outro software exigiria treinamento apropriado para uso. - Abra o software de análise de citometria de fluxo, faça login no programa e, na guia Citômetro , execute o Fluidics Start up.
- Para criar um novo experimento, na guia experimento, clique em Nova Pasta e nomeie a pasta/experimento adequadamente.
- Clique em nova pasta para realçar e, em seguida, na guia experimento novamente, clique em Novo experimento e nomeie o experimento adequadamente.
- Para adicionar a primeira amostra/espécime, na guia experimento, clique em Nova amostra e nomeie essa amostra apropriadamente (nome da linhagem celular/amostra de controle/amostra experimental).
- Para adicionar uma amostra de tubo, realce a amostra (grupo) e, na guia experimento , clique em Novo tubo. Adicione o número apropriado de tubos e o nome.
- Para preparar os gráficos necessários, na guia planilha , abra uma nova planilha. Quando a nova janela da planilha aparecer, abra o seguinte usando a tela da planilha (passe o mouse sobre o logotipo/imagens para encontrar os nomes):
- Prepare um gráfico de mancha de pontos de dispersão para frente (FSC) versus dispersão lateral (SCC) para selecionar a população de interesse. Defina o primeiro portão identificando e selecionando a população de interesse (P1) em um gráfico de densidade de dispersão para frente e para o lado. Exclua os detritos, que constituem a população com o menor sinal de dispersão para a frente.
Observação : O parâmetro FSC detecta células ou eventos com base em seu tamanho e o SCC os discrimina com base em sua granularidade20. - Prepare um gráfico de mancha de pontos de área FSC (FSC-A) versus altura FSC (FSC-H) para selecionar a população de célula única. Defina a segunda porta excluindo as populações de células duplas, pois as células duplas afetam consideravelmente os resultados e as conclusões. Exclua duplicatas usando gráficos de densidade FSC-H versus FSC-A, em que células do mesmo tamanho mostram uma área e altura semelhantes. Assim, os singlets ficam agrupados diagonalmente e separados dos dubletos.
NOTA: FSC é aproximadamente proporcional ao tamanho da célula. Os pulsos de tensão são definidos como FSC-H, a intensidade do sinal, FSC-largura que reflete o tamanho da célula e a duração do sinal, e FSC-A, que é H × W. Doublets têm um valor duplo de largura e área; portanto, o gating para singlets é baseado na detecção de desproporções entre H, W e A causadas por dubletes. - Prepare um histograma do número de eventos contra o fluoróforo de interesse.
- Prepare um gráfico de mancha de pontos de dispersão para frente (FSC) versus dispersão lateral (SCC) para selecionar a população de interesse. Defina o primeiro portão identificando e selecionando a população de interesse (P1) em um gráfico de densidade de dispersão para frente e para o lado. Exclua os detritos, que constituem a população com o menor sinal de dispersão para a frente.
- Antes de iniciar a citometria de fluxo, certifique-se de que o painel de aquisição para controlar a aquisição da amostra, o inspetor e o citômetro para ajustar os parâmetros de tensão, bem como a planilha com todos os gráficos, estejam abertos.
NOTA: Pelo menos 100 μL de uma suspensão de 10 × 104 células em um tubo de citometria de fluxo são necessários para realizar a análise. Especialmente em caso de menores viabilidades, a coloração com iodeto de propídio pode ser realizada para selecionar a população celular viável21,22. - Para executar a primeira amostra, no lado esquerdo da tela, verifique se a seta que aponta para o tubo está verde. Se esta seta não estiver verde, clique na seta para torná-la verde.
- Usando uma pipeta, transfira cada amostra da placa preta de 96 poços para um tubo de citometria de fluxo. Execute a amostra de controle não tratada e não manchada em uma velocidade baixa.
- No painel de aquisição, escolha um número apropriado de eventos a serem registrados (30.000), altere a taxa de fluxo para baixa e clique em Adquirir Dados.
- Ajuste a tensão para os parâmetros FSC e SCC. Certifique-se de que a população celular esteja centralizada dentro do gráfico de pontos e que nenhuma célula esteja tocando nenhum dos eixos do gráfico para evitar a perda das células de interesse.
- Aumente a velocidade de aquisição para média ou alta para analisar as amostras mais rapidamente, mas não exceda mais de 200 eventos/s. Em seguida, clique em Registrar dados.
- Realizar o gating para P1 (Figura 4A) e a população unicelular (Figura 4B). Construa o histograma dos eventos contra o fluorocromo utilizado e selecione P1 com base nos dados (aloficocianina (APC) neste caso) (Figura 4C).
- Depois de ajustar a tensão, o controle e o registro dos dados, retire a amostra e clique em Avançar tubo.
- Insira a próxima amostra e repita os dados de gravação para todas as amostras de controle e teste (Figura 3).
- Depois que todos os dados forem coletados, lave o citômetro de fluxo executando três tubos de água sanitária a 50%, enxágue FACS e água ultrapura, cada um por 5 minutos a uma alta taxa de fluxo.
- Em seguida, no menu suspenso Citômetro , clique em Fluidics Shut Down ( Fluidics Shut Down).
- Antes de fechar o software e desligar a máquina e o computador, exporte os resultados como arquivos .fcs para uma unidade USB para transferi-los e analisá-los, da seguinte maneira:
- No software de análise, pressione o botão NEW para criar um novo documento e uma nova janela para lidar com a análise. Arraste os arquivos de exemplo para a nova janela.
- Clique duas vezes para abrir a amostra não manchada. Escolha a população P1, clique duas vezes na população P1 para criar um gráfico FSC-H versus FSC-A e porta a população de célula única.
- Clique duas vezes nas células individuais fechadas para criar um histograma de eventos contra o fluorocromo usado.
- Na janela original, selecione P1 e células individuais e arraste-as para Todas as amostras para que todas as amostras agora contenham o mesmo gating.
- Clique no botão Editor de layout para abrir a janela Layouts . Arraste duas amostras (controle e teste) uma sobre a outra para criar um histograma de sobreposição.
Resultados
Um aspecto importante da descoberta e desenvolvimento de novos medicamentos é garantir a seletividade e a especificidade do candidato a medicamento. Isso significa que o candidato a medicamento deve ser capaz de discriminar entre diferentes células e afetar apenas a população celular de interesse (seletividade). A seletividade é estudada usando linhagens celulares que diferem em termos de expressão da proteína de interesse. Neste estudo, as linhagens celulares MDA-MB-231 e HEK 293T foram escolhidas como células E...
Discussão
O principal desafio com o desenvolvimento de novos aptâmeros é a falta de diretrizes padrão que se aplicam a diferentes etapas desse processo. McKeague et al. demonstraram recentemente alguns dos desafios associados, que levam a apresentações pouco claras de dados em publicações e falha em replicar a pesquisa. Propuseram diretrizes fundamentais necessárias para a consideração na caracterização dos aptâmeros19. Um ensaio de ligação de aptâmeros é uma etapa crítica na triagem e/ou ...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Os autores reconhecem o financiamento do SEED do Instituto de Saúde Mental e Física e Tradução Clínica (IMPACT), o programa "Alfred Deakin Postdoctoral Research Fellowship" da Universidade de Deakin e a "Bolsa de Estudo do Programa de Treinamento em Pesquisa do Governo Australiano".
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes with attached lid | Sigma-Aldrich | T6649 | |
| 15 mL CellStar blue screw cap, conical bottom tube | Greiner Bio One | 188271 | |
| 5 mL serological pipettes | Greiner Bio One | 606180 | |
| BD FACSCanto II Flow Becton Dickinson Cytometer | Becton Dickinson | N/A | |
| BD FACSDiva V9.0 | BD Biosciences | N/A | |
| Bovine Serum Albumin (BSA), Lyophilized powder | Sigma-AldrichTM | A7906-50G | |
| Bright-line Hemocytometer | Sigma-Aldrich | Z359629 | |
| Dulbecco’s Modified Eagle Medium (DMEM) High Glucose Media Powder | Life Technologies | 12100046 | |
| Dulbecco’s Phosphate- Buffered Saline (DPBS) | Life Technologies | 21300025 | |
| FlowJo, LLC 10.8.1 | BD Biosciences | N/A | |
| Foetal Bovine Serum (FBS) | Bovogen | SFBS-F | |
| HEK293T | American Type Culture Collection | ACS-4500 | |
| Heracell 150i CO2 Incubator | Thermo Fisher Scientific | N/A | |
| Heraeus Megafuge 16R Centrifuge | Thermo Fisher Scientific | N/A | |
| Magnesium Chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| MDA-MB-231 | American Type Culture Collection | CRM-HTB-26 | |
| Microplate, PS, 96 well, F-bottom (Chimney well), Black | Greiner Bio One | 655076 | |
| MiniAmp Thermal Cycler | Thermo Fisher Scientific | A37834 | |
| Phosphate-Buffered Saline (PBS) tablets | Life Technologies | 18912014 | |
| Pyrogen- and RNase-free ultrapure water | Milli-Q | ||
| T75 Cell Culture flask | Cellstar | 658170 | |
| TENN | Integrated DNA Technologies | N/A | 5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3 |
| TEPP | Integrated DNA Technologies | N/A | 5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′ |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8508-5X1ML | |
| Trypan Blue Solution | Life Technologies | 15250061 | |
| Trypsin-EDTA | Gibco | 15400054 |
Referências
- Cancer. World Health Organization Available from: https://www.who.int/news-room/fact-sheets/detail/cancer#:~:text=Cancer%20is%20a%20leading%20cause.and%20rectum%20and%20prostate%20cancers (2022)
- Liu, J. K. H. The history of monoclonal antibody development - Progress, remaining challenges and future innovations. Annals of Medicine and Surgery. 3 (4), 113-116 (2014).
- Nakhjavani, M., Shigdar, S. Future of PD-1/PD-L1 axis modulation for the treatment of triple-negative breast cancer. Pharmacological Research. 175, 106019 (2022).
- Bukari, B., Samarasinghe, R. M., Noibanchong, J., Shigdar, S. L. Non-invasive delivery of therapeutics into the brain: the potential of aptamers for targeted delivery. Biomedicines. 8 (5), 120 (2020).
- Wu, X., Chen, J., Wu, M., Zhao, J. X. Aptamers: active targeting ligands for cancer diagnosis and therapy. Theranostics. 5 (4), 322 (2015).
- Feng, X., et al. The aptamer functionalized nanocomposite used for prostate cancer diagnosis and therapy. Journal of Nanomaterials. 2022, (2022).
- Huang, J., et al. Advances in aptamer-based biomarker discovery. Frontiers in Cell and Developmental Biology. 9, 571 (2021).
- Ashrafuzzaman, M. Aptamers as both drugs and drug-carriers. BioMed Research International. 2014, (2014).
- Nakhjavani, M., Samarasinghe, R. M., Shigdar, S. Triple-negative breast cancer brain metastasis: an update on druggable targets, current clinical trials, and future treatment options. Drug Discovery Today. , (2022).
- Macdonald, J., et al. Development of a bifunctional aptamer targeting the transferrin receptor and epithelial cell adhesion molecule (EpCAM) for the treatment of brain cancer metastases. ACS Chemical Neuroscience. 8 (4), 777-784 (2017).
- Macdonald, J., et al. Bifunctional aptamer-doxorubicin conjugate crosses the blood-brain barrier and selectively delivers its payload to EpCAM-positive tumor cells. Nucleic Acid Therapeutics. 30 (2), 117-128 (2020).
- Shigdar, S., Agnello, L., Fedele, M., Camorani, S., Cerchia, L. Profiling cancer cells by cell-SELEX: use of aptamers for discovery of actionable biomarkers and therapeutic applications thereof. Pharmaceutics. 14 (1), 28 (2021).
- Rahimizadeh, K., et al. Development of cell-specific aptamers: recent advances and insight into the selection procedures. Molecules. 22 (12), 2070 (2017).
- Chen, M., et al. Development of cell-SELEX technology and its application in cancer diagnosis and therapy. International Journal of Molecular Sciences. 17 (12), 2079 (2016).
- Shigdar, S., et al. The use of sensitive chemical antibodies for diagnosis: detection of low levels of EpCAM in breast cancer. PLoS One. 8 (2), 57613 (2013).
- Ni, J., et al. Role of the EpCAM (CD326) in prostate cancer metastasis and progression. Cancer and Metastasis Reviews. 31 (3), 779-791 (2012).
- Ni, J., et al. Epithelial cell adhesion molecule (EpCAM) is associated with prostate cancer metastasis and chemo/radioresistance via the PI3K/Akt/mTOR signaling pathway. The International Journal of Biochemistry & Cell Biology. 45 (12), 2736-2748 (2013).
- McKeague, M., Kruse, P. F., Patterson, M. K., et al. . Tissue Culture. , 395-397 (1973).
- McKeague, M., et al. The minimum aptamer publication standards (MAPS guidelines) for de novo aptamer selection. Aptamers. 6, 10-18 (2022).
- Schoofs, G., Van Hout, A., D'huys, T., Schols, D., Van Loy, T. A flow cytometry-based assay to identify compounds that disrupt binding of fluorescently-labeled CXC Chemokine ligand 12 to CXC Chemokine receptor 4. Journal of Visualized Experiments. (133), e57271 (2018).
- McKinnon, K. M. Flow cytometry: an overview. Current Protocols in Immunology. 120 (1), 1-11 (2018).
- Kamiloglu, S., Sari, G., Ozdal, T., Capanoglu, E. Guidelines for cell viability assays. Food Frontiers. 1 (3), 332-349 (2020).
- Ruscito, A., DeRosa, M. C. Small-molecule binding aptamers: Selection strategies, characterization, and applications. Frontiers in Chemistry. 4, 14 (2016).
- McKeague, M., et al. Comprehensive analytical comparison of strategies used for small molecule aptamer evaluation. Analytical Chemistry. 87 (17), 8608-8612 (2015).
- Henri, J., Bayat, N., Macdonald, J., Shigdar, S. A guide to using nucleic acid aptamers in cell based assays. Aptamers. 23, (2019).
- Mao, H., et al. The mechanism and regularity of quenching the effect of bases on fluorophores: the base-quenched probe method. Analyst. 143 (14), 3292-3301 (2018).
- McKeague, M., et al. Analysis of in vitro aptamer selection parameters. Journal of Molecular Evolution. 81 (5), 150-161 (2015).
- Chen, B., et al. Targeting negative surface charges of cancer cells by multifunctional nanoprobes. Theranostics. 6 (11), 1887 (2016).
- Shigdar, S., et al. RNA aptamers targeting cancer stem cell marker CD133. Cancer Letters. 330 (1), 84-95 (2013).
- Amraee, M., Oloomi, M., Yavari, A., Bouzari, S. DNA aptamer identification and characterization for E. coli O157 detection using cell based SELEX method. Analytical Biochemistry. 536, 36-44 (2017).
- Yu, X. -. X., et al. Selection and characterization of a novel DNA aptamer, Apt-07S specific to hepatocellular carcinoma cells. Drug Design, Development and Therapy. 14, 1535 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados