Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Essai de liaison à une protéine de surface cellulaire basé sur la cytométrie en flux pour évaluer la sélectivité et la spécificité d’un aptamère anticancéreux
Dans cet article
Résumé
Une étape nécessaire dans le développement d’aptamères anticancéreux est de tester sa liaison à la cible. Nous démontrons un test basé sur la cytométrie en flux pour étudier cette liaison, en soulignant l’importance d’inclure un aptamère témoin négatif et des cellules cancéreuses positives ou négatives pour cette protéine particulière.
Résumé
Un défi clé dans le développement d’un aptamère anticancéreux est de déterminer efficacement la sélectivité et la spécificité de l’aptamère développé par rapport à la protéine cible. En raison de ses nombreux avantages par rapport aux anticorps monoclonaux, le développement d’aptamères a acquis une énorme popularité parmi les chercheurs sur le cancer. L’évolution systématique des ligands par enrichissement exponentiel (SELEX) est la méthode la plus courante de développement d’aptamères spécifiques aux protéines d’intérêt. Suite à SELEX, un test de liaison rapide et efficace accélère le processus d’identification, confirmant la sélectivité et la spécificité de l’aptamère.
Cet article explique un test de liaison étape par étape basé sur la cytométrie en flux d’un aptamère spécifique de la molécule d’adhésion cellulaire épithéliale (EpCAM). La glycoprotéine transmembranaire EpCAM est surexprimée dans la plupart des carcinomes et joue un rôle dans l’initiation, la progression et les métastases du cancer. Par conséquent, c’est un candidat précieux pour l’administration ciblée de médicaments aux tumeurs. Pour évaluer la sélectivité et la spécificité de l’aptamère à l’EpCAM lié à la membrane, des cellules EpCAM positives et négatives sont nécessaires. En outre, un aptamère EpCAM non contraignant avec une longueur et une structure 2 dimensions (2D) similaires à l’aptamère de liaison EpCAM est requis. Le test de liaison comprend différents tampons (tampon de blocage, tampon de lavage, tampon d’incubation et tampon FACS) et étapes d’incubation.
L’aptamère est incubé avec les lignées cellulaires. Après les étapes d’incubation et de lavage, les cellules seront évaluées à l’aide d’un test de cytométrie en flux sensible. L’analyse des résultats montre la liaison de l’aptamère spécifique à EpCAM aux cellules EpCAM positives et non aux cellules EpCAM négatives. Dans les cellules EpCAM positives, cela est représenté comme un décalage de bande dans la liaison de l’aptamère EpCAM vers la droite par rapport au contrôle de l’aptamère non liant. Dans les cellules EpCAM négatives, les bandes correspondantes d’aptamères liant et non liant EpCAM se chevauchent. Cela démontre la sélectivité et la spécificité de l’aptamère EpCAM. Bien que ce protocole soit axé sur l’aptamère EpCAM, il est applicable à d’autres aptamères publiés.
Introduction
Le cancer reste l’une des principales causes de mortalité dans le monde1. Malgré l’amélioration significative du traitement du cancer au cours des dernières décennies, le développement de médicaments anticancéreux est toujours un sujet très débattu. En effet, la chimiothérapie, en tant que pilier du traitement du cancer, s’accompagne d’effets secondaires graves qui limitent l’observance du traitement par le patient. De plus, la résistance du cancer induite par la chimiothérapie au traitement a limité son application en tant que seul choix d’intervention médicale. L’application d’anticorps monoclonaux (mAbs) a introduit une réponse accrue aux traitements contre le cancer2. La raison d’être de l’utilisation des AcM était d’améliorer l’efficacité des agents chimiothérapeutiques et de minimiser leurs effets indésirables. Cependant, l’administration des AcM est également devenue un défi. Cela n’était pas seulement dû aux réactions immunologiques induites par AcM, mais aussi aux coûts de production élevés et dépendants des animaux et aux conditions de stockagedifficiles 3. L’introduction des aptamères dans les années 19904 a suscité de nouveaux espoirs dans le traitement du cancer, car l’application des aptamères pourrait relever les défis associés aux mAbs.
Les aptamères sont de courtes séquences d’acides nucléiques qui sont spécifiquement produites pour une certaine cible. L’évolution systématique des ligands par enrichissement exponentiel (SELEX) est une méthode courante dans la production d’aptamères. Dans SELEX, la protéine d’intérêt est incubée avec une bibliothèque de séquences nucléotidiques aléatoires, et à travers une série de cycles itératifs, l’aptamère spécifique à cette protéine est purifié. Les aptamères ont une sélectivité et une spécificité de cible similaires à celles des mAb, et par conséquent, le développement de médicaments dans ce domaine montre des applications futures prometteuses. Les aptamères spécifiques aux biomarqueurs du cancer pourraient être appliqués comme médicaments uniques et outils de diagnostic du cancer 5,6,7. En raison de leur structure nanométrique, ces aptamères pourraient également agir comme porteurs de médicaments pour délivrer des agents cytotoxiques spécifiquement à la tumeur8. Cela augmenterait l’efficacité de l’administration ciblée de médicaments et diminuerait les effets indésirables non ciblés associés à la chimiothérapie. De plus, ces nanomédicaments ont une pénétration tissulaire élevée, ce qui en fait un candidat souhaitable pour l’administration et le traitement de médicaments contre les tumeurs profondes. Les aptamères peuvent également être conçus pour cibler les transporteurs exprimés sur la barrière hémato-encéphalique (BHE) afin d’améliorer l’administration de médicaments aux tumeurs cérébrales9. Un bon exemple d’un tel aptamère sont les aptamères bifonctionnels, ciblant le récepteur de la transferrine (TfR)10 pour améliorer l’administration de médicaments à travers la BHE, et délivrant une charge utile de médicament cytotoxique aux cellules tumorales11.
Malgré tous les avantages des aptamères, le développement de médicaments dans ce domaine n’a pas encore produit de médicament anticancéreux commercialisé et efficace. L’une des raisons pourrait être le manque de méthodes standard et reproductibles qui pourraient être suivies à l’échelle mondiale par les chercheurs dans le domaine. Dans cet article, un protocole étape par étape d’un aptamère se liant à une protéine native exprimée à la surface de la cellule est démontré. Ce protocole est une étape préalable à l’évaluation préclinique des aptamères anticancéreux. Le test est effectué pour montrer la sélectivité et la spécificité de l’aptamère purifié recueilli à partir de SELEX ou d’une séquence d’aptamères publiée pour confirmer la sélectivité et la spécificité. Ce test basé sur la cytométrie en flux est un test rapide, fiable et sensible qui montre avec précision la sélectivité et la spécificité de l’aptamère, où l’aptamère est testé contre les protéines à la surface de la cellule12,13,14. Cette méthode est démontrée à l’aide de la liaison d’un aptamère spécifique à l’EpCAM illustrée dans cet article15. EpCAM, en tant que glycoprotéine transmembranaire, joue un rôle dans la signalisation, la progression, la migration et les métastases des cellules tumorales16,17. Pour montrer la sélectivité et la spécificité de cet aptamère, des cellules cancéreuses EpCAM positives et négatives ont été utilisées. L’aptamère spécifique à l’EpCAM développé précédemment, TEPP (5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′), et un aptamère témoin négatif, TENN (5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3), ont été utilisés comme aptamères de liaison et non liant EpCAM, respectivement10. L’extrémité 3' du TEPP et du TENN a été marquée avec un fluorophore TYE665.
TEPP est un aptamère bifonctionnel qui cible EpCAM d’une extrémité et TfR de l’autre. Cela a fait de TEPP un candidat approprié pour l’administration de médicaments aux tumeurs cérébrales EpCAM+ . À l’aide de son extrémité spécifique au TfR, le TEPP traverse la barrière hémato-encéphalique et, à l’aide de l’extrémité spécifique à l’EpCAM, trouve la tumeur et délivre sa cargaison (par exemple, les médicaments cytotoxiques) à la tumeur. TENN a une longueur et une structure 2D similaires à celles du TEPP, mais il n’a pas d’affinité pour l’EpCAM ou le TfR, et est donc un aptamère de contrôle négatif approprié. À l’aide de TEPP et de TENN, le test de la liaison d’un aptamère à la protéine cible à l’aide de la cytométrie en flux est présenté dans cet article. Ce protocole s’applique au développement d’aptamères spécifiques aux cellules. Elle est également applicable à d’autres analyses complémentaires et de confirmation des séquences d’aptamères disponibles dans la littérature. Le protocole peut également être utilisé par les nouveaux arrivants dans le domaine des aptamères qui envisagent d’utiliser un aptamère déjà publié à des fins de recherche et développement (R & D). Dans cet article, deux séquences d’aptamères disponibles dans la littérature sont étudiées.
Protocole
REMARQUE : Avant de commencer l’expérience, portez un équipement de protection individuelle, y compris une blouse de laboratoire, des gants et des lunettes de protection. Consultez le tableau des matériaux pour plus de détails sur les matériaux, les réactifs, l’équipement et les logiciels utilisés dans ce protocole.
1. Tampons requis pour le test
- Préparez les tampons requis pour cette expérience - le tampon SELEX requis pour le pliage des aptamères, le tampon de blocage (BB), le tampon de lavage (WB) et le tampon de liaison (BiB) (tableau 1) - fraîchement le jour de l’expérience et conservez-les sur la glace ou à 4 °C.
REMARQUE: Chaque aptamère nécessite une condition de pliage unique. Cela inclut le tampon SELEX et les conditions de température de pliage. Il faut prendre soin de reproduire complètement les méthodes de l’article original décrivant l’aptamère10. Dans cette expérience, tous les tampons sont préparés dans la solution saline tamponnée au phosphate (DPBS) de Dulbecco. Le volume tampon requis dans chaque expérience dépend du nombre de lignées cellulaires, du nombre de répétitions et du nombre de concentrations d’aptamères testées.
| Ingrédients | Volume requis | ||
| Article | Concentration | ||
| Tampon SELEX | MgCl2 | 5 mM | 50 μL par échantillon + 10 % d’erreur de pipetage |
| Blocage de la mémoire tampon | MgCl2 | 5 mM | 500 μL par lignée cellulaire |
| BSA a | 1 mg/mL | ||
| ARNt b | 0,1 mg/mL | ||
| FBS c | 10 % (v/v) | ||
| Tampon de lavage | MgCl2 | 5 mM | 1 mL pour le premier lavage + 100 μL par échantillon d’essai + 10 % d’erreur de pipetage |
| Tampon de liaison | MgCl2 | 5 mM | 50 μL par échantillon + 10 % d’erreur de pipetage |
| BSA | 2 mg/mL | ||
| Trna | 0,2 mg/mL | ||
| FBS (en anglais seulement) | 20 % (v/v) | ||
Tableau 1 : Tampons requis pour le test de liaison. un Albumine sérique bovine, bAcide ribonucléique de transfert, cSérum bovin fœtal.
2. Préparation des aptamères
REMARQUE: Les aptamères utilisés dans le test sont marqués avec une molécule rapporteur de fluorescence, et donc des précautions doivent être prises pour les protéger de la lumière.
- Avant l’expérience, préparer un stock de 100 μM (stock A) d’aptamères d’essai et de contrôle en utilisant de l’eau ultrapure exempte de pyrogène et de RNase (Figure 1).
NOTE: Pour une conservation à long terme, le stock A doit être conservé au congélateur à -20 °C. - Préparer le stock B comme concentration utile des aptamères en diluant le stock A à l’aide du tampon SELEX (tableau 1). Pour suivre ce protocole, diluer le stock A à un stock de 1 000 nM pour préparer le stock B (Figure 1).
- Pour préparer l’aptamère à la formation de la structure tridimensionnelle (3D), dans un tube de 250 μL, diluer le stock B avec le tampon SELEX pour préparer le volume et la concentration requis de l’aptamère pour le pliage.
REMARQUE: Les aptamères pliés seront exposés à un volume égal de cellules. Par conséquent, la concentration de l’aptamère réglé pour le pliage doit être 2x plus concentrée que la concentration finale souhaitée. Utilisez l’équation (1) pour calculer les volumes et les concentrations requis. N’oubliez pas de considérer un volume supplémentaire de 10% pour l’erreur de pipetage.
Stock de concentration A × Stock de volumeA = Stockde concentration B ×Stock de volumeB (1)
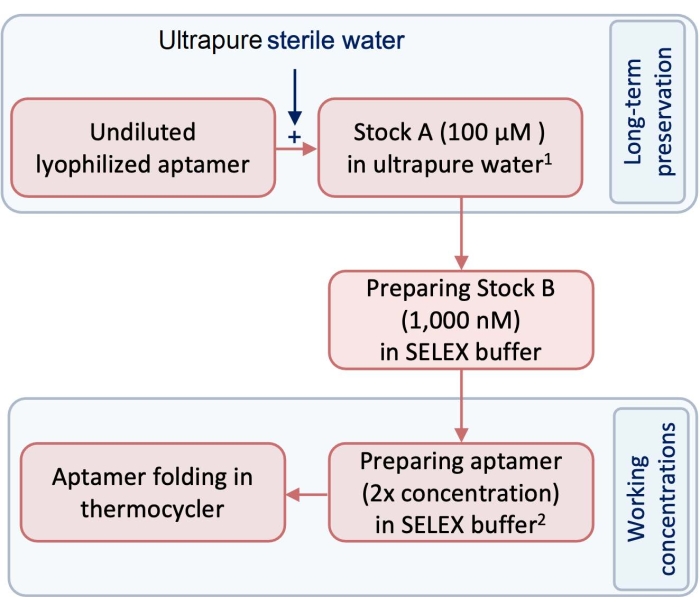
Figure 1 : Schéma montrant les étapes de préparation des aptamères. 1 Stock 1est stocké à -20 °C pour une conservation à long terme. deux Les concentrations de travail sont préparées dans un tampon SELEX et ne sont pas stockées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Maintien des cellules cancéreuses
REMARQUE: Avant de commencer l’étude, assurez-vous que les cellules sont à leur nombre de passages précoces, montrent leurs caractéristiques morphologiques typiques et sont exemptes de mycoplasmes. Pour tester la sélectivité et la spécificité de l’aptamère, des lignées cellulaires hautes, modérées et faibles/négatives exprimatrices de la protéine d’intérêt sont idéalement nécessaires.
- Ensemencer les cellules dans une fiole de culture T75 en utilisant les conditions de culture appropriées. Cultivez-les dans un incubateur humidifié à 5% de CO2 , à 37 °C.
REMARQUE : Dans cette étude, le milieu aigle modifié (DMEM) de Dulbecco complété par 10 % de sérum bovin fœtal (milieu complet) a été utilisé. - Lorsque les cellules atteignent ~80% de confluence, les faire passer dans une nouvelle fiole contenant un milieu complet frais.
REMARQUE: Selon la protéine d’intérêt et la lignée cellulaire, une confluence de 80% pourrait fournir une population cellulaire appropriée pour le test de liaison. Pour les lignées cellulaires de cette expérience, MDA-MB-231 et HEK 293T, une confluence de 80% convient. À ce stade, passez à la section 4, le dosage contraignant. Vérifiez toujours l’expression de la protéine d’intérêt, en utilisant des AcM spécifiques à cette protéine.
4. Dosage de liaison
REMARQUE : La figure 2 résume les étapes requises dans l’essai de liaison dans les cellules adhérentes.
- Dans une enceinte de biosécurité de classe II, recueillir les cellules de chaque fiole dans des tubes comme suit:
- Recueillir et jeter le média dans le flacon, ajouter 2 mL de PBS, l’étaler sur les cellules, puis recueillir et jeter le PBS. Répétez cette étape deux fois de plus pour supprimer toutes les traces de milieux qui peuvent inactiver la trypsine. Ajouter 1 mL de 0,25 % de trypsine/EDTA dans chaque fiole et incuber pendant 5 à 10 minutes à 37 °C. Visualiser le détachement des cellules au microscope.
- Ajouter 1 mL de milieu complet aux cellules et pipeter les cellules de haut en bas pour obtenir une suspension unicellulaire. Introduire les cellules dans un tube approprié et centrifuger à 200 × g pendant 5 min.
NOTE: Pour les cellules non adhérentes, recueillir les cellules dans un tube, centrifuger (200 × g, 5 min) et passer à l’étape 4.1.3. - Jeter le surnageant et remettre les cellules en suspension dans 1 mL de milieu frais. Compter les cellules en utilisant la coloration au bleu de trypan, en diluant un certain volume de suspension cellulaire avec du bleu de trypan. Répartir ~15 μL du mélange entre un hémocytomètre et un verre de couverture. Comptez les cellules comme décrit précédemment18, en utilisant l’équation (2):
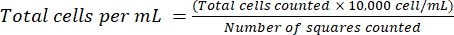 (2)
(2)
NOTA: Utiliser le volume minimum possible de suspension cellulaire et prendre note du facteur de dilution. Par exemple, mélanger des volumes égaux de suspension cellulaire et de bleu de trypan à 0,04% donne un facteur de dilution de 2. Assurez-vous d’une viabilité élevée (cellules vivantes / cellules totales × 100) de ~90% pour la plupart des lignées cellulaires adhérentes avant de continuer. Les cellules mortes absorbent non spécifiquement les aptamères et modifient les résultats19. Il est possible d’utiliser d’autres techniques de comptage de cellules, telles que l’utilisation d’un compteur de cellules. - Prélever le nombre requis de cellules, en vous assurant d’avoir 10 × 10à 4 cellules par échantillon d’essai. Envisagez un volume supplémentaire de 10% pour l’erreur de pipetage.
REMARQUE: Il est important de toujours garder le même nombre de cellules entre les expériences et les répétitions. - Incuber les cellules à 37 °C pendant 2 h pour permettre la stabilisation de la protéine d’intérêt sur la membrane cellulaire suite au décollement enzymatique.
NOTE: Cette période d’incubation peut différer selon la protéine d’intérêt.
- Au cours de cette incubation de 2 h :
- Réglez la température de la centrifugeuse à 4 °C. Laisser décongeler l’ARNt et les stocks d’aptamère à température ambiante ou sur de la glace. Pour protéger la molécule rapporteur de fluorescence, protégez les tubes aptamères de la lumière.
REMARQUE: Le rôle de l’ARNt est de bloquer les sites de liaison aux acides nucléiques. - Préparez le tampon SELEX, BB, WB et BiB (voir section 1), en les gardant tous sur la glace ou à 4 °C. Réglez la machine thermocycleuse sur un cycle vide. Placez une plaque noire de 96 puits et des tubes de cytométrie en flux sur de la glace.
REMARQUE: Le réglage du thermocycleur sur un cycle vide prépare le système de refroidissement et de chauffage et aide à générer des résultats plus reproductibles.
- Réglez la température de la centrifugeuse à 4 °C. Laisser décongeler l’ARNt et les stocks d’aptamère à température ambiante ou sur de la glace. Pour protéger la molécule rapporteur de fluorescence, protégez les tubes aptamères de la lumière.
- Après l’incubation de 2 h, centrifuger les cellules à 500 × g pendant 5 min. Jeter le surnageant et remettre les cellules en suspension dans 500 μL de BB. Incuber les cellules à 4 °C pendant 30 min avec un mélange intermittent.
- Pendant cette incubation de 30 minutes, effectuez le pliage des aptamères comme suit:
- Constituer les concentrations 2x des aptamères (voir rubrique 2), puis mélanger et incuber les aptamères dans la thermocycleuse, en fonction des conditions de pliage requises. Pour cet aptamère EpCAM, utilisez les conditions de pliage suivantes de 95 °C, 5 min, suivies de 22 °C, 10 min et 37 °C, 15 min.
REMARQUE : Incluez toujours un contrôle négatif (c.-à-d. un tampon SELEX sans aptamères).
- Constituer les concentrations 2x des aptamères (voir rubrique 2), puis mélanger et incuber les aptamères dans la thermocycleuse, en fonction des conditions de pliage requises. Pour cet aptamère EpCAM, utilisez les conditions de pliage suivantes de 95 °C, 5 min, suivies de 22 °C, 10 min et 37 °C, 15 min.
- Après l’incubation de 30 minutes, centrifuger les cellules (500 × g, 5 min, 4 °C), retirer le surnageant, ajouter 1 mL de WB et centrifuger à nouveau les cellules (500 × g, 5 min, 4 °C). Retirer le surnageant et remettre les cellules en suspension dans un volume approprié de BiB.
- Pipeter 50 μL des cellules remises en suspension dans chaque puits d’une plaque noire glacée de 96 puits. Gardez les cellules sur la glace pour inhiber l’internalisation de la protéine d’intérêt.
- Pipeter 50 μL des aptamères sur un volume de cellules de 50 μL, mélanger et incuber dans l’obscurité à 4 °C pendant 30 min. Centrifuger la plaque à 500 × g, 5 min, 4 °C, et retirer délicatement le surnageant.
- Remettez délicatement la pastille en suspension dans WB et centrifugez à 500 × g pendant 5 min. Répéter l’étape de lavage (4.7) 2x et remettre en suspension dans 100 μL de WB pour l’analyse cytométrique en flux.
REMARQUE : Voir la figure 3 pour un diagramme des interactions entre les aptamères et les cellules.

Figure 2 : Diagramme illustrant les étapes de la réalisation d’un test de liaison aptamère-protéine. Abréviations : SELEX = évolution systématique des ligands par enrichissement EXponentiel ; BB = tampon de blocage; WB = Tampon de lavage; BiB = Tampon de liaison. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Diagramme montrant les différents types de cellules et d’aptamères nécessaires pour effectuer le test de liaison des aptamères. Abréviation : EpCAM = molécule d’adhésion cellulaire épithéliale. Cette figure a été créée à l’aide de Biorender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
5. Cytométrie en flux et analyse des données
REMARQUE: Avant d’allumer le cytomètre en flux, assurez-vous qu’il n’y a pas de « bulles » dans les unités de filtration à membrane pour la solution d’arrêt, la solution de nettoyage et le liquide de gaine (0,9% NaCl). « Saigner » bulles s’il y a des bulles dans les capsules. Assurez-vous que le conteneur à déchets est vide et que les récipients de liquide de gaine, d’eau et d’eau de Javel à 1% dans de l’eau ultrapure sont pleins.
- Allumez le cytomètre en flux, puis l’ordinateur.
REMARQUE: Les détails de l’utilisation du cytomètre en flux expliqués ici sont spécifiques à la machine et au logiciel présentés dans la vidéo (voir le tableau des matériaux). D’autres logiciels nécessiteraient une formation appropriée pour être utilisés. - Ouvrez le logiciel d’analyse de cytométrie en flux, connectez-vous au programme et, sous l’onglet Cytomètre , exécutez Fluidics Start up.
- Pour créer une nouvelle expérience, sous l’onglet Expérience , cliquez sur Nouveau dossier et nommez le dossier /l’expérience de manière appropriée.
- Cliquez sur Nouveau dossier à mettre en surbrillance, puis sous l’onglet Expérience à nouveau, cliquez sur Nouvelle expérience et nommez l’expérience de manière appropriée.
- Pour ajouter le premier échantillon/échantillon, sous l’onglet Expérience , cliquez sur Nouveau spécimen et nommez ce spécimen de manière appropriée (nom de la lignée cellulaire/de l’échantillon témoin/de l’échantillon expérimental).
- Pour ajouter un échantillon de tube, mettez l’échantillon en surbrillance (groupe) et sous l’onglet expérience , cliquez sur Nouveau tube. Ajouter le nombre approprié de tubes et le nom.
- Pour préparer les graphiques requis, sous l’onglet feuille de calcul , ouvrez une nouvelle feuille de calcul. Une fois que la nouvelle fenêtre de feuille de calcul apparaît, ouvrez ce qui suit à l’aide de l’écran de la feuille de calcul (passez la souris sur le logo / les images pour trouver les noms):
- Préparez un graphique par transfert de points de la dispersion vers l’avant (FSC) par rapport à la dispersion latérale (SCC) pour sélectionner la population d’intérêt. Définissez la première porte en identifiant et en sélectionnant la population d’intérêt (P1) dans un diagramme de densité de dispersion vers l’avant et sur le côté. Exclure les débris, qui constituent la population ayant le signal de diffusion vers l’avant le plus faible.
REMARQUE: Le paramètre FSC détecte les cellules ou les événements en fonction de leur taille et le SCC les discrimine en fonction de leur granularité20. - Préparez un graphique par transfert de points de la surface FSC (FSC-A) par rapport à la hauteur FSC (FSC-H) pour sélectionner la population unicellulaire. Définissez la deuxième porte en excluant les populations de doublets cellulaires, car les cellules doublet affectent considérablement les résultats et les conclusions. Excluez les doublets en utilisant des diagrammes de densité FSC-H par rapport à FSC-A, où les cellules de même taille montrent une surface et une hauteur similaires. Par conséquent, les singlets sont regroupés en diagonale et séparés des doublets.
REMARQUE: FSC est à peu près proportionnel à la taille de la cellule. Les impulsions de tension sont définies comme FSC-H, l’intensité du signal, la largeur FSC qui reflète la taille de la cellule et la durée du signal, et FSC-A, qui est H × W. Les doublets ont une double largeur et valeur de surface; par conséquent, le contrôle pour les singulets est basé sur la détection des disproportions entre H, W et A causées par les doublets. - Préparez un histogramme du nombre d’événements contre le fluorophore d’intérêt.
- Préparez un graphique par transfert de points de la dispersion vers l’avant (FSC) par rapport à la dispersion latérale (SCC) pour sélectionner la population d’intérêt. Définissez la première porte en identifiant et en sélectionnant la population d’intérêt (P1) dans un diagramme de densité de dispersion vers l’avant et sur le côté. Exclure les débris, qui constituent la population ayant le signal de diffusion vers l’avant le plus faible.
- Avant de commencer la cytométrie en flux, assurez-vous que le tableau de bord d’acquisition pour contrôler l’acquisition de l’échantillon, l’inspecteur et le cytomètre pour ajuster les paramètres de tension, ainsi que la feuille de calcul avec tous les graphiques sont ouverts.
REMARQUE : Au moins 100 μL d’une suspension de 10 × 104 cellules dans un tube de cytométrie en flux est nécessaire pour effectuer l’analyse. En particulier en cas de viabilité inférieure, une coloration à l’iodure de propidium peut être effectuée pour sélectionner la population cellulaire viable21,22. - Pour exécuter le premier échantillon, sur le côté gauche de l’écran, assurez-vous que la flèche pointant vers le tube est verte. Si cette flèche n’est pas verte, cliquez sur la flèche pour la rendre verte.
- À l’aide d’une pipette, transférer chaque échantillon de la plaque noire à 96 puits dans un tube de cytométrie en flux. Faire fonctionner l’échantillon témoin non traité et non taché à basse vitesse.
- Dans le tableau de bord d’acquisition, choisissez un nombre approprié d’événements à enregistrer (30 000), définissez le débit sur faible, puis cliquez sur Acquérir des données.
- Ajustez la tension pour les paramètres FSC et SCC. Assurez-vous que la population cellulaire est centralisée dans le diagramme à points et qu’aucune cellule ne touche l’un ou l’autre axe du graphique pour éviter de perdre les cellules d’intérêt.
- Augmenter la vitesse d’acquisition à moyenne ou élevée pour analyser les échantillons plus rapidement mais ne pas dépasser plus de 200 événements/s. Ensuite, cliquez sur Enregistrer les données.
- Effectuez le contrôle pour P1 (Figure 4A) et la population unicellulaire (Figure 4B). Construire l’histogramme des événements par rapport au fluorochrome utilisé et sélectionner P1 en fonction des données (allophycocyanine (APC) dans ce cas) (Figure 4C).
- Après avoir réglé la tension, déclenché et enregistré les données, retirez l’échantillon et cliquez sur Next Tube (Tube suivant).
- Insérez l’échantillon suivant et répétez l’enregistrement des données pour tous les échantillons témoins et d’essai (Figure 3).
- Une fois toutes les données recueillies, lavez le cytomètre en flux en faisant fonctionner trois tubes d’eau de Javel à 50%, de rinçage FACS et d’eau ultrapure, chacun pendant 5 minutes à un débit élevé.
- Ensuite, dans le menu déroulant Cytomètre , cliquez sur Fluidics Shut Down.
- Avant de fermer le logiciel et d’éteindre la machine et l’ordinateur, exportez les résultats sous forme de fichiers .fcs vers un lecteur USB pour les transférer et les analyser, comme suit :
- Dans le logiciel d’analyse, appuyez sur le bouton NOUVEAU pour créer un nouveau document et une nouvelle fenêtre pour gérer l’analyse. Faites glisser les fichiers d’exemple dans la nouvelle fenêtre.
- Double-cliquez pour ouvrir l’échantillon non coloré. Choisissez la population P1, double-cliquez sur la population P1 pour créer un graphique FSC-H versus FSC-A, et fermez la population unicellulaire.
- Double-cliquez sur les cellules individuelles fermées pour créer un histogramme des événements par rapport au fluorochrome utilisé.
- Dans la fenêtre d’origine, sélectionnez P1 et cellules simples et faites-les glisser vers Tous les échantillons afin que tous les échantillons contiennent désormais le même point de contrôle.
- Cliquez sur le bouton Editeur de mise en page pour ouvrir la fenêtre Layouts . Faites glisser deux exemples (contrôle et test) l’un sur l’autre pour créer un histogramme de superposition.
Résultats
Un aspect important de la découverte et du développement de nouveaux médicaments est d’assurer la sélectivité et la spécificité du candidat médicament. Cela signifie que le candidat médicament devrait être capable de discriminer entre différentes cellules et n’affecter que la population cellulaire d’intérêt (sélectivité). La sélectivité est étudiée à l’aide de lignées cellulaires qui diffèrent en termes d’expression de la protéine d’intérêt. Dans cette étude, les lignées cellulaires...
Discussion
Le principal défi avec le développement de nouveaux aptamères est l’absence de lignes directrices standard qui s’appliquent aux différentes étapes de ce processus. McKeague et coll. ont récemment démontré certains des défis connexes, qui mènent à des présentations peu claires des données dans les publications et à l’échec de la reproduction de la recherche. Ils ont proposé des lignes directrices fondamentales nécessaires à la caractérisation des aptamères19. Un test de li...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs reconnaissent le financement SEED de l’Institute for Mental and Physical Health and Clinical Translation (IMPACT), le programme « Alfred Deakin Postdoctoral Research Fellowship » de l’Université Deakin et la « Australian Government Research Training Program Scholarship ».
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes with attached lid | Sigma-Aldrich | T6649 | |
| 15 mL CellStar blue screw cap, conical bottom tube | Greiner Bio One | 188271 | |
| 5 mL serological pipettes | Greiner Bio One | 606180 | |
| BD FACSCanto II Flow Becton Dickinson Cytometer | Becton Dickinson | N/A | |
| BD FACSDiva V9.0 | BD Biosciences | N/A | |
| Bovine Serum Albumin (BSA), Lyophilized powder | Sigma-AldrichTM | A7906-50G | |
| Bright-line Hemocytometer | Sigma-Aldrich | Z359629 | |
| Dulbecco’s Modified Eagle Medium (DMEM) High Glucose Media Powder | Life Technologies | 12100046 | |
| Dulbecco’s Phosphate- Buffered Saline (DPBS) | Life Technologies | 21300025 | |
| FlowJo, LLC 10.8.1 | BD Biosciences | N/A | |
| Foetal Bovine Serum (FBS) | Bovogen | SFBS-F | |
| HEK293T | American Type Culture Collection | ACS-4500 | |
| Heracell 150i CO2 Incubator | Thermo Fisher Scientific | N/A | |
| Heraeus Megafuge 16R Centrifuge | Thermo Fisher Scientific | N/A | |
| Magnesium Chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| MDA-MB-231 | American Type Culture Collection | CRM-HTB-26 | |
| Microplate, PS, 96 well, F-bottom (Chimney well), Black | Greiner Bio One | 655076 | |
| MiniAmp Thermal Cycler | Thermo Fisher Scientific | A37834 | |
| Phosphate-Buffered Saline (PBS) tablets | Life Technologies | 18912014 | |
| Pyrogen- and RNase-free ultrapure water | Milli-Q | ||
| T75 Cell Culture flask | Cellstar | 658170 | |
| TENN | Integrated DNA Technologies | N/A | 5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3 |
| TEPP | Integrated DNA Technologies | N/A | 5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′ |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8508-5X1ML | |
| Trypan Blue Solution | Life Technologies | 15250061 | |
| Trypsin-EDTA | Gibco | 15400054 |
Références
- Cancer. World Health Organization Available from: https://www.who.int/news-room/fact-sheets/detail/cancer#:~:text=Cancer%20is%20a%20leading%20cause.and%20rectum%20and%20prostate%20cancers (2022)
- Liu, J. K. H. The history of monoclonal antibody development - Progress, remaining challenges and future innovations. Annals of Medicine and Surgery. 3 (4), 113-116 (2014).
- Nakhjavani, M., Shigdar, S. Future of PD-1/PD-L1 axis modulation for the treatment of triple-negative breast cancer. Pharmacological Research. 175, 106019 (2022).
- Bukari, B., Samarasinghe, R. M., Noibanchong, J., Shigdar, S. L. Non-invasive delivery of therapeutics into the brain: the potential of aptamers for targeted delivery. Biomedicines. 8 (5), 120 (2020).
- Wu, X., Chen, J., Wu, M., Zhao, J. X. Aptamers: active targeting ligands for cancer diagnosis and therapy. Theranostics. 5 (4), 322 (2015).
- Feng, X., et al. The aptamer functionalized nanocomposite used for prostate cancer diagnosis and therapy. Journal of Nanomaterials. 2022, (2022).
- Huang, J., et al. Advances in aptamer-based biomarker discovery. Frontiers in Cell and Developmental Biology. 9, 571 (2021).
- Ashrafuzzaman, M. Aptamers as both drugs and drug-carriers. BioMed Research International. 2014, (2014).
- Nakhjavani, M., Samarasinghe, R. M., Shigdar, S. Triple-negative breast cancer brain metastasis: an update on druggable targets, current clinical trials, and future treatment options. Drug Discovery Today. , (2022).
- Macdonald, J., et al. Development of a bifunctional aptamer targeting the transferrin receptor and epithelial cell adhesion molecule (EpCAM) for the treatment of brain cancer metastases. ACS Chemical Neuroscience. 8 (4), 777-784 (2017).
- Macdonald, J., et al. Bifunctional aptamer-doxorubicin conjugate crosses the blood-brain barrier and selectively delivers its payload to EpCAM-positive tumor cells. Nucleic Acid Therapeutics. 30 (2), 117-128 (2020).
- Shigdar, S., Agnello, L., Fedele, M., Camorani, S., Cerchia, L. Profiling cancer cells by cell-SELEX: use of aptamers for discovery of actionable biomarkers and therapeutic applications thereof. Pharmaceutics. 14 (1), 28 (2021).
- Rahimizadeh, K., et al. Development of cell-specific aptamers: recent advances and insight into the selection procedures. Molecules. 22 (12), 2070 (2017).
- Chen, M., et al. Development of cell-SELEX technology and its application in cancer diagnosis and therapy. International Journal of Molecular Sciences. 17 (12), 2079 (2016).
- Shigdar, S., et al. The use of sensitive chemical antibodies for diagnosis: detection of low levels of EpCAM in breast cancer. PLoS One. 8 (2), 57613 (2013).
- Ni, J., et al. Role of the EpCAM (CD326) in prostate cancer metastasis and progression. Cancer and Metastasis Reviews. 31 (3), 779-791 (2012).
- Ni, J., et al. Epithelial cell adhesion molecule (EpCAM) is associated with prostate cancer metastasis and chemo/radioresistance via the PI3K/Akt/mTOR signaling pathway. The International Journal of Biochemistry & Cell Biology. 45 (12), 2736-2748 (2013).
- McKeague, M., Kruse, P. F., Patterson, M. K., et al. . Tissue Culture. , 395-397 (1973).
- McKeague, M., et al. The minimum aptamer publication standards (MAPS guidelines) for de novo aptamer selection. Aptamers. 6, 10-18 (2022).
- Schoofs, G., Van Hout, A., D'huys, T., Schols, D., Van Loy, T. A flow cytometry-based assay to identify compounds that disrupt binding of fluorescently-labeled CXC Chemokine ligand 12 to CXC Chemokine receptor 4. Journal of Visualized Experiments. (133), e57271 (2018).
- McKinnon, K. M. Flow cytometry: an overview. Current Protocols in Immunology. 120 (1), 1-11 (2018).
- Kamiloglu, S., Sari, G., Ozdal, T., Capanoglu, E. Guidelines for cell viability assays. Food Frontiers. 1 (3), 332-349 (2020).
- Ruscito, A., DeRosa, M. C. Small-molecule binding aptamers: Selection strategies, characterization, and applications. Frontiers in Chemistry. 4, 14 (2016).
- McKeague, M., et al. Comprehensive analytical comparison of strategies used for small molecule aptamer evaluation. Analytical Chemistry. 87 (17), 8608-8612 (2015).
- Henri, J., Bayat, N., Macdonald, J., Shigdar, S. A guide to using nucleic acid aptamers in cell based assays. Aptamers. 23, (2019).
- Mao, H., et al. The mechanism and regularity of quenching the effect of bases on fluorophores: the base-quenched probe method. Analyst. 143 (14), 3292-3301 (2018).
- McKeague, M., et al. Analysis of in vitro aptamer selection parameters. Journal of Molecular Evolution. 81 (5), 150-161 (2015).
- Chen, B., et al. Targeting negative surface charges of cancer cells by multifunctional nanoprobes. Theranostics. 6 (11), 1887 (2016).
- Shigdar, S., et al. RNA aptamers targeting cancer stem cell marker CD133. Cancer Letters. 330 (1), 84-95 (2013).
- Amraee, M., Oloomi, M., Yavari, A., Bouzari, S. DNA aptamer identification and characterization for E. coli O157 detection using cell based SELEX method. Analytical Biochemistry. 536, 36-44 (2017).
- Yu, X. -. X., et al. Selection and characterization of a novel DNA aptamer, Apt-07S specific to hepatocellular carcinoma cells. Drug Design, Development and Therapy. 14, 1535 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon