このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
Lifespan Machineを用いたハイスループット行動老化および寿命アッセイ(英語)
要約
イメージングプラットフォーム「The Lifespan Machine」は、大規模な個体群の生涯にわたる観察を自動化します。寿命、耐ストレス性、病因、および行動老化アッセイを実行するために必要な手順を示します。データの質と範囲により、研究者は生物学的および環境的変動が存在するにもかかわらず、老化への介入を研究することができます。
要約
一定の環境で飼育された遺伝的に同一の動物は、寿命の広い分布を示し、研究対象のすべての生物にわたって保存された老化に対する大きな非遺伝的、確率的側面を反映しています。この確率的要素は、老化を理解し、寿命を延ばしたり健康を改善したりする成功した介入を特定するために、研究者は実験動物の大規模な集団を同時に監視する必要があることを意味します。従来の手作業による死亡スコアリングでは、大規模な仮説検証に必要なスループットとスケールが制限されるため、ハイスループットの寿命アッセイのための自動化されたメソッドが開発されています。Lifespan Machine(LSM)は、線虫の生涯にわたる追跡のために、改造されたフラットベッドスキャナーとカスタム画像処理およびデータ検証ソフトウェアを組み合わせたハイスループットイメージングプラットフォームです。このプラットフォームは、大規模な動物集団から、前例のない規模で、経験豊富な研究者が手動で行うアッセイと同等の統計的精度と精度で、高度に時間分解された寿命データを生成することにより、大きな技術的進歩を構成します。最近では、加齢に伴う行動や形態の変化を定量化し、寿命に結びつけるためにLSMがさらに発展しています。ここでは、LSMを使用して自動化されたライフスパン実験を計画、実行、および分析する方法について説明します。さらに、行動データの収集と高品質の生存曲線の収集を成功させるために必要な重要なステップを強調します。
概要
老化は、生物の生理学的機能の低下を特徴とする複雑で多面的なプロセスであり、時間の経過とともに病気や死亡のリスクが高まります1。寿命は、出生または成人期の開始から死亡までの時間として測定され、老化の明確な結果2 と、集団間の相対的な老化率を測定するための間接的ではあるが厳密に定量的なプロキシを提供します3。老化研究は、介入に曝露された1つの集団と曝露されていない対照群の間の結果を比較するために、臨床試験と同様に寿命の正確な測定に依存することがよくあります。残念なことに、再現性の問題は老化研究に蔓延しており、統計的に検出力が不十分な実験 4や、環境の微妙な変化に対する寿命アッセイの固有の感度が原因であることがよくあります5。ロバストな実験には、大規模な母集団の複数回の反復が必要であり、このプロセスは、自動化6によって提供される実験のスケーラビリティから特に恩恵を受けます。
寿命アッセイの厳しい要求は、老化プロセス自体の予測不可能性に起因しています。同一の環境に収容された同質の個体は、異なる死亡時間と生理学的低下率を示し7、寿命には高度な確率性が関与していることを示唆している7,8。したがって、平均寿命や最大寿命の変化など、老化プロセスの定量的な変化を測定し、個人差から生じるバイアスを克服するには、大規模な集団が必要です。さらに、ハイスループットな寿命アッセイの能力は、生存曲線の形状と老化の動態のモデルの研究をサポートするために重要です9。
線虫 Caenorhabditis elegans は、その短い寿命、遺伝的トラクタビリティ、および迅速な生成時間により、老化研究にとって非常に貴重なモデルであり、ハイスループットな老化および寿命アッセイへの適合性を強調しています。伝統的に、 Cの寿命。 エレガンスは 、約50〜100匹の動物の同期した小さな個体群を固体媒体で経時的に追跡し、個々の死亡時刻を書き留めることによって測定されています。動物が年老いて運動能力を失うと、死亡時間を手作業で記録するには、動物を個別に突っ込み、頭や尾の小さな動きをチェックする必要があります。これは通常、退屈で面倒なプロセスですが、それを加速するための努力がなされています10、11、12。重要なことは、実験パイプラインの遅さは、老化の理解と検証された介入の有効性の進歩を妨げているということです。
定量的データに対する老朽化研究の要求を満たすために、マイクロ流体チャンバーからフラットベッドスキャナー13、14、15、16、17、18まで、データ収集を自動化するための多くの技術が開発されています。LSMは、ユーザーが自動分析を検証、修正、および改良できる広範なソフトウェアスイートと組み合わせた慎重な機器校正プロトコルの開発によって達成される、高精度で正確な寿命データを収集するための広範な最適化において他の方法とは異なります13。このソフトウェアは、原理的には多様なイメージングモダリティに適用できますが、実際には、ほとんどのユーザーは、環境の温度と湿度を微調整できるように改造されたフラットベッドスキャナーを使用しています - 寿命に大きな影響を与えるため、非常に重要な要素です19。LSMは、環境条件や遺伝子型に応じて、数日から数か月の間隔で20分ごとに線虫の画像を撮影します。生成されたデータは、手動アッセイのデータと比較してはるかに高い時間分解能であり、収集された画像は、生涯にわたって線虫の位置の永続的な視覚的記録を提供します。機械学習の手法を用いて、各個人に死の時間が自動的に割り当てられます。これらの結果は、「Worm Browser」と呼ばれるクライアントソフトウェアを使用して、手動で迅速に検証できます。そのハードウェアとソフトウェアの結果として、LSMは、経験豊富な研究者の手による手作業による死亡スコアリングと統計的に区別がつかない生存曲線を生成することができ、作業負荷の減少とスケーラビリティの向上という利点もあります13。
また、LSMの最新版では、線虫の生涯にわたる形態学的および行動学的データを収集し、各個体の寿命とともに報告することにより、行動老化の研究も可能になっています。特に、LSMは、個体の「健康寿命」を寿命とは異なるものとして定量化するためによく使用されるランドマークである、各動物の活発な運動停止(VMC)の時間を捉えます。LSMは、寿命と行動の老化データを同時に収集することにより、老化のさまざまな表現型の結果に異なる影響を与える可能性のある介入の研究をサポートします20。肉眼的に観察可能な様々な表現型を用いて、身体運動または咽頭ポンプ21、組織の完全性22、および運動速度または刺激誘発性回転17などの行動老化を研究することができる。異なる老化表現型間の比較は、老化プロセスの因果構造の分析をサポートすることができます。例えば、VMCと寿命の比較は、最近、線虫の2つの異なる老化プロセスを特徴づけるために用いられた23。
LSMは当初、線虫の寿命を測定するために開発されましたが、Cを含むさまざまな線虫種からの生存および行動データの収集をサポートしています。ブリッグサエ、C。トロピカリス、C。ジャポニカ、C。brenneri、および P.パシフィカス23。この技術は、寿命、ストレス耐性、および病原体抵抗性に対する生物学的および環境的介入の効果の研究を容易にし、RNA干渉の標的アッセイやオーキシン誘導性タンパク質分解システムなどの実験ツールと組み合わせることができます。今日まで、それは幅広い用途のために科学文献で使用されてきました6,24,25,26,27,28,29,30。
ここでは、寒天プレートを使用してLifespan Machine実験を行うためのプロトコルを、実験セットアップの初期段階から結果として得られる生存曲線の出力まで、段階的に概説します。LSMの特徴は、作業が高度に前倒しされていることであり、研究者の時間の大部分は実験のセットアップと、ごくわずかながら画像取得後の取得に費やされます。データ収集は実験の全期間にわたって完全に自動化されており、研究者は「ハンズフリー」で体験することができます。ここで説明するステップは、多くの異なるタイプの生存アッセイに共通しており、寿命、耐熱性、酸化ストレス、および病因アッセイに対して同じ実験セットアップが行われます。代表的な結果のセクションでは、最近発表された論文のデータのサブセットについて議論し、分析パイプラインの有効性を説明し、画像分析中の最も重要なステップを強調します23。
プロトコル
1. ソフトウェア要件とハードウェア要件
- フラットベッドスキャナー:原則として、LSMはさまざまなイメージングデバイスを使用して実装できます。スキャナーの変更とフォーカシングの詳細な手順は、他の場所で入手できます13.LSM ハードウェアを 補足図 1 に示します。
- データ解析ツール:LSMソフトウェアには、Linuxベースのスキャナ制御ソフトウェアパッケージ、Webブラウザベースのメタデータ管理パッケージ、WindowsおよびLinuxクライアント画像解析ソフトウェアパッケージの3つのコンポーネントがあります。GitHub リポジトリ (https://github.com/nstroustrup/lifespan) で公開されているソフトウェア ツールのインストール手順を参照してください。
- データ可視化・検証ソフトウェア:クライアントプログラムであるWorm Browserを使用して、実験のスケジュール設定、画像解析の検証、線虫の動きの手動アノテーション、生存データの出力を行います。Windows 7、Windows 8、およびWindows 10のワームブラウザにはバイナリ実行可能ファイルが提供され、ワームブラウザはLinuxまたはApple iOSのソースコードからコンパイルされます。インストールガイドは、上記のGitHubリポジトリで入手できます。
補足図1:寿命マシンのハードウェア。 ラバーマットにカットされた16個の開口部に下向きに配置された、ロードされたプレートを表示するための蓋が開いたフラットベッドスキャナーユニット1台。ゴムマットはガラススキャナーの表面に置かれます。画像解析時の問題を回避するために、プレートの側面に条件のラベルが書かれています。テープにデバイスの番号(「1」)や名前(「Jabba」)をマークすると、複数のスキャナーデバイスで作業するときに、後でサンプルの場所を簡単に確認できます。LSM ハードウェア コンポーネントの詳細については、別の場所 13 を参照してください。 このファイルをダウンロードするには、ここをクリックしてください。
2. 実験当日前の設営
- インキュベーターの温度校正:環境温度は Cの主要な決定要因です。 エレガンスの 寿命19.正確な結果を得るには、実験全体を通して一定に保たれた、慎重に校正された温度で画像取得を実行します。これを実現するには、実験開始の数日前に、動作中に各スキャナーの表面の温度を測定して校正します。他の場所31 に記載されているように、高精度の熱電対を使用してください( 材料表を参照)。
- スキャナープレートのレイアウト:実験を開始する前に、各スキャナーに最適な培養プレートのレイアウトを計画します。
メモ: この目的は、スキャナー間および各スキャナーの表面全体の温度変化に起因する交絡因子の導入を回避することです。スキャナは、その平均表面温度が微妙に異なり、さらに、その表面全体に微妙な温度バイアスを示す31。- スキャナープレートの位置:後続のデータ分析でこれらの熱効果を制御するには、スキャナーの位置に関する生物学的共変量をランダム化し、すべてのスキャナーでプレートの位置を標準化します。
- スキャナー全体のサンプル数:16枚のラバーマットレイアウト( 材料表を参照)を使用する場合は、各スキャナーに条件ごとに4枚のプレートを配置し、合計で少なくとも4台のスキャナーを配置します。これにより、各条件が複数のスキャナに分散され、データ分析中にスキャナ温度の交絡効果を特定して除去することができます13。この分析をより簡単にするために、各スキャナーに共通の参照条件(野生型サンプルなど)を含めます。
メモ: 一般に、スキャナーファンの配置により、ゴムマットの右上隅にあるプレートは乾燥しやすくなります。必要に応じて、この場所を空のままにします。
- プレートとサンプル調製
- 培養プレートの注入:スキャナー培養プレート( 材料表を参照)を最適に乾燥させるには、線虫をロードする4日前に寒天培地を注入します。スキャナーのフォーカスは、プレートに異なる量の寒天を添加できるように調整可能ですが、標準的なプレート容量は8 mLです。
注:特に大規模な実験では、蠕動ポンプでプレートを注ぐと役立つ場合があります。 - プレートの播種:実験開始の少なくとも2日前に、目的の細菌培養物をプレートに播種して、細菌芝生の適切な乾燥と成長を可能にします。典型的には、200μLの細菌培養物は、40匹の線虫を数週間養う芝生を形成するのに十分である。
注:イメージングに通常使用されるプレートは、標準的な培養ペトリ皿よりも密閉されています。したがって、実験室のフード内でプレートを播種して乾燥させることをお勧めします、通常約1時間、または適切に乾燥するまで。
- 培養プレートの注入:スキャナー培養プレート( 材料表を参照)を最適に乾燥させるには、線虫をロードする4日前に寒天培地を注入します。スキャナーのフォーカスは、プレートに異なる量の寒天を添加できるように調整可能ですが、標準的なプレート容量は8 mLです。
- 線虫の取り扱い
- 個体群サイズ:1枚のプレートで確実に画像化できる線虫の数は、遺伝子型、線虫がプレートされる年齢、および各プレートに添加される餌の量によって異なります。成人期初期に開始される寿命実験では、プレートごとに約40匹の動物をロードします。この数により、十分な食料が確保され、混雑が回避されます。
- 大規模な個体群の増加:個体群の拡大と同期に備えるには、選択した同期方法に適した線虫の個体群から始めます(以下を参照)。野生型N2動物の場合は成体32日目である最大産卵年齢で動物の同期を行うことを目指します。
注:成人期2日目の動物を用いて個体群を同期させるもう一つの理由は、個体群の不均一性に寄与する要因として母体の年齢を取り除くことである。母方の年齢は Cです。 エレガンスは 子孫の複数の適応度形質に影響を与えることが示されており、2日目の野生型動物は「最高品質の」子孫を産みます33。 - 年齢同期の実行: 正確な結果を得るには、動物の年齢をできるだけ正確に同期させます。このプロトコルでは、年齢同期は、修正された次亜塩素酸塩処理34を使用して行われる。他の方法には、卵のレイオフ、L1幼虫の停止、またはL4幼虫の手動摘み取りによる同期が含まれる場合があります。
注:次亜塩素酸塩処理による年齢同期の場合、各成体雌雄同体から3〜4個の卵を取得することを期待してください。 - 子孫のいない個体群の維持:L4後期の線虫を5-フルオロ-2'-デオキシウリジン(FUdR)に曝露することにより、子孫のない個体群を維持します35。
注:低用量では、FuDRは、生殖細胞系に肉眼的に目に見える変化を引き起こしたり、卵母細胞の産生速度を変えたりすることなく、胚の発生に致命的です。他の方法には、温度無菌変異体の使用、滅菌RNAiコンストラクトの使用、または単に生殖後の老化まで待って線虫をイメージング用のプレートに移す方法が含まれます。 - 個体群の移動:何千匹もの動物をプレート間で移動させる場合、白金/イリジウムワイヤーを含む標準的なプロトコルは面倒になります。線虫の液体懸濁液を含む方法は、移動を容易にし、それらをより効率的にする。M9 + Mg緩衝液(Na2HPO4 42.27 mM、KH2PO4 22.05 mM、NaCl 85.56 mM、MgSO4 1 mM)で線虫を回収し、線虫が重力によって沈降したら総体積を減らし、線虫をイメージングに使用したプレートにすばやく移します。
注:液体懸濁液で線虫を移すと、各プレートに移される動物の数にばらつきが生じる可能性があります。実験のばらつきを避けるために、各プレート上の線虫の数と一致するようにしてください。 - 介入の適用:実験中に画像取得を停止して再開すると、画像解析が複雑になります(ディスカッションを参照)。したがって、LSM実験は、必要な線虫処理がすべて完了してから開始してください。
- ゴムマットの滅菌:多数のマットをアルミホイルで個別に包むことにより、同時にオートクレーブします。
注意: ラバーマットは、真菌や細菌の汚染物質の蓄積を避けるために、使用の合間に滅菌する必要があります。通常使用されるほとんどの種類のゴムは、積極的なエタノール処理によって劣化します。
3. 実験当日の設営
- プレートサポートとスキャナーガラスの準備:プレートの取り扱いを簡素化するために、ペトリ皿をスキャナー表面に直接セットするのではなく、ガラス板で支えられたゴムマットを使用して所定の位置に保持します( 材料表を参照)。個体群はこのガラスを通して画像化されるため、ガラスを清潔に保ち、防曇、疎水性、滅菌コーティングで処理してください( 材料表を参照)。
- プレートをインキュベーターに入れる前に、防曇ガラスクリーナーでプレートサポートガラスの両側の表面を清掃してください。
- プレートを支持ガラスに装着する前に、保護用の疎水性ガラス処理( 材料表を参照)を施して、ラバーマットと接触するガラス側の曇りを最小限に抑えます。このトリートメントをよく広げ、次のステップに進む前に5〜10分間グラスの上に置いておきます。塗布後は激しく洗浄して残留物を取り除きます。
- 70%エタノールを塗布して、ゴムマットと接触するガラスの表面を消毒します。1分または2分放置してから、布またはペーパータオルで取り除きます。
- スキャナーへのプレートのセット
- まず、処理したプレートサポートガラスの上にオートクレーブ処理したゴムマットを置きます。
- 装填した線虫のイメージングに使用したプレートから蓋を外し、ガラス面に面したゴムマットの位置に置きます。たとえば、上に別のガラス板を追加して平らになるようにするか、各プレートの上部を軽くたたくなどして、すべてのプレートの周りにゴムマットがしっかりと密閉されていることを確認してください(緩んだプレートはわずかに動いてガラスにぶつかり、音がしますが、しっかりと固定されたプレートはタップしても動きません)。
注意: プレートサポートガラスの各シートに、プレートの内容物と目的のスキャナーに関する情報をマーキングテープで個別にラベル付けすると便利です。このデータは、実験後にプレートの位置に関する潜在的な曖昧さを解決するために使用できます。 - プレートをスキャナーにセットする前に、スキャナーファンのプラグを抜いて、プレートのロード中に実験者の指を保護します。
- プレートとプレートを支えているガラス板をスキャナーの表面にそっとスライドさせます。
注意: ラバーマットに直接圧力をかけると、マットがプレートサポートガラスを横切ってスライドするため、避けてください。マットがスライドすると、プレートがマットから外れることがよくあります。 - スキャナーファンを再度アクティブにし、前面ファンと側面ファンに電力が供給されていることを目視で確認します。スキャナーの電源がオフになっている場合は、この時点で電源を入れます。
4. プリイメージ取得
注:画像取得中のソフトウェアベースのすべての手順をまとめた包括的なフローチャートを 図1に示します。
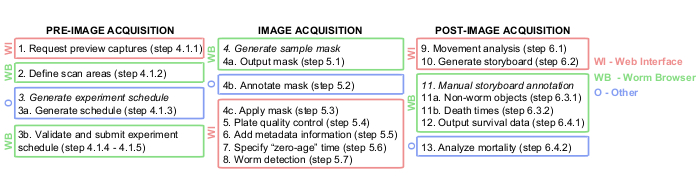
図1:Lifespan Machine画像解析パイプラインのグラフィカルな概要。 イメージ取得前、取得中、および取得後の手順は、主にWebインターフェイス(WI、赤)とワームブラウザ(WB、緑)で実行されます。手順3aのTXTドキュメント、手順4bのPhotoshopまたは同等のもの、手順13のJMPまたは同等のものなど、一部のステップは他のプラットフォーム(O、青色)で実行されます。 この図の拡大版をご覧になるには、ここをクリックしてください。
- 画像取得のセットアップ: 実験スケジュールと各スキャナーのプレートの位置を指定するファイルを生成して、画像取得を構成します。
注: このファイルには、実験のタイトル、画像キャプチャの頻度、実験の合計時間などの重要なメタデータが含まれています。通常、このファイルは生成されません de novo ただし、以前の実験の実験ファイルがテンプレートとして再利用されます。ユーザーの最初の実験では、テンプレート ファイル (補足ファイル1).- プレビューキャプチャをリクエストする:各スキャナーのすべてのプレートの位置を指定します。このプロセスを高速化するために、いくつかのツールが用意されています。まず、スキャナーを使用して、「プレビューキャプチャ」と呼ばれるスキャナー表面全体の画像を取得します。プレビューキャプチャ画像に、画像化する各プレートの表面全体がはっきりと写っており(図2A)、縞やプレートエッジのトリミングがないことを確認します。
- Webインターフェイスを使用して、メインページの[画像取得]セクションを見つけ、[キャプチャデバイスと画像サーバー]というリンクをたどります。そのページで、[Image Capture and Processing Servers]ボックスの[Search for New Devices (i.e., scanners)]ボタンをクリックします。サーバーの横にある [log] リンクをクリックして、スキャナーの検出の進行状況を監視します。
メモ: この手順を実行する前に、目的のスキャナーの電源が「オン」になっていて、サーバーに接続されていることを確認してください。 - Web インターフェイスの同じページで、サーバーに接続されている各スキャナが [Image Capture Devices ] ボックスに表示されていることを確認します。デバイスが正常に検出された場合、「現在のステータス」の下の「欠落」ラベルは表示されなくなります。新しくセットされたプレートを含む各スキャナーに対応するチェックボックスを選択します。
- [Image Capture Devices] セクションの下部にある [Request Preview Capture] ボタンをクリックします。1分または2分以内に、スキャナーが点灯し、スキャンが開始されます。
注意: ガラスサポートシートの初期位置は、多くの場合、すべてのプレートを表示可能な範囲に収めるために調整する必要があります。位置は、プレビューキャプチャ画像を検査し、プレート位置を調整し、新しいプレビューキャプチャ画像を再撮影することで修正できます。スキャンの進行が極端に遅い場合(1回のキャプチャスキャンで数分)、またはプレビューキャプチャ画像に長い白い縞が含まれている場合は、プレート、ゴムマット、またはその他の物体がスキャナーのキャリブレーション領域内で光を遮っていることを示しています(スキャナー表面の白い矢印で示されます)。すべてのオブジェクトは、ガラス支持ガラスのみがこの領域を占めるように再配置する必要があります。
- Webインターフェイスを使用して、メインページの[画像取得]セクションを見つけ、[キャプチャデバイスと画像サーバー]というリンクをたどります。そのページで、[Image Capture and Processing Servers]ボックスの[Search for New Devices (i.e., scanners)]ボタンをクリックします。サーバーの横にある [log] リンクをクリックして、スキャナーの検出の進行状況を監視します。
- スキャン領域の定義:Worm Browserの次の手順に従って、プレビューキャプチャ画像を解析し、それらを1つの合成画像に組み立てて、データ解析用の各プレートの位置を指定します。結果の画像が 図2Bのようになっていることを確認します。
- まず、Worm ブラウザを使用して、メニュー オプションの [ファイル] > [画像を開く] を選択して各プレビュー キャプチャ 画像を開き、目的の画像を選択します。
- 各イメージで、プレートのある領域の列をクリックして選択します (ラバー マットに 16 個のプレート位置がある場合は、4 列を選択します)。
注:スキャン領域は、広い領域ではスキャナーのキャプチャが遅くなり、線虫の動きによって画像がぼやけるため、背の高い列(幅の広い行ではない)として指定する必要があります。 - すべての画像を定義したら、メニュー項目の [画像取得]>[スキャン領域の定義]>[選択したスキャン領域をディスクに保存 ]を選択し、目的の場所を選択して、領域仕様をディスクにエクスポートします。
- 実験スケジュールを生成します。
- 補足ファイル 1 の形式に従って、実験名、スキャナーの各列の物理的な位置 (手順 4.1.2.3 で生成されたスキャン領域ファイルからコピー)、実験の合計時間、および画像キャプチャ頻度を含むファイルをアセンブルし、これを TXT ファイルと XML ファイルの両方として保存します。
- 次に、Worm Browser で [ Image Acquisition] > [Submit Experiment Schedule] をクリックし、生成された XML ファイルを選択します。ワームブラウザは、スケジュールの要約を出力するか、実験を実行するかを尋ねます。[ Generate a Summary File] をクリックします。
- 概要ファイルを検証する: 実験スケジュールを送信すると、Worm Browser はスケジュールの概要を出力します。この概要は画面に表示され、ディスクに書き込まれます。それを読んで、スケジュールされたキャプチャの日付、およびスキャナーの場所、名前、数を確認します。
- 実験スケジュールの送信: 概要ファイルに満足したら、メニュー オプションの [画像取得] > [実験スケジュールの送信] を選択して、実験スケジュールの XML ファイルを Worm ブラウザに再度ロードします。ワームブラウザは、スケジュールの概要を出力するか、実験を実行するかを2回目に尋ねます。今回は [実行]をクリックします。
注: 実験を送信してから数分後に、Web インターフェイスを使用して、実験が正常に送信され、すべてのスキャナーでスキャンが収集されていることを確認するのが賢明です。特に大規模な実験では、最初の数回のスキャンが見落とされるのが普通です。 - Web インターフェイスで実験を整理する: ビジー状態のスキャナー クラスターでは、さまざまなユーザーによって収集された何百もの実験データセットが生成される場合があります。このリストを整理するには、実験を個別のグループ(たとえば、実験を担当するユーザーの名前に対応する)に割り当てます。
- 新しいグループを作成するか、既存のグループを変更する: ウェブ管理画面で [画像取得] ボックスの下にある [テスト グループの管理] をクリックして、新しいグループを作成します。表示される新しいページで、[新しいグループの作成]に目的の名前を追加し、[作成]をクリックします。既存のグループの名前を変更するには、同じボックスの [既存のグループの変更] の横にある目的のグループを選択し、[変更] を選択します。
- テストをグループに割り当てる: 新しいテストを特定のグループに割り当てるには、ウェブ インターフェイスに移動し、目的のテストを見つけます(デフォルトでは、テスト リストの下部にある [グループなし ] グループに割り当てられます)。実験セクションの右側にある [編集] と表示されているリンクをクリックし、プルダウン リストを使用して使用するグループの名前を選択します。次に、 [ 保存] を選択します。
- テストをキャンセルするには:
注:LSMは、実験スケジュールで最終スキャンが指定されるまで、自律的に動作し続けます。実験スケジュールが完了すると、LSM はデフォルトでスキャンの収集を続行しますが、 自動スキャンと呼ばれるプロセスで画像データをすぐに破棄します。これらの自動スキャンは、スキャナーの電源が切れて冷却されるのを防ぎ、同じスペースで実行されている他の実験(ただし別の実験から)が他のスキャナーのシャットダウンの影響を受けないように、標準温度プロファイルを維持するために実行されます。- 自動スキャンの停止: Web インターフェイスで進行中の実験から自動スキャンを停止するには、目的の実験の横にある [編集 ] をクリックし、[ 保留中のスキャンのキャンセル] をクリックして、[ スケジュールされたキャプチャのキャンセル] を選択します。
- プレビューキャプチャをリクエストする:各スキャナーのすべてのプレートの位置を指定します。このプロセスを高速化するために、いくつかのツールが用意されています。まず、スキャナーを使用して、「プレビューキャプチャ」と呼ばれるスキャナー表面全体の画像を取得します。プレビューキャプチャ画像に、画像化する各プレートの表面全体がはっきりと写っており(図2A)、縞やプレートエッジのトリミングがないことを確認します。
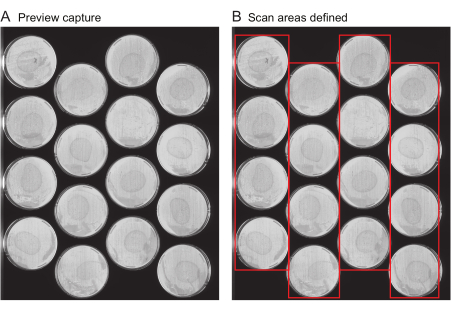
図2:キャプチャ画像のプレビューとスキャン領域の選択。 (A)実験のスキャナーごとに、プレビューキャプチャ画像が生成されます。 (B)一度に1列のプレート(赤いボックス)を選択することで、スキャン速度が向上し、領域が広すぎるスキャンの結果としてワームのモーションブラーが防止されます。 この図の拡大版をご覧になるには、ここをクリックしてください。
5. 画像取得
注: 次の手順は、実験の実行中または終了後に実行できます。
- 実験のマスクファイルを出力する:スキャナーからの生の画像データには、処理する必要のない領域(プレートの外側の領域)が多数含まれています。各プレートに個別に分析の焦点を絞るために、各スキャナーで各プレートが占める領域を指定する「マスク」が作成されます。スキャナーによって収集された画像に各プレートの位置をオーバーレイとして描画することで、このマスクを生成します。
- ワーム ブラウザを使用して、[ ファイル] > [現在の実験の選択] を選択して目的の実験を選択し、実験名をクリックします。
- 再度 Worm ブラウザーで [ Image Acquisition] > [Define Sample Masks] > [Generate Experiment Mask Composite] を選択し、マスクを目的の場所に保存します。結果のファイルが 図 3A のようになっていることを確認します。

図3:サンプルマスクを使用した各スキャナーのプレート位置の指定。図 1 に示すカラム選択内のプレートを独立して分析するには、画像マスク合成を生成して個々のプレートを識別する必要があります。(A)スキャナーのスキャンのキャプチャを画像操作ソフトウェアで開きます(スキャンされた選択範囲の上にあるスキャナーの名前「han」と、各列を指す「a-d」に注意してください)。(B)マスク合成の各プレートの位置をマークするマスク生成の個々のステップでは、背景を黒に設定し、(C)背景の拡大と縮小によってギザギザのエッジと選択されていないプレートのエッジを除去し、(D)前景プレートを選択し、領域全体を白いピクセルで塗りつぶす必要があります。 (E)LSMがスキャンされた行の個々のプレートを認識するために、行の各白い領域は、通常は明るさを増しながら、異なる階調のグレーで塗りつぶされます。(F)この段階で、マスクが保存されます(Photoshopで生成された場合は、レイヤーを指定しないLZW圧縮)。その後、マスクはワームブラウザによってスキャンされ、ソフトウェアによるマスクの視覚化が生成されます。正しいマスクの視覚化では、プレートごとに 1 つの定義された正方形が表示され、中央に小さな十字が表示され、行ごとに異なる色が表示されます。この図の拡大版をご覧になるには、ここをクリックしてください。
- 実験のマスクに注釈を付ける: 前の手順で生成したファイルを画像操作プログラム(PhotoshopやGIMPなど)で開き、画像内の各プレートの位置をマークします。Photoshop を使用したすべてのマスク編集手順の概要を以下に説明します。
- Photoshop で、許容値を 0 に設定し、連続オプションを選択し、アンチエイリアスを選択解除した状態で 塗りつぶし ツールを選択します。灰色の背景をクリックして、完全に黒に設定します。結果の画像が 図3Bのようになることを確認します。
- 魔法の杖ツールを使用して黒い背景を選択し、エイリアシングをオフに設定し、許容値をゼロに設定し、連続オプションを選択します。エッジを滑らかにするには、[選択] をクリックして選択した背景を 30 ピクセル拡大し >変更>展開します。次に、[ Select > Modify > Contract] で選択範囲を 20 ピクセル縮小します。結果の画像が 図3Cのようになることを確認します。
- スムージングされた背景全体を黒いピクセルで塗りつぶすには、たとえば [塗りつぶし ] ツールの許容値を 255 に設定し、選択した領域を塗りつぶします。次に、[ 反転>選択 ]をクリックして選択を反転し、前景を選択します。たとえば、塗りつぶしツールの許容値を 255 に設定し、その領域を白で塗りつぶすことで、新しい領域全体を白いピクセルで塗りつぶします。結果の画像が 図3Dのようになっていることを確認します。
- 各プレートを 1 つの領域内に分けるには、各行を異なるグレーのグラデーションで塗りつぶし、明るさを上げます。これは、塗りつぶしツールを使用してから、許容値を 0 に設定して目的の色を選択することで実行できます。結果の画像が 図3Eのようになっていることを確認します。「レイヤー」を指定せずにLZW圧縮で保存します。
注: プレートの順序は、指定した領域の色によって設定されます。プレート 1 から 4 に上から下の順に名前を付けるには、各行の明るさを増すように色を指定します。 - Wormブラウザで、 Image Acquisition>Define Sample Masks > Analyze Plate Locations Drawn on Experiment Mask Compositeを選択し、前の手順で生成したファイルを選択します。ソフトウェアは、送信されたマスクを分析するのに少し時間がかかります。
- ワームブラウザにマスクの視覚化が表示されます。マスクにファイル内の潜在的なエラーがないか調べます。プレートの各行が異なる色で塗りつぶされ、色付きの長方形で輪郭が描かれていることを確認します。結果の画像が 図3Fのようになっていることを確認します。
注:1つのプレートに2つ以上の円が表示されている場合、または2つの円が同じ色を共有している場合は、手順5.2に戻ってマスクファイルを修正し、ワームブラウザに再度ロードします。 - マスクの可視化が正しいことを確認したら、Worm ブラウザーで [Image Acquisition] > [Define Sample Masks] > [Submit Analyzed Experiment Mask Composite to Cluster] を選択します。画像解析サーバは、実験中のすべてのプレートの位置を解析します。
- マスクの適用:LSMはマスクを使用して、生の画像データを個々のプレートに分割します。このプロセスを開始するには、Web インターフェイスを使用してマスク適用ジョブをスケジュールします。
- ジョブを投入する前に、すべてのスキャナーがマスク内の領域を識別していることを確認してください。Web インターフェイスのメイン ページに移動し、実験の名前と [画像分析] という名前の列を見つけて、[ 分析の実行] をクリックします。 [Experiment Samples ] のすべてのデバイスに領域が識別されていることを確認します。
- マスクを適用するには、同じインターフェイスで [ New Job for All Samples] をクリックします。 [Schedule a Job for Individual Images] (個々の画像のジョブのスケジュール) というボックスで、[ Apply Mask] (マスクの適用) ボックスを選択し、[ Save Job] (ジョブの保存) を選択します。
注:マスクは、すでにキャプチャされたすべての画像と、今後キャプチャされるすべての画像に適用されます。
- プレートの品質管理を実行します。
注:コンタミネーション、乾燥、または曇りに悩まされているプレートは、画像分析のこの段階で打ち切られます。汚染されたプレート、乾燥したプレート、曇ったプレート、最適なプレートの例を 図4に示します。打ち切りの他の理由には、飢餓、空のプレート、または不妊集団の幼虫が含まれます。無効なプレートには、機械が線虫として解釈することを目的とした複雑な形状が含まれていることがよくあります。このステップでは、画像解析の後のステップで処理時間が長くなるのを防ぐために、無効なプレートを除外することが重要です。- ウェブインターフェースを使用して、目的の実験と Annotate Plate Information という名前の列を見つけて、 画像別を選択することで、打ち切りするプレートを除外します。
- プレートを検査するには、[ 画像の表示]をクリックします。
注: 画像を表示する手順が正しく機能しない場合は、Linux サーバーが正しく設定されていない可能性があります。これを行う方法については、前述のGitHubリポジトリのソフトウェアインストールガイドを参照してください。 - プレートを除外するには、[ 検閲]チェックボックスをオンにし、[ 理由の打ち切り]という名前のドロップダウンボックスでオプションを選択して、各ページの [保存]をクリックします。
- メタデータ情報の追加: メタデータは、実験における各プレートの内容を記述します。この情報は、その後に生成されるすべての統計データファイルに含まれます。
- 菌株、遺伝子型、温度、食品などに関連するメタデータ情報を追加するには、ウェブインターフェースのメインページに移動し、目的の実験とプレート 情報に注釈を付ける列を見つけて、 位置別を選択します。
- ラベルを入力し、左下隅にある [ デバイスに保存 ] をクリックして、メタデータを保存するスキャナーを選択します。
- すべてのラベルを再入力せずに、異なるスキャナー間でメタデータを再利用するには、右上隅の [ デバイスから読み込む ] に移動し、メタデータを再利用するスキャナーを選択して、[ デバイスから読み込む] をクリックします。
- 「ゼロエイジ」時間を指定する: デフォルトでは、LSM は UNIX エポックの開始を基準にして時間を測定します。これはめったに都合が悪いので、基準時間の指定が必要です(たとえば、すべての個体が孵化または成体に達した日付)。
- ゼロ経過時間情報を指定するには、Web インターフェイスのメイン ページに移動し、目的の実験と [画像分析] という名前の列を見つけて、 [ 分析の実行] を選択します。
- 表示される新しいページで、 [ すべてのリージョンの新しいジョブ] を選択します。[ Update Region Information] ボックスで [ Time at at which Animals had 0 Age] を選択し、情報を追加して [ Update Selected Fields] をクリックします。
注:すべての動物が同じゼロ年齢時間を共有していない場合は、代わりに [特定の動物タイプの新しいジョブ]を選択し、グループごとに上記の手順を繰り返します。
- 線虫検出のスケジュール設定:LSMは、プレート上の線虫の位置に基づいて、各線虫の検出を自動化できます。
- 各画像の線虫の自動検出を開始するには、Web インターフェイスのメイン ページに移動し、目的の実験と [画像分析] という名前の列を見つけて、[ 分析の実行] を選択します。
- 表示される新しいページで、[ New Job for All Regions] をクリックし、[ Schedule a Job for Individual Images] ボックスをクリックして、[ Median Filter > Threshold] > [Worm Detection] > [Save Job] ボックスを選択します。これらのジョブは、キャプチャされた過去および将来のすべての画像に適用されます。
注:特定の系統または状態のみのジョブをスケジュールするには、代わりに 特定の動物タイプのジョブをクリックします。オブジェクトの分類は、LSM の長期ストレージディレクトリの Models サブフォルダに保存されているファイルとして指定された SVM モデルを使用して実行されます。LSM の V2.0 線虫検出パラメーター セットは、GitHub リポジトリからダウンロードできます。
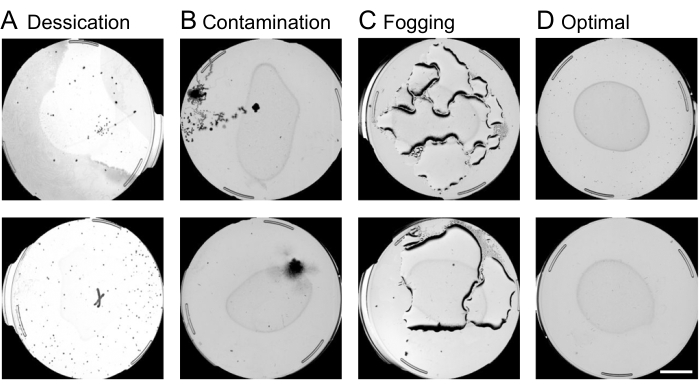
図4:ウェブインターフェースを使用したプレートの品質管理。 ワームの動きを分析する前に、ウェブインターフェース上で最適でないプレートを打ち切ることは、画像処理パイプラインを高速化するために重要です。除去の対象となるプレートの例としては、(A)乾燥、(B)汚染、(C)曇りなどの条件が挙げられます。(D)さらなる分析に含める最適なプレート。10 mm の縮尺記号がプレビュー キャプチャ イメージに重ね合わされます。 この図の拡大版をご覧になるには、ここをクリックしてください。
6. 画像取得後
注:ワームの検出が完了したら、実験から収集されたすべてのデータを経時的に集計して、各個体を生涯にわたって追跡し、すべての個体の死亡時間を特定する必要があります。実験に参加したすべての動物が死亡し、すべての寄生虫検出ジョブが完了するまで待ってから、次の手順を実行します。
- 移動分析をスケジュールします。
- 運動分析では、経時的なすべての実験データを統合して、死亡時間を推定します。このジョブを開始するには、Web インターフェイスのメイン ページに移動し、[ 画像解析] という名前の列で目的の実験を見つけます。[ 分析の実行] リンクを選択します。
- 表示される新しいページで、[ New Job for All Regions] リンクをクリックし、プルダウン メニューから [ Schedule a Job for an Entire Region] から [ Analyze Worm Movement] を選択し、[ Save Job] ボタンをクリックします。
- LSM画像取得サーバーは、すべてのプレートの動きの分析を自動的に開始します。
注: 移動分析は、単一の最大のジョブです。最新のマルチコアプロセッサでは、1か月の寿命実験から得られた各プレートの分析に20分以上かかることがあります。
- ストーリーボードの生成:移動分析が完了した後、LSMストーリーボードを使用すると、ユーザーは自動結果を手動で検証し、ソフトウェアが線虫の形態と行動について正しい仮定を行っていることを確認できます。
- Web インターフェイスのメイン ページで、目的の実験と [画像分析] という名前の列を見つけて、 [ 分析の実行] を選択します。表示される新しいページで、[ 新しい実験ジョブ] をクリックします。次に、[ Schedule a Job for an Entire Region (リージョン全体のジョブのスケジュール)] セクションで [ Generate Animal Storyboard (動物の絵コンテを生成)] を選択し、[ Save Job (ジョブの保存)] をクリックします。
- LSM がストーリーボードの生成を完了したら、ワーム ブラウザーに移動し、目的の実験を選択します。メイン メニューに戻り、[ Validation] > [Browse Entire Experiment > Immediately after Each Worm's Death] を選択します。
- ワーム ブラウザーのストーリーボードに注釈を付ける: ワーム ブラウザーの一般的なストーリーボードは次のようになります。 図5.
- 「ワーム以外の」オブジェクトに注釈を付ける
注: 移動中に検出された各オブジェクトは、推定死亡時間の順にストーリーボードに表示されます。ユーザーが特に指定しない限り、各オブジェクトは結果の生存曲線に含まれます。LSM オブジェクトの分類は、検出されない線虫の数を最小限に抑えるためのトレードオフとして、偽陽性率が高くなるように意図的に調整されています。線虫以外の物体の除外は、高品質の生存曲線を得るために重要です(代表的な結果を参照)。通常、ストーリーボードの最初と最後のページにはワーム以外のオブジェクトが多数あり、手動ですばやく一括で除外できます。- ストーリーボード上のワーム以外のオブジェクトを除外するには、オブジェクトのイメージを一度右クリックします。除外されたオブジェクトは、白いボックスで囲まれます。多数のオブジェクトを同時に除外するには、Ctrl キーを押しながら任意のオブジェクトを 1 回右クリックすると、ストーリーボード ページ上のすべてのオブジェクトが除外されます。
- 分析からオブジェクトを除外した後、もう一度 2 回右クリックすると、そのオブジェクトを再度含めることができます。
- 絵コンテの注釈中に作成した注釈を保存するには、[ 保存 ]ボタンをクリックします。注釈を付けている間は、進行状況を頻繁に保存することをお勧めします。
- 死亡時刻に注釈を付ける:
注:ユーザーの介入なしに、LSMはほとんどの集団の死亡時間を正確に推定します。ただし、各実験からランダムに抽出した個人のサブセットを手動で検証することにより、自動結果の精度を定期的に確認することをお勧めします。自動分析のエラーはこれらのグループに集まる傾向があるため、最も短命な個人と最も長い個人には特に注意を払う必要があります。- ストーリーボードの任意のオブジェクトを 1 回左クリックすると、新しいウィンドウが開き、そのオブジェクトに関する詳細な時系列情報が表示されます。このウィンドウでは、実験中に収集された対象物のすべての画像を検査し、対象物の動きのダイナミクスと形態を定量化することができます。同じインターフェイスを使用して、デスタイムに手動で注釈を付けます。デスタイムアノテーションのインターフェースを 図6に示します。
- 死亡時刻に手動で注釈を付けるには、死亡時刻に対応するポイントで下部のバーを左クリックします。スペースバーとキーボードの右矢印または左矢印を使用して時間枠を移動するか、目的の時間枠でバーを直接クリックします。
注: 視覚化は、時間の経過に伴う動物の移動状態を、X 軸が時間を示す水平棒グラフのように表します。ピンク/パープルのセクションは、オブジェクトが活発に動いている期間を示し、黄色は弱い動きを示し、赤は動かない動物を示し、緑は死に伴う膨張の期間を示します。線虫は、死に関連する特徴的な形態学的事象を示します:通常は死の前に起こり、その後、死亡中または直後に急速な体の拡張が続きます(図6B)。埃や影などのワーム以外の物体は、体の大きさにこれらのダイナミクスを示さず、代わりに、通常、時間の経過とともにサイズと強度が直線的に徐々に変化します。これらの異なる体サイズのダイナミクスは、迅速な分類と手動除外のための迅速で簡単な方法を提供します。 - 分析アプローチに応じて、死亡時間は、動作停止の時刻 (動作の視覚化の赤いバーの開始) または死亡に関連する拡大の時刻 (動作の視覚化の緑色のバー) と見なすことができます。収縮イベントと拡張イベントに手動で注釈を付けるには、目的の時間枠で下部のバーを右クリックします。
- 一部の初歩的な死亡率データの視覚化は、Worm Browser クライアント内でネイティブに提供されます。生存曲線と死亡時間診断は、ストーリーボードに存在する各集団について示されています(図7)。左側の生存曲線と、絵コンテの右側にある各個体の活発な運動停止(VMC)の時間と死亡の時間を比較した散布図を参照してください。
注:これらの結果は、特定の菌株、スキャナー、プレートなどの条件の下部バーをサブセットすることにより、さまざまな実験パラメータに従ってグループ化できます。個々のワームは、VMC対死亡時間プロットの個々のポイントを左クリックすることで、死亡時間に基づいて選択できます。
- 「ワーム以外の」オブジェクトに注釈を付ける
- 死亡率データのディスクへの書き込み: LSM は、CSV ファイルの形式で死亡率データを生成します。出力された生存曲線をプロットし、R、SAS、STATA、JMPなどの統計ソフトウェアで解析します。
- これらのファイルを生成するには、ワーム ブラウザで実験を選択し、メニューで [データ ファイル]、[Death Times] の順に選択し、[ Generate Death Times for Current Experiment] をクリックします。LSMは、実験の生存データを含む出力ファイルを生成し、結果ディレクトリに保存されます。
- ストーリーボードで手作業による注釈が実行されている場合は、手作業による注釈の処理方法を尋ねるプロンプトがワームブラウザに表示されます。「Immediately」をクリックすると、出力された死亡時刻ファイルに手書きの注釈が付けられます。
注: 死亡率データファイルは、imageserver.ini ファイルで指定された結果ディレクトリに書き込まれます。さまざまなファイルが書き込まれますが、最も一般的に使用されるバージョンは「survival_simple/survival=machine_hand」で、ストーリーボードを使用して作成されたすべての手動注釈が含まれています。 - 選択した統計ソフトウェアで死亡率データを分析します。
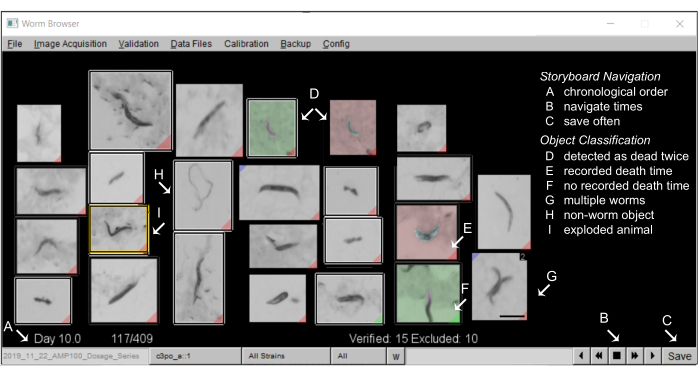
図5:Worm Browserの動物の絵コンテ (A)すべての静止したワームは、機械が注釈を付けた死亡時間の時系列で示されています。ストーリーボード内を移動するには、(B)右下隅のボタンを押し、(C)注釈を頻繁に保存します。(D)灰色以外の背景の画像は、2つの線虫の死(早期の死は緑、後の死は赤)を表しており、2匹の線虫が互いに近くで死んだ場合、または死んだ線虫が通りすがりの線虫によって移動され、2回死んだと検出された場合に発生します。 (E)画像の下隅にある赤いタグは、検出された死亡時間を持つワームを識別します。(F)緑色のタグは、オブジェクトが死亡時間を記録するのに十分な時間静止していなかった場所を示します。 (G)同じフレーム内の複数のワームは、Shiftキーを押しながら左クリックすることでフラグを立てることができます。(H) ワーム以外のオブジェクトは、右クリックで解析から除外されます。(I)爆発したワームは、対応する画像をクリックし(手作業による注釈ウィンドウが開きます)、Shiftキーを押しながら右クリックして「動物が爆発した」というメッセージが表示されるまで、分析から打ち切られます。 0.5 mm のスケール バーとラベルが Worm Browser ウィンドウのスクリーンショットに重ね合わされています。 この図の拡大版をご覧になるには、ここをクリックしてください。
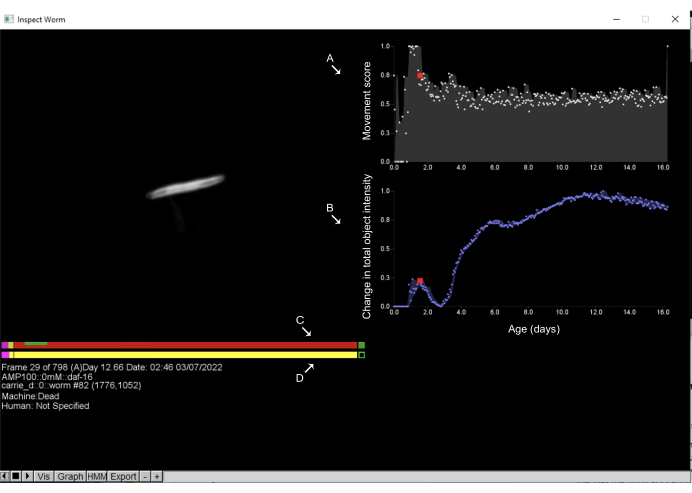
図6:Worm Browserでのオブジェクトの検査とデスタイムの注釈 Worm Browserストーリーボード上の任意のオブジェクトを左クリックすると、新しいインターフェースが開き、ユーザーはオブジェクトの動きのダイナミクスを調べることができます。右側には、物体の動きを定量化する(A)移動スコアが表示されます。これは、連続する観測間のピクセル強度の変化によって推定されます。また、右側には(B)物体の総強度の変化が表示され、物体の大きさの変化を数値化しています。左側のバーは、(C)機械による死亡時間の推定値を示し、下のバーは(D)人間の手作業による注釈です。バーの任意のポイントをクリックしてスペースキーを押すと、ユーザーはワームが画像化された時間枠を移動できます。これらのバーでは、ピンクは活発な動きに費やされた時間を表し、赤は死に費やされた時間を表し、黄色はその間のすべてを表しています。死亡時間後の膨張と収縮に費やされた時間は緑色で示されています。ラベルは、Worm Browserウィンドウのスクリーンショットに重ねて表示されます。 この図の拡大版をご覧になるには、ここをクリックしてください。
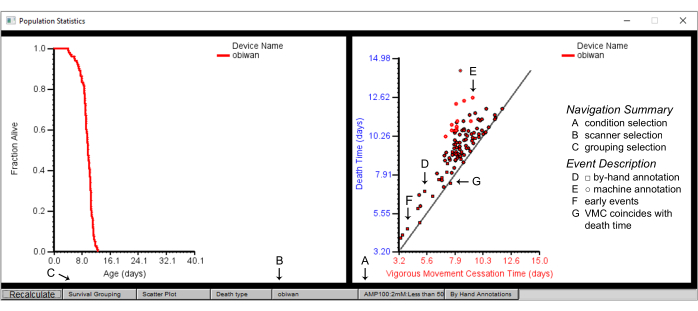
図7:Worm Browserの母集団要約統計量 スキャナーデバイス「obiwan」の個体群統計と、生存率のプロット(左パネル)と、活発な運動停止(VMC)時間対死亡時間の散布図(右パネル)。プロットされたのは、(A)1つの条件の詳細であり、(B)最初に(C)株による生存グループを選択することによって達成された1つのスキャナーから得られます。(D)散布図の正方形の図形は手作業で注釈が付けられたイベントを表し、(E)円形の図形は機械で注釈が付けられたイベントを示しています。(F)早期に発生する死亡イベント、または(G)活発な運動停止時間が死亡時間と一致するイベントでは、手作業による注釈が必要になることがよくあります。ラベルは、Worm Browserウィンドウのスクリーンショットに重ねて表示されます。 この図の拡大版をご覧になるには、ここをクリックしてください。
結果
ライフスパンアッセイにおける実験の再現性は困難であり、十分な統計的分解能を達成するには、厳密に制御された実験条件と大規模な集団の両方が必要です4,36。LSMは、高い時間分解能で一定の環境で動物の大規模な集団を調査するのに特に適しています。LSMの能力を実証し、分析の重要なステップを強調し、研究者が労働努力に優先順位を付け?...
ディスカッション
ここでは、最新バージョンのLifespan Machineを使用して実験を実行するための詳細でアクセス可能なプロトコルを提供します。私たちは、十分に分解された生存曲線を達成するための重要なステップは、画像取得後の非線虫オブジェクトを手動で除外することであることを示しました。手作業による死亡時間アノテーションは、生存曲線の全体的な形状にほとんど影響を与えず、手動アノテーシ...
開示事項
著者らは、競合する利害関係はないと宣言しています。
謝辞
rpb-2(cer135)対立遺伝子を作製してくれたJulian Cron氏とJeremy Vicencio氏(IDIBELL Barcelona)に感謝します。このプロジェクトは、欧州連合(EU)のHorizon 2020研究・イノベーションプログラム(助成金契約第852201号)に基づく欧州研究会議(ERC)、スペイン経済産業競争力省(MEIC)からEMBLパートナーシップ、Centro de Excelencia Severo Ochoa(CEX2020-001049-S、MCIN/AEI /10.13039/501100011033)、CERCAプログラム/カタルーニャ州政府、MEICエクセレンシア賞BFU2017-88615-P、 グレン医学研究財団からの賞。
資料
| Name | Company | Catalog Number | Comments |
| 1-Naphtaleneacetic acid (Auxin) | Sigma | N0640 | Solubilize Auxin in 1M potassium hydroxide and add into molten agar |
| 5-fluoro-2-deoxyuridine (FUDR) | Sigma | F0503 | 27.5 μg/mL of FUDR was used to eliminate progeny from populations on UV-inactivated bacteria |
| Glass cleaner | Kristal-M | QB-KRISTAL-M125ml | |
| Hydrophobic anti-fog glass treatment | Rain-X Scheibenreiniger | C. 059140 | |
| Rubber matt | Local crafstman | Cut on a high-strength EPDM rubber sheet stock | |
| Scanner glass | Local hardware supplier | 9" x 11.5" inch glass sheet | |
| Scanner plates | Life Sciences | 351006 | 50 mm x 9 mm, polystyrene petri dish |
| USB Reference Thermometer | USB Brando | ULIFE055500 | For calibrating temperature of scanners |
参考文献
- Harman, D. The aging process: Major risk factor for disease and death. Proceedings of the National Academy of Sciences of the United States of America. 88 (12), 5360-5363 (1991).
- Vaupel, J. W. Biodemography of human ageing. Nature. 464 (7288), 536-542 (2010).
- Mair, W., Goymer, P., Pletcher, S. D., Partridge, L. Demography of dietary restriction and death in Drosophila. Science. 301 (5640), 1731-1733 (2003).
- Petrascheck, M., Miller, D. L. Computational analysis of lifespan experiment reproducibility. Frontiers in Genetics. 8, 92 (2017).
- Lucanic, M., et al. Impact of genetic background and experimental reproducibility on identifying chemical compounds with robust longevity effects. Nature Communications. 8 (1), 14256 (2017).
- Banse, S. A., et al. Automated lifespan determination across Caenorhabditis strains and species reveals assay-specific effects of chemical interventions. Geroscience. 41 (6), 945-960 (2019).
- Herndon, L. A., et al. Stochastic and genetic factors influence tissue-specific decline in ageing C. elegans. Nature. 419 (6909), 808-814 (2002).
- Kirkwood, T. B., et al. What accounts for the wide variation in life span of genetically identical organisms reared in a constant environment. Mechanisms of Ageing and Development. 126 (3), 439-443 (2005).
- Stroustrup, N., et al. The temporal scaling of Caenorhabditis elegans ageing. Nature. 530 (7588), 103-107 (2016).
- Hamilton, B., et al. A systematic RNAi screen for longevity genes in C. elegans. Genes & Development. 19 (13), 1544-1555 (2005).
- Lee, S. S., et al. A systematic RNAi screen identifies a critical role for mitochondria in C. elegans longevity. Nature Genetics. 33 (1), 40-48 (2003).
- Cornwell, A. B., Llop, J. R., Salzman, P., Thakar, J., Samuelson, A. V. The replica set method: A high-throughput approach to quantitatively measure Caenorhabditis elegans lifespan. Journal of Visualized Experiments. (136), e57819 (2018).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: A quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Hulme, S. E., et al. Lifespan-on-a-chip: Microfluidic chambers for performing lifelong observation of C. elegans. Lab on a Chip. 10 (5), 589-597 (2010).
- Kerr, R. A., Roux, A. E., Goudeau, J. F., Kenyon, C. The C. elegans observatory: High-throughput exploration of behavioral aging. Frontiers in Aging. 3, 932696 (2022).
- Javer, A., Ripoll-Sánchez, L., Brown, A. E. Powerful and interpretable behavioural features for quantitative phenotyping of Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 373 (1758), 20170375 (2018).
- Miller, H., et al. Genetic interaction with temperature is an important determinant of nematode longevity. Aging Cell. 16 (6), 1425-1429 (2017).
- Bansal, A., Zhu, L. J., Yen, K., Tissenbaum, H. A. Uncoupling lifespan and healthspan in Caenorhabditis elegans longevity mutants. Proceedings of the National Academy of Sciences of the United States of America. 112 (3), E277-E286 (2015).
- Huang, C., Xiong, C., Kornfeld, K. Measurements of age-related changes of physiological processes that predict lifespan of Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 101 (21), 8084-8089 (2004).
- Garigan, D., et al. Genetic analysis of tissue aging in Caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Oswal, N., Martin, O. M., Stroustrup, S., Bruckner, M. A. M., Stroustrup, N. A hierarchical process model links behavioral aging and lifespan in C. elegans. PLoS Computational Biology. 18 (9), 1010415 (2022).
- Sen, I., et al. DAF-16/FOXO requires protein phosphatase 4 to initiate transcription of stress resistance and longevity promoting genes. Nature Communications. 11 (1), 138 (2020).
- Schiffer, J. A., et al. et al.Caenorhabditis elegans processes sensory information to choose between freeloading and self-defense strategies. Elife. 9, 56186 (2020).
- Bazopoulou, D., et al. Developmental ROS individualizes organismal stress resistance and lifespan. Nature. 576 (7786), 301-305 (2019).
- Guerrero-Rubio, M. A., Hernández-García, S., García-Carmona, F., Gandía-Herrero, F. Extension of life-span using a RNAi model and in vivo antioxidant effect of Opuntia fruit extracts and pure betalains in Caenorhabditis elegans. Food Chemistry. 274, 840-847 (2019).
- Janssens, G. E., et al. Transcriptomics-based screening identifies pharmacological inhibition of Hsp90 as a means to defer aging. Cell Reports. 27 (2), 467-480 (2019).
- Kasimatis, K. R., Moerdyk-Schauwecker, M. J., Phillips, P. C. Auxin-mediated sterility induction system for longevity and mating studies in Caenorhabditis elegans. G3: Genes, Genomes, Genetics. 8 (8), 2655-2662 (2018).
- Lin, X. -. X., et al. DAF-16/FOXO and HLH-30/TFEB function as combinatorial transcription factors to promote stress resistance and longevity. Nature Communications. 9 (1), 4400 (2018).
- Stroustrup, N., et al. The temporal scaling of Caenorhabditis elegans ageing. Nature. 530 (7588), 103-107 (2016).
- Byerly, L., Cassada, R., Russell, R. The life cycle of the nematode Caenorhabditis elegans: I. Wild-type growth and reproduction. Developmental Biology. 51 (1), 23-33 (1976).
- Perez, M. F., Francesconi, M., Hidalgo-Carcedo, C., Lehner, B. Maternal age generates phenotypic variation in Caenorhabditis elegans. Nature. 552 (7683), 106-109 (2017).
- Wilkinson, D. S., Taylor, R. C., Dillin, A. Analysis of aging in Caenorhabditis elegans. Methods in Cell Biology. 107, 353-381 (2012).
- Hosono, R. Sterilization and growth inhibition of Caenorhabditis elegans by 5-fluorodeoxyuridine. Experimental Gerontology. 13 (5), 369-373 (1978).
- Lithgow, G. J., Driscoll, M., Phillips, P. A long journey to reproducible results. Nature. 548 (7668), 387-388 (2017).
- Zhang, L., Ward, J. D., Cheng, Z., Dernburg, A. F. The auxin-inducible degradation (AID) system enables versatile conditional protein depletion in C. elegans. Development. 142 (24), 4374-4384 (2015).
- Baeriswyl, S., et al. Modulation of aging profiles in isogenic populations of Caenorhabditis elegans by bacteria causing different extrinsic mortality rates. Biogerontology. 11 (1), 53 (2010).
- Banse, S. A., Blue, B. W., Robinson, K. J., Jarrett, C. M., Phillips, P. C. The Stress-Chip: A microfluidic platform for stress analysis in Caenorhabditis elegans. PLoS One. 14 (5), e0216283 (2019).
- Banse, S. A., et al. Automated lifespan determination across Caenorhabditis strains and species reveals assay-specific effects of chemical interventions. Geroscience. 41 (6), 945-960 (2019).
- Swindell, W. R. Accelerated failure time models provide a useful statistical framework for aging research. Experimental Gerontology. 44 (3), 190-200 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved