Method Article
온도에 민감한 치명적인 돌연변이의 높은 처리량 로봇 보조 절연에
요약
Temperature-sensitive (ts) lethal mutants are valuable tools to identify and analyze essential functions. Here we describe methods to generate and classify ts lethal mutants in high throughput.
초록
Systematic identification and characterization of genetic perturbations have proven useful to decipher gene function and cellular pathways. However, the conventional approaches of permanent gene deletion cannot be applied to essential genes. We have pioneered a unique collection of ~70 temperature-sensitive (ts) lethal mutants for studying cell cycle regulation in the unicellular green algae Chlamydomonas reinhardtii1. These mutations identify essential genes, and the ts alleles can be conditionally inactivated by temperature shift, providing valuable tools to identify and analyze essential functions. Mutant collections are much more valuable if they are close to comprehensive, since scattershot collections can miss important components. However, this requires the efficient collection of a large number of mutants, especially in a wide-target screen. Here, we describe a robotics-based pipeline for generating ts lethal mutants and analyzing their phenotype in Chlamydomonas. This technique can be applied to any microorganism that grows on agar. We have collected over 3000 ts mutants, probably including mutations in most or all cell-essential pathways, including about 200 new candidate cell cycle mutations. Subsequent molecular and cellular characterization of these mutants should provide new insights in plant cell biology; a comprehensive mutant collection is an essential prerequisite to ensure coverage of a broad range of biological pathways. These methods are integrated with downstream genetics and bioinformatics procedures for efficient mapping and identification of the causative mutations that are beyond the scope of this manuscript.
서문
모델 생물의 체계적인 돌연변이 컬렉션의 표현형 특성은 세포의 복잡성을 해부에 대한 검증 된 방법입니다. 반수체 단세포 녹조류 인 Chlamydomonas reinhardtii에서 플랜트와 같은 유전자 세트를 가지고 있지만, 토지, 공장, 리니지 2에서 여러 게놈 중복 전에 육상 식물에서 분기. 원칙적으로, 유전자 중복 및 주로 반수체 수명 부족 크게 기능 상실 유전 학적 접근을 용이하게한다. 그러나, 관심 대상 유전자의 파괴로 인해 효율적인 상동 게놈 통합이 부족하는 것은 거의 불가능하다. 임의 삽입 성 장애 라이브러리는 지금까지 1,562 유전자 3을 나타내는 1935 매핑 중단의 배열 된 세트를 산출의 붕괴 사이트의 식별과 결합, 공사중입니다. 그러나, (null의 돌연변이를 생산하기 위해 일반적으로 예상)이 방법은 필수 유전자에 적용 할 수 없습니다. 온도에 민감한 (TS) 돌연변이 recovere 할 수있다필수 유전자 라, 그리고 최근의 방법은 돌연변이 유전자와 원인 병변을 효율적으로 식별 할 수 있습니다. 고온에서의 표현형 분석은 돌연변이 유전자의 기능에 대한 직접적인 정보를 제공한다. 우리는 세포주기 진행에 관여하는 유전자에 특히 초점을 맞추고, 클라 미도 모나스에서 ~ 70 필수적인 유전자 TS 치명적인 돌연변이의 분리 및 특성에보고 된 1,4-을 제어 할 수 있습니다.
TS 치명적인 화면은 수십 년 5, 6에 대한 미생물의 유전 적 분석의 주력이었다. 원칙적으로, 원하는 기능은 치사의 TS하는 변이 가능한 모든 유전자가 완전한 분석을 허용하는 적어도 하나의 돌연변이에 의해 식별되는 것을 의미하는 "포화 상태"에 접근한다. 그러나, 실제로, 여러 가지 요인이 포화에 접근을 제한 할 수 있습니다. 거의 모든 유전자가 고온에서 활성을 상실 돌연변이 될 수 있지만 먼저, 이러한 돌연변이의 회수 효율을 적어도 변한다크기 7,8의 순서. 따라서, 임의의 화면이 포화에 접근 오래 전에 "상용 고객 우대 프로그램"에 재발 히트를 데리러 시작합니다. TS 돌연변이는 일반적으로 기능의 감소가 발생하면서 둘째, 그들은 진정한 제한적인 온도에서 널 (null)하지 않을 수 있습니다 (반대로, 자주 허용 온도에서 정상적으로 작동하지 않습니다). 이 문제는 여러 대립 유전자를 비교하여 어느 정도 처리 할 수있다; 그들 모두는 공통의 표현형을 공유하는 경우,이 유전자의 간단한 비활성화의 결과를 반영 할 가능성이 높다. 여러 대립 유전자는 원인 병변 1의 최종 분자 식별에 매우 도움이됩니다. 그러나, "상용 고객 우대"문제는 거의 충돌하지 유전자의 여러 대립 유전자가 회복하기 어려울 수 있다는 것을 의미한다.
이러한 이유로 분리하고 TS 돌연변이 표현형을 특성화 강화 파이프 라인을 개발 하였다. 우리는 내가 지금까지 3000 TS 돌연변이를 수집200 새로운 후보 세포주기 변이 ncluding. 이미 가능성이 대부분 또는 모든 셀 필수 경로에 변이를 포함하는이 컬렉션의 분자 및 표현형 분석은 식물 세포 생물학에 새로운 통찰력과 가설을 제공해야한다. 중요한 것은,이 파이프 라인을 효율적 TS 돌연변이 모음을 구성하는 배지에서 성장하는 미생물에 적용될 수있다.
장비에 대한주의 사항 :이 로봇이 절차 (식민지 피커 및 이들의 조합으로 복제 도금 공 / 체리 피커)의 효율성을 위해 매우 중요하다. 피커는 일반적으로 금속 핀이 있습니다. 고른 식민지 (모델에 따라, 초 분) 일부 기간 동안이 핀에 공기에서 개최된다. 클라 미도 모나스 공기의 금속 핀 약 20 초에 죽는다. 이 유기체에 대한 모델 선택을 제한합니다. 정확도 문제. 우리의 식민지 피커는 합리적으로 정확하다; 그러나, 그것의 행동에 다소 폭력과 스프레이 기반의 교차 오염에 대한 몇 가지 가능성이있다. 그것은 높은 사용하지 않습니다어보다 384 밀도 (4.5 mm 센터 센터 간격); 이 밀도의 정밀도가 완전히 좋다. 복제 도금 / 체리 따기 로봇 (이러한 응용 프로그램에 사용되는 다른 첨부) 따기 훨씬 느리지 만 (6144에도이 매우 정확 로봇, 도전 인 1536 밀도 충분히 정확, 우리는이 밀도를 평가하지만 선출했다 ) 때문에 여러 가지 어려움을 사용합니다. 플레이트 등 고르지로드하는 경우 로봇이 잘 작동하지 않습니다. 옳은 일이 일어나고 확실하게 자리-확인하는 것이 중요하다; 물론, 로봇은 자동으로 실행되며 일반적으로 모든 처음 몇 판은 정확 잘하면 이동합니다.
프로토콜
1. UV 돌연변이 유발
- 9,10 매체 트리스 - 아세테이트 - 인산 (TAP) 200 직사각형 한천 플레이트 - (100)의 배치를 준비합니다. 며칠 앞서이 판을 준비하고 다음 단계에서 중단 된 세포의 빠른 흡수를 보장하기 위해 건조 벤치에 보관하십시오.
- 25 ° C에서 조명 아래 액체 TAP 100 ㎖에서 100 rpm으로 흔들어 - 문화 클라 미도 모나스 세포 최대 0.2 0.5 광학 밀도 (~ 이일 OD 750).
참고 : 제 구실을 하이 그로 필라의 R (하이 그로 마이신 B 내성을 부여)와 매트 + 파로 R (Paromomycin에 대한 내성을 부여) : UV의 돌연변이는 두 개의 유전 배경에서 독립적으로 수행된다. 항생제 선택은 두 교배형 상보 약물 저항을 제공하는, 임의이다. - 세포가 가능한, 건강 (수영 그대로), 오염없는 것을 확인하기 위해 현미경으로 문화의 각각의 샘플을 확인합니다.
참고 : 자란 문화 "충돌"과위상차 현미경의 "유령"으로 나타나는, 생존 능력을 잃게됩니다. 그들이 포화에 도달하면가는 통 문화를 보관하지 마십시오. 은 "유령"현상은 단계 1.3에서 쉽게 감지 할 수 있습니다. - 0.003 OD 750 문화를 희석. 균주는 운동성이 있고 빛에 반응 방향성 수영로, 균일 한 밀도를 보장하기 위해 알루미늄 호일로 병을 감싸.
- 600 생존자 식민지 판 (그림 1)에 형성 할 것이다 - 세포 살해를 차지, 200 있도록 계획 UV 량에 따라 서스펜션의 밀도를 조정합니다. 자외선 노출 시간에 대한 자세한 내용은 표 1을 참조하십시오.
- 오염을 방지하기 위해, 제조업체의 지시에 따라, 액체 분배기 맞는 작은 튜브 카세트 연결 및 멸균 세척 시리즈를 수행한다.
- 8 × 12 액체 디스펜서를 사용하여 직사각형 판 (그림 1에 2 ㎕를 각 4 × 96 방울을 분배 ). 액체의 얇은 시트로 모든 상품의 병합을 위해 바로 노광을 방지하기 위해 플레이트 커버 플레이트의 가장자리에 약간 탭.
- 매우 심지어 단일 세포의 분산을 위해 전술 한 바와 같이, 건판을 사용하여 신속하게 광에 응답하여 수영장으로부터 세포를 방지 판을 포함한다. 모든 액체가 흡수 될 때까지 커버 플레이트 레벨과 어두운 곳에서 보관하십시오.
- 생존자 중 TS-돌연변이의 최적의 수율을 제공하기 위해 경험적으로 결정 시간의 기간 동안 살균 UV 램프 (30w 살균 UV 관)에서 접시를 놓습니다. 99.9 %의 살인 - 90의 결과에 50cm - (40)의 거리에서 1.5 분 - 여기, 0.5 시간을 사용합니다. 1000 UV에 의한 점 돌연변이의 ~ 10 % 변화 코딩 시퀀스 - 생존자 (100)를 포함한다.
- 팩 즉시 11, 12,이 단계에서 어둠 속에서 작업을 복구로 인해 빛에 의존하는 DNA에 아무 복귀와 UV 조사의 최대 효능을 보장하기 위해조사 후 어두운 상자에 판.
- 실온에서 24 시간 - 8 어둠 속에서 판을 보관하십시오.
- 식민지를 형성하기 위해 조명과 21 °의 C 배양기에서 접시를 놓습니다. 콜로니 형성 10 일 ~ 소요됩니다. 액체의 응축을 지워하기 위해 비닐 봉지와 함께 접시를 포장하고 내부 흡수 용지를 추가해야합니다. 오염 물질이 판을 입력 할 증발 및 응축주기는 달리 액체 필름 경로를 제공 할 수 있습니다.
- 로봇 식민지 따기 (그림 2)에 대한 소스로 관련 스택에 접시를로드합니다. 직사각형 접시에 384 배열에 대한 식민지를 선택하고, 조명 (~ 일주)와 21 ° C에서 그들을 성장.
- 1536 어레이 (4 : 1)로 384 배열을 응축 복제 도금 로봇을 사용하여 (그림 3), 그리고 판은 21 ° C 배양기에서 2 ~ 3 일 동안 성장 할 수 있습니다.
- 각이 판에 1536-배열을 복제하고 (21 ° C 배양기에서 허용 한 장소온도)와 33 ° C 배양기 (제한 온도에 다른). 24 시간에 따라 미리 예열 플레이트의 새로운 세트에 33 ° C에서 접시를 복제하고, 33 ° C 배양기에 배치합니다.
주 :이 차 도금하는 이유는 셀들이 도금 후 처음 세포주기 체포 있더라도 그 치명적인 뮤턴트 경우 상당한 바이오 매스에 축적 할 수있는 화면 반전이다. 보조 복제본이 신호를 제거하고 크게 감도를 증가시킨다. - 33 ° C에서 성장의 3 일 21 ° C에서 성장 5 일 다음 디지털 카메라로 번호판을 촬영. 고정 프레임 판을 잡습니다. 배양 접시 (그림 4)와 함께 촬영 구 정렬 지표로 표시된 "그리드 플레이트"를 사용합니다. 교류 등의 사진 문화 판은 21 ° C, 33 ° C (33분의 21) 이미지를 쌍.
참고 : 야생형이 크게 증가하기 때문에 다른 배양 시간은 성장을 동일하게하기 위해 사용된다빠른 33 ° C에서 21 ° C보다.
TS 돌연변이 후보자 2. 식별 : 첫 화면
- 1536 배열에 배경 이미지 세그먼트를 제거하기 위해 정의 matlab에 이미지 분석 소프트웨어 쌍 33분의 21 판 이미지를 처리. 이 프로그램은 각 위치 (그림 5)에서 검출 된 바이오 매스 (총 픽셀 강도)를 결정합니다.
주 : (지침에 따라 SI 제공) 소프트웨어가 사용하는 배율 판 배향에서 1,536 어레이의 셀의 위치 (각각의 엔트리)를 결정하고 각 셀의 전체 픽셀의 강도를 계산하는 격자 플레이트 이미지를 사용 . 무트 (S (무트 33) / S (WT 33) / S : 각 돌연변이에 대한이 값은 다음 TS + 표준 및 온도 민감성의 정도에 21 ° C 상대에 필요한 성장을 결정하는 조정 가능한 매개 변수에 대한 비교로 정의 21) / S (WT 21), S는 신호 (픽셀입니다배경 차감 후 긴장)은 무트 개별 변이체이고 "WT"는 임의로 선택되지 않은 온도에 민감한 콜로니 TS + 표현형 있었다 (돌연변이 균주)이다. 21 ° C에서의 성장은 S (무트 21) / S (WT 21)로 정의된다. 이 첫 번째 화면에서 위음성 율을 낮게 유지하기 위해 (21 ° C에서 상대적으로 낮은 성장과 온도 감도의 상대적으로 낮은 수준을 허용) 편안한 선택 기준을 적용합니다. - (일반적으로 384 배열에) 로봇을 따기 단일 식민지에 대한 명령 파일과 소프트웨어에 의해 생성 선택한 식민지의 목록을로드합니다. 로봇 지침에 따라 소스 및 대상 접시를 준비하고 배열에 식민지 (그림 6)를 선택합니다.
참고 : 기존 선택기는를 .csv, .txt 인, 또는 .XLS로, 어떤 형식으로 명령 파일이 필요합니다. 모든 선택기 파일 구동 할 수있는 능력을 가질 것이다; 다른 형식은 MATLAB 코드 (제공되는 소스 코드)의 약간의 편집이 필요합니다. - ~ 5 일이 주식 판 성장을위한 21 °의 C 배양기에서 대상 플레이트를 놓습니다.
3. TS의 돌연변이를 확인 : 두 번째 화면
- 단계를 반복 2.1 고른 식민지를 다시 테스트합니다. 일반적으로 30 - UV 돌연변이 생존 초기 콜로니에 대해 5 %의 TS lethals - 콜로니의 50 %가 2 %의 수율에 대해, 명확 lethals의 TS를 다시 테스트한다.
- 반복 두 번 상영 된 TS lethals을 따기위한 단계 2.2,하지만 384 밀도 사각형 접시에 100 식민지의 블록으로 배열 식민지로 설계 수정 명령 파일을. 이것은 다음 단계에서 편리 현미경 검사를위한 것이다. 명령 파일 포맷은 MATLAB 코드에 의해 실행시에 특정된다.
- 제어 여러 WT의 식민지를 포함 ~ 5 일간 21 ° C에서 접시를 배치해야합니다.
4. 초기 표현형 결정
- 단계 3.2 배열 100 블록 플레이트를 복제하고 21 ° C 형 인큐에 배치울란바토르 2 ~ 일 동안 갓 성장 식민지를 얻을 수있다.
- 세 사본 100 블록 플레이트 (4.1)의 새로운 사본을 복제합니다. 컨트롤과 33 ° C 배양기에서 한 21 ° C를 인큐베이터에서 복사본 하나를 놓습니다.
- 로봇 설정에서 세 번째 사본 ( "판을 심사")를 놓고 긴 핀 멸균 물과 접촉하여 식민지를 발견.
참고 : 한천에 고정 클라 미도 모나스 세포가 로봇 현미경 검사에 사용할 수 몇 단일 세포, 세포의 비교적 조밀 한 자리로 처음 착륙하는 경향이있다. , 현미경 검사에 대한 식민지를 최적화 물 한 방울로 초기 전송 세포를 발견하기 (로봇 긴 핀에 의해 전송 된 물의 부피은 ~ 100 NL)입니다. 이것은 많은 고립 된 단일 세포는 초기 피닝 중심을 작은 반경 (~ 1mm)에 세포의 분산을 초래한다. - 시간 0에서 상영 판의 각 지점의 영역의 현미경 사진을 가지고 (단일 여전히 동안, 연속되는 세포) (그림 7)과 배양을 위해 33 ° C에서 접시를 놓습니다. 384 고밀도 어레이의 4.5 mm의 중심 간 간격으로 정지를 제공하도록 설정 변성 테트라 해부 현미경의 수동 단계 제어에 의해 화상의 위치를 결정한다.
- 33 ° C를 인큐베이터에서 심사 플레이트를 제거하고 변화하는 시점에서, 단계 4.4에서와 같이, (10 시간, 20 시간, 48 시간)을 현미경을. 무대, 판 홀더, 스테이지 컨트롤러를 확인하는 것은 정확하게 모든 시점에서 동일한 세포의 이미지를 얻기 위해 조정된다. 빠른 현미경 사진 수집 (~ 10 분)을 수행합니다.
- 는 TS 표현형을 확인하고 이미지 수집하는 동안 온도 변화가 TS 표현형에는 큰 영향이 없다는 것을 확인하기 위해 33 ° C 배양기에서 두 번째 복사본을 사용합니다.
- 현미경 이미지를 분석하고 원하는 기준에 따라 돌연변이 (그림 8)에 대한 선택합니다. 96 배열 한천 플레이트의 마지막 선택 세트를 발견. 확인 각플레이트는 같은 결합 유형 및 약제 내성의 돌연변이가 포함되어 있습니다.
- 각 플레이트를 들어, 마지막 두 개의 열 플러스 (쿼리 돌연변이)과 보완 분석에 대한 부정적인 (WT) 제어를 통합합니다.
새로운 돌연변이 5. 보완 및 연계 테스트에 "상용 고객 우대"
- 질소 무 생식 유도 매체 (10)에 배열 된 콜로니 대용량 전송 96- 웰 (각 콜로니의 밀도는 약 0.2이어야 OD) 마이크로 플레이트. 배우자를 허용 ~ 5 시간 동안 - (20 W 수은 전구 13) 빛 아래 번호판을 품어.
주 :이 단계는 간단한 도금 장치에 복제 로봇 또는 수동으로 설정하거나 수행 될 수있다. - 배우자에 대한 질소가없는 배우자 유도 매체 (10)와 튜브로 대체 저항 카세트를 품고 반대 짝짓기 유형의 쿼리를 일시 중단합니다.
- 상대 믹스트에서 표적 판의 샘플을 믹스20 μL (그림 9)의 URE 볼륨. 다음 ~ 빛 아래에서 10 분, 각 웰 회에서 자리 5 μL : 한 번 연결 테스트를위한 TAP 판에 한 번 보완 테스트를위한 TAP + 5 μM 파로 + 9 μm의 하이 그로 필라에.
- 하룻밤 21 ° C를 인큐베이터에서 연결 판을 품어, 다음 호일을 감싸. 접합 포자 형성 할 수 있도록 5 일간 어둠에 보관하십시오.
- 21 ° C에서 빛 십일 ~에 대한 보완-테스트 판을 품어.
주 : 약물 금액은 이중 이형 diploids의 생존을 할 수 있도록 교정,하지만 그들은 여전히 상대 haploids을 제거하기에 충분합니다. 이 금액은이 특정 프로젝트에 사용되는 표준 저항 카세트 통합에 대한 보정 하였다 새로운 통합으로, 용량은 재조정되어야한다. 대부분의 바이오 매스는 유동 세포 계측법 (그림 9)에 의해 diploids 것으로 확인하지만 아마 감수 분열과 이중 저항의 성장에서 haploids (의 변수 수준segregants)는 일반적으로도 관찰된다. - TS-표현형 식별을위한 두 개의 복사본으로 보완-테스트 판을 복제합니다. ~ 5 일간 33 ° C에서 21 ° C에서 하나의 사본과 사본을 배치합니다.
- TS-표현형에 대한 테스트 식민지로는 2.1 절 (그림 5)에서 설명했다. 쿼리로 보완되지 않은 식민지 가능성이 쿼리 (동일한 유전자의 돌연변이에 대한 이종 대립 유전자 diploids)와 동일한 유전자의 대립 유전자를 나타냅니다.
- 연동 테스트를 위해 단계 5.4 다음, ~ 7 일간 감수 분열과 반수체 segregants의 가지 수 있도록 빛을 다시 판을 이동.
- (카세트가 연결되지 않기 때문에, 반수체 자손의 25 %로 예측)을 두 번 방지 자손에 대한 선택하고 일주일 동안 21 ° C에서 부화 TAP + 10 μM 파로 10 μM 하이 그로 필라에 연동 테스트 판을 복제합니다.
참고 : 이중 저항 haploids에 대한 약물 투여 량을 늘립니다. - 테스트 식민지 TS-표현형에 대한, 같은 내가N 2.1 단계.
참고 : TS-표현형 쿼리와 같은 보완 그룹의 돌연변이 예상 (관찰)된다. 또한, 상기 결합 분석에서 TS-표현형 쿼리를 보완하는 돌연변이에 대해 질의 새로운 돌연변이 간의 긴밀한 결합을 반영 할 수있다. 테스트 쿼리를 보완 돌연변이 더 표현형 분석을 위해 궁극적 원인 병변의 생물 정보학 식별을 위해 파이프 라인을 입력 할 가능성이 새로운 유전자이다.
결과
우리는 클라 미도 모나스에 TS 돌연변이를 분리하는 가속 파이프 라인을 보여줍니다. 세포를 한천 플레이트에 분주하고, 곧 단일 셀 밀도 현미경 빠른 확인 후, 플레이트는 UV 조사 (도 1)이다. 일반적인 조사 콜로니를 확인하고 허용 온도 (그림 2)에서 성장 후 10 일 이후에 배열 형식으로 선택됩니다. 384 형식으로 생성 된 판은 1536 배열 (그림 3)에 병합됩니다. 조사 식민지를 수집이 첫 단계에서, 우리는 지금까지 ~ 20 만 식민지를 배열했다. 600 생존자 식민지 판에 형성 할 것이다 - 현탁액의 밀도는, 죽음을 차지, 200 있도록 계획 UV 량에 따라 조정됩니다. 세 UV 노출 시간 (1.5 분, 1 분, 0.5 분)와 세 밀도는 따라서 (표 1)을 시험 하였다. 경험적으로, 1.5 분의 노출 시간은 TS의 대부분을 수득- 돌연변이 (~ 50 %); 그러나, 지금까지 1 분은 세포주기 후보 대부분을 수득 하였다. 따라서, 향후 UV의 돌연변이 유발 원은 1 분으로 수행 하였다. 중요한 관심사는 초기 및 피킹 (특히 고밀도 형식)이 교차 오염시 튀는. 이 두 번 중복과 같은 돌연변이의 추가 표현형의 원인이 될 수 있습니다. 이러한 시나리오의 가능성을 최소화하기 위해 최적의 크기로 식민지를 수집하는데주의를 기울여야하고, 인접한 식민지 초기 분석에 포착되지 않도록해야합니다.
다음으로, 두 개의 연속 TS-표현형 분석 (그림 5)을 수행하고, 약 3,000 TS-돌연변이는 고립되었고, 표현형 시간 경과 현미경 (그림 8)에 의해 특징. 때문에 특정 기준을 충족 표현형에 대한 관심의 세포주기 연구에서 생물학적 초점에, 우리는 세대 각 대상에 두 개 이상의 대립 유전자를 수집에 관심이없는이자형. 따라서, 우리는 다운 스트림 파이프 라인 (그림 9)에서 높은 재발 유전자를 제거하기 위해 새로 수집 된 후보자에 대한 두 개 이상의 대립 유전자 (쿼리)에 이미 특징으로 유전자 보완 및 연계 시험을 수행 하였다.

그림 1 : 통일 Unmutagenized 세포와 UV 돌연변이 유발의 확산. (가) 384 × 2 μL 상품은 시약 디스펜서를 사용하여 한천 접시에 분배된다. (b) 임의의 플레이트는 단일 셀 밀도를 확인하기 위해 현미경으로 시험하고 1 분간 노출 UV 조사되어있다. 스케일 바는 50 μm의 =. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2. UV-돌연변이 식민지는 384 배열에 대한 고른. (a)는 UV 조사 된 단일 세포를 볼 수 식민지로 십일 ~ 재배된다. 스케일 바 = 2cm. (b) 이미지 분석 프로그램, 특정 파라미터에 따라 분리 콜로니를 인식 및 로봇 (384)로 플레이트를 배열. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

1536-배열 3. 병합 그림. 네 384 형식의 플레이트는 하나 1536 판에 병합됩니다; 한 번 성장, 그들은 TS-표현형에 대한 테스트를 다시 복제됩니다. 을 보려면 여기를 클릭하십시오이 그림의 더 큰 버전.
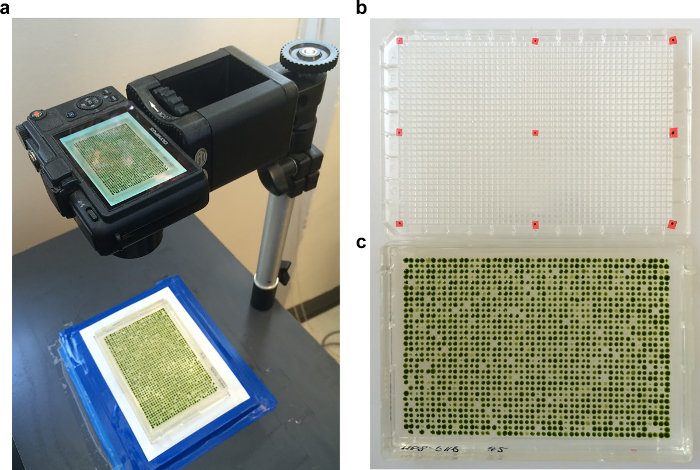
정량 4. 이미지를 그림. 일련의 모든 판은 동일한 배율 및 위치에서 촬영되도록 (가) 플레이트는 문서 사진 스탠드에서 프레임에 유지된다. (b) 첫 번째 이미지는 모서리를 중간에 배치 빨간 점 1536 웰 마이크로 티터 플레이트이다. 이 "그리드 플레이트"는 이미지 어레이의 위치를 정의한다. (c) 배양 한 후 1,536 어레이의 예. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

세미으로 TS-표현형의 그림 5. 확인-automated 이미지 분석. 플레이트 이미지는합니다 (SI 제공) 사용자 정의 MATLAB 소프트웨어로 분석된다. 이미지는 자동으로 셀 어레이 (96, 384 또는 1536)로 분할하고, 각 셀에서의 신호를 정량화 하였다. 21 ° C에서의 성장은 파란색으로 표시되며, 33 ° C에서의 성장은 노란색으로 표시됩니다. 33 ° C에 비해 21 ° C에서 상당히 높은 성장을 보이는 식민지 선택 및 녹색 점과 검은 색 사각형으로 표시됩니다 (TS + 표준화). 이러한 선택은 수동으로 편집 할 수 있습니다. 최종 선택은 식민지 따기 로봇에 의해 사용되는 파일로 전송됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

1 라운드 TS 치명적인 후보자의 그림 6. 로봇 따기. 체리 - pickiNG 로봇은 새로운 배열에 대한 후보를 선택하는 단계 2.1에 설명 된대로 생성 된 파일에서 지침을 따른다. 상단 패널은 살균 핀의 로딩을 보여줍니다. 바닥 판은 소스 접시 특정 콜로니로부터 정확한 피킹을 보여준다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

TS 치명적인 표현형의 초기 정렬 그림 7. 시간 경과 상영. 도 5에 도시 스크리닝 프로토콜의 두 라운드를 통과 클론 개별 세포를 현미경으로 해결 될 수있는 세포 밀도 판에 복제됩니다. 수정 된 테트라 해부 현미경은 현미경 0 시간, 10 시간, 20 시간의 배양 a를 한 후 촬영되는 위치를 식별하는 데 사용됩니다 t 33 ° C를. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
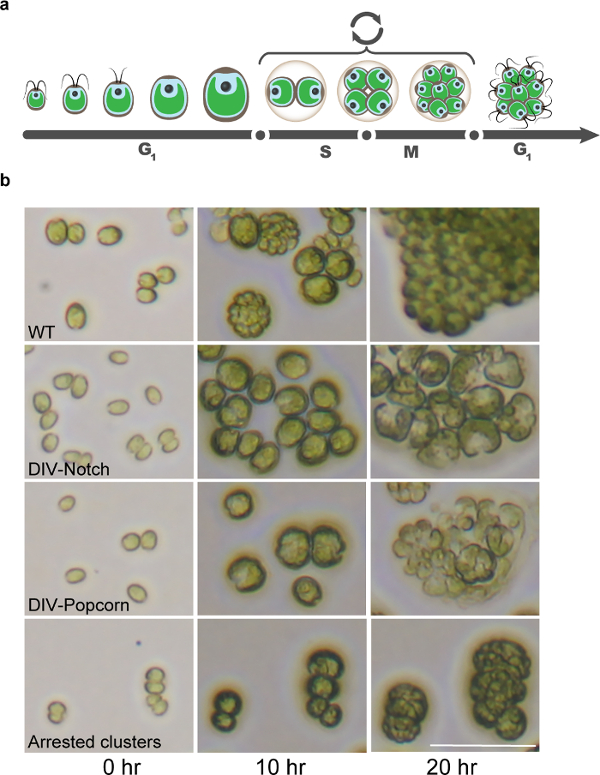
그림 8 : 타임 랩스 현미경에 의해 식별 된 클라 미도 모나스 세포주기의 돌연변이. (a)는 클라 미도 모나스 고유 세포주기의 그림 신생아 딸 세포의 해칭 결국 분열 SM 사이클,이어서 세포 성장을 특징으로 긴 G1 단계를 설명한다. (b) 현미경 이미지는 세포주기 및 유사 분열을 완료하지 전형적인 세포주기 변이의 확인 중에 WT 세포를 보여준다. 스케일 바는 50 μm의 =. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 9 : 보완 및 연동 테스트합니다. (a) 자주 반복 변이 유전자와 항생제 내성 (예를 들어, 하이 그로 필라) 질의 (예 파로) 제 항생제 내성 카세트 항구 대향 결합 형 변이체의 세트를 96 웰 플레이트에서 교차된다. (a에서 하부 왼쪽 패널) 핵산 염색 및 분석을 유동 세포 계측법에 의해 도시 된 바와 같이 보완 판 diploids 높은 분획을 얻었다. (나) 상대 혼합물 diploids의 보완을 위해 두 번 방지 감수 자손의 연동 모두 시험한다. 양성 대조군은 대향 결합 타입의 질의 자체이고, 음성 대조군은 WT이며 TS + 표현형을 도시하는 반면, 예상대로, 33 ℃에서의 TS-표현형을 나타낸다. 원 식민지 쿼리와 더 보완을 표시하지 않고있다 따라서 N쿼리 유전자 EW TS-대립 유전자. 전용 "상용 고객 우대 프로그램"은 보완 테스트 세트에 있기 때문에 이들은 일반적으로, 추가 특성에서 제외됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오. 0.5 '
| 시각 | OD | 식민지가 고른 (#) | TS-표현형 | 세포주기 후보들 |
| 1.5 ' | 0.012 | 6000 (평균 = ~ 200) | 200 (3.3 %) | 7 (3.5 %) |
| 1' | 0.003 | 11,000 | 131 (1.19 %) | 15 (11.4 %) |
| (평균 = ~ 360) | ||||
| 6E-04 | 9000 | 40 (0.45 %) | 1 (2.5 %) | |
| (평균 = ~ 300) |
표 1 : 조사 시간의 교정은 세포주기 돌연변이 후보자의 수율을 극대화합니다. 세 조사 시간 생존 콜로니 번호 (- 600 200)을 보장하기 위해 ODS를 대응 (30 판 각)을 시험 하였다.
토론
TS 치명적인 돌연변이 체의 고 수율의 격리를 위해 여기에 설명 된 파이프 라인은 클라 미도 모나스 게놈의 아마 모든 셀룰러 필수 경로가 표시되도록합니다. 잠재적 인 세포주기 유전자의 효율적인 수집을위한 반복적 인 "마일리지"대립 유전자의 제거를위한 두 가지 가장 중요한 단계는 다음과 같습니다 불완전한 세포주기와 2) 이미 확인에 대한 병렬 보완 분석에 대한 체포 표현형 특성 1) 일관된 정의 새로 고립 된 사람과 컬렉션을 확대하는 유전자를 쿼리합니다.
명암 사이클에 의해 동기화 할 때, 클라 미도 모나스는 DNA 복제 또는 세포 분열 (13)없이 일광 시간 및 셀 크기> 10 배 증가하는 동안 광합성을 성장한다. 밤의 개시와 거의 일치는 세포를 DNA 복제, 유사 분열 및 세포 분열 (도 8)를 교대로 여러 사이클을 겪는다. 이 규정 schem전자는 주로 세포의 성장 및 무결성과 세포 분열주기에 특히 필요한 유전자에 필요한 유전자들 간의 자연스러운 구별을 제공한다. 우리는 10 시간과 20 시간 시점 초기 거친 표현형은 1 컷에 대해 매우 유익한 것으로 나타났습니다. 이러한 이미지를 기반으로 현재 우리가 인식 TS 치명적인 돌연변이의 광범위한 클래스 1 (Tulin 및 크로스, 2014 년 SI 참조), 위치 : 노치, 팝콘, 원형, 소형, 중형, 초기 용해, 여러 사이클 (그림 8 ).
우리가에 초점을 세 가지 가장 중요한 범주는 노치, 팝콘, 그리고 라운드입니다. 은 "노치"및 "팝콘"표현형 대부분의 세포주기 관련 병변의 특징 인 것으로 앞서 도시 된 (예를 들어, 유사 분열 사이클린 의존성 키나아제, DNA 복제 기계, 및 토포 이소 머라 제 II) 1 하였다. 하나 (노치) 또는 복수의 모양 (팝콘) 초기하지만 실패 세포 분열의 명백한 비행기 편리 morphol입니다세포주기 개시 ogical 표시. 이러한 돌연변이는 일반적으로 10 시간 시점에서 WT 유사한 세포 부피가 증가하여, 거의 또는 전혀 결함이 성장을 나타낸다. 노치와 팝콘 표현형은 10 시간에서 분명하고 완전하게 20 시간으로 (자주 세포 용해와 관련된)을 개발하고 있습니다. "라운드"세포 따라서 대형 라운드 체포 세포를 산출, WT에 있지만 분명 초기 분할 평면 많이 감소 생산과 유사하게 성장한다. 이 범주에 이전 돌연변이는 미세 소관 기능 (튜 불린-접이식 보조 인자, 감마 - 튜 불린 링 단지) (1)에 필요한 anaphase (핵분열 말기) - 홍보 (14) 복잡하거나 유전자의 구성 요소로 떨어졌다. 이후의 시간에, 이러한 세포는 종종 현저한 세포 용해를 나타낸다.
"작은"과 "중간"세포는 WT (중간)에 비해 무시할 수 (소) 또는 훨씬 적은 중 성장한다. 현재까지 확인 된 이러한 돌연변이의 대부분은 주석 기본 cellula의 역할을 제안 유전자에 병변이R 성장 과정 (번역 또는 막 생물 발생). (:; : 감소 중간 WT 같은 라운드) 매체와 라운드 사이의 주요 미세한 차별은 10 시간에서 성장의 양에 달려있다. 중소기업 범주가 매우 크고 아마도 세포 경로의 큰 범위의 병변을 반영하기 때문에, 우리는 이러한 범주를 포화하려고하지 않습니다; 그러나, 우리는 다양한 경로에서 손실의 표현형을 이해하는 클래스의 대표를 식별 분자 싶지 않습니다. 두 배우지 범주는 다음과 같습니다 1) 초기 용균 이전에 세포 성장의 증거 2) 다수의 사이클, 10 시간 표시로 녹색 색상의 (손실, refractility의 손실) 무결성을 잃고 돌연변이. 세포는 장기 확산을 수행하기위한 완전한 무능력을 나타내고 있지만,도 10 및 20 시간에서 중량 마찬가지로 증식.
우리는 주로뿐만 아니라, 중소 라운드 세포를 "노치", "팝콘"및 "라운드"를 특성화 및 제외된다같은 새는 돌연변이 그 완전한 몇 세포 분열. 이는 성장과 막 무결성과 같은 기본적인 휴대폰 기능이 작동하는지 확인하고 분할 관련 유전자에 대한 가능성을 풍부하게하기 위해 주로이다. 이 방법은 경험적으로 효율적으로 발견되고; 그러나, 세포주기 유전자 실제 분할 전에,다면 발현과 이전 G1 추가적인 역할을 갖는 것으로 할 수있다. 우리는 드문 것으로 예상 이런 경우는 누락된다. 더 일반적으로, 우리는 높은 확률로 완전히 역기능 단백질로 인해 하나의 원인이되는 돌연변이입니다 균일 한 체포에 대한 목표로하고 있습니다. 그러나, 방금 설명한 것과 동일한 이유로, 여러 구속 포인트가있을 수 있으므로, 유연성 후보를 선택하는 것이 바람직하다.
새롭게 확인 된 유전자 수집 농축하기 위해, 선택된 후보 상보성에 대해 분석한다. 우리는 긍정적 인 컨트롤에서 음성 대조군에 (쿼리 돌연변이에 대한 쿼리) 및 TS +를 TS-을 필요로 (네가 윌리엄 이니WT에 대한 Y). 쿼리와 같은 보완 그룹의 새로운 돌연변이 TS-있습니다. 같은 보완 그룹의 구성원은 거의 변함없이 동일한 유전자의 분자 병변 (이 우리가 테스트 한 모든 이러한 유전자의 경우되었습니다) 반영한다. 따라서, 내용은 "자주 전단지,"이 기준은 추가 특성에 대한 배타적이다. 테스트 쿼리와 같은 보완 그룹에 없었다 돌연변이는 새로운 유전자 후보 또한 생물 정보학 및 실험 도구를 특징으로한다. 다른 보완 그룹으로 TS-대립 유전자의 높은 변수 복구는 잘 알려진 현상-이다가, 변동성 때문에 다른 유전자 사이 TS-하는 가변성의 위대한 본질적인 변화로, 푸 아송 소음보다 현저히 큰 경우이다. 고유 thermolability 차이가있을 수 있습니다 원인; 다른 단백질 크기; 대형으로 복잡한 안정 대 단량체 단백질의 존재; 및 돌연변이 핫스팟. 이것은 거의 순수한 nuisa입니다NCE. 그러나 한 결과 유리한 결과는 "상용"리스트 (목록의 대부분을 차지하는 몇 타겟으로) 긴 아니므 노동 집약적 보완 테스트 프로젝트의 나중 단계까지 방대한 사업 없다는 것이다.
보완적인 접근 방식으로, 우리는 연계 분석을 수행 하였다. 이 분석에서 두 번 강한 자손 선택하고 TS-표현형에 대한 시험한다. TS-표현형 예상 (관찰) 쿼리 또는 밀접하게 연결된 돌연변이 같은 보완 그룹에 돌연변이입니다. 테스트 된 각 유전자를 들어, WT 자손은 유전자의 거리에 따라 일정 확률 (TS + 표현형) 나타날 것으로 기대된다. 우리는이 지점에서 각각의 결합에 대한 약 100 zygospores가있는 것으로 추정된다. 100 % 감수 효율을 가정 할 때,이 때문에 M에 연결되지 않은 약물 내성 카세트에서 약 100 두 약제 내성 자손합니다 (감수 자손의 25 %, 감수 분열 당 네, 발생합니다endelian 상속). 이것은 또한 자손의 25 %가 두 번 돌연변이가 될 것 TS-돌연변이의 경우 것, 쿼리 및 테스트 돌연변이가 연결되지 않은 경우 25 % WT 될 것입니다. 따라서, 두 약제 내성 자손에서, 25 %는 WT (약 25 세포)입니다. 이것은 완전히 연결되지 않은 돌연변이의 경우입니다; 그러나, 중간 링크 (~ 20cm의 약 2 메가 또는 유전체 (115)의 2 % 이내)을 강하게 감소 시키거나 + TS 신호를 제거한다. 항생제 카세트 돌연변이 시험의 결합의 경우, 두 약물 내성 + haploids TS 매우 소량 존재한다. 이 모든 돌연변이가 주목 쉽게되는 비정상적인 결과를 테스트 보완에도 불구하고, 테스트 한 모든 돌연변이와 재결합하는 명백한 실패로 나타난다; 이러한 경우, 교잡은 문제를 해결하는 것이다.
종래 기술에서와 서열 분석에서 모두 우리 MO 있지만, 클라 미도 모나스의 세포주기 유전자의 유전자 약 500이 될 것으로 기대ST는하지만, 아마 모든 필수적이다. 우리는 추가적인 돌연변이 발사 필요성 이상의 돌연변이가 수집 될 때 채도 상승의 정도를 평가한다.
이 절차는 고유의 필수적인 생물학적 과정과이를 수행하는 유전자와 단백질 연구를 위해 설계되었습니다. 다른 방법은 필수 유전자 교란이 존재 생성하는 (예를 들어, 무작위 돌연변이 대립 유전자 (16), 조건부 전사 대립 유전자 (17), 또는 hypomorphic 대립 유전자 (18)의 변환). 그러나, 그들은 모두 강하게 식물 인 Chlamydomonas 억제되는 상동 재조합을 필요로한다. 클러스터 된 정기적으로 interspaced 짧은 회문 반복 (CRISPR) / Cas9 시스템은 유전자 변형 (19)를위한 강력한 도구로 설립되었습니다; 그러나, 클라 미도 모나스 (20)에 효율적으로 작업 할 수 아직. 비판적으로, 이러한 방법의 모든 대상에 대한 사전 지식이 필요합니다. 이것은 심각한 제한 사항입니다하나는 뭔가 새로운 것을 배울 수있는 가능성이하고자하는 경우! 우리의 접근 방식은 사전 지식의 독립적 필수적인 유전자를 식별 돌연변이를 얻을 것입니다. 따라서, 기술의 현재 수준에서 깊은 시퀀싱에 의해 유전자 식별 다음에 임의의 TS 돌연변이의 분리는 식물 superkingdom에서 미생물 세포 생물학에 신속하게 항목을 얻는 가장 효율적인 방법이 될 수 있습니다.
원인이되는 돌연변이의 식별이 논문의 범위를 벗어납니다 (코딩 시퀀스 변화하는 각각의 클론에 돌연변이가 100 ~ 중에서). 중량 화 분리 물 풀 한 깊은 순서는 유효하지만 노동 집약적이다. 균주 다수의 모든 변이를 결정하기위한 조합 적 풀 전략은 풀 소수 시퀀싱 한 후, 매우 비용 및 노동 효과적이다. 조합 중량 화 분리 물 시퀀싱을위한 새로운 전략 시뮬레이션 돌연변이 수십에서 원인 돌연변이의 식별을 허용하는 개발 중입니다(준비) 단일 염기 서열 실행에 ultaneously. 이러한 효율은 중요한 유전자 식별 단계는, 여기에 기재된 절차에 의해 가능하게되는 돌연변이의 매우 빠른 축적에 맞추어 허용하는 것은 매우 중요하다.
공개
저자는 유의 한 금융 이익을 선언합니다.
감사의 말
우리는 조언과 유용한 토론의 크로스 실험실 구성원 감사합니다. 이 작품은 PHS 5RO1 - GM078153에 의해에 의해 지원되었다 마이클 Breker에 시몬스 재단 주니어 펠로우 상.
자료
| Name | Company | Catalog Number | Comments |
| Equipment: | |||
| Hudson RapidPick colony picker | Hudson Robotics | ||
| MultiDrop Combi Reagent Dispenser | Thermo Scientific | 5840300 | |
| Small tube metal tip cassette | Thermo Scientific | 24073295 | |
| Singer RotoR replica-plating robot | Singer Instruments | Very essential for the process. For lower scale screenings you can use an in-house manual tool | |
| Singer single-colony picking attachment (‘Stinger’) | Singer Instruments | Can be picked manually, however for large scales it is nearly impossible | |
| Name | Company | Catalog Number | Comments |
| Materials: | |||
| SYTOX Green Nucleic Acid Stain | ThermoFisher Scientific | S7020 |
참고문헌
- Tulin, F., Cross, F. R. A microbial avenue to cell cycle control in the plant superkingdom. Plant Cell. 26, 4019-4038 (2014).
- Cross, F. R., Umen, J. G. The Chlamydomonas cell cycle. Plant J. 82, 370-392 (2015).
- Li, X., et al. An Indexed, Mapped Mutant Library Enables Reverse Genetics Studies of Biological Processes in Chlamydomonas reinhardtii. Plant Cell. 28, 367-387 (2016).
- Tulin, F., Cross, F. R. Cyclin-Dependent Kinase Regulation of Diurnal Transcription in Chlamydomonas. Plant Cell. 27, 2727-2742 (2015).
- Simchen, G. Cell cycle mutants. Annu. Rev. Genet. 12, 161-191 (1978).
- Hartwell, L. H., Culotti, J., Reid, B. Genetic control of the cell-division cycle in yeast. I. Detection of mutants. Proc. Natl. Acad. Sci. U. S. A. 66, 352-359 (1970).
- Li, Z., et al. Systematic exploration of essential yeast gene function with temperature-sensitive mutants. Nat. Biotechnol. 29, 361-367 (2011).
- Ben-Aroya, S., et al. Toward a comprehensive temperature-sensitive mutant repository of the essential genes of Saccharomyces cerevisiae. Mol Cell. 30, 248-258 (2008).
- Harris, E. . The Chlamydomonas Sourcebook: Introduction into Chlamydomonas and Its Laboratory Use. , (2008).
- Dutcher, S. K. Mating and tetrad analysis in Chlamydomonas reinhardtii. Methods Cell Biol. 47, 531-540 (1995).
- Gill, S. S., Anjum, N. A., Gill, R., Jha, M., Tuteja, N. DNA damage and repair in plants under ultraviolet and ionizing radiations. Sci World J. 2015, 250158 (2015).
- Liu, Z., Wang, L., Zhong, D. Dynamics and mechanisms of DNA repair by photolyase. Phys. Chem. Chem. Phys. 17, 11933-11949 (2015).
- Francis, D. Cell cycle control and plant development. Annual Plant Reviews, Volume 32. Ann. Bot. 101, 1049-1050 (2008).
- Zachariae, W., Nasmyth, K. Whose end is destruction: cell division and the anaphase-promoting complex. Genes Dev. 13, 2039-2058 (1999).
- Merchant, S. S., et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions. Science. 318, 245-250 (2007).
- Ben-Aroya, S., et al. Toward a comprehensive temperature-sensitive mutant repository of the essential genes of Saccharomyces cerevisiae. Mol. Cell. 30, 248-258 (2008).
- Mnaimneh, S., et al. Exploration of essential gene functions via titratable promoter alleles. Cell. 118, 31-44 (2004).
- Schuldiner, M., et al. Exploration of the function and organization of the yeast early secretory pathway through an epistatic miniarray profile. Cell. 123, 507-519 (2005).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827-832 (2013).
- Jiang, W., Brueggeman, A. J., Horken, K. M., Plucinak, T. M., Weeks, D. P. Successful transient expression of Cas9 and single guide RNA genes in Chlamydomonas reinhardtii. Eukaryot. Cell. 13, 1465-1469 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유