Method Article
결정 3'-테르미니와 에이즈-1 중 초기 단일 가닥 바이러스 성 DNA 분자의 순서 반대로 감염 된 세포에서 전사
요약
여기 우리는 초기 3'-테르미니의 공평한 결정 뿐만 아니라 단일 가닥 DNA 분자의 mutational 프로필을 제공 하는 깊은 시퀀싱 방법을 제시. 주요 응용 프로그램은 초기 retroviral 보완 DNAs (cDNAs) retroviral 반전 녹음 방송 과정 중에 생성 된 중간체의 특성 이다.
초록
바이러스 복제 중 핵 산 중간체의 모니터링 바이러스 DNA 합성에 효과 항 바이러스 화합물 및 호스트 세포 단백질의 행동의 메커니즘에 대 한 통찰력을 제공 합니다. 여기 우리가 정의 retroviral 반전 녹음 방송 중간체 생리의 컨텍스트 내에서 바이러스 감염의 수 있는 세포 기반, 높은-범위, 그리고 고해상도 분석 결과의 부족을 해결 합니다. 설명된 방법은 3'-테르미니 단일 뉴클레오티드 해상도 에이즈-1 감염 된 세포 내에서 초기 보완 DNA (cDNA) 분자의 캡처. 프로토콜 전체 세포 DNA, 바이러스 성 DNA 통해 하이브리드 캡처, 어댑터 결 찰, 젤 정화, PCR 증폭, 깊은 시퀀싱 및 데이터 분석에 의해 크기 분류의 대상된 농축의 수확을 포함 한다. 중요 한 단계 3'-DNA 테르미니 열 접합 기 분자의 효율적이 고 공평한 결 찰 이다. 설명된 방식 주어진된 샘플에 각 특정 길이의 역 증명서의 풍부를 결정합니다. 그것은 또한 어떤 잠재적인 돌연변이 역 증명서 및 그로 인하여 (내부) 시퀀스 변화에 대 한 정보를 제공합니다. 일반적으로, 분석 결과 DNA 3'-확장, 관련 된 모든 질문에 대 한 적합 한 서식 파일 시퀀스 알려져 있다 합니다.
서문
해 부하 고 바이러스 복제 완전히, 점점 더 세련 된 복제 캡처 기술을 이해 중간체가 필요 합니다. 특히, 감염 된 세포의 컨텍스트 내에서 바이러스 성 핵 산 종의 정확한 정의 날짜 필요가 많은 바이러스 성 복제 메커니즘 새로운 통찰력을, 제공할 수 있습니다 되었습니다 고립 생체 외에서 반응 검사. 대표적인 예는 레트로 바이러스, 인간 면역 결핍 바이러스 1 (hiv-1) 등에서 반전 녹음 방송 과정입니다. 에이즈-1 반전 녹음 방송, 동안 바이러스 성 효소 역전사 (RT) 복사 단일 가닥 RNA 게놈 이중 가닥 DNA의 다양 한 단계 순화 된 단백질으로 뇌관 확장 분석 실험에서 주로 공부 하 고 핵 되었습니다. 산1,2,3,,45. 기본 원리는 설치 되었다, 하는 동안 이러한 분석 모든 바이러스 및 세포질 구성 요소를 포함 하지 않습니다 그리고 관련된 요인의 생물학 관련 stoichiometries 반영 되지 않을 수 있습니다. 따라서, 우리는 그들의 정확한 cDNA 3'-테르미니 (즉, 그들의 정확한 길이 결정)와 생활의 감염의 맥락에서 뉴클레오티드 순서 반전 녹음 방송 중간체의 스펙트럼을 결정 하는 강력한 기술 설계 셀6. 시간 과정 실험 항 바이러스 분자 또는 단백질, 효율성 및 processivity DNA 합성에 영향을 미칠 수의 존재 등 다양 한 조건 하에서 증명서의 프로 파일을 비교에 이용 될 수 있다에서 데이터 수집 및 축적입니다. 이것은 종종 대상된 약물 설계 및 성공적인 치료 적 개입에 대 한 기초 자연 병원 체 라이프 사이클의 더 상세한 이해가 있습니다.
일련의 다음 빼기 가닥 강한 정지 라는 짧은 단일 가닥 cDNA 녹취 록을 생산 하는 RT에 의해 확장 된 게놈 RNA 서식 파일에 tRNA 뇌관의 어 닐 링에 의해 시작 하는 연속 이벤트를 구성 하는 V-1 반전 녹음 방송 (-sss) (참조 그림 1)입니다. 그 후,-sss cDNA에서에서 전송 됩니다 5'-긴 단말기 반복 (LTR)는 3'-LTR 게놈 RNA, 그것 anneals와 역할 계속된 RT에 대 한 입문서 중재 마이너스의 신장으로 가닥 DNA의 (반전 녹음 방송1 리뷰 보기 , 2 , 3 , 4).이 첫 번째 가닥 전송 반전 녹음 방송;의 속도 제한 단계 중 하나입니다 따라서,-sss cDNA 축적으로 알려져 있다. 감염 된 세포에 반전 녹음 방송 제품을 잡으려고 기본 워크플로 및 라이브러리 디자인 그림 2a에 설명 되어. 특정 뇌관 및 분석 프로토콜에 사용 되 고 표 1 에 나열 된 설정을 대상으로 모두 일찍 반전 전사 cDNA 중간체 23에서 ~ 650의 길이 범위 내에서 nt, 180-182 nt sss DNA를 포함 됩니다. 그러나, 전략에 적절 한 부 적응 하면 후반 반전 녹음 방송 제품 뿐만 아니라 다른 바이러스 및 시스템, 응용 프로그램 목표 3'-OH 포함 DNA 끝을 감지 하는 있습니다. 고려해 야 할 중요 한 제한 포함 최종 PCR 제품의 길이 범위 도서관; 특히, 서식 파일에는 오픈 3'-말단에 어댑터와 업스트림 뇌관 사이트 사이 거리 초과 ~ 1000 nt 것입니다 가능성이 덜 효율적으로 시퀀싱 할 잠재적으로 라이브러리 준비 (동안 기술 편견 오해 소개 자세한 내용 및 적응 제안에 대 한 토론 참조).
이전 3'-테르미니 핵 산 물가의 체계적인 측정 보고 기법 RNA, DNA, 분자에 집중 했다. 하나의 예제 3' 레이스 (cDNA 끝의 급속 한 확대)7, mRNA의 polyadenylation에 의존 하는. 또한, 어댑터 결 찰 기반 전략 RNA ligases 고용 개발 되었다, RLM-레이스 (RNA 리가 중재 경주)8 또는 레이스 (cDNA 끝의 결 찰 기반 확대)9는 포함. 그것은 결 찰 기반 확대는 어떤 바이어스도 자체 결 찰 반응에 의해 도입에 민감한 강조 하는 것이 중요입니다. 예를 들어 결 찰 3' 위치, 순서, 총 분자 길이, 또는 로컬 구조체에 특정 염기에 따라 더 효율적일 수 있습니다. 리가 환경 설정 같은 분자와 우리와 다른9,10관찰 판독에 허위 진술의 불완전 한 캡처 이어질. 여기에 설명 된 프로토콜의 어댑터 추가 단계 동안 결 찰 바이어스를 최소화 하기 위해 우리는 결 찰 전략의 수를 테스트 하 고 T4 DNA 리가 머리 핀 단일 가닥 DNA 접합 기의 사용을 발견 (곽 외에 의해 설명된대로. 11) 유일한 프로시저를 결 찰 효율 제어 oligonucleotides6특별히 선택 된 세트와 함께 평가 하는 때에 큰 차이가 발생 하지 않았다 양적 결 찰에 가까운 것. 이 결 찰 전략의 선택은, 그러므로,이 프로토콜의 성공에 주요 기능.
날짜 하려면, 감염 된 세포에 있는 에이즈-1 RT 진행의 모니터링 주로 성취 되어 고유 하 게 측정 하는 짧은 지 또는 긴지를 뇌관 프로브 세트를 사용 하 여 양이 많은 PCR (정량)와 다양 한 길이의 반전 녹음 방송 제품을 측정 하 여 (초기와 늦게, 각각) cDNA 제품12,,1314. 이 정량 접근은 셀룰러 시스템에서 반전 녹음 방송 과정의 본질적인 효율성을 결정 하는 적절 한, 출력 시퀀스 정보 없이 파생 되 고 상대적으로 낮은 해상도입니다. 우리의 새로운 접근, 기술 격차 최적화 된 어댑터 결 찰, PCR 중재 라이브러리 생성 및 깊은 시퀀싱 주소에 기반 하 고 양적 및 단일 뉴클레오티드에 에이즈-1 감염 시 반전 녹음 방송 모니터링 하는 기회 제공 해상도입니다.
우리 두 제안 된 모델에 대 한 HIV-1 제한 요소 APOBEC3G의 용량 (apolipoprotein B mRNA 편집 효소 촉매 polypeptide 같은 3 세대) 방해 하 사이 구별 하는 연구에서이 메서드의 유틸리티 설명가지고 바이러스 역 사본6의 생산입니다.
프로토콜
참고: 특정 시 약 및이 프로토콜에 사용 된 장비에 대 한 테이블의 자료 를 참조 하십시오.
1. 바이러스 생산 및 세포 감염
주의: 감염 에이즈-1만 승인된 biosafety 제약 실험실에서 처리 되어야 합니다.
참고:15,16인간 미 발달 신장 (HEK) 293T 세포에 설명 된 대로 1.1 단계, 표준 절차 이며 되었습니다의 과도 transfection에 의해 HIV-1 입자의 생산 기술 이전. 일반 세포 배양 절차는 기술 이전17.
- Hiv-1 바이러스 생산입니다.
- 293T 세포 Dulbecco의 수정이 글 중간 (DMEM) 10% 태아 둔감 한 혈 청 (FBS)와 1% 페니실린/스 (전체 DMEM) 37 ° C, 5% CO2 17위에서 설명한 대로 표준 셀 문화 인큐베이터에서 보완을 유지 합니다.
- 표준 층 류 조직 문화 후드에 성장 매체를 제거 하 고 293T 세포의 10 cm 근처 합칠 셀 문화 접시 (~1.2 x 107 셀) 미리 데워 진된 (37 ° C) trypsin의 3 개 mL를 추가 합니다. 다시 2-3 분 인큐베이터에에서 접시를 넣어.
- 접시는 인큐베이터에서 다시 조직 문화 후드에 고 추가 7 mL 전체 매체의. 플라스틱 아래로 접시 내에서 여러 번 셀 resuspend에. 새로운 10 cm 접시에 세포 현 탁 액의 2.5 mL을 추가 하 여 셀 1: 4를 분리 하 고 전체 매체의 7.5 mL와 함께 그것을 채울.
- 다음 날, proviral HIV-1 플라스 미드 DNA (pNL4.3) 등의 10 µ g 1 mL 혈 청 무료 최소한의 필수적인 매체의 혼합 하 고 1 µ g의 DNA 당 4.5 µ L에서 polyethylenimine (페이) 솔루션 (25000 mW, 1 mg/mL pH 7)를 추가 합니다. RT에서 10 분 동안 incubate을 293T 세포에 dropwise 추가 합니다.
- transfection, 후 24 h 매체를 제거 하 고 20 U/mL 매체에서 RNase 무료 DNase를 포함 하는 전체 DMEM의 6 mL와 함께 그것을 대체. 6 h 후 매체를 전체 DMEM의 10 mL 바꿉니다.
- transfection, 후 48 h는 상쾌한 수확 하 고 10 mL 주사기를 사용 하 여 15 mL 폴 리 프로필 렌 튜브에 0.22 μ m 필터를 통해 필터링.
- 얇은-벽으로 ultracentrifuge 오픈 탑 튜브를 버퍼링 하는 인산 염 (PBS) x 1에 메 마른 20% 자당의 2 개 mL를 추가 합니다. 천천히 표면에 뜨는 필터링 된 셀 자당 오버레이.
- ultracentrifuge을 사용 하 여 4 ° C에서 134,000 x g에서 1 h 15 분 동안 원심 분리기.
- ultracentrifuge에서 튜브를 신중 하 게 제거 합니다. 천천히 상쾌한와 자당은 흡입 또는 한 피 펫을 사용 하 여 이륙. 작은 피 펫을 사용 하 고 밖으로 마지막 자당 솔루션을 찍을 때 튜브를 기울기. 튜브의 하단에 있는 수송과 바이러스를 둡니다.
참고: 펠 릿 표시 되지 않습니다. - 1 x PBS의 200 µ L을 추가, 12 h 4에 대 한 냉장고에 두고, resuspend, 및-80 ° c.에서 20 µ L aliquots에 고정
- P24개 그 콘텐츠를 사용 하 여 결정 된 p24 HIV-1 항 ELISA 키트 (다음 제조업체의 지침).
- T-셀 라인 감염입니다.
- 불멸 하 게 T 세포 선 문화 (예., CEM SS 셀) 10 %FBS 1% 페니실린/스 (전체 RPMI)와 보충 로스웰 파크 기념 연구소 (RPMI) 1640 매체에. 2 x 106 셀/mL 12 잘 형식 셀 문화 접시에서와 함께 전체 RPMI의 1 mL와 함께 hemocytometer18 및 샘플 잘 당 종자 1 사용 하 여 셀을 계산 합니다.
- 에이즈-1 입자 추가 150 ng p24에 해당개 그 와 장소는 스윙으로 격판덮개 원심 회전-30 ° c.에 2000 x g에서 2 h 대 벤치탑 분리기에 원심 분리에 의해 감염 biocontainment 뚜껑 물통
- 원심 분리기에서 플레이트를 제거 하 고 37 ° C, 5% CO2표준 조직 문화 인큐베이터에서 1 h 동안 휴식 하자.
- 입력된 바이러스를 씻어, microcentrifuge 튜브 셀 정지 전송 및 500 x g 2 분 동안에 microcentrifuge에서 실시간 (RT)에 centrifuging 세포를 수집 합니다. 벗고는 상쾌한 셀 펠 렛을 방해 하지 않고.
- 미리 데워 진된 (37 ° C)의 1 mL에 셀 알 약 resuspend 살 균 1 x PBS. 두 번 더 원심 분리, 표면에 뜨는 제거 및 물의 resuspension 단계를 반복 합니다.
- 다시 원심는 상쾌한을 제거 하 고 전체 RPMI의 1 mL에 셀 펠 릿 resuspend. 새로운 12 잘 플레이트 한 잘에 각 정지를 추가 합니다.
- 1.2.4 단계에서 완료 6 h 바이러스 (4 h 후 원심 분리)의 초기 포스트 추가에서 원심 분리 하 여 세포를 수확. 제거 하 고 삭제는 상쾌한. 셀 알 약-80 ° C에서 냉동 하거나 DNA 추출에 대 한 직접 처리 될 수 있습니다.
2. DNA 추출, 에이즈-1 DNA 정량화, 및 하이브리드 캡처에 의해 농축
- 조직 배양 세포에 대 한 키트 매뉴얼에 따라 혈액과 조직 전체 DNA 추출 키트와 함께 전체 세포 DNA를 추출 합니다. 유일한 변화는 nuclease 없는 H의 200 µ L에서 차입 제공된 차입 버퍼 대신2O.
참고: chaotropic 세포의 용 해 버퍼 (키트의 "알 버퍼")와 가수분해의 추가, 후 샘플 수 biosafety 제약 실험실에서 제거 되며, 프로토콜의 나머지 부분에 대 한 표준 안전 수준의 실험실에서 처리. - 정량 여 에이즈-1 cDNA의 복사본 수를 결정 합니다.
- 2.1 단계에서 eluate의 17 µ L 고 DpnI 제한 효소의 1 µ L 함께 10 x 제한 효소 버퍼의 2 µ L을 추가. Transfection에서 어떤 잠재적인 잔여 입력된 플라스 미드 DNA를 제거 하는 37 ° C에서 1 시간에 품 어.
- 빼기-스트랜드 강한 스톱 cDNA 다음 뇌관 프로브 세트를 사용 하 여에 대 한 정량 수행: oHC64 (5 '-taactagggaacccactgc-3) 및 oHC65 (5 '-gctagagattttccacactg-3) oHC66를 조사 하 고 (5 '-FAM-acacaacagacgggcacacacta-TAMRA-3). 정량 Pcr 설치 및 정확한 조건 참조6,13에서 찾을 수 있습니다. CDNA 분자의 복사본 수를 결정 하는 표준 곡선으로 직렬 희석 pNL4.3 proviral 플라스 미드와 함께 수행.
참고: 예상된 수량에 대 한 토론을 참조 하십시오.
- 에이즈-1 DNA 하이브리드 캡처로 농축입니다.
참고:이 단계에서 그것은 모든 DNA 샘플에 대 한 낮은 핵 산 바인딩 속성 뿐만 아니라에 어로 졸 필터 피 펫 팁 microcentrifuge 튜브를 사용 하는 것이 좋습니다. 가능 하면 PCR 워크스테이션에서 작동 합니다. 달리 명시 되지 않는 모든 단계 및 시 약 실시간 (RT)에 있습니다.- 마그네틱 streptavidin 구슬의 마스터 믹스를 준비 하려면 단일 microcentrifuge 관으로 샘플 당 100 µ L 구슬 플라스틱. Microcentrifuge 튜브에 대 한 적합 한 자석에 튜브를 놓습니다.
- 구슬 튜브 (~ 1 분)의 자석 쪽을 향해 정착, 한 후 저장소 버퍼 오프, 자석에서 튜브를 제거 bind의 500 µ L에 구슬 resuspend 고 버퍼 (BW 버퍼, 5 mM Tris HCL pH 7.5, 0.5 m EDTA 세척 1 M NaCl)을 씻어.
- 튜브 자석에 다시, 제거는 상쾌한 놓고 500 µ L 카 세 인 솔루션을 추가 합니다. 자석의, resuspend 및 RT에서 10 분 동안 품 어 후 BW 버퍼와 세척.
참고: 세척은 자석에 튜브를 배치는 상쾌한 이륙, 튜브 자석, 오프는 버퍼를 추가 하 고 resuspending 합니다. - 튜브는 자석에 다시,는 상쾌한 벗어 놓고 BW 버퍼의 500 µ L에 구슬 resuspend. 추가할 각 캡처 biotinylated oligonucleotides의 50 pmol (참조 표 1, 3 oligos이 경우) 샘플 당. (예를 들어 5 DNA 샘플 처리 될 경우, 사용 하 여 각 oligonucleotide의 단계 2.3.1 및 250 pmol 자석 구슬의 500 µ L).
- RT에 30 분 이상 끝 믹서에 락을 하는 동안 품 어.
- 1 x 10 버퍼 (10 mM Tris HCl pH 8.0, 1 mM EDTA, 100 mM NaCl)의 500 µ L로 두 번 고정된 oligonucleotides와 구슬 워시.
- 샘플 당 1 x 10 버퍼의 10 µ L에 구슬 resuspend.
- 각 샘플에 대 한 1 개의 microcentrifuge 관 고 (2.1 단계)에서 구슬 정지의 10 µ L, DNA의 170 µ L를 추가 및 90 µ L 10 버퍼 x 3의. DNA를 변성을 2 분 동안 92 ° C에서 건조 열 블록에 품 어.
- 52 ° C로 설정 하는 다른 건조 열 블록 튜브를 이동한 1 h. 정기적으로 혼합 하는 반전에 대 한 품 어 (~ 매 10 분)이 부이 화 하는 동안.
- 10 버퍼 x 1의 500 µ L로 한번 세척 하 고 nuclease 무료 35 µ L에서 resuspend H2o.
- Elute, 92 ° c 2 분 동안 건조 열 블록에서 튜브를 품 어. 다음, 튜브 자석 (한 번에 1 개의 관)에 신속 하 게 이동합니다. 구슬 튜브의 측면에 묶여있다, 일단 에이즈-1 DNA 신선한 튜브를 포함 하는 상쾌한 전송.
- 옵션: 반복 정량 (단계 2.2.2에서에서 수행) 복구 된 HIV-1 cDNA를 결정 합니다.
참고: 예상된 수량에 대 한 토론을 참조 하십시오.
3. 어댑터 결 찰
- 어댑터를 준비
- 동결 건조 된 어댑터 resuspend (참조 표 1 "전체 곽 + MiSeq") nuclease 무료 H에서 100 µ M에서2o.
- 샘플 플러스 컨트롤 샘플, 결합 0.45 µ L x T4 DNA 리가 버퍼, 어댑터의 4 µ L nuclease 없는 H의 0.05 µ L 10의2오 십 분 동안 92 ° C에 고 각 진정 천천히.
참고: 옵션을 사용할 수 경우 조정 가능한 냉각 속도 (사용 2% 비율) PCR 기계를 사용 합니다. 이 92 ° C에서 16 ° c.를 약 30 분 소요 또는, 92 ° C에서 건조 열 블록을 사용 하 고 해제 합니다. 열 블록 실시간 다시 때 어댑터 mastermix를 꺼내 이 어댑터는 머리 핀 구조를 형성 하 게 하는 것입니다 ( 그림 2b참조).
- 세포에서 추출한 DNA 대신 합성된 oligonucleotides ( 표 2참조)의 집합으로 제어 반응 준비.
- 각 oligonucleotide의 100 µ M의 주식을 확인 합니다. 17 oligonucelotides의 각각의 1 µ L를 혼합 하 고 25 µ L의 최종 볼륨에 H2O는 아데닌 비율의 8 µ L를 추가 합니다.
- 희석 nuclease 무료 H에 믹스 1:2,500 직렬 희석에서2O. Nuclease 없는 H의 17.3 µ L 1 µ L 믹스의 결합 각 oligonucleotide 1.6 fmol에 있으면 단계 3.3.1에서에서 컨트롤 샘플 결 찰에 사용 하도록2O (0.026에 해당 60 µ L 반응에 nM).
- Ligations 설정
- PCR 튜브에서 60 µ L 최종 볼륨 반응 결합 T4 DNA 리가 x 10의 6 µ L에 대 한 버퍼, 40%의 24 µ L 말뚝, 5 M betaine, 어댑터 (pre-annelead 단계 3.1.2에서)의 4.5 µ L (400 pmol)의 6 µ L T4 DNA 리가 (2000000 단위/mL)의 1.2 µ L 및 (에서 DNA의 18.3 µ L 2.3.11 단계)
참고: 점성 솔루션 40% 등으로 특별 한 주의 정확한 볼륨을 유지 하는 말뚝. mastermix를 만들지 마십시오. - 수행 단계 3.3.1에서에서 하지만 준비 단계 3.2.2에서에서 제어 oligonucleotide 믹스와 같은 반응에 설정 합니다.
- 반응을 잘 혼합 하 고 하룻밤 16 ° C에서 PCR 기계에 품 어.
- PCR 튜브에서 60 µ L 최종 볼륨 반응 결합 T4 DNA 리가 x 10의 6 µ L에 대 한 버퍼, 40%의 24 µ L 말뚝, 5 M betaine, 어댑터 (pre-annelead 단계 3.1.2에서)의 4.5 µ L (400 pmol)의 6 µ L T4 DNA 리가 (2000000 단위/mL)의 1.2 µ L 및 (에서 DNA의 18.3 µ L 2.3.11 단계)
4. 어댑터 제거 및 크기 분리
- 젤 전기 이동 법을 변성 시키기
- Formamide 포함 된 DNA 젤 각 결 찰 반응 버퍼 로드의 30 µ L를 추가 합니다. Pipetting으로 잘 섞는다.
- PCR 기계에 94 ° C에서 2 분 동안 열 다음 즉시 얼음에 넣어.
- 적절 한 젤 탱크에 캐스트 6% 트리 스/borate/EDTA (TBE) 변성 우 레 아 polyacrylamide 젤 (10-잘 빗)를 배치 합니다. 1 x TBE (89 m m 89 m m 붕 산, 트리 스 베이스 2 mM EDTA) 실행 버퍼를 추가 하 고 미리 20 분 젤 250 V/최대 상수에서 실행.
- 주사기 및 바늘 21 G를 사용 하 여 버퍼를 실행 젤 주머니 세척 한다.
- 부하는 3 개의 우물 (잘 당 30 µ L)과 진한 파란색 염료 앞까지 20 분 (250 V/최대)에 대 한 실행으로 각 90 µ L 샘플에 대 한 중간 젤을 통해 서 이다.
- 얼룩 및 젤에서 절단 핵 산
- 21g 주사기 바늘 (sharps 작업 하는 동안 걸릴 주의)를 사용 하 여 바닥에 구멍을 파고에 의해 샘플 당 3 작은 microcentrifuge 튜브 (0.5 mL)를 준비 합니다. 2.0 mL microcentrifuge 관으로 각각 준비 된 튜브의 삽입 하 고 레이블 그들 샘플 이름과 "낮은", "중간" 또는 "높은".
- 밖으로 하 고 지레 젤 카세트를 엽니다. 아낌없이 소비 세 로드 샘플의 3 웰 스와 스트립을 면도날으로 세로 젤을 잘라. 젤 스트립 1 x TBE (약 30ml) 및 5 µ L cyanine 핵 산 얼룩의 컨테이너에 추가 합니다. 3-5 분 동안 품 어.
참고: 젤 추출 단계는 교차 오염에 특히 민감한. 그것은 젤 및 각 젤 얼룩에 대 한 별도, 깨끗 한 용기를 사용 하 여 1 샘플을 실행 하는 것이 좋습니다. 장갑 젤 입자 gloved 손가락에 문의 하는 경우에 바뀌어야 한다. - DdH2오와 철저 하 게 블루 빛 transilluminator의 표면을 청소합니다 얼룩 컨테이너에서 젤 조각을 하 고 라이트 박스에 추가 합니다.
- 오렌지색 필터를 통해 스테인드 핵 산 검사를 라이트 박스를 켜고.
참고: 어댑터는 일반적으로 오버 로드 된 나타나고 큰 "blob" 합자 HIV-1 dna를 연속으로 실행으로 실행 됩니다. - 아무 샘플 영역 있다면 젤의 면 버려야 새로운 면도날를 사용 하 여 로드 여전히 존재. 다음, 어댑터를 제거 하 고 낮은 부분을 젤을 어댑터 바로 위에 잘라. 마지막으로, 버려야 젤 등의 맨 약 1 m m 젤의 주머니, 자주 있는 높은 분자량 DNA의 날카로운 강렬한 신호.
- 샘플은 일반적으로 2 ~ 3 배를 포함 하는 나머지 젤 조각 분할 cm 3도 조각으로 가로 크기에: "낮은", "중간"와 "높은" 분자량 영역.
참고: 각 조각 지금 처리 됩니다 별도로 [즉., 있을 것입니다 3 튜브 (낮음, 중간, 높은)] 원래 샘플 당. - 3 젤 조각 각 작은 조각 (2 ~ 2 m m 입자 x)으로 잘라내어 준비한 0.5 mL microcentrifuge 튜브 (단계 4.2.1)에 그들을 전송.
- 젤 비 자금 만들려고 2 mL 튜브에 구멍을 통해 젤 조각 집어넣은 1 분 오픈 뚜껑 최고 속도로 회전 합니다. 어떤 젤 입자 0.5 mL 튜브의 하단에 남아 있으면 바늘 이나 피 펫 팁을 사용 하 여 수동으로 2 mL 튜브에 그들을 전송.
- DNA 추출
- 젤 비 자금에 요소 젤 추출 버퍼 (0.5 M NH4채널3CO2, 1 mM EDTA, 0.2 %SDS)의 1 mL를 추가 합니다. 튜브 3 h의 최소 회전 (하룻밤 수락 가능 하다) RT 이상 끝 믹서에.
- 핀셋의 깨끗 한 세트를 사용 하 여 셀 루 로스 아세테이트 멤브레인 필터 (0.2 µ m), 막 막힘 방지와 원심 분리기 열 한 작은 둥근 유리 섬유 필터를 추가 하. 거꾸로 피 펫 팁과 장소에 필터를 넣어.
- 짧게 젤 비 자금 및 추출 버퍼는 microcentrifuge에서 2 mL 튜브를 회전 하 고 준비 된 필터 열에는 상쾌한의 700 µ L를 전송. 젤 비와 나머지 상쾌한 유지.
- 1 분 새로운 2.0 mL microcentrifuge 관으로는 flowthrough 전송에 대 한 최고 속도에서 microcentrifuge에 필터 열 원심
- 나머지 상쾌한 열을 다시 로드 합니다. 구하는 많은 액체 가능한 추출에서 비 자금을 하려고 합니다. 젤의 전송 문제가 아니다. 다시 회전 하 고 같은 추출 샘플의 flowthroughs 결합.
- DNA 강 수
- PolyA RNA의 3 µ L를 추가 (1 µ g / µ L, 운반대로), glycogen, 그리고 단계의 4.3.5는 flowthrough에 소 프로 파 놀의 0.7 mL의 1 µ L. 짧게 소용돌이 및 동결-80 ° C에서 하룻밤.
- -80 ° C 냉동 고에서 샘플을가지고 짧게 재개 하자. 냉각된 (4 ° C) microcentrifuge 및 최고 속도로 30 분 동안 스핀에 넣어.
- 제거 하 고 삭제는 상쾌한. 펠 릿을 제거 하지 않도록 매우 주의 수 있습니다. 그 펠 릿 그렇지 않으면 제거 될 것이 확실 하지 않은 경우 액체의 30 ~ 50 µ L를 남겨 주세요.
참고: 일반적으로 모든 "높은" 샘플 "중간"와 "낮은" 샘플 보다 더 눈에 띄는 펠 릿 표시. - 80% 에탄올의 800 µ L를 추가 합니다. 튜브와 최고 속도에서 1 분 동안 다시 스핀 반전. 피 펫과 에탄올의 대부분을 제거, 짧게 다시 튜브를 회전 하 고 더 많은 에탄올 작은 볼륨 피 펫을 제거 합니다.
- 모든 나머지 에탄올 55 ° C 건조 열 블록으로 오픈 뚜껑 튜브를 배치 하 여 증발 하자. 샘플은 때 건조 (2-4 분) 추가 20 µ L nuclease 무료 h2O와 DNA 펠 릿을 보장 하기 위해 튜브의 바닥 주위 확산 해산. -20 ° c.에 DNA 샘플을 저장할 수 있습니다.
5. PCR 증폭 및 라이브러리 준비
- DNA 중 합 효소 사전 믹스 20 µ L와 40 µ L PCR 반응의 18 µ L 시 켰 던 고 단계 4.4.5, 앞으로 뇌관 "MP1.0 + 22HIV"의 1 µ L에서에서 DNA redissolved (10µM) ( 표 1참조), 그리고 1 µ L 멀티플렉스 올리고 뇌관 (인덱스 뇌관 1 ~ 24)의 (참조 테이블의 엄마 terials).
참고: 각 샘플에 별도 PCR 반응, 하지만 같은 3 반응 (낮은, 중간, 높은) 색인 뇌관 실행. 원래 감염 샘플의 각각에 대 한 다른 인덱스를 사용 합니다.- 다음과 같은 조건 하에서 PCR 반응 실행: 94 ° C 변성, 2 분 후의 3 단계 PCR; 18 주기 15 94 ° C 변성, s 15 s 55 ° C 68 ° c.에 30 s 확장에서 소 둔
- 품질 관리 옵션으로 PCR 반응을 자동화 하는 높은 감도 젤 전기 이동 법 체계와 분석. 제조업체의 지시에 따라 실행 낮은, 중간 및 높은 샘플의 2 µ L를 가져가 라.
주: 2 개의 뇌관 표시 하 고 (실제 길이 다릅니다) 45과 95 nt에 대 한 계산 된 길이에 자주 실행 해야 합니다. 150 ~ 500 사이의 DNA를 감지 해야 합니다 또한, nt. 신호가 없는 경우, 그것은 2와 10 추가 주기 사이 추가 PCR 주기를 추가 하는 것이 좋습니다. 3.3.2 단계에서 만든 oligonucleotide 컨트롤 샘플에 대 한 추가 사이클을 추가 하지 마십시오. - 제거 하는 프라이 머는 상자성 구슬 기반 PCR 정리 시스템을 사용 합니다.
- 각 PCR 반응의 20 µ L를가지고 고 (이 시점에서 모든 샘플 혼합) 샘플 함께 수영장. 나머지 20 µ L 반응-20 ° c.에 백업으로 고정
- 상자성 구슬 구슬 솔루션의 볼륨 x 1.8와 풀링된 PCR 반응 혼합와 RT에 서 보자. Pipetting으로 혼합 하 고 5 분 동안 품 어.
참고: 예, 4 샘플 준비 하는 경우와 각 낮은, 중간, 높은 반응으로, 볼륨 것 20 µ L x 3 x 4 = 432 µ L 비드 솔루션 240 µ L PCR 반응. - Microcentrifuge 튜브 자석에 튜브를 넣어, ~ 1 분에 대 한 바인딩 및 폐기 하는 상쾌한 벗고 구슬. 자석에 튜브를 두고 80% 에탄올 500 µ L을 추가.
- 30에 대 한 에탄올을 두고 s, 다음 철저 하 게 벗어 버리고 nuclease 없는 H의 ~ 5 분 추가 40 µ L의 구슬 airdry2O, 자석, 오프 튜브 고 여러 번 위아래 플라스틱.
- 자석에 다시 5 분 넣어에 대 한 서 스 펜 션 튜브를 두고, 구슬, 측면에 정착 하 고 새로운 튜브에는 상쾌한 전송. 이것은 라이브러리 이다. 10 µ L 품질 컨트롤 aliquot을-20 ° c.에 나머지를 고정
6. 도서관 평가
- 라이브러리 품질, 농도, 및 몰 결정 합니다.
- Fluorometric 정량화 방법을 사용 합니다. 1 µ L 및 높은 감도 dsDNA 분석 결과 키트 제조 업체의 지침에 따라 라이브러리의 3 µ L를 측정 합니다.
참고: 일반적인 농도 1, 10 ng / µ L 사이. - 높은 감도 의해 도서관 DNA 분자량 스펙트럼 측정 자동화 젤 전기 이동 법 (단계 5.2) 위에서 설명한 대로.
- 사용 자동된 젤 전기 이동 법 분석 라이브러리의 평균 분자량을 계산 라이브러리 nuclease 무료 H에 희석2O 4 nM. 모든 인덱스는 고유한으로 여러 라이브러리를 결합할 수 있습니다.
- Fluorometric 정량화 방법을 사용 합니다. 1 µ L 및 높은 감도 dsDNA 분석 결과 키트 제조 업체의 지침에 따라 라이브러리의 3 µ L를 측정 합니다.
- 옵션 낮은 처리량 품질 관리
- 확대를 위한 벡터 라이브러리 분자를 삽입 하19 를 복제 하는 TA를 DNA 도서관을 주제. 키트의 지침에 따라, ~ 10-20 밖으로 성장 식민지와 추출 DNA 통해 miniprep 프로토콜,20여기에 설명 된 대로.
- 로컬 시퀀싱 서비스와 삽입 원하는 V-1를 포함 하는 확인을 사용 하 여 시퀀스 벡터 파생 시퀀스 및 라이브러리 특정 어댑터.
7. 높은 처리량 시퀀싱 실행
- 시퀀싱 플랫폼 제공 하는 상용 소프트웨어와 시퀀싱 샘플 시트를 만듭니다.
- 선택 된 시퀀싱 키트를 나타냅니다. 일반적으로 150 주기 키트를 선택 하지만 다른 원하는 읽기 길이 따라 적당 하다.
- 응용 프로그램 워크플로로 "Fastq만"을 선택 합니다. 24 인덱스 (키트 설명서에 표시 된) 멀티플렉스 oligonucleotide 키트에 포함 된 서식 파일 중 하나를 선택 합니다.
- 선택 "25 nt" Read1에 대 한 및 "125 nt" Read2에 대 한. 계속 6 nt 읽기 단일 인덱스에 대 한.
참고: 내부 분석에만 Read2 분석에 사용 됩니다. Read1 25의 최소한 유지 nt 시퀀싱 플랫폼 알고리즘 목적.
- 정확 하 게 미리 실행된 라이브러리 준비 및 설치에 대 한 제조업체의 지침을 따릅니다. 최대 20 선택 오후 집중력과 매우 낮은 복잡성은 도서관으로 15 %PhiX 스파이크 사용.
8. 데이터 분석
- 패스 필터 백분율 및 평균 Q30 품질 평가 점수 시퀀싱 플랫폼 제조업체의 지침에 따라 허용 되는 경우 확인 하십시오.
참고: 패스 필터는 일반적으로 > 90% 그리고 Q30 점수는 일반적으로 > 80%. - 다운로드는.는 제조업체의 시퀀싱 허브에서 fastq.gz 파일.
- 시퀀싱 스크립트 설정
- "AnalysisXYZ" 라는 새 디렉토리 (폴더)를 만들고이 디렉터리에 모든 소스 코드 파일 (parse_sam.pl, rc_extract.pl, parse.sh)를 다운로드 하려면 https://github.com/malimlab/seqparse로 이동 합니다.
- 같은 디렉터리에 http://bowtie-bio.sourceforge.net/index.shtml에서 짧은 읽기 aligner Bowtie, 1.1.2 버전을 다운로드 합니다.
- 다운로드 "AnalysisXYZ" 라는 "Bowtie-1.1.2" 내에서 하위 디렉터리를 만듭니다. 이 디렉터리 내 하위 디렉터리 "인덱스" 열고 6 확장명의 파일을.ebwt으로 구성 된 템플릿 제공된 시퀀스를 다운로드 합니다.
- FASTQ/A 짧은 읽기 "AnalysisXYZ" 디렉토리에 http://hannonlab.cshl.edu/fastx_toolkit/download.html에서 툴킷 fastx 0.0.13 전처리를 다운로드 합니다.
- "문서" 디렉토리로 Samtools (https://sourceforge.net/projects/samtools/files/) 및 bam readcount (https://github.com/genome/bam-readcount)를 다운로드 하십시오.
- 이동 합니다. 8.2, 모든 단계에서 다운로드 한 fastq.gz 파일을 "AnalysisXYZ" 디렉터리에 2s (끝나는... _R2_001.fastq.gz) 읽기.
- 명령 콘솔/터미널을 엽니다. Cd 명령을 사용 하 여 현재 디렉터리 "AnalysisXYZ"로 이동 합니다. 스크립트를 실행 하려면 "./parse.sh" 입력 합니다.
- 총 읽기 건의 모든 샘플에 대 한 요약 된.csv 파일을 찾아, 길이 조정 수, 읽기 및 읽기 수, 정규화로 "분석 XYZ" 디렉터리에서 parse_results 라는 디렉터리에 각 샘플에 대 한 기본 변형을 파일.
참고: 분석 과정에 대 한 자세한 내용은 토론을 참조 하십시오. 스크립트는 뉴클레오티드 635까지 각 뉴클레오티드 에이즈-1NL4.3 강한 중지 시퀀스와 첫 번째 가닥 전송에 대 한 전체 읽기 csv 파일을 반환합니다. 지도로 50000 100000 고유 읽기는 일반적으로 항 바이러스 단백질 또는 화합물 없이 표시 된 셀 숫자와 바이러스 inocula 감염에서 샘플에서 관찰 됩니다. Oligonucleotide 컨트롤 샘플은 일반적으로 100000 200000 읽기를 생성합니다.
결과
이 문서에서 설명 하는 기술은 HIV-1 반전 녹음 방송 투여 인간의 단백질 APOBEC3G에 의해 (A3G)의 억제를 기본 메커니즘을 해결 하기 위해 광범위 한 연구에 적용 된6. 그림 3 은 CEM-SS T-세포에 감염 된 vif에서 샘플에서 프로토콜을 채용 후 얻은 대표적인 결과-A3G의 존재 또는 부재에 부족 한 HIV-1. 고유한 읽기의 총 수 같은 6 어떤 PCR 중복 항목을 필터링 후 각 샘플에서 얻은 nt 바코드와 동일한 길이 (제공 하는 분석 소프트웨어에서 수행 됨)에 그림 3는그려집니다. A3G의 증가 수준을 RT 중재 cDNA 합성 이전 설명 하 고 측정 하는 정량6,13,,2122에 A3G의 억제 효과 반영 하는 총 읽기 수를 줄일 수 있습니다. 그림 3b, 첫 번째 182 내 각 가능한 길이에 분자의 분수 nt 표시 됩니다. A3G의 부재에 에이즈-1 감염에 대 한 가장 풍부한 종 주 180은 nt 중지 강력한 분자 자체, 일부 축적으로 읽기의 짧은 범위에 (23 ~ 40 nt) (파란 그래프 히스토그램 탑). 이 프로필의 짧은, 날카로운 증가 잘린 몇 가지 매우 구체적인, 재현할 수 위치에서 cDNA의 분자는 A3G 변경의 추가 감지 (중간과 더 낮은 그래프). A3G cytidine deaminase, 시 토 신-uridine (C T로 식별) 이므로 돌연변이 cDNA에서 A3G 감염 virions21,,2324에 때 발생 합니다. 얻은 시퀀싱 정보를 사용 하 여 C T 돌연변이의 비율 (빨간 점선)를 같은 그래프에 플롯 이었다. Mutational 프로필은 모든 독특한 읽기 결합에서 파생 된와 각 뉴클레오티드의 범위는 달라 집니다 주목 한다. 그러나, 시퀀스 정보와 관련 다시 각 분자 수는 특정 3'-말단과 상관 필요한 경우. 제공 하는 데이터는 Pollpeter 그 외 여러분 에서 찍은 6 과 mutational 간의 상관 관계 및 cDNA 길이 프로 파일 때문에 검색 및 분열 세포에 의해 deaminated cDNA의 DNA 복구 기계 시연 했다.
3'-매핑 방법에 대 한 긍정적인 제어 쉽게 알려진된 순서, 길이, 및 농도의 합성 oligonucleotides의 수영장을 처리 하 여 생산 수 있습니다. 이 컨트롤은 단계 3.3.2에서에서 어댑터 결 찰에 추가 하 고 모든 멀티플렉스 라이브러리에 포함 되어야 하는 것이 좋습니다. 컨트롤 샘플에서 가져온 데이터에만 매우 사소한 배경 읽기와 예상된 입력된 비율에서 모든 oligonucleotides 있어야 합니다. 그림 4 는 17 화학적 합성된 oligonucleotides의 긍정적인 컨트롤 집합의 결과 (시퀀스, 표 2참조)는 아데닌 비율에 혼합 했다. 예상 대로, 모든 분자에만 작은 변화 (상단 그래프)와 동등한 풍부에 가까운 나타납니다. 하지는 oligonucleotide로 표현 했다-sss DNA 시퀀스 내에서 대부분 위치 반환 하지만 읽기 수 0, 1 또는 2 nt 실제 제어 oligonucleotides 보다 짧은 부 종을 관찰 합니다. 우리는 이러한 작은 종 더 조사 하지 않은 하지만 저하 또는 불완전 한 제품 구매에 제공된 oligonucleotide 주식에 잠재적으로 존재 나타내는 가정 (oligonucleotides HPLC 정화는으로 지시 했다는 제조 업체 > 80% 순도를 나타냅니다). 아래쪽 그래프 실행, 어디 변이 약간 높은 17 oligonucleotides 사이 이며 더 이상 제어 분자 보다 효율적으로 검색 되는 전체 길이와 상관 관계가 다음 짧은 것 들 다른 라이브러리에서 컨트롤 샘플을 보여 줍니다. 이 PCR 반응에 또는 삽입 크기 최적은 MiSeq 시퀀싱 하는 동안 클러스터링에 사소한 편견 때문일 수 있습니다 하 고 특히 광범위 한 삽입 범위를 운반 하는 라이브러리에서 발생할 수 있습니다. 이 바이어스를 해결 하기 위해 기본적인 방법은 분자 길이 (핑크 라인)을 상호 연결 하는 바이어스를 나타내는 기울기에 따라 정규화 요소의 응용 프로그램입니다. 필요한 계산 분석 프로그램에 포함 되어 있습니다 (프로토콜에 8.3 단계 참조).

그림 1: V-1의 첫 번째 단계 반전 전사 보여주는 다이어그램. 과정 개시와 바이러스 성 cDNA (블루, 2 단계)의 연신 율을 허용 하는 게놈 바이러스 성 RNA (1 단계)에서 뇌관 바인딩 사이트 (PBS)에 tRNA(Lys,3) (오렌지)의 어 닐 링로 시작 합니다. 수반, 템플릿 게놈 RNA RT (3 단계)의 RNaseH 활동에 의해 저하 됩니다. 반전 녹음 방송 과정에서 첫 번째 전체 중간 마이너스 물가 강한-정거장입니다 (-) sss cDNA, RT 촉매 중 합은 5'-말단 gRNA 반복 (R) 지역 (3 단계)에 도달 하면 완료입니다. 중간 (-) sss는 3'-말단 게놈 RNA 서식의 보완 3'-긴 단말기 반복 (LTR) R 영역 어 닐 링에 의해 전송 됩니다. 여기에서 중 합 (4 단계)를 계속합니다. 설명된 방법에 반전 녹음 방송 진행 초기 바이러스 성 cDNA (파란색)의 정확한 길이 매핑하여 결정 됩니다. PPT, polypurine로; U5, 독특한 5'-시퀀스; U3, 독특한 3'-시퀀스 이 그림은 이전 간행물6에서 재 공포. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
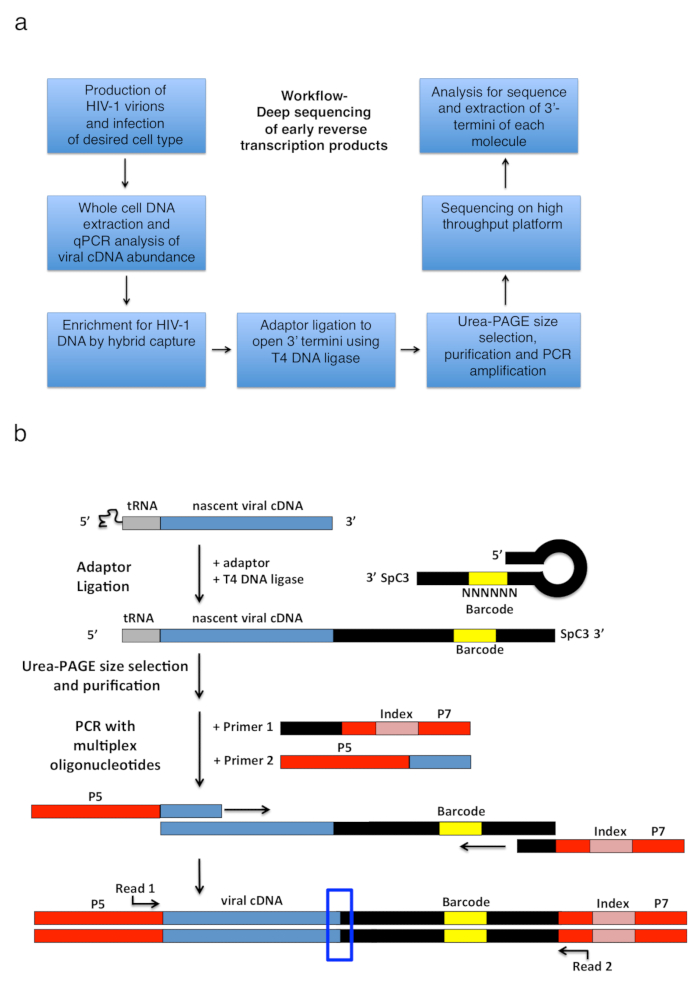
그림 2 : 워크플로 개요 및 어댑터 결 찰 및 PCR 확대 전략의 설계도. (a) 워크플로 개요 설명 기술의 주요 단계 3'-테르미니 에이즈-1의 결정을 반대로 감염 된 세포에서 성적. 그림6이전 간행물 적응입니다. (b)는 접합 기의 결 찰 및 PCR 확대 전략의 회로도 정화 하 고 이전 단계에서 다양 한 길이의 초기 cDNA 분자는 T4 DNA 리가 사용 하 여 단일 가닥 DNA 어댑터에 출혈. 머리 핀 어댑터 (명명 된 "전체 곽 + MiSeq", 표 1참조) 디자인 곽 외에 의해 영감을 했다. 11. 어댑터는 임의의 운반 6 nt 바코드 시퀀스, 결 찰을 촉진 하기 위하여 기본 쌍 수 하 고 동시에 독특한 읽기에 대 한 식별자 역할. 3'-테르미니 어댑터의 스페이서 (SpC3) 각자 결 찰을 방지 하기 위해 수행 합니다. 합자 제품 초과 어댑터에서 변성 시키기 polyacrylamide 젤 전기 이동 법 (페이지)에 의해 구분 됩니다. 젤에 핵 산 스테인드 되며25에서일 우물을 어댑터에서 지역에서 3 개의 별도, 동등한 크기 젤 조각으로 잘라. 차입 후, 강 수, 물의 resuspension, 제품은 어댑터의 알려진된 순서를 단련 하는 뇌관으로 증폭 PCR (뇌관 1, 멀티플렉스 oligonucleotide 키트 재료의 표참조)와 첫 번째 22를 들고 뇌관 에이즈-1의 nt 5'-LTR 시퀀스 직후 tRNA (뇌관 2, MP1.0 + 22HIV). 5'-테르미니 선택한 뇌관의 동일한 라이브러리에서 실행 하는 개별 샘플을 구분 하는 인덱스 시퀀스 뿐만 아니라 선택한 시퀀싱 플랫폼 (P5 및 P7)에 대 한 어댑터를 수행 한다. 시퀀싱의 시작 지점을 읽기 뇌관 표시 됩니다. 파란색 상자는 원래 3'-테르미니 캡처된 분자의 결정에 대 한 관심의 영역을 나타냅니다. 이 그림은 이전 간행물6에서 적응. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
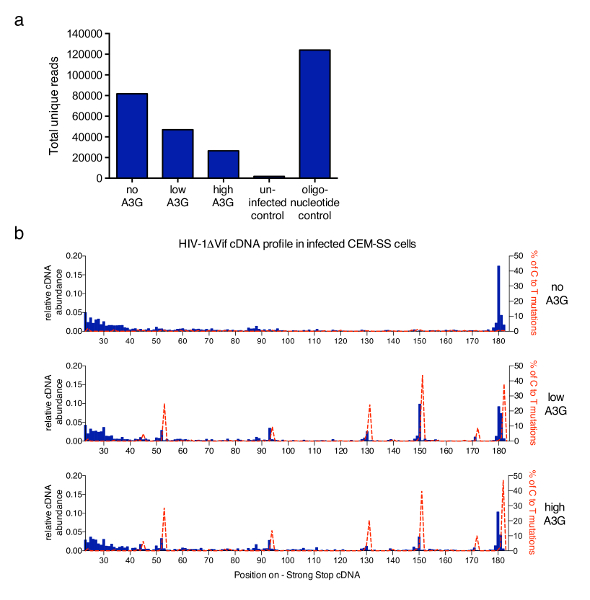
그림 3 : 대표적인 결과. (a) 총 읽기 수 설명 프로토콜 처리 대표 샘플입니다. 그들의 3'-테르미니 첫 번째 635 내와 에이즈-1 분자의 고유한 읽기로 확인 된 모든 시퀀스를 포함 하는이 마이너스의 nt 물가 cDNA (PPT, 최대 그림 1참조). A3G를 실행 하지 않을 에이즈-1 감염 A3G cDNA 합성을 억제 하 고 그로 인하여 총 읽을 수 감소 하는 반면 읽기의 가장 높은 번호를 생성 합니다. 감염 되지 않은 세포 합성 oligonucleotides 집합이 긍정적인 제어를 제공 하는 동안 부정적인 컨트롤로 제공 됩니다. b) nt 위치 23 182 사이 각 길이 대 한 cDNAs의 상대적인 풍부 (sss 전장 cDNA는 180 182 nt) 에이즈-1NL4.3 의 시퀀스 (x 축) 파란색 히스토그램 (왼쪽된 y 축에 규모)에 표시 됩니다. CDNA의 관계 되는 풍부는 시퀀스 182nt를 측정 하는 모든 읽기의 합으로 나눈-sss cDNA 순서 내에서 주어진된 뉴클레오티드에 종료 또는 더 적은의 절대 수에서 계산 했다. 빨간색 파선에은 읽기 들고 각각 위치 (-오른쪽 y 축에 규모)에 C-T/U 돌연변이의 비율입니다. 그림 3 b 는 이전 간행물6에서 재 공포. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
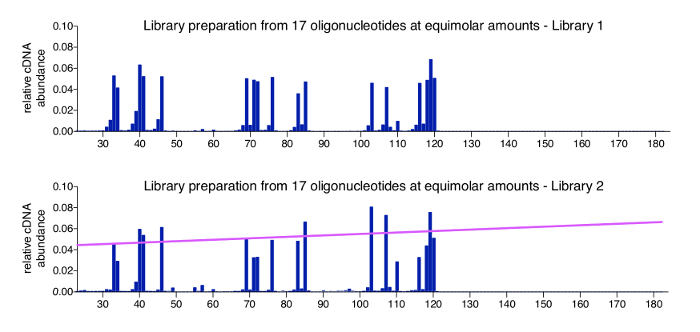
그림 4 : 제어 샘플의 대표적인 결과. 아데닌 금액 17 다른 길이 합성 oligonucleotides의 수영장에 대 한 두 개의 프로필은입니다. 이러한 oligonucleotides 에이즈-1NL4.3 에서 시퀀스 있고 다양 한 길이 커버 하는 3'-뉴클레오티드로 존재 하는 모든 4 기지 선정 됐다 ( 표 2참조). 위쪽 그래프 그림 3는에서 긍정적인 컨트롤 샘플을 보여 줍니다. 분자 길이 나는 오픈 3'-테르미니 향해 아무 중요 한 바이어스 검출 된다. 하단 그래프는 시퀀싱에 작은 길이 바이어스 생산 실행 다른 라이브러리를 보여 줍니다. 이 경우에, 그것은 크기 바이어스를 나타내는 기울기 (분홍색으로 표시)에서 파생 되는 정규화 요소를 적용 하는 것이 좋습니다. 이 그림은 이전 간행물6에서 재 공포. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 올리고 이름 | Nt에 길이 | 시퀀스 | 목적 | 제조업체 (정화) | ||||||||||||||
| 전체 곽 + MiSeq | 61 | 5'-포-tgaagagcctagtcgctgttcannnnnnctgcccatagagagatcggaagagcacacgtct-SpC3-3' | 어댑터 | IDT DNA 기술 (HPLC) | ||||||||||||||
| 2xBiotin SS 미끼 | 40 | 5'-비오 틴-cagtgtggaaaatctctagcagtggcgcccgaacagggac-비타민 b 복합체-3' | 하이브리드 캡처 | MWG Eurofins (HPLC) | ||||||||||||||
| Ss biotin 1-16 | 22 | 5'-cagtgtggaaaatctctagcag-BiTEG-3' | 하이브리드 캡처 | MWG Eurofins (HPLC | ||||||||||||||
| Biotin tRNA + CTG | 16 | 5'-cagtggcgcccgaaca-BITEG-3' | 하이브리드 캡처 | MWG Eurofins (HPLC) | ||||||||||||||
| MP1.0 + 22HIV | 82 | 5'-aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatctcactgctagagattttccacactg-3' | PCR 증폭 | MWG Eurofins (HPLC | ||||||||||||||
표 1: 등 길이, 시퀀스, 활용 하는 수정 설명 프로토콜에서 oligonucleotides의 테이블. 테이블은6이전 간행물 적응. 이 테이블을 excel 파일로 다운로드 하려면 여기를 클릭 하십시오.
| 올리고 이름 | Nt에 길이 | 시퀀스 | 제조업체 (정화) | |||||||||||||
| HTP 죄수 긴 C | 120 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagctttattgaggcttaagc-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 죄수 긴 G | 119 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagctttattgaggcttaag-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 죄수 긴 T | 116 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagctttattgaggctt-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 콘 긴 A | 118 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagctttattgaggcttaa-3' | MWG Eurofins (HPLC) | |||||||||||||
| C 중반 HTP 콘 | 76 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcac-3' | MWG Eurofins (HPLC) | |||||||||||||
| G (a) 중반 HTP 콘 | 71 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacg-3' | MWG Eurofins (HPLC) | |||||||||||||
| G (b) 중간 HTP 콘 | 72 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgg-3' | MWG Eurofins (HPLC) | |||||||||||||
| A 중간 HTP 콘 | 69 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacaga-3' | MWG Eurofins (HPLC) | |||||||||||||
| T 중반 HTP 콘 | 85 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactt-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 콘 짧은 A | 40 | 5'-ctgctagagattttccacactgactaaaagggtctgaggga-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 죄수 짧은 T | 33 | 5'-ctgctagagattttccacactgactaaaagggt-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 죄수 짧은 G | 41 | 5'-ctgctagagattttccacactgactaaaagggtctgaggg-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 죄수 짧은 C | 34 | 5'-ctgctagagattttccacactgactaaaagggtc-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 콘 46 (T) | 46 | 5'-ctgctagagattttccacactg actaaaagggtctgagggatctct-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP Con 83 (C) | 83 | 5'-ctgctagagattttccacactg actaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactac-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP Con 103 (C) | 103 | 5'-ctgctagagattttccacactg actaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagc-3' | MWG Eurofins (HPLC) | |||||||||||||
| HTP 콘 107 (A) | 107 | 5'-ctgctagagattttccacactg actaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagcttta-3' | MWG Eurofins (HPLC) | |||||||||||||
표 2: 긍정적인 컨트롤 샘플으로 사용 하는 17 합성 컨트롤 oligonucleotides의 표. 상위 13 oligonucleotides 크기에 따라 선정 됐다 [긴 (116 ~ 120 nt), 중간 (69 ~ 85 nt), 짧은 (33 41 nt)] 그들의 3'-테르미니에 뿐만 아니라. 테이블은6이전 간행물 적응. 이 테이블을 excel 파일로 다운로드 하려면 여기를 클릭 하십시오.
토론
빠르고, 안정적이 고 비용 효율적인 딥 시퀀싱의 가용성 시퀀싱 기반 분석에 훌륭한 깊이 허용 하는 생명 과학의 분야에서 여러 측면을 혁명 있다. 나머지 도전 시퀀싱 라이브러리는 혁신적인 디자인 및 대표의 창조에 있다. 여기는 초기 바이러스 성 cDNA 분자, 특히 에이즈-1 반전 녹음 방송 과정의 중간체를 잡으려고 프로토콜에 설명 합니다.
이 전략에서 가장 중요 한 단계 양적이 고 공평한 방법으로 한 어댑터는 오픈 3'-테르미니에의 결 찰입니다. 두 ssDNA 테르미니 사이 ligations의 효율성, 둘 다 간-및 intramolecular, 조사 및 다양 한 애플 리 케이 션11,26,27,,2829에 대 한 최적화 되었습니다. 3.3 단계에 설명 된 조건에서 T4 DNA 리가와 머리 핀 어댑터를 사용 하 여의 선택은 우리가 다른 ligases, 어댑터 및 대표 하는 합성 oligonucleotides의 결 찰에 대 한 시 약을 평가 하는 경험적 최적화의 결과 에이즈-1 시퀀스 (표 2) (데이터 표시 되지 않음). 이러한 생체 외에서 시험 반응에서 우리 확인 T4 DNA 리가 머리 핀 접합 기의 결 찰 중재 곽 외에 설명 된 대로. 11, 매우 낮은 바이어스를가 하 고 초과에서 어댑터를 사용할 때 수락자 분자의 완전 한 결 찰 근처 달성 한다. 결 찰 효율은 멀티플렉스 뇌관 시스템에 대 한 호환 되는 어댑터를 렌더링을 뉴클레오티드 순서의 추가 의해 영향을 받는 ( 그림 4참조). 비교에서는, 우리가 발견 한 내 5' DNA/RNA 리가 ("A 리가" 참조 테이블의 자료 여기에 비해 정확한 ligases), 인 한 조작된 RNA 리가 일부 개발 된 ssDNA 수락자로 결 찰 효율 개선 27, RNA 리가 (이 하 "리가 B") 하지만 단일 기본 길이 차이 [표 2 oligonucleotides 사이 결 찰 효율에 강한 차이와 중요 한 바이어스를 했다 보다 두 ssDNA 분자 ligating에서 실제로 더 효과적 이었습니다. ; HTP G (a)와 (b) 중반 죄수]. 또한, 우리 발견만 최소한의 편견 반응에는 무작위로 5'-테르미니 들고 어댑터와 함께 "리가 C" (전략 "리가 C"의 알려진된 뉴클레오티드 바이어스를 오프셋 하는 데 사용; 딩 외예를 참조 하십시오. 그러나 30)., "리가 C"-중재 intermolecular ligations 완료, 렌더링 T4 DNA 리가 시스템 우수한 선택 했다.
프로토콜의 과정 및 긍정적이 고 부정적인 컨트롤의 포함 몇 가지 품질 관리 단계 분석 결과 계속 하기 전에 잠재적인 문제 탐지에 대 한 허용 하 고 노력을 해결 하기 위한 지침을 제공. 2.2.2 및 2.3.12 단계에서 정량 quantifications 입력된 자료의 양이 충분 한지 확인 합니다. 전형적인 cDNA에서 약 10, 000에 300, 000 µ L 당 200 µ L (단계 2.1)에서 차입 범위에 복사 번호. 하이브리드 캡처 단계 전체 에이즈-1의 일부 손실 될 수 있습니다 cDNA 수량을 농축 하 여 전후 genomic DNA를 정할 적절 한 뇌관을 사용 하 여 결정 될 수 있는 세포 DNA에 특정 HIV-1 cDNA의 강한 농축 초래 한다 하지만 정량 Pcr 또는 총 DNA 농도 측정 하 여. 복구 된 HIV-1 cDNA 하이브리드 캡처 단계 후 입력의 적어도 10% 되어야 합니다. 낮은 시작 물자 그렇지 않으면 성공적인 oligonucleotide 긍정적인 통제를 설명할 수 있습니다 (단계 3.3.2 참조) 읽기 샘플에서 달성만 국한. 낮은 읽기 전체 숫자 또한 MiSeq 어댑터 없이 없는 DNA의 존재로 인해 라이브러리 농도의 과대평가 의해 설명 될 수 있었다. 이 낮은 클러스터 밀도 귀 착될 것입니다 그리고 fluorometric 분석 실험에 의해 DNA 총계 뿐만 아니라 정량 하 여 라이브러리에 있는 에이즈-1 시퀀스의 농도 결정 하 여 향상 시킬 수 있습니다. 방법의 매우 민감한 성격 때문에 특별 한 주의 낮은 수준의 오염 뿐만 아니라 실험실 장비에서 (특히에서 높은 농도 제어 oligonucleotide 주식) 다른 샘플에서 둘 다 피하기 위하여 취해야 한다. UV 살 균 PCR 워크스테이션에서이 점에서 유리 하다. 마지막 도서관 (6.1.2 단계)의 자동화 된 젤 전기 이동 법은 더 품질 관리 조치 이다. 일반적으로 관찰 하는 핵 산 크기 범위는 150 ~ 500 nt. 뇌관 PCR 후 고 정화 (단계 5.2에서에서 참고) 수 결 석 하기 전에 선택적 컨트롤에서 검출 될 수 있는 사이입니다. 대표적인 결과 샘플 강도 곡선 피크는 약 160에서 170 nt와 두 번째 선명 피크 약 320 ~ 350 nt. 이 가능성이 상대적으로 짧은 (1 ~ 20 nt 삽입 길이) 역방향 성적표와 전체 길이 강한 정지 (180 182 nt 삽입 길이)에서 자주 본 더 높은 풍부를 반영 한다 (그림 3b).
제시 프로토콜 및 선택 된 뇌관은 초기 HIV-1 반전 녹음 방송 구조에 대 한 특정, 방법은 오픈 3'-테르미니 DNA의 결정을 목표로 모든 연구에 일반적으로 적용 됩니다. 다른 컨텍스트에서 필요한 주요 수정 하이브리드 캡처 및 뇌관 디자인 전략에 대 한 방법 있을 것입니다. 예를 들어 대상이 늦은 HIV-1 성적 증명서에 적응 될 경우, 많은 수는 cDNA의 길이 어 닐 링 하는 다른 캡처 biotinylated oligonucleotides의 권장 것 그리고 하이브리드 캡처 단계에서 손실 가능성이 줄어듭니다. 소개에서 설명 했 듯이, 그것은 한계는 3'-테르미니 바이어스의 다른 소스를 피하기 위해 감지 하는 범위를 설계할 때 고려 하는 것이 중요. 첫째, 있을 수 있습니다 편견 PCR 반응에는 어댑터와 템플릿은 훨씬 다양 한 길이의 경우입니다. 둘째, 시퀀싱 플랫폼 여기 사용 (예: MiSeq)는 최적의 클러스터링 원하는 삽입 길이 범위 고 훨씬 짧고 긴 제품 수 있습니다 하지 시퀀싱 할 같은 효율으로. 부분에서,이 계산, 선형 길이 바이어스에 대 한 수정 계수를 계산 하 여 이루어졌다 해결할 수 있습니다 ( 그림 4, 하단 그래프 참조). 그러나, 3'-테르미니 매핑을 원하는의 영역이 긴 경우 (> 1000 nt), 그것은 더 합자 녹취 록과 함께 반응 고 여러 업스트림 뇌관을 사용 하 여 3'-테르미니 섹션에서 평가 하는 것이 좋습니다.
분석 프로그램 자체 고정된 어댑터 시퀀스에 인접 한 HIV-1 시퀀스의 마지막 뉴클레오티드 모두는 물론 모든 돌연변이 식별 하기 위해 모든 기지의 기본 변화 분석의 특정 목적을 위해 작성 되었습니다. 다음을 구성 하는 개별 단계: 첫째, 어댑터 시퀀스 fastx 0.0.13 툴킷;를 사용 하 여 손질은 다음, (의미는 바코드를 포함 하 여 동일한 시퀀스) 중복 되는 모든 시퀀스는 제거 됩니다. 모든 나머지 독특한 읽기 다음 Bowtie (http://bowtie-bio.sourceforge.net/index.shtml)를 사용 하 여 3 개의 기초에서 설정 최대 불일치와 에이즈-1 시퀀스에 정렬 됩니다. 서식 파일 시퀀스의 첫 번째 635 구성 에이즈-1 cDNA (NL4.3 스트레인)-sss 시퀀스 및 polypurine 트랙 (U5-R-U3-PPT; 그림 1참조)까지 첫 번째 가닥 전송 제품의 nt. 따라서, 제공 된 소프트웨어 및 서식 파일 (V-1NL4.3의 초기 역 증명서의 감지) 같은 응용 프로그램에 대 한 메서드를 사용 하는 경우 직접 적당만 하다. 다른 대상 시퀀스에 대 한 만들 수 조정 해야한다. 3'-테르미니 각 읽기에 대 한의 위치 맞춤에 위치에 의해 결정 되었다. 각 위치에 대 한 기본 호출 기록 하 고 돌연변이 속도 변화, 읽기는 서로 다른 길이의 긴 삽입 하지 적용 될 수 있습니다 완전히 Read2에 125 기반 시퀀싱으로 각 베이스의 전체 범위에서 계산 됩니다.
결론, 설명된 방법 연구의 많은 유형 위한 유용한 도구가 될 믿습니다. 확실 한 응용 프로그램 기본 투여 약물 또는 셀룰러 제한 요소를 통해 반전 녹음 방송 억제 메커니즘의 조사를 포함 합니다. 그러나,만 비교적 작은 조정 3'-테르미니 다른 단일 가닥 DNA 바이러스 중간체, parvovirus 복제에서 예를 들어, 나타나는 내 매핑 시스템에 맞게 해야 한다. 또한, 방법, 특히 그것의 최적화 된 결 찰 단계, 원칙 elongations 세포 이중 가닥 DNA에 의해 촉매를 포함 하 여 모든 3'-DNA 확장 특성에 대 한 라이브러리 준비 디자인의 핵심 부분을 제공할 수 있습니다. polymerases입니다.
공개
저자 들은 아무것도 공개 선언 합니다.
감사의 말
저자 인정 말림 실험실, 루이스 아폴로, Jernej Ule, 그리고 레 베 카 Oakey의 구성원에서 지원 합니다. 저자는 왕의 대학 런던 게놈 센터에서 매트 Arno와 데 비 휴즈 대학 대학 런던 (UCL)에 감사, 연구소 시설에 대 한 신경과 다음 세대 시퀀싱, MiSeq 시퀀싱에 대 한 실행 됩니다. 일 부여 계약 영국 의료 연구 위원회 (G1000196와 미스터/M001199/1 mm은), Wellcome 신뢰 (106223/Z/14/Z mm은 하), 유럽 위원회의 일곱 번째 기구 프로그램 (FP7 2007-2013 년)에 의해 지원 되었다 아니. PIIF-가-2012-329679 (D.P.), 하와는 보건 통해 남자와 세인트 토마스 ' NHS 재단 건강 연구 종합 메디컬 연구 센터 수상 국립 보건원 왕의 대학 런던 그리고 임금의 협력에 대 한 신뢰 대학 병원 NHS 기초 신망입니다.
자료
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | |
| Dulbecco's Modified Eagle's Medium | Gibco | 31966-021 | |
| Penicillin/Streptomycin | Gibco | 15150-122 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| HeraCell Vios 250i CO2 Incubator | Thermo Scientific | 51030966 | |
| Laminar flow hood - CAS BioMAT2 | Wolflabs | CAS001-C2R-1800 | |
| 10mm TC-treated culture dish | Corning | 430167 | |
| TrypLE™ Express (1x), Stable Trypsin Replacement Enzyme | Gibco | 12605-010 | |
| OptiMEM® (Minimal Essential Medium) | Gibco | 31985-047 | |
| HIV-1 NL4-3 Infectious Molecular Clone (pNL4-3) | NIH Aids reagent program | 114 | |
| Polyethylenimine (PEI) - MW:25000 | PolySciences Inc | 23966-2 | dissolved at 1mg/ml and adjusted to pH7 |
| RQ1- Rnase free Dnase | Promega | M6101 | |
| Filter 0.22 μm | Triple Red Limited | FPE404025 | |
| 15 mL polypropylene tubes | Corning | CLS430791 | |
| Sucrose | Calbiochem | 573113 | |
| Phosphate Buffered Saline (1x) | Gibco | 14190-094 | |
| Ultracentrifuge tubes | Beckman Coulter | 344060 | |
| Ultracentrifuge | Sorval | WX Ultra Series | Th-641 Rotor |
| Alliance HIV-1 p24 antigen ELISA kit | Perkin Elmer | NEK050001KT | |
| CEM-SS cells | NIH Aids reagent program | 776 | |
| Roswell Park Memorial Institute Medium | Gibco | 31870-025 | |
| CoStar® TC treated multiple well plates | Corning | CLS3513-50EA | |
| Benchtop centrifuge: Heraus™ Multifuge™ X3 FR | Thermo Scientific | 75004536 | |
| TX-1000 Swinging Bucket Rotor | Thermo Scientific | 75003017 | |
| Microcentrifuge: 5424R | Eppendorf | 5404000060 | |
| Total DNA extraction kit (DNeasy Blood and Tissue kit) | Qiagen | 69504 | |
| Nuclease free H2O | Ambion | AM9937 | |
| Cutsmart buffer | New England Biolabs (part of DpnI enzyme) | R0176S | |
| DpnI restriction enzyme | New England Biolabs | R0176S | |
| Oligonucleotides for qPCR | MWG Eurofins | N/A | HPSF purification |
| TaqMan PCR Universal Mastermix | Thermo | 4304437 | |
| LoBind Eppendorf® tubes | Eppendorf | 30108078 | |
| Axygen™ aerosol filter pipette tips, 1000 μL | Fisher Scientific | TF-000-R-S | |
| Axygen™ aerosol filter pipette tips, 200 μL | Fisher Scientific | TF-200-R-S | |
| Axygen™ aerosol filter pipette tips, 20 μL | Fisher Scientific | TF-20-R-S | |
| Axygen™ aerosol filter pipette tips, 10 μL | Fisher Scientific | TF-10-R-S | |
| PCR clean hood | LabCaire | Model PCR-62 | |
| DynaMag™2-magnet | Thermo | 12321D | |
| Streptavidin MagneSphere® paramagnetic particles | Promega | Z5481 | |
| Casein | Thermo Scientific | 37582 | |
| End over end rotator, Revolver™ 360° | Labnet | H5600 | |
| Tris-Base | Fisher Scientific | BP152-5 | |
| Hydrochloric Acid | Sigma | H1758-100ML | |
| EDTA disodium salt dihydrate | Electran (VWR) | 443885J | |
| Sodium Chloride | Sigma | S3014 | |
| Dri-Block® Analog Block Heater | Techne | UY-36620-13 | |
| PCR tubes and domed caps | Thermo Scientific | AB0266 | |
| PCR machine | Eppendorf | Mastercycler® series | |
| T4 DNA ligase | New England Biolabs | M0202M | |
| 40% Polyethylene glycol solution (PEG) in H2O, MW: 8000 | Sigma | P1458-25ML | |
| Betaine solution, 5M | Sigma | B0300-1VL | |
| Gel loading buffer II (formamide buffer) | Thermo Scientific | AM8546G | |
| Precast 6% TBE urea gels | Invitrogen | EC6865BOX | |
| Mini cell electrophoresis system | Invitrogen, Novex | XCell SureLock™ | |
| Tris/Borate/EDTA solution (10x) | Fisher Scientific | 10031223 | |
| Needle 21 G x1 1/2 | VWR | 613-2022 | |
| SYBR Gold nucleic acid stain (10000x) | Life Technologies | S11494 | |
| Dark Reader DR46B transilluminator | Fisher Scientific | NC9800797 | |
| Ammonium acetate | Merck | 101116 | |
| SDS solution 20% (w/v) | Biorad | 161-0418 | |
| Centrifuge tube filter | Appleton Woods | BC591 | |
| Filter Glass Fibre Gf/D 10mm | Whatman (VWR) | 512-0427 | |
| polyadenylic acid (polyA) RNA | Sigma | 10108626001 | |
| Glycogen, molecular biology grade | Thermo Scientific | R0561 | |
| Isopropanol (2-propanol) | Fisher Scientific | 15809665 | |
| Ethanol, molecular biology grade | Fisher Scientific | 10041814 | |
| Accuprime™ Supermix I (DNA polymerase premix) | Life Technologies | 12342-010 | |
| NEBNext® Multiplex Oligo for Illumina (Index Primer Set 1 and 2) | New England Biolabs | E7335S; E7500S | |
| Tapestation D1000 Screentape High sensitivity | Agilent Technologies | 5067- 5584 | |
| Tapestation D1000 Reagents | Agilent Technologies | 5067- 5585 | |
| 2200 Tapestation - automated gel electrophoresis system | Agilent Technologies | G2965AA | |
| Agencourt® AMPure® beads XP | Beckman Coulter | A63880 | |
| Qubit™ dsDNA HS Assay Kit | Invitrogen | Q32851 | |
| Qubit™ 2.0 Fluorometer | Invitrogen | Q32866 | |
| Topo™ TA cloning Kit | Invitrogen | 450071 | |
| Sequencing platform: MiSeq System | Illumina | ||
| Experiment Manager (Sample sheet software) | Illumina | Note: Use TruSeq LT as a template | |
| Miseq™ Reagent kit V3 (150 cycle) | Illumina | MS-102-3001 | |
| Sequencing hub: Basespace | Illumina | https://basespace.illumina.com | |
| Ligase A: Thermostable 5’ App DNA/RNA ligase | NEB | M0319S | Not used in this protocol, but tested in optimization process with results described in the discussion. |
| Ligase B: T4 RNA ligase 1 | NEB | M0204 | Not used in this protocol, but tested in optimization process with results described in the discussion. |
| Ligase C: CircLigase | Epicentre | CL4111K | Not used in this protocol, but tested in optimization process with results described in the discussion. |
참고문헌
- Herschhorn, A., Hizi, A. Retroviral reverse transcriptases. Cellular and Molecular Life Sciences. 67 (16), 2717-2747 (2010).
- Hu, W. S., Hughes, S. H. HIV-1 reverse transcription. Cold Spring Harbor Perspectives in Medicine. 2 (10), (2012).
- Levin, J. G., Mitra, M., Mascarenhas, A., Musier-Forsyth, K. Role of HIV-1 nucleocapsid protein in HIV-1 reverse transcription. RNA Biology. 7 (6), 754-774 (2010).
- Menendez-Arias, L., Sebastian-Martin, A., Alvarez, M. Viral reverse transcriptases. Virus Research. , (2016).
- Telesnitsky, A., Goff, S. P., Coffin, J. M., Hughes, S. H., Varmus, H. E. . Retroviruses. , (1997).
- Pollpeter, D., et al. Deep sequencing of HIV-1 reverse transcripts reveals the multifaceted antiviral functions of APOBEC3G. Nature Microbiology. 3 (2), 220-233 (2018).
- Frohman, M. A., Dush, M. K., Martin, G. R. Rapid production of full-length cDNAs from rare transcripts: amplification using a single gene-specific oligonucleotide primer. Proceedings of the National Academy of Sciences of the United States of America. 85 (23), 8998-9002 (1988).
- Liu, X., Gorovsky, M. A. Mapping the 5' and 3' ends of Tetrahymena thermophila mRNAs using RNA ligase mediated amplification of cDNA ends (RLM-RACE). Nucleic Acids Research. 21 (21), 4954-4960 (1993).
- Ince, I. A., Ozcan, K., Vlak, J. M., van Oers, M. M. Temporal classification and mapping of non-polyadenylated transcripts of an invertebrate iridovirus. Journal of General Virology. 94, 187-192 (2013).
- Hafner, M., et al. RNA-ligase-dependent biases in miRNA representation in deep-sequenced small RNA cDNA libraries. RNA. 17 (9), 1697-1712 (2011).
- Kwok, C. K., Ding, Y., Sherlock, M. E., Assmann, S. M., Bevilacqua, P. C. A hybridization-based approach for quantitative and low-bias single-stranded DNA ligation. Analytical Biochemistry. 435 (2), 181-186 (2013).
- Abram, M. E., Tsiang, M., White, K. L., Callebaut, C., Miller, M. D. A cell-based strategy to assess intrinsic inhibition efficiencies of HIV-1 reverse transcriptase inhibitors. Antimicrobial Agents and Chemotherapy. 59 (2), 838-848 (2015).
- Bishop, K. N., Verma, M., Kim, E. Y., Wolinsky, S. M., Malim, M. H. APOBEC3G inhibits elongation of HIV-1 reverse transcripts. PLoS Pathogens. 4 (12), 1000231 (2008).
- Zack, J. A., Haislip, A. M., Krogstad, P., Chen, I. S. Incompletely reverse-transcribed human immunodeficiency virus type 1 genomes in quiescent cells can function as intermediates in the retroviral life cycle. Journal of Virology. 66 (3), 1717-1725 (1992).
- Adachi, A., et al. Production of acquired immunodeficiency syndrome-associated retrovirus in human and nonhuman cells transfected with an infectious molecular clone. Journal of Virology. 59 (2), 284-291 (1986).
- Shah, V. B., Aiken, C. In vitro uncoating of HIV-1 cores. Journal of Visualized Experiments. (57), (2011).
- JoVE Science Education Database. Science Education Database: Basic Methods in Cellular and Molecular Biology: Passaging Cells. Journal of Visualized Experiments. , (2018).
- JoVE Science Education Database. JoVE Science Education Database: Basic Methods in Cellular and Molecular Biology: Using a Hemocytometer to Count Cells. Journal of Visualized Experiments. , (2018).
- Zhou, M. Y., Gomez-Sanchez, C. E. Universal TA cloning. Current Issues in Molecular Biology. 2 (1), 1-7 (2000).
- Zhang, S., Cahalan, M. D. Purifying plasmid DNA from bacterial colonies using the QIAGEN Miniprep Kit. Journal of Visualized Experiments. (6), 247 (2007).
- Mangeat, B., et al. Broad antiretroviral defence by human APOBEC3G through lethal editing of nascent reverse transcripts. Nature. 424 (6944), 99-103 (2003).
- Gillick, K., et al. Suppression of HIV-1 infection by APOBEC3 proteins in primary human CD4(+) T cells is associated with inhibition of processive reverse transcription as well as excessive cytidine deamination. Journal of Virology. 87 (3), 1508-1517 (2013).
- Harris, R. S., et al. DNA deamination mediates innate immunity to retroviral infection. Cell. 113 (6), 803-809 (2003).
- Zhang, H., et al. The cytidine deaminase CEM15 induces hypermutation in newly synthesized HIV-1 DNA. Nature. 424 (6944), 94-98 (2003).
- Konig, J., et al. iCLIP--transcriptome-wide mapping of protein-RNA interactions with individual nucleotide resolution. Journal of Visualized Experiments. (50), (2011).
- Troutt, A. B., McHeyzer-Williams, M. G., Pulendran, B., Nossal, G. J. Ligation-anchored PCR: a simple amplification technique with single-sided specificity. Proceedings of the National Academy of Sciences of the United States of America. 89 (20), 9823-9825 (1992).
- Zhelkovsky, A. M., McReynolds, L. A. Structure-function analysis of Methanobacterium thermoautotrophicum RNA ligase - engineering a thermostable ATP independent enzyme. BMC Molecular Biology. 13 (24), (2012).
- Li, T. W., Weeks, K. M. Structure-independent and quantitative ligation of single-stranded DNA. Analytical Biochemistry. 349 (2), 242-246 (2006).
- Gansauge, M. T., et al. Single-stranded DNA library preparation from highly degraded DNA using T4 DNA ligase. Nucleic Acids Research. 45 (10), 79 (2017).
- Ding, Y., Kwok, C. K., Tang, Y., Bevilacqua, P. C., Assmann, S. M. Genome-wide profiling of in vivo RNA structure at single-nucleotide resolution using structure-seq. Nature Protocols. 10 (7), 1050-1066 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유