JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
집단 운동 특성을 분석하기 위해 1차 마우스 배아 성 성 메센키메 세포의 격리 및 시간 경과 이미징
요약
우리는 2차원 (2D) 성장 및 상처 복구 분석의 시간 경과 화상 진찰을 위한 1 차적인 마우스 배아 성 구형 중간엽 세포의 격리 그리고 문화를 위한 프로토콜을 제시합니다. 우리는 또한 세포 스트림 형성 및 방향 운동성을 결정하기 위하여 시간 경과 화상 진찰 데이터의 분석을 위한 방법론을 제공합니다.
초록
입맛의 발달은 혀 옆에 있는 양측 의 성술 선반의 수직 성장을 수반하는 역동적인 과정이며 혀 위의 고도 와 융합이 뒤따릅니다. 이 프로세스에 있는 결점은 갈라진 구개, 일반적인 출생 결함으로 이끌어 내습니다. 최근 연구에 따르면 성구 선반 고도에는 선반의 방향을 수직에서 수평으로 변환하는 리모델링 프로세스가 포함됩니다. 이 동적 리모델링에서 성구 선반 중간엽 세포의 역할은 연구하기 어려웠습니다. 시간 경과-이미징 기반 정량 분석은 최근 1차 마우스 배아 성 구종 중간엽(MEPM) 세포가 집단 운동으로 자가 조직할 수 있음을 보여주기 위해 사용되었습니다. 정량적 분석은 미각 고도 결함이있는 마우스 모델에서 돌연변이 MEPM 세포의 차이를 식별 할 수 있습니다. 이 논문은 E13.5 배아에서 MEPM 세포를 분리하고 배양하는 방법을 설명하며, 특히 시간 경과 이미징을 위해- 그리고 스트림 형성, 모양 정렬 및 방향의 지속성을 위한 측정을 포함하여 집단 운동의 다양한 세포 특성을 결정합니다. 그것은 MEPM 세포고도의 동적 과정에서 구성 선반 mesenchyme의 역할을 연구하기위한 프록시 모델 역할을 할 수 있음을 posit. 이러한 정량적 방법을 사용하면 두개골 면안면 분야의 조사관이 대조군 및 돌연변이 세포의 집단 운동 특성을 평가하고 비교할 수 있으며, 이는 팔성 선반 고도 동안 중간엽 리모델링에 대한 이해를 증대시깁니다. 더욱이, MEPM 세포는 일반적으로 집단 세포 운동의 조사를 위한 희소한 중간엽 세포 모형을 제공한다.
서문
미각 발달은 성기사 발생에 결함이 갈라진 입맛-고립된 케이스에서 또는 증후군1의수백의 일환으로 생기는 일반적인 출생 결함으로 이끌어 내면서 광범위하게 연구되고 있다1,2. 배아 입맛의 발달은 배아 조직의 운동과 융합을 수반하는 동적 과정입니다. 이 과정은 네 가지 주요 단계로 나눌 수 있습니다 : 1) 성술 선반의 유도, 2) 혀 옆에 있는 성구 선반의 수직 성장, 혀 위의 성구 선반의 3) 고도, 그리고 4) 중간선1,3,4에서성구 선반의 융합. 지난 수십 년 동안 많은 마우스 돌연변이체가갈라진 구개5,6,7,8을나타내는 것으로 확인되었습니다. 이러한 모델의 특성화는 성구 선반 유도, 증식 및 융합 단계의 결함을 나타내고 있습니다. 그러나, 성구 선반 고도 결함은 드물다. 따라서, 성구 선반 고도의 역학을 이해하는 것은 연구의 흥미로운 영역입니다.
성병 선반 고도 결함이있는 일부 마우스 돌연변이체의 신중한 분석은 성병 선반의 매우 전방 영역이 뒤집는 것처럼 보이는 것을 보여주는 현재 모델로 이어졌으며, 성골 선반의 수직적 또는 "리모델링"은 입맛1,3,4의중간에서 후방 부위로발생합니다. 9,10,11. 성구 선반의 내측 가장자리 상피는 이 리모델링에 필요한 신호를 시작하고, 이는 그 후 성구 선반 mesenchyme에 의해 구동됩니다. 최근, 많은 연구자들은 태아 선반(12,13)과관련된 일시적인 경구 접착력을 보여 마우스 모델에서 성구 선반 고도 지연을 확인했다. 중간엽 리모델링은 수평 방향으로 벌지를 만드는 세포의 재구성을 포함하며 동시에 수직 방향9,10,14에서성구 선반을 후퇴시킵니다. 성병 선반 고도 및 근본적인 중간엽 리모델링에 영향을 미치기 위해 제안된 여러 메커니즘 중세포증식(15,16,17,화학작용성 그라데이션18,및 세포외 매트릭스 구성요소19,20)이있다. 중요한 질문이 발생: Specc1l-결핍마우스에서 관찰된 성환 선반 고도 지연은 또한 부분적으로 성병 선반 리모델링의 결함으로 인해, 그리고 이 리모델링 결함은 1차 MEPM 세포의 행동에 본질적인 결함에서 나타날 수있는 21?
1차 MEPM 세포는 유전자 발현22,23,24,25,26, 27,28,29,및증식(30,31) 및 이주를 포함하는 몇 가지 연구를 위해 두개안면 분야에서 사용되어왔다. 그러나 집단 세포 행동 분석에 대한 것은 없습니다. MEPM 세포의 시간 경과 이미징은 MEPM 세포가 집단운동(21)의방향 이동 및 형성된 밀도 의존세포 스트림-특성을 표시한다는 것을 보여주기 위해 2D 배양 및 상처 수리 소약으로 수행되었다. 더욱이, Specc1l 돌연변이 세포는 더 좁은 세포 스트림을 형성하고 높게 가변적인 세포 이주 궤적을 보여주었습니다. 이러한 조정된 운동성의 부족은 Specc1l 돌연변이배아(13,21)의미각 고도 지연에 기여하는 것으로 여겨진다. 따라서, 1차 MEPM 세포를 이용한 이러한 비교적 간단한 작용체는 성술 선반 고도 동안 중간엽 리모델링을 연구하기 위한 프록시역할을 할 수 있다. 이 논문은 2D 및 상처 복구 분석을 위한 1차 MEPM 세포의 격리 및 배양뿐만 아니라 시간 경과 이미징 및 분석을 설명합니다.
프로토콜
동물과 관련된 모든 실험은 KUMC 기관 동물 관리 및 사용 위원회가 승인 한 프로토콜로 수행되었으며 지침 및 규정에 따라 수행되었습니다 (프로토콜 번호 : 2018-2447).
1. 수확 E13.5 배아
- CO2 흡입 챔버를 사용 하거나 기관 동물 관리 및 사용 위원회에 의해 승인 된 절차에 의해 임신 한 여성 마우스를 안락사. 즉시 해부로 진행합니다.
- 피부와 복리료를 제거하여 복강의 열등한 절반을 노출합니다. E13.5 배아를 포함하는 자궁의 두 뿔을 소비합니다.
- 자궁을 미리 37°C멸균 인산염 완충식식염(PBS)에 잠깐 배치하여 과도한 혈액, 머리카락 또는 기타 이물질을 헹구는다. 멸균 PBS로 채워진 멸균 10cm 접시에 자궁을 놓습니다.
- 작은 가위를 사용하여 자궁벽을 잘라 자궁의 길이를 따라 각 배아를 노출시켰습니다. 배아를 둘러싼 노른자 주머니를 제거하지만 필요한 경우 지노티핑을 위해 저장하십시오. 배아가 제거되면 각 배아를 PBS로 채워진 12웰 플레이트의 자체 우물에 놓습니다.
2. 배아에서 성구 선반의 해부(그림 1)
참고: 스테인레스 스틸 해부 기구 (재료의 표참조)는 100 % 에틸 알코올 (EtOH)의 비커에 먼저 악기를 배치한 다음 10 s에 대한 350 °C의 기기 소독기에 배치 한 다음 100 % EtOH의 두 번째 비커로 냉각하여 각 배아를 처리 한 후 살균합니다.
- 멸균 천공 숟가락을 사용하여, 10 % 태아 소 세럼 (FBS), L-글루타민 (4 mM L-Glu), 항생제 - 페니실린 및 성병 절제술 (50 단위)를 포함하는 덜벡코의 최소 필수 매체 (DMEM)로 구성된 MEPM 배양 매체로 채워진 새로운 10cm 접시에 배아를 배치합니다.
- 멸균 가위를 사용하여 턱 선 바로 아래에 배아를 참수하십시오(그림 1A,빨간색 점선). 살균 된 미세 #5 개의 집게의 1 점을 입에 삽입하여 아래턱을 제거하여 뺨 내부에 유지하십시오. 삽입된 포스의 점을 두개골 뒤쪽으로 나갈 때까지 밀어 넣습니다.
- 도 1B의노란색 선을 따라 집게를 방향을 지정하여 집게의 반대편(배아 의 외부)이 외이도 바로 위에 떠있는 다음 집게를 꼬집어 조직을 잘라냅니다. 필요한 경우, 핀치에 의해 완전히 절단되지 않은 조직을 절단하기 위해 지금 폐쇄 된 집게의 솔기를 따라 또 다른 미세 한 집게를 실행합니다.
- 배아 헤드의 반대편에 대한 이전 단계를 반복한다. 핀치 컷 절차를 계속하여 두개골의 아래턱, 혀 및 열등한 부분을 완전히 제거하고 성술 선반을 노출하십시오.
- 도 1C(녹색선)에 도시된 것처럼 눈 바로 위를 절단하여 두개골의 두개골을 제거합니다. 머리를 왼쪽 또는 오른쪽에 놓고 작은 스테인리스 스틸 가위의 지점을 배아의 눈 높이 바로 위에 두개골 앞과 뒤에 배치하여 이렇게 하십시오. 가위의 빠른 스니핑으로 두개골 의 상단을 잘라, 이후 단계에서 안정성에 중요 할 것이다 평평한 표면을 만들고 그 측면에서 볼 때 그림 1D처럼 보일 것이다.
- 머리의 나머지 부분을 거꾸로 놓고, 머리의 우수한 측면 (두개골 제거) 접시의 바닥에 평평하게 놓여, 이는 성추 선반 제거를위한 안정적인 표면을 제공 할 것이다. 잠시 동안 발성 선반을 식별하여, 현재 노출되어 위를 향하고 있으며 머리의 앞쪽 절반에 있는 중앙 홈의 양쪽에 두 개의 올려진 능선으로나타납니다(그림 1E).
- 선반을 제거하는 동안 그것을 고정하기 위해 접시에 머리의 나머지 부분을 고정합니다. 머리의 비강 부위 근처의 조직을 통해 미세 포셉의 1 점을 삽입하여, 성추 선반에 전방, 그리고 두개골의 베이스를 통해 집게의 다른 점을 삽입하여, 후신선반에. 이 것들을 제자리에 고정시키면서, 성추선반의 절제를 수행한다.
- 한 손으로 머리를 고정하고, 두 선반 중 하나를 먼저 제거하고, 선반의 측면 표면의 기지에서 조직에 미세 집게의 두 번째 쌍의 두 점을 삽입하고 조직을 잘라 꼬집습니다(도 1F). 선반의 내측 표면 의 베이스를 따라 반복한 다음 선반의 전방 및 후방 끝에서 머리에서 분리하십시오.
- 선반을 부드럽게 들어 올려 필요에 따라 추가 핀치를 만들어 주변 조직에서 선반을 완전히 제거합니다.
- 두 번째 성구 선반을 제거하기 위해 이전 두 단계를 반복합니다.
- 이제 주변 조직에서 풀려나 PBS(그림G)에배치된 성술 선반을 통해 멸균 플라스틱 전구 이송 파이펫을 사용하여 파이펫에 선반을 그리고 약 500μL의 PBS와 함께 1.5mL 마이크로센트심분리기 튜브로 옮긴다. 나머지 쓰레기는 같은 방식으로 처리될 때, 성구 선반이 들어 있는 튜브를 얼음 위에 보관하십시오.
참고: 또는, 선반은 해부 직후 (얼음에 배치하는 대신) 예온 트립신 (0.25 %)을 포함하는 1.5mL 미세 원심 분리기 튜브에 배치 할 수 있습니다. 샘플은 더 신선하지만 샘플을 집합적으로 치료하는 것과는 달리 각 개별 샘플에 대한 모든 진행 단계에 주의를 기울여야 합니다.
3. MEPM 세포의 배양
참고: 여기에 설명된 조건하에서, 미각 상피 세포는 순수한 입맛 중간엽 세포 배양의 결과로 첫번째 통로에서 살아남지 않습니다. 멸균 기술을 사용하여 조직 배양 후드의 모든 단계를 수행하십시오.
- 1.5mL 튜브에서 PBS를 흡인하고 폐기하여 그 과정에서 선반을 버리지 않도록 주의하십시오. 즉시 전천(37°C) 트립신(0.25%)의 200 μL을 각 튜브에 추가합니다. 트립시니화를 가속화하기 위해 1000 μL 파이펫 팁을 사용하여 트립신에서 선반을 위아래로 간단히 피펫합니다.
- 37°C에서 5분 동안 튜브를 배양한 다음 각 샘플을 다시 위아래로 파이프하여 조직을 분해합니다. 37°C에서 또 다른 5분 동안 튜브를 배양하고, 조직의 해리를 완료하기 위해 다시 한 번 위아래로 파이프를 배양한다.
참고: 선반은 완전히 또는 거의 완전히 해리되고 남아있는 조직의 눈에 보이는 덩어리없이 트립신에 중단해야합니다. - 각 1.5mL 튜브에 MEPM 배양 배지(단계 2.1)의 800 μL을 추가합니다. 원심 분리기 는 200 × g에서 1.5 mL 튜브를 5 분 동안 세포에 펠릿합니다. 상체를 제거하고, MEPM 배양 배지의 1mL에서 세포 펠릿을 재놓는다.
- MEPM 세포를 MEPM 배양 배지를 포함하는 6웰 조직 배양 처리 플레이트로 플레이트. 세포가 5% CO2를가진 인큐베이터에서 37°C에서 12시간 동안 플라스틱 표면에 부착하도록 한다.
참고 : 하룻밤 잠복 후, 세포의 대다수 (~90%)가 부착됩니다. 이 시점에서 부착 된 세포는 삼각형 또는 약간 길쭉한 모양으로 상당히 균일하게 보입니다. - 매일 배지를 부드럽게 흡입하여 배지를 변경하고 칼슘이나 마그네슘 없이 1mL의 따뜻한 멸균 PBS로 즉시 교체하십시오. PBS를 흡인하고, 미리 따뜻해진 MEPM 배양 배지의 3mL로 대체한다.
- 일단 100% 컨서물이 되면 세포를 통과합니다.
참고: MEPM 세포는 거의 매일 숫자를 두 배로 늘려야 합니다.- 세포를 통과하려면, 기존 배지를 부드럽게 흡인하고, 칼슘이나 마그네슘없이 따뜻한 PBS로 즉시 교체하십시오 . PBS를 흡인하고, 0.25% 트립신의 0.5mL로 대체한다.
- ~5분 동안 37°C에서 배양하거나, 셀이 접시 표면에서 분리될 때까지 손으로 부드럽게 흔들리면 배양합니다. 세포가 분리되면 트립시화 된 세포에 미리 따뜻해지는 MEPM 배양 배지 5 mL을 즉시 추가하십시오.
- 10mL 세로지학적 파이프를 사용하여 15mL 원판 튜브에서 세포를 부드럽게 수집하고, 200 × g에서 튜브를 원심분리하여 5분간 젤렛세포를 펠릿합니다. 트립신 및 배지를 흡인시키고, 미리 따뜻진 MEPM 배양 배지의 3mL에서 세포를 재보선한다. 셀 1mL을 6웰 접시의 단일 웰에 부드럽게 파이프하고, MEPM 배양 배지의 2mL를 추가하여 총 부피를 3mL로 가져옵니다.
참고: 이것은 세포의 1:3 분할을 구성합니다. MEPM은 최대 3회까지 통과될 수 있다. MEPM의 파종 밀도는 다소 유연하며, 존재하는 세포의 수는 배양 용기에 따라 다릅니다. 그러나 MEPM은 너무 드물게 분할할 때 제대로 확산되지 않으며, 일단 부착되면 새로운 요리에 20-25% 이상 부유해야 합니다.
4. MEPM 세포의 냉동 보존
- 일단 트립시화 MEPM 세포는 펠릿화되면, MEPM 배양 배지에서 세포를 다시 중단하여 ~ 1 × 106 세포/mL의 농도를 얻습니다. 세포를 극저온으로 파이프하고, 세포 육수에 5% 디메틸설프리산화물의 최종 농도를 추가한다. 극저온을 캡, 잠시 반전하여 혼합하고, 즉시 1 °C / 분의 속도로 냉각 동결 용기에 바이알을 배치합니다.
- 하룻밤 동안 -80 °C 냉동고에 쿨러를 놓습니다. 다음 날, 장기간 보관을 위해 극저온을 액체 질소 탱크로 이동합니다.
- 해빙 저온 보존 MEPM 세포
- 액체 질소 탱크에서 극저온을 제거하고 내용이 액체가 되기 시작할 때까지 실온에서 해동하십시오. 미리 워진 MEPM 배양 배지의 9mL를 포함하는 15mL 원추형 튜브에 내용을 비웁니다.
- 200 × g에서 튜브를 원심분리하여 세포를 펠릿합니다. MEPM 배양 배지의 1mL에서 세포를 다시 중단하고, 세포를 6웰 플레이트의 단일 웰로 배관한다. 따뜻한 MEPM 배양 매체 2mL를 추가하여 총 볼륨을 3mL로 가져옵니다.
- 5% CO2를가진 인큐베이터에서 37°C에서 세포를 배양한다. 매일 매체를 변경합니다.
5. MEPM 세포의 라이브 이미징 - 2D 집단 이주 분석서(그림 2)
- 라이브 이미징에 사용할 접시를 준비합니다.
- 작은 수술 가위 또는 날카로운 메스를 사용하여 멸균 2 웰 실리콘 인서트를 ~ 1mm 높이로 줄입니다. 사용 중인 각 샘플에 대한 인서트를 준비합니다.
- 집게를 사용하여 6웰 플레이트의 우물 중앙에 단축된 2웰 실리콘 인서트를 배치합니다. 모든 가장자리를 따라 아래로 눌러 완전히 부착되었는지 확인합니다.
- 이 프로토콜의 섹션 4.3의 단계를 수행하여 저온 보존 된 세포를 해동합니다. MEPM 셀을 계산하고, 잘 당 40-50 μL MEPM 배양 배지의 총 부피로 단축 된 실리콘 인서트의 종자 300 세포 / mm2. 5% CO2를가진 인큐베이터에서 37°C에서 하룻밤 사이에 세포를 배양한다.
- 다음 날, 라이브 타임 랩스 이미징을 준비합니다.
- 단계 별 인큐베이터 및 자동 이미징 기능을 갖춘 위상 대비 현미경을 사용하십시오. 배양 배지의 증발을 줄이기 위해 무대 위 인큐베이터 저장소에 물을 추가; 온도를 37°C및 CO2 ~5%로 설정합니다. 무대 인큐베이터에 6웰 접시를 넣기 전에 습도가 쌓이려면 ~30분 간 허용하십시오.
- 시간 경과 이미징에 대해 다음과 동등한 설정을 사용합니다.
- 4x 목표를 선택하여 넓은 시야 및 위상 대비 필터를 갖습니다.
참고: 자동 초점, 자동 찾기 샘플, z 스택 및 자동 조명은 일반적으로 필요하지 않습니다. - 단축된 실리콘 인서트의 루멘을 캡처하기 위해 우물 당 두 개의 미세한 필드를 선택합니다. 모든 이미징 위치가 올바른 초점을 맞출 수 있는지 확인합니다.
참고: 이미징 중에 이미지 평면을 조정할 수 있지만 이러한 조정은 일반적으로 필요하지 않습니다. - 원하는 이미지 출력 파일 유형을 선택한 다음 원하는 대로 자동 비디오 생성 및 워터마크와 같은 이미징 후 옵션을 선택하거나 선택 해제합니다. 해당하는 경우 단계 대비를 이미징 모드로 선택합니다.
참고: 워터마크를 사용하면 후속 이미지 처리 단계가 방해가 될 수 있습니다. - 레코딩 기간을 72h로 설정합니다. 프로그램을 설정하여 10분마다 이미지를 캡처합니다.
참고: 일반적으로 이미징의 48 h만 필요하지만 이미 촬영 된 이미지를 잃지 않고 72 h 마크 전에 언제든지 이미징을 중지 할 수 있습니다. - 환경 챔버가 5.3.1에서 필요에 따라 작동하는지 확인하십시오. 이러한설정(루틴)을저장하고 이미징을 시작합니다.
- 4x 목표를 선택하여 넓은 시야 및 위상 대비 필터를 갖습니다.
- 72h(또는 지정된 시간)까지 이미징을 계속합니다.
6. 상처 수리 분석에서 MEPM 세포의 라이브 이미징(그림 3)
- 라이브 이미징에 사용할 플레이트를 준비합니다. 집게를 사용하여 멸균 2웰 실리콘 인서트를 6웰 플레이트 의 중앙에 놓고 모든 가장자리를 따라 눌러 완전히 부착되도록 합니다. 사용 중인 모든 샘플에 대해 2웰 인서트를 하나씩 준비합니다.
- 이 프로토콜의 섹션 4.3의 단계를 수행하여 저온 보존 된 세포를 해동합니다. MEPM 세포를 계산하고 필요한 경우 세포를 적어도 350 세포/μL에 농축합니다.
- 종자 1400 셀/mm2 실리콘 인서트에 100 μL의 부피당 MEPM 배양 배지. 5% CO2를가진 인큐베이터에서 37°C에서 48h의 세포를 배양하고, 매일 배지를 변경한다.
- 48 h 후 섹션 5.3 및 5.4에 설명 된 라이브 타임 랩스 이미징에 대한 현미경을 준비합니다. 무대 인큐베이터에 세포를 넣기 직전에 미리 따뜻해지는 MEPM 배양 배지 3mL을 우물(삽입 외부)에 추가한 다음 실리콘 인서트를 조심스럽게 제거합니다.
참고: 2개의 챔버를 분리하는 벽은 "상처"인 간격을 남깁니다. - 5.4에 설명된 대로 시간 경과 이미징을 시작하십시오.
- 더 높은 배율(예: 10배) 목표를 사용합니다. 상처 닫기를 캡처하려면 각 상처에 따라 5개의 시야 필드를 선택하여 상처가 이미지의 수직 축과 평행하도록 합니다.
- 72 h 후에 또는 상처가 완전히 닫혔을 때 화상 진찰을 중단하십시오.
7. 시간 경과 이미지 시퀀스의 전산 분석
참고: 파이썬 인터프리터, C 컴파일러 및 셸과 같은 표준 계산 도구가 장착된 컴퓨터에서 다음 절차를 수행합니다(재료 표참조).
- 통합 분석
참고: 이 절차는 희소한 배양 내의 세포 증식을 추정하거나 상처 폐쇄 실험을 정량화하는 데 사용할 수 있습니다. 셀이 차지하는 영역을 감지하려면 이미지 밝기의 로컬 표준 편차에 세분화 임계값이 적용됩니다. 코드는 이전에 Wu 외33 및 Neufeld 외34에 의해 설명되었으며 http://github.com/aczirok/cellconfluency 사용할 수 있습니다.- 이미지의 분할 임계값을 결정합니다. 예를 들어 임계값 4가 있는 세분화를 보려면 명령을 발행합니다.
segment.py -i 인아웃 이미지.jpg -S 4 -테스트 출력.jpg
그런 다음출력(출력.jpg)을확인합니다.
참고: 임계값이 너무 낮으면 현미경 검사의 배경 영역이 셀 로 구분됩니다. 대조적으로, 임계값이 너무 높으면 셀 커버 영역은 그렇게 분류되지 않습니다. 최적 임계값은 두 오류를 최소한으로 유지합니다. - 제공된 area.sh 스크립트를 사용하여 이미지 시퀀스에 대한 인플루엔자 값을 계산합니다.
area.sh -S 4 img_001.jpg img_002.jpg .... > 합류.dat
참고: 차별 임계값 4는 1단계에서 확인되고 결과는 파일 컨플루엔트에 저장됩니다.dat. 이미지를 적절한 폴더로 정렬하면 이미지 파일 이름 목록을 와일드카드 표기함으로 대체할 수 있습니다.
area.sh -S4 *.jpg > 합류.dat - 상처 폐쇄 실험의 경우, 시간-상처 에지 전파 속도 V의 함수로서 백분율로 표현된 세포 커버 영역의 크기인 인플루엔자 데이터 A(t)를변환합니다.
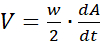
여기서 w는 필드의 폭을 나타내고 dA/dt는 A(t), 즉 셀 커버 영역의 팽창 속도의 시간 유도체이다.
- 이미지의 분할 임계값을 결정합니다. 예를 들어 임계값 4가 있는 세분화를 보려면 명령을 발행합니다.
- 세포 운동성 지도
- 세포 운동성을 특성화하기 위해 PIV(입자 심상 속도 측정)를 실행하고, 이미지 쌍 사이의 로컬 이동 "광학 흐름"의 정도를 추출하지만 개별 세포를 식별하지는 않습니다.
참고: 여기서, 50 μm의 초기 창 크기가 사용되며, Zamir 외35 및 Czirok 외36에의해 자세히 설명된 바와 같이 초기 창 크기는 50 μm의 초기 창 크기입니다. PIV 해석은 각 이미지 프레임 t 및 위치(이미지 내) x에 대해 속도 필드 v(x,t)를 생성합니다. - 7.1항에서 결정된 바와 같이 v(x,t)에서 세포 운동성의 평균 속도를 세포 점유 영역을 통해 계산된 공간 평균으로 추출한다.
- 세포 운동성을 특성화하기 위해 PIV(입자 심상 속도 측정)를 실행하고, 이미지 쌍 사이의 로컬 이동 "광학 흐름"의 정도를 추출하지만 개별 세포를 식별하지는 않습니다.
- 수동 셀 추적
참고: PIV 분석은 세포 운동성에 대한 자동 평가를 제공하지만 개별 셀의 동작에 집중하려면 수동 추적이 필요한 경우가 많습니다. 여러 도구가 이 기능을 제공하지만 초기 배치 후 수동으로 배치된 마커를 수정할 수 있고 추적을 시간에 따라 앞뒤로 수행할 수 있는 경우 매우 유용합니다.- 궤적 세그먼트 삭제와 같은 기본 편집기 기능을 제공하는 사용자 지정 개발 파이썬 도구(http://github.com/donnagreta/cm_track)로 셀 추적을 수행합니다.
참고: 이 수동 추적 도구는 텍스트 파일에서 시간 내에 셀 I의 위치 P(i,t)를 생성하고
cm_track.py -i 이미지/ -o 트랙.dat
시간 경과 이미지가 폴더 이미지/이고 위치 데이터가 파일 트랙에서 수집되는경우.dat(그림 4).
- 궤적 세그먼트 삭제와 같은 기본 편집기 기능을 제공하는 사용자 지정 개발 파이썬 도구(http://github.com/donnagreta/cm_track)로 셀 추적을 수행합니다.
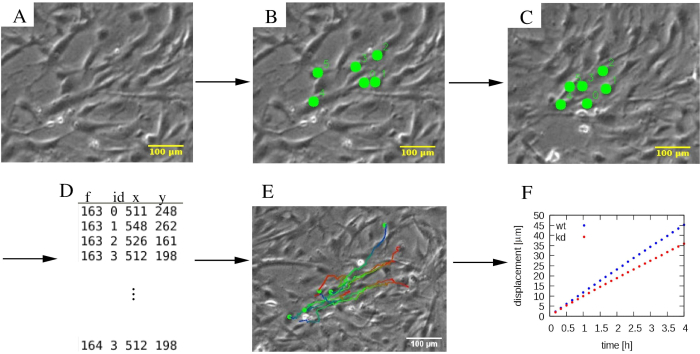
도 4: 개별 세포 궤적의 분석. (A)위상 대비 시간 경과 현미경 사진은(B, C)세포 (녹색 점)를 표시하는 수동 추적 절차를 실시한다. (D)셀 위치(x,y)는 각 세포에 대해 ID로 구별되며 각 프레임 f.(E)궤적에 대해 적혈구에 겹쳐서 시간정보를 나타내기 위해 색으로 구분될 수 있다. 예를 들어, 각 궤적에서 파란색에서 빨간색색 팔레트는 점진적으로 나중에 궤적 세그먼트를 나타내며 빨간색과 파란색은 각각 초기 및 최종 셀 위치를 표시합니다. (F)평균 제곱 변위와 같은 궤적의 다양한 통계적 특성은, 발췌및 다양한 세포 집단의 운동성을 특성화하기 위해 사용될 수 있으며, 이 예에서는 야생형(wt, blue), 및 녹다운(kd, red) MEPM 세포를 포함한다. 스케일 바는 100 μm을 나타냅니다.
- 두 번째 도구를 사용하여 이미지에 대한 오버레이 궤적을 호출하여
visdat.py -d 트랙.dat -i 이미지/ -o 오버레이/ -l999 -r3 -C2 - 폴더 오버레이에 오버레이된 궤적을 통해 이미지를 수집합니다.
참고: 이 예제에서는 셀 위치 데이터가 파일 트랙에 저장되며.dat시간 경과 이미지 시퀀스는 폴더 이미지 내에 있습니다. 나머지 매개변수는 그려진 궤적(-l), 심볼(-r)의 크기 및 사용된 색 구성표(-C)의 최대 길이를 제어합니다.
- 개별 세포 궤적 의 분석
- 총 경로 길이에 의한 궤적을 특성화
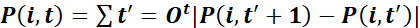 ,
,
상처쪽으로 그물 변위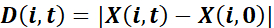 ,
,
여기서 X는 상처에 수직방향으로 P의 투영을 나타낸다: 상처가 y축과 평행할 때 위치의 x 좌표. 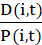 각 셀 i 및 시간 점 t37에대한 지침 효율을계산합니다. 이러한 단일 세포 측정의 인구 평균에 의해 배양을 특성화하고, 적절한 시점t에서평가한다.
각 셀 i 및 시간 점 t37에대한 지침 효율을계산합니다. 이러한 단일 세포 측정의 인구 평균에 의해 배양을 특성화하고, 적절한 시점t에서평가한다.
- 총 경로 길이에 의한 궤적을 특성화
- 세포의 집단 스트리밍 모션
- 이전에 설명된 바와 같이, 움직이는 세포의 부근에 있는 다른 세포의 평균 속도에 의해 세포 운동의 국소 공간 상관관계를 특징짓는다38,39.
참고: 계산 코드는 https://github.com/aczirok/flowfield 사용할 수 있습니다.- 참조 시스템을 앞, 후면, 왼쪽 및 오른쪽에 각 벡터 v(x,t)(그림 5A)에정렬합니다. 참조 시스템의 적절한 공간 셀에 각각의 주변 셀 또는 PIV 속도 벡터를 할당한다(도5B). 각 벡터가 참조 시스템(도5C,D)의원점이 될 때 프로시저를 반복하여 지정된 속도 벡터가 여러 저장소에 할당되도록 합니다.
참고: 데이터 포인트가 벡터 앞과 다른 데이터 점의 왼쪽에 있을 수 있습니다. - 참조 시스템을 공통 방향으로회전(도 5C,F)로회전하여 풀(그림5G).
참고: 풀로 된 속도 데이터의 각 빈의 평균은 공간상관관계를 나타내는 속도 벡터(U)이다: 평균은 공유 속도 성분(도5H)의척도이다. - U(x) 흐름 필드를 기하급수함수로
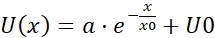 맞춥니다. 세 가지 피팅 매개변수 중에서 x0, 상관 관계길이(도 5I,J)에초점을 맞추어 국소 속도 상관관계가 사라지는 특징적인 거리이다.
맞춥니다. 세 가지 피팅 매개변수 중에서 x0, 상관 관계길이(도 5I,J)에초점을 맞추어 국소 속도 상관관계가 사라지는 특징적인 거리이다.
- 참조 시스템을 앞, 후면, 왼쪽 및 오른쪽에 각 벡터 v(x,t)(그림 5A)에정렬합니다. 참조 시스템의 적절한 공간 셀에 각각의 주변 셀 또는 PIV 속도 벡터를 할당한다(도5B). 각 벡터가 참조 시스템(도5C,D)의원점이 될 때 프로시저를 반복하여 지정된 속도 벡터가 여러 저장소에 할당되도록 합니다.
- 이전에 설명된 바와 같이, 움직이는 세포의 부근에 있는 다른 세포의 평균 속도에 의해 세포 운동의 국소 공간 상관관계를 특징짓는다38,39.
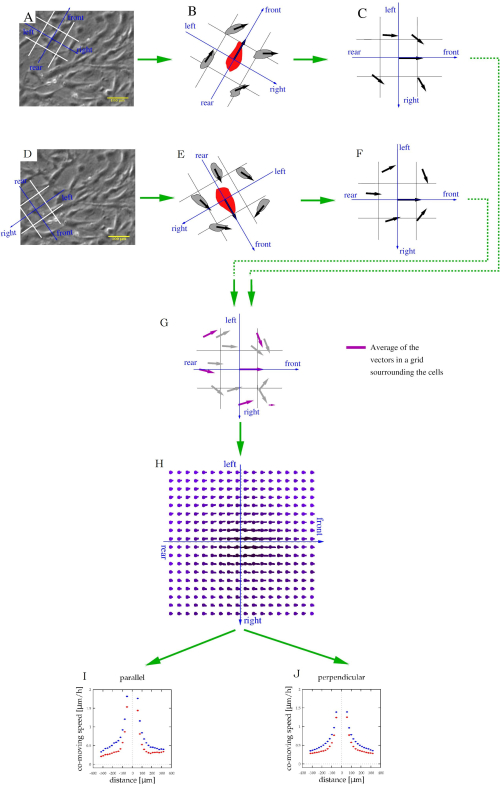
그림 5: 배양 된 세포의 스트림 형성의 특성화. (A,D) 도 4A에서 위상 대비 시간 경과 이미지는 세포 움직임을 식별하는 데 사용됩니다. 각 움직이는 셀에 대해, 참조(파란색) 및 공간 쓰레기통(white)의 프레임은 인접한 세포를 전면, 후면, 왼쪽 또는 오른쪽에 있는 것으로 분류하기 위해 공동 정렬되었다. (B,E) 인접 세포(black vectors)의 속도는 동일한기준(C,F)과관련이 있었다. 이 절차는 각 셀 및 시간 점에 대해 반복되었습니다. (G)이러한 국소 정보를 풀링한 후, 각 쓰레기통에는 평균 적인 모틸세포에 비해 다양한 위치에서 평균 동시 이동 속도(마젠타 화살표)를 결정할 수 있는 다중 속도 벡터(gray)가 포함됩니다. (H)평균 속도 맵은 이동 셀에 비해 다양한 위치에서 일반적인 세포 속도를 특성화합니다. (I,J) 마지막으로, 이 필드는 전방(병렬) 축을 따라, 또한 좌우(수직) 축을 따라 샘플링되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
성구 선반의 해부는 그림 1에설명되어 있습니다. 절개 순서는 조직의 미끄러짐을 최소화하도록 설계되었습니다. 머리제거(도 1A,B)에따라 아래턱이 제거됩니다(도1B,C). 머리의상부(도 1C,D)의절개는 거꾸로 배치할 때 조직을 안정화하기 위해 수행된다(도1E...
토론
성구 선반 고도는 수직에서 수평 리모델링 이벤트1,3,4,9,11을구성한다. 이 리모델링 과정은 성구 선반 중간엽 세포가 정성있게 행동하도록 요구한다고 가정합니다. 야생형 MEPM 세포를 가진 분석은 이 세포 행동이 본질적이고21의양수일 수 있다는 것을 보여?...
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 프로젝트는 국립 보건원 보조금 DE026172 (I.S.), GM102801 (A.C.)에 의해 부분적으로 지원되었다. I.S.는 또한 생물 의학 연구 우수성 센터 (COBRE) 보조금 (국립 일반 의학 연구소 P20 GM104936), 캔자스 IDeA 생체 의학 연구 우수 보조금 (국립 일반 의료 과학 P20 GM103418), 캔자스 지적 발달 장애 연구 센터 (KIDDRC) 보조금 (KIDDRC) 보조금 (KidDRC) 및 국립 보건 연구소 (키드 르 크)에 의해 부분적으로 지원되었다 HD090216).
자료
| Name | Company | Catalog Number | Comments |
| Beaker, 250 mL (x2) | Fisher Scientific | FB-100-250 | |
| CO2 | Matheson Gas | UN1013 | |
| Conical tubes, 15 mL (x1) | Midwest Scientific | C15B | |
| Debian operating system | computational analysis of time-lapse images | ||
| Dulbecco's Modified Eagles Medium/High Glucose with 4 mM L-Glutamine and Sodium Pyruvate | Cytiva Life Sciences | SH30243.01 | |
| EtOH, 100% | Decon Laboratories | 2701 | |
| EVOS FL Auto | ThermoFisher Scientific | AMAFD1000 | |
| EVOS Onstage Incubator | ThermoFisher Scientific | AMC1000 | |
| EVOS Onstage Vessel Holder, Multi-Well Plates | ThermoFisher Scientific | AMEPVH028 | |
| Fetal Bovine Serum | Corning | 35-010-CV | |
| Fine point #5 Stainless Steel Forceps (x2) | Fine Science Tools | 11295-10 | Dissection |
| Instrument sterilizer bead bath | Fine Science Tools | 18000-45 | |
| Microcetrifuge tubes, 1.5mL | Avant | 2925 | |
| Micro-Dissecting Stainless Steel Scissors, Straight | Roboz | RS-5910 | Dissection |
| NucBlue (Hoechst) Live Ready Probes | ThermoFisher Scientific | R37605 | |
| Penicillin Streptomycin Solution, 100x | Corning | 30-002-CI | |
| Silicone Insert, 2-well | Ibidi | 80209 | |
| Small Perforated Stainless Steel Spoon | Fine Science Tools | MC17C | Dissection |
| Spring Scissors, 4 mm | Fine Science Tools | 15018-10 | |
| Sterile 10 cm dishe(s) | Corning | 430293 | |
| Sterile 12-well plate(s) | PR1MA | 667512 | |
| Sterile 6-well plate(s) | Thermo Fisher Scientific | 140675 | |
| Sterile PBS | Corning | 21-031-CV | |
| Sterile plastic bulb transfer pipette | ThermoFisher Scientific | 202-1S | |
| Trypsin, 0.25% | ThermoFisher Scientific | 25200056 |
참고문헌
- Bush, J. O., Jiang, R. Palatogenesis: morphogenetic and molecular mechanisms of secondary palate development. Development. 139 (2), 231-243 (2012).
- Mossey, P. A., Little, J., Munger, R. G., Dixon, M. J., Shaw, W. C. Cleft lip and palate. Lancet. 374 (9703), 1773-1785 (2009).
- Lan, Y., Xu, J., Jiang, R. Cellular and molecular mechanisms of palatogenesis. Current Topics in Developmental Biology. 115, 59-84 (2015).
- Li, C., Lan, Y., Jiang, R. Molecular and cellular mechanisms of palate development. Journal of Dental Research. 96 (11), 1184-1191 (2017).
- Gritli-Linde, A. The etiopathogenesis of cleft lip and cleft palate: usefulness and caveats of mouse models. Current Topics in Developmental Biology. 84, 37 (2008).
- Meng, L., Bian, Z., Torensma, R., Vonden Hoff, J. W. Biological mechanisms in palatogenesis and cleft palate. Journal of Dental Research. 88 (1), 22-33 (2009).
- Dixon, M. J., Marazita, M. L., Beaty, T. H., Murray, J. C. Cleft lip and palate: understanding genetic and environmental influences. Nature Reviews Genetics. 12 (3), 167-178 (2011).
- Kousa, Y. A., Schutte, B. C. Toward an orofacial gene regulatory network. Developmental Dynamics. 245 (3), 220-232 (2016).
- Jin, J. Z., et al. Mesenchymal cell remodeling during mouse secondary palate reorientation. Developmental Dynamics. 239 (7), 2110-2117 (2010).
- Yu, K., Ornitz, D. M. Histomorphological study of palatal shelf elevation during murine secondary palate formation. Developmental Dynamics. 240 (7), 1737-1744 (2011).
- Chiquet, M., Blumer, S., Angelini, M., Mitsiadis, T. A., Katsaros, C. Mesenchymal remodeling during palatal shelf elevation revealed by extracellular matrix and F-actin expression patterns. Frontiers in Physiology. 7, 392 (2016).
- Paul, B. J., et al. ARHGAP29 mutation is associated with abnormal oral epithelial adhesions. Journal of Dental Research. 96 (11), 1298-1305 (2017).
- Hall, E. G., et al. SPECC1L regulates palate development downstream of IRF6. Human Molecular Genetics. 29 (5), 845-858 (2020).
- Walker, B. E., Fraser, F. C. Closure of the secondary palate in three strains of mice. Journal of Embryology and Experimental Morphology. 4 (2), 176-189 (1956).
- Jin, J. Z., Li, Q., Higashi, Y., Darling, D. S., Ding, J. Analysis of Zfhx1a mutant mice reveals palatal shelf contact-independent medial edge epithelial differentiation during palate fusion. Cell Tissue Research. 333 (1), 29-38 (2008).
- Kouskoura, T., et al. The etiology of cleft palate formation in BMP7-deficient mice. PLoS One. 8 (3), 59463 (2013).
- Lan, Y., Zhang, N., Liu, H., Xu, J., Jiang, R. Golgb1 regulates protein glycosylation and is crucial for mammalian palate development. Development. 143 (13), 2344-2355 (2016).
- He, F., et al. Wnt5a regulates directional cell migration and cell proliferation via Ror2-mediated noncanonical pathway in mammalian palate development. Development. 135 (23), 3871-3879 (2008).
- Lan, Y., Qin, C., Jiang, R. Requirement of hyaluronan synthase-2 in craniofacial and palate development. Journal of Dental Research. 98 (12), 1367-1375 (2019).
- Yonemitsu, M. A., Lin, T. Y., Yu, K. Hyaluronic acid is required for palatal shelf movement and its interaction with the tongue during palatal shelf elevation. Developmental Biology. 457 (1), 57-68 (2020).
- Goering, J. P., et al. SPECC1L-deficient palate mesenchyme cells show speed and directionality defect. Scientific Reports. 11 (1), 1452 (2021).
- Fantauzzo, K. A., Soriano, P. PI3K-mediated PDGFRalpha signaling regulates survival and proliferation in skeletal development through p53-dependent intracellular pathways. Genes and Development. 28 (9), 1005-1017 (2014).
- Vasudevan, H. N., Soriano, P. SRF regulates craniofacial development through selective recruitment of MRTF cofactors by PDGF signaling. Developmental Cell. 31 (3), 332-344 (2014).
- Vasudevan, H. N., Mazot, P., He, F., Soriano, P. Receptor tyrosine kinases modulate distinct transcriptional programs by differential usage of intracellular pathways. Elife. 4, 07186 (2015).
- Gao, L., et al. 2,3,7,8-Tetrachlorodibenzo-p-dioxin and TGFbeta3-mediated mouse embryonic palatal mesenchymal cells. Dose Response. 17 (1), 1559325818786822 (2019).
- Iyyanar, P. P. R., Nazarali, A. J. Hoxa2 inhibits bone morphogenetic protein signaling during osteogenic differentiation of the palatal mesenchyme. Frontiers in Physiology. 8, 929 (2017).
- Jiang, Z., Pan, L., Chen, X., Chen, Z., Xu, D. Wnt6 influences the viability of mouse embryonic palatal mesenchymal cells via the beta-catenin pathway. Experimental and Therapeutic Medicine. 14 (6), 5339-5344 (2017).
- Liu, X., et al. Negative interplay of retinoic acid and TGF-beta signaling mediated by TG-interacting factor to modulate mouse embryonic palate mesenchymal-cell proliferation. Birth Defects Research Part B: Developmental and Reproductive Toxicology. 101 (6), 403-409 (2014).
- Bush, J. O., Soriano, P. Ephrin-B1 forward signaling regulates craniofacial morphogenesis by controlling cell proliferation across Eph-ephrin boundaries. Genes & Development. 24 (18), 2068-2080 (2010).
- Mo, J., Long, R., Fantauzzo, K. A. Pdgfra and Pdgfrb genetically interact in the murine neural crest cell lineage to regulate migration and proliferation. Frontiers in Physiology. 11, 588901 (2020).
- He, F., Soriano, P. A critical role for PDGFRalpha signaling in medial nasal process development. PLoS Genetics. 9 (9), 1003851 (2013).
- Fantauzzo, K. A., Soriano, P. Generation of an immortalized mouse embryonic palatal mesenchyme cell line. PLoS One. 12 (6), 0179078 (2017).
- Wu, K., Gauthier, D., Levine, M. D. Live cell image segmentation. IEEE Transactions on Biomedical Engineering. 42 (1), 1-12 (1995).
- Neufeld, Z., et al. The role of Allee effect in modelling post resection recurrence of glioblastoma. PLoS Computational Biology. 13 (11), 1005818 (2017).
- Zamir, E. A., Czirok, A., Rongish, B. J., Little, C. D. A digital image-based method for computational tissue fate mapping during early avian morphogenesis. Annals of Biomedical Engineering. 33 (6), 854-865 (2005).
- Czirok, A., et al. Optical-flow based non-invasive analysis of cardiomyocyte contractility. Scientific Reports. 7 (1), 10404 (2017).
- Biggs, L. C., et al. Interferon regulatory factor 6 regulates keratinocyte migration. Journal of Cell Science. 127, 2840-2848 (2014).
- Czirok, A., Varga, K., Mehes, E., Szabo, A. Collective cell streams in epithelial monolayers depend on cell adhesion. New Journal of Physics. 15, 75006 (2013).
- Szabo, A., et al. Collective cell motion in endothelial monolayers. Physical Biology. 7 (4), 046007 (2010).
- Gulyas, M., Csiszer, M., Mehes, E., Czirok, A. Software tools for cell culture-related 3D printed structures. PLoS One. 13 (9), 0203203 (2018).
- Soderholm, J., Heald, R. Scratch n' screen for inhibitors of cell migration. Chemistry & Biology. 12 (3), 263-265 (2005).
- Riahi, R., Yang, Y., Zhang, D. D., Wong, P. K. Advances in wound-healing assays for probing collective cell migration. Journal of Laboratory Automation. 17 (1), 59-65 (2012).
- Svensson, C. M., Medyukhina, A., Belyaev, I., Al-Zaben, N., Figge, M. T. Untangling cell tracks: Quantifying cell migration by time lapse image data analysis. Cytometry Part A. 93 (3), 357-370 (2018).
- Fantauzzo, K. A., Soriano, P. PDGFRbeta regulates craniofacial development through homodimers and functional heterodimers with PDGFRalpha. Genes & Development. 30 (21), 2443-2458 (2016).
- Rafi, S. K., et al. Anti-epileptic drug topiramate upregulates TGFβ1 and SOX9 expression in primary embryonic palatal mesenchyme cells: Implications for teratogenicity. PLoS ONE. , (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유