É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Fluorescência dupla In situ Hibridação nas Seções cérebro fresco
Neste Artigo
Resumo
Este protocolo envolve um não-radioativo In-situ Processo de hibridação que permite a identificação simultânea de duas espécies de transcrição, com uma resolução única célula, em seções finas do cérebro dos vertebrados.
Resumo
Aqui nós descrevemos uma versão modificada de uma dupla de fluorescência
Protocolo
Este protocolo foi desenvolvido e aperfeiçoado com base no padrão radioativos e não radioativos métodos de hibridização in-situ, anteriormente desenvolvido por nós e outros para detectar uma ou duas espécies transcrição no tecido cerebral 1-7. O protocolo descrito a seguir tem um comprimento total de 2 ou 3 dias, dependendo do número de interrupções processual escolhida pelo usuário final. Todas as etapas detalhadas abaixo devem ser realizados à temperatura ambiente, com exceção da etapa de hibridização riboprobe e pós-hibridação lavagens. As soluções e buffers exigido para todos os passos envolvidos neste método podem ser encontrados no final do presente protocolo.
1. Preparação do tecido e Seccionamento
- Decapitar e extrair o cérebro do sujeito rapidamente, e colocá-lo em um molde de plástico de tamanho adequado.
- Cobrir o cérebro com Tissue-Tek meio de incorporação e rapidamente coloque o molde de plástico em um banho dry-ice/alcohol para o congelamento rápido. Tecido congelado pode ser armazenado a -80 ° C até o uso.
- Utilizando um criostato, recolher 2 ou 3 seções do cérebro por slide em Superfrost cobrado Além disso slides. Espessura das seções devem ser 10-12 mM. Lâminas podem ser armazenadas a -80 ° C até o uso.
2. Preparação de Sephadex G50 Colunas para Purificação Probe
"Colunas Sephadex podem ser adquiridos de fontes comerciais, no entanto, oferecemos a seguir uma alternativa de baixo custo para a geração de colunas, o que será necessário para a purificação da sonda.
- Quantidade adequada de hidrato de Sephadex G50 em pó com RNase-free, água DEPC-tratados (por exemplo, 2 g de pó em 100 ml de água tratada DEPC-), brevemente solução misturar e armazenar à temperatura ambiente para precipitar o excesso Sephadex G50.
- Remover o sobrenadante (camada superior de água), após precipitação Sephadex G50.
- Repita o processo acima 3-5 vezes.
- Após a lavagem final, re-suspender a Sephadex G50 solução em tampão TE (1:1), e armazenar a 4 ° C até o uso.
- Lugar autoclavado lã de vidro para uma seringa estéril 1 ml e comprimi-lo com o êmbolo para fazer uma camada compacta na parte inferior, e em seguida coloque a seringa em um tubo Falcon 15 ml.
- Misture bem a solução de Sephadex G50 em TE. Encha a seringa / coluna com esta solução.
- Centrifugar a coluna por 30 segundos a 1000 rpm.
- Repita este procedimento até que a coluna é quase completamente preenchida com Sephadex G50 contas.
- Aplicar 200 mL de tampão de lavagem coluna para a coluna e centrifugar por 2 min a 1000 rpm. Descartar por escoamento.
- Aplicar 200 mL de tampão de bloqueio coluna para a coluna e gire-a para 2 min a 1000 rpm. Repita este passo 4-5 vezes para equilibrar coluna. As colunas podem ser armazenados a 4 ° C até o uso se selado com Parafilm.
3. Rotulagem e Purificação de Riboprobes
Abaixo detalhamos a geração e purificação de riboprobe único. Para dFISH, a preparação de cada sonda irá envolver a mesma metodologia, exceto que uma das sondas será marcada com digoxigenina (DIG) marcadas UTP enquanto o outro com biotina marcadas UTP.
- Preparar um concentrado (> 150 ng / mL) e purificado solução linearizada cDNA de interesse para gerar um sentido (controle) ou riboprobes antisense.
- Em um microtubo de 1,5 ml, adicionar 0,5-1 mg de purificada modelo de cDNA, 2 mL de tampão 5X sonda rotulagem, 1 ml de 10X DIG (ou biotina), rotulagem mix, 0,5 mL de RNasin e 1 ml da RNA polimerase adequada , e trazer o volume final da solução para 10 ml com RNAse-free (DEPC-tratados) de água.
- Incubar a solução em um banho de água a 37 ° C por 2 horas.
- Adicionar 1 ml de tRNA (estoque, 20 mcg / mL), e 39 mL de tampão coluna de bloqueio para a solução.
- Prepare o Sephadex G50 coluna para purificação da sonda. Para este fim, adicionar 50 mL tampão de bloqueio para a coluna e gire-a por 10 segundos a 1000 rpm.
Repita este passo 2-3 vezes para equilibrar a coluna (ou seja, o total de 50 mL aplicada à coluna são recuperados após o ciclo de centrifugação). - Aplique a solução sonda para a coluna de Sephadex G50 posição, um tubo de microcentrífuga novo na parte inferior da coluna, e girá-lo por 3 min a 1000 rpm para obter o purificada 50 ml de solução riboprobe.
- Avaliar a qualidade e rendimento de riboprobe rotulados usando um padrão de formaldeído-agarose gel RNA, ou um espectrofotômetro.
4. Pós-fixação Acetilação e Hibridização
- Remove seções do freezer -80 ° C e permitir uma temperatura ambiente para equilibrar. Posteriormente, incubar seções em um frio, solução de paraformaldeído recém-made de 3% para 5 min.
- Brevemente lavar seções em phosphate solução salina tamponada (PBS) por duas vezes.
- Desidratar seções através de uma série de álcool normal (70, 95 e 100%; 2 min cada), e deixe-os secar ao ar.
- Incubar seções em uma solução de acetilação de 10 min.
- Enxágüe seções 3 vezes em 2X SSPE.
- Desidratar seções, mais uma vez através da série de álcool padrão descrito acima, e permitir-lhes-ar seco.
- Prepare um volume adequado de solução de hibridação e adicionar tanto riboprobes para a solução (concentração da sonda: 1 ng / mL para cada sonda). Volume total da solução de hibridização é determinada com base no número de seções para ser hibridizado (16 ml de solução de hibridização por seção).
- Aplicar volume adequado da solução de hibridização para o tecido e lamínula slides garantir que nenhum ar overlay bolhas no tecido.
- Coloque as lâminas em porta-lâminas de metal. Titular mergulhe em um banho de óleo mineral regulado a 65 ° C durante a noite. Garantir que as lamínulas são virados para cima (ou seja, o aspecto lateral do compartimento de slides deve estar em contato com o fundo do recipiente de óleo mineral).
5. Pós-hibridação Lava
- No dia seguinte, remova cuidadosamente o suporte de slides do banho de óleo mineral e enxaguá-lo brevemente em clorofórmio para remover o excesso de óleo do slides.
- Coloque as lâminas em uma solução de SSPE 2X de 5-10 min. Lamínulas devem separar de slides enquanto em solução.
- Transferir as lâminas para uma nova solução de SSPE 2x, e mantê-los por 1 hr em temperatura ambiente.
- Transferência de seções em uma solução contendo 2X SSPE além de formamida 50%. A temperatura desta solução deve coincidir com a temperatura utilizada no processo de hibridização durante a noite. Mantenha slides nesta solução por 1,5 hr.
- Transferência de seções para uma solução de SSPE 0,1 x pré-aquecida à mesma temperatura da hibridação. Incubar seções nesta solução por 30 min. Repita este passo para um adicional de 30 min.
6. Detecção e Visualização de Riboprobes
- Transferência de slides para o buffer TNT em que o peróxido de hidrogênio 0,3% foi adicionado, por 10 min.
- Lave os cortes em TNT buffer, 3 vezes (10 min cada).
- Usando uma caneta DAKO, desenhe um bem ao redor da área que contém as seções do cérebro. Importante, devem ser tomadas precauções para garantir que as seções não seca.
- Aplicar 150 mL de tampão TNB a cada slide, e incubar seções nesta solução por 30 min, em câmara úmida.
- Remoção da solução TNB excesso de slides de inclinação.
- Aplicar 150 mL de solução contendo TNB conjugado com peroxidase de anticorpos anti-DIG e slides armazenar em câmara úmida por 2 horas. Concentração de anticorpos deve ser determinada individualmente para cada transcrição de interesse antes da realização dFISH.
- Lave os cortes em TNT buffer; 3 vezes, 10 min cada.
- Aplicar 150 mL de Alexa 594-conjugados solução tiramida trabalhando para cada slide, e armazená-los em câmara úmida por 1 hora. Esta solução deve ser preparada de acordo com as instruções do fabricante.
- Lave os cortes em TNT buffer; 3 vezes, 10 min cada.
- Seções incubar em tampão TNT para que o peróxido de hidrogênio 0,3% foi adicionado para 10-30 min.
- Lave os cortes em TNT buffer; 3 vezes, 10 min cada.
- Aplicar 150 mL de tampão TNB por lâmina, e mantê-los em câmara úmida por 30 min.
- Remoção da solução TNB excesso de slides de inclinação.
- Adicionar 150 mL de solução contendo TNB conjugado com peroxidase de anticorpos anti-biotina, e slides armazenar em câmara úmida por 2 horas.
- Lave os cortes em TNT buffer; 3 vezes, 10 min cada.
- Aplicar 150 mL de uma Alexa 488-conjugados solução tiramida trabalhando para os slides e seções incubar por 1 hora. Esta solução deve ser preparada de acordo com as recomendações do fabricante.
- Lave os cortes em TNT buffer; 3 vezes, 10 min cada.
- Adicionar 150 mL de solução de Hoechst (1:1000 em TNT buffer) para as seções e mantê-los na câmara úmida por 2 min.
- Lavar as seções em TNT buffer; 3 vezes, 5 min cada.
- Seções lamela com uma média de fluorescência compatível com a montagem (por exemplo, Vectashield ou prolongar Antifade).
7. Resultados representante
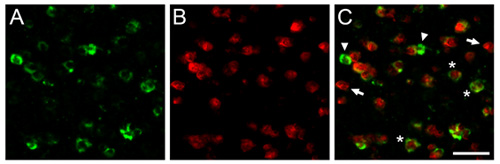
Figura 1. Mostramos aqui os resultados obtidos dFISH representante no cérebro de tentilhões-zebra. Mostrados são fotomicrografias obtidas a partir da nidopallium caudomedial (NCM), o análogo songbird do córtex auditivo de mamíferos. Seções do cérebro foram hibridizados com uma riboprobe biotinilado contra parvalbumina (A), um marcador para uma sub-população de neurônios inibitórios, e um DIG marcado ribosonda dirigida contra o dependente de atividade do gene Zenk (B), um marcador confiável para a canção-driven neurônios. C) Sobreposição de (A) e (B) mostra uma população de neurônios inibitórios que são ativadas pela experiência auditiva. Flechas e setas indicam células marcadas exclusivamente com cada um dos dois riboprobes, e asteriscos mostram neurônios representante co-expressar tanto transcrições de interesse. Barra de escala = 25 mm.
Discussão
Nós temos usado esse protocolo para estudar como o cérebro dos vertebrados é neuroquimicamente e funcionalmente organizado, e para determinar como comportamentalmente relevante impacto estímulos sensoriais as máquinas genômica de neurônios no cérebro adulto 80-10. Temos utilizado com sucesso este método em tecido cerebral de camundongos, ratos e pássaros, mas antecipamos que este protocolo será facilmente adaptável às seções do cérebro obtidas a partir de uma variedade de espécies de vertebra...
Divulgações
Agradecimentos
Trabalho suportado por concessões de NIH / NIDCD ea Fundação Schmitt para RP.
Materiais
Soluções
- Tampão da coluna de lavagem: 10 mM Tris-HCl, 0,15 M NaCl, 0,05 mM EDTA, 50 mcg / mL tRNA, SDS 0,1% em 50 ml de água DEPC-tratada.
- Tampão da coluna de bloqueio: 10 mM Tris-HCl, 50 mM NaCl, 0,1 mM EDTA em 50 ml de água DEPC-tratada.
- TNT buffer: 60 ml de 1 M Tris-HCl, 18 ml de 5 M NaCl e 1,8 ml de Triton X-100 em 600 ml de água tratada DEPC-.
- TNB buffer: 100 mM Tris-HCl, 8,3 mg / mL BSA, 0,15 M NaCl e Triton 3% X-100 em DEPC tratados com água.
- TE buffer: 10 mM Tris-HCl, pH 7,5, acrescido de 1 mM EDTA, pH 7.5.
- Acetilação solução: 2,7 ml de trietanolamina mais 0,5 ml de anidrido acético em 200 ml de água tratada DEPC-.
- Solução de hibridização: formamida 50%, 2X SSPE, 2 mg / mL tRNA, 1 mg / mL BSA e 1 mg / mL poli A em água DEPC-tratada. Ela precisa de 16 l por seção.
Referências
- Jin, L., Lloyd, R. V. In situ hybridization: methods and applications. J Clin Lab Anal. 11, 2-9 (1997).
- Stoler, M. H. In situ hybridization. Clin Lab Med. 10, 215-236 (1990).
- Komminoth, P., Werner, M. Target and signal amplification: approaches to increase the sensitivity of in situ hybridization. Histochem Cell Biol. 108, 325-3233 (1997).
- Kessler, C. The digoxigenin:anti-digoxigenin (DIG) technology--a survey on the concept and realization of a novel bioanalytical indicator system. Mol Cell Probes. 5, 161-205 (1991).
- Panoskaltsis-Mortari, A., Bucy, R. P. In situ hybridization with digoxigenin-labeled RNA probes: facts and artifacts. Biotechniques. 18, 300-307 (1995).
- Qian, X., Lloyd, R. V. Recent developments in signal amplification methods for in situ hybridization. Diagn Mol Pathol. 12, 1-13 (2003).
- Mello, C. V., Jarvis, E. D., Denisenko, N., Rivas, M. Isolation of song-regulated genes in the brain of songbirds. Methods Mol Biol. 85, 205-217 (1997).
- Pinaud, R. GABAergic neurons participate in the brain's response to birdsong auditory stimulation. Eur J Neurosci. 20, 1318-1330 (2004).
- Velho, T. A., Pinaud, R., Rodrigues, P. V., Mello, C. V. Co-induction of activity-dependent genes in songbirds. Eur J Neurosci. 22, 1667-1678 (2005).
- Tremere, L. A., Jeong, J. K., Pinaud, R. Estradiol shapes auditory processing in the adult brain by regulating inhibitory transmission and plasticity-associated gene expression. J Neurosci. 29, 5949-5963 (2009).
- Pinaud, R., Mello, C. V., Velho, T. A., Wynne, R. D., Tremere, L. A. Detection of two mRNA species at single-cell resolution by double-fluorescence in situ hybridization. Nat Protoc. 3, 1370-1379 (2008).
- Pinaud, R., Jeong, J. K. Duplex fluorescence in situ hybridization in the study of gene co-regulation in the vertebrate brain. Methods Mol Biol. 611, 115-129 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados