É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Modificado Sistema Yeast-Two-Hybrid para identificar proteínas Interagindo com o Fator de Crescimento progranulina
Neste Artigo
Resumo
Nós modificamos o fermento convencional de dois híbridos de rastreamento, uma ferramenta eficaz na identificação genética de interação de proteínas. Esta modificação marcadamente encurta o processo, reduz a carga de trabalho, eo mais importante, reduz o número de falsos positivos. Além disso, esta abordagem é reproduzível e confiável.
Resumo
Progranulina (PGRN), também conhecido como precursor granulin epithelin (GEP), é um fator de crescimento 593-amino-ácido autócrina. PGRN é conhecida por desempenhar um papel crítico em uma variedade de processos fisiológicos e de doença, incluindo a embriogênese precoce, uma cicatrização de feridas, inflamação 2, 3 e 4 de defesa do hospedeiro. PGRN também funciona como um fator neurotrófico 5, e as mutações no gene PGRN resultando em perda parcial da demência frontotemporal causa proteína PGRN 6, 7. Nossos estudos recentes têm levado ao isolamento de PGRN como um importante regulador da cartilagem desenvolvimento e degradação 8-11. Embora PGRN, descoberto há quase duas décadas, desempenha um papel crucial em várias condições fisiológicas e patológicas, os esforços para explorar as ações de PGRN e entender os mecanismos envolvidos foram significativamente prejudicada pela nossa incapacidade de identificar a sua ligação ao receptor (s). Para resolver este problema, desenvolvemos um modicados levedura híbrida de dois (MY2H) abordagem baseada no mais comumente usados GAL4 sistema 2-híbrido baseado. Comparado com o convencional levedura tela dois híbridos, MY2H reduz drasticamente o processo de tela e reduz o número de falsos positivos clones. Além disso, esta abordagem é reproduzível e confiável, e temos empregada com sucesso este sistema para isolar as proteínas de ligação de iscas diversas, inclusive de canal iônico, 12 de proteínas da matriz extracelular 10, 13, e fator de crescimento 14. Neste artigo, descrevemos este procedimento MY2H experimental em detalhe usando PGRN como um exemplo que levou à identificação de TNFR2 conhecido como o primeiro receptor PGRN associada 14, 15.
Protocolo
1. Informação de fundo
A levedura sistema de hibridação é uma poderosa técnica genética usado para descobrir interações proteína-proteína 16, 17. Vários tipos de sistemas híbridos de 2, como LexA sistemas baseados, o Sistema de Recrutamento Sos e bactérias ou células de mamíferos baseadas em dois híbridos, são comerciais disponíveis, este trabalho concentra-se especificamente sobre as modificações dos mais comumente usados GAL4 base sistema de levedura 2-híbrido. Resumidamente, o método é baseado nas propriedades da proteína de levedura que consiste GAL4 de domínios separáveis responsável pela ativação do DNA-binding e transcricional. A proteína isca é expressa como uma fusão para o GAL4 domínio de ligação ao DNA (DNA-BD), enquanto as proteínas presas são expressos como fusões para o domínio de ativação GAL4 (AD). Interação entre isca e proteínas de fusão presa leva à transcrição de ativações GAL4-binding sites que contêm genes repórter que estão integrados no g de fermentoenome. O princípio da Y2H é ilustrado na figura. 1 e do procedimento experimental está resumido na figura. 2.
2. Materiais necessários e soluções
- Meio de Crescimento YPD (uma mistura de peptona, extrato de levedura e dextrose em proporções ideais para o cultivo de cepas mais Saccharomyces cerevisiae).
- Bases mínima SD (Minimal sintética definida (SD) bases incluem uma base de nitrogênio levedura, sulfato de amônio, e uma fonte de carbono, dextrose. Dropout (DO) suplementos podem ser adicionados à Base SD mínima para fazer uma média, sintético definido sem o especificado nutrientes).
- Leu /-Trp Dropout (DO) complementar (que contém todos os aminoácidos essenciais, exceto para leucina e triptofano)
- -His/-Leu/-Trp/-Ura Dropout (DO) Suplemento (contendo todos os aminoácidos essenciais, exceto para leucina, triptofano, histidina, e uracil)
- Luria caldo (LB) (Triptona 10 g / L, extrato de levedura 5 g / L, NaCl 5 g / L)
- X-Gal (5-bromo-4-cloro-3-indolil-β-D-galactopiranosídeo) solução: preparado como um de 20 mg / ml de solução em N, N-Dimetilformamida
- TE 10X (100 mM Tris-HCl (pH 7,5), 10 mM EDTA, autoclavado)
- Herring sonicado ou DNA do esperma de salmão, cozido (10 mg / ml)
- LiAc 10X (1 acetato de lítio M, autoclavada)
- 50% PEG-3350, solução esterilizada por filtração,
- 3-Amino-1, 2, 4-triazol (3AT)
- Canamicina
- Ampicilina
- Células ELECTROMAX DH5α
- Z buffer: 16,1 g Na2HPO4 7H2O (ou 8,52 g anidro), 5,5 g NaH2PO4 H2O (ou 4,8 ganhydrous), 0,75 g KCl, 0,246 g MgSO4 7H2O (ou 0,12 g anidro), dissolvido em 1 L de água, autoclavada e destilada ajustada para pH 7.0.
3. Geração de isca (pDBLeu-PGRN)
Um fragmento de cDNA encoding PGRN falta peptídeo sinal (aa21-588) foi clonado direcionalmente para o I-Não Sal I locais do vetor pDBLeu (a ProQuest sistema de dois híbridos, Invitrogen),mantendo o mesmo quadro de leitura da tradução como o domínio de DNA GAL4 Binding para gerar pDBLeu-PGRN.
- Amplificação do fragmento de cDNA de PGRN descrito acima por PCR usando primers projetado para conter sítios de restrição (Sal I no 5'end e não eu ao 3'end) para permitir que no quadro-fusion.
- Gel purificar o produto da PCR, digestão com endonucleases Sal I e Not I.
Prepare o DB vector pDBLeu restrição por digestão dupla com o endonucleases de restrição mesmo. - Ligadura do fragmento PGRN restrição na pDBLeu linearizada vetor e transformar em DH5α com a seleção para LB + canamicina a 25 mcg / ml.
- Verificar a correcção da construção de seqüenciamento de DNA.
4. Transformação de pequena escala de isca plasmídeo
- Inocular 5 ml de YPD com uma colônia de Mav203 (Invitrogen), agitar durante a noite a 30 ° C.
- Diluir a cultura durante a noite em 50 ml de YPD. Crescer um additional 2-4 horas.
- Agregar as células a 3000 rpm por 5 min à temperatura ambiente. Ressuspender o sedimento em 40 ml de água, autoclavada destilada.
- Re-agregar as células. Ressuspender em 2 ml de solução I, incubar a RT 10 min.
Solução I: 0,5 ml 10xLiAc, 0,5 ml 10xTE, 4 ml H 2 O - Dispensar o DNA de tubos: 2-3 mL de pDBLeu-PGRN (0.25μg/μl) e 10 ml de desnaturado DNA do esperma, cortado salmão (10μg/μl). Adicionar 100μl células de levedura, misturar bem.
- Adicionar 700μl de solução II, misture bem. Incubar a 30 ° C por 30 min. Solução II: 0,2 ml 10xLiAc, 0,2 ml 10xTE, 1,6 ml 50% PEG3350.
- Choque térmico a 42 ° C por 15 min.
- Pellet por 2 min. Desprezar o sobrenadante. Ressuspender o sedimento em 200 mL de água, autoclavada destilada.
- Placa de suspensão para SD-Leu placas com diluições em série. Incubar as placas a 30 ° C por 2-3 dias.
5. Validação Bait
Antes de executar simª tela de dois híbridos, teste pDBLeu-PGRN para a auto-ativação e determinar os níveis de expressão basal do gene repórter HIS3. Este teste determina se iscas ativam a transcrição e se a auto-ativação pode ser neutralizada por inibidores. 3-Amino-1, 2, 4-triazol (3-AT) é um inibidor competitivo do produto HIS3 gene e pode ser usado para titular o nível mínimo de HIS3 expressão necessária para o crescimento em histidina deficiente mídia.
- Transformar o pDBLeu-PGRN em cepa de levedura Mav203, placa a transformação para SD-Leu placas e incubar por 48-72 h, a 30 ° C.
- Colônias de patch de cada transformação usando palitos de dente autoclavado para SD-Leu-Seu placas contendo 3-AT em concentrações de 10 mM, 25 mM, 50 mM, 75 mM e 100 mM. 3-AT é um inibidor competitivo da enzima HIS3 e iscas apenas que exibem auto-ativação será capaz de crescer na presença dos 3-AT.
- Cepas isca que crescem em placas containing mM 100 3-AT não são adequados para uso na tela dois híbridos. Para iscas que podem ser usados, use o mais baixo 3-AT concentração que inibe o crescimento celular. Em muitos casos, 25 mM de 3-AT é usado para a seleção.
6. triagem biblioteca de cDNA
O pDBLeu-PGRN plasmídeo é introduzido MaV203 usando uma transformação em pequena escala, como descrito acima. Introduzir pPC86-biblioteca (Invitrogen) em MaV203 (pDBLeu-PGRN), o procedimento descrito abaixo tipicamente dá ~ 4 x 10 4 colônias com 0,5 mg de DNA plasmídeo biblioteca. Portanto, 2,5 x 10 6 transformantes de levedura exigirá ~ 30,0 mg pPC86-cDNA DNA plasmídeo biblioteca, 25 transformações, e cinqüenta placas de 10 cm (SD-Trp-Leu-His-Ura 3 AT).
- Prepare o número apropriado de 10 cm 3 SD-Trp-Leu-His-Ura AT placas (50 para o procedimento abaixo). Também preparar pelo menos quatro de 10 cm SD-Leu-Trp placas para estimar o número de transformantes.
- Transformar a biblioteca emo MaV203 contendo pDBLeu-PGRN de acordo com o procedimento descrito abaixo.
- Suspender várias colônias isoladas de MaV203 (pDBLeu-PGRN) em ~ 100μl de água, destilada autoclavada e espalhá-los em uma placa de 10 cm SD-Leu. Repita o procedimento para uma placa SD-Leu segundo. Incubar ambas as placas por 18-24 horas a 30 ° C.
- Raspe e completamente suspender as células em 10 ml de água, autoclavada destilada. Adicionar um volume suficiente de suspensão celular a 500 ml de meio YPD líquido num balão de dar uma OD de 600 ~ 0,1.
- Verifique se o OD é de aproximadamente 0,1 após a inoculação.
- Agite a 30 ° C até o OD atinge 600 ~ 0.4.
- Prepare frescos:
- 110 ml 1X TE / LiAc através da combinação de 11 ml TE 10X, 11 ml LiAc 10X, e 88 ml de água autoclavada.
- 16 ml PEG / LiAc combinando 1,6 ml TE 10X, 1,6 ml LiAc 10X, e 12,8 ml 50% PEG-3350.
- Agregar as células em 3000g por 5 min à temperatura ambiente.
- Descartar tele sobrenadantes e delicadamente ressuspender o sedimento por pipetagem cima e para baixo em 100 ml de água, autoclavada destilada à temperatura ambiente.
- Centrifugar a 3.000 g por 5 min à temperatura ambiente.
- Desprezar o sobrenadante das células centrifugadas e ressuspender o pellet celular em 50 ml 1X solução TE / LiAc.
- Centrifugar a 3.000 g por 5 min à temperatura ambiente.
- Remover o sobrenadante e ressuspender o sedimento em um volume final de 2,5 ml de 1X solução TE / LiAc.
- Realizar 25 transformações. Combine 2,5 células ml, 125 mL (10μg/μl) desnaturados, DNA de esperma de salmão cortado, e 30 mg biblioteca de cDNA. Misture suavemente pipetando cima e para baixo. Adicionar 15 ml de solução de PEG / LiAc e misture delicadamente. Alíquota em 25 autoclavado tubos de 1,5 ml de microcentrífuga de 700 mL cada.
- Incubar por 30 min em banho-maria a 30 ° C.
- Choque térmico durante 15 min em banho-maria a 42 ° C.
- Centrifugar a 6.000 g por 1 min em temperatura ambiente. Remova cuidadosamente o supersobrenadante. Delicadamente ressuspender cada pellet em 400 mL de água, destilada autoclavada por pipetagem cima e para baixo.
- Placa de 200 mL da mistura de transformação em cada transformação única de 10 cm de SD-Trp-Leu-His-Ura 3 AT (3AT concentração: 25 mM) placas usando uma barra de spread, de modo 25 autoclavado 1,5 ml tubos de microcentrífuga pode fazer 50 placas (SD-Trp-Leu-His-Ura 3 AT).
- Incubar as placas para os dias 10/05 a 30 ° C.
- Para estimar a eficiência de transformação da reação, diluições em série de uma reação (1:40; 1:400 e 1:4000) são semeadas em SD-Leu-Trp prato. Após incubação por 3 dias a 30 ° C, o número de colônias é contado eo número total de transformantes calculado.
7. X-gal ensaio
- Transferência de colônias para placa de YPD; incubar a 30 ° C durante a noite.
- Lugar arredondado membrana de nitrocelulose na placa de YPD, certifique-se que não há bolhas entre a membrana ea placa de YPD. Após 1-2 min, make se todas as colônias são transferidos para membranas (papel de nitrocelulose).
- Coloque o lado colônia de membrana em barco feito com papel de alumínio.
- Colocar a membrana em nitrogênio líquido, esperar 20 segundo, e submergir em nitrogênio líquido por alguns minutos.
- Retire membrana para descongelar.
- Enquanto isso, misture o reagente seguinte:
1,5 ml de buffer Z
20μl X-gal (20 mg / ml) - Queda Z buffer / X-gal, numa placa de Petri, coloque papel Whatman em cima disso.
- Coloque cuidadosamente o papel de nitrocelulose em cima de papel Whatman.
- Incubar a 37 ° até colônias azuis aparecem.
8. Ensaio retransformação
Proteínas de fusão Prey (AD-Y) isolados de triagem biblioteca deve manter a interação com a proteína de fusão isca (DB-X) para induzir os genes relatório, e os clones retransformação de rapina e isca construir em levedura pode ainda eliminar falsos positivos e facilitar adicionaranálise itional.
- Isolar DNA plasmidial de cepas de leveduras potencialmente contendo proteínas que interagem.
- Transformar DNA em células de E. coli DH5α, a chapa do transformações no LB 100 mg / ml ampicilina placas para seletivamente isolar a biblioteca pPC86-cDNA, e incubar durante a 37 ° C.
- Escolha diversas colônias da placa para colocar em LB 100 mcg / ml caldo de ampicilina.
- Prepare miniprep DNA e examinar por análise de restrição de casal com Sal I e Not I.
- Co-transform a presa plasmídeo e isca plasmídeo em MaV203, a chapa do misturas de transformação na SC-Leu-Trp placas e incubar durante 2-3 dias a 30 ° C.
- Escolher três diferentes colônias da placa SC-Leu-Trp, execute X-gal de Ensaio.
9. Seqüenciamento e análise bioinformática
Seqüenciar o DNA plasmídeo que foi isolado provavelmente verdade clones positivos, compare essas seqüências aos do GenBank utilizando o BLAST programa, e identificar os clones de dois híbridos, que correspondem a genes conhecidos. Dados de sequenciamento mostrou que dois dos 12 positivos obtidos foram acima da superfície celular TNFR2 (TNFRSF1B/CD120b; Adesão # NM_130426). Além disso, a interação entre PGRN e TNFR foi verificada usando vários ensaios de interação proteína-proteína, incluindo in vitro em fase sólida ensaio de ligação, Co-imunoprecipitação, plasmon de superfície análise de ressonância, e ensaio de citometria de fluxo 14.
10. Resultados representante
O fluxograma da triagem é descrito na fig. 3. Normalmente 50-100 candidatos clone positivo será obtido nesta etapa. Inicialmente isolados 54 candidatos clone positivo entre 2,5 milhão transformantes selecionados com a isca PGRN. Candidatos clone positivos foram verificados, em seguida, através da realização de X-gal ensaio. Normalmente, cerca de 50% de falsos positivos clones são removidos através de X-gal ensaio. Obtivemos 23 clones positivos candid ates nesta etapa para a isca PGRN (Fig. 4). Retransformação dos clones presa e construção de isca para a levedura ainda eliminar falsos positivos e clones que ainda ativar os genes repórter provavelmente representam os verdadeiros positivos. Normalmente, cerca de 50% candidatos clone positivo será removido. Finalmente isolado 12 clones positivos que interagem com PGRN em levedura.
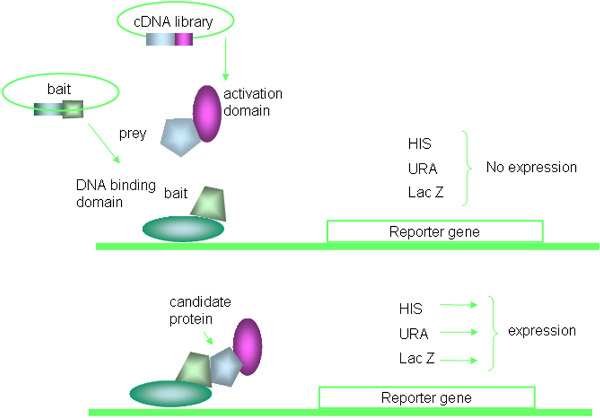
Figura 1. Princípio da levedura sistema de dois híbridos

Figura 2. Pipeline de identificação de parceiros de ligação às proteínas usando o sistema de levedura híbrida de dois

Figura 3. Fluxograma de levedura triagem dois híbridos biblioteca.rge.jpg "target =" _blank "> Clique aqui para ver uma versão em tamanho normal da imagem.

Figura 4. Ensaio de beta-galactosidase de candidatos clone positivo. A clones, positivos obtidos a partir do ecrã biblioteca foram transferidas para placa de YPD e incubadas a 30 ° C durante a noite; B, Todas as colônias na chapa YPD foram transferidas para membranas de nitrocelulose e beta-galactosidase ensaio realizado.
Access restricted. Please log in or start a trial to view this content.
Discussão
Triagem levedura híbrida de dois provou ser uma ferramenta eficaz na identificação de interação proteína 16, 17. Em comparação com outras abordagens para a identificação de proteínas de ligação de parceiros, como a bioquímica co-purificação de proteínas e chips, levedura híbrida de dois sistemas é uma abordagem sensível genética que pode ser usado para screening um grande número de seqüências de codificação em um experimento relativamente simples, em Além disso, ele detecta a intera...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado por verbas de pesquisa NIH K01AR053210, R01AR061484 e uma bolsa da Fundação Nacional de Psoríase.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| ProQuest sistema de dois híbridos | Invitrogen | 10835 | |
| YPD Meio de Crescimento | Clontech | 630409 | |
| YPD Agar Médio | Clontech | 630410 | |
| Base de Dados de agar mínimo SD | Clontech | 630412 | |
| -Leu DO Suplemento | Clontech | 630414 | |
| -Leu/-Trp DO Suplemento | Clontech | 630417 | |
| -His/-Leu/-Trp/-Ura DO Suplemento | Clontech | 630425 | |
| 3-Amino-1 ,2,4-triazol (3AT) | Sigma-Aldrich | A8056 | |
| Luria caldo (LB) | Sigma-Aldrich | L3022 | |
| X-Gal | Invitrogen | 15520-034 | |
| DNA de esperma de salmão sonicado | Stratagene | 201190 | |
| Ampicilina | AMRESCO | 0339 | |
| Sulfato de canamicina | Invitrogen | 11815-024 | |
| Eficiência subclonagem ™ DH5α ™ células competentes | Invitrogen | 18265-017 | |
| Trizma ® de base | Sigma-Aldrich | T6066 | |
| Acetato de lítio | Sigma-Aldrich | L4158 | |
| Ácido etilenodiaminotetracético | Sigma-Aldrich | ED | |
| Membrana de nitrocelulose | Bio-Rad | 162-0115 | |
| 10 cm de placa de Petri | ITI Scientific | CT-903 | |
| Incubadora (30 ° C) | ATR (Ecotron) |
Referências
- He, Z. Progranulin is a mediator of the wound response. Nat. Med. 9, 225-229 (2003).
- Kessenbrock, K. Proteinase 3 and neutrophil elastase enhance inflammation in mice by inactivating antiinflammatory progranulin. J. Clin. Invest. 118, 2438-2447 (2008).
- Zhu, J. Conversion of proepithelin to epithelins: roles of SLPI and elastase in host defense and wound repair. Cell. 111, 867-878 (2002).
- Yin, F. Exaggerated inflammation, impaired host defense, and neuropathology in progranulin-deficient mice. J. Exp. Med. 207, 117-128 (2010).
- Van Damme, P. Progranulin functions as a neurotrophic factor to regulate neurite outgrowth and enhance neuronal survival. J. Cell. Biol. 181, 37-41 (2008).
- Baker, M. Mutations in progranulin cause tau-negative frontotemporal dementia linked to chromosome 17. Nature. 442, 916-919 (2006).
- Cruts, M. Null mutations in progranulin cause ubiquitin-positive frontotemporal dementia linked to chromosome 17q21. Nature. 442, 920-924 (2006).
- Guo, F. Granulin-epithelin precursor binds directly to ADAMTS-7 and ADAMTS-12 and inhibits their degradation of cartilage oligomeric matrix protein. Arthritis Rheum. 62, 2023-2036 (2010).
- Feng, J. Q. Granulin epithelin precursor: a bone morphogenic protein 2-inducible growth factor that activates Erk1/2 signaling and JunB transcription factor in chondrogenesis. FASEB. J. 24, 1879-1892 (2010).
- Xu, K. Cartilage oligomeric matrix protein associates with granulin-epithelin precursor (GEP) and potentiates GEP-stimulated chondrocyte proliferation. J. Biol. Chem. 282, 11347-11355 (2007).
- Liu, C. J. The role of ADAMTS-7 and ADAMTS-12 in the pathogenesis of arthritis. Nat. Clin. Pract. Rheumatol. 5, 38-45 (2009).
- Liu, C., Dib-Hajj, S. D., Waxman, S. G. Waxman, S.G. Fibroblast growth factor homologous factor 1B binds to the C terminus of the tetrodotoxin-resistant sodium channel rNav1.9a (NaN). J. Biol. Chem. 276, 18925-18933 (2001).
- Liu, C. J. ADAMTS-7: a metalloproteinase that directly binds to and degrades cartilage oligomeric matrix protein. FASEB. J. 20, 988-990 (2006).
- Tang, W. The Growth Factor Progranulin Binds to TNF Receptors and Is Therapeutic Against Inflammatory Arthritis in Mice. Science. 332, 478-484 (2011).
- Wu, H., Siegel, R. M. Progranulin Resolves Inflammation. Science. 332, 427-428 (2011).
- Hollenberg, S. M. Identification of a new family of tissue-specific basic helix-loop-helix proteins with a two-hybrid system. Mol. Cell. Biol. 15, 3813-3822 (1995).
- Vojtek, A. B., Hollenberg, S. M., Cooper, J. A. Mammalian Ras interacts directly with the serine/threonine kinase Raf. Cell. 74, 205-214 (1993).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados