Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Modifizierten Hefe-Zwei-Hybrid-System, um Proteine interagieren mit dem Growth Factor Progranulin Identifizieren
In diesem Artikel
Zusammenfassung
Wir haben die konventionelle Hefe-Zwei-Hybrid-Screening, ein wirksames Instrument bei der Identifizierung genetischer Protein-Interaktion verändert. Diese Modifikation deutlich verkürzt den Prozess, reduziert den Arbeitsaufwand und vor allem, reduziert die Anzahl von Fehlalarmen. Darüber hinaus ist dieser Ansatz reproduzierbar und zuverlässig.
Zusammenfassung
Progranulin (PGRN), auch als granulin epithelin Vorstufe (GEP) bekannt, ist ein 593-Aminosäuren-autokrine Wachstumsfaktor. PGRN ist bekannt, dass eine kritische Rolle in einer Vielzahl von physiologischen und Krankheitsprozessen spielen, einschließlich der frühen Embryogenese, Wundheilung 1, Entzündung 2, 3, und Host-Sicherheitsebene 4. PGRN auch als neurotrophen Faktor 5, und Mutationen in der PGRN Gens zu einer teilweisen Verlust der PGRN Protein verursachen frontotemporale Demenz 6, 7. Unsere jüngsten Studien haben auf die Isolierung von PGRN als ein wichtiger Regulator des Knorpels Entwicklung und Abbau 8-11 geführt. Obwohl PGRN, entdeckte vor fast zwei Jahrzehnten, spielt eine entscheidende Rolle bei vielen physiologischen und pathologischen Bedingungen, die Bemühungen um die Aktionen der PGRN nutzen und verstehen die beteiligten Mechanismen wurden erheblich durch unsere Unfähigkeit behindert, seine Bindung Rezeptor (en) zu identifizieren. Um dieses Problem anzugehen, haben wir ein modiFied Hefe-Zwei-Hybrid (MY2H) Ansatz, der auf die am häufigsten verwendeten GAL4 basierten 2-Hybrid-System. Gegenüber der konventionellen Hefe-Zwei-Hybrid-Screen, MY2H verkürzt den Bildschirm und reduziert die Anzahl der falsch-positiven Klone. Darüber hinaus ist dieser Ansatz reproduzierbar und zuverlässig, und wir haben erfolgreich dieses System bei der Isolierung der Proteine aus verschiedenen Ködern, darunter Ionenkanal 12, extrazellulären Matrix Protein 10, 13, und Wachstumsfaktor 14 verwendet. In diesem Papier beschreiben wir diese MY2H experimentellen Verfahren im Detail mit PGRN als ein Beispiel, das zur Identifizierung von TNFR2 als der erste bekannte PGRN-assoziierten Rezeptors 14, 15 geführt.
Protokoll
1. Hintergrundinformationen
Die Hefe-Zwei-Hybrid-System ist eine leistungsfähige genetische Technik verwendet, um Protein-Protein-Interaktionen 16, 17 zu entdecken. Mehrere Arten von 2-Hybrid-Systeme, wie lexA-basierte Systeme, die Sos Recruitment System, und Bakterien-oder Säugetierzellen basierenden 2-Hybriden sind kommerziell verfügbar, dieses Papier konzentriert sich insbesondere auf die Änderungen der am häufigsten verwendeten GAL4 Basis Hefe 2-Hybrid-System. Kurz gesagt, ist die Methode, auf die Eigenschaften der Hefe GAL4-Protein, das von trennbaren Domänen verantwortlich für die DNA-Bindung und Transkriptionsaktivierung zusammensetzt. Der Köder-Protein als Fusionsprotein mit der GAL4 DNA-bindende Domäne (DNA-BD) exprimiert, während die Beute Proteine als Fusionen mit der GAL4-Aktivierungsdomäne (AD) ausgedrückt werden. Die Interaktion zwischen Köder und Beute Fusionsproteinen führt zur transkriptionellen Aktivierung des GAL4-Bindungsstellen, die Reporter-Gene, die in der Hefe g integriert sindenome. Das Prinzip der Y2H ist in Abb. dargestellt. 1 und das experimentelle Vorgehen ist in Abb. zusammengefasst. 2.
2. Benötigte Materialien und Lösungen
- YPD Growth Medium (eine Mischung aus Pepton, Hefeextrakt und Dextrose in optimalen Proportionen für den Anbau von den meisten Saccharomyces cerevisiae-Stämme).
- Minimal SD Bases (Minimal synthetischen definiert (SD)-Basen gehören ein Hefestickstoffbase, Ammoniumsulfat und eine Kohlenstoffquelle, Dextrose. Dropout (DO) Ergänzungen zu den Minimal SD Base kann hinzugefügt werden, um ein synthetisches, definiert Medium ohne die angegebene machen Nährstoffe).
- Leu /-Trp Dropout (DO) zu ergänzen (mit allen essentiellen Aminosäuren außer Leucin und Tryptophan)
- -His/-Leu/-Trp/-Ura Dropout (DO) Supplement (enthält alle essentiellen Aminosäuren außer Leucin, Tryptophan, Histidin und Uracil)
- Luria Broth (LB) (Trypton 10 g / L, Hefeextrakt 5 g / L, NaCl 5 g / L)
- X-Gal (5-Brom-4-Chlor-3-Indolyl-β-D-galactopyranosid) Lösung: vorbereitet als 20 mg / ml Stammlösung in N, N-Dimethylformamid
- 10X TE (100 mM Tris-HCl (pH 7,5), 10 mM EDTA, autoklaviert)
- Beschallt Herings-oder Lachssperma DNA, gekocht (10 mg / ml)
- 10X LiAc (1 M Lithium-Acetat, autoklaviert)
- 50% PEG-3350-Lösung, Filter-sterilisiert
- 3-Amino-1, 2, 4-Triazol (3AT)
- Kanamycin
- Ampicillin
- ElectroMax DH5 Zellen
- Z-Puffer: 16,1 g Na2HPO4 7H2O (oder 8,52 g wasserfrei), 5,5 g NaH2PO4 H2O (oder 4,8 ganhydrous), 0,75 g KCl, 0,246 g MgSO4 7H2O (oder 0,12 g wasserfrei), in 1 L autoklaviert, destilliertem Wasser gelöst und auf pH-Wert 7,0.
3. Bait Generation (pDBLeu-PGRN)
Ein cDNA-Fragment, das PGRN fehlt Signalpeptid (aa21-588) wurde gerichtet in die Sal I-Not I Stellen des pDBLeu Vektor (die ProQuest Two-Hybrid System, Invitrogen) kloniert,Beibehaltung der Übersetzung Leseraster wie die GAL4 DNA-bindende Domäne zu pDBLeu-PGRN generieren.
- Amplify der cDNA-Fragment von PGRN oben beschrieben durch PCR mit Oligonukleotid-Primer entworfen, um Restriktionsschnittstellen (Sal I am 5'-Ende und Not I am 3'-Ende), um in-frame-Fusion ermöglichen enthalten.
- Gel reinigt die PCR-Produkt, Endonukleasen verdaut mit Einschränkung Sal I und Not I
Bereiten Sie den DB-Vektor pDBLeu durch Doppelklick Restriktionsverdau mit den gleichen Restriktionsendonukleasen. - Ligieren die Beschränkung PGRN Fragment in den linearisierten Vektor pDBLeu und verwandeln sich in DH5 mit Auswahl für LB + Kanamycin bei 25 pg / ml.
- Überprüfen Sie, ob die Korrektur des Konstrukts durch DNA-Sequenzierung.
4. Kleine Transformation der Köder-Plasmid
- Inoculate 5 ml YPD mit einer Kolonie von Mav203 (Invitrogen), schütteln über Nacht bei 30 ° C.
- Verdünnen Sie die Übernacht-Kultur in 50 ml YPD. Wachsen Sie ein Additional 2-4 Stunden.
- Pellet die Zellen bei 3000 rpm für 5 min bei RT. Das Pellet in 40 ml autoklaviertem, destilliertem Wasser.
- Re-Pellet den Zellen. Resuspendieren in 2 ml Lösung I, bei RT 10 min inkubieren.
Lösung I: 0,5 ml 10xLiAc, 0,5 ml 10xTE, 4 ml H 2 O - Dispense die DNA zu Röhren: 2-3 pl pDBLeu-PGRN (0.25μg/μl) und 10 ul denaturierte, gescherte Lachssperma-DNA (10μg/μl). Add 100 &mgr; Hefezellen, gut mischen.
- Fügen Sie 700μl Lösung II, gut mischen. Inkubation bei 30 ° C für 30 min. Lösung II: 0,2 ml 10xLiAc, 0,2 ml 10xTE, 1,6 ml 50% PEG3350.
- Hitzeschock bei 42 ° C für 15 min.
- Pellet für 2 min. Überstand verwerfen. Das Pellet in 200 ul autoklaviert, destilliertes Wasser.
- Platte der Suspension auf SD-Leu-Platten mit Verdünnungsreihen. Die Platte bei 30 ° C für 2-3 Tage.
5. Bait-Validierung
Vor der Durchführung yeast-Zwei-Hybrid-Bildschirm Test pDBLeu-PGRN zur Selbsthilfe Aktivierung und bestimmen die basale Expression des HIS3-Reportergens. Dieser Test bestimmt, ob Köder Transkription zu aktivieren und ob die Selbst-Aktivierung kann durch Inhibitoren neutralisiert werden. 3-Amino-1, 2, 4-triazol (3-AT) ist ein kompetitiver Inhibitor des HIS3-Gen-Produkt und kann verwendet werden, um das Mindestniveau der HIS3 Ausdruck für das Wachstum auf Histidin-defizienten Medien erforderlich titriert werden.
- Transformieren Sie die pDBLeu-PGRN in Hefe Mav203 Stamm, Platte der Transformation auf SD-Leu-Platten und Inkubation für 48-72 h bei 30 ° C.
- Patch-Kolonien von jeder Transformation mit Hilfe von Zahnstochern auf autoklaviert SD-Leu-His-Platten mit 3-AT bei Konzentrationen von 10 mm, 25 mm, 50 mm, 75 mm und 100 mM. 3-AT ist ein kompetitiver Inhibitor des HIS3 Enzym und nur Köder, die Selbst-Aktivierung aufweisen können, in der Gegenwart des 3-AT wachsen.
- Bait-Stämme, die auf den Platten containi wachsenng 100 mM 3-AT sind nicht geeignet für den Einsatz im Zwei-Hybrid-Screen. Für Köder, die verwendet werden können, verwenden Sie den niedrigsten 3-AT Konzentration, die das Zellwachstum hemmt. In vielen Fällen ist 25 mM 3-AT für das Screening verwendet.
6. cDNA-Bank-Screening
Die pDBLeu-PGRN Plasmid in MaV203 eingeführt mit einer kleinen Veränderung, wie oben beschrieben. Zur Einführung pPC86-Bibliothek (Invitrogen) in MaV203 (pDBLeu-PGRN), das nachstehend beschriebene Verfahren in der Regel gibt ~ 4 x 10 4 Kolonien mit 0,5 ug Plasmid-Bibliothek DNA. Daher werden 2,5 x 10 6 Hefetransformanten erfordern ~ 30,0 pg pPC86-cDNA-Bibliothek Plasmid-DNA, 25 Transformationen und fünfzig 10-cm-Platten (SD-Leu-Trp-His-Ura 3 AT).
- Bereiten Sie die entsprechende Anzahl von 10-cm SD-Leu-Trp-His-Ura 3 AT-Platten (50 für das Verfahren weiter unten). Bereiten Sie zudem mindestens vier 10-cm SD-Leu-Trp-Platten für die Schätzung der Zahl der Transformanten.
- Transformieren Sie die Bibliothek indie MaV203 mit pDBLeu-PGRN nach dem unten beschriebenen Verfahren.
- Suspend mehrere isolierte Kolonien MaV203 (pDBLeu-PGRN) in ~ 100 &mgr; autoklaviert, destilliertem Wasser und breitete sie auf einen 10-cm SD-Leu Platte. Wiederholen Sie den Vorgang für eine zweite SD-Leu Platte. Inkubieren beide Platten für 18-24 h bei 30 ° C.
- Kratzen und vollständig die Zellen in 10 ml autoklaviert, destilliertes Wasser. Fügen Sie ein ausreichendes Volumen an Zellsuspension zu 500 ml Flüssigkeit YPD-Medium in einem Kolben bis zu einer OD 600 von ~ 0,1 geben.
- Stellen Sie sicher, dass die OD ~ 0,1 ist nach der Impfung.
- Schütteln bei 30 ° C, bis die OD 600 ~ 0,4 erreicht.
- Bereiten Sie frisch:
- 110 ml 1X TE / LiAc durch die Kombination von 11 ml 10X TE, 11 ml 10X LiAc, und 88 ml Wasser autoklaviert.
- 16 ml PEG / LiAc durch die Kombination von 1,6 ml 10X TE, 1,6 ml 10X LiAc und 12,8 ml 50% PEG-3350.
- Pellet die Zellen bei 3000g für 5 min bei Raumtemperatur.
- Discard ter Überstände und sanft das Pellet durch Auf-und Abpipettieren in 100 ml autoklaviert, destilliertem Wasser bei Raumtemperatur.
- Zentrifugation bei 3000 g für 5 min bei Raumtemperatur.
- Verwerfen Sie den Überstand der zentrifugierten Zellen und Zellpellet in 50 ml 1X TE / LiAc Lösung.
- Zentrifugation bei 3000 g für 5 min bei Raumtemperatur.
- Entfernen Sie die Überstände und das Pellet in einem Endvolumen von 2,5 ml 1X TE / LiAc Lösung.
- Führen Sie 25-Transformationen. Kombinieren Sie 2,5 ml Zellen, 125 ul (10μg/μl) denaturiert, gescherte Lachssperma DNA und 30 ug cDNA-Bibliothek. Vorsichtig mischen durch Auf-und Abpipettieren. 15 ml PEG / LiAc Lösung und vorsichtig mischen. Aliquot in 25 autoklaviert 1,5-ml Reaktionsgefäße von 700 ul jeder.
- Inkubieren für 30 min in einem 30 ° C Wasserbad.
- Hitzeschock für 15 min in einem 42 ° C Wasserbad.
- Zentrifuge bei 6.000 g für 1 min bei Raumtemperatur. Entfernen Sie vorsichtig die supernatant. Vorsichtig resuspendieren jedes Pellet in 400 ul autoklaviert, destilliertem Wasser mit einer Pipette auf und ab.
- Plate 200 ul Transformation Mischung aus jeder Transformation auf einzelne 10-cm SD-Leu-Trp-His-Ura 3 AT (3AT Konzentration: 25 mM)-Platten mit einem Spread bar, So 25 autoklaviert 1,5-ml Reaktionsgefäße kann 50 Platten machen (SD-Leu-Trp-His-Ura 3 AT).
- Die Platten für 5-10 Tage bei 30 ° C.
- Werden auf SD-Leu-Trp ausplattiert; Um die Transformationseffizienz der Reaktion wurden serielle Verdünnungen von einer Reaktion (1:400 und 1:4000 1:40) zu schätzen. Nach Inkubation für 3 Tage bei 30 ° C, die Anzahl der Kolonien gezählt und die Gesamtzahl der Transformanten berechnet.
7. X-gal-Assay
- Transfer-Kolonien auf YPD Platte; inkubieren bei 30 ° C über Nacht.
- Gerundet Nitrozellulosemembran auf der YPD Platte; stellen Sie sicher, es gibt keine Blasen zwischen der Membran und der YPD Platte. Nach 1-2 min, make, dass alle Kolonien sind an Membranen (Nitrocellulose-Papier) übertragen.
- Legen Sie die Membran Kolonie Seite nach oben im Boot mit Alufolie Papier.
- Legen Sie die Membran in flüssigen Stickstoff, warten Sie 20 Sekunden, und tauchen in flüssigen Stickstoff für ein paar Minuten.
- Nehmen Sie Membran zu tauen.
- Mittlerweile mischen die folgenden Reagenzien:
1,5 ml Z-Puffer
20 &mgr; l X-gal (20 mg / ml) - Drop-Z-Puffer / X-gal in einer Petrischale, legen Whatman-Papier obendrein.
- Vorsichtig Nitrocellulosepapier oben auf Whatman-Papier.
- Bei 37 ° bis blaue Kolonien erscheinen.
8. Retransformation Test
Prey-Fusionsproteine (AD-Y) von Bibliotheks-Screening isoliert sollten weiterhin die Interaktion mit Köder-Fusionsprotein (DB-X), um den Bericht Gene zu induzieren, und die Rückverwandlung des Beute-Klone und Köder in Hefe-Konstrukt kann weitere beseitigen Fehlalarme und erleichtern hinzufügenitional Analyse.
- Isolieren Plasmid-DNA aus Hefe-Stämme potentiell interagierende Proteine enthalten.
- Transform-DNA in E. coli DH5 Zellen Platte die Transformationen auf LB 100 pg / ml Ampicillin-Platten, um selektiv isolieren pPC86-cDNA-Bibliothek, beimpft und über Nacht bei 37 ° C.
- Wählen Sie mehrere Kolonien von der Platte in LB setzen 100 ug / ml Ampicillin Brühe.
- Bereiten Miniprep DNA und durch einen Doppelklick Restriktionsanalyse mit Sal I und Not I untersuchen
- Co-Transformation die Beute Plasmid und Köder-Plasmid in MaV203, Platte der Transformation Mischungen auf SC-Leu-Trp-Platten und Inkubation für 2-3 Tage bei 30 ° C.
- Wählen Sie drei verschiedene Kolonien von der SC-Leu-Trp Platte, führen X-gal-Assay.
9. Sequenzierung und bioinformatische Analyse
Sequence die Plasmid-DNA, die aus wahrscheinlich wahre positive Klone isoliert wurde, vergleichen Sie diese Sequenzen wie in der GenBank unter Verwendung der BLAST-Programm, und diejenigen identifizieren, two-hybrid-Klone, die zu bekannten Genen entsprechen. Sequencing Daten zeigten, dass zwei von den 12 positiven oben erhaltenen Zelloberfläche TNFR2 (TNFRSF1B/CD120b; Accession # NM_130426) wurden. Darüber hinaus wurde das Zusammenspiel zwischen PGRN und TNFR überprüft mittels verschiedener Protein-Protein Interaktion Assays, einschließlich In-vitro-Solid-Phase-Bindungsassay, Co-Immunpräzipitation, Surface-Plasmon-Resonanz-Analyse und Durchflusszytometrie-Assay 14.
10. Repräsentative Ergebnisse
Der Ablaufplan des Screening ist in Abb. dargestellt. 3. In der Regel 50-100 positive Klon Kandidaten werden bei diesem Schritt erreicht werden. Wir haben uns zunächst isoliert 54 positive Klon Kandidaten unter 2,5 Millionen Transformanten mit dem PGRN Köder gezeigt. Positive Klon Kandidaten wurden dann durch Ausführen X-gal-Assay überprüft. Typischerweise sind etwa 50% falsch-positive Klone mittels X-gal-Assay entfernt. Wir erhalten 23 positiven Klon candid Ates auf diesen Schritt für die PGRN Köder (Abb. 4). Rücktransformation der Beute Klone und Köder Konstrukt in Hefe weiter zu reduzieren False Positives und Klone, die noch aktivieren Sie die Reporter-Gene wahrscheinlich den wahren Positiven. Typischerweise werden etwa 50% positive Klon Kandidaten entfernt werden. Wir haben endlich isoliert 12 positive Klone, die mit PGRN in Hefe interagieren.
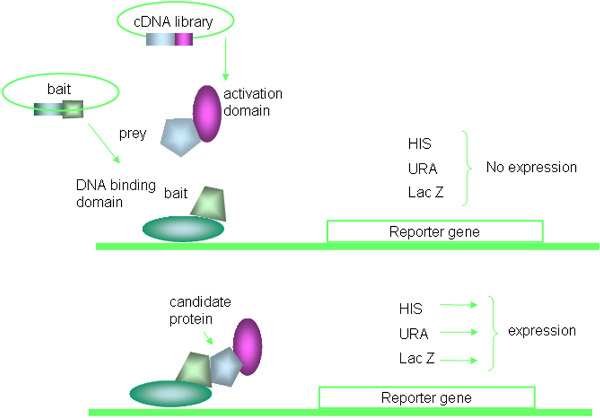
Abbildung 1. Prinzip des Hefe-Zwei-Hybrid-System

Abbildung 2. Pipeline identifizieren Protein Bindungspartner mit Hefe-Zwei-Hybrid-System

Abbildung 3. Ablaufschema Screening Hefe-Zwei-Hybrid-Bibliothek.rge.jpg "target =" _blank "> hier klicken, um einen Full-Size-Version dieses Bildes zu sehen.

Abbildung 4. Beta-Galactosidase Assay positiver Klon Kandidaten. A, Positive Klone aus der Bibliothek-Bildschirm erhalten wurden YPD-Platte transferiert und bei 30 ° C über Nacht; B, Alle Kolonien auf YPD-Platte wurden auf Nitrocellulose-Membranen übertragen und beta-Galaktosidase-Assay durchgeführt.
Diskussion
Hefe-Zwei-Hybrid-Screening hat sich als wirksames Instrument bei der Identifizierung von Protein-Interaktion 16, 17 sein. Im Vergleich mit anderen Ansätzen zur Identifizierung von Protein-Bindungspartner, wie biochemische co-Reinigungs-und Protein-Chips, Hefe-Zwei-Hybrid-System ist ein sensibler genetischer Ansatz, der für das Screening sehr hohe Zahl von kodierenden Sequenzen in einer relativ einfachen Experiment verwendet werden können; in Zudem erkennt es die in vivo Interaktion und braucht keine kompli...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde vom NIH Forschungsstipendien K01AR053210, R01AR061484 und einen Zuschuss von der National Psoriasis Foundation finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
| ProQuest Two-Hybrid System | Invitrogen | 10835 | |
| YPD Growth Medium | Clontech | 630409 | |
| YPD Agar Medium | Clontech | 630410 | |
| Minimal SD-Agar-Basis | Clontech | 630412 | |
| -Leu DO Supplement | Clontech | 630414 | |
| -Leu/-Trp DO Supplement | Clontech | 630417 | |
| -His/-Leu/-Trp/-Ura DO Supplement | Clontech | 630425 | |
| 3-Amino-1 ,2,4-triazol (3AT) | Sigma-Aldrich | A8056 | |
| Luria Broth (LB) | Sigma-Aldrich | L3022 | |
| X-Gal | Invitrogen | 15520-034 | |
| Ultraschall behandelter Lachssperma DNA | Stratagene | 201190 | |
| Ampicillin | AMRESCO | 0339 | |
| Kanamycinsulfat | Invitrogen | 11815-024 | |
| Subklonierung Efficiency ™ DH5a ™ Competent Cells | Invitrogen | 18265-017 | |
| Trizma ® base | Sigma-Aldrich | T6066 | |
| Lithium-Acetat | Sigma-Aldrich | L4158 | |
| Ethylendiamintetraessigsäure | Sigma-Aldrich | ED | |
| Nitrocellulosemembran | Bio-Rad | 162-0115 | |
| 10-cm-Petrischale | ITI Scientific | CT-903 | |
| Incubator (30 ° C) | ATR (Ecotron) |
Referenzen
- He, Z. Progranulin is a mediator of the wound response. Nat. Med. 9, 225-229 (2003).
- Kessenbrock, K. Proteinase 3 and neutrophil elastase enhance inflammation in mice by inactivating antiinflammatory progranulin. J. Clin. Invest. 118, 2438-2447 (2008).
- Zhu, J. Conversion of proepithelin to epithelins: roles of SLPI and elastase in host defense and wound repair. Cell. 111, 867-878 (2002).
- Yin, F. Exaggerated inflammation, impaired host defense, and neuropathology in progranulin-deficient mice. J. Exp. Med. 207, 117-128 (2010).
- Van Damme, P. Progranulin functions as a neurotrophic factor to regulate neurite outgrowth and enhance neuronal survival. J. Cell. Biol. 181, 37-41 (2008).
- Baker, M. Mutations in progranulin cause tau-negative frontotemporal dementia linked to chromosome 17. Nature. 442, 916-919 (2006).
- Cruts, M. Null mutations in progranulin cause ubiquitin-positive frontotemporal dementia linked to chromosome 17q21. Nature. 442, 920-924 (2006).
- Guo, F. Granulin-epithelin precursor binds directly to ADAMTS-7 and ADAMTS-12 and inhibits their degradation of cartilage oligomeric matrix protein. Arthritis Rheum. 62, 2023-2036 (2010).
- Feng, J. Q. Granulin epithelin precursor: a bone morphogenic protein 2-inducible growth factor that activates Erk1/2 signaling and JunB transcription factor in chondrogenesis. FASEB. J. 24, 1879-1892 (2010).
- Xu, K. Cartilage oligomeric matrix protein associates with granulin-epithelin precursor (GEP) and potentiates GEP-stimulated chondrocyte proliferation. J. Biol. Chem. 282, 11347-11355 (2007).
- Liu, C. J. The role of ADAMTS-7 and ADAMTS-12 in the pathogenesis of arthritis. Nat. Clin. Pract. Rheumatol. 5, 38-45 (2009).
- Liu, C., Dib-Hajj, S. D., Waxman, S. G. Waxman, S.G. Fibroblast growth factor homologous factor 1B binds to the C terminus of the tetrodotoxin-resistant sodium channel rNav1.9a (NaN). J. Biol. Chem. 276, 18925-18933 (2001).
- Liu, C. J. ADAMTS-7: a metalloproteinase that directly binds to and degrades cartilage oligomeric matrix protein. FASEB. J. 20, 988-990 (2006).
- Tang, W. The Growth Factor Progranulin Binds to TNF Receptors and Is Therapeutic Against Inflammatory Arthritis in Mice. Science. 332, 478-484 (2011).
- Wu, H., Siegel, R. M. Progranulin Resolves Inflammation. Science. 332, 427-428 (2011).
- Hollenberg, S. M. Identification of a new family of tissue-specific basic helix-loop-helix proteins with a two-hybrid system. Mol. Cell. Biol. 15, 3813-3822 (1995).
- Vojtek, A. B., Hollenberg, S. M., Cooper, J. A. Mammalian Ras interacts directly with the serine/threonine kinase Raf. Cell. 74, 205-214 (1993).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten