È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Modificato lievito-sistema del doppio ibrido per identificare proteine che interagiscono con il fattore di crescita progranulina
In questo articolo
Riepilogo
Abbiamo modificato il lievito tradizionale doppio ibrido di screening, un efficace strumento genetico per identificare interazioni proteiche. Questa modifica riduce notevolmente il processo, riduce il carico di lavoro e, soprattutto, riduce il numero di falsi positivi. Inoltre, questo approccio è riproducibile ed affidabile.
Abstract
Progranulina (PGRN), noto anche come precursore granulin epithelin (GEP), è un fattore 593-amino-acido crescita autocrino. PGRN è nota per svolgere un ruolo critico in una varietà di processi fisiologici e malattie, tra cui l'embriogenesi precoce, la guarigione della ferita 1, infiammazioni 2, 3, e ospitare difesa 4. PGRN funziona anche come un fattore neurotrofico 5, e le mutazioni nel gene PGRN con conseguente perdita parziale della proteina causano demenza frontotemporale PGRN 6, 7. I nostri studi recenti hanno portato all'isolamento di PGRN come un importante regolatore di sviluppo della cartilagine e del degrado 8-11. Anche se PGRN, scoperto quasi due decenni fa, gioca un ruolo cruciale in molteplici condizioni fisiologiche e patologiche, gli sforzi per sfruttare le azioni di PGRN e comprendere i meccanismi coinvolti sono stati significativamente ostacolata dalla nostra incapacità di identificare il suo legame al recettore (s). Per risolvere questo problema, abbiamo sviluppato un modicato doppio ibrido di lievito (MY2H) approccio basato sulla più comunemente utilizzati GAL4 base 2-sistema ibrido. Rispetto al tradizionale doppio ibrido di lievito schermo, MY2H riduce drasticamente il processo di schermo e riduce il numero di cloni falsi positivi. Inoltre, questo approccio è riproducibile e affidabile, e abbiamo impiegato con successo questo sistema per isolare le proteine leganti di esche, tra cui ioni di canale 12, proteine della matrice extracellulare 10, 13, e del fattore di crescita 14. In questo lavoro, si descrive questa procedura MY2H sperimentale in dettaglio utilizzando PGRN come un esempio che ha portato alla identificazione di TNFR2 come la prima nota PGRN associata recettore 14, 15.
Protocollo
1. Informazioni di base
Il sistema del doppio ibrido di lievito è una potente tecnica genetica utilizzata per scoprire interazioni proteina-proteina 16, 17. Diversi tipi di 2-sistemi ibridi, come i sistemi basati su lexa, il sistema di reclutamento Sos, e batteri o cellule di mammifero a base di 2-ibridi, sono commerciali disponibili, questo articolo si concentra in particolare sulle modifiche dei più comunemente usati GAL4 base lievito 2-sistema ibrido. In breve, il metodo si basa sulle proprietà della proteina GAL4 lievito che si compone di domini separabili responsabile per l'attivazione del DNA-binding e trascrizionale. La proteina esca è espresso come una fusione a DNA-binding GAL4 dominio (DNA-BD), mentre le proteine prede sono espressi come fusioni al dominio GAL4 attivazione (AD). L'interazione tra esca e proteine di fusione preda porta alla attivazione trascrizionale di GAL4 siti di legame contenente geni reporter che sono integrati nella g di lievitoenome. Il principio di Y2H è illustrato nella fig. 1 e la procedura sperimentale è sintetizzato nella fig. 2.
2. Materiali necessari e le soluzioni
- La crescita media YPD (una miscela di peptone, estratto di lievito, destrosio e in proporzioni ottimali per la coltivazione maggior parte dei ceppi Saccharomyces cerevisiae).
- Minime basi SD (Minimal sintetico definito (SD) basi includono una base di azoto lievito, solfato di ammonio, e una fonte di carbonio, destrosio. Dropout (DO), gli integratori possono essere aggiunti alla base minimale SD per fare una sintesi, un terreno specifico manca specificato nutrienti).
- Leu / Trp-Dropout (DO) supplemento (che contiene ogni amminoacido essenziale ad eccezione di leucina e triptofano)
- Dropout -His/-Leu/-Trp/-Ura (DO) Supplemento (contenente ogni amminoacido essenziale tranne che per la leucina, triptofano, istidina, e uracile)
- Luria brodo (LB) (Triptone 10 g / L, estratto di lievito 5 g / l, NaCl 5 g / L)
- X-Gal (5-bromo-4-cloro-3-indolil-β-D-galattopiranoside) soluzione: pronta come una dose di 20 mg / ml di soluzione concentrata in N, N-Dimetilformammide
- TE 10X (100 mM Tris-HCl (pH 7,5), 10 mM EDTA, autoclavato)
- Herring sonicato o sperma DNA di salmone, bolliti (10 mg / ml)
- LIAC 10X (1 acetato di litio M, autoclavato)
- 50% PEG-3350 la soluzione, sterilizzata per filtrazione
- 3-Amino-1, 2, 4-triazolo (3AT)
- Kanamicina
- Ampicillina
- ELECTROMAX DH5α cellule
- Z buffer: 16,1 g Na2HPO4 7H2O (o 8,52 g anidro), 5,5 g NaH2PO4 H2O (pari al 4,8 ganhydrous), 0,75 g di KCl, 0,246 g MgSO4 7H2O (o 0,12 g anidro), sciolto in 1 L autoclave, acqua distillata e regolato pH 7,0.
3. Bait generazione (pDBLeu-PGRN)
Un frammento di cDNA PGRN codifica manca peptide segnale (aa21-588) è stato clonato direzionale in I-Sal non ho siti del vettore pDBLeu (ProQuest il sistema del doppio ibrido, Invitrogen),mantenendo lo stesso frame di lettura traduzione come dominio di GAL4 DNA Binding per generare pDBLeu-PGRN.
- Amplificare il frammento di cDNA PGRN sopra descritti mediante PCR utilizzando primer oligonucleotidi progettati per contenere siti di restrizione (Sal io al 5'end e non io a 3'end) per consentire in-frame di fusione.
- Gel purificare il prodotto di PCR, digestione con endonucleasi di restrizione Not I e Sal I.
Preparare il DB vettore pDBLeu dalla digestione di restrizione doppio con enzimi di restrizione stessa. - Legare il frammento PGRN restrizione nel pDBLeu vettore linearizzato e trasformare in DH5α con selezione per LB + kanamicina a 25 mcg / ml.
- Verificare la correzione del costrutto di sequenziamento del DNA.
4. Piccola scala di trasformazione di esche plasmide
- Inoculare 5 ml di YPD con una colonia di Mav203 (Invitrogen), agitare una notte a 30 ° C.
- Diluire la cultura durante la notte in 50 ml di YPD. Crescere un additional 2-4 ore.
- Agglomerare le cellule a 3000 rpm per 5 minuti a temperatura ambiente. Risospendere il pellet in 40 ml di autoclave, acqua distillata.
- Re-agglomerare le cellule. Risospendere in 2 ml di soluzione di I, incubare a RT 10 min.
Soluzione I: 10xLiAc 0,5 ml, 0,5 ml 10xTE, 4 ml di H 2 O - Dispensare il DNA per tubi: 2-3 ml di pDBLeu-PGRN (0.25μg/μl) e 10 ml di denaturato, il DNA degli spermatozoi di salmone tranciati (10μg/μl). Aggiungere 100μl cellule di lievito, mescolare bene.
- Aggiungere 700μl soluzione II, mescolare bene. Incubare a 30 ° C per 30 min. Soluzione II: 0,2 ml 10xLiAc, 10xTE 0,2 ml, 1,6 ml, 50% PEG3350.
- Shock termico a 42 ° C per 15 min.
- Pellet per 2 min. Scartare il surnatante. Risospendere il pellet in 200 l autoclave, acqua distillata.
- Piatto la sospensione su SD-Leu piatti con diluizioni seriali. Incubare la piastra a 30 ° C per 2-3 giorni.
5. Bait convalida
Prima di eseguire sì° schermo doppio ibrido, test pDBLeu-PGRN per l'auto-attivazione e determinare i livelli basali di espressione del gene HIS3 reporter. Questo test determina se le esche attivare la trascrizione e se l'auto-attivazione può essere neutralizzato da inibitori. 3-Amino-1, 2, 4-triazolo (3-AT) è un inibitore competitivo del HIS3 gene-prodotto e può essere utilizzato per titolare il livello minimo di espressione HIS3 necessari per la crescita di istidina carenti di mezzi di comunicazione.
- Trasformare il pDBLeu-PGRN in Mav203 ceppo di lievito, la piastra di trasformazione su SD-Leu piastre e incubare per 48-72 ore a 30 ° C.
- Colonie patch di ogni trasformazione con stuzzicadenti autoclave su SD-Leu-His piastre contenenti 3-IN a concentrazioni di 10 mm, 25 mm, 50 mm, 75 mm e 100 mm. 3-AT è un inibitore competitivo dell'enzima HIS3 ed esche solo che mostrano auto-attivazione sarà in grado di crescere in presenza di 3-AT.
- Ceppi esca che crescono sulle piastre containing 100 mM 3-AT non sono adatti per l'utilizzo in doppio ibrido schermo. Per le esche che possono essere utilizzati, utilizzare la più bassa concentrazione di 3-A che inibisce la crescita cellulare. In molti casi, 25 mm di 3-AT è usato per lo screening.
6. biblioteca di screening di cDNA
Il plasmide pDBLeu-PGRN viene introdotto in MaV203 utilizzando una piccola trasformazione come descritto sopra. Per introdurre pPC86-library (Invitrogen) in MaV203 (pDBLeu-PGRN), la procedura descritta di seguito dà tipicamente ~ 4 x 10 4 colonie con 0,5 mg di DNA plasmidico biblioteca. Quindi, 2,5 x 10 6 trasformanti lievito richiederà ~ 30,0 mcg pPC86-cDNA DNA biblioteca plasmide, 25 trasformazioni, e cinquanta piastre da 10 cm (SD-Leu-Trp-His-Ura 3 AT).
- Preparare il numero appropriato di 10 cm di SD-Leu-Trp-His-Ura +3 al piatti (50 per la procedura di seguito). Anche preparare almeno quattro di 10 cm SD-Leu-Trp piastre per stimare il numero di trasformanti.
- Trasformare la libreria inil MaV203 contenente pDBLeu-PGRN secondo la procedura descritta di seguito.
- Sospendere diverse colonie isolate di MaV203 (pDBLeu-PGRN) in ~ 100μl autoclave, acqua distillata e diffonderle su una di 10 cm SD-Leu piatto. Ripetere la procedura per un secondo SD-Leu piatto. Incubare entrambe le piastre per 18-24 ore a 30 ° C.
- Raschiare e sospendere completamente le cellule in 10 ml in autoclave, acqua distillata. Aggiungere un volume sufficiente di sospensione cellulare a 500 ml di liquido di media YPD in un pallone per dare un OD 600 del ~ 0.1.
- Verificare che l'OD è ~ 0.1 dopo l'inoculazione.
- Agitare a 30 ° C fino a quando il diametro esterno raggiunge i 600 ~ 0.4.
- Preparare freschi:
- 110 ml 1X TE / LIAC unendo 11 ml 10X TE, 11 LIAC ml 10X, e 88 ml di acqua in autoclave.
- 16 ml PEG / LIAC combinando 1,6 ml 10X TE, 1,6 ml LIAC 10X, e 12,8 ml al 50% PEG-3350.
- Agglomerare le cellule a 3000g per 5 minuti a temperatura ambiente.
- Scartare tha surnatanti e delicatamente risospendere il pellet pipettando su e giù in 100 ml in autoclave, acqua distillata a temperatura ambiente.
- Centrifugare a 3000 g per 5 minuti a temperatura ambiente.
- Eliminare il surnatante delle cellule centrifugato e risospendere il pellet cellulare in 50 ml 1X TE / LIAC soluzione.
- Centrifugare a 3000 g per 5 minuti a temperatura ambiente.
- Rimuovere il surnatante e risospendere il pellet in un volume finale di 2,5 ml 1X TE / LIAC soluzione.
- Eseguire 25 trasformazioni. Unire celle 2,5 ml, 125 microlitri (10μg/μl) denaturati, tranciate DNA dello sperma di salmone, e 30 mcg biblioteca di cDNA. Mescolare delicatamente pipettando su e giù. Aggiungere 15 ml di PEG / LIAC soluzione e mescolare delicatamente. Aliquota in 25 autoclave da 1,5 ml tubi microcentrifuga di 700 microlitri ciascuno.
- Incubare per 30 minuti in un bagno d'acqua 30 ° C.
- Shock termico per 15 minuti in un bagno d'acqua ° 42 C.
- Centrifugare a 6.000 g per 1 minuto a temperatura ambiente. Rimuovere con attenzione il supersupernatante. Risospendere delicatamente ciascun pellet in 400 microlitri in autoclave, acqua distillata pipettando su e giù.
- Piastra da 200 microlitri miscela trasformazione da ogni trasformazione sul singolo di 10 cm SD-Leu-Trp-His-Ura 3 AT (3AT concentrazione: 25 mm) con piastre di una barra di diffusione, quindi 25 in autoclave da 1,5 ml, tubi microcentrifuga può fare 50 piastre (SD-Leu-Trp-His-Ura 3 AT).
- Incubare le piastre per 5-10 giorni a 30 ° C.
- Per stimare l'efficienza della trasformazione della reazione, diluizioni seriali di una reazione (1:40, 1:400 e 1:4000) sono placcati su SD-Leu-Trp piatto. Dopo incubazione per 3 giorni a 30 ° C, il numero di colonie viene contato e il numero totale di trasformanti calcolato.
7. X-gal assay
- Trasferimento colonie alla piastra YPD; incubare a 30 ° C durante la notte.
- Luogo arrotondati membrana di nitrocellulosa sul piatto YPD, assicurarsi che non vi siano bolle tra la membrana e la piastra YPD. Dopo 1-2 minuti, macerto ke tutte le colonie vengono trasferite su membrane (carta di nitrocellulosa).
- Posizionare il lato colonia membrana in barca fatta di carta foglio di alluminio.
- Mettere la membrana in azoto liquido, attendere 20 secondi, e sommergere in azoto liquido per un paio di minuti.
- Estrarre membrana a scongelare.
- Nel frattempo mescolare il reagente seguenti:
1,5 ml di buffer Z
20μl X-gal (20 mg / ml) - Caduta Z buffer / X-gal in una capsula di Petri, luogo di carta Whatman per di più.
- Con attenzione la carta di nitrocellulosa posto sopra la carta Whatman.
- Incubare a 37 ° fino a quando le colonie blu appaiono.
8. Ritrasformazione test
Proteine di fusione Prey (AD-Y) isolato da uno screening della libreria dovrebbe mantenere l'interazione con proteine di fusione esca (DB-X) per indurre i geni relazione e la ritrasformazione di cloni prede ed esche costruire nel lievito può inoltre eliminare i falsi positivi e facilitare aggiungereanalisi itional.
- Isolare il DNA plasmidico da ceppi di lievito che possono contenere proteine che interagiscono.
- Trasforma DNA in E. coli DH5α cellule, piastra le trasformazioni su LB 100 mcg / ml piastre ampicillina per isolare selettivamente i pPC86-cDNA biblioteca, e incubare una notte a 37 ° C.
- Raccogliere numerose colonie dalla piastra di mettere in LB 100 mcg / ml brodo ampicillina.
- Preparare miniprep DNA ed esaminare l'analisi di restrizione doppia con Sal io e non io
- Co-trasformare la preda plasmide e esca plasmide in MaV203 la targhetta del miscele di trasformazione su SC-Leu-Trp piastre e incubare per 2-3 giorni a 30 ° C.
- Scegli tre colonie di diverso da SC-Leu-Trp piastra, eseguire X-gal saggio.
9. Sequenziamento e l'analisi bioinformatica
Sequenza del DNA plasmide che è stato isolato da probabile cloni veri positivi, confrontare queste sequenze a quelle del GenBank utilizzando il BLAST programma, e identificare i due cloni ibridi che corrispondono a geni noti. Dati di sequenziamento ha dimostrato che due dei 12 positivi ottenuti in precedenza erano superficie cellulare TNFR2 (TNFRSF1B/CD120b; adesione # NM_130426). Inoltre, l'interazione tra PGRN e TNFR è stata verificata utilizzando varie proteina-proteina saggi di interazione, anche in vitro in fase solida test vincolante, Co-immunoprecipitazione, Surface Plasmon analisi di risonanza, e citometria a flusso saggio 14.
10. Rappresentante Risultati
Il diagramma di flusso dello screening è indicato nella fig. 3. Tipicamente 50-100 candidati clone positivo si ottiene in questa fase. Inizialmente abbiamo isolato 54 candidati clone positivo tra 2,5 milioni trasformanti screening con l'esca PGRN. Candidati clone positivi sono stati poi verificati eseguendo X-gal test. Tipicamente, circa il 50% dei cloni falsi positivi vengono rimossi tramite X-gal test. Abbiamo ottenuto 23 clone positivo candida Ates a questo passaggio per l'esca PGRN (Fig. 4). Ritrasformazione dei cloni preda e costruire esche in lieviti ulteriormente eliminare i falsi positivi e cloni che ancora attivare i geni giornalista probabilmente rappresentano l'veri positivi. Tipicamente, circa il 50% dei candidati clone positivo verrà rimosso. Abbiamo finalmente isolati 12 cloni positivi che interagiscono con PGRN nel lievito.
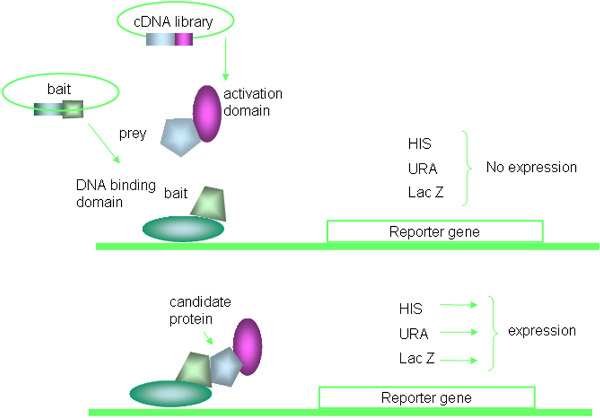
Figura 1. Principio di lievito sistema di doppio ibrido

Figura 2. Pipeline di identificazione di partner di legame con le proteine utilizzando lievito sistema del doppio ibrido

Figura 3. Diagramma di flusso di lievito di screening a due ibridi biblioteca.rge.jpg "target =" _blank "> Clicca qui per vedere una versione full-size di questa immagine.

Figura 4. Beta-galattosidasi saggio di candidati clone positivo. A, cloni positivi ottenuti dallo schermo della libreria sono stati trasferiti alla piastra YPD e incubato a 30 ° C durante la notte; B, tutte le colonie su piastra YPD sono stati trasferiti su membrane di nitrocellulosa e beta-galattosidasi test eseguiti.
Access restricted. Please log in or start a trial to view this content.
Discussione
Doppio ibrido di lievito di screening ha dimostrato di essere uno strumento efficace per identificare interazioni proteina 16, 17. Rispetto ad altri approcci per identificare le proteine di legame partner, come la biochimica di co-purificazione e proteine chip, lievito sistema del doppio ibrido è un approccio sensibile genetico che può essere utilizzato per lo screening numero molto elevato di sequenze codificanti in un esperimento relativamente semplice, in Inoltre, rileva l'interazione in ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato finanziato dal NIH assegni di ricerca K01AR053210, R01AR061484 e una borsa di National Psoriasis Foundation.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | |
| ProQuest sistema del doppio ibrido | Invitrogen | 10835 | |
| YPD crescita media | Clontech | 630409 | |
| YPD AGAR | Clontech | 630410 | |
| Minimo SD agar Base | Clontech | 630412 | |
| Leu-DO Supplemento | Clontech | 630414 | |
| -Leu/-Trp DO Supplemento | Clontech | 630417 | |
| -His/-Leu/-Trp/-Ura DO Supplemento | Clontech | 630425 | |
| 3-Amino-1 ,2,4-triazolo (3AT) | Sigma-Aldrich | A8056 | |
| Luria brodo (LB) | Sigma-Aldrich | L3022 | |
| X-Gal | Invitrogen | 15520-034 | |
| Sonicato Salmon Sperm DNA | Stratagene | 201190 | |
| Ampicillina | AMRESCO | 0339 | |
| Kanamicina solfato | Invitrogen | 11815-024 | |
| Efficienza subcloning ™ DH5α ™ cellule competenti | Invitrogen | 18265-017 | |
| Trizma ® di base | Sigma-Aldrich | T6066 | |
| Acetato di litio | Sigma-Aldrich | L4158 | |
| Etilendiammintetraacetato | Sigma-Aldrich | ED | |
| Membrana di nitrocellulosa | Bio-Rad | 162-0115 | |
| 10-cm scatola di Petri | ITI scientifico | CT-903 | |
| Incubatore (30 ° C) | ATR (Ecotron) |
Riferimenti
- He, Z. Progranulin is a mediator of the wound response. Nat. Med. 9, 225-229 (2003).
- Kessenbrock, K. Proteinase 3 and neutrophil elastase enhance inflammation in mice by inactivating antiinflammatory progranulin. J. Clin. Invest. 118, 2438-2447 (2008).
- Zhu, J. Conversion of proepithelin to epithelins: roles of SLPI and elastase in host defense and wound repair. Cell. 111, 867-878 (2002).
- Yin, F. Exaggerated inflammation, impaired host defense, and neuropathology in progranulin-deficient mice. J. Exp. Med. 207, 117-128 (2010).
- Van Damme, P. Progranulin functions as a neurotrophic factor to regulate neurite outgrowth and enhance neuronal survival. J. Cell. Biol. 181, 37-41 (2008).
- Baker, M. Mutations in progranulin cause tau-negative frontotemporal dementia linked to chromosome 17. Nature. 442, 916-919 (2006).
- Cruts, M. Null mutations in progranulin cause ubiquitin-positive frontotemporal dementia linked to chromosome 17q21. Nature. 442, 920-924 (2006).
- Guo, F. Granulin-epithelin precursor binds directly to ADAMTS-7 and ADAMTS-12 and inhibits their degradation of cartilage oligomeric matrix protein. Arthritis Rheum. 62, 2023-2036 (2010).
- Feng, J. Q. Granulin epithelin precursor: a bone morphogenic protein 2-inducible growth factor that activates Erk1/2 signaling and JunB transcription factor in chondrogenesis. FASEB. J. 24, 1879-1892 (2010).
- Xu, K. Cartilage oligomeric matrix protein associates with granulin-epithelin precursor (GEP) and potentiates GEP-stimulated chondrocyte proliferation. J. Biol. Chem. 282, 11347-11355 (2007).
- Liu, C. J. The role of ADAMTS-7 and ADAMTS-12 in the pathogenesis of arthritis. Nat. Clin. Pract. Rheumatol. 5, 38-45 (2009).
- Liu, C., Dib-Hajj, S. D., Waxman, S. G. Waxman, S.G. Fibroblast growth factor homologous factor 1B binds to the C terminus of the tetrodotoxin-resistant sodium channel rNav1.9a (NaN). J. Biol. Chem. 276, 18925-18933 (2001).
- Liu, C. J. ADAMTS-7: a metalloproteinase that directly binds to and degrades cartilage oligomeric matrix protein. FASEB. J. 20, 988-990 (2006).
- Tang, W. The Growth Factor Progranulin Binds to TNF Receptors and Is Therapeutic Against Inflammatory Arthritis in Mice. Science. 332, 478-484 (2011).
- Wu, H., Siegel, R. M. Progranulin Resolves Inflammation. Science. 332, 427-428 (2011).
- Hollenberg, S. M. Identification of a new family of tissue-specific basic helix-loop-helix proteins with a two-hybrid system. Mol. Cell. Biol. 15, 3813-3822 (1995).
- Vojtek, A. B., Hollenberg, S. M., Cooper, J. A. Mammalian Ras interacts directly with the serine/threonine kinase Raf. Cell. 74, 205-214 (1993).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon