É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Alto rendimento Gerador Protein Expression Usando uma plataforma microfluídica
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Apresenta-se um método de microfluidos para a expressão de arrays de proteínas. O dispositivo consiste de milhares de câmaras de reacção controladas por válvulas de micro-mecânicos. O dispositivo micro é acoplado a uma biblioteca de genes microarray-impresso. Estes genes são transcritos e traduzidos em seguida, on-chip, resultando numa matriz de proteína pronta para o uso experimental.
Resumo
Campos rápido crescimento, como a biologia de sistemas, exige o desenvolvimento e implementação de novas tecnologias, permitindo medições de alto rendimento e de alta fidelidade de grandes sistemas. Microfluídica promete cumprir muitos desses requisitos, como a realização de experimentos de alto throughput screening on-chip, abrangendo ensaios bioquímicos, biofísicos e baseada em células 1. Desde os primeiros dias de dispositivos microfluídicos, este campo tem evoluído drasticamente, levando ao desenvolvimento de integração em larga escala de microfluidos 2,3. Esta tecnologia permite a integração de milhares de válvulas micromecânica em um único dispositivo com uma pegada de porte grande (Figura 1). Nós desenvolvemos uma plataforma de alto rendimento microfluídicos para a geração de expressão in vitro de matrizes de proteína (Figura 2) chamado Ping (Proteína Gerador de rede de interação). Estas matrizes podem servir como um modelo para vários experimentostais como a proteína-proteína 4, proteína-RNA de 5 ou proteína ADN-6 interacções.
O dispositivo consiste de milhares de câmaras de reacção, que são programados individualmente utilizando um microarrayer. Alinhamento dos microarrays impresso para programas de dispositivos microfluídicos cada câmara com um único ponto eliminando a contaminação potencial ou reatividade cruzada Além disso, gerando microarrays utilizando técnicas de microarray padrão manchando também é muito modular, permitindo a arraying de proteínas, DNA 7 8, pequenas moléculas, e até mesmo as suspensões coloidais. O impacto potencial da microfluídica em ciências biológicas é significativo. Uma série de ensaios baseados em microfluídica já fornecido informações novas para a estrutura e função dos sistemas biológicos, e no campo da microfluídica continuarão a impactar biologia.
Protocolo
1. Fabricação de dispositivos
- Comprado DTPA-D SU-8 molde de controle e mofo SPR220-7 fluxo da microfluídica Stanford Fundição ( www.stanford.edu / grupo / fundição ).
- Expor os moldes de silicone de clorotrimetilsilano (TMCS) de vapor por 10 min para promover a libertação de elastómero após os passos de cozimento 9.
- Prepara-se uma mistura de elastómero de silicone com base e agente de cura (misturar bem), em duas diferentes proporções de 5:1 e 20:1 para o controle de fluxo e de moldes, respectivamente. Os rácios diferentes são necessários para a ligação apropriada de múltiplas camadas.
- Despeje a PDMS 05:01 sobre a camada de controlo (cerca de 5 mm de altura). Degas camada de controlo e cozê-la por 30 min a 80 ° C. Revestimento Spin (Laurell, EUA), a mistura a 20:1 PDMS sobre a camada de fluxo a 2.600 rpm durante 60 segundos e, em seguida, cozer a 80 ° C durante 30 min. A velocidade de rotação revestidor é optimizado para actuação da válvula, a uma pressãode 15 psi. Girando rápido resultará numa camada mais fina com uma pressão mais baixa activação e vice-versa. Os dispositivos que utilizam têm um limite de 25 psi no controle e 10 psi no fluxo, acima do qual desprendimento do substrato pode ocorrer.
- Separou-se a camada de controlo do molde. Faça isso devagar e ter cuidado para não descascar o padrão SU8. Em seguida, corte o dispositivo em torno de seu perímetro e furos para acessar os canais de controlo.
- Alinhe o fluxo e as camadas de controle manualmente sob um estereoscópio. Comece alinhando o canto superior esquerdo, a válvula de botão (camada de controlo) deve ser no meio da câmara de reacção (camada de fluxo). Em seguida, alinhe a primeira linha e gentilmente liberar a linha camada de controle por linha. Certifique-se de que todas as válvulas de botão está no meio das câmaras de reacção e de que as válvulas de entrada de endereço e atravessar os canais de fluxo na posição correcta. O processo pode ser repetido até que todas as linhas localmente estão alinhados. No final, libertar qualquer tensão no b PDMSy levantando-o cuidadosamente a partir dos lados e cantos. Não levante muito ou desalinhamento pode ocorrer.
- Asse durante 2 horas a 80 ° C.
- Corte em torno do perímetro do dispositivo de descascar e o dispositivo de camada dois (chip) a partir do molde de fluxo. Furos de acesso aos canais de fluxo (Figura 1).
2. Arraying DNA e Alinhamento Dispositivo
- Produzir genes sintéticos pela montagem PCR. Os genes sintéticos são compor de T7 promotor, sítio de ligação ao ribossoma (RBS), ORF com dois marcadores de epitopo (uma em cada extremidade), e um terminador de T7 4. Os genes podem variar em comprimento de 100 pb até, pelo menos, 5.000 pb.
- Preparar os genes sintéticos para arraying: preparar uma mistura de poli-etileno glycole (1,25%) e D-trealose di-hidratada (125 mg / ml) e 2 ul de distribuição por reacção em 384 poços de placa. Esta solução irá reduzir a ligação irreversível do DNA no vidro, bem como para a visualização durante alinhamento 10.
- Transferir os genes sintéticos para a placa de cavidades 384. Geralmente as concentrações de ADN pode variar de 10 ng / ul a 100 ng / ul concentração final. Adicionar dH 2 O para um volume final de 20 ul (depende microarrayer e tipo pino).
- Detectar uma série de genes sintéticos sobre substratos de vidro epoxi revestidos utilizando um microarrayer. Usamos MicroGrid 610 (Bio Robotics) com pinos SMT-S75 silicone (Parallel Synthesis, EUA). Contactar a impressão do ADN, com esses pinos específicas, resulta em manchas com diâmetro de cerca de 100 fim sobre a superfície de vidro. Cada pino de carga é suficiente para cerca de 100 pontos. Verifique se os campos de coluna e de linha correspondem ao dispositivo específico utilizado. O dispositivo usamos contém 16 colunas e 40 linhas com um arremesso de 680 mM por 320 pm, respectivamente.
- Alinhar manualmente o dispositivo de microfluidos para a matriz de gene sob um microscópio estereoscópico. A mancha de ADN deve estar no meio das câmaras de DNA. Iniciar o alinhamento, localizando a primeira fila de pontos below a primeira fila de câmaras de DNA. Em seguida, alinhar o resto das linhas de acabamento com ajuste de precisão de todo o dispositivo. Uma vez que a matriz é alinhado cuidado deve ser tomado para aliviar qualquer stress na PDMS, levantando-a localmente. Se houver tensão deixado no PDMS não pode ligar bem ao microarray.
- Finalmente, o dispositivo de ligação para a lâmina de vidro por incubação durante a noite numa placa quente a 80 ° C.
3. Priming e ativar o dispositivo
- As válvulas na camada de controle são comandados a partir do computador utilizando o LabVIEW. O script LabVIEW controla uma série de válvulas de solenóide através de um micro caixa de comando electrónica (adquirido a partir de Stanford Microfluidics Foundry). O bloco de válvulas solenóide está ligado a ar comprimido, os quais controlam o fluxo de ar e pressão em que as válvulas de controlo do dispositivo.
- Conectar o dispositivo ao colector de válvulas de solenóide através de um tubo de plástico flexível com um diâmetro interno de 0,02 "(Tygon) E pino de aço inoxidável (New England pequenos tubos Corporation).
- Encher os tubos com DDW e inserir o pino nos furos de acesso da camada de controlo. Certifique-se de que cada um dos tubos está ligado ao seu canal de controlo correspondente.
- Execute o aplicativo LabVIEW no computador para ativar os canais de controle. Através da activação da válvula a partir do script LabVIEW, que aplica a pressão do ar, o que por sua vez, vai empurrar a água para os canais de controlo de PDMS. Definir a pressão de ar de 5 psi. Recomenda-se para encher o 'sandwich' e as válvulas de endereço em primeiro lugar, a fim de identificar conversas cruzadas entre os canais de controlo de dispositivos defeituosos. Em caso de conversas cruzadas, atuação da linha de controle "sanduíche" levará à atuação do pescoço ou linhas de controle de botão. Isto é semelhante a um curto-circuito eléctrico. Um dispositivo com a diafonia é defeituoso e não pode ser utilizado.
- Depois de todos os canais de controlo e as válvulas estão cheios de DDW, as válvulas são preparadas e ready para bloquear os canais de fluxo abaixo delas. Aumentar a pressão de ar de 15 psi. Ativação da válvula é controlado a partir de um programa LabVIEW através de um conjunto de chaves liga / desliga conectados a válvulas solenóides individuais. Cada switch controla uma linha de controle específico através da sua válvula solenóide correspondente. Ligando o botão do interruptor (no script) será aplicada pressão de ar no tubo correspondente. A pressão do ar vai empurrar o DDW na linha de comando e resultará na expansão das válvulas microfluídicos, bloqueio do canal por baixo. Desligar uma válvula de controlo, irá libertar a pressão do ar e, posteriormente, libertar o bloqueio do canal de fluxo respectivo.
- Para assegurar que todas as válvulas estão abertas, ligar um tubo a uma das entradas de fluxo de ar e fluxo para dentro do dispositivo em psi 4-5. Isso vai liberar quaisquer válvulas pegajosas da lâmina de vidro.
- Finalmente, o dispositivo está preparado e pronto. Feche todas as entradas e 'pescoço' válvulas, para iniciar a química de superfície.
- De forma a facilitar a auto-montagem de uma matriz de proteína sobre a superfície e impedir a adsorção não específica no interior do dispositivo microfluidico, a superfície é modificada quimicamente:
- A fluir através de um componente do dispositivo, ligar um novo tubo com a solução necessária para um dos canais de escoamento do dispositivo. Ligue o lado livre do tubo, para o colector de manual e abrir o fluxo de pressão de ar (5 psi).
- Carregar 40 ul de biotinilado-BSA (1 ug / uL) em um novo tubo e fluxo aproximadamente metade dela durante 20 min através do dispositivo, o BSA irá ligar à superfície de epóxi.
- Use Hepes (50 mM), para lavar o substrato que não reagiu, entre cada uma das etapas da química de superfície diferentes.
- Estrepavidina fluxo de 25 ul (0,5 ug / uL) durante 20 minutos, no topo do biotinilado-BSA.
- Lavar com Hepes, durante 5 minutos.
- Feche o "botão" da válvula e fluir o resto do biotinilada-BSA (como descritocama, supra), a passivação da superfície circundante do botão.
- Lavar com Hepes, durante 5 minutos.
- Libertar o "botão" da válvula e do fluxo de 30 ul de penta-His-biotina (0,16 ug / uL) durante 20 minutos. O anticorpo irá ligar-se a estreptavidina o exposto, especificamente para a área sob o "botão" criação de uma matriz anti-His.
5. Protein Expression
- Expressar as proteínas no dispositivo usando coelho transcrição rápida de reticulócitos acoplados e reação de tradução. A expressão da proteína a partir dos genes sintéticos manchado no dispositivo cria uma matriz de proteínas prontas para usar. Por exemplo, uma tal utilização é para uma tela de ligação às proteínas.
- Abra o 'pescoço' válvulas e fluxo de reticulócito de coelho transcrição rápida acoplados e reação de tradução através do dispositivo para a câmara de DNA. Em seguida, fechar as válvulas "sanduíche" e separar cada gene de seu ambiente. Incubar o dispositivo sobre uma placa de aquecimento durante 2,5 horas a 30 ° C. Expresso proteins vai difundir a partir da câmara de DNA para a câmara de reacção espontânea (pescoço de válvula é aberta) e se ligam ao anticorpo anti-His sob o "botão" válvula de imobilização da proteína através da sua extremidade C-terminal tag.
- Rotular as proteínas com C-myc anticorpo Cy3. O anticorpo irá ligar ao seu epítopo correspondente localizado na extremidade N-terminal da proteína.
- Determinar os níveis de expressão de proteínas com um scanner de microarray (LS Reloaded, Tecan) usando um laser de 532 nm e 575 nm de emissão de filtro.
6. Resultados representativos
1. Fabricação de dispositivos
Um projeto gráfico para o dispositivo foi criado pelo AutoCAD com base em nossas necessidades experimentais. O projeto foi impressa em um filme de transparência por um setter imagem de alta resolução. Esta transparência serve como um fotomáscara em fotolitografia contato. Uma técnica microusinagem de superfície foi usado para criar modelos de 3-D de uma pastilha de silício determinado pelos padrões inscritos no tele máscaras utilizadas.
O dispositivo de microfluidos foi fabricado em silicone do molde de fundição de elastómero de silicone de polidimetilsiloxano (PDMS, SYLGARD 184, Dow Corning, EUA) (Figura 1). Cada dispositivo é constituído por duas camadas de PDMS alinhados, o fluxo e a camada de controlo. O molde foi primeiramente exposta a clorotrimetilsilano (TMCS, Aldrich) de vapor durante 10 minutos para promover a libertação de elastómero após os passos de cozimento. Uma mistura de elastómero de silicone com base e agente de cura foi preparado em duas proporções diferentes de 5:1 e 20:1 para o controle de fluxo e de moldes, respectivamente. A camada de controlo foi desgaseificada e cozido durante 30 min a 80 ° C. A camada de fluxo foi inicialmente revestido girar (Laurell, EUA) a 2600 rpm durante 60 segundos e aquecidas a 80 ° C durante 30 min. A camada de controle foi separado de seu molde e furos de controle de acesso de canal foram perfurados. Em seguida, as camadas de escoamento e de controlo foram alinhadas manualmente sob um estereoscópio e cozidos durante 2 horas a 80 ° C. O dispositivo de duas camadas foram descascadas from o molde de fluxo e de fluxo de canais de acesso buracos foram perfurados.
2. Descrição do Dispositivo
A capacidade do dispositivo pode variar desde 500 até 10.000 células unitárias (Figura 2). O dispositivo consiste em duas camadas; camada de escoamento (cinzento) e controlo de camada (cor). A camada de controlo contêm uma variedade de válvulas com diferentes funções. Cada célula unitária, na camada de fluxo, é composto de um DNA e uma câmara de reacção e é controlado por três tipos de válvulas de micromecânica; 'pescoço', 'botão', e 'sandwich' (figura 2A). "Gene sintético» A spotted depositado no interior da câmara de DNA é bloqueada a partir da câmara de reacção através do "pescoço" da válvula (verde). Lavagem de captura e mecânico de proteínas de superfície ligados na câmara de reacção é efectuada pelo "botão" da válvula (azul). A "sanduíche" de válvula permite que cada reacção ocorra na sua própria unidade de célula (vermelho). As válvulas de endereço pode dividir o dispositivo até 8 seções independentes, por diferent ensaio condição. Além disso, a camada de controlo contêm válvulas de entrada que permitem que o fluxo de fluido escolhido para a camada de fluxo. Os canais de controle de PDMS são cheios com ADD. Quando a válvula é accionado o aumento de pressão (15 psi) resulta na expansão do PDMS. Em locais onde a membrana é fina o suficiente (ou seja, cruzando de controle e linhas de fluxo), este é suficiente para bloquear completamente a linha de fluxo. Altura da célula unitário médio é de 10 um, e volume médio das células da unidade é menor do que 1 nl.
3. , Expressão de proteínas e Detecção
As proteínas foram expressas no dispositivo utilizando coelho transcrição rápida de reticulócitos acoplados e reacção de tradução (Promega). A expressão das proteínas criado um array de proteínas prontas para uso em uma tela de ligação (Figura 3). Uma mistura de expressão (12,5 uL) foi carregado para o dispositivo e, em seguida, inundado para as câmaras de DNA, abrindo as válvulas de "pescoço". Em seguida, os "sanduíche" válvulas foramfechado deixando cada célula unitária separada das suas células vizinhas e do dispositivo foi incubada numa placa de aquecimento durante 2,5 horas a 30 ° C. Proteínas expressas difundem através da câmara de gene para dentro da câmara de reacção, em que a sua extremidade C-terminal tag Sua podia ligar o anticorpo anti-His (Qiagen) localizada sob o "botão" válvula de imobilização da proteína para a superfície. As proteínas foram marcadas com um C-myc Cy3 (Sigma), que se ligou ao seu epítopo correspondente localizado na extremidade N-terminal da proteína e rotularam. Níveis de expressão de proteína foram determinadas com um scanner de microarray (LS Reloaded, Tecan) usando um laser de 532 nm e 575 nm de filtro. A matriz de proteína resultante consiste em vários níveis de expressão da proteína. Normalmente, cerca de 20% de uma biblioteca de genes não conseguem expressar a níveis detectáveis. Nenhuma correlação com o tamanho da proteína foi observada 3. Níveis de fundo foram determinados utilizando câmaras que não foram manchadas com o ADN. Assim, os sinais das câmaras de proteína correspondentes devem-se a ser noise ou adsorção não específica dos anticorpos de rotulagem.
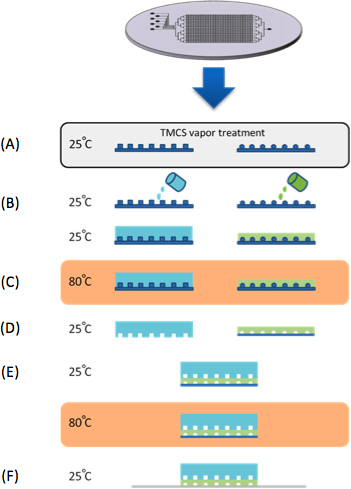
Figura 1. Fabrication chip. (A) foram tratados com moldes vapores TMCS por 10 min. (B) PDMS é fundido no controle e fluxo de moldes de silicone. (C) Tanto o controle de fluxo e de moldes são cozidas em 80 ° C durante 30 min. (D) A camada de controlo do molde desenrolada, cortada ao tamanho e as entradas de controlo são perfuradas. (E) A camada de controle é alinhada com a camada de fluxo sob um estereoscópio e em seguida cozido em 80 ° C durante 2 horas. Em paralelo com a fabricação de dispositivos PDMS, uma série de genes sintéticos são microarrayed sobre substratos de vidro revestidos com epóxi (CEL Associates). (F) O dispositivo é então alinhado com o DNA microarray prendendo os "genes sintéticos" dentro das câmaras de DNA.

Figura 2. Uma foto do Dispositivo PING. (A) O dispositivo consiSão de duas camadas alinhados PDMS (controle e fluxo). A camada de escoamento contém câmaras paralelas de ADN e de reacção (a cinzento) controladas por válvulas de micromecânica ("botão", "sandwich" e "pescoço") na camada de controlo. (B) As camadas são alinhados a um microarray de impresso, que cada câmara de programas de DNA com uma única mancha. Isso elimina qualquer contaminação potencial.

Figura 3. Imagens de fluorescência de uma proteína de matriz criada com um chip microfluídico. Os genes de impressos foram expressos para proteínas dentro do dispositivo (na câmara de DNA) e foram puxadas para baixo para a superfície (abaixo do "botão" de válvula na câmara de reacção) através da sua C-terminal tag dele. A expressão proteica foi detectada utilizando o seu C-myc N-terminal tag e um anticorpo específico de fluorescência. Intensidades de sinal diferentes indicam diferentes níveis de expressão da proteína. Un-manchado câmaras serviram como controle para o fundo leVels.
Discussão
Neste artigo, apresentamos um método para matrizes de geração de proteínas de alto rendimento, utilizando uma plataforma microfluídica. A geração da matriz é baseada em microarray de impressão de moldes de ADN in vitro e a expressão de proteína a partir do ADN no interior do dispositivo microfluidico.
Nossa plataforma microfluídica romance tem várias vantagens importantes sobre os métodos usados atualmente, que o tornam uma ferramenta promissora e geral para prot...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado por Marie Curie internacional reintegração subvenção.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagente / Equipamento | Companhia | Número de catálogo | |
| PDMS-184 SYLGARD | Dow Corning EUA | ESSEX-DC | |
| Clorotrimetilsilano (TMCS | Sigma-Aldrich | C72854 | |
| Epóxi substratos de vidro revestidos | CEL Associates EUA | VEPO-25C | |
| Poli etileno glycole (PEG) | Sigma-Aldrich | 81260 | |
| D-trealose di-hidratada | Sigma-Aldrich | T9531 | |
| Biotinilado-BSA | Perfurar | PIR-29130 | |
| Neutravidina | Perfurar | 31050 | |
| penta-His-biotina | Qiagen | 34440 | |
| Hepes | Biological Industries | 03-025 1B | |
| TNT-T7 | Promega | L5540 | |
| C-myc anticorpo Cy3 | Sigma-Aldrich | ||
| Caixa de controle | Stanford microfluídica Fundição | ||
| Molde | Stanford microfluídica Fundição | ||
| Pin | New England pequenos tubos Corporação | ||
| Tygon tubulação microbore | Tygon | S-54-HL | |
| Microarrayer | Bio Robótica | MicroGrid 610 | |
| Pinos de silicone | Paralelo Syntese | SMT-S75 |
Referências
- Maerkl, S. J. Integration column: Microfluidic high-throughput screening. Integrative biology quantitative biosciences from nano to macro. 1, 19-29 (2009).
- Hong, J. W., Quake, S. R. Integrated nanoliter systems. Nature. 21, 1179-1183 (2003).
- Unger, M. A Monolithic Microfabricated Valves and Pumps by Multilayer Soft Lithography. Science. 288, 113-116 (2000).
- Gerber, D., Maerkl, S. J., Quake, S. R. An in vitro microfluidic approach to generating protein-interaction networks. Nature. 6, 71-74 (2009).
- Einav, S. Discovery of a hepatitis C target and its pharmacological inhibitors by microfluidic affinity analysis. Nature. 26, 1019-1027 (2008).
- Fordyce, P. M. De novo identification and biophysical characterization of transcription-factor binding sites with microfluidic affinity analysis. Nature Biotechnology. 28, 962-967 (2010).
- Zhu, H. Global analysis of protein activities using proteome chips. Science (New York, N.Y.). 293, 2101-2105 (2001).
- Ramachandran, N. Self-assembling protein microarrays. Science (New York, N.Y.). 305, 86-90 (2004).
- Zhong, J. F. A microfluidic processor for gene expression profiling of single human embryonic stem cells. Lab on a chip. 8, 68-74 (2008).
- Kusnezow, W., Hoheisel, J. D. Solid supports for microarray immunoassays. Journal of molecular recognition JMR. 16, 165-176 (2003).
- Lundin, M., Monne, M., Widell, A., Von Heijne, G., Persson, M. A. A. Topology of the membrane-associated hepatitis C virus protein NS4B. Journal of virology. 77, 5428 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados