Method Article
Isolamento de cromatina por purificação de RNA (CHIRP)
Neste Artigo
Resumo
CHIRP é uma técnica recente e rápida de mapa genômico sítios de ligação de RNAs não-codificantes longos (lncRNAs). O método tira partido da especificidade dos oligonucleótidos anti-sentido ladrilhos para permitir que a enumeração de sítios lncRNA-bound genómicos.
Resumo
RNAs não-codificantes longos são os reguladores de cromatina principais estados para processos biológicos importantes, como a compensação de dosagem, imprinting e expressão de genes de desenvolvimento 1,2,3,4,5,6,7. A recente descoberta de milhares de lncRNAs em associação com os complexos de cromatina específicos de modificação, tal como o complexo Repressiva Polycomb 2 (PRC2) que medeia a histona H3 lisina 27 trimethylation (H3K27me3), sugere as funções gerais para lncRNAs numerosos estados na gestão de cromatina em um gene específico moda 8,9. Enquanto alguns lncRNAs são pensados para trabalhar em cis em genes vizinhos, lncRNAs outros trabalhar em trans para regular genes localizados distantes. Por exemplo, Drosophila lncRNAs roX1 e roX2 regiões ligam numerosos no cromossomo X de células masculinas, e são críticas para a compensação de dosagem 10,11. No entanto, as localizações exatas dos seus locais de ligação não são conhecidos em alta resolução. Da mesma forma, humana lncRNA hotair pode afectar PRC2 ocupação em hundreds de genes do genoma 3,12,13, mas como especificidade o objectivo é claro. LncRNAs também pode servir como suportes modulares para recrutar o conjunto de complexos de proteínas múltiplas. O clássico trans-acting RNA andaime é o RNA TERC que serve como o modelo e andaime para a telomerase complexo 14; hotair também pode servir como um andaime para PRC2 e um desmetilase H3K4 complexo 13.
Estudos anteriores de mapeamento de ocupação de RNA em cromatina revelaram percepções substanciais 15,16, mas apenas com um único locus do gene de cada vez. Os locais de ocupação da maioria dos lncRNAs não são conhecidos, e os papéis de lncRNAs na regulação da cromatina foram principalmente inferida a partir dos efeitos indiretos da perturbação lncRNA. Assim como imunoprecipitação de cromatina seguido por microarray ou seqüenciamento profunda (CHIP CHIP ou CHIP-seq, respectivamente) tem melhorado muito a nossa compreensão de interações DNA-proteína em escala genômica, aqui nós ilustrar um Recenqüentemente publicado estratégia para mapear ocupação RNA longo de todo o genoma em alta resolução 17. Este método de isolamento, cromatina pela RNA Purificação (CHIRP) (Figura 1), baseia-se na captura de afinidade de alvo lncRNA: Complexo de cromatina por ladrilhos anti-sentido-oligos, que gera então um mapa de sítios de ligação genómicos com uma resolução de várias centenas de bases com sensibilidade elevada e baixa do fundo. CHIRP é aplicável a muitos lncRNAs porque o desenho de afinidade sondas é simples, dada a sequência de RNA e não requer conhecimento da estrutura do RNA ou domínios funcionais.
Protocolo
1. Projeto Sonda
Design DNA anti-senso de lado a lado sondas para a recuperação seletiva de RNA alvo por piar.
- Projeto sondas anti-senso oligo usando o designer sonda on-line em singlemoleculefish.com 18.
- Use esses parâmetros: número de sondas = 1 / sonda de 100 pb de comprimento RNA, 2) Alvo% GC = 45, comprimento Espaçamento 4) = 60-80; 3) Comprimento Oligonucleotide = 20. Quebre RNA em segmentos se muito tempo para o designer. Omitir regiões de repetições ou homologia extensa.
- Ordem sondas anti-senso de DNA com BiotinTEG em 3-primeiro-final.
- Sondas etiqueta de acordo com as suas posições ao longo do RNA. Separá-los em dois grupos para que a piscina ", mesmo" contém todas as sondas de numeração 2, 4, 6, etc, e na piscina "estranho" contém sondas de numeração 1, 3, 5, etc Diluir pool de sondas para concentração de 100 mM e armazenar a -20 ° C.
- Todas as experiências são para ser feita usandoambas as caixas, que servem como controles internos para o outro. Sinal dependente de ARN real estaria presente em ambos os pools, enquanto sonda específicas ruídos seria único para cada pool. Isto aplica-se tanto para CHIRP-qPCR e CHIRP-seq.
2. Células Colheita
Recolher células que serão usados para a experiência CHIRP.
- Crescer as células em placas de cultura de tecidos ou frascos até à confluência. Lavar com salina tamponada com fosfato (PBS) e uma vez trypsinize. Têmpera de tripsina com> volume 2x de mídia, pipeta cima e para baixo para retirar as células e ressuspender em suspensão de células individuais. Transferir todos os meios e as células ressuspensas em tubos de 50 ml Falcon. 20 milhões de células são tipicamente suficiente para uma amostra CHIRP.
- Girar em células 800RCF durante 4 min. Meios aspirado e ressuspender 40 milhões de células em 40 ml de PBS, combinar os tubos, se necessário. Girar em células 800RCF durante 4 min. Decantar PBS, aspirar cuidadosamente sobre um ângulo que o líquido restante.
3. Cross-link células e recolher pellet celular
Crosslink coletadas células com glutaraldeído para preservar RNA-cromatina interações e preparar pellet celular.
- Execute todas as etapas à temperatura ambiente.
- Prepare glutaraldeído 1% em PBS à temperatura ambiente. Preparar 10 ml por 10 milhões de células (0,4 ml de caldo de glutaraldeído a 25% + 9,6 mL de PBS). O glutaraldeído deve ser utilizado fresco.
- Toque fundo de tubos Falcon de desalojar pelotas. Ressuspender o sedimento de células em glutaraldeído a 1%, começando com um pequeno volume para evitar pedaços, em seguida, o início até ao volume total. Inverta para misturar. Reticulação durante 10 min à temperatura ambiente num agitador de ponta a ponta ou rotador.
- Extingue-se a reacção de reticulação com um volume de 1/10th de 1,25 M de glicina, à temperatura ambiente durante 5 min.
- Girar em 2000RCF durante 5 min. Aspirar o sobrenadante e lava-pelete com 20 mL de PBS gelado uma vez, girando a 2000RCF durante 5 min.
- Aspirar e ressuspender o wincinerada, reticulada pelete com 1 ml de PBS, refrigerada por 20 milhões de células. Transfira cada ml a um tubo Eppendorf e giram a 2000RCF durante 3 min a 4 ° C. Remover PBS, tanto quanto possível, com a ponta da pipeta com cuidado.
- Flash-congelar os peletes de células em azoto líquido e armazenada a -80 ° C indefinidamente.
4. Lise Celular
Lisar células reticulados para preparar lisado celular.
- Descongelar sedimentos celulares congelados à temperatura ambiente. Toque difícil desalojar e misturar o pellet celular. Girar para baixo o sedimento em 2000RCF durante 3 min a 4 ° C. Use uma forte ponta da pipeta 10 ul para remover qualquer PBS restante.
- Em uma balança eletrônica (precisão de 1 mg) tara de um tubo de Eppendorf vazio (os nossos tubos de pesar 1.060 gramas muito consistente). Pese cada pellet e gravar o seu peso. Um prato centímetros completa 15 de células HeLa reticulados tipicamente pesa 100 mg.
- Tampão de Lise suplemento (massa 10X de pellet, por exemplo, 1 ml para 100 mg) com fresh inibidor da protease, PMSF e Superase-in (veja a lista de tampão em anexo). Misture bem.
- Adicionar 10X volume de Tampão de Lise completado, para cada tubo e ressuspender o sedimento. Para as pelotas pequenas <25 mg, ressuspender em 250 ul de tampão de lise suplementado. Suspensão deve ser suave. Se não, dividir suspensão em 500 uL alíquotas e usar uma motorizada pelete misturador para quebrar grumos. Proceder de imediato à sonicação.
5. Sonicação
DNA de cisalhamento por sonicando lisados de células reticuladas.
- Sonicar lisado celular em Bioruptor em 15 tubos Falcon ml. Use <1,5 lisado ml em cada tubo, e para mais rapidamente sonicação, sonicar não mais do que dois tubos de cada vez.
- Sonifique em um banho de água 4 ° C a mais alta definição com 30 segundos, 45 segundos OFF intervalos de pulso. Verifique lisado cada 30 min. Continuar sonicando até que o ligado celular não é mais turvo. Isso pode demorar tão pouco como 30 minutos e até 4 horas. O númerode tubos, o volume da amostra, a temperatura do banho, eo período de tempo de sonicação irá afectar o tempo que o processo leva. Tubos provavelmente sonicar em taxas diferentes, de modo juntam-los em conjunto a cada 30 min e redistribuir em tubos originais para assegurar a homogeneidade. Nota: o glutaraldeído-reticulado células demorar muito mais para sonicate do que seus equivalentes de formaldeído.
- Quando lisado transforma claro, transferir 5 uL lisado para um novo tubo Eppendorf. Adicione 90 uL de DNA de proteases K (PK) Buffer (ver lista buffer) e 5 PK uL. Vortex para misturar e girar rapidamente. Incubar durante 45 min a 50 ° C.
- Extrair DNA com Qiagen kit de purificação PCR. Eluir o DNA em 30 uL Tampão eluição Qiagen (EB) e verificar o tamanho do DNA em 1% gel de agarose. Se grandes quantidades de DNA é o esfregaço 100-500 pb, sonicação está completa. Se não, continuar a sonicate.
- Centrifugue as amostras sonicadas em 16100RCF durante 10 min a 4 ° C. Combine sobrenadantes, alíquotas em amostras de 1 ml e congelar-in Flash nitroge líquidon. Armazenar a -80 ° C.
6. CHIRP
Hibridizar sondas de DNA biotiniladas para o RNA e isolar cromatina ligada.
- Tubos de descongelamento de cromatina à temperatura ambiente.
- Preparar o tampão de hibridação (ver lista de buffer, prepare 2 ml por ml de cromatina). Vórtice para misturar.
- Para uma amostra CHIRP típico usando 1 ml de lisado, remover 10 uL para RNA INPUT e 10 uL de ADN para INPUT e local em tubos de Eppendorf. Manter em gelo até posterior utilização.
- Transferir 1 cromatina mL a 15 mL tubo Falcon. Adicionar 2 mL Tampão de Hibridização a cada tubo. Para o volume total <1,5 ml, utilizar tubos Eppendorf.
- Descongele sondas à temperatura ambiente. Nanodrop sondas para verificar quantidade se você não tê-lo usado em um longo tempo (100 sondas iM deve especificação ~ 500-600 ng / ul usando configuração DNA de cadeia simples). Adicionar volume apropriado de sondas específicas para os tubos (100 pmol por sonda 1 mL da cromatina, 1 uL de 100 sonda pmol / uL por 1 mL de cromatina).Misture bem. Incubar a 37 ° C durante 4 horas com agitação.
- Com 20 min restante para a hibridação, preparar os grânulos C-1 magnéticos (armazenada a 4 ° C). Use de 100 L por 100 pmol de sondas. Lava-se com 1 mL Tampão de Lise não suplementado três vezes, utilizando a tira de íman DynaMag-2 a esferas separadas a partir de tampão.
- Contas Ressuspender em volume original de tampão de lise e Suplemento com PMSF fresco, PI e Superase-in. Após 4 h de reacção de hibridação está completa, adicionar 100 grânulos uL a cada tubo. Misture bem. Incubar a 37 ° C durante 30 min com agitação.
- Preparar tampão de lavagem (5 mL por amostra). Vórtice para misturar. Pré-aquecido a 37 ° C. Adicionar PMSF antes da utilização.
- Lavar grânulos com 1 mL de tampão de lavagem por cinco vezes. Na primeira lavagem, utilizar-15 DynaMag tira magnética a esferas separadas, decantar, e ressuspender em 1 mL de tampão de lavagem. Transferir volume para tubo Eppendorf de 1,5 mL. Incubar a 37 ° C com agitação durante 5 min.
- Em lavagens subseqüentes, girar cada tubo em um minicentrifuge, Definir amostra em DynaMag-2 tira magnética durante 1 min. Decantar amostra, seque os pingos com um Kimwipe, ressuspender em 1 ml de tampão de lavagem. Incubar a 37 ° C com agitação durante 5 min. Repetir para cinco lavagens totais.
- Na última lavagem, ressuspender as esferas bem. Retirar 100 uL e reserve para o isolamento de RNA. Reserve uL 900 para fração de DNA. Coloque todos os tubos em DynaMag-2 fita magnética e remover o tampão de lavagem. Gire todos os tubos para baixo brevemente; colocá-los na faixa ímã. Retire o último pedaço de tampão de lavagem completamente com uma afiada ponta de pipeta 10 ul.
7. RNA Isolation
Extrair fracção de ARN a partir de amostras Chirp para quantificar por qRT-PCR.
- Tome 100 l amostras de contas e 10 amostra de entrada uL RNA. Adicionam-se 85 uL RNA tampão pH 7,0 PK para o RNA INPUT. Contas Ressuspender em 95 uL de RNA PK pH 7,0. Adicionar 5 K Proteinease uL e incubar a 50 ° C durante 45 min com ponta a ponta de agitação.
- Resumidamente girar todos os tubos eferver amostras durante 10 min em bloco de aquecimento a 95 ° C.
- Arrefeça amostras em gelo, adicionar 500 uL TRIzol vórtice, vigorosamente, durante 10 seg. Incubar a temperatura ambiente durante 10 min. Conservar a -80 ° C ou vá para a etapa 4.
- Adicionar 100 uL de clorofórmio para TRIzol amostras tratadas. Vortex vigorosamente durante 10 seg. Girar em 16100RCF numa centrífuga de bancada durante 15 min a 4 ° C.
- Remover ~ 400 uL sobrenadante aquoso, evitando orgânica e de interface.
- Adicionar 600 uL (1,5 de volume) de etanol a 100% e misturar bem. Centrifugue a amostra através MIRNeasy colunas mini. Lave 1x com ERP (MIRNeasy mini-kit), 2x por RPE com o protocolo do fabricante. Eluir com 30 uL sem nuclease H2O (NFH 2 O).
- Tratar o produto de eluição com a DNA-RNA livre por protocolo do fabricante. Após a reacção está completa, aquecer a amostra durante 15 minutos a 65 ° C para inactivar qualquer completamente DNase restante.
- Use 1 RNA isolado por microlitro bem para qRT-PCR para confirmar lncRNArecuperação. GAPDH é frequentemente utilizado como um controlo negativo.
8. Isolamento de DNA
Extrair DNA de amostras de fração Chirp para identificar por seqüenciamento ou quantificar por qPCR.
- Preparar o tampão de eluição DNA (ver lista de buffer), 150 ul por amostra, incluindo entrada DNA.
- Adicionar 1 0μL RNase A (10 mg / mL) e 10 uL de RNase H (10 U / ul) por ml de tampão de eluição de DNA, e vórtice para misturar.
- Ressuspender cada amostra de grânulos em 150 uL de Tampão de Eluição com DNA RNases. (Ressuspender ENTRADA DNA em 140 uL) Incubar a 37 ° C durante 30 min com agitação.
- Contas separadas e sobrenadante em DynaMag-2 tira magnética. Remover o sobrenadante e adicionar aos tubos etiquetados.
- Prepara-se uma segunda alíquota de tampão de eluição de ADN com 10 uL de RNase A (10 mg / mL) e RNaseH (10 U / mL) exactamente como é feito em 8,2). Adicionar 150 uL de cada amostra (incluindo entrada do ADN), incubar e remover o sobrenadante. Colete todos sobrenadante (should ser ~ uL 300).
- Adicionar 15 PK uL de cada amostra. Incubar a 50 ° C durante 45 min com agitação.
- Pré-girar amarelo tubos fase de bloqueio de gel (5PRIME). Transferir as amostras de DNA a tubos de bloqueio de fase de gel, e adicionam-se 300 uL PhOH: Clorofórmio: isoamilo por amostra. Agita-se vigorosamente durante 10 min, e girar para baixo em uma centrífuga de bancada a 16100RCF durante 5 min a 4 ° C. Tome aquosa a partir do topo (~ uL 300). Adicionar 3 GlycoBlue uL, 30 NaOAc uL, e 900 uL de EtOH a 100%. Misture bem e armazenar a -20 ° C durante a noite.
- Girar amostras a 16100RCF durante 30 min a 4 ° C.
- Decante o sobrenadante cuidadosamente. Adicionar 1 mL de EtOH a 70% e vórtice para misturar. Girar em 16100RCF durante 5 min. Remover o sobrenadante com uma pipeta. Deixe secar por 1min. Ressuspender em 30 uL EB.
- Amostras de DNA está pronto para análise por qPCR ou preparação de bibliotecas de alto rendimento de sequenciamento por protocolo Illumina.
10. Os resultados representativos
A Figura 1 descreve o fluxo de trabalho piar. Uma experiência CHIRP bem sucedida tipicamente enriquece RNA alvo significativamente ao longo do não-específicos RNAs. A Figura 2 mostra o enriquecimento de humano telomerase RNA (TERC) a partir de células HeLa sobre GAPDH, um RNA abundantes celulares que serve como um controlo negativo. Maioria dos RNAs TERC (~ 88%) presentes na célula foram puxados para baixo através da realização de CHIRP, enquanto que apenas 0,46% de GAPDH RNA foi recuperado, demonstrando um factor de enriquecimento de ~ 200 vezes. Sondas inespecíficas, tais como sondas de segmentação LacZ RNA, que não se expressa em células de mamíferos (Figura 2), pode ser usado como adicionais controlos negativos.
Regiões de ADN que se espera que se ligam a lncRNA alvo são tipicamente enriquecida ao longo de regiões negativas, quando medido por qPCR. A Figura 3 mostra qPCR validação de quatro hotair ligados a sítios em fibroblastos de prepúcio humanos primários que determinados através da realização de CHIRP-seq na mesma linha celular, enquanto TERC e GAPDH DNA sítios siRVE como regiões de controlo negativo. Tanto o "mesmo" ea sonda "estranha" define rendeu enriquecimento comparável de espera hotair ligados sites sobre regiões negativas, marca registrada dos verdadeiros lncRNA sítios de ligação.
Alta capacidade de seqüenciamento de DNA CHIRP enriquecido produz um mapa global de lncRNA sítios de ligação. A Drosophila lncRNA roX2 é conhecido para interagir com o cromossoma X de uma maneira que é necessária para a compensação de dosagem. A Figura 4 mostra roX2 perfil de ligação ao longo de um secção do cromossoma X. Tanto o "mesmo" e "estranha" as amostras já foram seqüenciados e seus ruídos originais foram eliminados para produzir uma faixa de sinais que se sobrepõem. Cada "pico" aqui indica um site de forte roX2 vinculativo. A faixa completa ea lista de roX2 genes alvo têm sido descritas na Chu et ai. 2011 17.
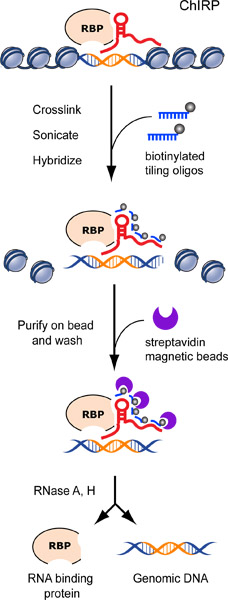
Figura Fluxograma 1. Do procedi CHIRPDure. Cromatina é reticulado para lncRNA: aductos de proteína in vivo biotinilados sondas azulejos são hibridizados para alvejar lncRNA, e os complexos de cromatina são purificados utilizando esferas de estreptavidina magnéticos, seguido por lavagens rigorosas.. Nós eluir lncRNA DNA ligado ou proteínas com um coquetel de RNase A e H. A seqüência lncRNA suposta ligação está esquematizado na laranja. Anteriormente publicado em Chu et ai. 2011. 17
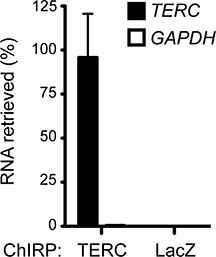
Enriquece a Figura 2. Chirp para o homem TERC RNA. TERC-asDNA sondas recuperar ~ 88% de RNA celular TERC e GAPDH indetectável. LacZ-asDNA sondas são utilizados como controles negativos e recuperar nem RNAs. A média + dp são mostrados. Anteriormente publicado em Chu et ai. 2011. 17
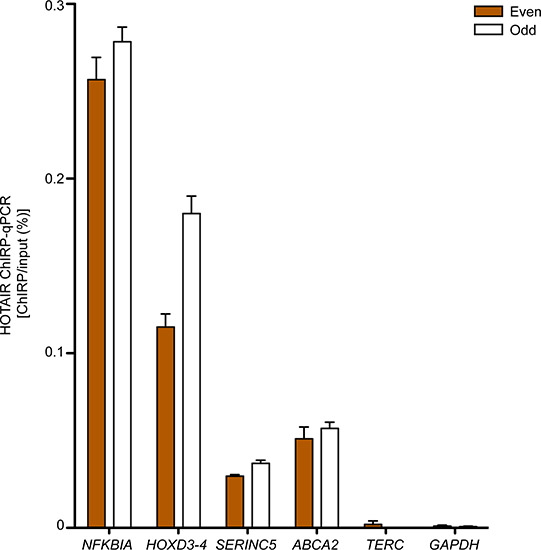
Figura 3. Hotair CHIRP-qPCR em humano primário parafibroblastos Eskin. NFKBIA, HOXD3-4, SERINC5 e ABCA2 são regiões que interagem com hotair. TERC e GAPDH serviram como controles negativos. A média + dp são mostrados. Anteriormente publicado em Chu et ai. 2011. 17
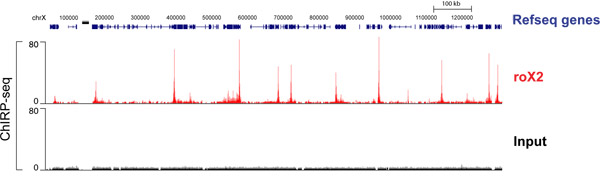
Figura 4. CHIRP-seq de dados de roX2 RNA em células de Drosophila SL2. "Mesmo" e "estranho" foram seqüenciados em separado; seus dados mesclar para refletir apenas picos comuns em ambos. A faixa fundida é mostrado. Anteriormente publicado em Chu et ai. 2011. 17
Discussão
Aqui descrevemos CHIRP-seq, um método de mapeamento em sites in vivo lncRNA ligação do genoma. Os principais parâmetros para o sucesso são as piscinas de divisão de azulejos sondas de oligonucleotídeos e reticulação glutaraldeído. A concepção de sondas de afinidade é simples, dada a sequência de RNA e não requer conhecimento prévio da estrutura do RNA ou domínios funcionais. O nosso sucesso com roX2, TERC, e hotair - três RNAs bastante diferentes em duas espécies - sugere que CHIRP-seq provavelmente generalizáveis para lncRNAs muitos. Tal como com todas as experiências, os controlos de cuidados e adequada são necessários para interpretar os resultados. LncRNA diferente pode exigir titulação de condições, ea mudança criteriosa de condições, tais como seleção de sondas diferentes de afinidade ou reticuladores, pode realçar diferentes aspectos da RNA-cromatina interações. Como ChIP seguintes, nem todos os eventos de ligação são, necessariamente, funcional, e estudos adicionais são necessários para verificar as conseqüências biológicas da RNA occupancy em cromatina. No entanto, prevemos muitas aplicações interessantes desta tecnologia para os pesquisadores de outros cromatina associados lncRNAs, cujo número agora na casa dos milhares 8,9. Assim como ChIP-seq abriu a porta para a exploração do genoma de interações DNA-proteína, CHIRP-seq estudos do "RNA interactoma" pode revelar muitos novos caminhos da biologia.
Divulgações
C. Chu e Chang HY são nomeados como inventores sobre um pedido de patente com base neste método.
Agradecimentos
Agradecemos T. Hung, MC. Tsai, O. Manor, E. Segal, M. Kuroda, T. Swigut, e I. Shestopalov para as discussões. Apoiado pela Agência de Ciência, Tecnologia e Pesquisa de Cingapura (CC), NIH-R01 e R01 CA118750-HG004361 (HYC), e do Instituto de Medicina Regenerativa da Califórnia (HYC). HYC é um cientista de carreira precoce do Howard Hughes Medical Institute.
Materiais
| Name | Company | Catalog Number | Comments |
| Buffer List: | |||
| Dissolve a pellet of complete protease inhibitor in 1 ml water as 50x stock. Make 100 mM PMSF in isopropanol (100x stock). Superase-in is used as 200x stock. Store all at -20 °C. | |||
| Lysis Buffer: | |||
| 50 mM Tris-Cl pH 7.0 10 mM EDTA 1% SDS Always add PMSF, P.I. and Superase-in fresh before use except when washing beads | |||
| Proteinase K Buffer (for DNA) | |||
| 100 mM NaCl 10 mM TrisCl pH 8.0 (For RNA use pH 7.0) 1 mM EDTA 0.5% SDS Add 5% by volume Proteainse K (Ambion AM2546 20 mg/ml) fresh before use | |||
| Hybridization Buffer | |||
| 750 mM NaCl 1% SDS 50 mM Tris-Cl pH 7.0 1 mM EDTA 15% formamide (store in the dark at 4 °C) Always add PMSF, P.I. and Superase-in fresh before use | |||
| Wash Buffer | |||
| 2x NaCl and Sodium citrate (SSC) (diluted from 20x SSC Invitrogen stock) 0.5% SDS Always add PMSF fresh before use | |||
| DNA elution Buffer | |||
| 50 mM NaHCO3 1% SDS | |||
| Table of specific reagents and equipment: | |||
| Glutaraldehyde (EM grade) | Sigma-Aldrich | G5882-10x10ml | |
| Motorized pellet mixer | VWR international | V8185-904 | |
| Protease inhibitor | Roche Group | 11873580001 | |
| PMSF | Sigma-Aldrich | 78830 | |
| Superase-in | Ambion | AM2696 | |
| Bioruptor | Diagenode | UCD-200 | |
| Falcon tubes (for sonication) | Corning | 430790 | |
| Proteinase K | Ambion | AM2546 | |
| PCR purification kit | Qiagen | 28106 | |
| C-1 magnetic beads | Invitrogen | 65002 | |
| PMSF | Sigma-Aldrich | P7626-25G | |
| DynaMag-15 magnet | Invitrogen | 123-01D | |
| DynaMag-2 magnet | Invitrogen | 123-21D | |
| MIRNeasy mini kit | Qiagen | 217004 | |
| Rnase H | Epicentre Biotechnologies | R0601K | |
| Rnase A | Sigma-Aldrich | R4875-100MG | |
| Phase Lock Gel Heavy | 5 PRIME | 2302810 | |
| Trizol | Invitrogen | 15596-018 | |
| Phenol:chloroform:Isoamyl | Invitrogen | 15593-031 | |
| Chloroform | Ricca | RSOC0020-1C | |
| GlycoBlue | Ambion | AM9515 | |
| Glycine | JT Baker | 4057-06 | |
| PBS, pH 7.4 | Invitrogen | 10010-049 | |
| Elution Buffer (EB) | Qiagen | 19086 | |
| 20x SSC | Invitrogen | 15557-036 | |
| 10% SDS | Invitrogen | 15553-027 | |
| DNA-free | Ambion | AM1906 | |
| Buffer kit | Ambion | AM9010 | |
| Formamide | Invitrogen | 15515-026 | |
Referências
- Koziol, M. J., Rinn, J. L. RNA traffic control of chromatin complexes. Curr. Opin. Genet. Dev. 20, 142-148 (2010).
- Mercer, T. R., Dinger, M. E., Mattick, J. S. Long non-coding RNAs: insights into functions. Nat. Rev. Genet. 10, 155-159 (2009).
- Rinn, J. L. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 129, 1311-1323 (2007).
- Zhao, J., Sun, B. K., Erwin, J. A., Song, J. J., Lee, J. T. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science. 322, 750-756 (2008).
- Kelley, R. L. Epigenetic spreading of the Drosophila dosage compensation complex from roX RNA genes into flanking chromatin. Cell. 98, 513-522 (1999).
- Pandey, R. R. Kcnq1ot1 antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation. Mol. Cell. 32, 232-246 (2008).
- Wang, K. C. A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression. Nature. 472, 120-124 (2011).
- Khalil, A. M. Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression. Proc. Natl. Acad. Sci. U.S.A. 106, 11667-11672 (2009).
- Zhao, J. Genome-wide identification of polycomb-associated RNAs by RIP-seq. Mol. Cell. 40, 939-953 (2010).
- Meller, V. H., Wu, K. H., Roman, G., Kuroda, M. I., Davis, R. L. roX1 RNA paints the X chromosome of male Drosophila and is regulated by the dosage compensation system. Cell. 88, 445-457 (1997).
- Franke, A., Baker, B. S. The rox1 and rox2 RNAs are essential components of the compensasome, which mediates dosage compensation in Drosophila. Mol. Cell. 4, 117-122 (1999).
- Gupta, R. A. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature. 464, 1071-1076 (2010).
- Tsai, M. C. Long noncoding RNA as modular scaffold of histone modification complexes. Science. 329, 689-693 (2010).
- Zappulla, D. C., Cech, T. R. RNA as a flexible scaffold for proteins: yeast telomerase and beyond. Cold Spring Harb. Symp. Quant. Biol.. 71, 217-224 (2006).
- Nagano, T. The Air noncoding RNA epigenetically silences transcription by targeting G9a to chromatin. Science. 322, 1717-1720 (2008).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature. 32, 623-626 (2002).
- Chu, C., Qu, K., Zhong, F. L., Artandi, S. E., Chang, H. Y. Genomic Maps of Long Noncoding RNA Occupancy Reveal Principles of RNA-Chromatin Interactions. Mol. Cell. , (2011).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5, 877-879 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados