Method Article
Isolamento di cromatina Purificazione RNA (disturbo)
In questo articolo
Riepilogo
CHIRP è una tecnica innovativa e rapida mappa genomica siti di legame di RNA non codificanti lunghi (lncRNAs). Il metodo sfrutta la specificità di oligonucleotidi anti-senso di affiancamento per consentire l'enumerazione di lncRNA-bound siti genomici.
Abstract
RNA non codificanti lunghi sono regolatori chiave degli Stati cromatina per importanti processi biologici quali la compensazione del dosaggio, imprinting, e l'espressione genica di sviluppo 1,2,3,4,5,6,7. La recente scoperta di migliaia di lncRNAs in associazione con specifici complessi di modifica della cromatina, come Polycomb Complex repressiva 2 (PRC2) che media l'istone H3 lisina 27 trimethylation (H3K27me3), suggerisce i ruoli di massima per la gestione lncRNAs numerosi stati della cromatina in un gene specifico moda 8,9. Mentre alcuni lncRNAs sono pensati per lavorare in cis sui geni vicini, lncRNAs altri lavorano in trans per regolare i geni si trovano lontano. Per esempio, Drosophila lncRNAs roX1 e roX2 legano numerose regioni del cromosoma X delle cellule maschili, e sono fondamentali per la compensazione del dosaggio 10,11. Tuttavia, le posizioni esatte dei loro siti di legame non sono note ad alta risoluzione. Allo stesso modo, umano lncRNA Hotair può influire sulla occupazione PRC2 hundreds di geni genoma 3,12,13, ma come specificità si ottiene non è chiaro. LncRNAs può anche servire come strutture modulari di assumere l'assemblaggio di complessi proteici multipli. Il classico trans-agente RNA scaffold è il TERC RNA che serve come modello e scaffold per la telomerasi complesso 14; Hotair può anche servire come impalcatura per PRC2 e H3K4 demetilasi complesso 13.
Precedenti studi di mappatura occupazione RNA a cromatina hanno rivelato notevoli intuizioni 15,16, ma solo in un locus singolo gene alla volta. I siti di occupazione della maggior parte dei lncRNAs non sono noti, ei ruoli dei lncRNAs nella regolazione della cromatina sono stati in gran parte dedotta dagli effetti indiretti della perturbazione lncRNA. Proprio come immunoprecipitazione della cromatina seguita da microarray o sequenziamento profondo (ChIP-chip o ChIP-seq, rispettivamente) ha notevolmente migliorato la nostra comprensione di interazioni proteina-DNA su scala genomica, qui illustrare un RecenTLY pubblicato strategia per mappare lunga occupazione RNA genome-wide ad alta risoluzione 17. Questo, Isolamento metodo cromatina di purificazione dell'RNA (CHIRP) (Figura 1), si basa sulla cattura affinità di bersaglio lncRNA: complesso cromatina allineando-oligo antisenso, che genera un mappa genomica di siti di legame ad una risoluzione di diverse centinaia di basi con alta sensibilità e basso background. CHIRP è applicabile a molti lncRNAs perché il progetto di affinità sonde è semplice data la sequenza di RNA e non richiede alcuna conoscenza della struttura del RNA o domini funzionali.
Protocollo
1. Sonda di progettazione
Design anti-senso DNA piastrellatura sonde per il recupero selettivo di RNA bersaglio CHIRP.
- Progettazione anti-senso sonde oligo con il progettista in linea sonda singlemoleculefish.com 18.
- Utilizzare questi parametri: numero di sonde sonda = 1/100 bp di lunghezza RNA, 2) sulla destinazione GC = 45; 3) la durata oligonucleotide = 20; 4) Lunghezza distanza = 60-80. Rompere RNA in segmenti, se troppo a lungo per il progettista. Omettere regioni di ripetizioni o di una ampia omologia.
- Ordina anti-senso sonde di DNA con BiotinTEG a 3-prime.
- Sonde etichetta secondo le loro posizioni lungo l'RNA. Separarli in due gruppi in modo che il pool di "anche" contiene tutte le sonde di numerazione 2, 4, 6, ecc e la piscina "dispari" contiene sonde di numerazione 1, 3, 5, ecc Diluire pool di sonde a 100 micrometri e la concentrazione conservare a -20 ° C.
- Tutti gli esperimenti devono essere eseguite usandoentrambe le piscine, che servono come controlli interni per l'altro. Reale RNA-dipendente segnale sarebbe presente da entrambe le piscine, mentre la sonda specifici rumori sarebbe unico per ogni pool. Questo vale sia per CHIRP-qPCR e CHIRP-seq.
2. Raccogliere cellule
Raccogliere cellule che verranno utilizzati per l'esperimento CHIRP.
- Coltivare cellule in piastre di coltura di tessuto o di palloni confluenza. Risciacquo con tampone fosfato salino (PBS) e una volta Tripsinizzare. Quench tripsina con volume> 2x dei mezzi di comunicazione, pipetta su e giù per staccare le cellule e risospendere in sospensione singola cella. Trasferire tutti i media e le cellule risospese in 50 ml provette Falcon. 20 milioni di cellule sono in genere sufficienti per un campione CHIRP.
- Spin cellule a 800RCF per 4 min. Aspirare il terreno e risospendere 40 milioni di cellule in 40 ml di PBS, unire tubi se necessario. Spin cellule a 800RCF per 4 min. Decantare PBS, aspirare accuratamente in un angolo il liquido rimanente.
3. Cross-Link Cells e raccogliere pellet cellulare
Crosslink raccolte le cellule con glutaraldeide per preservare le interazioni RNA-cromatina e preparare pellet di cellule.
- Eseguire tutti i passaggi a temperatura ambiente.
- Preparare glutaraldeide 1% in PBS temperatura ambiente. Preparare 10 ml per 10 milioni di cellule (0,4 mL magazzino glutaraldeide 25% + 9,6 mL PBS). Glutaraldeide deve essere usato fresco.
- Toccare fondo di tubi Falcon per rimuovere pellets. Risospendere il pellet cellulare in 1% glutaraldeide, a partire da un piccolo volume per evitare blocchi, poi ricaricare fino a tutto volume. Capovolgere. Crosslink per 10 minuti a temperatura ambiente su un end-to-end shaker o dei rotatori.
- Raffreddare bruscamente la reazione di reticolazione con volume 1/10th di 1,25 M glicina a temperatura ambiente per 5 min.
- Spin a 2000RCF per 5 min. Aspirare il surnatante e pellet lavaggio con 20 mL PBS volta raffreddato, filatura a 2000RCF per 5 min.
- Aspirare e risospendere il wincenerito, cross-linked pellet con 1 ml di PBS refrigerata per 20 milioni di cellule. Trasferire ogni ml di una provetta Eppendorf e centrifugare a 2000RCF per 3 min a 4 ° C. Rimuovere il più possibile con PBS punta della pipetta accuratamente.
- Flash-congelare i pellet cellulari in azoto liquido e conservare a -80 ° C a tempo indeterminato.
4. Lisi cellulare
Lisare cellule reticolati per preparare lisato cellulare.
- Scongelare pellet cellulari surgelati a temperatura ambiente. Toccare difficili da rimuovere e miscelare il pellet di cellule. Rallenta il pellet a 2000RCF per 3 min a 4 ° C. Utilizzare una forte punta di 10 microlitri pipetta per rimuovere ogni residuo PBS.
- Su una bilancia elettronica (precisione di 1mg) tarare la massa di un tubo vuoto Eppendorf (i nostri tubi pesano 1.060 grammi molto coerente). Pesare ciascuna pellet e registrare il suo peso. Un piatto completo di 15 cm di cellule HeLa reticolati pesa in genere 100 mg.
- Supplemento Lysis Buffer (10 volte la massa di pellet, ad esempio, 1 ml per 100 mg) con Fresh inibitore della proteasi, PMSF e Superase-in (vedi elenco allegato buffer). Mescolare bene.
- Aggiungi 10X tampone di lisi del volume integrata in ogni provetta e risospendere il pellet. Per le piccole palline <25 mg, risospendere in 250 microlitri di buffer di lisi integrato. Sospensione deve essere liscia. In caso contrario, dividere la sospensione in 500 pl aliquote e utilizzare un mixer motorizzato pellet per rompere grumi. Procedere immediatamente a sonicazione.
5. Sonicazione
Shear DNA da lisati cellulari sonicazione reticolati.
- Sonicare lisato cellulare in Bioruptor in 15 ml provette Falcon. Utilizzare <1,5 ml di lisato in ciascuna provetta, e per una più rapida sonicazione, sonicare non più di due tubi per volta.
- Sottoporre ad ultrasuoni in un bagno di acqua 4 ° C alla massima impostazione con 30 secondi, 45 secondi OFF intervalli di impulso. Controllare lisato ogni 30 min. Continuare sonicazione finché il lisato cellulare è più torbida. Questa operazione potrebbe richiedere un minimo di 30 min e fino a 4 ore. Il numerodi tubi, il volume del campione, la temperatura del bagno, e il periodo di tempo sonicazione che influenzano la durata del processo richiede. Tubi probabilmente ultrasuoni a velocità differenti, in modo da mettere in comune tra loro ogni 30 min e ridistribuire in tubi originali per assicurare l'omogeneità. Nota: glutaraldeide-reticolato cellule richiedere molto più tempo per sonicare equivalenti di formaldeide.
- Quando lisato diventa chiaro, cedere il 5 ul lisato in una nuova provetta Eppendorf. Aggiungere 90 ul DNA proteasi K (PK) Buffer (vedi elenco buffer) e 5 PK ul. Vortex per miscelare e centrifugare brevemente. Incubare per 45 min a 50 ° C.
- Estrarre DNA con il kit di purificazione PCR Qiagen. DNA Eluire in 30 microlitri Buffer Elution Qiagen (EB) e controllare le dimensioni del DNA su gel di agarosio 1%. Se massa della striscio DNA è 100-500 bp, sonicazione è completa. In caso contrario, continuano ad ultrasuoni.
- Centrifugare i campioni sonicati a 16100RCF per 10 minuti a 4 ° C. Combina surnatanti, le aliquote in 1 ml di campioni e flash-congelamento in azoto, liquidon. Conservare a -80 ° C.
6. CHIRP
Ibridano sonde di DNA a RNA biotinilati e isolare cromatina legata.
- Scongelare i tubi della cromatina a temperatura ambiente.
- Preparare il tampone di ibridazione (vedi elenco buffer, preparare 2 ml per ml della cromatina). Vortex per mescolare.
- Per un esempio tipico CHIRP utilizzando 1 ml di lisato, 10 L per rimuovere INPUT RNA e 10 L per INPUT DNA e posto in provette Eppendorf. Tenere in ghiaccio fino al successivo utilizzo.
- Trasferire 1 cromatina mL a 15 mL provetta Falcon. Aggiungere 2 Tampone di ibridazione mL in ciascun tubo. Per volume totale <1,5 ml, utilizzare provette Eppendorf.
- Scongelare sonde a temperatura ambiente. NanoDrop sonde per controllare importo, se non lo avete utilizzato in un tempo lungo (100 sonde pM dovrebbe spec ~ 500-600 ng / ul utilizzando singola impostazione filamento di DNA). Aggiungere il volume appropriato di sonde per tubi specifici (sonda 100 pmol per 1 mL cromatina, 1 ml di 100 pmol / ul sonda per 1 mL cromatina).Mescolare bene. Incubare a 37 ° C per 4 ore con agitazione.
- Con 20 min rimanente per ibridazione, preparare le perline magnetiche C-1 (conservato a 4 ° C). Utilizzare 100 pl per 100 pmol di sonde. Lavare con 1 mL di tampone di lisi senza supplementi per tre volte, utilizzando il DynaMag-2 striscia magnetica di perline separate da buffer.
- Risospendere le sfere in volume originale di tampone di lisi e Supplemento fresca PMSF, PI e Superase-in. Dopo 4 ore reazione di ibridazione è completa, aggiungere 100 microlitri perline in ciascun tubo. Mescolare bene. Incubare a 37 ° C per 30 minuti con agitazione.
- Preparare il tampone di lavaggio (5 mL per campione). Vortex per mescolare. Pre-riscaldare a 37 ° C. Aggiungere PMSF prima dell'uso.
- Lavare perline con 1 mL di tampone di lavaggio cinque volte. Al primo lavaggio, utilizzare DynaMag-15 nastro magnetico a grani separati, decantare, e risospendere in 1 ml di tampone di lavaggio. Trasferire il volume di 1,5 ml provetta Eppendorf. Incubare a 37 ° C con agitazione per 5 min.
- Il lavaggi successivi, spin down ogni tubo su un minicentrifuge, Set di prova sulla DynaMag-2 striscia magnetica per 1 min. Campione di decantare, asciugare eventuali gocce con un Kimwipe, risospendere in 1 ml di tampone di lavaggio. Incubare a 37 ° C con agitazione per 5 min. Ripetere l'operazione per cinque lavaggi totali.
- In ultimo lavaggio, risospendere le sfere bene. Togliere 100 pl e mettere da parte per l'isolamento di RNA. Riserva 900 microlitri per frazione di DNA. Posizionare tutte le provette su DynaMag-2 banda magnetica e rimuovere il tampone di lavaggio. Centrifugare tutte le provette premuto brevemente, metterli su striscia magnetica. Rimuovere l'ultimo bit di tampone di lavaggio completamente con un netto 10 puntale ul.
7. RNA isolamento
Estratto frazione di RNA da campioni CHIRP per quantificare da qRT-PCR.
- Prendete 100 campioni ul tallone e 10 L campione di input RNA. Aggiungere 85 ul RNA PK Buffer pH 7,0 a RNA INPUT. Risospendere le sfere in 95 microlitri di buffer pH 7,0 RNA PK. Aggiungere 5 K ul Proteinease e incubare a 50 ° C per 45 min con end-to-end agitazione.
- In breve lo spin-down tutti i tubi ecampioni bollire per 10 min in blocco di calore a 95 ° C.
- Raffreddare i campioni sul ghiaccio, aggiungere 500 microlitri TRIzol, mescolare vigorosamente nel vortex per 10 sec. Incubare a temperatura ambiente per 10 min. Conservare a -80 ° C o di procedere al punto 4.
- Aggiungere 100 ul al cloroformio Trizol campioni trattati. Energicamente per 10 sec. Spin a 16100RCF su una centrifuga da banco per 15 min a 4 ° C.
- Rimuovere ~ 400 microlitri surnatante acquoso, evitando organico e interfaccia.
- Aggiungere 600 microlitri (1,5 volume) 100% di etanolo e mescolare bene. Spin campione attraverso MIRNeasy colonne mini. Lavare 1x RWT (MIRNeasy mini kit), 2x con RPE al protocollo del produttore. Eluire con 30 microlitri priva di nucleasi H 2 O (NFH 2 O).
- Trattare l'eluato RNA con DNA-free per protocollo del produttore. Dopo la reazione è completa, riscaldare il campione per 15 minuti a 65 ° C per inattivare completamente qualsiasi residuo DNasi.
- Utilizzare 1 RNA ul isolato per bene per qRT-PCR per confermare lncRNArecupero. GAPDH viene spesso utilizzato come controllo negativo.
8. DNA Isolation
Estratto frazione di DNA da campioni CHIRP mediante sequenziamento per identificare o quantificare da qPCR.
- Preparare il tampone di eluizione del DNA (vedi elenco buffer), 150 ml per campione, con ingresso DNA.
- Aggiungere 1 0μL RNasi A (10 mg / ml) e 10 L RNasi H (10 U / pl) per ml di tampone di eluizione DNA, e vortex per miscelare.
- Risospendere ogni campione di perle in 150 microlitri di tampone di eluizione del DNA con RNasi. (DNA INPUT risospendere in 140 pl) incubare a 37 ° C per 30 minuti con agitazione.
- Perle separate e il surnatante DynaMag-2 striscia magnetica. Rimuovere il surnatante e aggiungere ai tubi etichettati.
- Preparare una seconda aliquota di tampone di DNA eluizione con 10 L RNasi A (10 mg / mL) e RNaseH (10 U / mL) esattamente come fatto in 8,2). Aggiungere 150 ul di ogni campione (comprese INPUT DNA), incubare e rimuovere il surnatante. Raccogliere tutte le surnatante (spallad essere ~ 300 mL).
- Aggiungere 15 PK ul a ciascun campione. Incubare a 50 ° C per 45 minuti con agitazione.
- Pre-spin down giallo phase-lock tubi di gel (5PRIME). Trasferimento campioni di DNA di phase-lock tubi di gel, e aggiungere 300 ml PhOH: Cloroformio: isoamile per campione. Agitare vigorosamente per 10 min, e girare su una centrifuga da banco a 16100RCF per 5 min a 4 ° C. Prendere acquoso dalla parte superiore (~ 300 pl). Aggiungere 3 GlycoBlue uL, NaOAc 30 uL, e 900 microlitri EtOH 100%. Mescolare bene e conservare a -20 ° C per una notte.
- Spin campioni a 16100RCF per 30 minuti a 4 ° C.
- Decantare il surnatante con cura. Aggiungere 1 ml di EtOH 70% e vortex per miscelare. Spin a 16100RCF per 5 min. Rimuovere il surnatante con la pipetta. Lasciare asciugare all'aria per 1min. Risospendere in 30 ul EB.
- Campioni di DNA sono pronti per l'analisi mediante qPCR o nella preparazione di high-throughput sequencing librerie per protocollo Illumina.
10. Risultati rappresentativi
Figura 1 descrive il flusso di lavoro CHIRP. Un esperimento CHIRP successo arricchisce tipicamente RNA bersaglio significativamente nel non-specifici RNA. Figura 2 mostra arricchimento della telomerasi RNA umano (TERC) da cellule HeLa oltre GAPDH, un RNA abbondanti cellulari che serve come controllo negativo. La maggior parte degli RNA TERC (~ 88%) presenti nella cellula sono state abbattute eseguendo CHIRP, mentre solo il 0,46% del GAPDH RNA è stato recuperato, dimostrando un fattore di arricchimento di circa 200 volte. Le sonde non specifici quali sonde di targeting LacZ RNA, che non è espressa in cellule di mammifero (Figura 2), può essere usato come addizionali controlli negativi.
Regioni di DNA prevede di impegnare la lncRNA destinazione sono in genere arricchiti sulle regioni negativi quando misurata dal qPCR. La figura 3 mostra la convalida qPCR di quattro Hotair-bound siti primari fibroblasti del prepuzio umano che siamo determinati eseguendo CHIRP-seq nella linea stessa cella, mentre TERC e GAPDH DNA siti di sérve come regioni di controllo negativo. Sia "anche" e la sonda "dispari" imposta arricchimento prodotto comparabile attesi Hotair-bound siti su regioni negativi, un segno distintivo di veri lncRNA siti di legame.
High-throughput sequenziamento del DNA CHIRP arricchito produce una mappa globale di lncRNA siti di legame. La Drosophila lncRNA roX2 è noto per interagire con il cromosoma X in un modo che è necessaria la compensazione del dosaggio. Figura 4 mostra roX2 profilo di legame su una sezione del cromosoma X. Sia "anche" e "dispari" i campioni sono stati sequenziati e le loro unici rumori sono stati eliminati per produrre una traccia di segnali sovrapposti. Ogni "picco" indica qui un sito di forte roX2 vincolante. La pista e la lista delle roX2 geni bersaglio sono stati descritti in Chu et al. 2011 17.
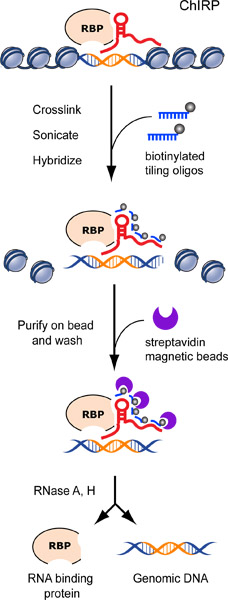
Figura 1. Diagramma di flusso della procedura CHIRPdure. La cromatina è reticolata ad lncRNA: addotti proteici in vivo Biotinylated sonde piastrelle sono ibridate per indirizzare lncRNA e complessi della cromatina sono purificati utilizzando perline streptavidina magnetici, seguiti da lavaggi stringenti.. Noi eluire DNA legato lncRNA o proteine, con un cocktail di RNasi A e H. è schematizzata Una sequenza putativa lncRNA vincolante in arancione. Precedentemente pubblicato in Chu et al. 2011 17.
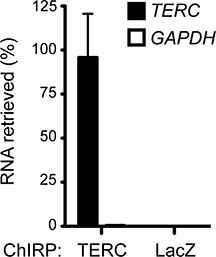
Figura 2. Arricchisce CHIRP per l'uomo TERC RNA. TERC-asDNA sonde recuperare ~ 88% dei cellulari TERC RNA non rilevabile e GAPDH. LacZ-asDNA sonde vengono utilizzati come controlli negativi e recuperare né RNA. Media + sd vengono visualizzati. Precedentemente pubblicato in Chu et al. 2011 17.
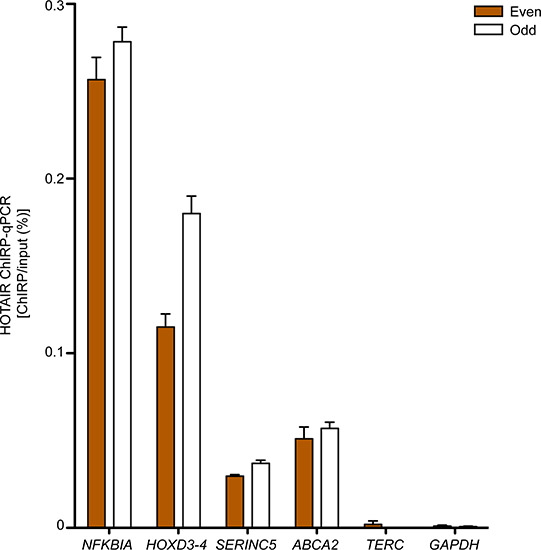
Figura 3. Hotair CHIRP-qPCR in umana primaria perfibroblasti Eskin. NFKBIA, HOXD3-4, SERINC5 e ABCA2 sono regioni che interagiscono con Hotair. TERC e GAPDH serviti come controlli negativi. Media + sd vengono visualizzati. Precedentemente pubblicato in Chu et al. 2011 17.
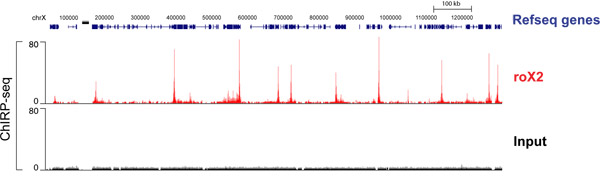
Figura 4. CHIRP-seguenti dati di roX2 SL2 RNA in cellule di Drosophila. "Anche" e "dispari" sono stati sequenziati separatamente, i loro dati si fondono in modo da riflettere solo picchi comuni in entrambi. La pista è mostrato unito. Precedentemente pubblicato in Chu et al. 2011 17.
Discussione
Qui abbiamo descritto CHIRP-seq, un metodo di mappatura in vivo i siti di legame lncRNA genome-wide. I parametri chiave per il successo sono le piscine separate di piastrelle sonde oligonucleotidiche e reticolazione glutaraldeide. Il disegno di sonde per affinità è semplice data la sequenza di RNA e non richiede alcuna conoscenza preventiva della struttura del RNA o domini funzionali. Il nostro successo con roX2, TERC e Hotair - tre RNA piuttosto differenti in due specie - suggerisce che CHIRP-seq è probabile generalizzabile a molte lncRNAs. Come per tutti gli esperimenti, i controlli per la cura e propria sono tenuti a interpretare i risultati. LncRNA diversi possono richiedere la titolazione delle condizioni, giudizioso e il cambiamento delle condizioni, come la selezione di sonde affinità diverse o reticolanti, può mettere in luce aspetti diversi di interazioni RNA-cromatina. Come ChIP-seq, non tutti gli eventi vincolanti sono necessariamente funzionali, e sono necessari ulteriori studi per accertare le conseguenze biologiche di RNA occupancy sulla cromatina. Tuttavia, si prevede molte interessanti applicazioni di questa tecnologia per i ricercatori di altri cromatina associate lncRNAs, che ora si contano a migliaia 8,9. Proprio come ChIP-seq ha aperto la porta per la genome-wide esplorazioni di interazioni proteina-DNA, CHIRP-seq studi della "RNA interattoma" può rivelare molte nuove vie della biologia.
Divulgazioni
C. Chu e HY Chang sono denominati come inventori su una domanda di brevetto basato su questo metodo.
Riconoscimenti
Ringraziamo T. Hung, MC. Tsai, O. Manor, E. Segal, M. Kuroda, T. Swigut e I. Shestopalov per le discussioni. Supportato da parte dell'Agenzia di Scienza, Tecnologia e Ricerca di Singapore (CC), NIH R01-R01-CA118750 e HG004361 (HYC), e la California Institute for Regenerative Medicine (HYC). HYC è uno scienziato inizi della carriera del Howard Hughes Medical Institute.
Materiali
| Name | Company | Catalog Number | Comments |
| Buffer List: | |||
| Dissolve a pellet of complete protease inhibitor in 1 ml water as 50x stock. Make 100 mM PMSF in isopropanol (100x stock). Superase-in is used as 200x stock. Store all at -20 °C. | |||
| Lysis Buffer: | |||
| 50 mM Tris-Cl pH 7.0 10 mM EDTA 1% SDS Always add PMSF, P.I. and Superase-in fresh before use except when washing beads | |||
| Proteinase K Buffer (for DNA) | |||
| 100 mM NaCl 10 mM TrisCl pH 8.0 (For RNA use pH 7.0) 1 mM EDTA 0.5% SDS Add 5% by volume Proteainse K (Ambion AM2546 20 mg/ml) fresh before use | |||
| Hybridization Buffer | |||
| 750 mM NaCl 1% SDS 50 mM Tris-Cl pH 7.0 1 mM EDTA 15% formamide (store in the dark at 4 °C) Always add PMSF, P.I. and Superase-in fresh before use | |||
| Wash Buffer | |||
| 2x NaCl and Sodium citrate (SSC) (diluted from 20x SSC Invitrogen stock) 0.5% SDS Always add PMSF fresh before use | |||
| DNA elution Buffer | |||
| 50 mM NaHCO3 1% SDS | |||
| Table of specific reagents and equipment: | |||
| Glutaraldehyde (EM grade) | Sigma-Aldrich | G5882-10x10ml | |
| Motorized pellet mixer | VWR international | V8185-904 | |
| Protease inhibitor | Roche Group | 11873580001 | |
| PMSF | Sigma-Aldrich | 78830 | |
| Superase-in | Ambion | AM2696 | |
| Bioruptor | Diagenode | UCD-200 | |
| Falcon tubes (for sonication) | Corning | 430790 | |
| Proteinase K | Ambion | AM2546 | |
| PCR purification kit | Qiagen | 28106 | |
| C-1 magnetic beads | Invitrogen | 65002 | |
| PMSF | Sigma-Aldrich | P7626-25G | |
| DynaMag-15 magnet | Invitrogen | 123-01D | |
| DynaMag-2 magnet | Invitrogen | 123-21D | |
| MIRNeasy mini kit | Qiagen | 217004 | |
| Rnase H | Epicentre Biotechnologies | R0601K | |
| Rnase A | Sigma-Aldrich | R4875-100MG | |
| Phase Lock Gel Heavy | 5 PRIME | 2302810 | |
| Trizol | Invitrogen | 15596-018 | |
| Phenol:chloroform:Isoamyl | Invitrogen | 15593-031 | |
| Chloroform | Ricca | RSOC0020-1C | |
| GlycoBlue | Ambion | AM9515 | |
| Glycine | JT Baker | 4057-06 | |
| PBS, pH 7.4 | Invitrogen | 10010-049 | |
| Elution Buffer (EB) | Qiagen | 19086 | |
| 20x SSC | Invitrogen | 15557-036 | |
| 10% SDS | Invitrogen | 15553-027 | |
| DNA-free | Ambion | AM1906 | |
| Buffer kit | Ambion | AM9010 | |
| Formamide | Invitrogen | 15515-026 | |
Riferimenti
- Koziol, M. J., Rinn, J. L. RNA traffic control of chromatin complexes. Curr. Opin. Genet. Dev. 20, 142-148 (2010).
- Mercer, T. R., Dinger, M. E., Mattick, J. S. Long non-coding RNAs: insights into functions. Nat. Rev. Genet. 10, 155-159 (2009).
- Rinn, J. L. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 129, 1311-1323 (2007).
- Zhao, J., Sun, B. K., Erwin, J. A., Song, J. J., Lee, J. T. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science. 322, 750-756 (2008).
- Kelley, R. L. Epigenetic spreading of the Drosophila dosage compensation complex from roX RNA genes into flanking chromatin. Cell. 98, 513-522 (1999).
- Pandey, R. R. Kcnq1ot1 antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation. Mol. Cell. 32, 232-246 (2008).
- Wang, K. C. A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression. Nature. 472, 120-124 (2011).
- Khalil, A. M. Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression. Proc. Natl. Acad. Sci. U.S.A. 106, 11667-11672 (2009).
- Zhao, J. Genome-wide identification of polycomb-associated RNAs by RIP-seq. Mol. Cell. 40, 939-953 (2010).
- Meller, V. H., Wu, K. H., Roman, G., Kuroda, M. I., Davis, R. L. roX1 RNA paints the X chromosome of male Drosophila and is regulated by the dosage compensation system. Cell. 88, 445-457 (1997).
- Franke, A., Baker, B. S. The rox1 and rox2 RNAs are essential components of the compensasome, which mediates dosage compensation in Drosophila. Mol. Cell. 4, 117-122 (1999).
- Gupta, R. A. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature. 464, 1071-1076 (2010).
- Tsai, M. C. Long noncoding RNA as modular scaffold of histone modification complexes. Science. 329, 689-693 (2010).
- Zappulla, D. C., Cech, T. R. RNA as a flexible scaffold for proteins: yeast telomerase and beyond. Cold Spring Harb. Symp. Quant. Biol.. 71, 217-224 (2006).
- Nagano, T. The Air noncoding RNA epigenetically silences transcription by targeting G9a to chromatin. Science. 322, 1717-1720 (2008).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature. 32, 623-626 (2002).
- Chu, C., Qu, K., Zhong, F. L., Artandi, S. E., Chang, H. Y. Genomic Maps of Long Noncoding RNA Occupancy Reveal Principles of RNA-Chromatin Interactions. Mol. Cell. , (2011).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5, 877-879 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon