Method Article
Isolement de la chromatine par purification d'ARN (CHIRP)
Dans cet article
Résumé
CHIRP est une nouvelle technique et rapide à la carte génomique des sites de liaison de l'ARN non codantes de longues (lncRNAs). La méthode tire parti de la spécificité des oligonucléotides carrelage anti-sens pour permettre le dénombrement des sites génomiques lncRNA assortis.
Résumé
ARN non codant longues sont des régulateurs clés de états de la chromatine pour des processus biologiques importants tels que la compensation de dosage, d'impression, et l'expression des gènes de développement 1,2,3,4,5,6,7. La découverte récente de milliers de lncRNAs en association avec des complexes spécifiques modification de la chromatine, comme complexe Polycomb répressive 2 (PRC2) qui assure la médiation histone H3 lysine 27 triméthylation (H3K27me3), suggère grands rôles pour de nombreuses lncRNAs dans la gestion des états de chromatine dans un gène spécifique la mode 8,9. Alors que certains sont lncRNAs pensé à travailler en cis sur les gènes voisins, d'autres travaillent dans lncRNAs trans pour réguler les gènes situés loin. Par exemple, chez la drosophile lncRNAs roX1 et roX2 régions lient de nombreux sur le chromosome X des cellules mâles, et sont essentiels pour 10,11 compensation de dosage. Toutefois, les emplacements exacts de leurs sites de liaison ne sont pas connus à haute résolution. De même, l'homme lncRNA Hotair peut affecter PRC2 d'occupation sur les hes centaines de gènes du génome entier 3,12,13, mais comment la spécificité est atteint n'est pas claire. LncRNAs peut également servir comme des échafaudages modulaires pour recruter l'assemblage des complexes protéiques multiples. Le classique agissant en trans ARN échafaud est le TERC ARN qui sert de modèle et d'échafaudage pour la télomérase complexe 14; Hotair peut aussi servir comme un échafaudage pour PRC2 et une déméthylase H3K4 complexe 13.
Des études antérieures de cartographie d'occupation ARN à la chromatine ont permis de mieux comprendre substantielles 15,16, mais seulement à un seul locus à la fois. Les sites d'occupation de la plupart des lncRNAs ne sont pas connus, et les rôles de lncRNAs en matière de réglementation chromatine ont été la plupart du temps induite par les effets indirects de la perturbation lncRNA. Tout comme immunoprécipitation de la chromatine suivie par microarray ou séquençage en profondeur (ChIP-chip ou ChIP-seq, respectivement) a grandement amélioré notre compréhension des interactions protéine-ADN sur une échelle génomique, ici, nous illustrer un recenTLY publié stratégie pour cartographier l'occupation à long génome à ARN à l'échelle à haute résolution 17. Cette méthode, d'isolement de la chromatine par purification d'ARN (CHIRP) (Figure 1), est basée sur la capture d'affinité de la cible lncRNA: complexe de la chromatine par carrelage antisens-oligos, qui génère alors une carte de la génomique des sites de liaison à une résolution de plusieurs centaines de bases avec de sensibilité et d'arrière-plan faible. CHIRP est applicable à de nombreux lncRNAs parce que la conception de sondes affinité est simple compte tenu de la séquence d'ARN et ne nécessite aucune connaissance de la structure de l'ARN ou des domaines fonctionnels.
Protocole
1. Conception de la sonde
Conception d'ADN anti-sens de carrelage sondes pour la récupération sélective de l'ARN cible par Chirp.
- Sondes de conception oligo anti-sens en utilisant le concepteur de sonde en ligne à singlemoleculefish.com 18.
- Utilisez ces paramètres: nombre de sondes = 1 / sonde de 100 pb de longueur ARN; 2) Cible GC% = 45; longueur Espacement 4) = 60-80; 3) Longueur = 20 oligonucléotides. Pause d'ARN dans les segments s'il est trop long pour le concepteur. Omettre régions de répétitions ou d'homologie étendue.
- Sondes d'ADN de commande anti-sens avec BiotinTEG à 3-Premier fin.
- Des sondes marqueurs en fonction de leurs positions le long de l'ARN. Séparez-les en deux poules de sorte que le "même" pool contient toutes les sondes au nombre de 2, 4, 6, etc, et le «bizarre» pool contient des sondes au nombre de 1, 3, 5, etc Diluer pool de sondes à une concentration de 100 uM et conserver à -20 ° C.
- Toutes les expériences doivent être effectuées à l'aidedeux piscines, qui servent de contrôles internes pour l'autre. Réel ARN-dépendante du signal serait présent à partir de deux piscines, tandis que la sonde spécifiques bruits serait propre à chaque piscine. Ceci s'applique à la fois pour chirp-qPCR et CHIRP-seq.
2. Les cellules de récolte
Recueillir les cellules qui seront utilisées pour l'expérience gazouiller.
- Cultiver les cellules dans des plaques de culture de tissus ou des flacons à la confluence. Rincer avec un tampon phosphate salin (PBS) et une fois trypsiniser. Quench trypsine avec un volume> 2x des médias, une pipette de haut en bas pour déloger les cellules et remettre en suspension seule cellule. Transférez tous les médias et les cellules remises en suspension dans 50 ml tubes Falcon. 20 millions de cellules sont généralement suffisante pour un échantillon gazouiller.
- Spin cellules à 800RCF pendant 4 min. Aspirer les médias et remettre 40 millions de cellules dans 40 ml de PBS, de combiner les tubes si nécessaire. Spin cellules à 800RCF pendant 4 min. Décanter PBS, aspirer délicatement sur un angle du liquide restant.
3. Croix-lier des cellules et collecte culot cellulaire
Crosslink recueillies cellules avec du glutaraldéhyde à préserver l'ARN-chromatine interactions et de préparer culot cellulaire.
- Effectuez toutes les étapes à température ambiante.
- Préparer glutaraldéhyde à 1% à température ambiante PBS. Préparer 10 ml par 10 millions de cellules (0,4 ml de bouillon de glutaraldéhyde à 25% + 9,6 ml de PBS). Glutaraldéhyde doit être utilisée fraîche.
- Appuyez sur fond de tubes Falcon pour déloger pellets. Resuspendre culot cellulaire dans du glutaraldéhyde à 1%, en commençant par un petit volume pour éviter morceaux, puis compléter à plein volume. Mélanger par inversion. Crosslink pendant 10 minutes à température ambiante sur un agitateur de bout en bout ou des rotateurs.
- Arrêter la réaction de réticulation avec le volume de 1/10e de 1,25 M de glycine à la température ambiante pendant 5 min.
- Centrifuger à 2000RCF pendant 5 min. Aspirer le surnageant et laver culot avec 20 ml refroidi PBS fois, tourne à 2000RCF pendant 5 min.
- Aspirer et remettre en suspension le wcendré, réticulé culot avec 1 ml de PBS par réfrigéré 20 millions de cellules. Transférer chaque ml dans un tube Eppendorf et centrifuger à 2000RCF pendant 3 min à 4 ° C. Retirez autant que possible avec du PBS pointe de la pipette avec soin.
- Flash geler les culots cellulaires dans l'azote liquide et stocker à -80 ° C indéfiniment.
4. Lyse cellulaire
Lyser les cellules réticulées à préparer un lysat cellulaire.
- Décongeler les culots de cellules congelés à température ambiante. Tapez difficile à déloger et mélanger le culot cellulaire. Isoler le culot à 2000RCF pendant 3 min à 4 ° C. Utilisez une forte pointe de la pipette 10 ul de supprimer tout reste PBS.
- Sur une balance électronique (précision de 1 mg) la tare de la masse d'un tube vide Eppendorf (nos tubes pèsent 1.060 grammes très régulièrement). Peser chaque pastille et enregistrer son poids. Un plat complet de 15 cm de cellules HeLa réticulés pèse généralement 100 mg.
- Tampon de lyse Supplément (10X la masse de granulés de bois, par exemple 1 ml pour 100 mg) avec fresquesh Inhibiteur de la protéase, PMSF et Superase-in (voir la liste des tampons ci-jointe). Mélangez bien.
- Ajouter 10X tampon de lyse du volume complété à chaque tube et remettre le culot. Pour de petites boulettes <25 mg, remettre en suspension dans 250 ul de tampon de lyse complétée. La suspension doit être lisse. Si ce n'est pas, il faut diviser la suspension en 500 aliquotes et utiliser un véhicule motorisé à granulés mélangeur pour briser les mottes. Procéder immédiatement à une sonication.
5. Sonication
ADN cisaillement par sonication réticulés lysats cellulaires.
- Soniquer lysat cellulaire dans Bioruptor dans 15 ml tubes Falcon. Utilisation <1,5 lysat ml dans chaque tube, et pour accélérer la sonication, sonication pas plus de deux tubes à la fois.
- Soniquer dans un bain d'eau à 4 ° C au réglage le plus élevé avec 30 secondes ON, 45 secondes OFF intervalles d'impulsions. Vérifiez lysat toutes les 30 min. Continuer jusqu'à ce que le traitement aux ultrasons lysat cellulaire est plus trouble. Cela peut prendre aussi peu que 30 minutes et jusqu'à 4 heures. Le nombredes tubes, le volume d'échantillon, la température du bain, et la période de temps de sonication se influer sur la durée du processus prend. Tubes sera probablement sonication à des taux différents, afin de les mettent en commun toutes les 30 min et de le redistribuer dans des tubes d'origine pour assurer l'homogénéité. Remarque: le glutaraldéhyde réticulé cellules prendre beaucoup plus de temps à sonication que leurs équivalents de formaldéhyde.
- Lorsque lysat tourne clair, transférer 5 lysat ul dans un nouveau tube Eppendorf. Ajouter 90 uL d'ADN protéase K (PK) de tampon (buffer voir la liste) et 5 PK ul. Vortex pour mélanger et centrifuger brièvement. Incuber pendant 45 min à 50 ° C.
- Extraire l'ADN avec le kit Qiagen PCR purification. ADN Eluer dans 30 uL tampon d'élution Qiagen (EB) et vérifier la taille de l'ADN sur gel d'agarose 1%. Si en vrac du frottis d'ADN est 100-500 pb, sonication est terminée. Si non, continuer à sonication.
- Centrifuger les échantillons à soniqués 16100RCF pendant 10 min à 4 ° C. Combinez surnageants, aliquotes dans des échantillons de 1 mL et flash-gel dans nitroge liquiden. Conserver à -80 ° C.
6. CHIRP
Hybrider des sondes d'ADN à l'ARN biotinylés et d'isoler la chromatine liée.
- Décongeler complètement les tubes de la chromatine à la température ambiante.
- Préparer un tampon d'hybridation (voir la liste des tampons, de préparer 2 ml par ml de la chromatine). Vortex pour mélanger.
- Pour un échantillon typique de CHIRP avec 1 ml de lysat, retirez 10 uL pour INPUT ARN et 10 uL d'ADN pour INPUT et le lieu dans des tubes Eppendorf. Gardez sur la glace jusqu'à ce une utilisation ultérieure.
- Transférer 1 ml chromatine à 15 ml Falcon tube. Ajouter 2 ml de tampon d'hybridation à chaque tube. Pour volume total <1,5 ml, utiliser des tubes Eppendorf.
- Décongeler sondes à température ambiante. Sondes Nanodrop pour vérifier le montant si vous ne l'avez pas utilisé dans un temps long (100 uM sondes devraient spec ~ 500-600 ng / ul en utilisant unique paramètre brin d'ADN). Ajouter le volume approprié de sondes dans des tubes spécifiques (100 pmol de sonde pour 1 ml de la chromatine, 1 ul de 100 pmol / ul sonde par 1 ml chromatine).Mélangez bien. Incuber à 37 ° C pendant 4 heures avec agitation.
- Avec 20 min en restant pour l'hybridation, préparer les perles C-1 magnétiques (conservé à 4 ° C). Utiliser 100 pi par 100 pmol de sondes. Laver avec 1 ml tampon de lyse sans complément trois fois, en utilisant la bande magnétique dynamag-2 à des billes distinctes de la mémoire tampon.
- Remettre les billes en volume initial de tampon de lyse, supplément avec des produits frais PMSF, PI et Superase en. Après 4 h réaction d'hybridation est terminée, ajouter 100 ul perles à chaque tube. Mélangez bien. Incuber à 37 ° C pendant 30 min avec agitation.
- Préparer le tampon de lavage (5 ml par échantillon). Vortex pour mélanger. Préchauffer à 37 ° C. Ajouter PMSF avant utilisation.
- Laver perles avec 1 ml de tampon de lavage cinq fois. Sur le premier lavage, utilisez dynamag-15 bande magnétique à des billes séparées, de décantation, et remettre en suspension dans 1 ml de tampon de lavage. Transfert de volume à 1,5 ml tube Eppendorf. Incuber à 37 ° C avec agitation pendant 5 min.
- Sur les lavages suivants, tourner en bas de chaque tube sur un minicentrifuge, Mis en échantillon sur dynamag-2 bande magnétique pendant 1 min. Décanter l'échantillon, essuyer les gouttes avec un Kimwipe, remettre en suspension dans une solution de lavage ml. Incuber à 37 ° C avec agitation pendant 5 min. Répétez l'opération pour cinq lavages au total.
- Au dernier lavage, remettre en suspension les perles bien. Prélever 100 ul et mis de côté pour l'isolement d'ARN. Réserve 900 pi de la fraction d'ADN. Placer tous les tubes sur dynamag-2 bande magnétique et éliminer le tampon de lavage. Spin tous les tubes vers le bas brièvement; les placer sur bande magnétique. Retirez le dernier bit du tampon de lavage complètement avec une forte pointe de la pipette 10 ul.
7. RNA
Extrait fraction d'ARN à partir d'échantillons CHIRP pour quantifier par qRT-PCR.
- Prenez 100 ul d'échantillons de perles et un échantillon de 10 ul ENTREE ARN. Ajouter 85 ul d'ARN PK tampon pH 7,0 à l'ARN de départ. Remettre les billes dans 95 ul d'ARN 7,0 PK pH tampon. Ajouter 5 K Proteinease ul et incuber à 50 ° C pendant 45 min avec de bout en bout en secouant.
- En bref spin down tous les tubes etfaire bouillir les échantillons pendant 10 min sur le bloc de chaleur à 95 ° C.
- Réfrigérer les échantillons sur la glace, ajouter 500 ul TRIzol, homogénéiser vigoureusement au vortex pendant 10 secondes. Incuber à température ambiante pendant 10 min. Conserver à -80 ° C ou passez à l'étape 4.
- Ajouter 100 ul de chloroforme à TRIzol échantillons traités. Vortex vigoureusement pendant 10 sec. Centrifuger à 16100RCF sur une centrifugeuse de paillasse pendant 15 min à 4 ° C.
- Retirer ~ 400 pi de surnageant aqueux, organique et en évitant l'interface.
- Ajouter 600 ul (1,5 volume) d'éthanol à 100% et bien mélanger. Spin échantillon à travers MIRNeasy colonnes mini. Laver 1x avec RTT (MIRNeasy mini kit), 2x avec RPE selon le protocole du fabricant. Éluer avec 30 uL sans nucléase H 2 O (NFH 2 O).
- Traiter l'éluat ARN avec l'ADN-libre par le protocole du fabricant. Après la réaction est terminée, chauffer l'échantillon pendant 15 minutes à 65 ° C pour inactiver complètement toute restante DNase.
- Utilisez 1 ARN ul par puits pour isoler qRT-PCR pour confirmer lncRNAextraction. GAPDH est souvent utilisé comme contrôle négatif.
8. Isolement d'ADN
Extrait fraction d'ADN à partir d'échantillons CHIRP à identifier par séquençage ou quantifier par qPCR.
- Préparer un tampon d'élution d'ADN (voir la liste des tampons), 150 ul par échantillon, y compris l'entrée d'ADN.
- Ajouter 1 0μL RNase A (10 mg / ml) et 10 ul RNase H (10 U / pl) par ml de tampon d'élution d'ADN, et le vortex pour mélanger.
- Remettre en suspension chaque échantillon de billes dans 150 pi de tampon d'élution d'ADN avec RNases. (INPUT ADN Resuspendre dans 140 uL) Incuber à 37 ° C pendant 30 min avec agitation.
- Perles séparées et le surnageant sur dynamag-2 bande magnétique. Éliminer le surnageant et ajouter à tubes étiquetés.
- Préparer une seconde partie aliquote de tampon d'élution d'ADN avec 10 uL Une RNase (10 mg / ml) et RNaseH (10 U / uL) exactement comme fait à la section 8.2). Ajouter 150 ul de chaque échantillon (y compris l'entrée d'ADN), incuber, et retirer le surnageant. Collectionnez-les tous surnageant (should être ~ 300 pi).
- Ajouter 15 uL PK à chaque échantillon. Incuber à 50 ° C pendant 45 min avec agitation.
- Pré-spin down jaune tubes de gel à verrouillage de phase (5PRIME). Transfert des échantillons d'ADN dans les tubes de gel à verrouillage de phase, et ajouter 300 ul PhOH: chloroforme: isoamyle par échantillon. Agiter vigoureusement pendant 10 min, et centrifuger dans une centrifugeuse de paillasse à 16100RCF pendant 5 min à 4 ° C. Prenez aqueuse à partir du haut (~ 300 pi). Ajouter 3 GlycoBlue ul, 30 ul de NaOAc, et 900 ul EtOH 100%. Mélangez bien et conserver à -20 ° C pendant la nuit.
- Spin échantillons à 16100RCF pendant 30 min à 4 ° C.
- Décanter le surnageant avec précaution. Ajouter 1 ml EtOH 70% et le vortex pour mélanger. Centrifuger à 16100RCF pendant 5 min. Enlever le surnageant à la pipette. Sécher à l'air pendant 1min. Remettre en suspension dans 30 pl EB.
- Des échantillons d'ADN sont prêtes pour l'analyse par qPCR ou de la préparation des bibliothèques de séquençage à haut débit par le protocole d'Illumina.
10. Les résultats représentatifs
Figure 1 représente le flux de travail gazouiller. Une expérience réussie CHIRP enrichit généralement ARN cible de façon significative par rapport aux non-spécifiques des ARN. La figure 2 montre l'enrichissement de la télomérase humaine ARN (TERC) à partir de cellules HeLa sur GAPDH, un ARN cellulaire abondant qui sert de contrôle négatif. La majorité des ARN TERC (~ 88%) présents dans la cellule ont été tiré vers le bas en effectuant CHIRP, alors que seulement 0,46% de la GAPDH ARN a été récupéré, ce qui démontre un facteur d'enrichissement de ~ 200 fois. Sondes non spécifiques tels que des sondes ciblant l'ARN LacZ, qui n'est pas exprimé dans les cellules de mammifères (Figure 2), peut être utilisé comme d'autres témoins négatifs.
Des régions d'ADN attendues pour lier le lncRNA cible sont généralement enrichies au cours régions négatifs lorsqu'elle est mesurée par qPCR. La figure 3 montre la validation qPCR de quatre Hotair assortis sites dans des fibroblastes primaires de prépuce humain que nous avons déterminé en effectuant CHIRP-seq dans la même lignée cellulaire, tandis que TERC et GAPDH l'ADN des sites soirve que les régions de contrôle négatif. Les deux «même» et «bizarre» la sonde fixe donné l'enrichissement comparable attendus Hotair assortis sites sur les régions négatives, une caractéristique des véritables lncRNA sites de liaison.
Séquençage à haut débit de l'ADN enrichi CHIRP donne une carte mondiale des sites de liaison lncRNA. La drosophile lncRNA roX2 est connue pour interagir avec le chromosome X d'une manière qui est nécessaire pour la compensation de dosage. La figure 4 montre le profil de liaison roX2 sur une section du chromosome X. Les deux "même" et "impairs" échantillons ont été séquencés et leurs bruits uniques ont été éliminés pour produire une piste de signaux qui se chevauchent. Chaque «pic» indique ici un site fort de roX2 contraignant. La piste complète et la liste des roX2 gènes cibles ont été décrits dans Chu et al. 2011 17.
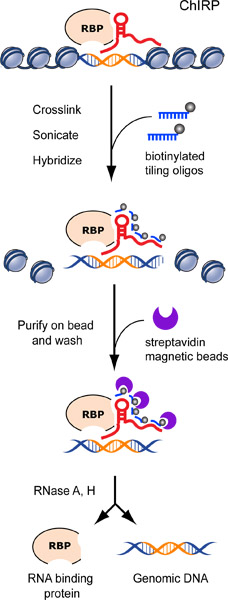
Diagramme Figure 1. De la procédure CHIRPcédure. Chromatine est réticulée à lncRNA: adduits protéiques in vivo biotinylés sondes carrelage sont hybridées à cibler lncRNA et complexes chromatiniens sont purifiés en utilisant des billes de streptavidine magnétiques, suivie par des lavages strictes.. Nous éluer lncRNA ADN lié ou des protéines avec un cocktail de la RNase A et H. Une séquence lncRNA putatif de liaison est schématisée en orange. Précédemment publié dans Chu et al. 2011 17.
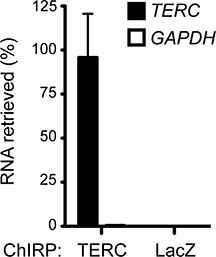
Figure 2. Enrichit CHIRP pour l'homme TERC ARN. TERC-asDNA sondes de récupérer environ 88% de l'ARN cellulaire TERC et indétectable GAPDH. LacZ-asDNA sondes sont utilisés comme témoins négatifs et de récupérer ni ARN. Moyenne + sd sont présentés. Précédemment publié dans Chu et al. 2011 17.
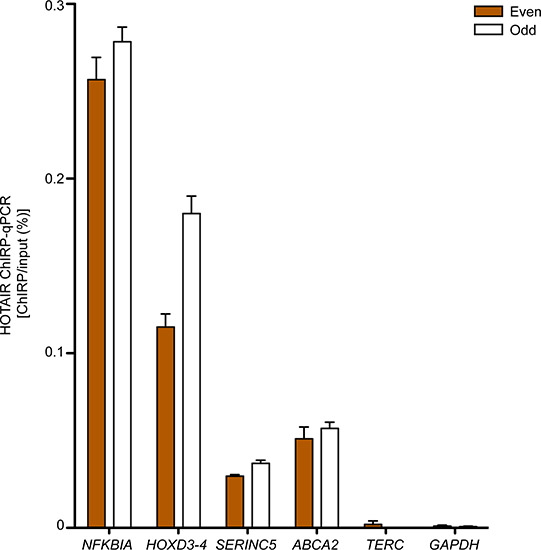
Figure 3. Hotair chirp-qPCR humaine primaire pourfibroblastes Eskin. NFKBIA, HOXD3-4, SERINC5 et ABCA2 sont les régions qui interagissent avec Hotair. TERC et GAPDH ont servi de témoins négatifs. Moyenne + sd sont présentés. Précédemment publié dans Chu et al. 2011 17.
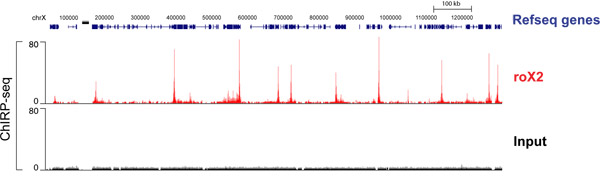
Figure 4. CHIRP-seq de données de roX2 ARN dans les cellules de drosophile SL2. "Même" et "bizarre" ont été séquencés séparément; leurs données fusionnent pour ne tenir compte que des pics communs à la fois. La piste fusionnée est montré. Précédemment publié dans Chu et al. 2011 17.
Discussion
Ici, nous avons décrit CHIRP-seq, une méthode de cartographie des sites de liaison in vivo lncRNA l'échelle du génome. Les paramètres clés de la réussite sont les piscines fendues de carrelage sondes oligonucléotidiques et de réticulation au glutaraldéhyde. La conception de sondes affinité est simple compte tenu de la séquence d'ARN et ne nécessite aucune connaissance préalable de la structure de l'ARN ou des domaines fonctionnels. Notre succès avec roX2, TERC, et Hotair - trois ARN assez différentes dans les deux espèces - suggère que CHIRP-seq est probable généralisables à de nombreux lncRNAs. Comme avec toutes les expériences, les contrôles de soins et de bonne sont tenus d'interpréter les résultats. LncRNA différents peuvent nécessiter des conditions de titrage, et les changements judicieux de conditions, telles que la sélection de sondes d'affinité différentes ou des agents de réticulation, peut mettre en évidence différents aspects de l'ARN-chromatine interactions. Comme ChIP-seq, tous les événements de liaison ne sont pas nécessairement fonctionnelle, et des études supplémentaires sont nécessaires pour déterminer les conséquences biologiques de l'ARN occupancy sur la chromatine. Néanmoins, nous prévoyons de nombreuses applications intéressantes de cette technologie pour les chercheurs d'autres chromatine associés lncRNAs, qui sont actuellement au nombre des milliers 8,9. Tout comme ChIP-seq a ouvert la porte à l'échelle du génome des explorations de interactions ADN-protéines, chirp-études suivants de la "interactome ARN" peut révéler beaucoup de nouvelles avenues de la biologie.
Déclarations de divulgation
C. Chu et HY Chang sont nommés comme inventeurs sur une demande de brevet sur la base de cette méthode.
Remerciements
Nous remercions T. Hung, MC. Tsai, O. Manor, E. Segal, M. Kuroda, T. Swigut, et I. Shestopalov pour les discussions. Soutenu par l'Agence de la Science, la Technologie et de la recherche de Singapour (CC), NIH R01 et R01-CA118750-HG004361 (HYC), et le California Institute for Regenerative Medicine (HYC). HYC est un scientifique en début de carrière de l'Institut médical Howard Hughes.
matériels
| Name | Company | Catalog Number | Comments |
| Buffer List: | |||
| Dissolve a pellet of complete protease inhibitor in 1 ml water as 50x stock. Make 100 mM PMSF in isopropanol (100x stock). Superase-in is used as 200x stock. Store all at -20 °C. | |||
| Lysis Buffer: | |||
| 50 mM Tris-Cl pH 7.0 10 mM EDTA 1% SDS Always add PMSF, P.I. and Superase-in fresh before use except when washing beads | |||
| Proteinase K Buffer (for DNA) | |||
| 100 mM NaCl 10 mM TrisCl pH 8.0 (For RNA use pH 7.0) 1 mM EDTA 0.5% SDS Add 5% by volume Proteainse K (Ambion AM2546 20 mg/ml) fresh before use | |||
| Hybridization Buffer | |||
| 750 mM NaCl 1% SDS 50 mM Tris-Cl pH 7.0 1 mM EDTA 15% formamide (store in the dark at 4 °C) Always add PMSF, P.I. and Superase-in fresh before use | |||
| Wash Buffer | |||
| 2x NaCl and Sodium citrate (SSC) (diluted from 20x SSC Invitrogen stock) 0.5% SDS Always add PMSF fresh before use | |||
| DNA elution Buffer | |||
| 50 mM NaHCO3 1% SDS | |||
| Table of specific reagents and equipment: | |||
| Glutaraldehyde (EM grade) | Sigma-Aldrich | G5882-10x10ml | |
| Motorized pellet mixer | VWR international | V8185-904 | |
| Protease inhibitor | Roche Group | 11873580001 | |
| PMSF | Sigma-Aldrich | 78830 | |
| Superase-in | Ambion | AM2696 | |
| Bioruptor | Diagenode | UCD-200 | |
| Falcon tubes (for sonication) | Corning | 430790 | |
| Proteinase K | Ambion | AM2546 | |
| PCR purification kit | Qiagen | 28106 | |
| C-1 magnetic beads | Invitrogen | 65002 | |
| PMSF | Sigma-Aldrich | P7626-25G | |
| DynaMag-15 magnet | Invitrogen | 123-01D | |
| DynaMag-2 magnet | Invitrogen | 123-21D | |
| MIRNeasy mini kit | Qiagen | 217004 | |
| Rnase H | Epicentre Biotechnologies | R0601K | |
| Rnase A | Sigma-Aldrich | R4875-100MG | |
| Phase Lock Gel Heavy | 5 PRIME | 2302810 | |
| Trizol | Invitrogen | 15596-018 | |
| Phenol:chloroform:Isoamyl | Invitrogen | 15593-031 | |
| Chloroform | Ricca | RSOC0020-1C | |
| GlycoBlue | Ambion | AM9515 | |
| Glycine | JT Baker | 4057-06 | |
| PBS, pH 7.4 | Invitrogen | 10010-049 | |
| Elution Buffer (EB) | Qiagen | 19086 | |
| 20x SSC | Invitrogen | 15557-036 | |
| 10% SDS | Invitrogen | 15553-027 | |
| DNA-free | Ambion | AM1906 | |
| Buffer kit | Ambion | AM9010 | |
| Formamide | Invitrogen | 15515-026 | |
Références
- Koziol, M. J., Rinn, J. L. RNA traffic control of chromatin complexes. Curr. Opin. Genet. Dev. 20, 142-148 (2010).
- Mercer, T. R., Dinger, M. E., Mattick, J. S. Long non-coding RNAs: insights into functions. Nat. Rev. Genet. 10, 155-159 (2009).
- Rinn, J. L. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 129, 1311-1323 (2007).
- Zhao, J., Sun, B. K., Erwin, J. A., Song, J. J., Lee, J. T. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science. 322, 750-756 (2008).
- Kelley, R. L. Epigenetic spreading of the Drosophila dosage compensation complex from roX RNA genes into flanking chromatin. Cell. 98, 513-522 (1999).
- Pandey, R. R. Kcnq1ot1 antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation. Mol. Cell. 32, 232-246 (2008).
- Wang, K. C. A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression. Nature. 472, 120-124 (2011).
- Khalil, A. M. Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression. Proc. Natl. Acad. Sci. U.S.A. 106, 11667-11672 (2009).
- Zhao, J. Genome-wide identification of polycomb-associated RNAs by RIP-seq. Mol. Cell. 40, 939-953 (2010).
- Meller, V. H., Wu, K. H., Roman, G., Kuroda, M. I., Davis, R. L. roX1 RNA paints the X chromosome of male Drosophila and is regulated by the dosage compensation system. Cell. 88, 445-457 (1997).
- Franke, A., Baker, B. S. The rox1 and rox2 RNAs are essential components of the compensasome, which mediates dosage compensation in Drosophila. Mol. Cell. 4, 117-122 (1999).
- Gupta, R. A. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature. 464, 1071-1076 (2010).
- Tsai, M. C. Long noncoding RNA as modular scaffold of histone modification complexes. Science. 329, 689-693 (2010).
- Zappulla, D. C., Cech, T. R. RNA as a flexible scaffold for proteins: yeast telomerase and beyond. Cold Spring Harb. Symp. Quant. Biol.. 71, 217-224 (2006).
- Nagano, T. The Air noncoding RNA epigenetically silences transcription by targeting G9a to chromatin. Science. 322, 1717-1720 (2008).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature. 32, 623-626 (2002).
- Chu, C., Qu, K., Zhong, F. L., Artandi, S. E., Chang, H. Y. Genomic Maps of Long Noncoding RNA Occupancy Reveal Principles of RNA-Chromatin Interactions. Mol. Cell. , (2011).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5, 877-879 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon