Method Article
Хроматина Изоляция очистки РНК (чирпа)
В этой статье
Резюме
CHIRP это новый и быстрый метод для отображения геномных участков связывания длинных некодирующих РНК (lncRNAs). Метод использует специфику антисмысловые олигонуклеотиды плитки, чтобы перечисление lncRNA связанные сайты генома.
Аннотация
Длинные РНК некодирующих являются ключевыми регуляторами хроматина государств для важных биологических процессов, таких как дозовой компенсации, печать, и развития генной экспрессии 1,2,3,4,5,6,7. Недавнее открытие тысяч lncRNAs в связи с конкретной модификации хроматина комплексов, таких как Polycomb Репрессивные Комплекс 2 (PRC2), который является посредником гистона H3 лизин 27 триметилирование (H3K27me3), предлагает широкую роль для многочисленных lncRNAs хроматина в управлении государства в конкретных генов мода 8,9. Хотя некоторые lncRNAs, как считается, работают в странах СНГ на соседние гены, другие lncRNAs работать в транс, чтобы регулировать отдаленно расположенных генов. Например, у дрозофилы lncRNAs roX1 и roX2 связывают многочисленные области на Х-хромосоме мужских клеток и имеют решающее значение для дозовой компенсации 10,11. Тем не менее, точные места их сайтов связывания не известны в высоком разрешении. Кроме того, человек lncRNA Hotair может повлиять на размещение PRC2 чundreds генов генома 3,12,13, но как специфика достигается не ясна. LncRNAs может также служить в качестве модульных лесов на работу сборки нескольких белковых комплексов. Классический транс-действующие РНК леса является TERC РНК, которая служит в качестве шаблона и леса для теломеразы комплекс 14; Hotair может также служить каркасом для PRC2 и H3K4 деметилазы комплекс 13.
Предыдущие исследования отображения РНК на размещение хроматина выявили существенные идеи 15,16, но только на одного локуса гена одновременно. Размещение сайтов наиболее lncRNAs не известны, и роль в регуляции lncRNAs хроматина были в основном выведены из косвенных последствий возмущение lncRNA. Так же, как хроматин иммунопреципитации следует микрочипов или глубокой последовательности (Chip-чипа или чип-далее, соответственно) значительно улучшило наше понимание белок-ДНК взаимодействий на геномную масштаба, здесь мы проиллюстрируем recenTLY опубликованы стратегия для отображения длительный размещение РНК генома с высоким разрешением 17. Этот метод, хроматина Выделение РНК очистки (чирпа) (рис. 1), основаны на близости захвата целевой lncRNA: хроматин комплекса плитки антисмысловых олигонуклеотидов, которые затем генерирует карту генома сайты связывания с разрешением в несколько сотен баз Высокая чувствительность и низкий фон. CHIRP распространяется на многие lncRNAs, потому что проект сродства-зондов является простым, учитывая последовательность РНК и не требует знания структуры РНК или функциональных областей.
протокол
1. Зонд Design
Дизайн антисмысловые ДНК плитки зонды для селективного извлечения РНК мишени чириканье.
- Дизайн антисмысловые олиго зондов с помощью онлайн-конструктора зонд на singlemoleculefish.com 18.
- Используйте следующие параметры: количество проверок = 1 зонд / 100 б.п. длины РНК; 2) Целевая GC% = 45, 3) олигонуклеотидов длиной = 20, 4) Расстояние длина = 60-80. Перерыв на сегменты РНК, если слишком долго для дизайнера. Пропустить регионов повторяет или обширные гомологии.
- Заказ антисмысловые ДНК-зондов с BiotinTEG на 3-премьер конца.
- Этикетка датчиков в зависимости от их позиции по РНК. Разделите их на две бассейнов, так что "даже" пул содержит все зонды 2 нумерации, 4, 6 и т.д., и "нечетных" пул содержит датчики нумерацию 1, 3, 5 и т.д. Развести бассейн зондов до 100 мкм, концентрация и хранить при -20 ° C.
- Все эксперименты должны быть выполнены с использованиеми бассейнов, которые служат в качестве внутреннего контроля друг за друга. Реальная РНК-зависимой сигнал будет присутствовать обоих бассейнов, в то время как зонд конкретных шумов будет уникальным для каждого бассейна. Это относится как к CHIRP-КПЦР и чирпа-след.
2. Урожай клетки
Сбор клеток, который будет использован для эксперимента чириканье.
- Рост клеток в культуре ткани пластин или колбы до слияния. Полоскание с фосфатным буферным раствором (PBS) и один раз trypsinize. Угашайте трипсина с> 2x объем информации, пипетки вверх и вниз, чтобы выбить клеток и ресуспендируют в суспензии отдельных клеток. Перенесите все средства массовой информации и ресуспендированного клеток в 50 мл пробирки Falcon. 20000000 клетки, как правило, достаточное для одного образца чириканье.
- Спиновая клеток 800RCF в течение 4 минут. Аспирата СМИ и Ресуспендируйте 40 миллионов клеток в 40 мл PBS, объединить трубы, если необходимо. Спиновая клеток 800RCF в течение 4 минут. Переливать PBS, тщательно аспирации на угол оставшуюся жидкость.
- Выполните все шаги, при комнатной температуре.
- Подготовить 1% глутаральдегида при комнатной температуре PBS. Подготовить 10 мл на 10 миллионов клеток (0,4 мл 25% глутаральдегида акции ± 9,6 мл PBS). Глутаральдегид необходимо использовать свежие.
- Нажмите нижнюю часть трубы Сокол выбить шарики. Ресуспендируют осадок клеток в 1% глутаральдегида, начиная с небольшого объема, чтобы избежать куски, а затем вверх до полного объема. Обратить перемешать. Crosslink в течение 10 минут при комнатной температуре на конец в конец шейкер или ротатор.
- Утолить сшивания реакции с 1/10th объем 1,25 М глицин при комнатной температуре в течение 5 мин.
- Спиновая на 2000RCF в течение 5 мин. Аспирируйте супернатант и промывки гранул с 20 мл охлажденной PBS раз, вращающийся на 2000RCF в течение 5 мин.
- Аспирируйте и ресуспендируют Wоподзоленные, сшитые гранулы с 1 мл охлажденной PBS на 20 миллионов клеток. Передача каждого мл пробирку Эппендорфа и спина в 2000RCF течение 3 мин при 4 С. Удалить столько PBS это возможно, с кончика пипетки осторожно.
- Flash-заморозить клетки гранул в жидком азоте и хранят при температуре -80 ° C до бесконечности.
- Размораживайте гранулы клетки при комнатной температуре. Нажмите трудно вытеснить и перемешайте осадок клеток. Спином вниз осадок на 2000RCF в течение 3 мин при 4 ° C. Используйте острый 10 мкл наконечник пипетки, чтобы удалить оставшиеся PBS.
- На электронных весах (с точностью до 1 мг) таре массой пустой трубы Эппендорф (наши трубы весом 1,060 грамма очень последовательно). Взвесить каждый шарик и записать свой вес. Полный 15 см блюдо из сшитого клеток HeLa обычно весит 100 мг.
- Дополнение лизис буфера (10X масса гранул, например, 1 мл на 100 мг) с Фресч ингибитор протеазы, PMSF и Superase-в (см. прилагаемый список буфера). Все хорошо перемешать.
- Добавить в 10 раз объем дополнен лизис буфера в каждую пробирку и ресуспендируют осадок. Для небольших гранул <25 мг, ресуспендируют в 250 мкл дополнен лизис буфера. Подвеска должна быть гладкой. Если нет, то разделить суспензии в 500 мкл аликвоты и использовать моторизованные гранул смеситель, чтобы разбить сгустки. Немедленно приступить к обработке ультразвуком.
- Разрушать ультразвуком Клеточный лизат в Bioruptor в 15 мл пробирок Falcon. Используйте <1,5 мл лизат в каждой трубке, и для более быстрой обработки ультразвуком, разрушать ультразвуком не более двух труб одновременно.
- Разрушать ультразвуком в 4 ° С на водяной бане при максимальных настройках с 30 секунд, 45 секунд с интервалом импульса. Проверьте лизата каждые 30 мин. Продолжайте, пока sonicating Клеточный лизат больше не мутная. Это может занять всего 30 минут и больше, чем 4 часа. Номертруб, объем образца, температура ванны, а также период времени ультразвука повлияет, как долго этот процесс занимает. Трубы, скорее всего, разрушать ультразвуком с разной скоростью, так что объединить их вместе, каждые 30 минут и распространять в оригинальной трубы для обеспечения однородности. Примечание: глутаральдегида сшитый клетки занять значительно больше времени, чтобы разрушать ультразвуком, чем формальдегид эквиваленты.
- Когда лизат станет прозрачной, передают 5 мкл лизата в новую пробирку Эппендорфа. Добавить 90 мкл ДНК протеазы К (PK) буфера (см. список буферов) и 5 мкл ПК. Vortex перемешать и спином вниз кратко. Инкубировать 45 мин при 50 ° C.
- Извлечение ДНК Qiagen комплект очистки ПЦР. Элюции ДНК в 30 мкл буфера элюирования Qiagen (EB) и проверить ДНК размером в 1% агарозном геле. Если большая часть ДНК мазок 100-500 б.п., ультразвука является полным. Если нет, продолжаем разрушать ультразвуком.
- Центрифуга ультразвуком образцов 16100RCF в течение 10 мин при 4 ° C. Комбинат супернатантах, аликвоту в 1 мл пробы и флэш-замораживание в жидком nitrogeл. Хранить при температуре -80 ° C.
- Оттепель труб хроматина при комнатной температуре.
- Подготовка гибридизация буфера (см. список буферов, готовить 2 мл на мл хроматина). Vortex перемешать.
- Для типичного образца CHIRP использованием 1 мл лизат, удалить 10 мкл РНК вход и 10 мкл на вход ДНК и места в трубах Эппендорф. Хранить на льду до дальнейшего использования.
- Передача 1 мл хроматина до 15 мл трубку Falcon. Добавить 2 мл буфера гибридизации в каждую пробирку. В общем объеме <1,5 мл, использовать Эппендорф труб.
- Таяние зондов при комнатной температуре. Nanodrop зонды проверить сумму, если вы не использовали его в течение длительного времени (100 мкМ зонды должны спецификации ~ 500-600 нг / мкл с помощью одного параметра ДНК). Добавить соответствующий объем зондов для конкретных труб (100 пмоль зонд в 1 мл хроматина, 1 мкл 100 пмоль / мкл зонда на 1 мл хроматина).Все хорошо перемешать. Инкубировать при температуре 37 ° С в течение 4 часов при встряхивании.
- С 20 мин, оставшееся гибридизации, подготовить C-1 магнитных шариков (хранить при температуре 4 ° С). Используйте 100 мкл на 100 пмоль зондов. Промыть 1 мл unsupplemented лизис буфера в три раза, используя DynaMag-2 магнит полосы для отдельных бусы из буфера.
- Ресуспендируйте бусины в первоначальный объем лизирующего буфера; дополнение со свежими PMSF, PI и Superase в. После 4 часов гибридизации реакция завершена, добавить 100 мкл бисер в каждую пробирку. Все хорошо перемешать. Инкубировать при температуре 37 ° С в течение 30 мин при встряхивании.
- Подготовка промывочного буфера (5 мл на пробу). Vortex перемешать. Предварительно теплой до 37 ° C. Добавить PMSF перед использованием.
- Вымойте бисер с 1 мл промывочного буфера в пять раз. На первой стирки, использовать DynaMag-15 магнитной полосы на отдельные шарики, переливать, и ресуспендируют в 1 мл промывочного буфера. Передача объема до 1,5 мл трубки Эппендорф. Инкубировать при температуре 37 ° C при встряхивании в течение 5 мин.
- В последующие мойки, спином вниз каждую пробирку по minicentrifuge, Установить образца на DynaMag-2 магнитной полосой в течение 1 мин. Переливать образца, стереть любые капли с Kimwipe, ресуспендируют в 1 мл промывочного буфера. Инкубировать при температуре 37 ° C при встряхивании в течение 5 мин. Повторяйте в течение пяти общего стирок.
- Наконец стирки, ресуспендируют бисером хорошо. Удалить 100 мкл и отложите для выделения РНК. Резервный 900 мкл ДНК фракции. Поместите все трубы на DynaMag-2 магнитной полосой и удаления промывочного буфера. Спиновая все трубы вниз кратко, разместить их на магнит полосы. Удаление последнего бита буфера для промывки полностью с резким 10 мкл наконечником пипетки.
- Возьмите по 100 мкл шарик образцов и 10 мкл РНК вход образца. Добавить 85 мкл РНК ПК буфера рН 7,0 с РНК INPUT. Ресуспендируйте бисером в 95 мкл РНК 7,0 PK буфера рН. Добавьте 5 мкл К Proteinease и инкубировать при температуре 50 ° C в течение 45 минут с конца в конец трясутся.
- Кратко спином вниз все трубы икипятить образцы в течение 10 минут на тепловых блоков при температуре 95 ° C.
- Охладите образцы на льду, добавить 500 мкл TRIzol, вихревые энергично в течение 10 сек. Инкубируйте при комнатной температуре в течение 10 мин. Хранить при температуре от -80 ° C или перейдите к шагу 4.
- Добавить 100 мкл хлороформа TRIzol обработанных образцов. Vortex энергично в течение 10 сек. Спиновая на 16100RCF на настольных центрифуг в течение 15 мин при 4 ° C.
- Удалить ~ 400 мкл водного супернатант, избегая органических и интерфейс.
- Добавить 600 мкл (1,5 объема) 100% этилового спирта и хорошо перемешать. Спиновая образца через MIRNeasy мини столбцов. Вымойте 1x с RWT (MIRNeasy мини комплект), 2x с НПП в соответствии с протоколом производителя. Элюции 30 мкл нуклеазы свободного H 2 O (NFH 2 O).
- Лечить элюата РНК с ДНК свободными в соответствии с протоколом производителя. После завершения реакции, нагрева образца в течение 15 минут при температуре 65 ° C полностью инактивирует любые оставшиеся ДНКазы.
- Используйте 1 мкл РНК изолировать одну скважину для QRT-PCR анализ, чтобы подтвердить lncRNAпоиска. GAPDH часто используется в качестве отрицательного контроля.
- Подготовить буфер элюирования ДНК (см. список буферов), 150 мкл на пробу, в том числе ДНК INPUT.
- Добавьте 1 0μL РНКазы (10 мг / мл) и 10 мкл РНКазы Н (10 ед / мкл) в мл буфера элюции ДНК, и вихрь перемешать.
- Ресуспендируйте каждого образца из бисера в 150 мкл буфера элюции ДНК с РНКазы. (Вход Ресуспендируйте ДНК в 140 мкл) инкубировать при температуре 37 ° С в течение 30 мин при встряхивании.
- Отдельные бусины и супернатант на DynaMag-2 магнитной полосой. Удалить супернатант и добавить к помечены труб.
- Подготовка второго аликвоту ДНК буфера элюции 10 мкл РНКазы (10 мг / мл) и RNaseH (10 ед / мкл) точно так, как сделано в 8.2). Добавить 150 мкл каждого образца (в том числе ДНК INPUT), инкубировать, и удалите супернатант. Соберите все супернатант (Shoulг составляет ~ 300 мкл).
- Добавьте 15 мкл ПК для каждого образца. Инкубировать при температуре 50 ° С в течение 45 мин при встряхивании.
- Предварительно спином вниз желтой фазе блокировки гель в тубах (5PRIME). Передача образцов ДНК в фазе блокировки труб гель, и добавить 300 мкл PhOH: хлороформ: изоамиловый в образце. Встряхивать в течение 10 минут, и спином вниз на настольных центрифуг на 16100RCF в течение 5 мин при 4 ° C. Возьмите водный сверху (~ 300 мкл). Добавить 3 мкл GlycoBlue, 30 мкл NaOAc и 900 мкл 100% этанола. Все хорошо перемешать и хранить при температуре -20 ° С в течение ночи.
- Спиновая образцов 16100RCF в течение 30 мин при 4 ° C.
- Переливать супернатант тщательно. Добавить 1 мл 70% этанола и вихревые перемешать. Спиновая на 16100RCF в течение 5 мин. Удалить супернатант пипетки. Воздух сухой в течение 1 мин. Ресуспендируйте в 30 мкл EB.
- Образцы ДНК готовы для анализа КПЦР или подготовки высокой пропускной последовательности библиотек в соответствии с протоколом Illumina.
3. Кросс-ссылки клетки и собирать осадок клеток
Crosslink собранных клеток глутаральдегида сохранить РНК-хроматина взаимодействия и подготовки осадок клеток.
4. Лизиса клеток
Лизировать клетки сшиты подготовить Клеточный лизат.
5. Разрушение ультразвуком
Сдвига ДНК sonicating сшитый лизатов клеток.
6. CHIRP
Скрещиваться биотинилированного зонда ДНК в РНК и изолировать связанные хроматина.
7. Выделение РНК
Извлечение РНК часть из образцов для количественного CHIRP по QRT-PCR.
8. Выделение ДНК
Извлечение ДНК фракции CHIRP образцы определить по последовательности или количественно по КПЦР.
10. Представитель Результаты
Рисунок 1 изображает рабочего чириканье. Успешный эксперимент CHIRP обычно обогащает РНК-мишени значительно более неспецифической РНК. На рисунке 2 показано обогащение человеческой теломеразы РНК (TERC) из клеток HeLa по GAPDH, обильные клеточной РНК, которая служит в качестве отрицательного контроля. Большинство TERC РНК (~ 88%) в клетке были снесены, выполняя чириканье, в то время как только 0,46% из GAPDH РНК была получена, демонстрируя обогащения в ~ 200 раз. Неспецифические зондов, таких как датчики ориентации LacZ РНК, которая выражается не в клетках млекопитающих (рис. 2), могут быть использованы в качестве дополнительного отрицательного контроля.
Участки ДНК, как ожидается, связывать целевую lncRNA, как правило, обогащенный более негативный регионов, когда измеряется КПЦР. На рисунке 3 показано КПЦР проверки четырех Hotair связанных сайтов в первичных человеческих фибробластов крайней плоти, которые мы определили, выполняя CHIRP, далее в той же клеточной линии, в то время TERC и GAPDH ДНК сайтах себеRVE как отрицательный области управления. И "даже" и "нечетных" Зонд устанавливается дали сопоставимых обогащению ожидалось Hotair связанных сайтов по отрицательным регионов, отличительной чертой истинного lncRNA-сайты связывания.
Высокая пропускная способность секвенирования ДНК CHIRP обогащенного дает глобальную карту lncRNA-сайты связывания. Drosophila lncRNA roX2, как известно, взаимодействует с X-хромосомой в манере, которая необходима для компенсации дозы. На рисунке 4 показана roX2 обязательных профилей на участке Х-хромосомы. И "даже" и "нечетных" образцы были последовательно и их уникальные шумы были устранены, чтобы произвести учет перекрытия сигналов. Каждый «пик» здесь указывает на сильную сайте roX2 обязательным. Полный трек и список roX2 генов-мишеней были описаны в Чуйской и соавт. 2011 17.
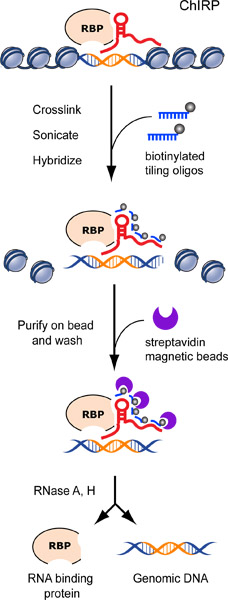
Рисунок 1. Блок-схема CHIRP процедурДюре. Хроматина сшивают в lncRNA: белковых аддуктов в естественных условиях Биотинилированные плитки зонды гибридизации с целевой lncRNA и хроматина комплексы очищают, используя магнитные шарики стрептавидином, а затем строгий стирок.. Мы элюировать lncRNA связанной ДНК или белки с коктейлем РНКазы и H. предполагаемого lncRNA обязательной последовательности схематически в оранжевый цвет. Ранее опубликованные в Чуйской и соавт. 2011 17.
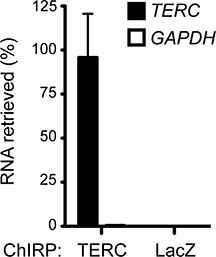
Рисунок 2. CHIRP обогащает для человека TERC РНК. TERC-asDNA зондов получить ~ 88% клеточной РНК и TERC обнаружить фермента. LacZ-asDNA зонды используются в качестве отрицательного контроля и получить ни РНК. Среднее + SD показаны. Ранее опубликованные в Чуйской и соавт. 2011 17.
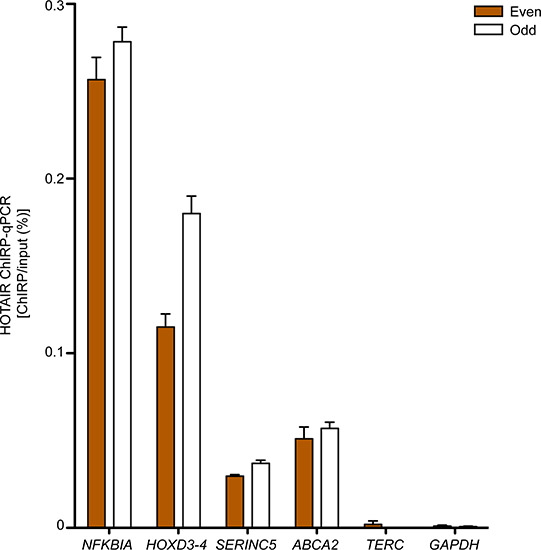
Рисунок 3. Hotair CHIRP-КПЦР в первичных человеческих дляЭскин фибробластов. NFKBIA, HOXD3-4, SERINC5 и ABCA2 регионы, которые взаимодействуют с Hotair. TERC и GAPDH служил отрицательный контроль. Среднее + SD показаны. Ранее опубликованные в Чуйской и соавт. 2011 17.
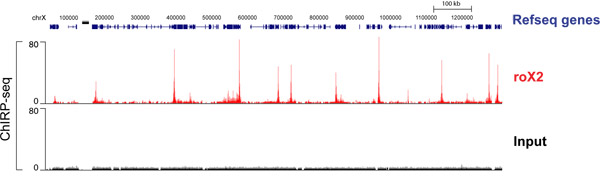
Рисунок 4. CHIRP, далее данные roX2 РНК в клетках дрозофилы SL2. "Even" и "нечетных" были последовательно отдельно, их объединения данных, чтобы отразить лишь общие пики в обоих. Объединенный трек показано на рисунке. Ранее опубликованные в Чуйской и соавт. 2011 17.
Обсуждение
Здесь мы описали CHIRP-след, метод отображения в естественных условиях lncRNA сайты связывания генома. Основные параметры для успеха раскол пула плитки олигонуклеотидных зондов и глутаральдегида сшивания. Конструкция близость-зондов является прямым дана последовательность РНК и не требует предварительных знаний о структуре РНК и функциональных областей. Наш успех roX2, TERC и Hotair - три различных РНК, а у двух видов - предполагает, что CHIRP, далее, скорее всего, обобщению многих lncRNAs. Как и во всех экспериментах, ухода и надлежащего управления, необходимые для интерпретации результатов. Различные lncRNA может потребоваться титрование условий и разумное изменение условий, таких как выбор различных датчиков близости или сшивающие агенты, могут выделить различные аспекты РНК-хроматина взаимодействия. Как ChIP, далее, не все события привязки обязательно функциональным, и необходимы дополнительные исследования, чтобы выяснить биологические последствия РНК оccupancy на хроматина. Тем не менее, мы предвидим множество интересных применений этой технологии для исследователей других хроматина связанных lncRNAs, число которых в 8,9 тысячи. Просто как чип-след открыл двери для генома исследования ДНК-белковых взаимодействий, CHIRP-последующие исследования "РНК интерактома" может выявить много новых направлений биологии.
Раскрытие информации
С. Чу и HY Чанг названы в качестве изобретателей на патентную заявку на основе этого метода.
Благодарности
Мы благодарим Т. Хунг, MC. Цай, О. Manor, Е. Сигал, М. Курода, Т. Swigut, И. Шестопалов для обсуждения. Поддерживаемый Агентством по науке, технологиям и исследованиям Сингапура (CC), NIH R01-R01 и CA118750-HG004361 (HYC) и Калифорнийского института регенеративной медицины (HYC). HYC является ранний ученый Карьера Медицинского института Говарда Хьюза.
Материалы
| Name | Company | Catalog Number | Comments |
| Buffer List: | |||
| Dissolve a pellet of complete protease inhibitor in 1 ml water as 50x stock. Make 100 mM PMSF in isopropanol (100x stock). Superase-in is used as 200x stock. Store all at -20 °C. | |||
| Lysis Buffer: | |||
| 50 mM Tris-Cl pH 7.0 10 mM EDTA 1% SDS Always add PMSF, P.I. and Superase-in fresh before use except when washing beads | |||
| Proteinase K Buffer (for DNA) | |||
| 100 mM NaCl 10 mM TrisCl pH 8.0 (For RNA use pH 7.0) 1 mM EDTA 0.5% SDS Add 5% by volume Proteainse K (Ambion AM2546 20 mg/ml) fresh before use | |||
| Hybridization Buffer | |||
| 750 mM NaCl 1% SDS 50 mM Tris-Cl pH 7.0 1 mM EDTA 15% formamide (store in the dark at 4 °C) Always add PMSF, P.I. and Superase-in fresh before use | |||
| Wash Buffer | |||
| 2x NaCl and Sodium citrate (SSC) (diluted from 20x SSC Invitrogen stock) 0.5% SDS Always add PMSF fresh before use | |||
| DNA elution Buffer | |||
| 50 mM NaHCO3 1% SDS | |||
| Table of specific reagents and equipment: | |||
| Glutaraldehyde (EM grade) | Sigma-Aldrich | G5882-10x10ml | |
| Motorized pellet mixer | VWR international | V8185-904 | |
| Protease inhibitor | Roche Group | 11873580001 | |
| PMSF | Sigma-Aldrich | 78830 | |
| Superase-in | Ambion | AM2696 | |
| Bioruptor | Diagenode | UCD-200 | |
| Falcon tubes (for sonication) | Corning | 430790 | |
| Proteinase K | Ambion | AM2546 | |
| PCR purification kit | Qiagen | 28106 | |
| C-1 magnetic beads | Invitrogen | 65002 | |
| PMSF | Sigma-Aldrich | P7626-25G | |
| DynaMag-15 magnet | Invitrogen | 123-01D | |
| DynaMag-2 magnet | Invitrogen | 123-21D | |
| MIRNeasy mini kit | Qiagen | 217004 | |
| Rnase H | Epicentre Biotechnologies | R0601K | |
| Rnase A | Sigma-Aldrich | R4875-100MG | |
| Phase Lock Gel Heavy | 5 PRIME | 2302810 | |
| Trizol | Invitrogen | 15596-018 | |
| Phenol:chloroform:Isoamyl | Invitrogen | 15593-031 | |
| Chloroform | Ricca | RSOC0020-1C | |
| GlycoBlue | Ambion | AM9515 | |
| Glycine | JT Baker | 4057-06 | |
| PBS, pH 7.4 | Invitrogen | 10010-049 | |
| Elution Buffer (EB) | Qiagen | 19086 | |
| 20x SSC | Invitrogen | 15557-036 | |
| 10% SDS | Invitrogen | 15553-027 | |
| DNA-free | Ambion | AM1906 | |
| Buffer kit | Ambion | AM9010 | |
| Formamide | Invitrogen | 15515-026 | |
Ссылки
- Koziol, M. J., Rinn, J. L. RNA traffic control of chromatin complexes. Curr. Opin. Genet. Dev. 20, 142-148 (2010).
- Mercer, T. R., Dinger, M. E., Mattick, J. S. Long non-coding RNAs: insights into functions. Nat. Rev. Genet. 10, 155-159 (2009).
- Rinn, J. L. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 129, 1311-1323 (2007).
- Zhao, J., Sun, B. K., Erwin, J. A., Song, J. J., Lee, J. T. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science. 322, 750-756 (2008).
- Kelley, R. L. Epigenetic spreading of the Drosophila dosage compensation complex from roX RNA genes into flanking chromatin. Cell. 98, 513-522 (1999).
- Pandey, R. R. Kcnq1ot1 antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation. Mol. Cell. 32, 232-246 (2008).
- Wang, K. C. A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression. Nature. 472, 120-124 (2011).
- Khalil, A. M. Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression. Proc. Natl. Acad. Sci. U.S.A. 106, 11667-11672 (2009).
- Zhao, J. Genome-wide identification of polycomb-associated RNAs by RIP-seq. Mol. Cell. 40, 939-953 (2010).
- Meller, V. H., Wu, K. H., Roman, G., Kuroda, M. I., Davis, R. L. roX1 RNA paints the X chromosome of male Drosophila and is regulated by the dosage compensation system. Cell. 88, 445-457 (1997).
- Franke, A., Baker, B. S. The rox1 and rox2 RNAs are essential components of the compensasome, which mediates dosage compensation in Drosophila. Mol. Cell. 4, 117-122 (1999).
- Gupta, R. A. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature. 464, 1071-1076 (2010).
- Tsai, M. C. Long noncoding RNA as modular scaffold of histone modification complexes. Science. 329, 689-693 (2010).
- Zappulla, D. C., Cech, T. R. RNA as a flexible scaffold for proteins: yeast telomerase and beyond. Cold Spring Harb. Symp. Quant. Biol.. 71, 217-224 (2006).
- Nagano, T. The Air noncoding RNA epigenetically silences transcription by targeting G9a to chromatin. Science. 322, 1717-1720 (2008).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature. 32, 623-626 (2002).
- Chu, C., Qu, K., Zhong, F. L., Artandi, S. E., Chang, H. Y. Genomic Maps of Long Noncoding RNA Occupancy Reveal Principles of RNA-Chromatin Interactions. Mol. Cell. , (2011).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5, 877-879 (2008).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены