Method Article
RNA 정제에 의한 염색질 절연 (처프)
요약
처프 긴 noncoding RNAs의 게놈에 바인딩 사이트 (lncRNAs)를 매핑하는 소설 및 신속한 기술입니다. 방법은 lncRNA 바인딩된 게놈 사이트의 열거를 허용하는 안티 센스가 원래의 oligonucleotides의 특이성을 이용합니다.
초록
긴 noncoding의 RNAs에는 투여 보상, 각인, 그리고 발달 유전자 발현 1,2,3,4,5,6,7 같은 중요한 생물 학적 과정 동안 염색질 상태 중 핵심 규제입니다. mediates 히스톤 H3 라이신 27 trimethylation (H3K27me3)가 유전자에 특정한에서 염색질 상태를 관리하는 수많은 lncRNAs에 대한 광범위한 역할을 제시하는 등 Polycomb 억압 단지 2 (PRC2)와 같은 특정 염색질의 수정 단지와 협회의 lncRNAs 수천명의 최근 발견 패션 8,9. 일부 lncRNAs은 인근의 유전자에있는 CIS에서 일할 것으로 생각되고 있지만, 다른 lncRNAs가 먼에 위치한 유전자를 조절하는 트랜스으로 작동합니다. 예를 들어, Drosophila lncRNAs roX1과 남성 세포 X의 염색체에 roX2 구속 수많은 지역 및이 복용량 보상 10,11 위해 중요합니다. 그러나 그들의 구속력이 사이트의 정확한 위치는 높은 해상도로 알려져 있지 않습니다. 마찬가지로, 인간 lncRNA HOTAIR은 H에 PRC2 인 영향을 미칠 수유전자 게놈 차원 3,12,13,하지만 어떻게 특이성이 이루어지면의 undreds가 불분명합니다. LncRNAs 또한 여러 단백질 단지의 어셈블리를 채용한 모듈형 공사장 공중 발판이 될 수 있습니다. 클래식 트랜스 연기 RNA 비계 14 복잡한 telomerase를위한 템플릿 및 발판 역할 TERC RNA이며 HOTAIR도 PRC2위한 발판와 13 단지 H3K4 demethylase로 검색할 수 있습니다.
염색질에서 RNA의 인지도를 선행 연구는 상당한 통찰력에게 15,16을 보여주지만, 한 번에 하나의 유전자 현장에있다. 대부분의 lncRNAs의 인 사이트는 알려져 있지되며, 염색질 조절에 lncRNAs의 역할은 주로 lncRNA의 섭동의 간접적인 영향으로부터 유추되었습니다. 염색질의 immunoprecipitation는 microarray 또는 깊은 시퀀싱 (각각 칩 칩 또는 칩 seq) 뒤에 것처럼 크게 게놈 규모의 단백질-DNA 상호 작용에 대한 우리의 이해를 개선하고있다, 우리가 recen을 보여주는tly 고해상도 17 살의 긴 RNA의 인 게놈 전체를 매핑하기위한 전략을 발표했다. 다음과 함께 수백 개의 기지의 해상도 게놈 바인딩 사이트의지도를 생성하는 안티 센스-oligos을 기와로 염색질 복합 : RNA의 정제 (처프) (그림 1)에 의해이 메소드는, 염색질 격리는 대상 lncRNA의 친화 캡쳐를 기반으로 고감도와 배경 쌉니다. 친화력 - 프로브의 설계는 RNA 시퀀스 주어진 간단하고 RNA의 구조 또는 기능적 도메인에 대한 지식을 필요로하지 않기 때문에 처프 다수 lncRNAs에 적용됩니다.
프로토콜
1. 프로브 디자인
디자인 안티 센스 DNA는 짹짹에 의해 RNA 대상의 선별 검색을 위해 프로브를 기와.
- 에서 온라인 프로브 디자이너를 사용하여 설계 안티 - 감각의 oligo 프로브 singlemoleculefish.com 18.
- 이러한 매개 변수를 사용하여 프로브 RNA의 길이 = 1 프로브 / 100 BP의 수 2) 대상의 GC % = 45; 3) Oligonucleotide 길이 = 20; 4) 간격의 길이 = 60-80을. 너무 오래 경우 디자이너에 대한 세그먼트로 RNA 봐라. 반복이나 다양한 상동 지역이 생략하십시오.
- 3 소수 끝에 BiotinTEG과 주문 안티 - 감각의 DNA 프로브.
- RNA를 따라 자신의 위치에 따라 라벨 프로브. "심지어는"수영장이 모든 프로브에게 번호 2, 4, 6, 등등을 포함하고 "이상한"수영장 1 번호 프로브, 3, 5를 포함 있도록하는 두 개의 수영장으로 그들을 분리 등 100 μm의 농도에 탐사선의 수영장을 희석하고 -20 ° C.에 저장
- 모든 실험 사용하여 수행할 수 있습니다서로에 대한 내부 통제의 역할을 둘 다 풀. 프로브 특정 소음 각 풀에 고유 될하면서 리얼 RNA-의존 신호는 두 개의 수영장 선물이 될 것입니다. 이것은 짹짹-qPCR와 짹짹-seq 모두에 적용됩니다.
2. 수확 셀
짹짹 실험에 사용됩니다 세포를 수집합니다.
- 조직 배양 플레이트 또는 confluency에 flasks에서 세포를 성장. 인산염과 헹굼 한번 식염수 (PBS)을 버퍼링하고 trypsinize. 세포를 이동시키다 및 단일 세포 현탁액으로 resuspend 아래로, 미디어> 2X 볼륨과 피펫을 트립신을 끄다합니다. 50 ML 팔콘 튜브로 모든 미디어 및 resuspended 세포를 전송합니다. 20,000,000 세포는 일반적으로 한 짹짹 샘플 충분합니다.
- 4 분 동안 800RCF에서 세포를 봐. 필요한 경우 PBS 40 ML에서 대기음 매체와 resuspend 40,000,000 세포는 튜브를 결합. 4 분 동안 800RCF에서 세포를 봐. 가만히 따르다 PBS는 조심스럽게 각도 남은 액체에 기음.
- 실온에서 모든 단계를 수행합니다.
- 방 온도를 PBS에 1 % 글루 타 알데히드를 준비합니다. 10000000 전지 (0.4 ML 25 % 글루 타 알데히드 주식 + 9.6 ML PBS) 당 10 ML을 준비합니다. 글루 타 알데히드는 신선한 사용해야합니다.
- 알약을 꺼내려 팔콘 튜브의 바닥을 누릅니다. Resuspend 세포 펠렛은 1 % 글루 타 알데히드에 청크를 피하기 위해 작은 볼륨으로 시작, 그때 전체 볼륨까지 맨. 믹스하는 반전. 엔드 - 투 - 엔드 흔드는 또는 회전에 실온에서 10 분 대한 Crosslink.
- 5 분을위한 상온에서 1.25 M 글리신의 1/10th 볼륨 가교 반응을 끄다.
- 5 분 동안 2000RCF에서 봐. 20 ML은 5 분 동안 2000RCF에서 방적 한번 PBS를 차게로 뜨는 및 세차 펠렛을 기음.
- W를 기음과 resuspend재를 떨어서, 교차 연결된 펠릿 20,000,000 셀 당 한 ML 냉장 PBS로. 주의 깊게 피펫 팁으로 가능한 한 많은 PBS를 제거 4 C.에서 3 분 동안 2000RCF에 Eppendorf 튜브와 스핀 각 ML을 전송합니다.
- -80에서 액체 질소와 저장소에있는 세포 알약을 플래시 동결 ° C 무기한.
- 실온에서 냉동 세포 알약을 녹여. 세포 펠렛을 이동시키다하고 혼합하기 위해 최선을 누릅니다. 4에 3 분 ° C.에 대해 2000RCF에 펠렛을 던가 모든 남아있는 PBS를 제거하는 샤프 10 μl 피펫 팁을 사용합니다.
- 전자 잔액 (1mg까지 정확하게) 비어있는 Eppendorf 튜브의 살갈퀴 질량에서 (우리의 튜브는 매우 일관되게 1.060 그램 무게). 각 펠렛를 저울질하고 체중을 기록합니다. 가교 HELA 세포의 전체 15cm 요리는 일반적으로 100 MG 무겁죠.
- fres와 보충의 용해 완충액 (펠렛의 10X 질량, 100mg에 대한 예 : 1 ML)H 단백 분해 효소 억제제, PMSF와 Superase 인은 (첨부된 버퍼 목록을 참조). 잘 섞는다.
- 각각의 튜브로 10X 볼륨 보충 용해 완충액을 넣고 펠렛을 resuspend. 작은 알약 들어 <250 μl 보충 용해 완충액에 resuspend 25 밀리그램. 서스펜션은 부드럽게해야합니다. 그렇지 않다면, 500 μl aliquots으로 정학을 분할하고 대단히 짧은 시간을 깨고 동력 펠렛 믹서를 사용합니다. sonication 즉시 진행합니다.
- 15 ML 팔콘 튜브에 Bioruptor의 세포 lysate를 Sonicate. 각 튜브에 <1.5 ML의 lysate를 사용하여 빠른 sonication을 위해, 시간에 더 이상 둘을 제외 튜브 sonicate.
- 펄스 간격에서 45 초 30 초 최고 설정에서 4 ° C의 물을 욕조에 Sonicate. 매 30 분 lysate 확인합니다. 세포 lysate가 더 이상 흐린되지 않습니다 때까지 sonicating 계속합니다. 이것은 빠르면 30 분, 많은 4와 같은 시간이 걸릴 수 있습니다. 번호튜브, 샘플 볼륨, 목욕 온도 및 sonication 시간의 기간은 프로세스가 걸리는 시간에 영향을줍니다. 튜브 가능성이 서로 다른 속도로 sonicate 때문에 함께 균등을 보장하기 위해 원래의 튜브에 매 30 분 및 재배포 그들을 풀 것이다. 참고 : 글루 타 알데히드 - 가교 세포가 포름 알데히드 등가물보다 sonicate 데 시간이 더 크게 가져가라.
- lysate 분명 켜면, 새로운 Eppendorf 튜브에 5 μL lysate를 전송합니다. 90 μL의 DNA 프로 테아제 K (PK) 버퍼 (버퍼리스트 참조) 5 μL PK를 추가합니다. 와동은 믹스 앤 간단히 다운 스핀합니다. 50 45 분 동안 품어 ° C.
- Qiagen PCR 정화 키트에 DNA를 추출합니다. Elute의 30 μL Qiagen의 용출 버퍼 (EB)의 DNA와 1 % 아가로 오스 겔에 DNA의 크기를 확인합니다. DNA의 비방의 일괄 100-500 BP 경우, sonication이 완료됩니다. 그렇지 않을 경우 sonicate로 진행합니다.
- 4에서 10 분 동안 16100RCF에서 sonicated 샘플을 원심 ° C. 액체 nitroge에 1 ML 샘플 및 플래시 동결로 supernatants, 나누어지는을 결합N. -80시 스토어 ° C.
- 상온에서 염색질의 해동 튜브.
- (염색질의 ML 당 2 ML 준비, 버퍼리스트 참조) 하이브리드화 버퍼를 준비합니다. 혼합할 수있는 와동.
- lysate 1 ML을 사용하여 일반적인 짹짹 샘플의 경우 Eppendorf 튜브에있는 DNA의 입력과 장소에 대한 RNA의 입력과 10 μL 10 μL를 제거합니다. 추가 사용까지 얼음에 보관.
- 15 ML 팔콘 튜브 1 ML의 염색질을 전송합니다. 각 튜브에 두 ML의 하이브리드화 버퍼를 추가합니다. 총 볼륨의 <1.5 ML는 Eppendorf 튜브를 사용합니다.
- 실온에서 프로브를 녹여. 당신은 오랫동안 그것을 사용하지 않은 경우에는 금액을 확인 Nanodrop 프로브 (100 μm의 프로브해야 사양 ~ 단일 스트랜드의 DNA 설정을 사용 500-600 NG / μl). 특정 튜브 (1 ML 염색질 당 100 pmol 프로브, 1 ML 염색질 당 100 μL / pmol 프로브 1 μL)에 탐사선의 적절한 볼륨을 추가합니다.잘 섞는다. 떨고있는 4 시간 동안 37 ° C에서 품어.
- 20 분이 하이브 리다이 제이션을 위해 남아있는 상태에서 C-1 자석 구슬 (4 ° C에 저장) 준비합니다. 프로브의 100 pmol 당 100 μL를 사용하십시오. 한 ML unsupplemented 용해 버퍼와 버퍼에서 별도의 구슬로 DynaMag-2 자석 스트립을 사용하여 세 번 .. 내사랑.
- 용해 완충액의 원래 볼륨에서 Resuspend 구슬, 신선한 PMSF, PI 및 Superase 인과 보충. 4 시간의 하이브 리다이 제이션 반응이 완료되면 각각의 튜브에 100 μL 구슬을 추가합니다. 잘 섞는다. 떨고있는 30 분 동안 37 ° C에서 품어.
- (샘플 당 5 ML) 버퍼 씻어 준비합니다. 혼합할 수있는 와동. 37까지 사전 따뜻한 ° C. 사용하기 전에 PMSF를 추가합니다.
- 한 ML 세척 버퍼 다섯 번으로 구슬을 씻는다. 첫 번째 세척에서 1, ML 세척 버퍼에 별도의 구슬, 가만히 따르다, 그리고 resuspend에 DynaMag-15 마그네틱 스트립을 사용합니다. 1.5 ML Eppendorf 튜브에 볼륨을 전송합니다. 5 분 동안 흔들어으로 37 ° C에서 품어.
- 이후 세차장에서 minicentrifuge 각 튜브를 돌려서, 1 분 DynaMag-2 마그네틱 스트립에 샘플을 설정합니다. 가만히 따르다 예제 1 ML 세척 버퍼에 resuspend Kimwipe로 모든 drips 닦아냅니다. 5 분 동안 흔들어으로 37 ° C에서 품어. 다섯 총 세차장에 대해 반복합니다.
- 마지막으로 세척 장에서 잘 구슬 resuspend. 100 μL를 제거하고 RNA 격리를 위해 별도로 설정합니다. DNA 분획을위한 준비 900 μL. DynaMag-2 마그네틱 스트립에있는 모든 튜브를 삽입하고 세차 버퍼를 제거합니다. 간단히 아래 모든 튜브 던가, 자석 스트립에 넣어. 날카로운 10 μl 피펫 팁으로 완전히 세척 버퍼의 마지막 비트를 제거합니다.
- 100 μL 비드 샘플과 10 μL RNA 입력 샘플을 가져가라. 입력 RNA 85 μL RNA PK 버퍼 산도 7.0을 추가합니다. 95 μL RNA PK 버퍼 산도 7.0의 Resuspend 구슬. 엔드 - 투 - 엔드 떨고있는 45 분 동안 50 ° C에서 5 μL Proteinease의 K 및 부화를 추가합니다.
- 간단히 모든 튜브를 돌려서95에서 열 블록에 10 분 동안 샘플을 끓여서 ° C.
- 얼음에 샘플을 풀어, 10 초 동안 적극적으로 500 μL TRIzol, 와동을 추가합니다. 10 분 동안 상온에서 품어. -80시 쇼핑몰 ° C 또는 4 단계로 진행합니다.
- TRIzol 처리 샘플에 100 μL의 클로로포름을 추가합니다. 10 초에 대한 노골적인 소용돌이. 4 ° C.에 15 분 benchtop 원심 분리기에 16100RCF에 스핀
- 유기 및 인터페이스를 피하 ~ 400 μL 수성 표면에 뜨는을 제거합니다.
- 600 μL (1.5 볼륨) 100 % 에탄올을 넣고 잘 섞는다. MIRNeasy 미니 칼럼을 통해 샘플을 봐. RWT (MIRNeasy 미니 키트), 제조 업체의 프로토콜마다 RPE와 2X와 함께 1X 씻으십시오. 30 μL nuclease없는 H 2 O (nfH 2 O)로 Elute.
- RNA의 eluate 제조 업체의 프로토콜마다 DNA를 무료로 드셔보세요. 반응이 완료되면, 65에서 15 분 동안 시료를 가열 ° C가 완전히 남아있는 DNase를 inactivate 수 있습니다.
- 1 μL RNA를 사용 lncRNA을 확인 qRT-PCR 분석에 당 잘 격리검색. GAPDH는 종종 부정적인 컨트롤로 사용됩니다.
- DNA를 용출 버퍼 (버퍼리스트 참조), DNA의 입력을 포함한 샘플 당 150 μl를 준비합니다.
- 섞어 한 0μL RNase (10 밀리그램 / ML)와 10 μL RNase H (10 U / μl)의 DNA 용리 버퍼의 ML 당, 그리고 소용돌이를 추가합니다.
- RNases있는 DNA를 용출 버퍼 150 μL의 구슬 각각의 샘플을 Resuspend. (140 μL의 Resuspend의 DNA 입력) 떨고있는 30 분 동안 37 ° C에서 품어.
- 별도의 구슬과 DynaMag-2 마그네틱 스트립에 뜨는. 뜨는을 제거하고 레이블 튜브에 추가합니다.
- 10 μL RNase (10 밀리그램 / ML)와 RNaseH (10 U / μL) 그대로 8.2에서 수행)와 함께 DNA의 용출 버퍼의 두 번째 나누어지는을 준비합니다. 각 샘플 (DNA 입력 포함), 알을 품다 150 μL를 추가하고 뜨는 제거합니다. 모든 뜨는 (shoul를 수집D) ~ 300 μL 수 있습니다.
- 각 시료 15 μL PK를 추가합니다. 떨고있는 45 분 동안 50 ° C에서 품어.
- 노란색 위상 잠금 젤 튜브 (5PRIME)를 다운 사전 돌리다. 위상 잠금 젤 튜브에의 DNA 샘플을 전송, 300 μL PhOH 추가 샘플 당 Isoamyl : 클로로포름합니다. 10 분 동안 적극적으로 흔들, 4 ° C.에 5 분 대한 16100RCF에서 benchtop 원심 분리기에 다운 스핀 상단 (~ 300 μL)에서 수성을 가져가라. 3 μL GlycoBlue, 30 μL NaOAc, 900 μL 100 % EtOH를 추가합니다. 하룻밤 -20 ° C에서 잘하고 매장 섞는다.
- 4에서 30 분 동안 16100RCF에서 샘플을 던가 ° C.
- 신중하게 가만히 따르다 뜨는. 한 ML 70 % EtOH와 섞어 소용돌이를 추가합니다. 5 분 동안 16100RCF에서 봐. 피펫으로 뜨는을 제거합니다. 공기 1min 동안 건조. 30 μL EB에 Resuspend.
- DNA 샘플은 qPCR 또는 Illumina 프로토콜 당 높은 처리량 시퀀싱 라이브러리의 준비에 의한 분석을 위해 준비가되어 있습니다.
3. 크로스 링크 셀 및 셀 펠릿를 수집
Crosslink는 RNA-염색질 상호 작용을 유지하고 세포 펠렛을 준비하는 글루 타 알데히드와 세포를 수집했습니다.
4. 세포 용해
세포 lysate를 준비하는 가교 세포를 Lyse.
5. Sonication
가교 세포 lysates를 sonicating에 의한 전단의 DNA.
6. 짹짹
바운드 염색질을 RNA와 분리 biotinylated DNA 프로브를 잡종.
7. RNA 격리
qRT-PCR에 의해 quantitate하는 처프 샘플에서 RNA 분획을 추출합니다.
8. DNA의 격리
시퀀싱에 의해 식별하거나 qPCR에 의해 quantitate하는 처프 샘플에서 DNA 분획을 추출합니다.
10. 대표 결과
그림 1 짹짹의 흐름을 묘사. 성공 짹짹 실험은 일반적으로 상당히 아닌 특정 RNAs를 통해 대상 RNA를 enriches. 그림 2 GAPDH 이상 HELA 세포로부터 인간 telomerase RNA (TERC), 부정적인 제어 역할을 풍부한 세포 RNA의 농축을 보여줍니다. GAPDH RNA의 단지 0.46 %가 검색되었습니다 반면 세포에 존재 TERC RNAs (~ 88%)의 대부분은 ~ 200 배로 농축 계수를 보여주 처프를 수행 타고 내려오게되었다. 같은 포유 동물 세포 (그림 2)로 표현되지 않습니다 LacZ RNA를 타겟팅 프로브와 같은 특이 현상이 프로브가 추가로 부정적인 컨트롤로 사용할 수 있습니다.
대상 lncRNA를 바인딩 것으로 예상의 DNA 영역은 일반적으로 qPCR로 측정 부정적인 영역 통해 풍부합니다. 그림 3은 우리가 같은 세포 라인에서 짹짹-seq를 수행하여 결정하는 주요 인간 포피의 섬유아 세포에서 네번 HOTAIR 바인딩된 사이트 qPCR 검증을 보여줍니다, 반면 TERC와 GAPDH 유전자 사이트 SE부정적인 통제 지역으로 rve. 모두 "도"와 "이상한"탐사선은 부정적인 지역, 진정한 lncRNA 바인딩 사이트의 특징 이상 예상 HOTAIR 바인딩된 사이트 굴복 비교 농축을 설정합니다.
짹짹 강화 DNA의 높은 처리량 시퀀싱은 lncRNA 바인딩 사이트의 글로벌지도를 얻을. Drosophila lncRNA roX2은 복용량 보상에 필요한 방식으로 X-염색체와 상호 작용하는 것으로 알려져 있습니다. 그림 4는 X의 염색체의 일부분 이상 roX2 바인딩 프로필을 보여줍니다. 모두 "도"와 "이상"샘플은 순서가되어 그들의 독특한 소리가 중복되는 신호의 추적을 생산하기 위해 제거되었습니다. 각각의 "피크"는 여기 roX2 바인딩의 강력한 사이트를 나타냅니다. 전체 트랙과 roX2 대상 유전자의 목록은 추 외에 설명되어있다. 2,011 17가.
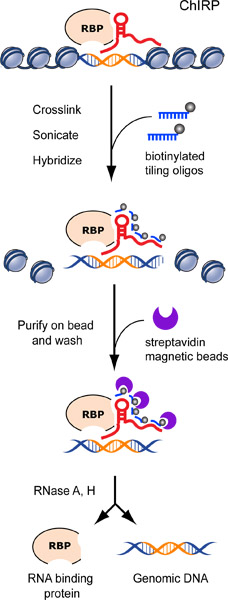
짹짹 proce의 그림 1. 흐름 차트dure. Biotinylated 기와 프로브는 lncRNA을 대상으로 hybridized되는 생체내에 단백질 부가물을하고, 염색질의 단지는 엄격한 세차장 뒤에는, 자기 streptavidin 비즈를 사용하여 정화됩니다. 염색질은 lncRNA에 가교됩니다. 우리는 putative lncRNA 바인딩 순서가 오렌지색으로 schematized되고 Rnase와 헤의 칵테일과 함께 lncRNA 바운드의 DNA 또는 단백질을 elute. 이전 추 외 년에 출판된. 2,011. 17
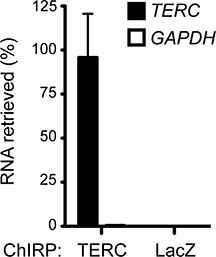
인간 TERC RNA 그림 2. 짹짹의 enriches. TERC-asDNA 프로브 세포 TERC RNA 및 탐지 GAPDH의 ~ 88 % 검색할 수 있습니다. LacZ-asDNA 프로브는 부정적인 컨트롤로 사용도 RNAs를 검색할 수 있습니다. 민 + SD가 표시됩니다. 이전 추 외 년에 출판된. 2,011. 17
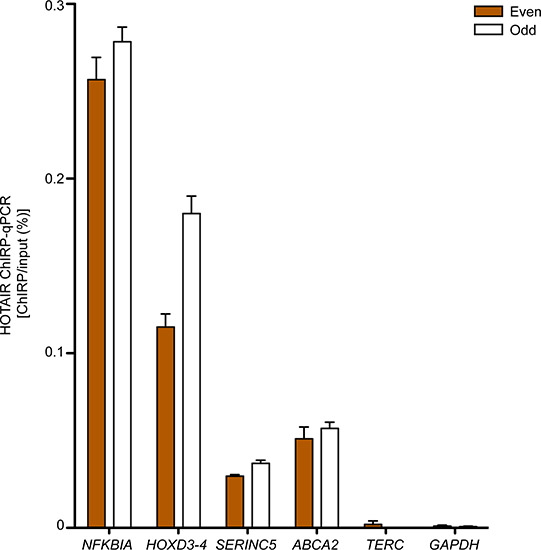
그림 3. 기본 인간의 HOTAIR 짹짹-qPCR을위한eskin의 섬유아 세포. NFKBIA, HOXD3-4, SERINC5 및 ABCA2가 HOTAIR와 상호 작용하는 지역입니다. TERC와 GAPDH는 부정적인 컨트롤을 역임했습니다. 민 + SD가 표시됩니다. 이전 추 외 년에 출판된. 2,011. 17
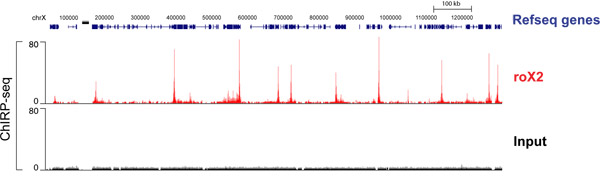
그림 4. Sl2 Drosophila 세포의 roX2 RNA의 짹짹-seq 데이터입니다. "비록"과 "이상한"가 별도로 순서가 있었던 것은 그들의 데이터는 모두 전용 일반 봉우리를 반영하도록 병합합니다. 병합된 트랙이 나타납니다. 이전 추 외 년에 출판된. 2,011. 17
토론
여기 짹짹-seq, 생체내 lncRNA 구속력이 사이트는 게놈 전체에 매핑하는 방법을 설명했다. 성공을위한 주요 파라미터는 oligonucleotide 프로브 및 글루 타 알데히드 crosslinking을 기와의 분할 수영장입니다. 친화력 - 프로브의 설계는 RNA 시퀀스 주어진 간단하고 RNA의 구조 또는 기능적 도메인에 대한 사전 지식이 필요하지 않습니다. 두 종족에 세보다는 다른 RNAs - - roX2, TERC 및 HOTAIR와의 성공 짹짹-seq 많은 lncRNAs 대한 가능성 generalizable 것을 제안합니다. 모든 실험과 마찬가지로, 관리 및 적절한 컨트롤이 결과를 해석해야합니다. 다른 lncRNA는 조건 적정과 같은 서로 다른 선호도 프로브 또는 crosslinkers의 선택과 같은 조건의 의식 변화를 요구할 수 있습니다, RNA-염색질 상호 작용의 다양한 측면을 강조할 수 있습니다. 칩 seq 마찬가지로, 모든 바인딩 이벤트가 반드시 기능적 있으며, 추가적인 연구는 RNA O의 생물 학적 결과를 확인하는 데 필요한염색질에 ccupancy. 그럼에도 불구하고, 우리는 수천 8,9의 현재 번호를 다른 염색질 - 관련 lncRNAs의 연구자들은이 기술의 많은 흥미로운 애플 리케이션을 예견한다. 칩 seq는 DNA-단백질 상호 작용의 게놈 차원의 탐험을 위해 문을 열었습니다 것처럼, "RNA interactome"의 짹짹-seq 연구는 생물학의 많은 새로운 길을 보여줄 수 있습니다.
공개
C. 추와 HY 장은이 방법을 바탕으로 특허 출원의 발명가로 지정됩니다.
감사의 말
우리는 T. 숙취, MC 감사합니다. 영, O. 매너 E. Segal, M. 구로다, T. Swigut 및 토론을위한 나 Shestopalov. 과학, 기술, 싱가포르 (CC), NIH R01-CA118750 및 R01-HG004361 (HYC) 및 재생 의료 (HYC)의 캘리포니아 공과 대학 연구 기관 지원. HYC는 하워드 휴즈 의학 연구소의 조기 채용 과학자이다.
자료
| Name | Company | Catalog Number | Comments |
| Buffer List: | |||
| Dissolve a pellet of complete protease inhibitor in 1 ml water as 50x stock. Make 100 mM PMSF in isopropanol (100x stock). Superase-in is used as 200x stock. Store all at -20 °C. | |||
| Lysis Buffer: | |||
| 50 mM Tris-Cl pH 7.0 10 mM EDTA 1% SDS Always add PMSF, P.I. and Superase-in fresh before use except when washing beads | |||
| Proteinase K Buffer (for DNA) | |||
| 100 mM NaCl 10 mM TrisCl pH 8.0 (For RNA use pH 7.0) 1 mM EDTA 0.5% SDS Add 5% by volume Proteainse K (Ambion AM2546 20 mg/ml) fresh before use | |||
| Hybridization Buffer | |||
| 750 mM NaCl 1% SDS 50 mM Tris-Cl pH 7.0 1 mM EDTA 15% formamide (store in the dark at 4 °C) Always add PMSF, P.I. and Superase-in fresh before use | |||
| Wash Buffer | |||
| 2x NaCl and Sodium citrate (SSC) (diluted from 20x SSC Invitrogen stock) 0.5% SDS Always add PMSF fresh before use | |||
| DNA elution Buffer | |||
| 50 mM NaHCO3 1% SDS | |||
| Table of specific reagents and equipment: | |||
| Glutaraldehyde (EM grade) | Sigma-Aldrich | G5882-10x10ml | |
| Motorized pellet mixer | VWR international | V8185-904 | |
| Protease inhibitor | Roche Group | 11873580001 | |
| PMSF | Sigma-Aldrich | 78830 | |
| Superase-in | Ambion | AM2696 | |
| Bioruptor | Diagenode | UCD-200 | |
| Falcon tubes (for sonication) | Corning | 430790 | |
| Proteinase K | Ambion | AM2546 | |
| PCR purification kit | Qiagen | 28106 | |
| C-1 magnetic beads | Invitrogen | 65002 | |
| PMSF | Sigma-Aldrich | P7626-25G | |
| DynaMag-15 magnet | Invitrogen | 123-01D | |
| DynaMag-2 magnet | Invitrogen | 123-21D | |
| MIRNeasy mini kit | Qiagen | 217004 | |
| Rnase H | Epicentre Biotechnologies | R0601K | |
| Rnase A | Sigma-Aldrich | R4875-100MG | |
| Phase Lock Gel Heavy | 5 PRIME | 2302810 | |
| Trizol | Invitrogen | 15596-018 | |
| Phenol:chloroform:Isoamyl | Invitrogen | 15593-031 | |
| Chloroform | Ricca | RSOC0020-1C | |
| GlycoBlue | Ambion | AM9515 | |
| Glycine | JT Baker | 4057-06 | |
| PBS, pH 7.4 | Invitrogen | 10010-049 | |
| Elution Buffer (EB) | Qiagen | 19086 | |
| 20x SSC | Invitrogen | 15557-036 | |
| 10% SDS | Invitrogen | 15553-027 | |
| DNA-free | Ambion | AM1906 | |
| Buffer kit | Ambion | AM9010 | |
| Formamide | Invitrogen | 15515-026 | |
참고문헌
- Koziol, M. J., Rinn, J. L. RNA traffic control of chromatin complexes. Curr. Opin. Genet. Dev. 20, 142-148 (2010).
- Mercer, T. R., Dinger, M. E., Mattick, J. S. Long non-coding RNAs: insights into functions. Nat. Rev. Genet. 10, 155-159 (2009).
- Rinn, J. L. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 129, 1311-1323 (2007).
- Zhao, J., Sun, B. K., Erwin, J. A., Song, J. J., Lee, J. T. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science. 322, 750-756 (2008).
- Kelley, R. L. Epigenetic spreading of the Drosophila dosage compensation complex from roX RNA genes into flanking chromatin. Cell. 98, 513-522 (1999).
- Pandey, R. R. Kcnq1ot1 antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation. Mol. Cell. 32, 232-246 (2008).
- Wang, K. C. A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression. Nature. 472, 120-124 (2011).
- Khalil, A. M. Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression. Proc. Natl. Acad. Sci. U.S.A. 106, 11667-11672 (2009).
- Zhao, J. Genome-wide identification of polycomb-associated RNAs by RIP-seq. Mol. Cell. 40, 939-953 (2010).
- Meller, V. H., Wu, K. H., Roman, G., Kuroda, M. I., Davis, R. L. roX1 RNA paints the X chromosome of male Drosophila and is regulated by the dosage compensation system. Cell. 88, 445-457 (1997).
- Franke, A., Baker, B. S. The rox1 and rox2 RNAs are essential components of the compensasome, which mediates dosage compensation in Drosophila. Mol. Cell. 4, 117-122 (1999).
- Gupta, R. A. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature. 464, 1071-1076 (2010).
- Tsai, M. C. Long noncoding RNA as modular scaffold of histone modification complexes. Science. 329, 689-693 (2010).
- Zappulla, D. C., Cech, T. R. RNA as a flexible scaffold for proteins: yeast telomerase and beyond. Cold Spring Harb. Symp. Quant. Biol.. 71, 217-224 (2006).
- Nagano, T. The Air noncoding RNA epigenetically silences transcription by targeting G9a to chromatin. Science. 322, 1717-1720 (2008).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature. 32, 623-626 (2002).
- Chu, C., Qu, K., Zhong, F. L., Artandi, S. E., Chang, H. Y. Genomic Maps of Long Noncoding RNA Occupancy Reveal Principles of RNA-Chromatin Interactions. Mol. Cell. , (2011).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5, 877-879 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유