Method Article
Visualização da ATP sintase Dimers na mitocôndria por Electron Cryo-tomografia
Neste Artigo
Resumo
Nós apresentamos um protocolo de como coletar e eletrônica processo de crio-tomograms de toda mitocôndrias. A técnica fornece informações detalhadas sobre a estrutura, função e organização de grandes complexos de proteínas de membrana em membranas biológicas nativas.
Resumo
Crio-tomografia de electrões é uma ferramenta poderosa na biologia estrutural, capaz de visualizar a estrutura tridimensional de amostras biológicas, tais como células, organitos, vesículas de membrana, ou vírus em detalhe molecular. Para conseguir isso, a amostra aquosa é rapidamente vitrificados em etano líquido, que preserva-lo em um estado congelado hidratado close-to-natal. Ao microscópio eletrônico, série de inclinação são registrados à temperatura do azoto líquido, a partir do qual tomografias 3D são reconstruídos. A razão sinal-para-ruído do volume tomográfico é inerentemente baixa. Reconhecível, características recorrentes são reforçadas por média subtomogram, pelo qual subvolumes individuais são cortados, alinhados e média para reduzir o ruído. Desta forma, 3D mapeia com uma resolução de 2 nm ou melhor pode ser obtida. Um ajuste de estruturas de alta resolução disponíveis para o volume 3D, em seguida, produz modelos atômicos de complexos de proteínas em seu ambiente nativo. Aqui nós mostramos como usamos elétron crio-tomography para estudar a organização in situ de grandes complexos de proteínas de membrana de mitocôndrias. Nós descobrimos que as sintases de ATP são organizadas em filas ao longo de dímeros ápices altamente curvas da cristas membrana interna, ao passo que o complexo I está distribuído de forma aleatória nas regiões da membrana de cada lado das filas. Por subtomogram média obtivemos uma estrutura da ATP sintase mitocondrial dímero dentro da membrana de cristas.
Introdução
As mitocôndrias são o poder de casas da célula. Através da conversão de um gradiente de protões electroquímico através da membrana mitocondrial interna na energia de ligação química, o ATP sintase mitocondrial produz a maior parte do ATP que impulsiona os processos celulares. A fim de compreender os mecanismos por trás de conversão de energia mitocondrial, precisamos determinar a estrutura da ATP sintase in situ, e para saber como ele é organizado e distribuído na membrana mitocondrial interna. Embora as estruturas de alta resolução da maior parte dos componentes da sintase ATP mitocondrial de 1-3 e mapas de baixa resolução de todo o complexo de 4 estão disponíveis, é importante estabelecer a estrutura e a conformação da enzima activa na membrana. A distribuição da ATP sintase, na membrana mitocondrial interna tem sido amplamente consideradas como aleatória, mas um início encontrando 5 e os nossos próprios resultados iniciais indicaram que 6 isto não é tele caso. Estudos subsequentes do nosso grupo e outros 7 confirmaram que o ATP sintase é disposta em linhas longas de dímeros ao longo das nervuras curvas de força nas cristas da membrana mitocondrial interna 8, enquanto que as bombas de protões na cadeia de transporte de electrões parece estar localizado em ambos os lados das linhas 9. Este arranjo tem implicações importantes para os mecanismos de conversão de energia mitocondrial.
A técnica que usamos para determinar esse arranjo é elétron crio-tomografia (crio-ET). Cryo-ET é atualmente o único método que oferece tridimensional (3D) volumes precisos de células, compartimentos celulares ou organelas na resolução molecular. Cryo-ET é particularmente adequado para o estudo de grandes complexos de membranas biológicas, pois as membranas aparecem com bom contraste e são fáceis de localizar em volumes tomográficos 3D.
Outros métodos para estudar a estrutura 3D de células ou organelles não fornecer detalhes molecular. Super-resolução microscopia de luz 10, 11 é excelente em revelar a posição ou a distância entre marcadores emissores de luz ligados a proteínas de interesse com uma precisão de dezenas de nm, mas não revela a estrutura da própria proteína, mesmo em baixa resolução . Microscopia eletrônica de transmissão de cortes seriados de 12 ou de imagem de bloco-face por microscopia eletrônica de varredura 13 de amostras biológicas de plástico incorporado proporcionam vistas de baixa resolução de volumes celulares, mas também não revelam detalhes molecular. Microscopia de força atômica 14 pode, em princípio, oferecer resolução molecular ou mesmo atômico, mas apenas na superfície dos objetos sobre um suporte atomicamente plana e sólida. Finalmente, a tomografia de raios X ou dispersão de 15 impulsos de raios-X a partir de lasers intensos de electrões livres é improvável para revelar a estrutura de grandes complexos, objectos aperiódicos tais como células completas ou organelos no m 16resolução olecular no futuro previsível. Assim, no momento, não há alternativa a Cryo-ET para estudos da estrutura 3D de células ou organelas com resolução nanométrica.
Cryo-ET é o método de escolha para a análise da estrutura e a conformação dos conjuntos de proteínas associadas à membrana, incluindo o complexo do poro nuclear 17, a espiga 18 da gripe complexos, e os flagelos proteínas de motor 22, 23 mas também a organização das células bacterianas inteiras 19 e a entrada de vírus patogénicos tais como o VIH em células de 20-23. Cryo-ET é inestimável para a visualização de proteínas filamentosas e suas interações na célula, incluindo os filamentos de actina 24 ou axonemas 25. A resolução pode ser aumentada para 2 nm ou melhor, pela subtomogram média de 26, segundo o qual subvolumes de repetidas, feições regulares são cortadas de um volume tomográfica e média por partícula única imagem protécnicas cessamento.
Cryo-ET envolve a aquisição de uma série de imagens de projeção de uma amostra fina (<250 nm) tomadas em diferentes ângulos de inclinação em um microscópio eletrônico de transmissão (TEM). A amostra deve ser fina para que os elétrons, que interagem fortemente com a matéria, estão espalhados por mais de uma vez. Espalhamento múltiplo faz com que as imagens resultantes de difícil interpretação e reduz o contraste. Imagens da área da amostra seleccionada estão alinhadas umas em relação às outras e projectado para um espaço 3D por um programa de computador apropriado, gerando um volume 3D do espécime. O alinhamento das imagens é auxiliada por marcadores fiduciais de ouro, os quais são misturados com a amostra antes da congelação. Idealmente marcadores fiduciais 10 ou mais uniformemente distribuídas deve estar presente em cada imagem para obter um bom alinhamento.
Para observar detalhes molecular, as amostras são mergulhar congelado em etano líquido, que preserva seu estado hidratado nativa. Congelamento em líquidoetano é tão rápido (~ 10 5 ° C / s) 27 que a água não se cristalizam, mas permanece em um estado como vidro vitrificado. Cristal de gelo danos formação de estruturas biológicas sensíveis. Como amostras biológicas sofrem de danos por radiação, há um limite para o número total de eventos de dispersão da amostra pode tolerar. As imagens são, assim, adquirido no modo de baixa dose: Uma área de interesse é identificado com pequeno aumento (1,500X) com uma dose de elétrons abaixo de 1 e - / nm 2 (modo de busca). A imagem é focalizada em uma ampliação maior, fora da área de interesse (modo de focagem). Só quando uma imagem é adquirida, a área de interesse, é irradiada com uma dose mais elevada de electrões (modo de exposição).
Aqui, apresentamos uma visão geral de como coletar e elétrons processo crio tomografias, usando ATP sintase dímeros na membrana mitocondrial interna como um exemplo. O protocolo a seguir descreve como preparar mitocôndria para crio-ET, como configurare recolha de uma série de inclinação com uma dose total de electrões específico, e como processar a série de inclinação para obter um volume 3D da área de interesse. Uma visão geral do procedimento é ilustrado na figura 1.
Protocolo
1: Preparação de mitocôndrias de células ou tecidos por centrifugação diferencial
Esta secção descreve um processo geral para o isolamento de mitocôndrias intactas a partir de vários organismos eucariotas. A precisão componentes do tampão e velocidades de centrifugação devem ser otimizados para cada tecido / espécies estudadas.
- Romper as células em tampão isotónico (por exemplo, sacarose 250 mM, HEPES 10 mM, pH 7,4), usando um moinho de vidro e de talão (micélio do fungo) 28, a digestão enzimática da parede celular (Saccharomyces cerevisiae), 29, de um homogeneizador de rolamento de esferas (única -cell eucariotos / cultura células / nematóides) 30, ou um liquidificador (tecidos animais ou vegetais) 31.
- Remover os detritos celulares por filtração através de musselina seguido de centrifugação a baixa velocidade (2000 xg, 4 ° C, 10 min).
- Recolhe sobrenadante e de sedimento mitocôndria por centrifugação a alta velocidade (9000 xg, 4 ° C, 10 min).
- Quando necessário, utilizar um passo de gradiente de densidade isotónico para purificação adicional da fracção mitocondrial 32.
2 Preparação de mitocôndrias para Electron Cryo-tomografia
A seção a seguir descreve como obter amostras congeladas hidratado para crio-ET. NOTA: O método envolve a utilização de azoto líquido extremamente frio e etano, que pode provocar queimaduras graves na pele. Óculos de segurança e luvas de proteção crio devem ser usados. Etano líquido, que também é inflamável, devem ser tratados de uma coifa.
- Ressuspender mitocôndrias peletizadas em 250 mM de trealose, tampão HEPES 10 mM a pH 7,4 a uma concentração de aproximadamente 5 mg / ml de proteína total.
- Grids EM holey carbono Glow-descarga, laterais de carbono para cima, em um dispositivo de vácuo de acordo com as instruções do fabricante.
- Liquefazer alguns mililitros de etano dirigindo uma corrente de gás de etano para o lado interior de um cool de azoto líquidorecipiente de alumínio ed.
- Misture suspensão fiducial ouro proteína A conjugada 1: 1 com suspensão mitocondrial e aplicar imediatamente 3 mL de uma grade EM brilho de descarga realizada em pinça.
- Coloque as pinças em um dispositivo de vitrificação, por exemplo, uma guilhotina caseira. Seque o excesso de líquido com uma fatia de papel de filtro (~ 5 segundos ou até que o líquido parar de espalhar) e mergulhar imediatamente a grade em etano líquido soltando o gatilho.
- Transferir a rede a partir de etano líquido em azoto líquido. Durante a transferência, remover o excesso de etano a partir da grelha com papel de filtro. Colocar a ponta de um pedaço em forma de cunha de papel de filtro no etano líquido. Como o etano líquido sobe, arraste suavemente a grade se o papel de filtro, mas mantê-lo abaixo da frente líquido. Etano líquido é removido da rede por acção capilar e uma vez todo o etano líquido foi removido, transferir imediatamente a grade em azoto líquido.
- Coloque a grade vitrificados em uma caixa de armazenamento de rede, umnd armazenar sob azoto líquido para posterior utilização como apropriado.
3 Gravação de Tomographic Tilt Series
A seção a seguir descreve como configurar e coletar uma série de inclinação tomográfica de uma mitocôndria com um microscópio eletrônico de Polara equipado com um filtro de energia pós-coluna e câmera CCD. Protocolos similares são usados com todos os crio microscópios eletrônicos equipados com câmeras CCD ou detectores de elétrons diretos.
- Alinhe o Microscópio
- Insira teste espécime por exemplo, grafite e as ilhas de ouro em filme de carbono holey.
- Selecione o modo de pesquisa no sistema de baixa dose do microscópio.
- Trazer amostra para a altura eucentric. Este é o ponto de movimento xy mínima ao inclinar o suporte de amostras. Centralize um ponto de interesse a 0 ° de inclinação, incline a fase de 20 ° e recentrar a ponto de alterar o z-altura. Retornar para 0 ° e repita até desvio lateral é minimizado.
- Selecionemodo de exposição. Escolha ampliação desejada para a coleta de uma tomografia (por exemplo, 25,000X no detector ≈ 0,6 nm espécime tamanho do pixel).
- Escolha a abertura do condensador pequeno (50-70 mm) e selecione um tamanho de ponto e intensidade do feixe de modo que o feixe é apenas mais largo que o dispositivo de imagem e dá uma leitura de pixel de 60 e - / pixel (CCD) ou 14 e - / pixel / seg (detector eletrônica direta, modo de contagem).
- Centro de abertura do condensador.
- Encontre Gaussian por exemplo foco, ponto de contraste mínimo. Redefinir leitura microscópio desfocar e pontos de articulação correta e centro de rotação de acordo com as instruções do fabricante.
- Disque desfocar desejado para a gravação de tomografia. NOTA: alta desfocagem (8 mm) aumenta o contraste, mas reduz a resolução, enquanto que a partir de desfocagem (2-4 mm) aumenta a resolução à custa de contraste.
- Ao longo de um buraco vazio, gerar uma nova referência de ganho e alinhar filtro de energia de acordo com o fabricante doinstruções.
- Alinhe modos de pesquisa e de exposição. No modo de exposição, centralizar um ponto de interesse e alternar para o modo de pesquisa. Seleccionar ampliação de 1,500X (0,033 uM / pixel da amostra no detector) e de desfocagem de 100 mM (para o aumento de contraste). Traga ponto de interesse para o centro utilizando bobinas de deslocamento de imagem.
- No modo de pesquisa, ajustar o tamanho do local e intensidade do feixe de modo que o feixe é apenas mais largo que o dispositivo de imagem e dá uma leitura de pixel de ~ 20 e - / pixel (CCD) ou ~ 8 e - / pixel / seg (detector eletrônica direta, modo de contagem).
- Encontrar uma boa área de Specimen
- Insira o grid com mitocôndrias congelados hidratado para o microscópio eletrônico à temperatura do azoto líquido (consulte as instruções do fabricante EM).
- No modo de busca, procure a rede para áreas de espessura do gelo e amostras de qualidade adequado. Dê uma busca de imagens 6 seg de áreas promissoras para determinar a adequação para a coleta de tomografia. Both da membrana mitocondrial interna e externa deve ser visível neste ampliação.
- Gravação de um Tilt Series tomográfica
- Uma vez que uma boa área espécime é encontrado, incline a fase de ± 60 ° para determinar o alcance máximo de inclinação que está disponível sem qualquer obstrução da exposição ou foco área por barras de grade ou pedaços de gelo.
- Em um buraco cheio de gelo perto de aparência semelhante, mudar para o modo de exposição e ajustar o tempo de intensidade do feixe ou de aquisição de imagem de modo que cada imagem gravada tem uma dose de elétrons de 30-50 e - / pixel para CCDs ou 6-8 e - / pixel / s detectores de elétrons diretos, modo de contagem.
- Calcule a razão de distribuição dose (I 0 / I 60), dividindo a média de contagem eletrônica de uma imagem de 1 seg adquiriu a 0 ° com o de uma imagem de 60 ° para. Esta relação descreve o aumento do tempo de exposição necessário para manter uma contagem constante de elétrons por imagem com o aumento do ângulo de inclinação (tempo de exposição = 1 / cos (α) n, onde (I 0 / I 60) = 2 n). A relação também serve como uma boa indicação da espessura do gelo. Boas tomograms de mitocôndrias são geralmente gravados com um I 0 / I 60 = 2,3-2,6.
- Ao longo de um buraco vazio, adquirir uma imagem de 1 segundo no modo de exposição e observe a contagem de elétrons por um 2. Levando-se em conta a relação de distribuição de dose, calcular o número total de imagens que podem ser gravadas para uma dose total de elétrons específico (por exemplo, <40 e - / a 2 para a determinação da estrutura e ~ 160 e - / a 2 para a morfologia).
- Determinar o intervalo de inclinação apropriado para a recolha de tomograma dividindo a gama total de inclinação (por exemplo, 120 ° para ± 60 °), com o número total de imagens calculadas em 3.3.4.
- Configurar e gravar uma tomografia com os parâmetros determinados acima, usando software de coleta de dados automática apropriado 33, 34. Série Tilt são geralmente iniciado em ± 20 ° e passar por 0 ° antes de atingir altas inclinações, a fim de maximizar o conteúdo de informação de imagens de baixa inclinação, que é destruído pelo aumento da dose de elétrons.
4 Criação e Segmentação de Volumes tomográficos
Esta seção descreve como volumes tomográficos da mitocôndria são gerados a partir de séries de inclinação e como os volumes estão presentes para a visão geral.
- Salve a série tomográfica de inclinação para um diretório apropriado. Gerar uma pilha de imagens e converter para um formato de arquivo adequado, com software de código aberto, como dm2mrc ou tif2mrc (pacote Imod), que convertem .dm3, .dm4 ou .tif para pilhas MRC. pilhas MRC são necessários para a reconstrução tomográfica com Imod 35. Outros pacotes requerem diferentes formatos.
- Alinhe as imagens e gerar uma tomografia, seguindo os passos descritos no tutorial Imod ( http://bio3d.colorado.edu/imod/doc/etomoTutorial.html ).
- Melhorar o contraste da tomografia usando o filtro anisotrópico de difusão não-linear distribuído com Imod. Este filtro funciona bem para as membranas e as partículas associadas com a membrana, tais como ATP-sintase.
- Para a visualização, o segmento manualmente tomograma usando programas disponíveis comercialmente, por exemplo, Amira. Atribuir voxels correspondentes à membrana interna ou externa e gerar uma superfície. Usando a opção de clicker no plugin EM-pacote para AMIRA 36 marcar a localização de partículas ATP sintase.
5. Subtomogram cálculo da média da ATP sintase dímeros e Montagem de Estruturas X-Ray
A seção a seguir descreve como médias subtomogram da ATP sintase dímeros podem ser obtidas.
- Usando as partículas marcadas como entrada e um pacote de software apropriado, como 'ParteEstimativa igo para o programa Electron Tomography ", calcular uma média subtomogram.
- Para uma estimativa de resolução, comparar duas determinados independentemente médias subtomogram por Fourier shell correlação 37.
- Se disponíveis, doca conhecido estruturas de raios-X para a média subtomogram por corpo rígido encaixe, de forma manual ou usando rotinas de atracação sequenciais automáticas como as do programa Chimera 38.
Resultados
Electron crio tomogramas de mitocôndrias revelam claramente a morfologia 3D da organela (Figura 2). Segmentação manual das membranas num volume tomográfico ilustra a estrutura das cristas em uma mitocôndria. Por imagiologia mitocôndrias de diferentes estirpes de levedura que não possuem a eliminar certos componentes da proteína, o efeito destas proteínas na morfologia cristas podem por avaliada. Figura 3 mostra uma mitocôndria de uma estirpe de levedura sem ATP sintase e da subunidade. Este componente do complexo da sintase ATP é necessária para a dimerização da ATP sintase mitocondrial. As mitocôndrias desta estirpe não têm a cristas lamelar normal das mitocôndria tipo selvagem (Figura 2) e, em vez de conter um número de compartimentos da membrana interior. Estes compartimentos são ou desprovido de cristas ou conter pequenas invaginações da membrana em forma de balão (Figura 3).
Em tomograms com bom contraste, grandes complexos de proteínas mitocondriais, neste caso ATP sintase dímeros, são facilmente visíveis (Figura 4; Filme 1). As estruturas dos complexos pode ser determinada a 2-3 nm de resolução média subtomogram (Figura 5; Filme 2). Os volumes médios podem ser colocados de volta na tomografia, a fim de avaliar a organização dos complexos individuais em relação uns aos outros e a outros complexos de proteínas na membrana (Figura 6; filme 3).
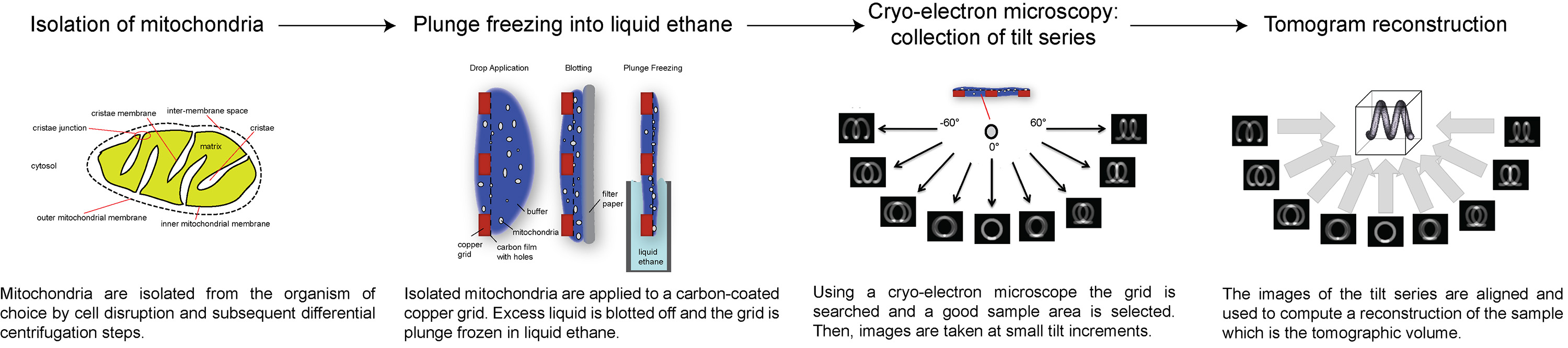
Figura 1 Fluxograma mostrando as etapas do elétron crio-tomografia. Clique aqui para ver a imagem ampliada.

Figura 2 Morfologia de uma mitocôndria de wildtype S. Cerevisiae. Fatia Central através de um volume tomográfico de um tipo selvagem S. mitocôndria cerevisiae (esquerda) e do volume rendeu-superfície correspondente (direita). O volume segmentada da membrana externa é mostrado em cinza e os volumes do limite interior e cristas membranas em azul claro. Adaptado de Davies et al 8. Clique aqui para ver a imagem ampliada.

Figura 3. mitocôndrias a partir de um S. Cerevisiae estirpe falta uma subunidade necessária paraATP sintase dimerização. Cortam o volume tomográfica (esquerda) e acompanha o volume processado-superfície (à direita) de uma mitocôndria de uma S. Cerevisiae sem a subunidade de proteína e necessária para a ATP sintase dimerização. Quando comparada com a Figura 2, a mitocôndria da estirpe mutante não tem o cristas lamelar normal da mitocôndria tipo selvagem. Em vez disso, a mitocôndria tem muitos compartimentos da membrana interna com ou sem cristas ou cristas em forma de balão. Assim elétron crio-tomografia destaca alterações na morfologia da membrana devido a deleções de genes. Adaptado de Davies et al 8. Clique aqui para ver a imagem ampliada.

Figure 4 Mitocôndria a partir do fungo P. anserina. cortam o volume tomográfica (à esquerda) e do volume-rendido superfície de acompanhamento (à direita) de uma mitocôndria do fungo filamentoso P. anserina. Neste tomografia, filas de 10 partículas nm (setas amarelas) estão localizados acima cumes de membrana altamente curvas no cristas da membrana interna (ver Filme 1). Estas partículas foram identificadas como dímeros ATP sintase por subtomogram média. De Davies et al 9. Clique aqui para ver a imagem ampliada.
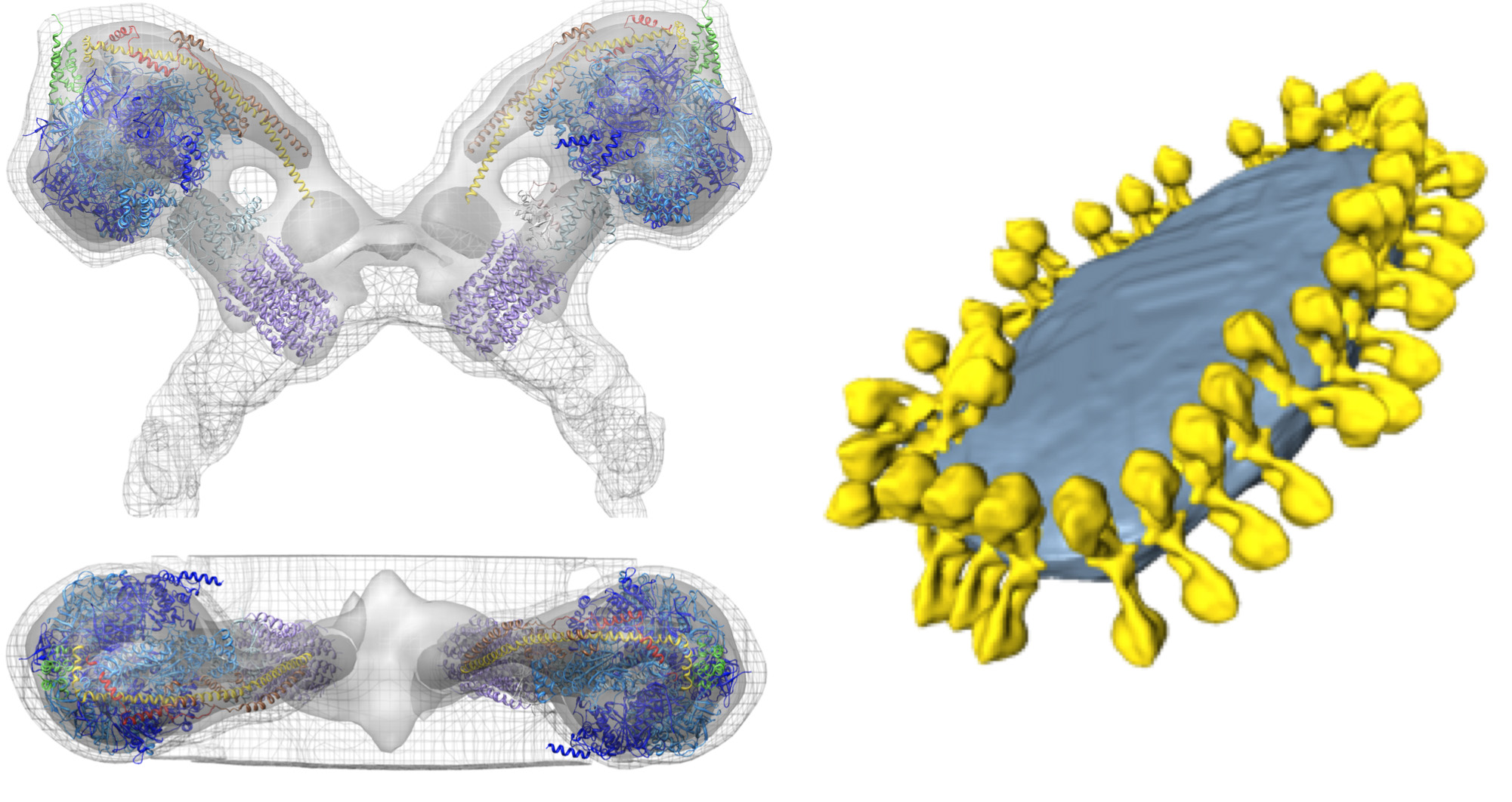
Figura 5 Estrutura e organização da ATP sintase mitocondrial. Vista lateral e superior mostrando a densidade eletrônica de um dímero ATP sintase de S. c erevisiae conforme determinado pela média subtomogram com modelos atômicos embutidos (esquerda). Mitocondrial vesícula de membrana interior que mostra a organização da ATP sintase dímeros em filas (direita). A figura foi gerada pelo posicionamento da média subtomogram do dímero sintase ATP em volume segmentada da vesícula de membrana, usando as coordenadas calculadas durante média. Adaptado de Davies et al 8. Modelos atômicos: F 1 / rotor-ring [PDB: 2WPD] 39 (azul e roxo); oligomicina sensível conferindo proteína OSCP [PDB: 2BO5] 40 (verde); fragmento de talo periférico [PDB: 2CLY] 1 com resíduos N-terminais de [PDB: 2WSS] 2 (amarelo e vermelho) (ver filme 2). Clique aqui para ver a imagem ampliada.
_upload / 51228 / 51228fig6highres.jpg "width =" 500 "/>
Figura 6 crista isolada a partir de uma vesícula de P. anserina mitocôndria. fatias de um volume tomográfica (à esquerda) e do volume-rendido superfície de acompanhamento (à direita) de uma vesícula crista de P. anserina. Densidades de proteínas que saem da membrana são claramente visíveis. As densidades indicadas por setas amarelas são dímeros ATP sintase, conforme identificado por subtomogram média. Setas verdes apontam para densidades identificadas pela marcação de anticorpos como NADH desidrogenase (complexo 1, para mais detalhes ver 9). A segmentação das densidades de proteína revela a sua organização na cristas, com os dímeros da ATP sintase (vermelho e amarelo) que formam as linhas ao longo das arestas de cristas altamente curvas e os complexos de NADH desidrogenase (verde) em regiões da membrana de cada lado das linhas (ver Filme 3). De Davies et al 9.28 / "target =" 51228fig7highres.jpg _blank "> Clique aqui para ver a imagem ampliada.
Filme 1. Electron crio-tomografia de um P. anserina mitocôndria. O filme mostra fatias sucessivas através de um volume tomográfica tomado de uma mitocôndria do fungo filamentoso P. anserina. Fileiras de ATP sintase são indicados por setas amarelas. A, o volume segmentada-rendido superfície mostra a localização da ATP sintase (esferas vermelho) em relação à estrutura 3D de cristas. Membrana externa, cinza; membrana de limite interior, azul transparente; cristas membranas, azul opaco. De Davies et al 9. Ver também Figura 4. Clique aqui para ver o vídeo.
Filme 2. média Subtomogram do dímero ATP sintase mitocondrial de S. Cerevisiae com modelos atômicos embutidos. A média foi decalculada a partir de 121 subvolumes. A densidade é apresentada em três níveis de contorno: 1s - malha, 2s - cinza claro e cinza escuro - 3s. Modelos atômicos foram ajustados para a densidade usando a rotina ajuste seqüencial em Chimera. Modelos atômicos: F 1 / rotor-ring [PDB: 2WPD] 39 (azul e roxo); oligomicina sensível conferindo proteína OSCP [PDB: 2BO5] 40 (verde); fragmento de talo periférico [PDB: 2CLY] 1 com resíduos N-terminais de [PDB: 2WSS]. 2 (amarelo e vermelho) Clique aqui para ver o vídeo.
Adaptado de Davies et al 8. Ver também figura 5.
Filme 3 isolado a partir de uma vesícula de cristas P. anserina mitocôndria. fatias sucessivas através do volume tomográfico são mostrados, seguido pelo volume rendeu-superfície segmentada. Densidades de proteína salientes do membrane são claramente visíveis. Densidades vermelhas e amarelas são dímeros ATP sintase. Densidades verdes são NADH desidrogenase (complexo I), conforme determinado por marcação com anticorpos. De Davies et al 9. Ver também Figura 6. Clique aqui para ver o vídeo.
Discussão
O protocolo aqui apresentado fornece uma introdução para crio-ET e subtomogram média das mitocôndrias, mas, essencialmente, o mesmo procedimento pode ser aplicado a qualquer outro compartimento da célula ou membrana. Para obter os melhores dados possíveis, os passos críticos durante o procedimento são a preparação da amostra, a estratégia de aquisição de processo de congelamento de mergulho e dados. Qualidade da amostra, o que é crítico para o sucesso, depende de um protocolo de congelação otimizado para garantir a espessura do gelo adequado, que é de suma importância para o bom contraste da imagem. A melhor estratégia de aquisição de dados depende do instrumento e amostra. Os parâmetros a serem otimizados incluem doses de elétrons por imagem, sistema de inclinação e desfocar. Adquirir um bom tomografia de uma boa amostra faz tudo ainda mais etapas de processamento mais fácil e garante um resultado final satisfatório.
Cryo-ET combinada com média subtomogram e os ajustes do modelo atômico fornece detalhes de como complexos de proteínas são organizadas em their ambiente celular nativo. A técnica é igualmente adequado para investigar a estrutura de grandes complexos de proteínas de membrana, tais como a cadeia respiratória supercomplexos (1,7 MDA), dímeros ATP sintase (2x500 kDa), ou o complexo do poro nuclear (~ 120 MDa) 8, 9, 17. A organização de ATP dímeros sintase em linhas não pode ser observada por meio de técnicas de alta-resolução, tais como cristalografia de raios-X, RMN ou de uma única partícula de crio-EM, porque as linhas de dímeros são interrompidos por extracção com detergente, o que é um passo necessário para o isolamento e purificação de complexos de proteínas de membrana.
O arranjo da ATP sintase, em fileiras de dímeros de cristas ao longo de arestas é um princípio organizador universal de mitocôndrias em todas as espécies. Os complexos de bombeamento de prótons da cadeia de transporte de elétrons, em particular complexo I (NADH desidrogenase), estão localizados nas áreas de membrana cada lado das linhas 8,9. Esta organização da cadeia respiratória tem profundo impacto na bioenergética mitocondrial. Se as linhas de dímeros não podem formar, devido à ausência de subunidades de proteínas específicas do dímero, as células exibem tempos de vida mais prolongados e reduzida aptidão celular, como observada no mutante de levedura mostradas na Figura 3, 41. Com o crescente interesse em doenças mitocondriais, um detalhada compreensão da base molecular que regula ultra-estrutura e função mitocondrial é de suma importância. Crio-tomografia Electron fornece uma ligação entre a estrutura da proteína foi determinada por meio de métodos de alta resolução, e a distribuição e a disposição dessas proteínas na membrana na escala dos nanómetros. Isso faz com que crio-ET uma ferramenta essencial para a compreensão da estrutura e função mitocondrial na saúde e na doença.
Novos desenvolvimentos e melhorias técnicas em crio-ET incluir estratégias de rotulagem proteína para identificar a posição de protein subunidades em complexos macromoleculares ou a localização das proteínas distintas menores ou menos (<0,5 MDa) em células. Além disso, métodos de processamento EM híbrido, que combinam subtomogram média com a análise de uma única partícula ou reconstrução helicoidal recentemente determinado estruturas de proteínas para ~ 8 a 42, 43. Estes métodos de processamento são atualmente restrita a proteínas purificadas, que são bem separados no gelo ou formam conjuntos helicoidais e não são actualmente aplicáveis a ambientes celulares lotados como mitocôndrias e cristas membranas. Para a coleta de dados, as novas ferramentas de computação e hardware estão sendo desenvolvidas para permitir a aquisição tomografia automatizado, aumentar o contraste da imagem e reduzir a dose total de elétrons necessária. A única limitação fundamental na crio-ET é danos da radiação com a amostra pelo feixe de elétrons. Isso significa que apenas doses muito baixas de elétrons pode ser usado para gravar cada imagem de uma série tomográfica inclinação, resultando em mau sinal-trazão que, em última análise limita a resolução alcançável o-ruído. Os novos detectores de elétrons direta, lançado menos de um ano atrás, estão a revolucionar a área de de uma única partícula de crio-EM 44, 45. Estes novos detectores de proporcionar maior contraste e melhor resolução em doses mais baixas de elétrons. Para elétron crio-tomografia, isso significa que a série de inclinação com tamanhos de etapa menores ou tomografias eixo mesmo duplas podem ser coletadas sem preocupações com danos excessivos de radiação (uma imagem CCD é equivalente em dose de elétrons para 5 imagens detector eletrônica direta). As grandes quantidades de dados produzidos por esses detectores de criar seus próprios desafios em manipulação de dados, processamento e armazenamento, que terão de ser superados.
Além disso, as placas de fase, que trabalham em princípios semelhantes aos usados rotineiramente em microscopia de luz para melhorar o contraste de fase, estão sendo desenvolvidos atualmente para microscopia eletrônica de transmissão 46, 47. Isto deve permitir radiografias para ser recolhido mais perto de se concentrar e, portanto, a maior resolução, enquanto que, ao mesmo tempo preservando as características de baixa resolução necessária para o alinhamento e interpretação dos volumes tomográficos. Tomados em conjunto, esses avanços tecnológicos irá expandir a gama de questões biológicas que podem ser abordados por crio-ET.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este trabalho foi apoiado pela Sociedade Max Planck (KMD, BD, AWM, TB, TBB, DJM, e WK), o Cluster de Frankfurt "complexos macromoleculares" Excelência financiado pelo Deutsche Forschungsgemeinschaft (WK) e um pós-doutorado EMBO Long-Term Fellowship (VAMG).
Materiais
| Name | Company | Catalog Number | Comments |
| Sucrose | Roth | 4621.2 | |
| Trehalose | Sigma-Aldrich | T9449 | |
| HEPES | Sigma-Aldrich | H3375 | |
| Zymolase | biomol | Z1000.1 | |
| Density gradient medium | Sigma-Aldrich | D1556 or P1644 | |
| Protein A gold | Aurion | 810.111 | 10 nm |
| Ethane | Air Liquide | P0502S02R0A001 | |
| Nitrogen | Air Liquide | I4150RG | |
| Consumables | |||
| Filter paper | Whatman, GE Healthcare | 1004 090 | Nr 4 |
| Tweezers | Dumont | T506 | Non-magnetic, pattern #5 |
| EM combined test grid | Plano GmbH | S142 | grapite and gold islands on holey carbon film |
| Holey carbon grids | Quantifoil Micro Tools GmbH or C-flat | R2/2 300 Cu mesh | |
| Cryo grid boxes | Plano GmbH | 160-42 | Alternative home-made metal boxes |
| Potter | Sigma-Aldrich | P7984 | |
| Blender | Waring | 8011S | |
| Glass bead mill - bead beater | BioSpec Products | 1107900-105 | Including 0.5 mm beads |
| Ball-bearing (Balch) homogeniser | isobiotec | H8 | |
| Centrifuge | Thermo Scientific | 46915 | Sorvall RC6+ |
| Glow-discharge apparatus | Bal-Tec A.G. | CTA 010 | |
| Grid vitrification device | FEI, Gatan or Leica | Alternative home-made plunger | |
| Transmission electron microscope | FEI or Jeol | 300 keV | |
| Direct electron detector | Gatan | K2 summit | 4k x 4k pixels, counted mode |
| Charge coupled device (CCD) | Gatan | US4000 | 4k x 4k pixels (alternative to Direct electron detector) |
| Energy filter | Gatan or Jeol | ||
| Software | |||
| Image acquisition software | Gatan | ||
| Tomogram acquisition software | Gatan, FEI, USCF, or UC-Boulder | 4 alternatives | |
| Tomogram processing software | FEI, UCSF or UC-Boulder | 3 alternatives | |
| Subtomogram averaging software | UC-Boulder, or University of Basel | 2 alternatives | |
| Tomogram visualization software | UC-Boulder, FEI, or University of Utah | 3 alternatives | |
| Visualization software plugin | Goethe university | http://www.biophys.uni-frankfurt.de/frangakis/ | |
| Structure visualization software | UCSF | http://www.cgl.ucsf.edu/chimera/ | |
| Numerical calculation software | MathWorks | http://www.mathworks.de/academia/ | |
Referências
- Dickson, V. K., Silvester, J. A., Fearnley, I. M., Leslie, A. G., Walker, J. E. On the structure of the stator of the mitochondrial ATP synthase. The EMBO journal. 25, 2911-2918 (2006).
- Rees, D. M., Leslie, A. G., Walker, J. E. The structure of the membrane extrinsic region of bovine ATP synthase. Proceedings of the National Academy of Sciences of the United States of America. 106, 21597-21601 (2009).
- Watt, I. N., Montgomery, M. G., Runswick, M. J., Leslie, A. G., Walker, J. E. Bioenergetic cost of making an adenosine triphosphate molecule in animal mitochondria. Proceedings of the National Academy of Sciences of the United States of America. 107, 16823-16827 (2010).
- Baker, L. A., Watt, I. N., Runswick, M. J., Walker, J. E., Rubinstein, J. L. Arrangement of subunits in intact mammalian mitochondrial ATP synthase determined by cryo-EM. Proceedings of the National Academy of Sciences of the United States of America. 109, 11675-11680 (2012).
- Allen, R. D., Schroeder, C. C., Fok, A. K. An investigation of mitochondrial inner membranes by rapid-freeze deep-etch techniques. The Journal of cell biology. 108, 2233-2240 (1989).
- Strauss, M., Hofhaus, G., Schroder, R. R., Kühlbrandt, W. Dimer ribbons of ATP synthase shape the inner mitochondrial membrane. The EMBO journal. 27, 1154-1160 (2008).
- Dudkina, N. V., Oostergetel, G. T., Lewejohann, D., Braun, H. P., Boekema, E. J. Row-like organization of ATP synthase in intact mitochondria determined by cryo-electron tomography. Biochimica et biophysica acta. 1797, 272-277 (2010).
- Davies, K. M., Anselmi, C., Wittig, I., Faraldo-Gomez, J. D., Kühlbrandt, W. Structure of the yeast F1Fo-ATP synthase dimer and its role in shaping the mitochondrial cristae. Proceedings of the National Academy of Sciences of the United States of America. 109, 13602-13607 (2012).
- Davies, K. M., et al. Macromolecular organization of ATP synthase and complex I in whole mitochondria. Proceedings of the National Academy of Sciences of the United States of America. 108, 14121-14126 (2011).
- Wurm, C. A., et al. Nanoscale distribution of mitochondrial import receptor Tom20 is adjusted to cellular conditions and exhibits an inner-cellular gradient. Proceedings of the National Academy of Sciences of the United States of America. 108, 13546-13551 (2011).
- Schermelleh, L., Heintzmann, R., Leonhardt, H. A guide to super-resolution fluorescence microscopy. The Journal of cell biology. 190, 165-175 (2010).
- Austin, J. R. 2nd, Staehelin, L. A. Three-dimensional architecture of grana and stroma thylakoids of higher plants as determined by electron tomography. Plant physiology. 155, 1601-1611 (2011).
- Ohta, K., et al. Beam deceleration for block-face scanning electron microscopy of embedded biological tissue. Micron. 43, 612-620 (2012).
- Buzhynskyy, N., Sens, P., Prima, V., Sturgis, J. N., Scheuring, S. Rows of ATP synthase dimers in native mitochondrial inner membranes. Biophysical journal. 93, 2870-2876 (2007).
- Larabell, C. A., Nugent, K. A. Imaging cellular architecture with X-rays. Current opinion in structural biology. 20, 623-631 (2010).
- Miao, J., Hodgson, K. O., Sayre, D. An approach to three-dimensional structures of biomolecules by using single-molecule diffraction images. Proceedings of the National Academy of Sciences of the United States of America. 98, 6641-6645 (2001).
- Maimon, T., Elad, N., Dahan, I., Medalia, O. The human nuclear pore complex as revealed by cryo-electron tomography. Structure. 20, 998-1006 (2012).
- Meyerson, J. R., et al. Determination of molecular structures of HIV envelope glycoproteins using cryo-electron tomography and automated sub-tomogram averaging. Journal of visualized experiments : JoVE. (58), (2011).
- Chen, S., et al. Electron cryotomography of bacterial cells. Journal of visualized experiments : JoVE. (39), (2010).
- Bharat, T. A., et al. Structural dissection of Ebola virus and its assembly determinants using cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 109, 4275-4280 (2012).
- Bennett, A. E., et al. Ion-abrasion scanning electron microscopy reveals surface-connected tubular conduits in HIV-infected macrophages. PLoS pathogens. 5, e1000591(2009).
- Liu, J., et al. Cellular architecture of Treponema pallidum: novel flagellum, periplasmic cone, and cell envelope as revealed by cryo electron tomography. Journal of molecular biology. 403, 546-561 (2010).
- Liu, J., et al. Intact flagellar motor of Borrelia burgdorferi revealed by cryo-electron tomography: evidence for stator ring curvature and rotor/C-ring assembly flexion. Journal of bacteriology. 191, 5026-5036 (2009).
- Patla, I., et al. Dissecting the molecular architecture of integrin adhesion sites by cryo-electron tomography. Nature cell biology. 12, 909-915 (2010).
- Heuser, T., et al. Cryoelectron tomography reveals doublet-specific structures and unique interactions in the I1 dynein. Proceedings of the National Academy of Sciences of the United States of America. 109, E2067-E2076 (2012).
- Briggs, J. A. Structural biology in situ-the potential of subtomogram averaging. Current opinion in structural biology. 23, (2013).
- Cheng, D., Mitchell, D. R. G., Shieh, D. B., Braet, F. Current Microscopy Contributions to Advances in Science and Technology. Méndez-Vilas, A. 2, Formatex. (2012).
- Brust, D., et al. Cyclophilin D links programmed cell death and organismal aging in Podospora anserina. Aging cell. 9, 761-775 (2010).
- Gregg, C., Kyryakov, P., Titorenko, V. I. Purification of mitochondria from yeast cells. Journal of visualized experiments : JoVE. (30), (2009).
- Bhaskaran, S., et al. Breaking Caenorhabditis elegans the easy way using the Balch homogenizer: an old tool for a new application. Analytical biochemistry. 413, 123-132 (2011).
- Pavlov, P. F., Rudhe, C., Bhushan, S., Glaser, E. In vitro and in vivo protein import into plant mitochondria. Methods in molecular biology. 372, 297-314 (2007).
- Graham, J. M., et al. Ch. 3, Current protocols in cell biology. Bonifacino, J. S. , John Wiley and Sons. (2001).
- Zheng, S. Q., et al. UCSF tomography: an integrated software suite for real-time electron microscopic tomographic data collection, alignment, and reconstruction. Journal of structural biology. 157, 138-147 (2007).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of structural biology. 152, 36-51 (2005).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of structural biology. 116, 71-76 (1996).
- Pruggnaller, S., Mayr, M., Frangakis, A. S. A visualization and segmentation toolbox for electron microscopy. Journal of structural biology. 164, 161-165 (2008).
- Scheres, S. H., Chen, S. Prevention of overfitting in cryo-EM structure determination. Nature methods. 9, 853-854 (2012).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. Journal of computational chemistry. 25, 1605-1612 (2004).
- Dautant, A., Velours, J., Giraud, M. F. Crystal structure of the Mg.ADP-inhibited state of the yeast F1c10-ATP synthase. The Journal of biological chemistry. 285, 29502-29510 (2010).
- Carbajo, R. J., et al. Structure of the F1-binding domain of the stator of bovine F1Fo-ATPase and how it binds an alpha-subunit. Journal of molecular biology. 351, 824-838 (2005).
- Paumard, P., et al. The ATP synthase is involved in generating mitochondrial cristae morphology. The EMBO journal. 21, 221-230 (2002).
- Bharat, T. A., et al. Structure of the immature retroviral capsid at 8 A resolution by cryo-electron microscopy. Nature. 487, 385-389 (2012).
- Bartesaghi, A., Lecumberry, F., Sapiro, G., Subramaniam, S. Protein secondary structure determination by constrained single-particle cryo-electron tomography. Structure. 20, 2003-2013 (2012).
- Bai, X. C., Fernandez, I. S., McMullan, G., Scheres, S. H. Ribosome structures to near-atomic resolution from thirty thousand cryo-EM particles. eLife. 2, e00461(2013).
- Li, X., et al. Electron counting and beam-induced motion correction enable near-atomic-resolution single-particle cryo-EM. Nature methods. , (2013).
- Danev, R., Nagayama, K. Optimizing the phase shift and the cut-on periodicity of phase plates for TEM. Ultramicroscopy. 111, 1305-1315 (2011).
- Walter, A., et al. Practical aspects of Boersch phase contrast electron microscopy of biological specimens. Ultramicroscopy. 116, 62-72 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados