Method Article
Isolamento de comunidades de bacterianas intracelulares único gerado a partir de um modelo murino de infecção do trato urinário para análise de célula única a jusante
Neste Artigo
Resumo
Este protocolo descreve um método simples de isolar células epiteliais única, infectados-bexiga de um modelo murino de infecção do trato urinário.
Resumo
Neste artigo, descrevem um procedimento utilizado para isolar comunidades bacterianas intracelulares individuais de um mouse que tem sido experimentalmente infectadas no trato urinário. O protocolo pode ser dividido em três seções: a infecção, colheita de células epiteliais da bexiga e micropipetting de boca para isolar células epiteliais infectadas individuais. As células epiteliais isoladas contém células bacterianas viáveis e é quase livre de contaminação de bactérias extracelulares, tornando-a ideal para análise de célula única a jusante. O tempo gasto desde o início da infecção, a obtenção de uma única comunidade bacteriana intracelular é cerca de 8 h. Este protocolo é barato para implantar e usa materiais amplamente disponíveis, e esperamos que também pode ser utilizada em outros modelos de infecção para isolar células infectadas única de misturas de célula, mesmo se essas células infectadas são raras. No entanto, devido a um risco potencial em micropipetting boca, este procedimento não é recomendado para agentes altamente infecciosos.
Introdução
Infecções do trato urinário (ITU) são uma das mais comuns infecções bacterianas. Estima-se 40-50% das mulheres são esperados para experimentar pelo menos uma infecção do trato urinário (ITU) durante sua vida1. Um dos principais agentes de UTI é uropathogenic e. coli (UPEC), que é responsável por mais de 70% de simples UTIs2. Além disso, aproximadamente um quarto do que aqueles que têm uma infecção urinária, terá uma infecção recorrente, muitas vezes causada pela mesma tensão, apesar do tratamento antibiótico adequado3. A alta incidência de ITU representa um peso substancial nos sistemas de saúde, custando mais de US $ 2 bilhões por ano nos E.U.4. Além disso, o uso de antibióticos para tratar infecções do trato urinário também leva a taxas crescentes de resistência aos antibióticos, o que é uma preocupação de saúde pública5.
Portanto, foi colocado um grande esforço para compreender os mecanismos pelos quais UPEC infecta o trato urinário, bem como sua capacidade de causar infecções recorrentes6,7,8. Em particular, um modelo do rato da infecção tem sido usado para examinar o bacteriano e host características que contribuem para a UTI8. Este modelo de rato tem a vantagem de ser aplicável a sem modificações clínicas cepas isoladas de pacientes humanos. Este modelo também levou à descoberta de caminhos bacterianas druggable potencialmente importantes para o estabelecimento de UTI, tais como o tipo 1 pilus9 e ferro aquisição sistemas10.
Comparado a esses sucessos em estudar os eventos iniciais na UTI, conhecimento dos mecanismos subjacentes ITU recorrente continua a faltar11. Uma hipótese é que UPEC escapa a terapia antibiótica e provoca infecções na bexiga, formando comunidades bacterianas intracelulares (GRG) dentro das células epiteliais da bexiga. GRG têm sido identificados em modelos murino de infecção e no humano UTI pacientes12,13. A presença de IBCs em amostras de urina de pacientes UTI pediátricas tem sido associada com maiores taxas de recorrência de14,15. No entanto, isolando o GRG e estudar as bactérias dentro deles tem provado para ser tecnicamente desafiador devido à sua raridade; Estima-se que uma bexiga murino infectada normalmente só tem 10-100 GRG16. Além disso, as células epiteliais da bexiga são relativamente grandes (50-120 µm)17, tornando-se desafiador para implantar fluorescência assistida célula classificação (FACS) dado que os bocais de FACS típicos são projetados com diâmetros de 70 µm ou 100 µm. Assim, as células tão grandes quanto as células epiteliais da bexiga frequentemente são removidas por filtração antes da FACS para evitar entupimento do fluidics.
Nosso laboratório descrito recentemente um método geral e econômico para isolar as raras células infectadas de misturas tais como células epiteliais raspadas do bexiga18. Para efetivamente isolar o GRG, usamos tradicional boca pipetagem. Micropipetting de boca é uma técnica que tem sido muito utilizada para micromanipulação de células únicas e embriões para análise a jusante19,20,21,22,23, 24 , 25. tradicional boca pipetagem de grandes volumes de líquido (em mililitros) tem sido muitas vezes a causa de laboratório relacionados a acidentes, e a técnica justamente tem sido rejeitada pela maioria da comunidade de investigação fora do tradicional embriologia e aplicações de célula única. Nosso protocolo é inspirado pelas versões única célula desta técnica19,20, que mitigar riscos, fornecendo um buffer grande (> 2 mL) de ar entre o pesquisador e a amostra em comparação com o volume de líquido transferido (< 1 Μ L). Esse método também aproveita o controle fino essa boca micropipetting fornece, que se traduz em um baixo volume final de solução transferida e alta pureza de células isoladas circundantes. A técnica utiliza materiais baratos (<$ 50) e, portanto, deve ser viável implementar em todos os laboratórios.
Este protocolo visual descreve nossa técnica de isolamento do IBC, fornecendo uma referência para auxiliar outros pesquisadores buscando replicar essa técnica. O pesquisador terá acesso a um microscópio fluorescente dissecação (ou equipamento semelhante) que pode ser usado para visualizar células epiteliais individuais e as bactérias fluorescentes durante a imagem ao vivo, com um palco de imagens aberto e acessível para micropipetting (consulte a Tabela de materiais para os detalhes do microscópio usado, embora outros modelos de instrumento equivalente também podem ser utilizados). Enquanto este protocolo incidirá sobre GRG, em um modelo murino de UTI, métodos semelhantes devem ser aplicáveis para isolar células infectadas de suspensões celulares em outros modelos de infecção.
Protocolo
Todos os métodos descritos aqui sobre manipulação de animais foram aprovados pelo cuidado institucional do Animal e Comissão de utilização (IACUC) do Instituto genoma de Singapura e centro de recursos biológicos da Agência de ciência, tecnologia e pesquisa, Singapura.
1. Mouse infecção
-
Preparação de capilares de vidro
- Uma fonte de fogo aberto (bico de Bunsen ou lamparina) de luz.
- Mantenha um vidro capilar com as duas mãos por apertando firmemente as duas extremidades e, em seguida, no meio do tubo de calor uniformemente até que o vidro se macio. Gire o capilar suavemente e para trás ao longo de seu eixo para ajudar na mesmo aquecimento do vidro.
- Remover o capilar de vidro da fonte de calor e imediatamente separe as mãos, mantendo a aderência em ambas as extremidades do tubo. O comprimento ideal final do capilar puxado é 3-5 cm mais comprido do que um unpulled capilar para garantir o diâmetro interno apropriado para isolar as células epiteliais da bexiga único.
Atenção: O tubo ainda permanece extremamente quente por um período de tempo, então Reserve o capilar em uma superfície de calor-seguro por alguns minutos esfriar antes de prosseguir para a próxima etapa. - Verifique para ver se o meio do capilar de vidro tornou-se mais estreito (Figura 1A) e que o interior do tubo é ainda oco (Figura 1B). Para o isolamento de GRG, um tamanho de furo de 200-400 µm é utilizável.
- Pega uma das extremidades do capilar puxou com uma mão. Segure um par de pinças com a outra mão e usá-lo para pegar o copo puxado capilar no seu ponto mais estreito. Certifique-se de que a pinça está empunhada com força suficiente para segurar o capilar firmemente sem esmagá-lo.
- Use um movimento de rotação rápido pela mão segurando a pinça para agarrar o capilar puxado no ponto mais estreito para criar um micropipetting boca capilar.
Nota: Usar os dedos em vez de fórceps também é aceitável, desde que proteção adequada de cacos de vidro é usada. - Repita as etapas 1.1.2-1.1.6 pelo menos 4 vezes mais para produzir suficiente capilares de reposição micropipetting e fornecer uma gama de diâmetros para isolamento do IBC. Se mais de um grupo de infecção é antecipado, prepare-se 5 capilares micropipetting adicionais para cada grupo adicional de infecção.
Atenção: Não se esqueça de desligar a chama aberta. - Coloque os capilares puxados em uma placa de Petri de 100mm e expor o prato à radiação UV durante 30 min esterilizar os capilares.
- Substitua a tampa sobre o prato de Petri após esterilização UV e armazenar o prato de petri com os capilares à temperatura ambiente.
-
Preparação de cateteres antes da infecção
- Prepare-se cateteres urinários para infecção conforme descrito no Hung et al8 e Conover et al26 , pelo menos um dia antes da infecção.
-
Preparação de fluorescente uropathogenic e. coli cultura
- Crescer o selecionado uropathogenic fluoróforo-expressando bacteriana estirpe de acordo com protocolos estabelecidos.
Nota: As escolhas de tensão e de fluoróforo em grande parte dependem do microscópio e estirpes disponíveis em laboratórios individuais. Neste exemplo, usamos uma estirpe derivada de UTI89, que é um clínico isolado originalmente de um paciente com cistite recorrente. Esta estirpe, SLC-638, carrega um plasmídeo (pSLC-77) que expressa a resistência de ambos vsfGFP-9 e canamicina18. SLC-638 é cultivado em caldo LB a 37 ° C, suplementado com 50 canamicina µ g/mL. - Raia tensão SLC-638 em um Luria Bertani (LB)-placa de ágar suplementado com 50 canamicina µ g/mL. Incube a placa a 37 ° C durante a noite.
- (Opcional) Ver os a placa na dissecação microscópio para confirmar a expressão de marcadores fluorescentes antes de selecionar uma colônia.
- Utilizando uma ansa de inoculação bacteriana, transferi a colônia selecionada para um Erlenmeyer de 125 mL, com 10 mL de caldo LB suplementado com 50 canamicina µ g/mL. Incube o frasco estaticamente a 37 ° C por 24 h.
- Subcultura as bactérias de colocar o balão ao tirar 10 µ l de cultura do frasco e diluir em 10 mL de caldo LB suplementado com 50 canamicina µ g/mL em um frasco de 125ml fresco (uma diluição de 1: 1000). Incube a este segundo frasco estaticamente a 37 ° C, para outro 24h.
- Spin para baixo a cultura bacteriana por 5 min a 5.000 x g e 4 ° C.
- Decante o sobrenadante e ressuspender o bacteriana em PBS frio em OD600 de 0,5.
Nota: Apesar de que pode variar de cultura para cultura, normalmente, 1 mL de cultura estática dá cerca de 4-5 mL de OD600 = 0,5 cultura bacteriana. O volume total de inóculo bacteriano necessário para cada tensão pode ser calculado da seguinte forma: 50 µ l e é necessária para cada rato 50 µ l é para encher a cabeça da agulha. Recomenda-se um adicional de 10-20% (mínimo de 50 µ l) do inóculo para contabilizar o volume morto na seringa. - Use a mistura restante bacteriana para determinar o título de infecção conforme descrito no Hung et al.8.
Nota: Esta etapa pode ser adiada por algumas horas, armazenando a mistura bacteriana a 4 ° C.
- Crescer o selecionado uropathogenic fluoróforo-expressando bacteriana estirpe de acordo com protocolos estabelecidos.
-
Modelo murino de infecção do trato urinário
- Infectam os ratos, conforme descrito por Hung et al8, com um grupo experimental para todas as estirpes de fluorescente e. coli, cultivada no ponto 1.3.
Nota: Consulte também Conover et al26 de assistência visual. - Observe o momento da inoculação bacteriana para o mouse ou gaiola.
- Repita infecções para todo o grupo experimental.
Nota: O cateter de infecção pode ser reutilizado para ratos todos no mesmo grupo. - Repita os passos de 1.4.1-1.4.3 para cada grupo experimental planejado, garantindo que um cateter fresco e novo gel lubrificante é preparado para cada grupo.
Nota: Para experimentos com um grande número de animais, é melhor dividir os animais em grupos de cinco e escalonamento das infecções tal que cada grupo está infectado cerca de 30 min a 1 h separado. Isto fornecerá o tempo suficiente para as próximas etapas (seções 2 e 3).
- Infectam os ratos, conforme descrito por Hung et al8, com um grupo experimental para todas as estirpes de fluorescente e. coli, cultivada no ponto 1.3.
2. bexiga célula epitelial da colheita para obter uma suspensão de células
-
Colheita e invertendo bexigas murino
- Prepare-se três tubos de 50ml cónico preenchidos com 45 mL de etanol a 70% para esterilização de equipamentos cirúrgicos.
- Em dois dos tubos preparados na etapa 2.1.1, coloque um par de tesouras e um par de pinças cada. Coloque dois pares de pinças (um de preferência mais estreito e com ponta arredondada, para a inversão da bexiga) para o terceiro tubo.
Nota: As ferramentas no primeiro tubo serão usadas na região externa, as ferramentas na segunda serão usadas na colheita de bexiga, e os dois pares de pinças na última vontade de tubo usado em inversão de bexiga. - Às 6 h pós infecção, eutanásia os ratos infectados, de acordo com protocolos estabelecidos de IACUC da instituição.
Nota: Nosso protocolo IACUC pede eutanásia através de deslocamento cervical realizada enquanto o mouse está sob anestesia (isoflurano). - Colocar os animais plana em suas costas e use um frasco do pulverizador enchido com etanol a 70% para esterilizar sua área abdominal.
- Usando um par de pinças e tesoura cirúrgica do primeiro tubo (preparado na etapa 2.1.2), abertura faz uma pequena incisão transversal na pele cerca de 1 cm acima da uretra. Expandir a incisão na diagonal para os membros superiores do mouse, criando um corte em forma de V ao longo de todo o anterior do mouse, que expõe o conteúdo do peritônio. Certifique-se de que durante esse processo, a tesoura não corta os intestinos do mouse (Figura 2A, B).
- Alternar para o segundo conjunto de ferramentas (preparado na etapa 2.1.2). Usando as lâminas da tesoura ou os eixos da pinça, empurre suavemente para baixo nas almofadas de gordura próximo a região pélvica do mouse.
Nota: Essa etapa faz com que a bexiga se projetam para fora e garante visibilidade para a colheita. - Pega a bexiga exposta no ápice com um par de pinças (Figura 2C).
- Mantendo um aperto firme no ápice da bexiga com a pinça, corte e livre a bexiga do resto do animal (cortando a uretra e ureteres) usando a tesoura cirúrgica. Não liberar a pinça segurando a bexiga ainda.
- Mudança de tesouras para a pinça arredondada mais estreita do terceiro cónico tubo (da etapa 2.1.2), introduza a ponta de um eixo da pinça arredondada na abertura da bexiga onde só foi cortado na etapa anterior (Figura 2D). Com a ponta da pinça arredondada com segurança inserida na abertura da bexiga, liberar o par de pinças de preensão do ápice da bexiga e devolvê-lo para o segundo tubo cónico.
- Usando o segundo par de pinças do terceiro tubo, vire ligeiramente a bexiga "inside out", primeiro puxando a extremidade externa da boca da bexiga longe a pinça arredondada (Figura 2D, seta 1) e guiando-os ao redor e sobre a outra ponta do a pinça arredondada (Figura 2D, seta 2).
Nota: A ação pode ser comparada a remoção de uma meia de um pé e puxando-o sobre o outro.- Durante o processo de inversão, manter o primeiro par arredondado de fórceps quase completamente fechado. Isso fornece suficiente liberdade de movimentos para retirar a bexiga do primeiro eixo da pinça arredondada, mas também traz o segundo eixo da pinça arredondada mais perto à primeira e permite a bexiga ser transferido facilmente. O resultado final desta etapa é que a bexiga deve acabar sendo invertido e na ponta do eixo do segundo do primeiro par de pinças(Figura 2).
- Usando o segundo par de pinças, delicadamente persuadi a bexiga invertida a ponta da pinça em 1 mL de PBS frio.
Nota: (Opcional) Este é o momento oportuno para tirar fotos de todo o invertido, infectada da bexiga para observar a frequência geral e distribuição de GRG, se for o caso.
- Repita as etapas 2.1.1-2.1.10 para cada bexiga no grupo experimental (até um máximo de cinco animais).
-
Raspagem de células epiteliais da bexiga
- Usando dois pares de pinças de limpar, lixe ligeiramente a parte externa da bexiga invertida (que é a camada interna de células epiteliais). O PBS circundante deve aparecer mais turva como raspagem receitas e células epiteliais são liberadas para a solução de PBS.
- (Opcional) Visualmente, confirme que a raspagem de bexiga liberou as células em solução usando um microscópio de dissecação. Figura 3 A, B). O PBS deve aparecer nublado a olho nu, e as células epiteliais da bexiga individuais raspado podem ser vistas na ampliação de 10x.
- Repita as etapas 2.2.1-2.2.2 para cada bexiga colhida da etapa 2.1.
3. intracelular isolamento da comunidade bacteriana (IBC): boca pipetagem de GRG
Nota: Todos os métodos descritos nesta seção foram submetidas a uma avaliação de risco institucional. Boca de pipetagem carrega o risco inerente de ingestão da solução que está sendo transferida. Este risco é mitigado em grande parte dos volumes de nanolitros que usa esse protocolo, e recomendamos que todos os usuários do salário de protocolo em atenção da precaução e notas práticas listadas aqui e na discussão.
-
Após as células têm sido raspada para a PBS, configurar o aparelho de micropipetting de boca (Figura 3C).
- Insira a extremidade mais grossa do vidro puxado capilar (unpulled final) no plug de borracha (branco final) do tubo aspirador.
- Inserir a extremidade estreita de uma ponta de pipeta de 1 mL em outra (vermelha) extremidade aberta do tubo aspirador, assegurando que não haja um ajuste apertado.
- Insira a extremidade estreita de um 2 mL pipeta de aspiração na extremidade aberta, mais larga da ponta da pipeta de 1 mL, novamente, garantindo que existe um apertado cabe.
Nota: A configuração resultante permite que o pesquisador pipeta de boca da extremidade mais larga, aberta da pipeta aspirar para criar uma força de sucção suave da extremidade estreita do tubo capilar no outro extremo do aparelho. - Teste o aparelho de micropipetting de boca final usando um prato de Petri de 100mm contendo água desionizada. Uma ligeira acção de sucção na extremidade aberta da pipeta aspirar (semelhante a beber uma bebida com um canudo) deve aumentar o nível de líquido no capilar, mas não causa a água desionizada a transbordar para o tubo do aspirador. Utilize uma mão para controlar o tubo capilar, enquanto usando a outra mão para ajustar a posição da caixa de Petri.
Nota: A força de sucção necessária para boca-Pipetar uma única IBC irá variar entre os pesquisadores. No entanto, é recomendável que cada pesquisador tentar esta técnica começar com uma sucção fraca e aumentá-la lentamente se nenhum líquido flui até os capilares. Não há nenhuma necessidade para uma força maior do que chupar um canudo para beber. Se não aparecer recolher líquido durante o teste na etapa 3.1.4 capilar, é possível que o tubo de aspiração ou capilar é obstruído e precisa ser substituído. É mais recomendado que todos os novos pesquisadores primeiro treino controlando sucção no aparato de pipetagem boca usando água esterilizada. Além disso, observe que o controle do volume retomado pelo aparato de pipetagem de boca é através do uso de língua do pesquisador. A língua pode finamente ajustar a força de sucção aplicada, bem como atuar como uma paragem de emergência. - Depois de atingir o sucesso absorção de líquido, teste a capacidade de expulsá-lo do capilar soprando suavemente na extremidade aberta da pipeta aspirar. Certifique-se de que não há bolhas são criadas no processo de expelir o líquido para evitar a contaminação dos GRG durante a etapa de 3.5.
Nota: Como com a sucção, a força de pressão positiva aplicada pelo pesquisador para expulsar o IBC no tubo de centrífuga irá variar entre os pesquisadores. É recomendável para os investigadores novos para este protocolo para a etapa prática 3.1 alguns dias antes da infecção real. Uma sugestão para praticar micropipetting boca é prática para transferir pequenos volumes de água esterilizada, misturado com algumas gotas de corante de comida (a visibilidade) usando o aparato de micropipeta de boca.
- Coloque a suspensão de células raspadas sob o microscópio de dissecação e identificar os GRG como grandes agregados fluorescentes (Figura 3A, B). A faixa ideal de ampliação é 20-40 x. Mergulhe a extremidade bem do capilar de vidro num tubo fresco de PBS para 1 s para reduzir a absorção do volume indesejado através da ação capilar.
- Olhando através do microscópio, identificar o IBC de interesse e traga lentamente a extremidade aberta do tubo capilar em direção o IBC. Use bem final do vidro capilar para varrer as células extras perto cada IBC para evitar a aspiração de duas ou mais células, ou para quebrar distante maiores agregados de células.
- Olhando através do microscópio, aplica uma pequena força de sucção na extremidade mais distante (pipeta aspirar) do aparato de micropipetting de boca para guiar o IBC o capilar de vidro.
- Depois de pegar o IBC, mover o capilar para um tubo de centrifugação vazio 1,5 mL e aplique uma ligeira pressão positiva para expulsar a gota e o IBC no tubo de centrífuga (Figura 3-D).
- Repita as etapas de 3.3-3.6 em como muitos GRG são necessários da bexiga atual, antes de prosseguir para a próximo da bexiga. Mude a aspiração pipetas e capilares com frequência para evitar a acumulação de saliva.
Atenção: Ao trabalhar com doenças infecciosas (ou clínicas) de cepas de bactérias, monitore constantemente o nível da solução no capilar. Não deixe que o nível de líquido sendo pipetado estouro da borda do capilar no tubo de aspiração. Se isso ocorrer, imediatamente mudar para um tubo aspirador diferentes e descartar o conjunto anterior acima. - Repita as etapas 3.2-3.6 em todas as bexigas colhidas, ou até que um número suficiente de amostras biológicas foram recolhido para o grupo experimental.
- (Opcional) Repita as etapas de 3.6 e 3.7 até todos experimental grupos (ou ratos) tem sido sacrificados e foram colhidas amostras biológicas suficientes de cada grupo.
Resultados
Além de confirmação (Figura 3-D), da presença de um único isolado IBC no tubo de coleta através do microscópio de dissecação, a pureza do GRG isolado também pode ser confirmada pela microscopia confocal. Como mostrado na Figura 4A, as células isoladas devem manchar tanto para Escherichia coli e uroplakin e são o tamanho esperado para GRG (50-120 µm)17. Além disso, a coloração de e. coli não está presente no líquido circundante. Com base em nossos dados, mais de 90% das células isoladas com esta técnica são GRG18. Após o isolamento, a presença e a viabilidade das células bacterianas em IBC individual podem ser confirmados através da enumeração de unidade (CFU) formadora de Colônia (Figura 4B) ou reação em cadeia da polimerase quantitativa (qPCR) para genômica equivalentes ( Figura 4 C). Figura 4C também demonstra que não infectadas células epiteliais isoladas com o mesmo protocolo não têm quantidades quantificáveis de bactérias. Com base nestes dados, estimamos que a gama de CFUs em um único IBC é de2-10 103 no modelo murino de infecção do trato urinário. Um dos principais objetivos do único isolamento IBC é realizar análises a jusante, tais como a sequenciação do ARN. Para verificar que o nosso método de isolamento é capaz de obter RNA de bactérias em GRG para análise, realizamos a quantificação de (qRT-PCR) reação em cadeia de polimerase transcrição reversa quantitativa de três genes (16S, cyoB e frdA) para um gama de GRG individualmente isolados e agrupados (Figura 4-D). Todos os dados mostrados na Figura 4 foi adaptado com permissão da Tatiana et al.18. Uma esquema de visão geral do nosso protocolo de isolamento IBC pode ser vista na Figura 5, que é reproduzida de Tatiana et al.18.

Figura 1 : Mão-puxado capilares retêm aberturas estreitas. (A) amostras de mão puxado os tubos capilares é exibida em um fundo preto para o contraste. De baixo para cima, um unpulled capilar, um capilar que não foi puxado em grau suficiente, um capilar que pode ser usado para a colheita de células epiteliais único da bexiga e um capilar que foi puxado muito fina (e, portanto, separado em duas partes) são mostrados. Uma régua de 15 cm é colocada na parte inferior da imagem para escala. O ponto estimado para estalar fora do capilar utilizável é indicado na figura pela seta vermelha. (B) imagem tirada com um microscópio de dissecação confirmando o diâmetro interno oco de um capilar puxado (parte inferior). Um unpulled capilar está posicionado acima para demonstrar a diferença de tamanho relativo dos dois capilares. Barra de escala = 4,0 mm. clique aqui para ver uma versão maior desta figura.
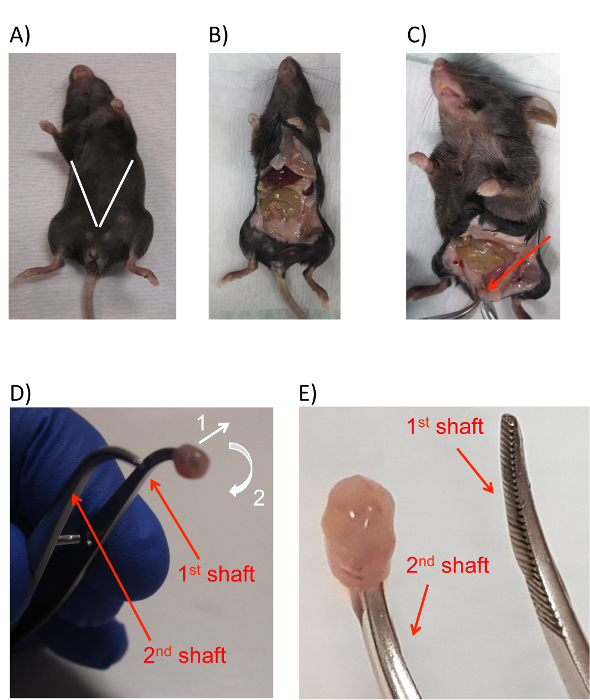
Figura 2 : Dissecação de rato para colher células epiteliais da bexiga. (A) uma imagem de um rato com linhas brancas adicionado para indicar a localização estimada e ângulo de incisões para expor a cavidade peritoneal murino e bexiga. Pós-incisão do (B) uma imagem do exposto rato cavidade peritoneal. (C), uma imagem do exposto bexiga (seta vermelha) projetando-se entre as almofadas de gordura. (D) uma imagem da bexiga murino com a ponta da pinça inserida o lúmen, com setas para indicar a direção do movimento necessário para inverter a bexiga. A bexiga primeiro é puxada um pouco para o exterior, então ao redor e fora o primeiro eixo da pinça. Os sentidos de movimento para ambas as ações são indicadas pelas setas brancas numeradas 1 e 2. (E) uma imagem mostrando a posição final da bexiga invertida inserida no segundo eixo da pinça. Os eixos da pinça são rotulados em ambos os painéis D e E com setas vermelhas e texto. Clique aqui para ver uma versão maior desta figura.
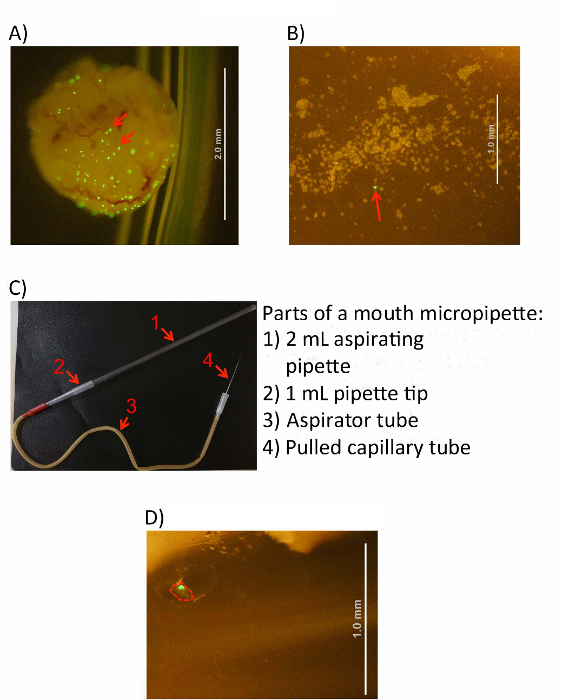
Figura 3 : IBC colheita de células da bexiga. (A) uma bexiga infectada e invertida em solução de PBS fria antes de raspagem de células. (B) uma imagem mostrando raspado de células da bexiga, como pode ser visto sob um microscópio. GRG podem ser identificados como grandes agregados fluorescentes verdes em ambas as imagens (ver setas vermelhas). Aparelhos de micropipetting de boca (C), uma imagem do concluído. A pipeta aspirar, ponta da pipeta, tubo de aspirador e tubo capilar puxado são identificadas com setas numeradas como indicado à direita. (D) uma imagem de um único isolado IBC dentro de um tubo de coleta de 1,5 mL (esboçado em vermelho). Barras de escala (conforme indicado) são representadas por linhas brancas em painéis A, B e D. , por favor clique aqui para ver uma versão maior desta figura.
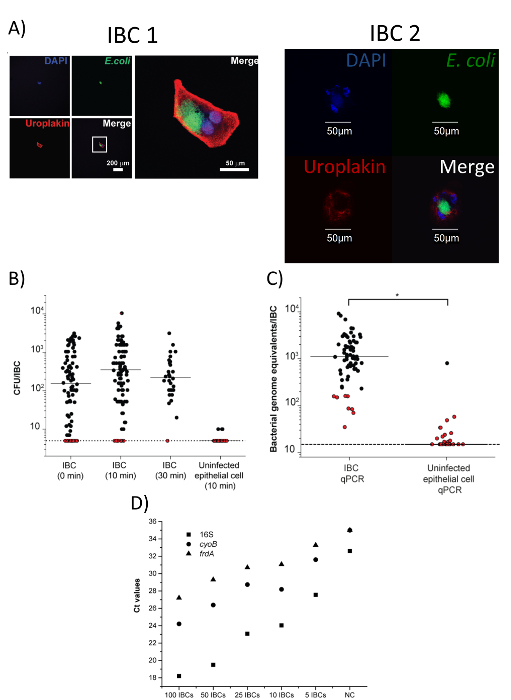
Figura 4 : GRG colhidos são puros e podem ser usados para análise a jusante. Esta figura foi modificada com a permissão da Tatiana et al18. Imagens do (A) de duas células GFP-positivo isoladas que foram manchadas com anti-uroplakin e antianticorpos dee. coli . A primeira célula (IBC 1) tem imagens de canais individuais (em baixo-ampliação) à esquerda, e uma imagem mesclada de alta ampliação é à direita. A segunda célula (IBC 2) é mostrada em alta ampliação em canais individuais e mesclados. Barras de escala são indicadas. DNA é manchado com 4, 6-diamidino-2-phenylindole (DAPI) e representada no canal azul. Anti -e. coli está manchada com um anticorpo secundário conjugado com isotiocianato de fluoresceína (FITC) e representada no canal verde. Anti-uroplakin está manchada com um anticorpo secundário conjugado com isotiocianato de diversos (TRITC) e representada no canal vermelho. (B) CFUs bacteriana de GRG isolados. GRG foram processados imediatamente, ou incubados em 0,1% Triton-X para 10 ou 30 min. em pool CFU as contagens de GRG individuais isolado de n = 3 experimentos separados são mostrados. Limite de detecção = 0,7 log10 CFUs/IBC. Plotados no limite de detecção de pontos vermelhos indicam amostras para as quais não há colônias foram recuperadas. Todas as amostras que contenham IBC não são significativamente diferentes (p > 0,05, teste de Mann-Whitney); as células epiteliais não infectadas são significativamente diferentes a partir dos dados do IBC (10 min) (p < 0,001, teste de Mann-Whitney). (C) qPCR quantificação das bactérias em GRG individuais e células epiteliais não infectadas após uma incubação de 10 min em 0,1% Triton-X (*, p < 0,0001, teste de Mann-Whitney, n = 4). Limite de detecção = 1,18 log10 genoma bacteriano equivalentes/IBC. Pontos vermelhos indicam as amostras para as quais não há colônias foram recuperadas na Titulagem no painel b. (D) quantificação dos genes 16S rRNA, cyoB e frdA para números diferentes de GRG individualmente isolados e agrupados (n = 1 experimento; cada um ponto indica a média de 3 repetições de técnicas). NC = sem controle negativo do DNA. Clique aqui para ver uma versão maior desta figura.
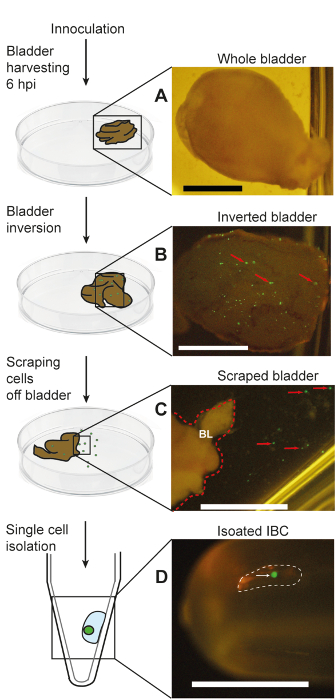
Figura 5 : Um esquema e seus associadas fotografias representando o isolamento de GRG através de micropipetting boca de infectados bexigas ratos. Esta figura é reproduzida de Tatiana et al.18. (A), A colheita inteira bexiga; (B) um invertido bexiga toda expondo o GFP expressando GRG; (C) um close-up da borda de uma bexiga raspada mostrando GRG individuais em suspensão no buffer adjacente; (D) um único isolado IBC pipetado em um tubo. As setas vermelhas no painel B indicam exemplos de GRG GFP-positivo na superfície luminal da bexiga. A linha pontilhada vermelha no painel C indica a borda direita da bexiga invertida (indicada como "BL"); as setas vermelhas no painel C indicam aparente individual GFP-positivo as células epiteliais que têm sido raspadas da superfície da bexiga. Linha pontilhada branca no painel D indica uma gotícula de micropipetted submicrolitro contendo um IBC isolado, que é indicado por uma seta branca. Escala de barras = 2 mm. clique aqui para ver uma versão maior desta figura.
Discussão
Descrevemos o protocolo permite o isolamento de GRG único de um modelo murino de UTI. Este protocolo isola GRG contendo bactérias intracelulares viáveis, que podem ser verificadas pelo cultivo de UFC. Os resultados do protocolo em bactérias intracelulares de GRG com pouca contaminação por bactérias extracelulares, permitindo mais a caracterização de ambas as bactérias e hospedam a célula de um IBC (Figura 4C). Mostramos também que as bactérias de um IBC único podem ser usadas em aplicações a jusante como qPCR (Figura 4C), sugerindo que nossa técnica pode ser usado para processo de IBCs para outras análises in vitro. Por reunir as bactérias de sómente 5 GRG colhidas, mais demonstramos nossa capacidade de realizar análise de qRT-PCR em três genes de bactérias, sugerindo que o RNA de boa qualidade pode ser colhida da bactéria em nossos GRG isolados (Figura 4-D). Combinados, os dados que temos mostrado indicam que realizando análise de todo o genoma RNA (tais como a sequenciação do ARN) em GRG único pode ser possível usar esta técnica de isolamento.
Neste protocolo, temos focado na ponto 6 h de tempo porque é quando IBC números de pico nas bexigas de pretos 6 ratos infectados pelo UTI8927. Além disso, também usamos um sistema de cultura bacteriana estático para aumentar o nível de expressão de pilus tipo 1 em UTI89. A expressão de pilus tipo 1 é crítica para Escherichia coli anexar e infectar a bexiga células epiteliais28. No entanto, essa expressão é fortemente regulamentado29 e pistas ambientais são conhecidas para alterá-lo30. Para manter um fenótipo de infecção consistente e um número suficiente de GRG, nós recomendamos usar uma cultura bacteriana estático de 2 x 24 h (ligeiramente modificada de Hung et al.8) e o ponto de tempo de infecção de 6 h, quando trabalhando com previamente testado Escherichia coli cepas como NU14 e UTI8928,29. No entanto, é possível que essas variáveis precisará ser ajustado em outras cepas UTI ou em outras cepas de ratos para obter o número ideal de GRG de cada infecção.
Enquanto o protocolo de Hung et al8 usa apenas ratos fêmeas, outros protocolos estabelecidos para o estabelecimento de infecção do trato urinário em ratos masculinos têm sido relatados31. Neste modelo, cistite em camundongos machos também seguiu o caminho do IBC. Como a bexiga de ratos machos e fêmeas é similar em tamanho, prevemos que nosso protocolo de isolamento do IBC pode ser usado em camundongos machos infectados também.
A tecnologia relativamente simples utilizada neste protocolo também garante que ele pode ser implantado na maioria dos laboratórios. Uma das principais etapas envolvidas neste protocolo é o puxando de capilares de vidro para criar microcapilares para selecionar o tipo de célula de interesse. Esta etapa permite flexibilidade nos diâmetros dos microcapilares criado, e, portanto, o método pode ser estendido para vários tipos de células de destino diferente. No entanto, devido à variação inerente na criação destes capilares, deve ter cuidado para garantir que o diâmetro final está em uma escala utilizável. Se os capilares são muito estreitos, eles não conseguem pegar a célula de interesse, mas se eles são feitos muito largos, várias células poderiam ser Selecionadodas em uma única tentativa. Além disso, a utilização de uma chama aberta durante o processo de puxar os capilares acarreta um risco inerente de queimaduras e de incêndio, para que o pesquisador, a tentativa de criar microcapilares deve ter cuidado para evitar a ocorrência de tais eventos. Para reduzir a variabilidade, bem como o risco de fogo aberto que envolvido em fazer estes capilares, o pesquisador poderia fazer uso de uma micropipeta tradicional puxando a máquina, como aqueles usados para experimentos eletrofisiológicos (e.g., PC-100, Narishige grupo). Como estas máquinas fazem uso da gravidade ou plataformas robóticas para puxar os capilares, elas podem ser adaptadas para atender às necessidades do modelo de infecção. No entanto, a vasta gama de micropipeta puxando máquinas disponíveis significará que o pesquisador individual terá de passar por alguma tentativa e erro para determinar o diâmetro capilar final adequado para uso com este protocolo.
O protocolo apresentado faz uso de bactérias expressando uma etiqueta fluorescente para identificar visualmente o IBC. Assim, esta técnica é limitada pela capacidade dos pesquisadores para modificar geneticamente o organismo infeccioso. Especificamente para UPEC, IBC-formando cepas como CFT073 e NU14 foram com sucesso transformadas com GFP-expressando plasmídeos32,33,34; Estas, portanto, devem ser utilizáveis no mesmo protocolo. Com base na área do rato bexiga (70 mm2)35, o comprimento de células epiteliais individuais (50-120 µm)17e a frequência de GRG em um único bexiga16, uma estimativa conservadora para a incidência de GRG é sobre 1 em 1.000 células (ou 0,1%). Esta estimativa mostra a utilidade do nosso protocolo de isolamento de célula alvo eventos raros. A precisão de seleção de célula através de nosso protocolo e a ampla gama de diâmetros capilares que pode ser puxado para sugerir que este protocolo pode ser usado para isolar bactérias intracelulares de outros modelos in vivo e in vitro de infecção. Na verdade, nós usaram com sucesso esta técnica para isolar células epiteliais infectadas bexiga cultivadas (dados não mostrados).
Um do mais tecnicamente desafiador passos no protocolo está invertendo a bexiga para expor as células epiteliais por raspagem. Encontramos que também é possível fazer uma incisão na bexiga para splay-lo para fora para raspagem. No entanto, devido deve ser cuidado para reduzir os danos às células epiteliais da bexiga durante o processo de abrir; Idealmente, um único corte deve ser utilizado para splay abrir a bexiga. Além disso, o corte deve ser feito em PBS frio, para evitar a perda acidental de células ou tecido da bexiga durante o processo.
Boca de pipetagem dos GRG neste protocolo fornece maior controle sobre o processo de seleção de células, bem como limitar o volume final da solução transferido juntamente com a célula. O controle fino e grande separação da solução da boca dos pesquisadores também maximiza a segurança do pesquisador, como os volumes transferidos estão dentro o nanolitros a gama microlitro. Em contraste, a nossa experiência com a micropipeta moderna é que ela tende a transferir mais líquido circundante e as células com o IBC, podendo levar a contaminação com bactérias extracelulares luminal. Nossa constatação que boca pipetagem fornece um desempenho mais elevado sobre outra célula única métodos de isolamento também foi relatada por outros laboratórios22,23,24. Além de isolamento de célula única, boca pipetagem nem foi usado na célula única electroporation para neurônios25, que mais demonstra a utilidade e o controle de minutos que um investigador treinado pode atingir com a técnica. No entanto, a segurança é de importância primordial, e sugerimos potenciais medidas adicionais que podem ser tomadas dependendo dos patógenos sendo usados: (i) a extensão do buffer entre o pesquisador e o material biológico, por exemplo, usando uma aspiração de ar pipeta com um volume maior (por exemplo, 5 mL), ou (ii) adicionando um filtro físico como a lã de algodão dentro da pipeta aspirar agir como uma barreira adicional.
Em casos onde uma avaliação de risco leva à conclusão de que ainda é muito arriscado boca pipetagem, configurações robóticas comercialmente disponíveis (tais como aqueles usados para nanoinjections) podem ser combinadas com as outras seções de nossa técnica para fornecer um método mais seguro para isolar as células infectadas de populações mistas. Deve-se notar que a nossa experiência com o uso de um micromanipulador robótica demonstrou uma diminuição da taxa de isolamento de IBC em comparação com boca pipetagem, como a grande variação intra experimental em tamanho de células epiteliais da bexiga torna desafiador para o usuário de um braço robótico para determinar a força necessária para buscar o único GRG. No entanto, continua a ser uma opção viável, embora mais caro, para aqueles que trabalham com agentes altamente infecciosos da doença.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Esta pesquisa foi apoiada pela Fundação Nacional de pesquisa, gabinete, Singapura do primeiro-ministro, sob seu regime de comunhão de pesquisa NRF (NRF prêmio n. NRF-RF2010-10); Conselho de pesquisa médica nacional do Ministério da saúde a Singapura (NMRC/CIRG/1358/2013); e o Instituto do genoma de Singapura (GIS) / agência de ciência, tecnologia e pesquisa (A * STAR).
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL eppendorf tube | For static bacterial culture and OD measurement | ||
| 100% ethanol | For Alcohol Burner | ||
| 15 mL conical tube | For static bacterial culture and OD measurement | ||
| 1 mL Tuberculin Syringe | BD Biosciences | 302100 | |
| 3% Bacterial Agar | For static bacterial culture and OD measurement | ||
| 70% ethanol | For static bacterial culture and OD measurement | ||
| Aesculap anatomic forceps | Braun/Kruuse | BD222R | For initial dissection of mouse (skin, fascia) |
| Alcohol Burner | Wheaton | 237070 | |
| Aspirating pipette | BD Biosciences | 357558 | |
| Aspirator tube | Sigma-Aldrich | A5177 | |
| Bacterial loops | For static bacterial culture and OD measurement | ||
| Benchtop centrifuge | Eppendorf | 5424 | Any centrifuge for 1.5ml eppendorf tubes |
| Conical flasks | For static bacterial culture and OD measurement | ||
| Digital camera for microscope | Olympus | DP71 | For image capture and harvesting of IBCs. Any other fluorescent microscope with a GFP channel will suffice |
| Glass Capillaries | Kimax | 6148K07 | |
| Iris Scissors STR SS 110MM | Braun | BC110R | |
| Isoflurane (Isothesia) | Henry Schein Animal Health | 29405 | |
| Kanamycin Sulfate | Calbiochem | 420311 | For static bacterial culture and OD measurement |
| LB broth (Miller) | Thermo/Gibco | 10855021 | For static bacterial culture and OD measurement |
| Light source unit for microscope | Olympus | LG-PS2 | For image capture and harvesting of IBCs. Any other fluorescent microscope with a GFP channel will suffice |
| Lubricant | KY | Any similar commercial medical lubricant will suffice | |
| Macro fluorescence microscope | Olympus | MVX10 | For image capture and harvesting of IBCs. Any other fluorescent microscope with a GFP channel will suffice |
| Micropipette + micropipette tips | For static bacterial culture and OD measurement | ||
| PBS 1x | For static bacterial culture and OD measurement | ||
| Pipette controller + Pipettes | For static bacterial culture and OD measurement | ||
| Polyethylene Tubing | BD Intramedic | 427401 | |
| Precision Glide needle 30 G | BD Biosciences | 305107 | Possibly under new catalogue number (305106) |
| Splinter forceps curved | Braun | BD312R | |
| Spray bottle (for ethanol) | For static bacterial culture and OD measurement | ||
| Square cuvettes | Elkay | 127-1010-400 | For static bacterial culture and OD measurement |
| Sterilgard III Advance Safety Cabinet | Baker | SG403 | Any biosafety cabinet with a UV irridiator |
| Sterilin 90mm Standard Petri Dish | Thermo | 101VR20 | Any sterile petri dish |
| Stevens, vascular and tendon scissors, curved, delicate, 110 mm | Braun | OK366R | Recommended for harvesting of bladder |
| Surgical Scissors STR S/B 105MM | Braun | BC320R | |
| Tabletop Centrifuge | Eppendorf | 5810R | Any refridgerated centrifuge for 15ml conicals |
| WPA C08000 cell density meter | Biowave (Biochrom) | 80-3000-45 | For static bacterial culture and OD measurement |
Referências
- Barber, A. E., Norton, P. J., Spivak, A. M., Mulvey, M. A. Urinary Tract Infections: Current and Emerging Management Strategies. Clinical Infectious Diseases. 57 (5), 719-724 (2013).
- Flores-Mireles, A. L., Walker, J. N., Caparon, M., Hultgren, S. J. Urinary tract infections: epidemiology, mechanisms of infection and treatment options. Nature Reviews Microbiology. 13, 269-284 (2015).
- Foxman, B. Recurring urinary tract infection: incidence and risk factors. American Journal of Public Health. 80, 331-333 (1990).
- Foxman, B., Barlow, R., D'Arcy, H., Gillespie, B., Sobel, J. D. Urinary tract infection: self-reported incidence and associated costs. Annals of Epidemiology. 10 (8), 509-515 (2000).
- Zowawi, H. M., et al. The emerging threat of multidrug-resistant Gram-negative bacteria in urology. Nature Reviews Urology. 12, 570-584 (2015).
- Silverman, J. A., Schreiber, H. L., Hooton, T. M., Hultgren, S. J. From physiology to pharmacy: developments in the pathogenesis and treatment of recurrent urinary tract infections. Current Urology Reports. 14, 448-456 (2013).
- Sivick, K. E., Mobley, H. L. T. Waging war against uropathogenic Escherichia coli: winning back the urinary tract. Infection and Immunity. 78, 568-585 (2010).
- Hung, C. -. S., Dodson, K. W., Hultgren, S. J. A murine model of urinary tract infection. Nature Protocols. 4, 1230-1243 (2009).
- Cusumano, C. K., et al. Treatment and prevention of urinary tract infection with orally active FimH inhibitors. Science Translational Medicine. , (2011).
- Alteri, C. J., Hagan, E. C., Sivick, K. E., Smith, S. N., Mobley, H. L. T. Mucosal Immunization with Iron Receptor Antigens Protects against Urinary Tract Infection. PLoS Pathogens. , (2009).
- Mysorekar, I. U., Hultgren, S. J. Mechanisms of uropathogenic Escherichia coli persistence and eradication from the urinary tract. Proceedings of the National Academy of Sciences of the United States of America. , (2006).
- Hunstad, D. A., Justice, S. S. Intracellular lifestyles and immune evasion strategies of uropathogenic Escherichia coli. Annual Review of Microbiology. 64, 203-221 (2010).
- Rosen, D. A., Hooton, T. M., Stamm, W. E., Humphrey, P. A., Hultgren, S. J. Detection of intracellular bacterial communities in human urinary tract infection. PLoS Medicine. , (2007).
- Robino, L., et al. Detection of intracellular bacterial communities in a child with Escherichia coli recurrent urinary tract infections. Pathogens and Disease. 68 (3), 78-81 (2013).
- Robino, L., et al. Intracellular bacteria in the pathogenesis of Escherichia coli urinary tract infection in children. Clinical Infectious Diseases. 59 (11), 158-164 (2014).
- Schwartz, D. J., Chen, S. L., Hultgren, S. J., Seed, P. C. Population dynamics and niche distribution of uropathogenic Escherichia coli during acute and chronic urinary tract infection. Infection and Immunity. 79, 4250-4259 (2011).
- Keshtkar, A., Keshtkar, A., Lawford, P. Cellular morphological parameters of the human urinary bladder (malignant and normal). International Journal of Experimental Pathology. 88, 185-190 (2007).
- Duraiswamy, S., Chee, J. L. Y., Chen, S., Yang, E., Lees, K., Chen, S. L. Purification of Intracellular Bacterial Communities during Experimental Urinary Tract Infection Reveals an Abundant and Viable Bacterial Reservoir. Infection and Immunity. , (2018).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nature Protocols. 2, 739-752 (2007).
- Tang, F., et al. Deterministic and stochastic allele specific gene expression in single mouse blastomeres. PLoS One. , (2011).
- Wells, J. M., Melton, D. A. Early mouse endoderm is patterned by soluble factors from adjacent germ layers. Development. 127, 1563-1572 (2000).
- Guo, H., et al. Profiling DNA methylome landscapes of mammalian cells with single-cell reduced-representation bisulfite sequencing. Nature Protocols. 10 (5), 645-659 (2015).
- Zhao, R., et al. The establishment of clonally derived chicken embryonic fibroblast cell line (CSC) with high transfection efficiency and ability as a feeder cell. Journal of Cellular Biochemistry. , (2018).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nature Protocols. 5 (3), 516-535 (2010).
- Wiegert, J. S., Gee, C. E., Oertner, T. G. Single-Cell Electroporation of Neurons. Cold Spring Harbor Protocols. , (2017).
- Conover, M. S., Flores-Mireles, A. L., Hibbing, M. E., Dodson, K., Hultgren, S. J. Establishment and Characterization of UTI and CAUTI in a Mouse Model. Journal of Visualized Experiments. (100), 52892 (2015).
- Justice, S. S., et al. Differentiation and developmental pathways of uropathogenic Escherichia coli in urinary tract pathogenesis. Proceedings of the National Academy of Sciences of the United States of America. 101 (5), 1333-1338 (2004).
- Mulvey, M. A., et al. Induction and Evasion of Host Defenses by Type 1-Piliated Uropathogenic Escherichia coli. Science. 282 (5393), 1494-1497 (1998).
- Zhang, H., Susanto, T. T., Wan, Y., Chen, S. L. Comprehensive mutagenesis of the fimS promoter regulatory switch reveals novel regulation of type 1 pili in uropathogenic Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 113 (15), 4182-4187 (2016).
- Gally, D. L., Bogan, J. A., Eisenstein, B. I., Blomfield, I. C. Environmental regulation of the fim switch controlling type 1 fimbrial phase variation in Escherichia coli K-12: effects of temperature and media. Journal of Bacteriology. 175 (19), 6186-6193 (1993).
- Olson, P. D., Hruska, K. A., Hunstad, D. A. Androgens Enhance Male Urinary Tract Infection Severity in a New Model. Journal of the American Society of Nephrology. 27 (6), 1625-1634 (2016).
- Garofalo, C. K., et al. Escherichia coli from Urine of Female Patients with Urinary Tract Infections Is Competent for Intracellular Bacterial Community Formation. Infection and Immunity. 75 (1), 52-60 (2007).
- Berry, R. E., Klumpp, D. J., Schaeffer, A. J. Urothelial cultures support intracellular bacterial community formation by uropathogenic Escherichia coli. Infection and Immunity. 77 (7), 2762-2772 (2009).
- Holden, N., Totsika, M., Dixon, L., Catherwood, K., Gally, D. L. Regulation of P-fimbrial phase variation frequencies in Escherichia coli CFT073. Infection and Immunity. 75 (7), 3325-3334 (2007).
- Jost, S. P. Postnatal growth of the mouse bladder. Journal of Anatomy. 143, 39-43 (1985).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados