É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Caracterização de imagens de refletância interferométrica de partícula única de vesículas extracelulares individuais e dinâmica populacional
Neste Artigo
Resumo
Este protocolo apresenta imagens de refletância interferométrica de partícula única projetadas para medições abrangentes e multinível de tamanho de vesículas extracelulares (EV), contagem de EV, fenótipo EV e colocalização de biomarcadores EV.
Resumo
As vesículas extracelulares (EVs) são vesículas de tamanho nanométrico com uma bicamada lipídica que são secretadas pela maioria das células. Os EVs carregam uma infinidade de moléculas biológicas diferentes, incluindo proteínas, lipídios, DNA e RNA, e são postulados para facilitar a comunicação célula a célula em diversos tecidos e órgãos. Recentemente, os EVs têm atraído atenção significativa como biomarcadores para diagnósticos e agentes terapêuticos para várias doenças. Muitos métodos foram desenvolvidos para caracterização de EV. No entanto, os métodos atuais para análise de VE têm limitações diferentes. Assim, o desenvolvimento de métodos eficientes e eficazes para isolamento e caracterização de EV continua sendo uma das etapas cruciais para este campo de pesquisa de ponta à medida que amadurece. Aqui, fornecemos um protocolo detalhado descrevendo um sensor de imagem de refletância interferométrica de partícula única (SP-IRIS), como um método capaz de detectar e caracterizar EVs de fontes biológicas não purificadas e EVs purificadas por outras metodologias. Essa técnica avançada pode ser usada para medições abrangentes e de vários níveis para a análise do tamanho do EV, contagem de EV, fenótipo de EV e colocalização de biomarcadores.
Introdução
As vesículas extracelulares (EVs) são vesículas de membrana de tamanho nanométrico de origem celular que podem ser isoladas de vários fluidos biológicos, incluindo sangue, leite materno, saliva, urina, bile, suco pancreático e fluidos cefalorraquidianos e peritoneais. A derivação de EVs ocorre por meio de três mecanismos principais: apoptose, liberação via fusão de corpos multivesiculares com a membrana plasmática e bolhas da membrana plasmática1. Evidências de transferência EV de componentes de células doadoras para células e tecidos vizinhos ou distantes sugerem que esses pacotes fechados por membrana podem desempenhar papéis importantes em cascatas de sinalização parácrina, bem como de longa distância ou endócrina 1,2,3. Como os EVs podem fornecer um instantâneo do fenótipo de uma célula, o potencial para seu uso como ferramentas diagnósticas e terapêuticas para o tratamento de várias doenças tornou-se uma área ativa de pesquisa 4,5,6,7,8.
Muitos métodos voltados para a caracterização de EV foram desenvolvidos 9,10,11,12,13. A maioria desses métodos fornece informações exclusivas e valiosas sobre populações de EVs principalmente em massa. Embora um subconjunto dessas técnicas possa fornecer detalhes sobre substâncias dentro ou em EVs únicos, pode haver limitações para caracterizar EVs no nível de EV único. Por exemplo, a microscopia imunoeletrônica pode ser usada para entender EVs únicos e sua composição, mas essa técnica é de baixo rendimento, severamente limitada em sua capacidade de ser usada para descrever a dinâmica populacional e requer o desenvolvimento significativode métodos 14.
Recentemente, o desenvolvimento e a comercialização da técnica de sensor de imagem de refletância interferométrica de partícula única (SP-IRIS), por meio da plataforma ExoView, abriram a caracterização individual de EV usando um método automatizado de coleta de dados simples e de rotina. O núcleo dessa tecnologia é o chip, uma camada dupla de 1 cm x 1 cm Si/SiO2 , que permite a medição interferométrica de nanopartículas biológicas únicas. O chip é cultivado com um microarray de pontos de anticorpos funcionalizados individuais, permitindo a detecção multiplexada de até seis tipos diferentes de captura. Os chips padrão incluem os marcadores comuns de tetraspanina (CD81, CD63 e CD9) para captura durante a etapa de incubação, e o usuário pode adicionar pontos de captura personalizados adicionais para isolar populações distintas de EVs separadas das tetraspaninas. Após a etapa de incubação, cada ponto de captura vinculou muitos EVs a ele que expressam o marcador correspondente. Esses EVs capturados podem então ser simplesmente lavados, secos e digitalizados no leitor para quantificar o tamanho das vesículas ligadas ao local de captura entre 50-200 nm para fornecer uma distribuição de tamanho ponderada via SP-IRIS15. O sistema também oferece três canais de detecção fluorescente para imunomarcar os EVs capturados e fornece a intensidade fluorescente média, que não é limitada pelo tamanho, como medições SP-IRIS, e aspectos de colocalização para cada coloração fluorescente. Isso permite que o usuário defina populações de EVs únicos com base na exibição de quatro biomarcadores diferentes por EV (captura mais três marcadores imunofluorescentes). O sistema pode ir além da medição de proteínas de superfície com imunofluorescência, pois um protocolo de carga opcional permite ao usuário sondar proteínas internas dos EVs capturados e epítopos luminais de marcadores de superfície que abrangem a membrana, além de permitir que o usuário verifique a integridade da membrana EV. Neste artigo, fornecemos um protocolo detalhado descrevendo as etapas necessárias para obter dados consistentes sobre o tamanho e o número de EVs, com até quatro biomarcadores diferentes em um único nível de EV em grandes populações de EVs. Essa técnica pode ser usada em fluidos biológicos não processados e EVs isolados usando qualquer número de técnicas, como ultracentrifugação, ultrafiltração, agentes precipitantes, captura de imunoafinidade, microfluídica e cromatografia de exclusão de tamanho.
O protocolo descrito abaixo usa vesículas extracelulares (EV) derivadas de meios de cultura de células HEK 293 e do soro de camundongo usando um método de isolamento estabelecido16. O protocolo foi aplicado a vários outros fluidos biológicos, meio de cultura de células e vesículas extracelulares purificadas isoladas de fluidos biológicos. Este protocolo é dividido em um procedimento de dois dias com o fluxo de trabalho para um experimento típico mostrado na Figura 1.
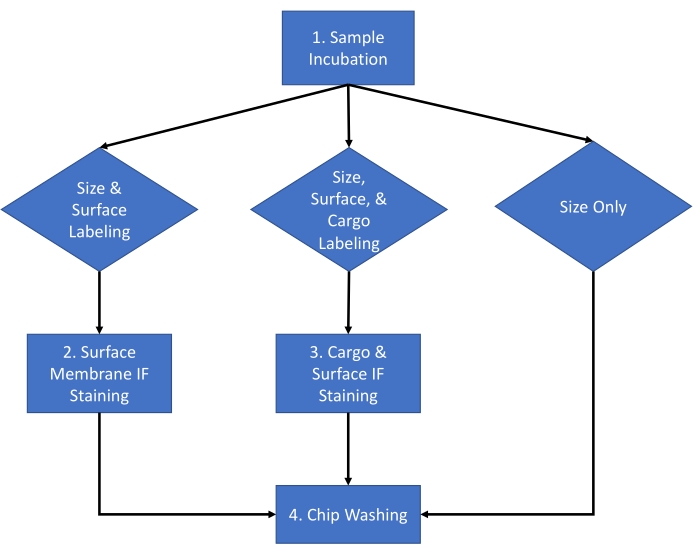
Figura 1: Fluxo de trabalho do ensaio. Fluxo de trabalho de ensaio para escolher o tipo de análise a ser concluída para a amostra entre tamanho e contagem, contagem de tamanho e coloração de superfície e contagem de tamanho e coloração de carga. Clique aqui para ver uma versão maior desta figura.
Protocolo
Amostras de soro foram coletadas de camundongos de acordo com um protocolo aprovado pelos Comitês Institucionais de Cuidados e Uso de Animais (IACUC) no Centro Médico da Universidade do Kansas (KUMC). O uso dessas amostras biológicas nesses experimentos também foi aprovado pelo KUMC.
1. Preparação da amostra (Dia 1)
- Determine a concentração de EV usando rastreamento de nanopartículas ou uma técnica equivalente.
- Diluir a amostra com a solução de incubação até uma concentração de 5 x 107-5 x 108 EVs/mL; é necessário um mínimo de 50 μL.
NOTA: Para amostras em que a concentração de EV é desconhecida, a proteína total a 1 μg/mL pode ser usada como medida substituta. Se for previsível que a amostra seja de baixa concentração, execute pelo menos uma diluição de 1:1 na solução de incubação antes de carregá-la. - Coloque uma placa de 24 poços em uma superfície plana, livre de vibrações e movimentos bruscos.
NOTA: Para aumentar o contraste, uma folha de papel branca pode ser colocada sob a placa. - Adicione água às áreas ao redor dos poços (Figura 2).
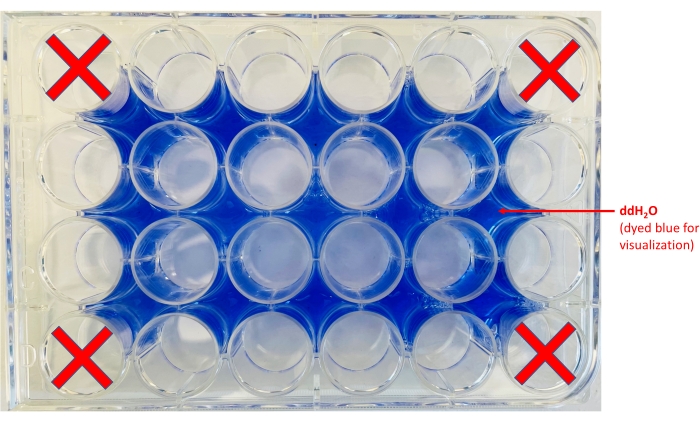
Figura 2: Layout da placa de 24 poços. Os locais de onde alíquota ddH2O (corante azul foi adicionado apenas para fins de visualização) e os poços nos quais os chips serão mantidos são mostrados. Clique aqui para ver uma versão maior desta figura.
2. Preparação e pré-digitalização de chips
- Remova a placa selada de 48 poços contendo cavacos da geladeira de 4-8 ° C e deixe-os atingir a temperatura ambiente (~ 15 min) antes de abrir a vedação.
NOTA: Isso é essencial para evitar condensação nos cavacos que podem danificar as manchas. Se for observada condensação na superfície ao remover o chip da embalagem, espere mais tempo antes de remover os outros. - Prossiga para a etapa 8 para recuperar o mandril para se preparar para a execução da pré-varredura (Figura 3).
NOTA: Os dados de pré-varredura serão usados para identificar quaisquer partículas detectáveis nos pontos de captura antes da incubação com a amostra para que possam ser removidas durante a etapa de análise.

Figura 3: Imagem do mandril usado para carregar o cavaco na máquina. Clique aqui para ver uma versão maior desta figura.
- Usando uma pinça, remova o número desejado de cavacos (um cavaco por amostra) da placa de 48 poços e carregue os cavacos diretamente no mandril para a execução da pré-varredura. Quando todos os chips destinados a serem usados para o experimento tiverem sido carregados e pré-digitalizados conforme descrito na etapa 8, prossiga para a etapa 2.4.
NOTA: Ao manusear chips, certifique-se de não tocar nos quadrados no centro, pois os pontos de captura de anticorpos estão nessa região e serão danificados se tocados pela pinça (Figura 4).

Figura 4: Chip e manuseio adequado do chip. (A) A linha pontilhada amarela indica a localização dos anticorpos detectados ou o lado funcional do chip. O ID do chip está localizado abaixo da linha ("58"). A figura também mostra o manuseio adequado. (B) Demonstra manuseio inadequado do chip. (C) Lado não funcional do chip. Clique aqui para ver uma versão maior desta figura.
- Coloque cada chip pré-escaneado com a superfície funcionalizada voltada para cima na placa pré-preparada de 24 poços. A superfície funcionalizada é facilmente reconhecida pela numeração distinta, grades de alinhamento e as três caixas pretas no meio. O lado não funcionalizado é uma superfície plana e uniforme prateada.
- Use uma pinça de ponta fina para garantir que cada cavaco esteja centralizado dentro do poço (Figura 5).
NOTA: Centralizar o cavaco dentro do poço é fundamental porque se o cavaco tocar a parede do poço, a amostra pode ser eliminada após o carregamento.

Figura 5: Demonstração da colocação adequada de cavacos no poço. (A) Os cavacos devem ser colocados no meio do poço, sem cantos tocando as laterais do poço. (B) Representação de colocação inadequada do chip, onde os cantos tocam as laterais do poço. Clique aqui para ver uma versão maior desta figura.
3. Carregamento e incubação do chip e da amostra
- Pipetar 35 μL da amostra preparada (do passo 1) para a microplaqueta.
NOTA: Tenha cuidado para não adicionar bolhas ou tocar no chip com a ponta da pipeta, pois isso pode impedir a distribuição uniforme da amostra no chip ou danificar os pontos de anticorpos no chip. A amostra deve se espalhar por toda a superfície do chip. No caso de a amostra se desprender da aparas durante o carregamento, adicionar uma amostra adicional ao alvéolo até um volume de 250 μL ou adicionar uma solução de incubação até 250 μL e observar o fator de diluição alterado para essa amostra. - Sele a placa, usando o filme da placa incluído no kit para evitar a evaporação da amostra.
- Incubar a amostra/chip durante a noite (~16 h) na placa selada à temperatura ambiente em uma área livre de vibrações ou movimentos.
NOTA: Após a incubação noturna, o usuário escolherá a opção apropriada do Dia 2 para sua pergunta de pesquisa. Se apenas o tamanho e a contagem de EV forem desejados, prossiga com a etapa 4 (Dia 2). Se a análise de vários marcadores de superfície for desejada, pule para a etapa 5 (Dia 2).
4. Determinando o tamanho e a contagem de EV (Dia 2)
- Adicione 1 mL da solução A na lateral de cada poço contendo um chip, tomando cuidado para não adicionar diretamente a solução no chip ou arranhar o chip com a ponta da pipeta.
- Coloque a placa em um agitador orbital girando a ~500 rpm por 3 min em temperatura ambiente.
NOTA: Se as lascas chacoalharem na placa, diminua imediatamente a velocidade de forma que o líquido fique girando, mas não haja barulho. - Remova 750 μL de líquido. Evite inclinar a placa durante a remoção de líquidos para evitar a secagem acidental do cavaco.
- Adicione 750 μL de Solução B usando a técnica descrita na etapa 4.1 e agite a ~ 500 rpm por 3 min em temperatura ambiente.
- Repita as etapas 4.3-4.4 mais duas vezes para um total de três lavagens com a Solução B.
- No final da última lavagem, remova 750 μL da solução, deixando 250 μL da Solução B no poço.
- Adicione 750 μL de água bidestilada (ddH2O) e agite a ~500 rpm por 3 min em temperatura ambiente.
- Encha uma placa de Petri de 10 cm com 50 mL de ddH2O e transfira um chip de cada vez do poço para a placa usando uma pinça.
NOTA: Tome cuidado para transferir o chip horizontalmente e certifique-se de que não seque. Até oito cavacos podem ser lavados antes de substituir o ddH2O. - No ddH2O, segure o chip pelas bordas usando uma pinça e gire no prato por três voltas para remover os detritos.
- Remova o chip em um ângulo de 45° para fora da água e coloque-o em papel absorvente com o ID do chip voltado para cima (Figura 6).
NOTA: As fichas agora estão prontas para serem lidas. Pule para a etapa 8 para digitalizar no leitor SP-IRIS.

Figura 6: Maneira correta de remover o cavaco da água ddH2O em um ângulo de 45°. (A) Vista de cima e (B) vista de lado demonstrando o ângulo em que remover o cavaco. Clique aqui para ver uma versão maior desta figura.
5. Preparação da solução de anticorpos (Dia 2)
- Adicione 300 μL de solução de bloqueio por chip em um tubo de tamanho apropriado (0,5 a 3 mL).
- Adicione 0,6 μL de anticorpo por 300 μL da solução de bloqueio. Misture delicadamente batendo no tubo e centrifugue rapidamente.
NOTA: Anticorpos para CD9, CD81 e CD63 humanos ou CD9, CD81 e CD63 murinos estão incluídos nos kits apropriados. Se outros anticorpos conjugados fluorescentes forem desejados, determine a concentração de coloração ideal usando uma titulação típica em uma faixa de 0,1 a 10 μg/mL com uma carga de amostra constante, como faria para citometria de fluxo.
6. Determinando o tamanho, a contagem e a fenotipagem do EV com coloração imunofluorescente
- Adicione 1 mL de Solução A na lateral de cada poço contendo um cavaco, tomando cuidado para não adicionar diretamente a solução no cavaco ou arranhar o cavaco com a ponta da pipeta.
- Coloque a placa em um agitador orbital girando a ~500 rpm por 3 min em temperatura ambiente.
NOTA: Se as lascas chacoalharem na placa, diminua imediatamente a velocidade de forma que o líquido fique girando, mas não haja barulho. - Remova 750 μL do líquido. Evite inclinar a placa durante a remoção de líquidos para evitar a secagem acidental do cavaco.
- Adicione 750 μL de Solução A usando a técnica descrita na etapa 6.1 e agite a ~ 500 rpm por 3 minutos em temperatura ambiente.
- Repita as etapas 6.3 e 6.4 duas vezes para um total de três lavagens com a Solução A.
- No final da última lavagem, remova 750 μL da solução, deixando 250 μL da Solução A no poço.
NOTA: Se o usuário desejar Coloração de Carga, prossiga para a etapa 7 neste ponto. Caso contrário, prossiga para a etapa 6.7 - Adicione 250 μL da solução de anticorpos (etapa 5) a cada chip no poço. Cubra a placa com papel alumínio para proteger da luz e agite por 1 h no agitador orbital à temperatura ambiente.
- Adicione 500 μL de Solução A, de modo que o volume total por poço seja ~ 1.000 μL.
- Remova imediatamente 750 μL e adicione 750 μL de solução A e agite a ~ 500 rpm no agitador orbital por 3 min em temperatura ambiente.
- Remova 750 μL da solução e adicione 750 μL da Solução B e agite a ~ 500 rpm por 3 minutos em temperatura ambiente.
- Repita a etapa 6.10 mais duas vezes para um total de três lavagens.
- Adicione 750 μL de água bidestilada (ddH2O) e agite a ~500 rpm por 3 min em temperatura ambiente.
- Encha uma placa de Petri de 10 cm com 50 mL de ddH2O e transfira um chip do poço para a placa usando uma pinça.
NOTA: Tome cuidado para transferir o chip horizontalmente e certifique-se de que não seque. Até oito cavacos podem ser lavados antes da troca por ddH2O fresco. - No ddH2O, segure o cavaco pelas bordas usando uma pinça e gire no prato por três rotações para remover os detritos.
- Remova o chip em um ângulo de 45° para fora da água e coloque-o em papel absorvente com o ID do chip voltado para cima (Figura 6).
NOTA: As fichas agora estão prontas para serem lidas. Pule para a etapa 8 (Coleta de dados) para obter instruções sobre como configurar os chips para digitalização no leitor.
7. Coloração de carga opcional
NOTA: Este protocolo permite a rotulagem simultânea de marcadores internos e de superfície.
- Com 250 μL de Solução A restantes em cada poço, adicione 250 μL de Solução C em cada poço.
- Coloque imediatamente no agitador orbital e ajuste para ~200 rpm por precisamente 10 min.
NOTA: Nesta etapa e na etapa 7.8 abaixo, tanto o tempo quanto a velocidade de agitação mais lenta são críticos; Certifique-se de que a velocidade dê apenas um redemoinho lento nos poços e use um cronômetro. - Após 10 minutos de incubação com a solução C, adicione 500 μL de solução A a cada poço.
- Remova imediatamente 750 μL e adicione 750 μL de Solução A e agite a ~500 rpm no agitador orbital por 3 min em temperatura ambiente.
- Repita a etapa 7.4 mais duas vezes para um total de três lavagens.
- No final da última lavagem, remova 750 μL da solução, deixando ~ 250 μL da solução no poço com o cavaco.
- Adicione 250 μL de Solução D a cada poço.
- Coloque imediatamente no agitador orbital ajustado para ~200 rpm por precisamente 10 min.
- Após 10 minutos de incubação com a solução C, adicione 500 μL de solução A a cada poço.
- Remova imediatamente 750 μL e adicione 750 μL de solução A e agite a ~ 500 rpm no agitador orbital por 3 min em temperatura ambiente.
- Repita a etapa 7.10 duas vezes para um total de três lavagens.
- Remova 750 μL da solução após a última lavagem e retorne à etapa 6.7 acima para coloração e conclusão do protocolo de ensaio.
8. Coleta de dados
NOTA: O procedimento para coletar dados dos chips usando o ExoView R100 é automatizado e não requer entradas do usuário. Instruções detalhadas podem ser encontradas no Guia do Usuário e no vídeo correspondente para carregar o suporte do chip, ou "mandril", e aquisição de dados17.
- Ligue a plataforma de caracterização EV usando os dois interruptores de energia na parte traseira do instrumento e, em seguida, inicie o software do scanner clicando duas vezes no ícone da área de trabalho.
NOTA: Quando o software do scanner for iniciado, o leitor será definido automaticamente para a tela inicial e, em seguida, solicitará ao usuário que "Abra a porta para carregar os chips". - Abra a porta na frente do leitor levantando a maçaneta prateada. Isso ejetará a platina, permitindo que o usuário acesse o mandril e configure uma nova varredura no software.
- Identifique o local onde o usuário deseja salvar os dados clicando na pasta Salvar e selecionando um local desejado para os dados a serem salvos.
NOTA: Muitas vezes é útil nomear o local de salvamento com algo perspicaz como o nome do experimento e, em seguida, criar duas subpastas, uma para pré-varreduras e outra para pós-varreduras. Isso facilita a localização e a correspondência dos dados durante a análise. - Para escanear os chips, localize os chipfiles no computador de controle.
NOTA: Os arquivos de chip são mapas do layout do ponto de anticorpo no chip em uso e permitem que o scanner saiba onde digitalizar em cada chip. Em cada kit, há uma chave USB com os arquivos de chip para os chips dentro. Eles devem ser salvos em um local no computador de controle após o recebimento do kit, onde os usuários possam encontrá-los de forma confiável. - Carregue os chips no mandril com o número nos cavacos voltado para a alça do mandril e, em seguida, no menu suspenso Chip no computador, selecione cada chip da lista de arquivos de chip e coloque-o no local correspondente apropriado no mandril virtual no software do scanner. Depois de concluído, um prompt na tela solicitando que o usuário coloque o mandril no palco aparecerá.
- Coloque o mandril carregado no palco; o alinhamento magnético no mandril irá movê-lo automaticamente para o local correto no palco e, em seguida, clique em OK ao lado de Colocar o mandril no palco. O scanner iniciará a rotina automatizada de coleta de dados.
NOTA: Os dados estão prontos para serem analisados assim que o software relatar o status de varredura de cada chip como bem-sucedido.
9. Análise dos dados
- Clique duas vezes em ExoView Analyzer na área de trabalho do PC de controle. Após a inicialização do software, clique no botão Dados de pré-varredura e selecione o local da pasta para o conjunto de dados de pré-varredura na seção 8.3.
- Clique no botão Dados do Postscan e selecione o local da pasta para o conjunto de dados do postscan na seção 8.3.
NOTA: Se os dados forem detectados corretamente em ambas as pastas, para pelo menos alguns dos chips, o software exibirá o número "X" de chips detectados ao lado do botão de localização do Chipfile. Quando salvos corretamente, os chips detectados devem corresponder ao número de chips verificados. - Clique em Avançar na parte inferior da área de carregamento de dados.
- No menu suspenso da metatabela, cada ficha terá uma célula. Insira os nomes das amostras, fatores de diluição e quais marcadores estão sendo corados em cada canal de detecção clicando na caixa correspondente e digitando as informações; quando concluído, clique em Avançar novamente.
NOTA: Esses metadados serão salvos com os dados. - Controle de qualidade de pré-formas (CQ) clicando na guia Desativar sob o botão CQ no canto superior esquerdo.
NOTA: Esse recurso permite que sondas e chips específicos sejam desligados para análise. O software fornecerá dois avisos diferentes sobre os dados, um para contagens altas que podem indicar que uma determinada captura está saturada e um alto coeficiente de variação (CV) que identifica quando uma das réplicas de um tipo de captura é diferente das outras.- Clique em um ponto de aviso para CV alto, examine os pontos que estão sendo chamados pelo software e veja se há danos físicos óbvios; quando o dano for identificado, clique no Número do Ponto para desativar esse ponto na análise.
- Repita até que todos os avisos tenham sido avaliados e clique em Avançar.
- Análise de corte de pré-forma clicando na guia Corte localizada ao lado da guia Desativar no botão CQ no canto superior esquerdo.
NOTA: Isso apresentará a resposta fluorescente do ponto de controle em um gráfico e oferecerá ao usuário duas configurações, um mínimo e um máximo para cada canal de cor (vermelho/verde/azul). Definir o valor de corte para cada canal de detecção fluorescente é o único ajuste de dados que precisa ser feito e é relativamente simples. É importante ressaltar que o ponto de corte final escolhido deve ser consistente em um experimento e, conforme observado, deve estar dentro das regras gerais para 300-400 u.a. em vermelho e verde, e 400-700 u.a. para azul em experimentos típicos; O software também possui guias codificados por cores para o usuário para essas faixas.- Clique na guia Cor Verde para carregar os dados desse canal e exibir os cortes atuais. Examinando o ponto de controle negativo do isotipo, pode-se ver eventos de detecção com intensidades de fluorescência muito baixas; Nesse caso, os usuários normalmente devem esperar definir o verde entre 300-400 UA.
- Aumente o mínimo para cada um dos canais de detecção até que a "Avg % Inc" (a % média das partículas detectadas no ponto de controle incluído acima do corte) sob a exibição de dados não tenha avisos conforme indicado pelo realce vermelho ou amarelo nessa célula.
NOTA: O valor máximo geralmente não precisa ser ajustado do padrão, mas pode ser reduzido para filtrar partículas brilhantes ou limitar a detecção fluorescente a uma faixa mais estreita. - Clique no botão Avançar .
- Aumente o mínimo para cada um dos canais de detecção até que a "Avg % Inc" (a % média das partículas detectadas no ponto de controle incluído acima do corte) sob a exibição de dados não tenha avisos conforme indicado pelo realce vermelho ou amarelo nessa célula.
- Clique na guia Cor Vermelha para carregar os dados desse canal e exibir os cortes atuais. Novamente, examinando o ponto de controle negativo do isotipo, pode-se ver eventos de detecção com intensidades de fluorescência muito baixas; Nesse caso, os usuários normalmente devem esperar definir o vermelho entre 300-400 UA. Siga os mesmos procedimentos observados nas etapas 9.6.1.1 a 9.6.1.3.
- Clique na guia Cor Azul para carregar os dados desse canal e exibir os cortes atuais. Mais uma vez, examinando o ponto de controlo negativo do isotipo, seguir os mesmos procedimentos indicados nos passos 9.6.1.1 a 9.6.1.3.
NOTA: O canal azul é único, pois o anticorpo no local é autofluorescente em níveis suficientes para exigir um corte ligeiramente maior de 400-700 u.a., mesmo para chips em branco.
- Clique na guia Cor Verde para carregar os dados desse canal e exibir os cortes atuais. Examinando o ponto de controle negativo do isotipo, pode-se ver eventos de detecção com intensidades de fluorescência muito baixas; Nesse caso, os usuários normalmente devem esperar definir o verde entre 300-400 UA.
- Após a conclusão da análise de corte, clique em Avançar e o gráfico do mapa de calor aparecerá, o que fornecerá uma visão geral da ligação de partículas no experimento. Existem várias ferramentas de visualização que o usuário pode usar para plotar os dados. Esses gráficos e dados brutos associados podem ser adicionados a um relatório clicando no botão Adicionar relatório de plotagem .
NOTA: O mapa de calor é a exibição de dados padrão que mostra uma visualização de todas as amostras, todas as capturas de uma detecção específica. A exibição padrão é o total que quantifica os EVs exclusivos vinculados a cada ponto de captura. - Clique no botão Exportar relatório e selecione um local para salvar o relatório.
NOTA: O relatório será aberto em um navegador. Localize o arquivo de planilha da Lista de partículas filtradas anexado ao relatório; esta é a informação a submeter à EVTRACK. - Continue adicionando relatórios usando as diferentes seleções de Amostras e Tipos de captura nos menus suspensos na parte superior da tela. Os dados exibidos são controlados com a seleção de botões de detecção de canal no canto superior esquerdo. Defina um fenótipo/seleção de amostra/canal de captura específico clicando em cada botão de cor para desativar (esmaecido) ou ativar (colorido) esse canal de detecção.
- Clique no botão Adicionar Plotagem ao Relatório para agregar as parcelas desejadas em um relatório.
- Clique em Exportar Relatório e defina o local de salvamento para o relatório final.
Resultados
A Figura 7 (painel esquerdo) mostra uma imagem composta de três cores de EVs derivadas de mídia condicionada HEK293 ligada ao ponto CD63 no chip e corada para CD81, CD63 e CD9 nos seguintes canais verde, vermelho e azul, respectivamente. A Figura 7 (painel superior direito) é uma imagem ampliada que mostra que cada um dos EVs capturados pode exibir a colocalização de uma ou mais cores com intensidades variadas em cada canal...
Discussão
Os métodos atuais de caracterização de EV dependem em grande parte de EVs purificados, o que é restrito pelas limitações experimentais atuais dos métodos de purificação de EV 9,10,11,12,13. A imagem de refletância interferométrica de partícula única (SP-IRIS) é uma tecnologia eficaz que pode eliminar as etapas de...
Divulgações
Clayton Deighan e George Daaboul são funcionários e acionistas da NanoView Biosciences Inc.
Agradecimentos
Este trabalho foi patrocinado em parte pelo Programa de Prêmio de Aquisição de Recursos e Equipamentos de Pesquisa da Escola de Medicina da Universidade do Kansas. PCG, LKC, FD e AR foram apoiados com fundos do NIA R21 AG066488-01.
Materiais
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
Referências
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356 (2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374 (2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386 (2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016 (2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246 (2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202 (2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728 (2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados