É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Metodologia para Inativação Metabolicamente de Bactérias para Pesquisa de Caenorhabditis elegans
Neste Artigo
Resumo
A fonte de alimento para Caenorhabditis elegans no laboratório é Escherichia coli viva. Como as bactérias são metabolicamente ativas, elas apresentam uma variável de confusão em estudos metabólicos e de drogas em C. elegans. Um protocolo detalhado para inativar metabolicamente bactérias usando paraformaldeído é descrito aqui.
Resumo
Caenorhabditis elegans é um organismo modelo comum para pesquisa em genética, desenvolvimento, envelhecimento, metabolismo e comportamento. Como C. elegans consomem uma dieta de bactérias vivas, a atividade metabólica de sua fonte alimentar pode confundir experimentos que procuram os efeitos diretos de várias intervenções sobre o verme. Para evitar os efeitos de confusão do metabolismo bacteriano, os pesquisadores de C. elegans usaram vários métodos para inativar metabolicamente as bactérias, incluindo irradiação ultravioleta (UV), morte por calor e antibióticos. O tratamento UV é relativamente baixo rendimento e não pode ser usado em cultura líquida porque cada placa deve ser examinada para matar bactérias com sucesso. Um segundo método de tratamento, o termo-matança, afeta negativamente a textura e a qualidade nutricional das bactérias, levando à parada do desenvolvimento de C. elegans. Finalmente, o tratamento com antibióticos pode alterar diretamente a fisiologia de C. elegans , além de prevenir o crescimento bacteriano. Este manuscrito descreve um método alternativo para inativar metabolicamente bactérias usando paraformaldeído (PFA). O tratamento com PFA faz ligações cruzadas de proteínas dentro das células bacterianas para prevenir a atividade metabólica, preservando a estrutura celular e o conteúdo nutricional. Este método é de alto rendimento e pode ser usado em cultura líquida ou placas sólidas, pois o teste de uma placa de bactérias tratadas com PFA para crescimento valida todo o lote. A inativação metabólica através do tratamento com PFA pode ser usada para eliminar os efeitos de confusão do metabolismo bacteriano em estudos de suplementação de drogas ou metabólitos, resistência ao estresse, metabolômica e comportamento em C. elegans.
Introdução
Caenorhabditis elegans foi originalmente proposto como organismo modelo em 19651 e desde então tem sido amplamente adotado em estudos de genética, desenvolvimento, comportamento, envelhecimento e metabolismo2. Devido ao seu grande tamanho de ninhada e cutícula transparente, C. elegans é particularmente adequada para triagem de alto rendimento com repórteres fluorescentes3. Seu curto ciclo de vida, reprodução hermafrodita e homologia genética com humanos também fazem de C. elegans um valioso sistema modelo para estudos sobre o desenvolvimento4 e biologia do envelhecimento5. Além disso, C. elegans são relativamente fáceis de manter. Os vermes podem ser cultivados em cultura líquida ou em placas sólidas de ágar e consumir uma dieta de bactérias vivas de Escherichia coli OP504.
No entanto, a fonte de alimento vivo de C. elegans pode confundir estudos de metabolismo, suplementação de drogas e comportamento. Como as bactérias vivas têm seu próprio metabolismo, as condições experimentais que afetam as bactérias também alteram os nutrientes e metabólitos disponíveis para os vermes. Por exemplo, diferenças nas concentrações bacterianas de ferro, aminoácidos e folato têm diversos efeitos sobre o desenvolvimento, fisiologia e vida útil de C. elegans 6. Muitas práticas comuns de laboratório podem provocar tais mudanças na composição de nutrientes e metabólitos produzidos pelo OP50. Especificamente, a exposição à 5-fluor-2'-desoxiuridina (FUdR), um composto comumente usado para prevenir a reprodução em C. elegans, provoca amplas alterações na expressão gênica do gene OP50, incluindo vias de biossíntese de aminoácidos7. Bactérias vivas também podem confundir estudos em que C. elegans são suplementados com pequenas moléculas porque as bactérias podem metabolizar parcial ou completamente os compostos ativos. Além disso, os efeitos dessas pequenas moléculas sobre as bactérias podem, por sua vez, alterar a fisiologia de C. elegans, como foi relatado com o fármaco que prolonga a vida útil da metformina8. Finalmente, bactérias vivas podem alterar o ambiente do verme de maneiras que alteram o comportamento, como secretar odorantes atraentes9, produzir neuromoduladores exógenos10 e criar gradientes de oxigênio em um gramado bacteriano denso11.
Para atenuar os efeitos de confusão do metabolismo bacteriano na pesquisa de C. elegans , vários métodos para matar bactérias foram desenvolvidos (Tabela 1). Três estratégias comuns para matar OP50 são irradiação UV, morte por calor e tratamento com antibióticos. Embora simples e relativamente de baixo custo, cada um desses métodos pode ter efeitos indesejáveis sobre bactérias e C. elegans. A eliminação por UV através de um reticulador UV12 é de baixo rendimento e a taxa é limitada pelo número de placas que podem caber no reticulante UV. Além disso, a eficácia da eliminação por UV pode variar de placa para placa dentro de um lote, e o teste de crescimento em todas as placas pode se tornar difícil em grandes experimentos. Matar o calor OP50 expondo a cultura a temperaturas de >60 °C vem com um conjunto separado de desafios. O calor elevado pode danificar nutrientes essenciais para o verme e destruir a estrutura celular das bactérias, criando uma textura mais macia que diminui a quantidade de tempo que os vermes passam no alimento13. Este método também não pode ser usado durante todo o ciclo de vida de C. elegans , pois vermes alimentados com bactérias mortas pelo calor podem parar no início do desenvolvimento13. O tratamento com antibióticos é um terceiro método comum para suprimir o metabolismo bacteriano14, mas os antibióticos também podem alterar o crescimento e o metabolismo do verme15.
Uma solução para eliminar os efeitos metabólicos de bactérias vivas, preservando a estrutura bacteriana e nutrientes essenciais, é matar a OP50 com paraformaldeído (PFA)16. O PFA é um polímero de formaldeído que pode fazer ligações cruzadas entre proteínas dentro das células17 para impedir a replicação bacteriana sem destruir estruturas celulares internas, como a membrana plasmática interna18. Devido a essa preservação da estrutura celular interna, as bactérias tratadas com PFA não exibem crescimento ou atividade metabólica, mas permanecem como fonte alimentar comestível e rica em nutrientes para C. elegans16. Aqui, um protocolo detalhado é fornecido que mostra como inativar metabolicamente bactérias usando paraformaldeído.
| Método | Materiais necessários | Escalonável? | Nutricional? | Efeitos sobre o verme? | ||||
| UV | Reticulante UV | Limitado por: | Sim | Efeitos variáveis na vida útil de NGM12, 23, 24 | ||||
| Número de placas que se encaixam no reticulante UV | Efeitos variáveis na vida útil do FUdR24, 26, 27 | |||||||
| Tempo de irradiação por placa | Diminuição da preferência alimentar16 | |||||||
| Capacidade de verificar o crescimento de cada placa8 | ||||||||
| Calor | Incubadora >60 °C | Sim | Não: destrói a parede celular, diminuição do valor nutricional | Prisão preventiva 13 | ||||
| Diminuição da preferência alimentar13 | ||||||||
| Prolonga a vida útil no NGM31 | ||||||||
| Antibióticos | Antibióticos (canamicina, carbenicilina, etc.) | Sim | Sim | Atrasa o crescimento e o desenvolvimento15 | ||||
| Prolonga a vida útil em meios líquidos19 | ||||||||
| Prolonga a vida útil no NGM15 | ||||||||
| PFA | 0,5% Paraformaldeído | Sim | Sim | Diminuição do tamanho da ninhadapequena 16 | ||||
| Aumento do tempo de desenvolvimentopequeno 16 | ||||||||
| Diminuição da preferência alimentar16 | ||||||||
Tabela 1. Comparações de métodos para matar OP50. UV-killing, heat-killing, antibiotico-treatment, e PFA-treatment têm efeitos variados sobre o estado nutricional das bactérias e a saúde dos vermes alimentados com bactérias tratadas. Esses métodos para inativar replicativamente E. coli também diferem em seus materiais necessários e escalabilidade.
Protocolo
1. Inoculação de bactérias
- Preparar o caldo Luria (LB) dissolvendo 10 g de triptona, 5 g de extrato de levedura e 10 g de cloreto de sódio (NaCl) em 950 mL de água destilada.
- Ajustar o pH do LB para 7,0 adicionando hidróxido de sódio (NaOH) 5M. Isso deve exigir apenas cerca de 0,2 mL de NaOH.
- Autoclave o meio LB ajustado ao pH em um ciclo líquido por 45 min a 15 psi. Deixe a solução arrefecer e armazenar à temperatura ambiente.
- Inocular uma única colônia de bactérias em 100 mL de LB em um frasco de Erlenmeyer de 500 mL. Cultivar as bactérias durante a noite em uma incubadora de agitação a 37 °C.
- Dependendo da saúde da colônia bacteriana, do tamanho do frasco e da velocidade com que o agitador está configurado, o tempo que leva para a bactéria crescer pode variar. Após ~14 h, verifique a densidade óptica (OD) das bactérias a 600 nm (OD600).
- Remova as bactérias do agitador quando o OD600 for 3,0 (1 x 109 unidades formadoras de colônias (UFC)/mL). Se o OD600 for inferior a 3,0, devolva o balão à incubadora do agitador até que o OD desejado seja atingido.
- Aliquot as bactérias em tubos cônicos de 50 mL e armazenar a 4 °C ou prosseguir para a próxima etapa.
2. Trabalhando com paraformaldeído
NOTA: A concentração de paraformaldeído (PFA) utilizada e a duração da exposição podem variar um pouco dependendo do clima, localização e tipo de bactéria a ser tratada. Um bom ponto de partida para OP50 é a exposição a PFA 0,5% por 1 h, enquanto PFA 0,25% por 1 h pode ser suficiente para HT115.
- Prepare 32% de estoque de PFA ou use solução de PFA de 32% comprada comercialmente. Use equipamentos de proteção individual (EPIs) adequados ao trabalhar com PFA. Use luvas e proteção ocular.
- Adicione PFA dentro de um exaustor de fumaça química com ventilação adequada. Descarte as lavagens e pontas de PFA contendo em recipientes apropriados para riscos químicos no exaustor.
3. Tratamento bacteriano com paraformaldeído
- Quando as bactérias atingirem um OD600 de 3,0, utilizar uma pipeta sorológica para transferir 50 mL para um novo frasco de Erlenmeyer de 250 mL. Guarde o resto para um controle ao vivo, um controle simulado (consulte a etapa 4) ou para tratar também com PFA, conforme necessário.
- Evite derramar de um frasco para outro e tenha cuidado para não salpicar as laterais do novo frasco com bactérias. As colónias do lado do frasco podem receber uma dose mais baixa de PFA.
- Na capa química, adicionar 781 μL de PFA 32% a 50 mL de bactérias para elevar a concentração final a 0,5%. Descarte a ponta utilizada em recipiente para resíduos sólidos de risco químico.
- Cobrir o balão com papel alumínio e regressar à incubadora de agitação a 37 °C durante 1 h. Após 1 h, retirar o balão da incubadora e prosseguir para o passo 5.
4. Controle simulado
- Quando as bactérias atingirem um OD600 de 3,0, use uma pipeta sorológica para transferir 50 mL da bactéria para um tubo cônico de 50 mL.
- Prossiga para o passo 5.3 para concluir os passos de lavagem semelhantes ao grupo tratado com PFA.
5. Lavar as bactérias para remover PFA residual
- Na capa química, utilizar uma pipeta sorológica para transferir as bactérias tratadas do frasco de Erlenmeyer para um tubo cônico de 50 mL. O uso de uma pipeta sorológica em vez de derramar as bactérias evitará a contaminação de quaisquer colônias bacterianas na borda do frasco que possam ter evitado o tratamento direto com PFA.
- Centrifuga tratou bactérias a aproximadamente 3000 x g por 20 min. Remova o sobrenadante descartando-o em um recipiente de risco químico de resíduos líquidos na coifa química.
- Adicione 25 mL de LB e vórtice para ressuspender o pellet bacteriano (Encher o tubo totalmente dificulta a ressuspensão do pellet). Repita a centrifugação e a ressuspensão do pellet 4x.
- Ressuspenda o pellet em volumes ideais para diferentes ensaios. Para os ensaios de vida útil, sementes de placas de 60 mm com 200 μL de bactérias ressuspendidas em 10 mL de LB. A ressuspensão das bactérias em 10 mL de LB resulta em uma concentração de 5x da cultura original de 50 mL.
- Conservar as bactérias a 4 °C.
6. Verificação da qualidade do crescimento bacteriano
- Após a lavagem final e ressuspensão, passe uma placa LB (usando uma ponta de pipeta estéril) com as bactérias preparadas. É uma boa prática riscar o LB usado para lavar e ressuspender em uma placa separada, bem como certificar-se de que o LB usado não foi contaminado.
- Coloque as placas em uma incubadora de 37 °C durante a noite. Verifique se há algum crescimento. As bactérias são consideradas replicativamente mortas quando as colônias não crescem na placa LB.
7. Verificação de qualidade do metabolismo bacteriano usando um respirômetro
- Após a lavagem final e ressuspensão do passo 5.4, confirmar se as bactérias estão metabolicamente mortas usando ferramentas disponíveis, como respirômetros19,20 e medindo a taxa basal de consumo de oxigênio (OCR).
- Preparar solução M9: Dissolver 3 g de fosfato de potássio monobásico (KH 2 PO 4), 6 g de fosfato de sódio dibásico (Na2HPO4) e 5 g de cloreto de sódio (NaCl) em 950 ml de água destilada. Autoclave em ciclo líquido por 45 min a 15 psi e, em seguida, deixe a solução esfriar até a temperatura ambiente. Adicionar 1 mL de sulfato de magnésio 1 M (MgSO4) e armazenar à temperatura ambiente.
- Hidratar o cartucho do respirômetro: Adicione 200 μL de calibrador a todos os poços de uma placa de 96 poços. Coloque o cartucho na placa de 96 poços e incube durante a noite em uma incubadora de 37 °C.
- Calibração do ensaio: No dia seguinte, coloque o cartucho hidratado na máquina e inicie a calibração.
- Configuração da placa de teste de ensaio: Usando uma nova placa de 96 poços, adicione 160 μL de M9 e 40 μL das bactérias preparadas (1 x 109 UFC/mL) para testar poços. Adicione 200 μL de M9 aos 4 poços de canto para usar como poços em branco. Adicionar 160 μL de M9 e 40 μL de LB utilizados para lavagem e ressuspensão para utilização como controlos negativos. Adicionar 200 μL de M9 ao restante dos poços que não serão utilizados.
- Execute o ensaio: Quando a calibração do cartucho estiver concluída, insira a placa de ensaio da etapa 7.5 na máquina para análise. As configurações incluem etapas para misturar, esperar, medir e fazer loop. Os resultados serão apresentados como taxa de consumo de oxigênio (OCR). As bactérias estão metabolicamente mortas e prontas para uso quando o OCR é zero.
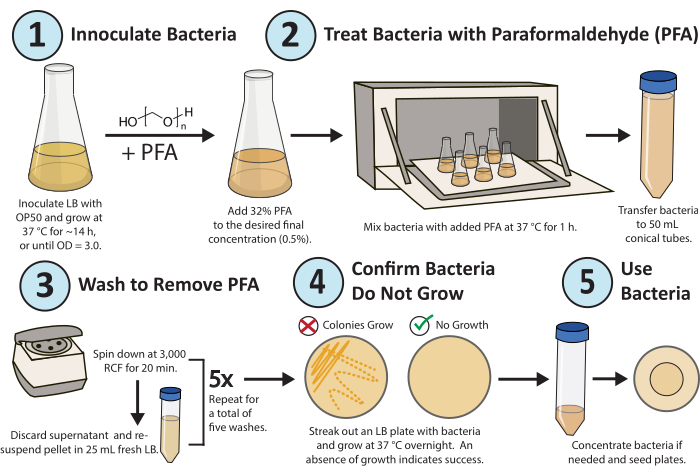
Gráfico 1. Fluxo de trabalho para tratamento com paraformaldeído. Uma única colônia de bactérias E. coli OP50 é cultivada durante a noite. PFA é adicionado a uma concentração final de 0,5%, e a cultura tratada com PFA é agitada por 1 h a 37 °C. Finalmente, o PFA é removido lavando a cultura com LB fresco 5x. Para confirmar que as bactérias tratadas são replicativamente inativas, retire uma placa LB das bactérias tratadas e cresça durante a noite. Clique aqui para ver uma versão maior desta figura.
Resultados
Um fluxo de trabalho detalhado do protocolo é mostrado na Figura 1. Um método de alto rendimento foi desenvolvido e otimizado para inativar consistentemente a replicação bacteriana (Figura 2A) e o metabolismo (Figura 2B) para estudos metabólicos e de drogas em pesquisas de C. elegans usando paraformaldeído16. O objetivo era determinar a menor concentração de PFA necessária e o menor tempo ne...
Discussão
Benefícios da morte por PFA em relação a outros métodos de eliminação bacteriana
O tratamento com PFA é um método de alto rendimento para prevenir o metabolismo bacteriano, mantendo uma fonte de alimento nutritivo para C. elegans. Matar bactérias através do tratamento com PFA tem múltiplas vantagens sobre outros métodos. Ao contrário do tratamento UV, onde cada placa deve ser testada para matar com sucesso, uma única placa de um lote de bactérias tratadas com PFA pode ser testa...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi financiado pelo NIH R21AG059117 e pelo Paul F. Glenn Laboratories for Biology of Aging Research da Universidade de Michigan. O SB foi financiado pela T32AG000114. O ESK foi financiado pela NSF DGE 1841052.
Materiais
| Name | Company | Catalog Number | Comments |
| Aluminum Foil | Staples | 2549291 | |
| Bunsen burner | VWR | 470121-700 | |
| Cell Density Meter | Denville | 80-3000-45 | |
| Centrifuge | Eppendorg | 5430 | |
| Chemical fume hood | Labcono | 975050411384RG | |
| Conincal tubes (50 mL) | Fisher | 339652 | |
| Cuvettes | Fisher | 14-955-127 | |
| E. coli OP50 | CGC | OP50 | |
| Erlenmyer flasks | Fisher | 250 mL: FB501250 500 mL: FB501500 1000 mL: FB5011000 | |
| Inoculation loop | Fisher | 22-363-605 | |
| LB Agar | Fisher | BP1425500 | |
| Liquid waste collection bottle | Thomas Scientific | 1230G50 | |
| Magnesium Sulfate (MgSO4) | Sigma | M7506 | |
| Paraformaldehyde (32%) | Electron Microscopy Sciences | 15714-S | Paraformaldehyde – methanol free solution |
| Pipettor | Eppendorf | Eppendorf Easypet 3 | |
| Plastic dishes (100 mm) | Fisher | FB0875712 | |
| Potassium Phosphate Monobasic (KH2PO4) | Fisher | P2853 | |
| Seahorse XF Calibrant | Agilent | 100840-000 | |
| Seahorse XFe96 Extracellular Flux Assay Kit and Cell Culture Microplate | Agilent | 101085-004 | |
| Serological pipettes (50 mL) | Genesee Scientific | 12-107 | |
| Shaker incubator | Thermo | 11 676 083 | |
| Sodium Chloride (NaCl) | Fisher | S640-3 | |
| Sodium Hydroxide (NaOH) | Fisher | S318500 | |
| Sodium Phosphate Dibasic Anhydrous (Na2HPO4) | Sigma | S374-500 | |
| Solid waste collection bucket | M&M Industries | 5.0 Gallon M1 Traditional Pail | |
| Tryptone | Genesee Scientific | 20-251 | |
| Vortex | Thermo | 11676331 | |
| Weighing balance | C Goldenwall | HZ10K6B | |
| Yeast Extract | Genesee Scientific | 20-255 |
Referências
- Riddle, D. L., Blumenthal, T., Meyer, B. J., Priess, J. R. C. . Elegans II. 33, (1997).
- Corsi, A. K., Wightman, B., Chalfie, M. A transparent window into biology: A primer on caenorhabditis elegans. WormBook. , 1-31 (2015).
- Kaletta, T., Hengartner, M. O. Finding function in novel targets: C. elegans as a model organism. Nature reviews. Drug discovery. 5 (5), 387-398 (2006).
- Meneely, P. M., Dahlberg, C. L., Rose, J. K. Working with worms: Caenorhabditis elegans as a model organism. Current Protocols Essential Laboratory Techniques. 19 (1), (2019).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Frontiers in Endocrinology. 11, 554994 (2020).
- Feng, M., Gao, B., Garcia, L. R., Sun, Q. Microbiota-derived metabolites in regulating the development and physiology of Caenorhabditis elegans. Frontiers in Microbiology. 14, 1035582 (2023).
- McIntyre, G., Wright, J., Wong, H. T., Lamendella, R., Chan, J. Effects of FUdR on gene expression in the C. elegans bacterial diet OP50. BMC Research Notes. 14 (1), 207 (2021).
- Cabreiro, F., et al. Metformin retards aging in c. Elegans by altering microbial folate and methionine metabolism. Cell. 153 (1), 228-239 (2013).
- Worthy, S. E., et al. Identification of attractive odorants released by preferred bacterial food found in the natural habitats of c. Elegans. PLoS One. 13 (7), e0201158 (2018).
- Chen, Y. C., Seyedsayamdost, M. R., Ringstad, N. A microbial metabolite synergizes with endogenous serotonin to trigger C. elegans reproductive behavior. Proceedings of the National Academy of Sciences of the United States of America. 117 (48), 30589-30598 (2020).
- Kim, D. H., Flavell, S. W. Host-microbe interactions and the behavior of Caenorhabditis elegans. Journal of Neurogenetics. 34 (3-4), 500-509 (2020).
- Gems, D., Riddle, D. L. Genetic, behavioral, and environmental determinants of male longevity in Caenorhabditis elegans. Genetics. 154 (4), 1597-1610 (2000).
- Qi, B., Kniazeva, M., Han, M. A vitamin-b2-sensing mechanism that regulates gut protease activity to impact animal's food behavior and growth. eLife. 6, e26243 (2017).
- Garigan, D., et al. Genetic analysis of tissue aging in caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Virk, B., et al. Folate acts in E. coli to accelerate C. elegans aging independently of bacterial biosynthesis. Cell Reports. 14 (7), 1611-1620 (2016).
- Beydoun, S., et al. An alternative food source for metabolism and longevity studies in Caenorhabditis elegans. Communications Biology. 4 (1), 258 (2021).
- Thavarajah, R., Mudimbaimannar, V. K., Elizabeth, J., Rao, U. K., Ranganathan, K. Chemical and physical basics of routine formaldehyde fixation. Journal of Oral and Maxillofacial Pathology. 16 (3), 400-405 (2012).
- Felix, H. Permeabilized and immobilized cells. Methods in Enzymology. 137, 637-641 (1988).
- Lobritz, M. A., et al. Antibiotic efficacy is linked to bacterial cellular respiration. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8173-8180 (2015).
- Nadanaciva, S., et al. Assessment of drug-induced mitochondrial dysfunction via altered cellular respiration and acidification measured in a 96-well platform. Journal of Bioenergetics and Biomembranes. 44 (4), 421-437 (2012).
- Shtonda, B. B., Avery, L. Dietary choice behavior in Caenorhabditis elegans. The Journal of Experimental biology. 209 (Pt 1), 89-102 (2006).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. Diet-induced developmental acceleration independent of tor and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- Kumar, S., et al. Lifespan extension in C. elegans caused by bacterial colonization of the intestine and subsequent activation of an innate immune response. Developmental Cell. 49 (1), 100-117 (2019).
- Nakagawa, H., et al. Effects and mechanisms of prolongevity induced by Lactobacillus gasseri sbt2055 in Caenorhabditis elegans. Aging Cell. 15 (2), 227-236 (2016).
- Kaeberlein, T. L., et al. Lifespan extension in Caenorhabditis elegans by complete removal of food. Aging Cell. 5 (6), 487-494 (2006).
- Beaudoin-Chabot, C., et al. The unfolded protein response reverses the effects of glucose on lifespan in chemically-sterilized C. elegans. Nature Communication. 13 (1), 5889 (2022).
- Komura, T., Takemoto, A., Kosaka, H., Suzuki, T., Nishikawa, Y. Prolonged lifespan, improved perception, and enhanced host defense of Caenorhabditis elegans by Lactococcus cremoris subsp. cremoris.Microbiology Spectrum. 10 (3), e0045421 (2022).
- Ye, X., Linton, J. M., Schork, N. J., Buck, L. B., Petrascheck, M. A pharmacological network for lifespan extension in Caenorhabditis elegans. Aging Cell. 13 (2), 206-215 (2014).
- Hastings, J., et al. Wormjam: A consensus C. elegans metabolic reconstruction and metabolomics community and workshop series. Worm. 6 (2), e1373939 (2017).
- O'Donnell, M. P., Fox, B. W., Chao, P. H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Stuhr, N. L., Curran, S. P. Bacterial diets differentially alter lifespan and healthspan trajectories in C. elegans. Communications Biology. 3 (1), 653 (2020).
- Dirksen, P., et al. Cembio - the Caenorhabditis elegans microbiome resource. G3 (Bethesda). 10 (9), 3025-3039 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados