Method Article
Mastócitos no microambiente do carcinoma hepatocelular conferem prognóstico favorável: um estudo retrospectivo usando o software de análise de imagem QuPath
Neste Artigo
Resumo
A presença de mastócitos na margem interna e áreas peritumorais do carcinoma hepatocelular após a ressecção confere um prognóstico favorável. Este estudo endossa o software de análise de imagens QuPath como uma plataforma promissora que pode atender à necessidade de reprodutibilidade, consistência e precisão em patologia digital.
Resumo
Os insights fornecidos pela detecção in situ de células imunes no carcinoma hepatocelular (CHC) podem apresentar informações sobre os resultados dos pacientes. Estudos que investigam a expressão e localização de células imunes dentro de tecidos tumorais estão associados a vários desafios, incluindo a falta de anotação precisa para regiões tumorais e seleção aleatória de campos de visão microscópicos. O QuPath é um software de código aberto e fácil de usar que pode atender à crescente necessidade de patologia digital na análise de imagens de lâmina inteira (WSI).
A infiltração de CHC e tecidos adjacentes por células dendríticas imaturas CD1a+ (iDCs), mastócitos CD117+ e células natural killer (NKs) NKp46+ foi avaliada imunohistoquimicamente em espécimes representativos de 67 pacientes com CHC submetidos à ressecção curativa. A fração de área (FA) das células coradas positivamente foi avaliada automaticamente em WSIs usando QuPath no centro do tumor (TC), margem interna (IM), margem externa (OM) e área peritumoral (PT). A significância prognóstica das células imunes foi avaliada quanto ao tempo de recorrência (TTR), sobrevida livre de doença (DFS) e sobrevida global (OS).
A FA dos mastócitos foi significativamente maior do que a FA das NKs, e a FA das iDCs foi significativamente menor em comparação com as NKs em cada região de interesse. FAs altas de mastócitos nas áreas IM e PT foram associadas a DFS mais longo. Além disso, a alta FA de mastócitos na MI foi associada a uma SG mais longa.
A análise assistida por computador usando este software é uma ferramenta adequada para obter informações prognósticas para células imunes infiltrantes de tumor (iDCs, mastócitos e NKs) em diferentes regiões do CHC após a ressecção. Os mastócitos exibiram a maior FA em todas as regiões de interesse (ROIs). Os mastócitos na região peritumoral e IM mostraram significância prognóstica positiva.
Introdução
A organização espacial e a abundância de células imunes infiltrantes de tumor comprovadamente afetam a sobrevida em diferentes tipos de câncer, incluindo o carcinoma hepatocelular (CHC) 1,2,3,4. O significado prognóstico dos linfócitos infiltrantes tumorais em cânceres foi demonstrado pela primeira vez em cortes corados com hematoxilina e eosina (H&E) 5,6. Em seguida, um estudo pioneiro de Galon et al. usando imuno-histoquímica (IHQ) demonstrou associações das densidades de células T CD3+ e CD8+ no tecido de câncer de cólon com prognóstico4.
A IHQ é um padrão-ouro para visualizar, quantificar e mapear células imunes dentro do tecido tumoral para posterior associação com resultados clínicos7. A IHQ apresenta várias vantagens, como baixo custo, ampla disponibilidade e compatibilidade com tecido embebido em parafina fixado em formalina (FFPE)8. No entanto, a avaliação precisa das células imunes coradas com IHC é um grande desafio. A pontuação tradicional em campos de visão microscópicos selecionados é demorada e não é mais suficiente para garantir a análise objetiva, reprodutível e de alta qualidade, essencial para a seleção de biomarcadores candidatos e correlação clínica confiável9. As varreduras de lâminas inteiras podem ser avaliadas como uma imagem inteira ou após subamostragem10.
A avaliação quantitativa computadorizada da abundância de células imunes em amostras de tecido pode garantir dados práticos, precisos, confiáveis e clinicamente relevantes11. O QuPath é um software gratuito e de código aberto que permite a análise de imagem digital de lâminas coradas com IHC e maximiza a quantidade de informações obtidas a partir de amostras individuais12.
Uma vez que as células dendríticas imaturas (iDCs), as células natural killer (NK) e os mastócitos estão envolvidos nas respostas imunes antitumorais e demonstraram se correlacionar com os resultados dos pacientes 13,14,15, apresentamos aqui um protocolo passo a passo para avaliar sua distribuição espacial no tecido FFPE CHC e explorar seu impacto prognóstico. O CD1a é expresso principalmente na membrana de células dendríticas imaturas, cuja densidade tem sido associada a desfechos clínicos em uma variedade de tumores humanos 16,17. Os mastócitos podem desempenhar um papel pró-tumoral ou antitumoral dentro do microambiente tumoral (TME)18. Eles podem apoiar a angiogênese e facilitar a metástase19. Por outro lado, foi relatado que os mastócitos podem mediar a apoptose das células tumorais por meio da produção de IL-420. A coloração anti-CD117 é comumente usada para visualizar e quantificar mastócitos dentro do tecido tumoral21. Acredita-se que as células NK contribuam para a vigilância e controle do CHC22, matando as células cancerígenas23. A expressão de NKp46 por NK é um parâmetro crucial para sua atividade antitumoral24. No entanto, as células NKp46+ NK correlacionaram-se positivamente com o diâmetro do tumor em pacientes com CHC25. Apesar disso, pouco se sabe sobre sua correlação com a sobrevida dos pacientes.
Nosso objetivo foi avaliar quantitativamente a abundância de CD1a+ iDCs, mastócitos CD117+ e células NKp46+ NK em diferentes regiões do CHC e destacar sua significância prognóstica. Um total de 70 pacientes consecutivos com CHC estágio I-IV confirmado patologicamente, que eram elegíveis para ressecção de acordo com as diretrizes do BCLC e foram submetidos à ressecção hepática com intenção curativa no Hospital Universitário de Pilsen entre 1997 e 2019, foram incluídos no estudo retrospectivo atual. Os relatórios anatomopatológicos dos pacientes foram revisados. Nenhum dos pacientes incluídos neste estudo apresentou metástases à distância nem recebeu terapia neoadjuvante, como quimioterapia ou radioterapia, antes da cirurgia. Um total de 3 pacientes com peças histológicas de baixa qualidade foram excluídos, e os 67 pacientes restantes foram incluídos no estudo (Tabela 1).
Protocolo
Este protocolo fez parte de um estudo realizado de acordo com os padrões éticos estabelecidos na Declaração de Helsinque (versão de 2013); foi aprovado pelo Comitê de Ética da Faculdade de Medicina e do Hospital Universitário de Pilsen (118/2021, 11 de março de 2021). A Figura 1 mostra um resumo dos métodos empregados.
1. Seleção de blocos de tecido FFPE
- Recupere identificadores de bloco FFPE de arquivos de pacientes com a ajuda de médicos ou patologistas.
- Solicite os blocos FFPE do arquivo de patologia local.
- Para cada paciente, selecione 2-3 blocos FFPE contendo tecido tumoral viável, preferencialmente com a área peritumoral circundante e fígado não tumoral.
NOTA: É aconselhável obter a opinião de um patologista especialista para esta seleção. A avaliação histológica do fígado não tumoral, juntamente com dados anamnésicos, clínicos e laboratoriais, foi usada para definir a etiologia subjacente do CHC.
2. Desparafinização e reidratação do tecido FFPE
- Corte uma ou duas seções de tecido de 4 μm de espessura de cada um dos blocos de tecido FFPE em um micrótomo.
NOTA: A espessura das seções pode estar entre 3 μm e 5 μm sem impacto perceptível de manchas; no entanto, uma espessura de 4 μm é o padrão. - Coloque as mechas em banho-maria (42°C) para remover as rugas.
- Monte as lâminas pegando-as e colocando-as em uma lâmina microscópica de vidro carregada positivamente. Em seguida, aplique as lâminas para secagem à temperatura ambiente (TA) por 24 h.
- Coloque as lâminas em um termostato (56 °C) por 1 h.
- Use um corador automático para desparafinizar as seções em solução de desparafinagem-1 (30 s a 72 °C), solução de desparafinagem-2 (10 s a 72 °C), solução de desparafinagem-3 (10 s em AT), etanol 96%-1 (10 s), etanol 96%-2 (10 s) e etanol 96%-3 (10 s).
- Use um corante automático para reidratar as seções na solução de lavagem-1 (10 s, AT), solução de lavagem-2 (10 s, AT) e solução de lavagem-3 (5 min, AT).
3. Realização de IHQ em um corante totalmente automatizado
NOTA: A ICC é realizada de acordo com o protocolo do fabricante. As etapas são explicadas brevemente abaixo.
- Método de recuperação de antígeno
- Recupere o antígeno usando o procedimento de recuperação de epítopos induzidos por calor (ER) em uma solução de ER com pH alto (por exemplo, Tris-EDTA): solução de ER-1 (10 s, AT), solução de ER-2 (10 s, AT), solução de ER-3 (20 min a 100 °C), solução de ER-4 (12 min, AT).
- Enxágue a lâmina em uma solução de lavagem por 3x (30 s cada) e 1x (3 min).
- Bloqueie a peroxidase endógena usando a solução de bloco de peróxido por 5 min em AT.
- Enxágue em uma solução de lavagem por 3x (30 s cada).
- Bloqueie a ligação inespecífica de anticorpos com um bloqueio de proteína para NKp46 apenas por 30 min.
- Ligação com anticorpos primários: Incubar com anticorpos primários por 15 min em AT, depois enxaguar com uma solução de lavagem por 3x (30 s cada).
- Para ligação secundária de anticorpos, siga as etapas descritas abaixo.
- Incubar com anticorpos secundários conjugados com peroxidase de rábano por 8 min em AT.
- Enxágüe com uma solução de lavagem por 2x (2 min cada) e 1x com água deionizada.
- Visualize a reação enxaguando as lâminas com solução de diaminobenzidina (DAB) e, em seguida, incubando-as com a solução DAB por 10 min no AT.
- Enxágue por 3x (30 s) com água deionizada.
- Contra-core as seções com hematoxilina de Mayer.
- Enxágue com água deionizada por 1x (30 s), solução de lavagem por 1x (30 s) e água deionizada por 1x (30 s).
- Use um corador de lâmina totalmente automatizado para desidratar as seções em etanol 70% (2 min), etanol 80% (2 min), etanol 96%-1 (3 min), etanol 96%-2 (3 min), etanol 100% (3 min), xileno-1 (4 min), xileno-2 (4 min) e xileno-3 (4 min).
- Use uma lamínula de vidro totalmente automatizada para montar as seções nas corrediças em um meio de montagem.
NOTA: Amostras de controle de tecido negativas e positivas (amígdalas) foram usadas por toda parte.
4. Obtenção de varreduras de lâminas inteiras
- Digitalize as lâminas coradas usando a objetiva de 20x e densidade moderada-alta de foco em um scanner de lâmina inteira.
5. Análise IHC usando o software
- Crie um projeto para as imagens IHC no software.
- Baixe e abra o QuPath-0.4.3 (ou superior).exe
- Crie uma nova pasta com um nome adequado.
- Clique no botão Criar projeto no canto superior esquerdo do software e selecione a pasta recém-criada.
- Arraste as imagens digitalizadas para a janela do software. Quando uma nova janela aparecer automaticamente, selecione Definir tipo de imagem > H-DAB e clique em Importar (Figura 2).
- Observe a lista de imagens na janela esquerda do menu; Clique duas vezes para abrir a imagem.
- Selecione no menu principal na parte superior Arquivo para salvar as imagens como um projeto.
NOTA: O software pode lidar com uma ampla variedade de formatos de imagem, incluindo muitos formatos de slides inteiros. No entanto, o formato tiff é preferido para evitar a perda de dados brutos e metadados associados.
- Anotar regiões de interesse
- Selecione a ferramenta Pincel ou a ferramenta Varinha no menu principal na parte superior para anotar a área do tumor. Para regiões descontínuas do tumor, siga as etapas 5.2.1.1-5.2.1.3.
- Anote-os separadamente.
- Pressione e segure a tecla Ctrl enquanto seleciona todas as regiões do tumor
- Clique com o botão direito do mouse e selecione Editar vários > Mesclar selecionados.
- Certifique-se de que a anotação do tumor esteja selecionada (as bordas têm uma cor amarela).
- Clique em Automatizar no menu principal e selecione Mostrar Editor de Scripts.
- Copie o script disponível no Arquivo Suplementar 1 ou em https://petebankhead.github.io/qupath/scripts/2018/08/08/three-regions.htmls e cole-o no Editor de Scripts e clique em Executar. As ROIs: centro tumoral (TC), margem interna (IM), margem externa (OM) e área peritumoral (PT) serão criadas e rotuladas automaticamente.
- Para obter uma maneira alternativa de criar regiões de interesse, siga as etapas 5.2.5.1-5.2.5.5.
- Selecione a ferramenta Polilinha no menu principal na parte superior para desenhar uma borda separando os ninhos de células malignas e o tecido não tumoral adjacente.
- Selecione a borda resultante. Selecione no menu principal na parte superior Objetos > anotação > Expandir anotações > 500 μm para Raio de expansão > Plana para Limite de linha. Ative Remover interior e ative Restringir ao pai.
- Clique com o botão direito do mouse na anotação resultante, Editar Divisão Única >. Duas anotações separadas serão estendidas automaticamente como regiões de 500 μm de largura em cada lado da borda.
- Nomeie as regiões anotadas adequadamente para IM e OM clicando com o botão direito do mouse na anotação na lista à esquerda, selecionando Definir Propriedades e inserindo o nome.
- O TC representa a área tumoral remanescente. Estender a área de 500 μm de TP adjacente ao OM utilizando o mesmo procedimento.
NOTA: As lâminas anotadas e as detecções de FA QuPath foram revisadas por um histopatologista especialista.
- Selecione a ferramenta Pincel ou a ferramenta Varinha no menu principal na parte superior para anotar a área do tumor. Para regiões descontínuas do tumor, siga as etapas 5.2.1.1-5.2.1.3.
- Otimização da classificação de pixels
- Selecione a ferramenta Retângulo no menu principal. Em seguida, anote uma região, que contém todos os tipos de pixels a serem distinguidos (por exemplo, hematoxilina, DAB e plano de fundo).
- No menu principal, selecione Analisar > pré-processamento > Estimar vetor de mancha.
- Quando a janela Editor de Manchas Visuais aparecer automaticamente, selecione Auto > OK e defina H-DAB estimado como o nome do vetor de manchas.
- Para um método alternativo, siga as etapas 5.3.4.1-5.3.4.3.
- Selecione a ferramenta Retângulo no menu principal e anote uma pequena região de um núcleo típico corado com hematoxilina.
- Selecione Imagem no menu à esquerda e clique duas vezes em Mancha 1 > Sim.
- Selecione a ferramenta Retângulo no menu principal e anote uma pequena região de coloração positiva com DAB. Selecione Imagem no menu à esquerda e clique duas vezes em Mancha 2 > Sim.
- Avaliação da fração de área de células imunes positivas usando a opção Classificação de Pixels .
- No menu principal, clique em Classificar > classificação de pixels > Criar limiar.
- Selecione Alta para a resolução > canal: DAB > Pré-filtro: Gaussiano > sigma: 0,5 > (Figura 3) Limiar: (0,2-0,3) > Acima do limite: Positivo > Abaixo do limiar: Negativo.
- Para a região, selecione Qualquer anotação, digite um nome para o classificador > Salvar > medida > OK. As células positivas para DAB serão alteradas para a cor vermelha (Figura 4).
- Copie os resultados do menu do lado esquerdo para uma planilha.
- Ou abra Medir no menu principal superior, selecione Mostrar medidas de anotação > Copiar para a área de transferência e cole os resultados em uma planilha.
- Para fechar a imagem, clique com o botão direito do mouse > Multi View > Close Viewer.
NOTA: Antes da quantificação, todos os artefatos eram eliminados. O limiar foi ajustado para cada caso com uma coloração positiva específica fraca. A exatidão das anotações e a classificação de pixels foram primeiro verificadas e depois revisadas por um histopatologista sênior. Todas as análises foram feitas de forma cega. Mais informações sobre a análise da IHQ usando o software podem ser encontradas em https://forum.image.sc/tag/QP.
6. Métodos estatísticos e análise de dados
- Os desfechos do estudo foram tempo de recorrência (TTR), sobrevida livre de doença (DFS) e sobrevida global (OS). Os pacientes sem recidiva ou óbito foram censurados no último acompanhamento.
NOTA: Os dados contínuos com distribuição não normal são expressos como mediana (min-max). As proporções são expressas como dados brutos (porcentagens). As frações de área das células imunes em diferentes regiões de interesse (ROIs) foram comparadas pela ANOVA de Friedman, seguida pelo teste de pares pareados de Wilcoxon com correção de Bonferroni. Devido à distribuição não paramétrica da maioria das variáveis, a correlação de Spearman foi utilizada para avaliar as associações entre pares de variáveis ordinais ou quantitativas. Para determinar o valor prognóstico dos preditores individuais para os desfechos, foi realizada uma análise de regressão de Cox univariada seguida de multivariada. Foram calculadas as taxas de risco (HR). HR estava mostrando o risco relativo para o grupo intermediário alto combinado em comparação com 1 para o grupo baixo. Todas as estimativas de sobrevida foram calculadas pelo método de Kaplan-Meier e comparadas entre os grupos pelo teste de log-rank. O GraphPad Prism 9.0 (GraphPad Software LLC) foi utilizado para as análises estatísticas. Um valor de p bilateral < 0,05 foi considerado estatisticamente significativo.
Resultados
As características demográficas e clínicas dos pacientes são apresentadas na Tabela 1. A mediana de idade dos pacientes foi de 69 anos, sendo a maioria do sexo masculino (77,6%). Em relação à etiologia do CHC, a hepatite crônica não viral foi a doença de base mais frequente, com prevalência de esteato-hepatite não alcoólica (EHNA) (23,9%). Apenas 15 pacientes (22,4%) tinham cirrose como doença de base. A maioria dos pacientes (68,7%) tinha TNM estágio I. Achados clínicos e anatomopatológicos mais detalhados foram descritos em nosso trabalho publicado anteriormente 2,26.
Resultados
No último acompanhamento, a recidiva tumoral foi observada em 29 (41,8%) pacientes, dos quais 89,7% apresentaram recidiva local e 38 (56,7%) pacientes foram a óbito. Aos 5 anos após a cirurgia, a proporção de SLD foi de 33,2%, a proporção OS foi de 49,4% e a proporção livre de recidiva (RFP) foi de 48,7% (Tabela 2).
Distribuição de células imunes em diferentes regiões de interesse
A proteína CD117 foi encontrada predominantemente na membrana citoplasmática de células arredondadas (Figura 5A). No CT e IM, eles estavam localizados principalmente no estroma e no espaço perivascular. Nas áreas de MO e TP, as células CD117+ foram observadas na cápsula tumoral e no espaço perivascular.
A proteína NKp46 foi encontrada principalmente na membrana citoplasmática de células arredondadas, que estavam presentes dentro de espaços semelhantes sinusóides no CT e IM, além de estarem associadas ao estroma (Figura 5B). Na área de MO e TP, células NKp46+ foram observadas em cápsulas ao redor de ninhos tumorais e no estroma dos tratos portais.
A proteína CD1a foi encontrada no CT e IM, principalmente na membrana citoplasmática de células arredondadas (espalhadas ou em agregados) no estroma, dentro e ao longo dos limites dos espaços sinusóides (Figura 5C). Na área de TP, CD1a+ DC foi observada dentro e ao longo dos limites dos sinusóides e dentro do epitélio biliar nas vias portais.
A FA dos mastócitos foi significativamente maior do que a FA das células NKp46+ (p < 0,001) (Figura 6). As CD1a+ iDCs em cada ROI apresentaram a menor FA em comparação com mastócitos e células NK (p < 0,001) (Figura 6). A FA das células CD117+ e NKp46+ em CT ou IM foi significativamente menor do que as da área de TP e MO (p < 0,001) (Figura 6). Para as células CD1a+, a FA não diferiu significativamente entre as regiões.
Associações significativas para células imunes individuais entre todas as ROIs são mostradas na Tabela 3. A Figura 7 mostra o mapa de calor para associações significativas entre diferentes células imunes em diferentes regiões. A FA de mastócitos CD117+ em CT correlacionou-se significativamente com a FA de CD1a no CT e IM.
Valor prognóstico das variáveis clínicas e anatomopatológicas
Entre as variáveis clínicas e anatomopatológicas, apenas a idade mais jovem foi associada a um maior risco de recorrência (HR = 0,96, IC: 0,93-0,99, p = 0,007), enquanto nenhuma variável foi associada a SLD e SG (Tabela 4).
Valores prognósticos de células imunes infiltrantes
Uma FA elevada de mastócitos CD117+ no MI foi associada a DFS mais longo (HR = 0,48, IC: 0,241 - 0,935, p = 0,031) e SG (HR = 0,34, IC: 0,167 - 0,703, p = 0,004) (Figura 8, Tabela 5). Além disso, uma FA elevada de mastócitos CD117+ na área de TP foi associada a DFS mais longo (HR = 0,48, IC: 0,247 - 0,945, p = 0,034) (Figura 8, Tabela 5). Por outro lado, as FAs das células iDC e NK não foram associadas a nenhum dos desfechos. Os mastócitos CD117+ na margem interna mantiveram associações significativas com DFS (HR = 0,46, IC: 0,23-0,91, p = 0,027) e OS (HR = 0,33, IC: 0,16-0,68, p = 0,003) após ajuste para o estágio TNM (Tabela 6). Quanto aos mastócitos na região do TP, a associação com DFS também permaneceu significativa (HR = 0,47, IC: 0,24-0,93, p = 0,029) (Tabela 6).
Figura 1: Esquema representativo para os métodos. Diagrama esquemático do fluxo de trabalho para identificação do papel prognóstico de mastócitos, células dendríticas e células natural killer no CHC. Abreviaturas: FFPE, embebido em parafina fixado em formalina; H&E, Hematoxilina e Eosina; ROIs, regiões de interesse. Clique aqui para ver uma versão maior desta figura.
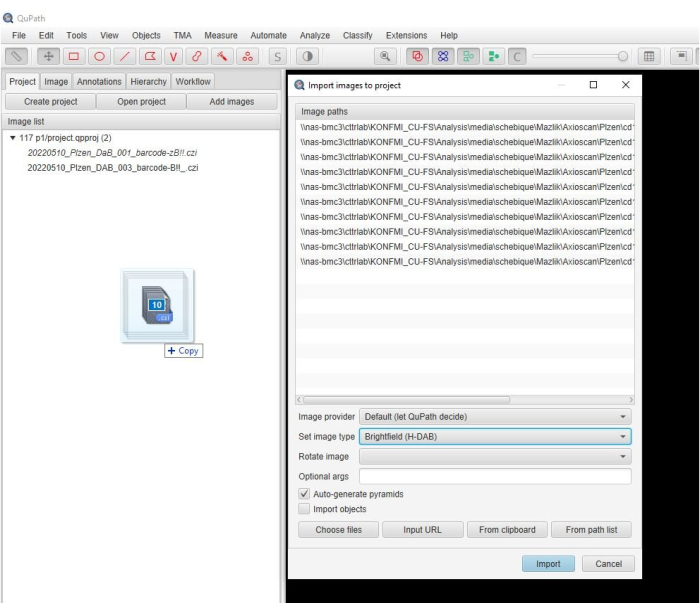
Figura 2: Captura de tela da janela do software. Uma captura de tela descritiva mostrando a etapa de importação de imagens para um projeto no software. Clique aqui para ver uma versão maior desta figura.
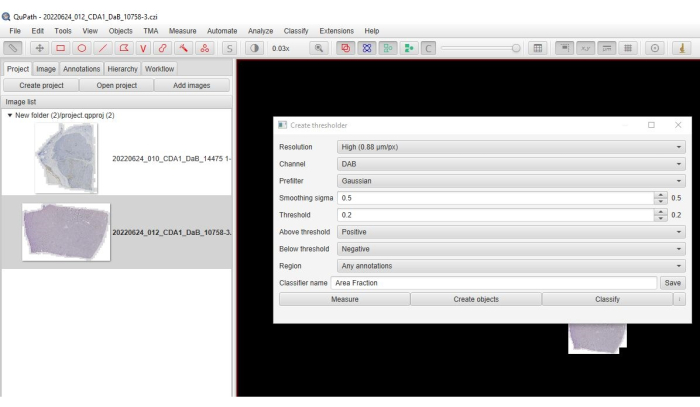
Figura 3: Captura de tela da opção de fração de área. Uma captura de tela do software mostrando os parâmetros selecionados para quantificar a fração de área da coloração positiva. Clique aqui para ver uma versão maior desta figura.
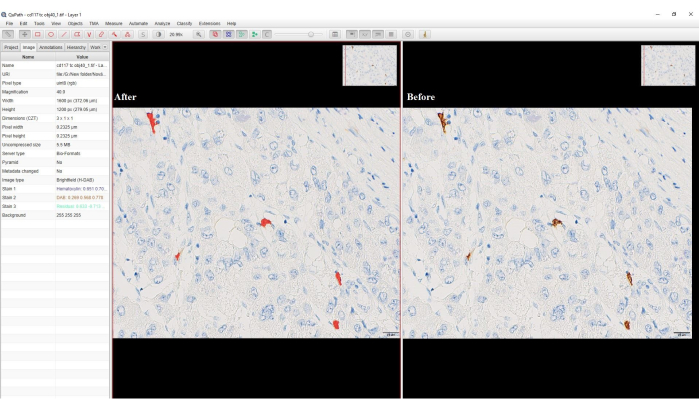
Figura 4: Captura de tela da opção de classificação de pixels. Captura de tela do software mostrando as células positivas DAB antes e depois da classificação de pixels. A coloração DAB acastanhada foi convertida para a cor vermelha após a classificação de pixels. Clique aqui para ver uma versão maior desta figura.
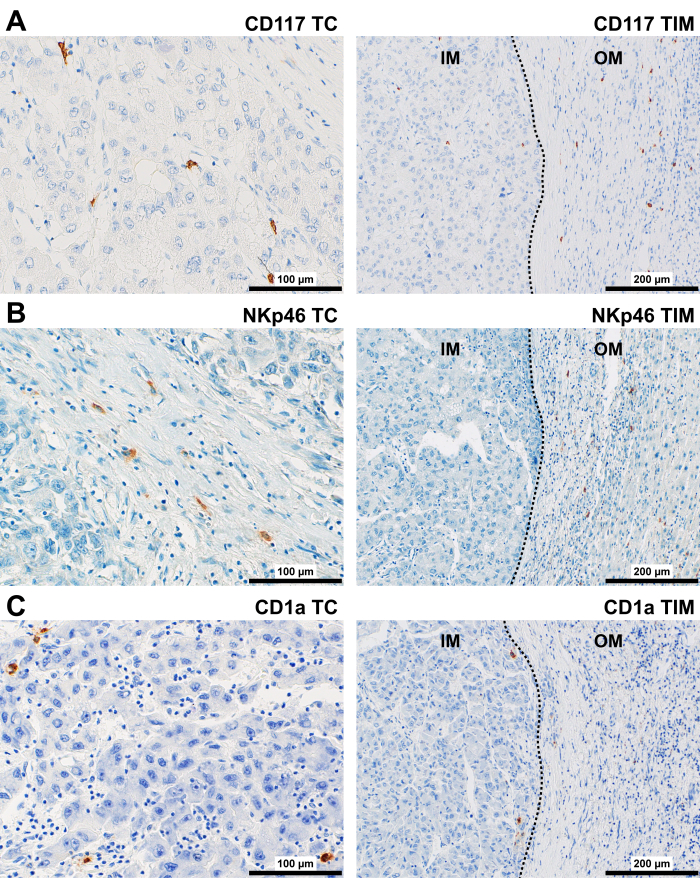
Figura 5: Imunocoloração representativa de células imunes inatas em pacientes com CHC. Imunomarcação representativa de (A) CD117+, (B) NKp46+ e (C) células CD1a+ em TC (400x) e TIM (200x) de CHC. Abreviaturas: CHC, carcinoma hepatocelular; CT, centro tumoral; TIM, margem invasiva do tumor, com margem interna (IM) e margem externa (MO). A linha pontilhada representa uma fronteira entre o tecido tumoral e não tumoral. Clique aqui para ver uma versão maior desta figura.
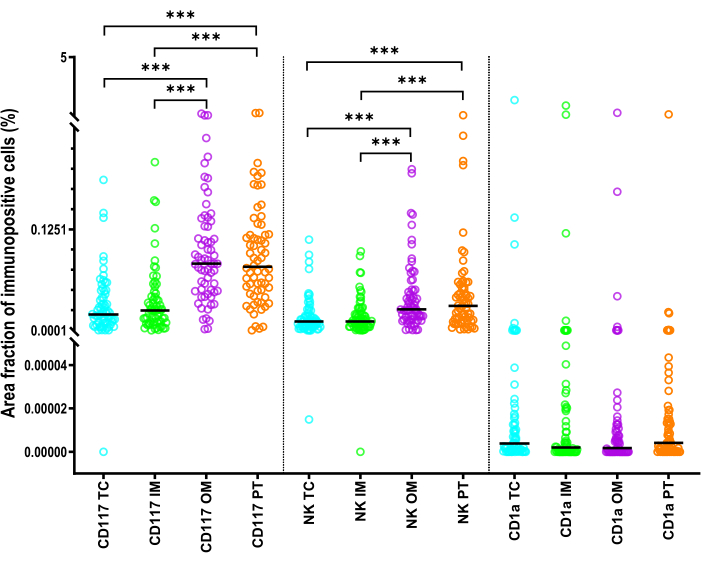
Figura 6: Estatísticas que descrevem a fração de área das células imunes inatas em pacientes com CHC. Estatísticas para a fração de área de células dendríticas CD1a +, mastócitos CD117 + e células NKp46 + NK) nas áreas TC, IM, OM e PT do CHC. As linhas pretas são medianas. O teste de pares pareados de Wilcoxon com correção de Bonferroni foi utilizado para comparações. Abreviaturas: CHC, carcinoma hepatocelular; CT, centro tumoral; como primário, margem interna; OM, margem externa; TP, área peritumoral. : p <0,001 Clique aqui para ver uma versão maior desta figura.
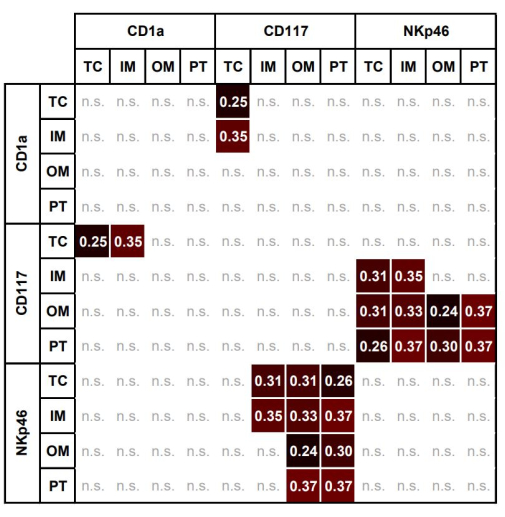
Figura 7: Mapa de calor de correlações significativas entre frações de área de mastócitos, DCs e NKs em diferentes regiões de interesse. Mapa de calor de correlações significativas entre frações de área de mastócitos CD117+, CD1a+ DCs e NKp46+NKs em TC, IM, OM e PT (Spearman ρ, p < 0,05). Abreviaturas: TC, centro tumoral; como primário, margem interna; OM, margem externa; TP, área peritumoral; DC, células dendríticas; NK, células assassinas naturais. Clique aqui para ver uma versão maior desta figura.
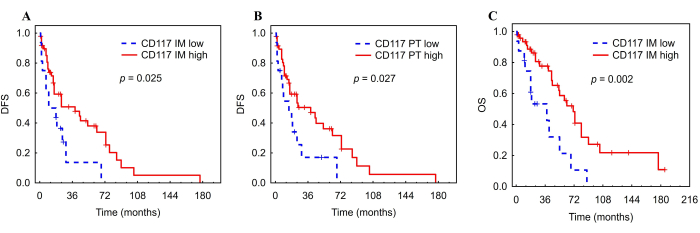
Figura 8: Curvas de Kaplan-Meier DFS e OS para mastócitos infiltrantes de tumor em pacientes com CHC. Análise de Kaplan-Meier de DFS e OS de acordo com FA baixa vs. alta de mastócitos infiltrantes de tumor na margem interna (A, C) e PT (B) do CHC. Abreviaturas: CHC, carcinoma hepatocelular; DFS, sobrevida livre de doença; como primário, invasivo interno; TP, área peritumoral. A figura foi adaptada com permissão de Ali et al.26. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Antecedentes clínicos dos pacientes com carcinoma hepatocelular incluídos. Antecedentes clínicos dos casos inscritos de carcinoma hepatocelular. Abreviaturas: DHGNA, doença hepática gordurosa não alcoólica. Clique aqui para baixar esta tabela.
Tabela 2: Probabilidade estimada de desfechos na análise de Kaplan-Meier. A probabilidade estimada de RFP, DFS e OS na análise de Kaplan-Meier. Abreviaturas: RFP, proporção livre de recorrência; DFS, sobrevida livre de doença; OS, sobrevida global. Clique aqui para baixar esta tabela.
Tabela 3: Correlação entre a fração de área de mastócitos infiltrantes de tumores, células dendríticas e células natural killer em diferentes ROIs. Correlação de Spearman (ρ) entre a fração de área de mastócitos infiltrantes de tumores, células dendríticas e células natural killer em diferentes ROIs, p < 0,038. Abreviaturas: TC, centro tumoral; como primário, margem interna; OM, margem externa; TP, área peritumoral; ROIs, regiões de interesse. Clique aqui para baixar esta tabela.
Tabela 4: Análise univariada de variáveis clínicas e patológicas associadas ao tempo de recorrência (TTR), sobrevida livre de doença (DFS) e sobrevida global (OS). " Não", sexo feminino ou "A" foram as categorias de referência para as variáveis dicotômicas. Valores em negrito indicam significância estatística no nível de p < 0,05. Abreviaturas: HR, taxa de risco; IC, intervalo de confiança; TTR, tempo até a recorrência; DFS, sobrevida livre de doença. Clique aqui para baixar esta tabela.
Tabela 5: Análise univariada da associação da fração de área de mastócitos CD117+, células CD1a+denderíticas e células NKp46+natural killer com TTR, DFS e OS por região individual de interesse usando a regressão de Cox (67 pacientes com CHC). A fração de área de células imunes por seção de área (mm2) foi convertida em percentis e, em seguida, categorizada em baixa (percentil 0-25) versus alta (percentil 25-100). As taxas de risco mostram o risco relativo em comparação com 1 para a FA baixa. Valores em negrito indicam significância estatística no nível de p < 0,05. Abreviaturas: AF, fração de área; DFS, sobrevida livre de doença; OS, sobrevida global; HR, taxa de risco. Clique aqui para baixar esta tabela.
Tabela 6: A fração de área de mastócitos CD117+ por ROI individual associada à sobrevida livre de doença e sobrevida global (análise multivariada). Análise multivariada da associação da fração de área de mastócitos CD117+ com DFS e OS na área IM e TP usando regressão de Cox (67 pacientes com CHC). A fração de área de células imunes por seção de área (mm2) foi convertida em percentis e então categorizada em baixa (percentil 0-24) versus alta (percentil 25-100). As taxas de risco mostram o risco relativo em comparação com 1 para a FA baixa. Valores em negrito indicam significância estatística no nível de p > 0,05. Abreviaturas: AF, fração de área; DFS, sobrevida livre de doença; OS, sobrevida global; HR, taxa de risco. Clique aqui para baixar esta tabela.
Arquivo Suplementar 1: Script para criação de ROI. Clique aqui para baixar este arquivo.
Discussão
Por meio do monitoramento da organização imunológica in situ no TME, o campo da imuno-oncologia pode adicionar novos biomarcadores prognósticos e preditivos do câncer. Nosso artigo publicado anteriormente sobre o papel das células imunes adaptativas no TME do CHC mostrou associações prognósticas positivas de células T CD3+ e CD8+, bem como células B CD20+ em ROIs selecionadas para o tempo de recorrência2. Aqui, o software de análise de imagem QuPath foi usado para avaliar a abundância de CD1a+ iDCs, mastócitos CD117+ e células NKp46+ NK em várias regiões distintas do CHC e avaliou sua significância prognóstica. Devido às formas irregulares de algumas células imunes inatas, sua quantificação pode ser imprecisa. É por isso que avaliar a fração de área de células imunopositivas foi a opção ideal. Entre os três tipos de células, apenas os mastócitos CD117+ demonstraram impacto prognóstico significativo: uma maior FA de mastócitos na MI e na área de TP foi associada a maior sobrevida.
Os mastócitos foram os mais abundantes em todas as ROIs, e a associação de sua FA na área peritumoral e na margem interna com melhor sobrevida reflete o papel antitumoral dos mastócitos. Os mastócitos podem ser atraídos para o TME por quimioatraentes liberados por células tumorais, como SCF ou CCL1518. Os mastócitos podem afetar a resposta antitumoral por citotoxicidade direta às células tumorais27 ou pela secreção de citocinas pró-inflamatórias que podem inibir o crescimento tumoral18. O aumento da densidade de mastócitos foi protetor contra a recorrência de câncer de próstata28, câncer gástrico27 e CHC após transplante de fígado29. Uma investigação abrangente consistindo em uma coorte maior de 245 pacientes com CHC encontrou uma correlação positiva entre infiltração de mastócitos mais significativa em amostras de tumor e sobrevida mais prolongada após a ressecção do tumor30. Rohr-Udilova et al. relataram resultados semelhantes de maiores densidades de mastócitos no tecido circundante do CHC; no entanto, apenas a densidade de mastócitos intratumorais foi associada a uma menor taxa de recorrência29.
Neste estudo, os mastócitos exerceram um efeito antitumoral apenas no fígado IM e TP. O TME de diferentes tumores é heterogêneo quanto à distribuição espacial das células imunes31. O significado prognóstico das células imunes no TME também está criticamente relacionado à sua distribuição espacial32,33. Diferentes abordagens para anotar a margem invasiva do tumor foram propostas, incluindo uma margem inteira de diferentes larguras 4,34,35, margens interna e externa, novamente com larguras diferentes36,37, com ou sem TP 2,7. Seguimos a metodologia padronizada e reprodutível do International Immunooncology Biomarkers Working Group para definir a margem invasiva como uma região de 1 mm centrada na borda que separa os ninhos de células malignas do tecido hospedeiro e representa o tumor central como a área tumoral remanescente38. Como o estudo anterior destacou diferenças significativas nos resultados da margem invasiva interna e externa2, essas regiões (cada uma com 500 μm de largura) foram analisadas separadamente. O perfil imunológico espacial da área peritumoral tem sua importância preditiva distinta 39,40 e, portanto, a área de TP também foi incluída.
A análise de CT, margem tumoral e PT hepática foi realizada em WSIs em oposição à opção de avaliar campos de visão selecionados nas respectivas regiões. A imagem de lâmina inteira é uma fonte potencialmente rica de dados41. Como não realizamos subamostragem, o "valor esperado verdadeiro" foi obtido de maneira eficiente no tempo10. Além disso, esse sistema foi razoável, pois foram analisadas apenas 1 ou 2 lâminas por bloco, o que permitiu evitar o viés de seleção de retalhos42. O fluxo de trabalho lento nos desafiou no estudo anterior, quando a estereologia foi aplicada para a quantificação de células imunes2.
A IHQ permite a localização direta da expressão de iDCs CD1a+, NKp46+NK e mastócitos CD117+ no tecido hepático, quantificação de suas distribuições e, portanto, subclassificação da coorte. A análise da IHQ confere a este estudo vantagens importantes sobre os ensaios bioquímicos em tecido solubilizado, o que pode levar a resultados falso-negativos quando apenas algumas células positivas para biomarcadores estão presentes43 e não refletem a reação regional das células imunes às características histopatológicas44. A IHQ também é preferível à análise em cortes corados com H&E. Estudos sobre câncer colorretal e câncer de pulmão de pequenas células mostraram que a avaliação de seções coradas com H & E pode fornecer biomarcadores robustos e quantitativos imunes a tumores45 , 46 , 47 ; no entanto, limitações para refletir a presença real de subtipos específicos ainda estão presentes.
A alta qualidade das seções de tecido e a coloração IHQ são um pré-requisito para uma avaliação precisa. Antecedentes altos, artefatos de borda e coloração específica, mas indesejável (por exemplo, coloração de células endoteliais dos espaços sinusoidais hepáticos com anticorpos anti-CD4) podem corromper todos os resultados. Um anticorpo CD56 foi a primeira escolha no estudo atual para detectar células assassinas naturais, mas foi substituído pelo anticorpo NKp46 devido à expressão de CD56 em ductos biliares imaturos. A seleção do marcador mais apropriado em termos de especificidade, padrão de coloração robusto e estabilidade em blocos FFPE representa outra questão a ser considerada. Por exemplo, CD117 foi escolhido em vez de triptase porque a expressão de triptase pode ser fortemente regulada negativamente, além disso, as células que não expressam CD117 não são mastócitos48.
O QuPath oferece a opção de suavizar as bordas da ROI do lado de fora e remover quaisquer artefatos, grandes vasos, tecido necrótico, coloração inespecífica, fundo e agregação de eritrócitos que não devem fazer parte da ROI. O operador precisa selecionar o ROI e pressionar a tecla "Alt" enquanto anota esses artefatos. A coloração inespecífica foi eliminada neste estudo como artefatos antes da quantificação para que não afetasse os resultados. No caso de coloração específica sistematicamente baixa ou alto fundo, o protocolo deve ser revisado para evitar possíveis vieses. O limiar foi ajustado para cada caso com uma coloração positiva específica fraca. Antes de qualquer uso rotineiro, o protocolo deve ser otimizado. Todas as análises devem ser feitas de forma cega e, idealmente, a precisão do limiar merece verificação cruzada.
O QuPath é uma solução de código aberto, intuitiva, extensível e fácil de usar para patologia digital e análise de imagens de lâminas inteiras9, testada anteriormente para análise de imagens em CHC49 e diferentes tipos de câncer50,51. A facilidade de operação do software para WSIs e as opções de anotação de regiões de interesse (por exemplo, áreas tumorais ou peritumorais) são as principais vantagens.
A aplicação de scripts existentes, modificados ou criados de novo pode acelerar substancialmente a análise. O script para criar anotações para ROIs foi aplicado, o que permitiu uma segmentação regional rápida e precisa em vez de delineamento manual. O script não é fixo e pode ser facilmente personalizado para margens inferiores a 500 μm para regiões com limites de tecido insuficientes. A largura da área IM, OM ou PT pode ser personalizada por meio do editor de script Automate > Show > adaptando a linha 30 Double Expand Margin Microns = 500 μm à largura disponível > Executar. Os ROIs também podem ser adicionados ou removidos. Em geral, os fluxos de trabalho no software não são fixos e o operador é livre para desenvolvê-los e modificá-los.
Uma infinidade de ferramentas de software disponíveis para análise de imagens patológicas, incluindo CellProfiler, ImageJ, Fiji, Microscopy Image Browser e outras, já estão disponíveis. No entanto, o QuPath se distingue por um conjunto de vantagens, como arquitetura de código aberto, interface amigável, recursos de personalização de algoritmos e integração de ferramentas avançadas de aprendizado de máquina. Esses recursos posicionam coletivamente este software de análise de imagem como uma escolha robusta para a análise diferenciada de tecidos cancerígenos no campo da patologia digital.
O QuPath apresentou a menor variabilidade em comparação com os softwares comerciais HALO (IndicaLab) e QuantCenter (3DHistech) para a detecção da expressão de Ki67 no câncer de mama52. O software também possui o potencial de ser instruído a executar um script para todas as imagens do projeto de maneira reproduzível de processamento em lote.
O QuPath nos permitiu criar dados quantitativos contínuos em vez de ordinais (semiquantitativos). Os dados ordinais obtidos por pontuação visual estão repletos de problemas devido à subjetividade na interpretação, variabilidade interobservador e baixa reprodutibilidade53. A prontidão da análise atual foi melhorada pelo fato de que indivíduos sem extensa formação em histopatologia e experiência em programação podem realizar análises automáticas, desde que a precisão das anotações seja verificada por um patologista. Um estudo sobre tumores de cabeça e pescoço demonstrou a adequação do QuPath para avaliação rápida e reprodutível do valor prognóstico de linfócitos infiltrantes de tumor CD57+50. O estudo também mostrou uma concordância substancial entre observadores humanos e o QuPath. Alta concordância entre observação manual e avaliação usando QuPath também foi apresentada em câncer oral54 e câncer de mama55. O artigo acima mencionado comparou a quantificação de IHC de 5 biomarcadores clínicos de câncer de mama usando QuPath e 2 produtos de software comerciais (Definiens Tissue Studio e inForm) com uma pontuação manual por um patologista. Ao longo deste estudo, o QuPath exibiu consistentemente o melhor desempenho, bem como o menor tempo de configuração e aplicação, mostrando-se assim uma excelente alternativa futura ao monitoramento visual e ao software comercial. Mesmo em comparação com o ImageJ, o software de código aberto mais conhecido para análise de imagens biomédicas, o QuPath se destaca em lidar com WSIs de grande porte56.
Existe um fórum online aberto para o QuPath, onde os usuários postam suas perguntas sobre métodos de patologia digital e apresentam uma comunidade ativa e engajada para apoiar o desenvolvimento de ferramentas para análise de imagens (https://forum.image.sc/tag/qupath).
A coorte utilizada neste estudo foi única e não representativa da população geral com CHC, pois abrangeu apenas pacientes elegíveis para ressecção hepática. Geralmente, uma minoria de pacientes com CHC (20%-30%) é elegível para ressecção hepática57. Essa também pode ser uma razão pela qual menos pacientes confirmaram cirrose, porque os pacientes com essa etiologia de CHC provavelmente tinham escores de Child-Pugh mais altos e não eram considerados ressecáveis. A coorte também foi bastante pequena e a maioria dos pacientes teve estágios iniciais de TNM da doença. Portanto, a extrapolação dos resultados para outras populações deve ser feita com cautela.
No estudo atual, a abundância de células imunes inatas no microambiente CHC foi estimada por meio da avaliação de sua FA, pois a contagem precisa de células imunes de formato irregular, como células dendríticas, pode ser difícil. Por outro lado, a fração de área das células imunes geralmente se correlaciona fortemente com suas densidades58. A marcação da imunoperoxidase fornece dados para a localização do antígeno, fração de área ou densidade das células que a expressam, mas a intensidade da coloração não está linearmente relacionada à quantidade de antígeno. Portanto, a IHQ não deve ser usada para avaliar quantitativamente o nível de expressão de uma proteína específica. A precisão do método também depende da uniformidade da espessura da seção, qualidade da imagem e resolução selecionada para classificação de pixels. Quaisquer resultados da análise de imagem devem ser validados e tratados com cautela. Como um único marcador não pode capturar totalmente a complexidade das células imunes, mais marcadores IHC e coloração multiplex são necessários para obter uma imagem confiável do TME imunológico do CHC. O uso de apenas um único marcador IHQ para fenotipagem de células imunes representa uma aproximação, e os resultados devem ser interpretados com cautela.
O uso do software de análise de imagem QuPath para avaliar as lâminas coradas com IHC nos permitiu avaliar a distribuição e a fração de área de iDCs locais, mastócitos e NKs em tumor e peritumor de pacientes com CHC e, em seguida, analisar suas relações com o prognóstico. A abundância de mastócitos na margem interna e no fígado peritumoral do CHC está associada a DFS e OS mais longos, destacando os efeitos antitumorais dessas células imunes inatas. O fluxo de trabalho analítico do QuPath é amigável, rápido e fácil de usar, com configurações padrão úteis e fácil exportação de dados. Este software foi endossado como uma plataforma promissora para análise de imagens digitais, que poderia atender à necessidade de reprodutibilidade, consistência e precisão em patologia digital.
Divulgações
Não há conflitos de interesse financeiros a serem divulgados.
Agradecimentos
Agradecemos as contribuições de Dom Ondřej Šebesta (Vinicna Microscopy Core Facility, Faculdade de Ciências, Charles University) para a digitalização de lâminas inteiras e o projeto "e-Infrastruktura CZ" (e-INFRA LM2018140), que nos forneceu os recursos computacionais para este estudo. Os técnicos Jana Dosoudilova e Jan Javurek são reconhecidos por sua excelente assistência técnica. Esta pesquisa foi financiada pelo programa de pesquisa e inovação Horizonte 2020 da União Europeia, concessão N°856620, e pelo Ministério da Saúde da República Tcheca, concessão AZV NU21-03-00506, e pelo Programa Cooperatio (Disciplinas cirúrgicas). A Vinicna Microscopy Core Facility é co-financiada pelo grande projeto RI da Czech-BioImaging LM2023050.
Materiais
| Name | Company | Catalog Number | Comments |
| Anti-CD1a | Leica Biosystems | PA0235 | Identifier- immature dendritic cells; RTU, Protocol F, ER2/20 min |
| Anti-CD117 | Leica Biosystems | PA0007 | Identifier- mast cells; RTU, Protocol F, ER2/20 min |
| BOND Plus Microscope Slides | Leica Biosystems, Germany | S21.2113.A | |
| BOND RXm | Leica Biosystems | 49.1501 | Fully Automated IHC Stainer |
| Bond Aspirating Probe Cleaning Kit | Leica Biosystems | CS9100 | |
| Bond Dewax Solution | Leica Biosystems | AR9222 | |
| Bond Polymer Refine Detection Kit | Leica Biosystems | DS9800 | |
| BondTM Epitope Retrieval 2 | Leica Biosystems | AR9640 | |
| BondTM Primary Antibody Diluent | Leica Biosystems | AR9352 | |
| BondTM Wash Solution 10X Concentrate | Leica Biosystems | AR9590 | |
| Computer Specifications: Intel(R) Core(TM) i5-10500 CPU @ 3.10GHz 3.10 GHz Installed RAM: 128 G | Intel | A 64-bit operating system that has Windows 7. Any computer with Java-based operating system and Excel available | |
| Coverslips | Leica Biosystems, Germany | 14071135636 | |
| CV Mount | Leica Biosystems, Germany | 14046430011 | |
| CV5030 Fully Automated Glass Coverslipper | Leica Biosystems | 149CVTS5025 | |
| Drying Oven UN30 | Memmert GmbH | UN30 | |
| GraphPad Prism 9.0 | GraphPad Software LLC | Version 12 | |
| Human NKp46/NCR1 Antibody, Monoclonal Mouse IgG2B Clone # 195314 | R&D Systems, Inc., United States | MAB1850 | Identifier- natural killer cells; Dilution 1:150, Protocol F + BLOK, ER2/20 min |
| Leica HI1210 - Water Bath | Leica Biosystems | 14041521466 | |
| Protein Block | Agilent Dako, United States | X0909 | |
| QuPath 0.3.2 or higher versions | version (QuPath v.0.3.2) | ||
| RM2235 Rotary Microtome | Leica Biosystems | 149AUTO00C1 | |
| ST5020 Multistainer Slide Stainer | Leica Biosystems | DEV-ST5010-CV5030 | |
| Statistica | StatSoft Inc. | version 7 | |
| Zeiss Axio Scan.Z1 | ScienceServices GmbH, Germany | 430038-9000-000 | Slide scanner |
Referências
- Du, M., Yin, Y. L., Xiao, L., Cai, Y. M., Ji, Y. Evaluating tumor-infiltrating lymphocytes in hepatocellular carcinoma using hematoxylin and eosin-stained tumor sections. World J Clin Cases. 10 (3), 856-869 (2022).
- Trailin, A., et al. T-and B-cells in the inner invasive margin of hepatocellular carcinoma after resection associate with favorable prognosis. Cancers (Basel). 14 (3), 604(2022).
- Xiao, N., et al. CD74+ macrophages are associated with favorable prognosis and immune contexture in hepatocellular carcinoma. Cancer Immunol Immunother. 71 (1), 57-69 (2022).
- Galon, J., et al. Type, density, and location of immune cells within human colorectal tumors predict clinical outcome. Science. 313 (5795), 1960-1964 (2006).
- Roxburgh, C. S. D., Salmond, J. M., Horgan, P. G., Oien, K. A., McMillan, D. C. Tumour inflammatory infiltrate predicts survival following curative resection for node-negative colorectal cancer. Eur J Cancer. 45 (12), 2138-2145 (2009).
- Klintrup, K., et al. Inflammation and prognosis in colorectal cancer. Eur J Cancer. 41 (17), 2645-2654 (2005).
- Kather, J. N., et al. Topography of cancer-associated immune cells in human solid tumors. Elife. 7, e36967(2018).
- Cho, J. Basic immunohistochemistry for lymphoma diagnosis. Blood Res. 57 (S1), 55-61 (2022).
- Bankhead, P., et al. QuPath: Open source software for digital pathology image analysis. Sci Rep. 7 (1), 16878(2017).
- Kolinko, Y., et al. Using virtual microscopy for the development of sampling strategies in quantitative histology and design-based stereology. Anat Histol Embryol. 51 (1), 3-22 (2022).
- Rodrigues, A., et al. Computer-assisted tumor grading, validation of PD-L1 scoring, and quantification of CD8-positive immune cell density in urothelial carcinoma, a visual guide for pathologists using QuPath. Surg Exp Pathol. 5, 12(2022).
- Hein, A. L., et al. QuPath digital immunohistochemical analysis of placental tissue. J Pathol Inform. 12, 40(2021).
- Lichterman, J. N., Reddy, S. M. Mast cells: A new frontier for cancer immunotherapy. Cells. 10 (6), 1270(2021).
- Veglia, F., Gabrilovich, D. I. Dendritic cells in cancer: the role revisited. Curr Opin Immunol. 45, 43-51 (2017).
- Wu, S. -Y., Fu, T., Jiang, Y. -Z., Shao, Z. -M. Natural killer cells in cancer biology and therapy. Mol Cancer. 19 (1), 120(2020).
- Kai, K., et al. Immunohistochemical analysis of the aggregation of CD1a-positive dendritic cells in resected specimens and its association with surgical outcomes for patients with gallbladder cancer. Transl Oncol. 14 (1), 100923(2021).
- Minesaki, A., Kai, K., Kuratomi, Y., Aishima, S. Infiltration of CD1a-positive dendritic cells in advanced laryngeal cancer correlates with unfavorable outcomes post-laryngectomy. BMC Cancer. 21 (1), 973(2021).
- Komi, D. E. A., Redegeld, F. A. Role of mast cells in shaping the tumor microenvironment. Clin Rev Allergy Immunol. 58 (3), 313-325 (2020).
- Maltby, S., Khazaie, K., McNagny, K. M. Mast cells in tumor growth: Angiogenesis, tissue remodelling and immune-modulation. Biochim Biophys Acta. 1796 (1), 19-26 (2009).
- Gooch, J. L., Lee, A. V., Yee, D. Interleukin 4 inhibits growth and induces apoptosis in human breast cancer cells. Cancer Res. 58 (18), 4199-4205 (1998).
- Brockmeyer, P., et al. High mast cell density indicates a longer overall survival in oral squamous cell carcinoma. Sci Rep. 7 (1), 14677(2017).
- Lee, H. A., et al. Natural killer cell activity is a risk factor for the recurrence risk after curative treatment of hepatocellular carcinoma. BMC Gastroenterol. 21 (1), 258(2021).
- Shimasaki, N., Jain, A., Campana, D. NK cells for cancer immunotherapy. Nat Rev Drug Discov. 19 (3), 200-218 (2020).
- Barrow, A. D., Martin, C. J., Colonna, M. The natural cytotoxicity receptors in health and disease. Front Immunol. 10, 909(2019).
- Guan, X., et al. Tumor-associated NK cells facilitate tumor growth via NKp46 in immunocompetent murine hepatocellular carcinoma. Immunol Lett. 258, 8-19 (2023).
- Ali, E., et al. Prognostic role of macrophages and mast cells in the microenvironment of hepatocellular carcinoma after resection. BMC Cancer. 24 (1), 142(2024).
- Lin, C., et al. Tryptase expression as a prognostic marker in patients with resected gastric cancer. Br J Surg. 104 (8), 1037-1044 (2017).
- Hempel, H. A., et al. Low intratumoral mast cells are associated with a higher risk of prostate cancer recurrence. Prostate. 77 (4), 412-424 (2017).
- Rohr-Udilova, N., et al. Morphometric analysis of mast cells in tumor predicts recurrence of hepatocellular carcinoma after liver transplantation. Hepatol Commun. 5 (11), 1939-1952 (2021).
- Lin, S. Z., et al. Prediction of recurrence and survival in hepatocellular carcinoma based on two cox models mainly determined by FoxP3+ regulatory T cells. Cancer Prev Res. 6 (6), 594-602 (2013).
- Jia, Q., Wang, A., Yuan, Y., Zhu, B., Long, H. Heterogeneity of the tumor immune microenvironment and its clinical relevance. Exp Hematol Oncol. 11 (1), 24(2022).
- Pyo, J. -S., Son, B. K., Lee, H. Y., Oh, I. H., Chung, K. H. Prognostic implications of intratumoral and peritumoral infiltrating lymphocytes in pancreatic ductal adenocarcinoma. Curr Oncol. 28 (6), 4367-4376 (2021).
- Yusa, T., et al. Survival impact of immune cells infiltrating peritumoral area of hepatocellular carcinoma. Cancer Sci. 113 (12), 4048-4058 (2022).
- Halama, N., et al. Localization and density of immune cells in the invasive margin of human colorectal cancer liver metastases are prognostic for response to chemotherapy. Cancer Res. 71 (17), 5670-5677 (2011).
- Zwing, N., et al. Analysis of spatial organization of suppressive myeloid cells and effector T cells in colorectal cancer-A potential tool for discovering prognostic biomarkers in clinical research. Front Immunol. 11, 550250(2020).
- Soeratram, T. T. D., et al. Prognostic value of T-cell density in the tumor center and outer margins in gastric cancer. Mod Pathol. 36 (9), 100218(2023).
- Gonzàlez-Farré, M., et al. Characterization and spatial distribution of the immune cell infiltrate in triple-negative breast cancer: a novel classification based on plasma cells and CD8+ T cells. Hum Pathol. 139, 91-105 (2023).
- Hendry, S., et al. Assessing tumor-infiltrating lymphocytes in solid tumors: A practical review for pathologists and proposal for a standardized method from the International Immunooncology Biomarkers Working Group: Part 1: Assessing the host immune response, TILs in invasive breast carcinoma and ductal carcinoma in situ, metastatic tumor deposits and areas for further research. Adv Anat Pathol. 24 (5), 235-251 (2017).
- Brück, O., et al. Spatial immunoprofiling of the intratumoral and peritumoral tissue of renal cell carcinoma patients. Mod Pathol. 34 (12), 2229-2241 (2021).
- Knebel, M., et al. Prognostic impact of intra- and peritumoral immune cell subpopulations in head and neck squamous cell carcinomas - comprehensive analysis of the TCGA-HNSC cohort and immunohistochemical validation on 101 patients. Front Immunol. 14, 1172768(2023).
- Lee, S., et al. Interactive classification of whole-slide imaging data for cancer researchers. Cancer Res. 81 (4), 1171-1177 (2021).
- Ciga, O., et al. Overcoming the limitations of patch-based learning to detect cancer in whole slide images. Sci Rep. 11 (1), 8894(2021).
- Rizzardi, A. E., et al. Quantitative comparison of immunohistochemical staining measured by digital image analysis versus pathologist visual scoring. Diagn Pathol. 7, 42(2012).
- Masuda, S., Nakanishi, Y. Application of immunohistochemistry in clinical practices as a standardized assay for breast cancer. Acta Histochem Cytochem. 56 (1), 1-8 (2023).
- Matsutani, S., et al. Tumor-infiltrating immune cells in H&E-stained sections of colorectal cancer tissue as a reasonable immunological biomarker. Anticancer Res. 38 (12), 6721-6727 (2018).
- Väyrynen, J. P., et al. Prognostic significance of immune cell populations identified by machine learning in colorectal cancer using routine hematoxylin and eosin-stained sections. Clin Cancer Res. 26 (16), 4326-4338 (2020).
- Zhou, G., et al. Clinical significance of tumor-infiltrating lymphocytes investigated using routine H&E slides in small cell lung cancer. Radiat Oncol. 17 (1), 127(2022).
- Horny, H. -P., Sotlar, K., Valent, P. Mastocytosis. Immunol Allergy Clin North Am. 34 (2), 315-321 (2014).
- Mi, H., Ho, W. J., Yarchoan, M., Popel, A. S. Multi-scale spatial analysis of the tumor microenvironment reveals features of cabozantinib and nivolumab efficacy in hepatocellular carcinoma. Front Immunol. 13, 892250(2022).
- de Ruiter, E. J., et al. Assessing the prognostic value of tumor-infiltrating CD57+ cells in advanced stage head and neck cancer using QuPath digital image analysis. Virchows Archiv. 481 (2), 223-231 (2022).
- Fanucci, K. A., et al. Image analysis-based tumor infiltrating lymphocytes measurement predicts breast cancer pathologic complete response in SWOG S0800 neoadjuvant chemotherapy trial. NPJ Breast Cancer. 9 (1), 38(2023).
- Acs, B., et al. Ki67 reproducibility using digital image analysis: an inter-platform and inter-operator study. Lab Invest. 99 (1), 107-117 (2019).
- Sobottka, B., et al. Establishing standardized immune phenotyping of metastatic melanoma by digital pathology. Lab Invest. 101 (12), 1561-1570 (2021).
- Moratin, J., et al. Digital pathology scoring of immunohistochemical staining reliably identifies prognostic markers and anatomical associations in a large cohort of oral cancers. Front Oncol. 11, 712944(2021).
- Bankhead, P., et al. Integrated tumor identification and automated scoring minimizes pathologist involvement and provides new insights to key biomarkers in breast cancer. Lab Invest. 98 (1), 15-26 (2018).
- Humphries, M. P., Maxwell, P., Salto-Tellez, M. QuPath: The global impact of an open source digital pathology system. Comput Struct Biotechnol J. 19, 852-859 (2021).
- Koh, J. H., et al. Liver resection versus liver transplantation for hepatocellular carcinoma within Milan criteria: a meta-analysis of 18,421 patients. Hepatobiliary Surg Nutr. 11 (1), 78-93 (2022).
- Eriksen, A. C., et al. Computer-assisted stereology and automated image analysis for quantification of tumor infiltrating lymphocytes in colon cancer. Diagn Pathol. 12 (1), 65(2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados