Method Article
간세포 암종의 미세환경에서의 비만 세포는 유리한 예후를 제공합니다: QuPath 이미지 분석 소프트웨어를 사용한 후향적 연구
요약
절제 후 간세포암의 내측 가장자리와 종양 주위 영역에 비만세포가 존재하면 유리한 예후를 얻을 수 있습니다. 이 연구는 QuPath 이미지 분석 소프트웨어를 디지털 병리학의 재현성, 일관성 및 정확성에 대한 요구를 충족할 수 있는 유망한 플랫폼으로 승인합니다.
초록
간세포 암종(HCC) 내 면역 세포의 현장 검출이 제공하는 통찰력은 환자 결과에 대한 정보를 제공할 수 있습니다. 종양 조직 내 면역 세포의 발현 및 국소화를 조사하는 연구는 종양 영역에 대한 정확한 주석이 부족하고 현미경 시야의 무작위 선택을 포함하여 여러 가지 문제와 관련이 있습니다. QuPath는 전체 슬라이드 이미지(WSI) 분석에서 디지털 병리학에 대한 증가하는 요구를 충족할 수 있는 사용자 친화적인 오픈 소스 소프트웨어입니다.
근치적 절제술을 받은 67명의 간세포암 환자의 대표 검체에서 CD1a+ 미성숙 수지상세포(iDC), CD117+ 비만세포 및 NKp46+ 자연살해세포(NK) 세포에 의한 간세포암 및 인접 조직의 침윤을 면역조직화학적으로 평가했습니다. 양성으로 염색된 세포의 면적 분율(AF)은 종양 센터(TC), 내부 마진(IM), 외부 마진(OM) 및 종양 주위(PT) 영역에서 QuPath를 사용하여 WSI에서 자동으로 평가되었습니다. 면역 세포의 예후 유의성은 TTR(Time to Recurrence), 무병 생존(DFS) 및 전체 생존(OS)에 대해 평가되었습니다.
비만세포의 AF는 NK의 AF보다 유의하게 높았으며, iDC의 AF는 각 관심 영역의 NK에 비해 유의하게 낮았다. IM 및 PT 영역에서 비만 세포의 높은 AF는 더 긴 DFS와 관련이 있었습니다. 또한 IM에서 비만 세포의 높은 AF는 더 긴 OS와 관련이 있었습니다.
이 소프트웨어를 사용한 컴퓨터 보조 분석은 절제 후 간세포의 다른 영역에서 종양 침투 면역 세포(iDC, 비만 세포 및 NK)에 대한 예후 정보를 얻는 데 적합한 도구입니다. 비만 세포는 모든 관심 영역(ROI)에서 가장 큰 AF를 나타냈습니다. 종양 주위 영역의 비만 세포와 IM은 긍정적인 예후 유의성을 보였다.
서문
종양에 침투하는 면역 세포의 공간적 조직과 풍부함은 다양한 암에서 생존에 영향을 미치는 것으로 입증되었으며, 간세포 암종(HCC)에는 1,2,3,4가 포함되었습니다. 암에서 종양 침윤 림프구의 예후 중요성은 헤마톡실린 및 에오신(H&E) 염색 섹션 5,6에서 처음으로 나타났습니다. 그 후, 면역조직화학(IHC)을 이용한 Galon 등의 선구적인 연구에서 대장암 조직에서 CD3+ 및 CD8+ T 세포의 밀도와 예후의 연관성을 입증했습니다4.
IHC는 임상 결과와의 추가적인 연관성을 위해 종양 조직 내 면역 세포를 시각화, 정량화 및 매핑하기 위한 황금 표준입니다7. IHC는 저렴한 비용, 광범위한 가용성, 포르말린 고정 파라핀 포매(FFPE) 조직과의 호환성과 같은 여러 가지 이점을 제공합니다8. 그러나 IHC 염색 면역 세포의 정확한 평가는 큰 과제입니다. 선택된 현미경 시야에서 기존의 스코어링은 시간이 많이 걸리며 후보 바이오마커 선택 및 신뢰할 수 있는 임상 상관 관계에 필수적인 고품질의 재현 가능하고 객관적인 분석을 보장하기에 더 이상 충분하지 않습니다9. 전체 슬라이드 스캔은 전체 이미지로 평가하거나서브샘플링 10 후에 평가할 수 있습니다.
조직 샘플에 있는 면역 세포의 풍부도에 대한 전산화된 정량적 평가는 실용적이고 정확하며 신뢰할 수 있고 임상적으로 관련된 데이터를 보장할 수 있습니다11. QuPath는 IHC 염색 슬라이드의 디지털 이미지 분석을 가능하게 하고 개별 샘플에서 얻은 정보의 양을 극대화하는 무료 오픈 소스 소프트웨어입니다12.
미성숙 수지상 세포(iDC), 자연살해(NK) 세포 및 비만 세포는 항종양 면역 반응에 관여하고 환자의 결과와 상관관계가 있는 것으로 나타났기 때문에 13,14,15 여기에서는 FFPE HCC 조직에서 이들의 공간적 분포를 평가하고 예후 영향을 탐색하기 위한 단계별 프로토콜을 제시합니다. CD1a는 주로 미성숙 수지상 세포의 막에서 발현되며, 그 밀도는 다양한 인간 종양에서 임상 결과와 관련이 있습니다16,17. 비만 세포는 종양 미세환경(TME) 내에서 전종양 또는 항종양 역할을 할 수 있습니다18. 그들은 혈관 신생을 지원하고 전이를 촉진할 수 있다19. 반대로, 비만세포는 IL-420의 생산을 통해 종양세포 사멸을 매개할 수 있는 것으로 보고되었습니다. Anti-CD117 염색은 일반적으로 종양 조직21 내의 비만 세포를 시각화하고 정량화하는 데 사용됩니다. NK 세포는 암세포23을 사멸시킴으로써 HCC22의 감시 및 제어에 기여하는 것으로 생각됩니다. NK에 의한 NKp46 발현은 항종양 활성에 중요한 파라미터이다24. 그러나 NKp46+ NK세포는 간세포암 환자에서 종양 직경과 양의 상관관계가 있었다25. 그럼에도 불구하고 환자 생존율과의 상관 관계에 대해서는 알려진 바가 거의 없습니다.
우리는 간세포암의 다양한 영역에서 CD1a+ iDC, CD117+ 비만세포 및 NKp46+ NK 세포의 풍부도를 정량적으로 평가하고 이들의 예후적 중요성을 강조하는 것을 목표로 했습니다. 1997년에서 2019년 사이에 BCLC 지침에 따라 절제술을 받을 자격이 있고 Pilsen University Hospital에서 치료 목적 간 절제술을 받은 병리학적으로 확인된 I-IV 병기 간세포암 환자 총 70명이 이번 후향적 연구에 포함되었습니다. 환자의 병리학 보고서를 검토하였다. 이 연구에 포함된 환자 중 원격 전이가 있거나 수술 전에 화학 요법 또는 방사선 요법과 같은 선행 요법을 받은 환자는 없었습니다. 질이 낮은 조직학적 검체를 가진 총 3명의 환자를 제외하였고, 나머지 67명의 환자를 연구에 포함시켰다(표 1).
프로토콜
이 의정서는 헬싱키 선언(2013년 버전)에 명시된 윤리 기준에 따라 수행된 연구의 일부였습니다. 플젠 의과대학 윤리위원회와 플젠 대학병원의 승인을 받았습니다(118/2021, 2021년 3월 11일). 그림 1 은 사용된 방법의 요약을 보여줍니다.
1. FFPE 조직 블록의 선택
- 치료 의사 또는 병리학자의 도움을 받아 환자 파일에서 FFPE 블록 식별자를 검색합니다.
- 로컬 병리학 아카이브에서 FFPE 블록을 요청합니다.
- 각 환자에 대해 생존 가능한 종양 조직을 포함하는 2-3개의 FFPE 블록을 주변 종양 주위 영역 및 비종양 간과 우선적으로 선택합니다.
알림: 이 선택에 대해서는 전문 병리학자의 의견을 얻는 것이 좋습니다. 비종양 간의 조직학적 평가와 기억소거학, 임상적, 실험실 데이터를 사용하여 간세포암의 기저 원인을 정의했습니다.
2. FFPE 조직의 탈파라핀화 및 재수화
- 마이크로톰의 각 FFPE 조직 블록에서 4μm 두께의 조직 절편 하나 또는 두 개를 절단합니다.
참고: 단면의 두께는 눈에 띄는 염색 영향 없이 3μm에서 5μm 사이일 수 있습니다. 그러나 4μm의 두께가 표준입니다. - 절편을 수조(42°C)에 넣어 주름을 제거합니다.
- 슬라이드를 집어 양전하를 띤 유리 현미경 슬라이드에 올려 놓습니다. 그런 다음 상온(AT)에서 24시간 동안 건조하기 위해 슬라이드를 적용합니다.
- 슬라이드를 온도 조절기(56°C)에 1시간 동안 놓습니다.
- 탈왁스 용액-1(72°C에서 30초), 탈왁스 용액-2(72°C에서 10초), 탈왁스 용액-3(AT에서 10초), 에탄올 96%-1(10초), 에탄올 96%-2(10초) 및 에탄올 96%-3(10초)의 절편을 탈파라핀화합니다.
- 자동 염색기를 사용하여 세척 용액-1(10초, AT), 세척액-2(10초, AT) 및 세척액-3(5분, AT)의 섹션을 재수화합니다.
3. 완전 자동화된 염색기에서 IHC 수행
알림: IHC는 제조업체의 프로토콜에 따라 수행됩니다. 단계는 아래에 간략하게 설명되어 있습니다.
- 항원 회수 방법
- pH가 높은 ER 용액(예: Tris-EDTA)에서 열 유도 에피토프 검색(ER) 절차를 사용하여 항원을 검색합니다: ER 용액-1(10초, AT), ER 용액-2(10초, AT), ER 용액-3(100°C에서 20분), ER 용액-4(12분, AT).
- 슬라이드를 세척액으로 3회(각 30초) 및 1회(3분) 헹굽니다.
- AT에서 5분 동안 Peroxide Block 용액을 사용하여 내인성 과산화효소를 차단합니다.
- 세척액으로 3회(각 30초) 헹굽니다.
- AT에서 30분 동안만 NKp46용 단백질 블록으로 항체의 비특이적 결합을 차단합니다.
- 1차 항체와 결합: 1차 항체와 함께 AT에서 15분 동안 배양한 후 세척액으로 3회(각 30초) 헹굽니다.
- 2차 항체 결합의 경우 아래 설명된 단계를 따르십시오.
- AT에서 8분 동안 양고추냉이 과산화효소 접합 2차 항체로 배양합니다.
- 세척액으로 2회(각 2분) 헹굽니다.
- 슬라이드를 디아미노벤지딘(DAB) 용액으로 헹구고 AT에서 10분 동안 DAB 용액으로 배양하여 반응을 시각화합니다.
- 탈이온수로 3x(30초) 동안 헹굽니다.
- Mayer의 헤마톡실린(hematoxylin)으로 절편을 대조염색합니다.
- 탈이온수 1x(30초), 세척액 1x(30초), 탈이온수 1x(30초)로 헹굽니다.
- 완전 자동화된 슬라이드 염색기를 사용하여 에탄올 70%(2분), 에탄올 80%(2분), 에탄올 96%-1(3분), 에탄올 96%-2(3분), 에탄올 100%(3분), 자일렌-1(4분), 자일렌-2(4분) 및 자일렌-3(4분) 섹션을 탈수합니다.
- 완전 자동화된 유리 커버슬리퍼를 사용하여 슬라이드의 섹션을 장착 매체에 장착합니다.
참고: 음성 및 양성(편도선) 조직 대조군 샘플이 전체적으로 사용되었습니다.
4. 전체 슬라이드 스캔 얻기
- 20x 대물렌즈와 중간 밀도의 고밀도 초점 렌즈를 사용하여 전체 슬라이드 스캐너에 초점을 맞춰 염색된 슬라이드를 스캔합니다.
5. 소프트웨어를 사용한 IHC 분석
- 소프트웨어에서 IHC 이미지에 대한 프로젝트를 만듭니다.
- QuPath-0.4.3 이상을 다운로드하여 엽니다.exe
- 적절한 이름으로 새 폴더를 만듭니다.
- 소프트웨어의 왼쪽 상단 모서리에 있는 프로젝트 만들기 버튼을 클릭하고 새로 만든 폴더를 선택합니다.
- 스캔한 이미지를 소프트웨어 창으로 드래그합니다. 새 창이 자동으로 나타나면 H-DAB> 이미지 유형 설정을 선택하고 가져오기 를 클릭합니다(그림 2).
- 메뉴의 왼쪽 창에 있는 이미지 목록을 관찰하십시오. 두 번 클릭하여 이미지를 엽니다.
- 상단의 기본 메뉴에서 File(파일 )을 선택하여 이미지를 프로젝트로 저장합니다 .
참고: 이 소프트웨어는 많은 전체 슬라이드 형식을 포함하여 다양한 이미지 형식을 처리할 수 있습니다. 그러나 원시 데이터 및 관련 메타데이터의 손실을 방지하기 위해 tiff 형식이 선호됩니다.
- 관심 영역에 주석 달기
- 상단의 메인 메뉴에서 브러시 도구 또는 지팡이 도구를 선택하여 종양 영역에 주석을 추가합니다. 종양의 불연속 영역의 경우 5.2.1.1-5.2.1.3단계를 따르십시오.
- 별도로 주석을 추가합니다.
- Ctrl 키를 누른 상태에서 모든 종양 영역을 선택합니다.
- 마우스 오른쪽 버튼을 클릭하고 Edit Multiple > Merge Selected를 선택합니다.
- 종양 주석이 선택되어 있는지 확인합니다(테두리는 노란색으로 표시됨).
- 메인 메뉴에서 Automate(자동화 )를 클릭하고 Show Script Editor(스크립트 편집기 표시)를 선택합니다.
- 보충 파일 1 또는 https://petebankhead.github.io/qupath/scripts/2018/08/08/three-regions.htmls 에서 사용할 수 있는 스크립트를 복사하여 스크립트 편집기에 붙여넣은 다음 실행을 클릭합니다. ROI: 종양 센터(TC), 내부 마진(IM), 외부 마진(OM) 및 종양 주변 영역(PT)이 자동으로 생성되고 레이블이 지정됩니다.
- 관심 영역을 만드는 다른 방법은 5.2.5.1-5.2.5.5단계를 수행합니다.
- 상단의 메인 메뉴에서 폴리라인 도구를 선택하여 악성 세포 둥지와 인접한 비종양 조직을 구분하는 테두리를 그립니다.
- 결과 테두리를 선택합니다. 상단의 기본 메뉴에서 Objects > Annotation > Expand annotations > 500 μm( 확장 반경 의 경우 500 μm) > Line Cap의 경우 Flat을 선택합니다. Remove Interior(내부 제거 )를 활성화하고 Constrain to parent(부모로 제한)를 활성화합니다.
- 결과 주석인 Edit Single > Split을 마우스 오른쪽 버튼으로 클릭합니다. 두 개의 개별 주석은 테두리의 각 측면에서 500μm 너비의 영역으로 자동 확장됩니다.
- 왼쪽 목록에서 주석을 마우스 오른쪽 단추로 클릭하고 속성 설정을 선택한 다음 이름을 입력하여 IM 및 OM에 맞게 주석이 추가된 영역의 이름을 지정합니다.
- TC는 남은 종양 영역을 나타냅니다. 동일한 절차를 사용하여 OM에 인접한 500μm 너비의 PT 영역을 확장합니다.
참고: 주석이 달린 슬라이드와 QuPath AF 검출은 전문 조직 병리학자가 검토했습니다.
- 상단의 메인 메뉴에서 브러시 도구 또는 지팡이 도구를 선택하여 종양 영역에 주석을 추가합니다. 종양의 불연속 영역의 경우 5.2.1.1-5.2.1.3단계를 따르십시오.
- 픽셀 분류 최적화Optimization of pixel classification
- 메인 메뉴에서 Rectangle 도구를 선택합니다. 그런 다음 구별할 모든 유형의 픽셀(예: hematoxylin, DAB 및 배경)을 포함하는 영역에 주석을 추가합니다.
- 메인 메뉴에서 Analyze > Preprocessing > Estimate Stain Vector를 선택합니다.
- Visual Stain Editor 창이 자동으로 나타나면 Auto > OK를 선택하고 H-DAB estimated를 stain vector의 이름으로 설정합니다.
- 다른 방법을 보려면 5.3.4.1-5.3.4.3단계를 수행합니다.
- 메인 메뉴에서 Rectangle 도구를 선택하고 hematoxylin으로 염색된 전형적인 핵의 작은 영역에 주석을 추가합니다.
- 왼쪽 메뉴에서 이미지를 선택하고 Stain 1 > Yes를 두 번 클릭합니다.
- 메인 메뉴에서 Rectangle 도구를 선택하고 DAB로 포지티브 염색의 작은 영역에 주석을 달 수 있습니다. 왼쪽 메뉴에서 이미지를 선택하고 Stain 2 > Yes를 두 번 클릭합니다.
- Pixel Classification 옵션을 사용한 양성 면역 세포의 면적 분율 평가.
- 메인 메뉴에서 Classify(분류) > Pixel classification(픽셀 분류) > Create thresholder(임계값 생성 )를 클릭합니다.
- 해상도 > 채널에 대해 High 를 선택합니다 . DAB > 프리필터: Gaussian > Sigma: 0.5 >(그림 3) 임계값: (0.2-0.3) 임계값 초과: 양수 > 임계값 미만: >음.
- 영역의 경우 Any Annotation(모든 주석)을 선택하고 Classifier(분류자)의 이름을 입력한 > Save > Measure(측정값) > OK(확인)를 선택합니다. DAB 양성 셀은 빨간색으로 바뀝니다(그림 4).
- 왼쪽 메뉴의 결과를 스프레드시트에 복사합니다.
- 또는 상단 메인 메뉴에서 Measure 를 열고 Show Annotation Measurements > Copy to Clipboard를 선택한 다음 결과를 스프레드시트에 붙여넣습니다.
- 이미지를 닫으려면 [Multi View]> 마우스 오른쪽 버튼으로 클릭하고 [뷰어 닫기]> 클릭합니다.
참고: 정량화하기 전에는 모든 아티팩트가 제거되었습니다. 임계값은 약한 특정 양성 염색이 있는 각 사례에 대해 조정되었습니다. 주석과 픽셀 분류의 정확성은 먼저 교차 점검한 다음 선임 조직 병리학자에 의해 검토되었습니다. 모든 분석은 블라인드 방식으로 수행되었습니다. 소프트웨어를 사용한 IHC 분석에 대한 자세한 내용은 https://forum.image.sc/tag/QP 에서 확인할 수 있습니다.
6. 통계적 방법 및 데이터 분석
- 연구 종점은 TTR(Time to Recurrence), 무병 생존(DFS) 및 전체 생존(OS)이었습니다. 재발이나 사망이 없는 환자는 마지막 추적 조사에서 검열되었습니다.
참고: 비정규적으로 분포된 연속형 데이터는 중앙값(최소-최대)으로 표시됩니다. 비율은 원시 데이터(백분율)로 표현됩니다. Friedman ANOVA는 서로 다른 관심 영역(ROI)에서 면역 세포의 면적 분율을 비교한 후 Wilcoxon 일치 쌍 테스트와 Bonferroni 보정을 비교했습니다. 대부분의 변수가 비모수 분포를 따르기 때문에 Spearman 상관 계수를 사용하여 순서 변수 또는 양적 변수 쌍 간의 연관성을 평가했습니다. 종점에 대한 개별 예측 변수의 예후 값을 결정하기 위해 단변수 다음에 다변수 Cox 회귀 분석을 수행했습니다. 위험비(HR)를 계산했습니다. HR은 결합된 중간 상위 그룹에 대한 상대 위험도를 나타낸 반면 낮은 그룹에 대한 상대 위험은 1이었습니다. 모든 생존 추정치는 Kaplan-Meier 방법에 의해 계산되었으며 로그 순위 검정에 의해 그룹 간에 비교되었습니다. 통계 분석에는 GraphPad Prism 9.0(GraphPad Software LLC)이 사용되었습니다. 양측 p-값 < 0.05는 통계적으로 유의한 것으로 간주되었습니다.
결과
환자의 인구 통계학적 특성 및 임상적 특성은 표 1에 제시되어 있습니다. 환자의 평균 연령은 69세였으며 대부분 남성(77.6%)이었다. 간세포암의 병인과 관련하여, 만성 비바이러스성 간염이 가장 빈번한 배경 질환이었으며, 비알코올성 지방간염(NASH)의 유병률(23.9%)이 높았다. 간경변증을 배경질환으로 가지고 있는 환자는 15명(22.4%)에 불과했다. 환자의 대다수(68.7%)가 TNM I기를 앓고 있었다. 보다 자세한 임상 및 병리학 결과는 이전에 발표된 연구 2,26에 설명되어 있습니다.
결과
마지막 추적관찰에서 29명(41.8%)의 환자에서 종양 재발이 관찰되었으며, 그 중 89.7%는 국소 재발이었고 38명(56.7%)은 사망했다. 수술 후 5년 시점에서 DFS의 비율은 33.2%, OS의 비율은 49.4%, RFP(Recurrence-Free Proportion)는 48.7%였다(Table 2).
다양한 관심 영역의 면역 세포 분포
CD117 단백질은 주로 둥근 세포의 세포질막에서 발견되었습니다(그림 5A). TC와 IM에서는 대부분 기질과 혈관 주위 공간에 국한되어 있었습니다. OM 및 PT 영역에서는 CD117+ 세포가 종양 캡슐과 혈관 주위 공간에서 관찰되었습니다.
NKp46 단백질은 주로 둥근 세포의 세포질막에서 발견되었으며, TC와 IM의 정현파와 같은 공간 내부에 존재하고 기질과 관련이 있었습니다(그림 5B). OM 및 PT 영역에서는 NKp46+ 세포가 종양 둥지 주변의 캡슐과 문맥 기질에서 관찰되었습니다.
CD1a 단백질은 TC와 IM에서 발견되었으며, 주로 기질의 둥근 세포(흩어져 있거나 응집된 형태)의 세포질막, 정현파와 같은 공간의 내부 및 경계를 따라 발견되었습니다(그림 5C). PT 영역에서, CD1a+ DC는 정현파의 경계 내부 및 따라, 그리고 문맥의 담도 상피 내에서 관찰되었습니다.
비만 세포의 AF는 NKp46+ 세포의 AF보다 훨씬 컸습니다(p < 0.001)(그림 6). 각 ROI의 CD1a+ iDC는 비만 세포 및 NK 세포에 비해 가장 낮은 AF를 보여주었습니다(p < 0.001)(그림 6). TC 또는 IM에서 CD117+ 및 NKp46+ 세포의 AF는 PT 영역 및 OM의 AF보다 유의하게 작았습니다(p < 0.001)(그림 6). CD1a+ 셀의 경우 AF는 지역 간에 큰 차이가 없었습니다.
모든 ROI 간의 개별 면역 세포에 대한 유의한 연관성이 표 3에 나와 있습니다. 그림 7 은 서로 다른 지역 내의 서로 다른 면역 세포 간의 유의미한 연관성에 대한 히트 맵을 보여줍니다. TC에서 CD117 + 비만 세포의 AF는 TC 및 IM에서 CD1a의 AF와 유의한 상관 관계가 있었습니다.
임상 및 병리학 변수의 예후 가치
임상변수와 병리변수에서는 나이가 어릴수록 재발 위험이 높았고(HR=0.96, CI: 0.93-0.99, p=0.007), DFS 및 OS와 관련된 변수는 없었다(표 4).
면역 세포 침윤의 예후 가치
IM에서 CD117+ 비만 세포의 높은 AF는 더 긴 DFS(HR=0.48, CI: 0.241 - 0.935, p=0.031) 및 OS(HR=0.34, CI: 0.167 - 0.703, p=0.004)와 관련이 있었습니다(그림 8, 표 5). 또한, PT 영역에서 CD117+ 비만 세포의 높은 AF는 더 긴 DFS와 관련이 있었습니다(HR = 0.48, CI: 0.247 - 0.945, p = 0.034)(그림 8, 표 5). 대조적으로, iDC 및 NK 세포의 AF는 어떤 결과와도 관련이 없었습니다. TNM 병기를 조정한 후 내측 가장자리의 CD117+ 비만세포는 DFS(HR=0.46, CI: 0.23-0.91, p=0.027) 및 OS(HR=0.33, CI: 0.16-0.68, p=0.003)와 유의한 연관성을 유지했다(표 6). PT 영역의 비만세포의 경우, DFS와의 연관성도 유의하게 유지되었다(HR=0.47, CI: 0.24-0.93, p=0.029)(표 6).
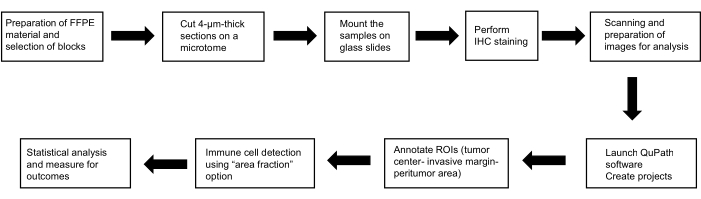
그림 1: 방법에 대한 대표 개략도. 간세포에서 비만세포, 수지상세포, 자연살해세포의 예후적 역할을 식별하기 위한 작업 흐름의 개략도. 약어 : FFPE, 포르말린 고정 파라핀 임베디드; H&E, 헤마톡실린 및 에오신; ROI, 관심 영역. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
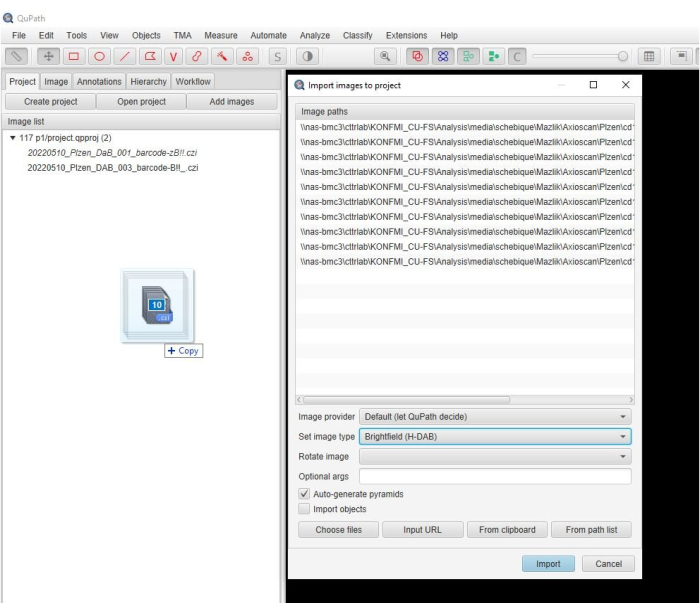
그림 2: 소프트웨어 창의 스크린샷 소프트웨어에서 프로젝트로 이미지를 가져오는 단계를 보여주는 설명 스크린샷입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
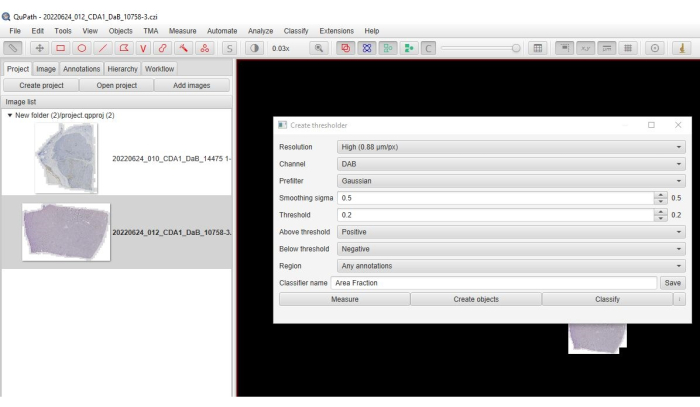
그림 3: 면적 분수 옵션의 스크린샷. 포지티브 염색의 면적 분율을 정량화하기 위해 선택한 매개변수를 보여주는 소프트웨어의 스크린샷. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
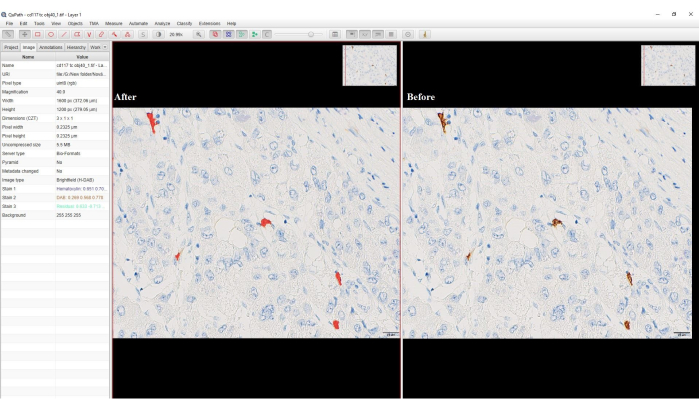
그림 4: 픽셀 분류 옵션의 스크린샷. 픽셀 분류 전후의 DAB 양성 세포를 보여주는 소프트웨어의 스크린샷. 갈색을 띤 DAB 염색은 픽셀 분류 후 빨간색으로 변환되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
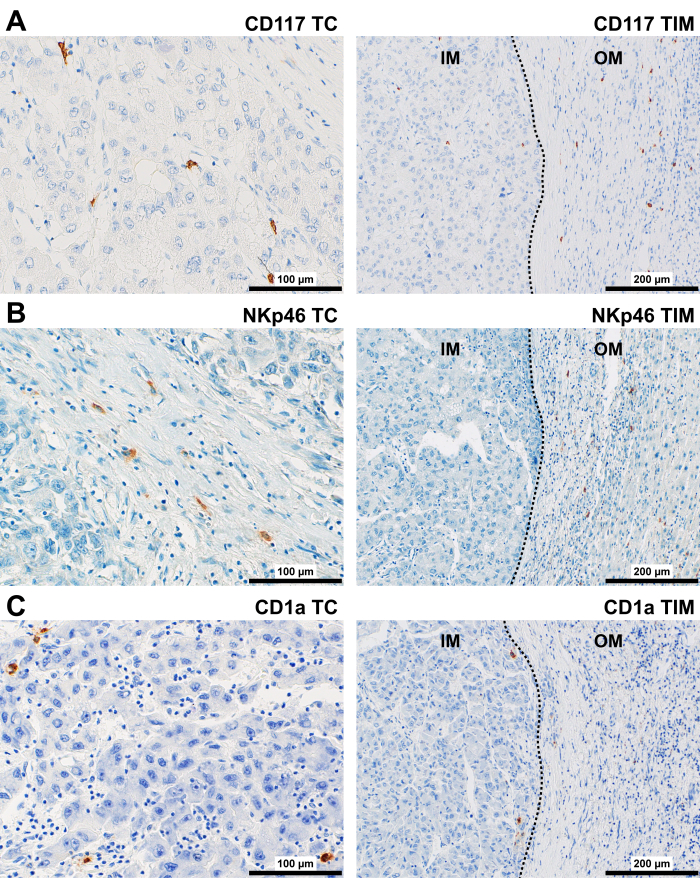
그림 5: 간세포암 환자에서 선천성 면역 세포의 대표적인 면역염색. 간세포암의 TC(400x) 및 TIM(200x)에서 (A) CD117+, (B) NKp46+ 및 (C) CD1a+ 세포의 대표적인 면역염색. 약어: HCC, hepatocellular carcinoma; TC, 종양 센터; TIM, 종양 침습성 마진, 내부 마진(IM) 및 외부 마진(OM). 점선은 종양과 비종양 조직 사이의 경계를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
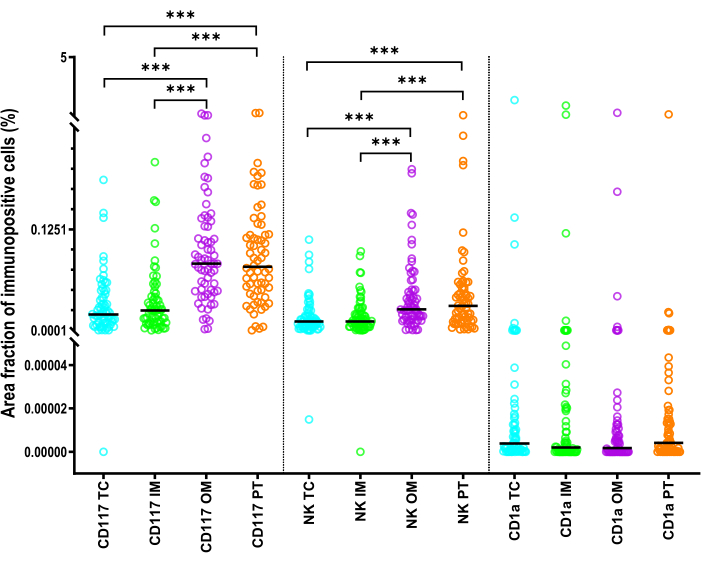
그림 6: 간세포암 환자에서 선천성 면역 세포의 면적 분율을 설명하는 통계. 간세포의 TC, IM, OM 및 PT 영역에서 CD1a+ 수지상세포, CD117+ 비만세포 및 NKp46+ NK 세포의 면적 분율에 대한 통계. 검은색 선은 중앙값입니다. Wilcoxon 일치 쌍 검정과 Bonferroni 보정이 비교를 위해 사용되었습니다. 약어: HCC, hepatocellular carcinoma; TC, 종양 센터; IM, 내부 여백; OM, 외부 여백; PT, 종양 주위 영역. : p <0.001 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
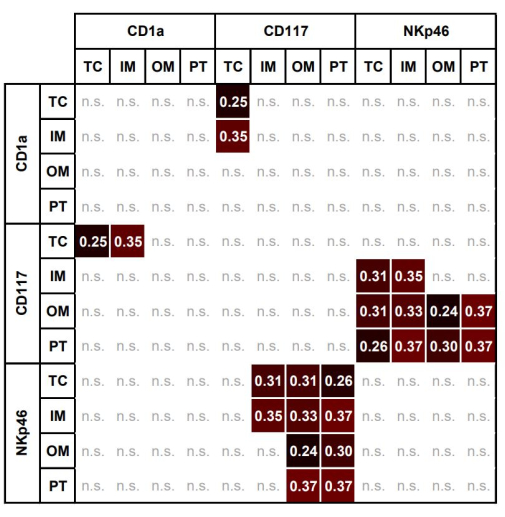
그림 7: 서로 다른 관심 영역에서 비만 세포, DC 및 NK의 면적 분율 간의 유의미한 상관 관계에 대한 히트 맵. TC, IM, OM 및 PT에서 CD117+ 비만 세포, CD1a+ DC 및 NKp46+NK의 면적 분율 간의 유의한 상관 관계에 대한 히트맵(Spearman ρ, p < 0.05). 약어: TC, tumor center; IM, 내부 여백; OM, 외부 여백; PT, 종양 주위 영역; DC, 수지상 세포; NK, 자연살해세포(natural killer cell). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
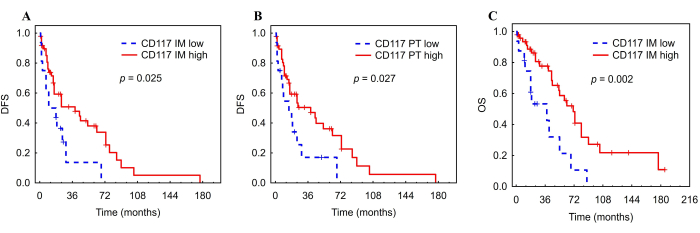
그림 8: 간세포암 환자의 종양 침윤 비만 세포에 대한 Kaplan-Meier DFS 및 OS 곡선. 간세포의 내측(A, C) 및 PT(B)에서 종양 침투 비만 세포의 낮은 AF와 높은 AF에 따른 DFS 및 OS의 Kaplan-Meier 분석. 약어: HCC, hepatocellular carcinoma; DFS, 무병 생존; IM, 내부 침습성; PT, 종양 주위 영역. 이 그림은 Ali et al.26의 허가를 받아 각색되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: 등록된 간세포암종 환자의 임상적 배경. 등록된 간세포암 사례의 임상 배경. 약어: NAFLD, 비알코올성 지방간 질환. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 2: Kaplan-Meier 분석에서 예상되는 결과 확률. Kaplan-Meier 분석에서 RFP, DFS 및 OS의 예상 확률입니다. 약어: RFP, 재발 없는 비율; DFS, 무병 생존; OS, 전체 생존. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 3: 서로 다른 ROI에서 종양 침윤 비만 세포, 수지상 세포 및 자연 살해 세포의 면적 분율 간의 상관 관계. 서로 다른 ROI에서 종양 침투 비만 세포, 수지상 세포 및 자연 살해 세포의 면적 분율 간의 Spearman 상관 관계(ρ), p < 0.038. 약어: TC, tumor center; IM, 내부 여백; OM, 외부 여백; PT, 종양 주위 영역; ROI, 관심 영역. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 4: TTR(Time to Recurrence), DFS(Disease-Free Survival) 및 OS(Overall Survival)와 관련된 임상 및 병리학 변수의 단변량 분석. 아니오", 여성, 성별 또는 "A"는 이분형 변수에 대한 참조 범주였습니다. 굵은 글씨체는 p < 0.05 수준에서 통계적 유의성을 나타냅니다. 약어: HR, 위험비; CI, 신뢰 구간; TTR, 재발 시간; DFS, 무병 생존. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 5: Cox 회귀분석을 사용하여 CD117+ 비만세포, CD1a+덴데라이트틱 세포 및 NKp46+자연살해세포와 개별 관심 영역별 TTR, DFS 및 OS의 면적 분율 연관성에 대한 단변량 분석(HCC 환자 67명). 면적 섹션당 면역 세포의 면적 분율(mm2)을 백분위수로 변환한 다음 낮은(0-25 백분위수) 대 높은(25-100 백분위수)로 분류했습니다. 위험비는 낮은 AF에 대한 1과 비교한 상대적 위험을 보여줍니다. 굵은 글씨체는 p < 0.05 수준에서 통계적 유의성을 나타냅니다. 약어: AF, 면적 분수; DFS, 무병 생존; OS, 전체 생존; HR, 위험 비율. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 6: 무병 생존 및 전체 생존과 관련된 개별 ROI당 CD117+ 비만 세포의 면적 분율(다변수 분석). Cox 회귀 분석을 사용하여 IM 및 PT 영역에서 CD117+ 비만 세포의 면적 분율과 DFS 및 OS의 연관성에 대한 다변수 분석(HCC 환자 67명). 면적 섹션당 면역 세포의 면적 분율(mm2)을 백분위수로 변환한 다음 낮은(0-24 백분위수) 대 높은(25-100 백분위수)로 분류했습니다. 위험비는 낮은 AF에 대한 1과 비교한 상대적 위험을 보여줍니다. 굵은 글씨체는 p > 0.05 수준에서 통계적 유의성을 나타냅니다. 약어: AF, 면적 분수; DFS, 무병 생존; OS, 전체 생존; HR, 위험 비율. 이 표를 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: ROI 생성을 위한 스크립트. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
TME에서 in situ 면역 조직을 모니터링함으로써 면역 종양학 분야는 새로운 암 예후 및 예측 바이오마커를 추가할 수 있습니다. 간세포암의 TME에서 적응면역세포의 역할에 대해 이전에 발표된 논문에서는 선별된 ROI에서 CD3+ 및 CD8+ T 세포와 CD20+ B 세포의 긍정적인 예후 연관성을 보여주었습니다2. 여기에서 이미지 분석 소프트웨어인 QuPath를 사용하여 간세포암의 여러 개별 영역에서 CD1a+ iDC, CD117+ 비만세포 및 NKp46+ NK 세포의 풍부도를 평가하고 이들의 예후 중요성을 평가했습니다. 일부 선천성 면역 세포의 불규칙한 모양으로 인해 정량화가 정확하지 않을 수 있습니다. 이것이 면역 양성 세포의 면적 분율을 평가하는 것이 최적의 선택이었던 이유입니다. 세 가지 세포 유형 중 CD117+ 비만 유의미한 예후 영향을 보였는데, IM 및 PT 영역에서 비만 세포의 AF가 높을수록 생존 기간이 길어졌습니다.
비만 세포는 모든 ROI에서 가장 풍부했으며, 종양 주위 영역과 내부 가장자리에서의 AF와 더 나은 생존율의 연관성은 비만 세포의 항종양 역할을 반영합니다. 비만 세포는 SCF 또는 CCL15와 같은 종양 세포 방출 화학 유인제에 의해 TME로 유인될 수 있다18. 비만세포는 종양세포에 대한 직접적인 세포독성을 통해 항종양 반응에 영향을 미칠 수 있으며27 종양 성장을 억제할 수 있는 전염증성 사이토카인을 분비함으로써 항종양 반응에 영향을 미칠 수 있다18. 증가된 비만세포 밀도는 간 이식 후 전립선암28, 위암27 및 간세포암의 재발을 예방하는 것으로 나타났다29. 245명의 HCC 환자로 구성된 대규모 코호트로 구성된 종합 조사에서 종양 샘플에서 더 중요한 비만 세포 침윤과 종양 절제 후 더 긴 생존 기간 사이에 긍정적인 상관관계가 있는 것으로 나타났습니다30. Rohr-Udilova 등은 주변 간세포 조직에서 비만 세포의 밀도가 더 높다는 유사한 결과를 보고했습니다. 그러나, 종양내 비만세포 밀도만이 더 낮은 재발률과 관련이 있었다29.
이 연구에서 비만세포는 IM 및 PT 간에서만 항종양 효과를 발휘했습니다. 서로 다른 종양의 TME는 면역 세포의 공간적 분포와 관련하여 이질적이다31. TME에서 면역 세포의 예후 중요성은 또한 공간 분포와 중요한 관련이 있습니다32,33. 종양 침습성 가장자리에 주석을 달기 위한 다양한 접근법이 제안되었는데, 이는 상이한 너비(4,34,35)의 전체 마진, 내부 및 외부 마진, 다시 상이한 너비(36,37)를 포함하며, PT 2,7의 유무에 관계없이. 국제 면역항암학 바이오마커 실무 그룹(International Immunooncology Biomarkers Working Group)의 재현 가능하고 표준화된 방법론에 따라 침습성 마진을 악성 세포 둥지를 숙주 조직에서 분리하는 경계 중심의 1mm 영역으로 정의하고 중심 종양을 나머지 종양 영역38로 표시했습니다. 이전 연구에서 내부 및 외부 침습적 마진2의 결과에서 상당한 차이가 강조된 이후 해당 영역(각 너비 500μm)을 별도로 분석했습니다. 종양 주위 영역의 공간 면역 프로파일링은 뚜렷한 예측 중요도(39,40)를 가지므로 PT 영역도 포함되었습니다.
TC, 종양 가장자리 및 PT 간의 분석은 각 영역에서 선택된 시야를 평가하는 옵션과 달리 WSI에서 수행되었습니다. 전체 슬라이드 이미징은 잠재적으로 풍부한 데이터 소스이다41. 서브샘플링을 수행하지 않았기 때문에 "실제 기대값"은 시간 효율적인 방식으로 얻어졌다10. 또한, 이 시스템은 블록당 1개 또는 2개의 슬라이드만 분석했기 때문에 합리적이었으며, 이를 통해 패치 선택42의 편향을 피할 수 있었습니다. 느린 워크플로우는 이전 연구에서 면역 세포의 정량화를 위해 스테리학을 적용했을 때 우리에게 도전이 되었습니다2.
IHC는 간 조직에서 CD1a+ iDC, NKp46+NK 및 CD117+ 비만 세포 발현의 직접 국소화, 분포 정량화 및 코호트의 하위 분류를 가능하게 합니다. IHC 분석은 이 연구에 가용화된 조직에서의 생화학적 분석에 비해 중요한 이점을 제공하며, 이는 소수의 바이오마커 양성 세포만 존재하고43 조직병리학적 특징에 대한 면역 세포의 지역적 반응을 반영하지 않을 때 위음성 결과를 초래할 수 있습니다44. IHC는 또한 H&E 염색 절면의 분석보다 선호됩니다. 대장암 및 소세포 폐암에 대한 연구는 H&E 염색 절편의 평가가 강력하고 정량적인 종양 면역 바이오마커를 제공할 수 있음을 보여주었습니다 45,46,47; 그러나 특정 하위 유형의 실제 존재를 반영하는 제한 사항은 여전히 존재합니다.
높은 품질의 조직 절편 및 IHC 염색은 정확한 평가를 위한 전제 조건입니다. 높은 배경, 가장자리 아티팩트 및 특이적이지만 바람직하지 않은 염색(예: 항-CD4 항체로 간 정현파 공간의 내피 세포 염색)은 모든 결과를 손상시킬 수 있습니다. CD56 항체는 이번 연구에서 자연살해세포를 검출하기 위한 첫 번째 선택이었지만 미성숙 담관에서 CD56이 발현되어 NKp46 항체로 대체되었습니다. FFPE 블록에서 특이성, 견고한 염색 패턴 및 안정성 측면에서 가장 적합한 마커를 선택하는 것은 고려해야 할 또 다른 문제입니다. 예를 들어, 트립타제 발현이 강하게 하향조절될 수 있기 때문에, CD117을 발현하지 않는 세포는 비만세포(mast cell)가 아니기 때문에 트립타제 대신에 CD117이 선택되었다(48).
QuPath는 외부에서 ROI의 경계를 매끄럽게 하고 ROI의 일부가 되어서는 안 되는 아티팩트, 대형 혈관, 괴사 조직, 비특이적 염색, 배경 및 적혈구의 응집을 제거할 수 있는 옵션을 제공합니다. 작업자는 ROI를 선택하고 해당 아티팩트에 주석을 추가하는 동안 "Alt" 키를 눌러야 합니다. 이 연구에서 비특이적 염색은 결과에 영향을 미치지 않도록 정량화 전에 인공물로 제거되었습니다. 체계적으로 낮은 특이 염색 또는 높은 백그라운드의 경우, 가능한 편향을 피하기 위해 프로토콜을 검토해야 합니다. 임계값은 약한 특정 양성 염색이 있는 각 사례에 대해 조정되었습니다. 일상적인 사용 전에 프로토콜을 최적화해야 합니다. 모든 분석은 블라인드 방식으로 수행되어야 하며 이상적으로는 임계값의 정확성을 교차 점검할 가치가 있습니다.
QuPath는 디지털 병리학 및 전체 슬라이드 이미지 분석9을 위한 사용자 친화적이고 직관적이며 확장 가능한 오픈 소스 솔루션으로, 이전에 HCC49 및 다양한 암50,51의 이미지 분석을 위해 테스트되었습니다. WSI용 소프트웨어의 쉬운 작동과 관심 영역(예: 종양 또는 종양 주변 영역)의 주석 옵션이 주요 장점입니다.
기존, 수정 또는 새로 생성된 스크립트를 적용하면 분석 속도가 크게 빨라질 수 있습니다. ROI에 대한 주석을 생성하는 스크립트가 적용되어 수동 묘사 대신 빠르고 정확한 지역 세분화가 가능해졌습니다. 스크립트는 고정되어 있지 않으며 조직 경계가 충분하지 않은 영역에 대해 500μm 미만의 여백으로 쉽게 사용자 지정할 수 있습니다. IM, OM 또는 PT 영역의 너비는 Automate > Show 스크립트 편집기 를 통해 사용자 정의할 수 > 30 Double Expand Margin Microns = 500 μm 라인을 실행 > 사용 가능한 너비로 조정할 수 있습니다. ROI를 추가하거나 제거할 수도 있습니다. 일반적으로 소프트웨어의 워크플로는 고정되어 있지 않으며 작업자는 이를 자유롭게 개발하고 수정할 수 있습니다.
현재 CellProfiler, ImageJ, Fiji, Microscopy Image Browser 등을 포함하여 병리학적 이미지 분석을 위한 다양한 소프트웨어 도구를 사용할 수 있습니다. 그러나 QuPath는 오픈 소스 아키텍처, 사용자 친화적인 인터페이스, 알고리즘 사용자 지정 기능 및 고급 머신 러닝 도구 통합과 같은 일련의 이점을 통해 차별화됩니다. 이러한 기능을 총체적으로 통해 이 이미지 분석 소프트웨어는 디지털 병리학 영역에서 암 조직의 미묘한 분석을 위한 강력한 선택으로 자리매김합니다.
QuPath는 유방암에서 Ki67 발현을 검출하는 데 있어 상용 소프트웨어인 HALO(IndicaLab) 및 QuantCenter(3DHistech)에 비해 변동성이 가장 낮았다52. 이 소프트웨어는 또한 재현 가능한 일괄 처리 방식으로 모든 프로젝트 이미지에 대한 스크립트를 실행하도록 지시할 수 있는 잠재력을 가지고 있습니다.
QuPath를 사용하면 순서(반정량적) 데이터 대신 정량적 연속 데이터를 생성할 수 있었습니다. 시각적 채점에 의해 얻어진 순서 데이터는 해석의 주관성, 관찰자 간 변동성, 낮은 재현성으로 인해 문제가 많다53. 병리학자가 주석의 정확성을 확인한 경우 광범위한 조직 병리학 배경 및 프로그래밍 전문 지식이 없는 개인도 자동 분석을 수행할 수 있다는 사실에 의해 현재 분석의 신속성이 향상되었습니다. 두경부 종양에 대한 연구는 CD57+ 종양 침윤 림프구50의 예후 가치에 대한 빠르고 재현성 있는 평가에 대한 QuPath의 적합성을 입증했습니다. 이 연구는 또한 인간 관찰자와 QuPath 사이에 상당한 일치가 있음을 보여주었습니다. QuPath를 사용한 수동 관찰과 평가 간의 높은 일치성은 구강암54 및 유방암55에서도 나타났습니다. 앞서 언급한 논문에서는 QuPath와 2개의 상용 소프트웨어 제품(Definiens Tissue Studio 및 inForm)을 사용하여 5개의 임상 유방암 바이오마커에 대한 IHC 정량화를 병리학자의 수동 채점과 비교했습니다. 이 연구를 통해 QuPath는 지속적으로 최고의 성능과 설정 및 적용에 가장 적은 시간을 보였으며, 따라서 시각적 모니터링 및 상용 소프트웨어에 대한 훌륭한 미래 대안임을 보여주었습니다. 생물 의학 이미지 분석을 위한 가장 잘 알려진 오픈 소스 소프트웨어인 ImageJ와 비교해도 QuPath는 대형 WSI56을 처리하는 데 탁월합니다.
QuPath를 위한 공개 온라인 포럼에서는 사용자가 디지털 병리학 방법에 대한 질문을 게시하고 이미지 분석(https://forum.image.sc/tag/qupath) 도구 개발을 지원하기 위한 적극적이고 참여적인 커뮤니티를 제공합니다.
이 연구에 사용된 코호트는 독특했으며 간 절제술이 가능한 환자만 포함했기 때문에 간세포암이 있는 일반 인구를 대표하지 않았습니다. 일반적으로 간세포암 환자 중 소수(20%-30%)가 간 절제술을 받을 수 있다57. 이는 또한 간경변증을 확인한 환자가 더 적은 이유일 수 있는데, 이는 간암의 병인을 가진 환자들이 아마도 더 높은 Child-Pugh 점수를 가지고 있었고 절제 가능한 것으로 간주되지 않았기 때문일 수 있습니다. 코호트는 또한 다소 작았고, 대부분의 환자들은 질병의 초기 TNM 단계를 가지고 있었다. 따라서 결과를 다른 모집단으로 외삽하는 것은 주의해서 이루어져야 합니다.
이번 연구에서는 수지상세포와 같은 불규칙한 형태의 면역세포를 정확하게 계수하는 것이 어려울 수 있기 때문에 AF 평가를 통해 간세포암 미세환경에서 선천면역세포의 풍부도를 추정했습니다. 반면에, 면역 세포의 면적 분율은 일반적으로 그들의 밀도와 강한 상관관계가 있다58. 면역페록시다제 라벨링은 항원, 면적 분율 또는 이를 발현하는 세포의 밀도의 국소화를 위한 데이터를 제공하지만, 염색 강도는 항원의 양과 비선형적으로 관련되어 있습니다. 따라서 특정 단백질의 발현 수준을 정량적으로 평가하기 위해 IHC를 사용해서는 안 됩니다. 이 방법의 정확도는 또한 단면 두께, 이미지 품질 및 픽셀 분류를 위해 선택한 해상도의 균일성에 따라 달라집니다. 이미지 분석의 모든 결과는 주의해서 검증하고 처리해야 합니다. 단일 마커는 면역 세포의 복잡성을 완전히 포착할 수 없기 때문에 간세포암의 면역 TME에 대한 신뢰할 수 있는 그림을 얻으려면 더 많은 IHC 마커와 다중 염색이 필요합니다. 면역 세포의 표현형에 단일 IHC 마커만 사용하는 것은 근사치를 나타내며 결과를 주의해서 해석해야 합니다.
QuPath 이미지 분석 소프트웨어를 사용하여 IHC 염색 슬라이드를 평가함으로써 간세포암 환자의 종양 및 종양 주위 종양에서 국소 iDC, 비만 세포 및 NK의 분포 및 면적 분율을 평가한 다음 예후와의 관계를 분석할 수 있었습니다. 간세포암의 내측 가장자리와 종양 주위 간에 풍부한 비만세포는 DFS 및 OS가 긴 것과 관련이 있으며, 이는 선천면역세포의 항종양 효과를 강조합니다. QuPath의 분석 워크플로우는 사용자 친화적이고, 빠르고, 사용하기 쉬우며, 유용한 기본 설정과 간편한 데이터 내보내기를 제공합니다. 이 소프트웨어는 디지털 병리학의 재현성, 일관성 및 정확성에 대한 요구를 충족할 수 있는 디지털 이미지 분석을 위한 유망한 플랫폼으로 승인되었습니다.
공개
공개해야 할 재정적 이해 상충은 없습니다.
감사의 말
전체 슬라이드 스캐닝에 대한 Mgr. Ondřej Šebesta(Vinicna Microscopy Core Facility, Faculty of Science)와 이 연구를 위한 계산 리소스를 제공한 "e-Infrastruktura CZ"(e-INFRA LM2018140) 프로젝트에 감사드립니다. 기술자 Jana Dosoudilova와 Jan Javurek은 탁월한 기술 지원으로 인정받고 있습니다. 이 연구는 유럽연합(EU)의 Horizon 2020 연구 및 혁신 프로그램(보조금 N°856620)과 체코 보건부(Ministry of Health, Grant AZV NU21-03-00506) 및 Cooperatio Program(외과 분야)의 자금 지원을 받았습니다. Vinicna 현미경 핵심 시설은 체코-바이오이미징(Czech-BioImaging)의 대규모 RI 프로젝트 LM2023050가 공동 출자합니다.
자료
| Name | Company | Catalog Number | Comments |
| Anti-CD1a | Leica Biosystems | PA0235 | Identifier- immature dendritic cells; RTU, Protocol F, ER2/20 min |
| Anti-CD117 | Leica Biosystems | PA0007 | Identifier- mast cells; RTU, Protocol F, ER2/20 min |
| BOND Plus Microscope Slides | Leica Biosystems, Germany | S21.2113.A | |
| BOND RXm | Leica Biosystems | 49.1501 | Fully Automated IHC Stainer |
| Bond Aspirating Probe Cleaning Kit | Leica Biosystems | CS9100 | |
| Bond Dewax Solution | Leica Biosystems | AR9222 | |
| Bond Polymer Refine Detection Kit | Leica Biosystems | DS9800 | |
| BondTM Epitope Retrieval 2 | Leica Biosystems | AR9640 | |
| BondTM Primary Antibody Diluent | Leica Biosystems | AR9352 | |
| BondTM Wash Solution 10X Concentrate | Leica Biosystems | AR9590 | |
| Computer Specifications: Intel(R) Core(TM) i5-10500 CPU @ 3.10GHz 3.10 GHz Installed RAM: 128 G | Intel | A 64-bit operating system that has Windows 7. Any computer with Java-based operating system and Excel available | |
| Coverslips | Leica Biosystems, Germany | 14071135636 | |
| CV Mount | Leica Biosystems, Germany | 14046430011 | |
| CV5030 Fully Automated Glass Coverslipper | Leica Biosystems | 149CVTS5025 | |
| Drying Oven UN30 | Memmert GmbH | UN30 | |
| GraphPad Prism 9.0 | GraphPad Software LLC | Version 12 | |
| Human NKp46/NCR1 Antibody, Monoclonal Mouse IgG2B Clone # 195314 | R&D Systems, Inc., United States | MAB1850 | Identifier- natural killer cells; Dilution 1:150, Protocol F + BLOK, ER2/20 min |
| Leica HI1210 - Water Bath | Leica Biosystems | 14041521466 | |
| Protein Block | Agilent Dako, United States | X0909 | |
| QuPath 0.3.2 or higher versions | version (QuPath v.0.3.2) | ||
| RM2235 Rotary Microtome | Leica Biosystems | 149AUTO00C1 | |
| ST5020 Multistainer Slide Stainer | Leica Biosystems | DEV-ST5010-CV5030 | |
| Statistica | StatSoft Inc. | version 7 | |
| Zeiss Axio Scan.Z1 | ScienceServices GmbH, Germany | 430038-9000-000 | Slide scanner |
참고문헌
- Du, M., Yin, Y. L., Xiao, L., Cai, Y. M., Ji, Y. Evaluating tumor-infiltrating lymphocytes in hepatocellular carcinoma using hematoxylin and eosin-stained tumor sections. World J Clin Cases. 10 (3), 856-869 (2022).
- Trailin, A., et al. T-and B-cells in the inner invasive margin of hepatocellular carcinoma after resection associate with favorable prognosis. Cancers (Basel). 14 (3), 604(2022).
- Xiao, N., et al. CD74+ macrophages are associated with favorable prognosis and immune contexture in hepatocellular carcinoma. Cancer Immunol Immunother. 71 (1), 57-69 (2022).
- Galon, J., et al. Type, density, and location of immune cells within human colorectal tumors predict clinical outcome. Science. 313 (5795), 1960-1964 (2006).
- Roxburgh, C. S. D., Salmond, J. M., Horgan, P. G., Oien, K. A., McMillan, D. C. Tumour inflammatory infiltrate predicts survival following curative resection for node-negative colorectal cancer. Eur J Cancer. 45 (12), 2138-2145 (2009).
- Klintrup, K., et al. Inflammation and prognosis in colorectal cancer. Eur J Cancer. 41 (17), 2645-2654 (2005).
- Kather, J. N., et al. Topography of cancer-associated immune cells in human solid tumors. Elife. 7, e36967(2018).
- Cho, J. Basic immunohistochemistry for lymphoma diagnosis. Blood Res. 57 (S1), 55-61 (2022).
- Bankhead, P., et al. QuPath: Open source software for digital pathology image analysis. Sci Rep. 7 (1), 16878(2017).
- Kolinko, Y., et al. Using virtual microscopy for the development of sampling strategies in quantitative histology and design-based stereology. Anat Histol Embryol. 51 (1), 3-22 (2022).
- Rodrigues, A., et al. Computer-assisted tumor grading, validation of PD-L1 scoring, and quantification of CD8-positive immune cell density in urothelial carcinoma, a visual guide for pathologists using QuPath. Surg Exp Pathol. 5, 12(2022).
- Hein, A. L., et al. QuPath digital immunohistochemical analysis of placental tissue. J Pathol Inform. 12, 40(2021).
- Lichterman, J. N., Reddy, S. M. Mast cells: A new frontier for cancer immunotherapy. Cells. 10 (6), 1270(2021).
- Veglia, F., Gabrilovich, D. I. Dendritic cells in cancer: the role revisited. Curr Opin Immunol. 45, 43-51 (2017).
- Wu, S. -Y., Fu, T., Jiang, Y. -Z., Shao, Z. -M. Natural killer cells in cancer biology and therapy. Mol Cancer. 19 (1), 120(2020).
- Kai, K., et al. Immunohistochemical analysis of the aggregation of CD1a-positive dendritic cells in resected specimens and its association with surgical outcomes for patients with gallbladder cancer. Transl Oncol. 14 (1), 100923(2021).
- Minesaki, A., Kai, K., Kuratomi, Y., Aishima, S. Infiltration of CD1a-positive dendritic cells in advanced laryngeal cancer correlates with unfavorable outcomes post-laryngectomy. BMC Cancer. 21 (1), 973(2021).
- Komi, D. E. A., Redegeld, F. A. Role of mast cells in shaping the tumor microenvironment. Clin Rev Allergy Immunol. 58 (3), 313-325 (2020).
- Maltby, S., Khazaie, K., McNagny, K. M. Mast cells in tumor growth: Angiogenesis, tissue remodelling and immune-modulation. Biochim Biophys Acta. 1796 (1), 19-26 (2009).
- Gooch, J. L., Lee, A. V., Yee, D. Interleukin 4 inhibits growth and induces apoptosis in human breast cancer cells. Cancer Res. 58 (18), 4199-4205 (1998).
- Brockmeyer, P., et al. High mast cell density indicates a longer overall survival in oral squamous cell carcinoma. Sci Rep. 7 (1), 14677(2017).
- Lee, H. A., et al. Natural killer cell activity is a risk factor for the recurrence risk after curative treatment of hepatocellular carcinoma. BMC Gastroenterol. 21 (1), 258(2021).
- Shimasaki, N., Jain, A., Campana, D. NK cells for cancer immunotherapy. Nat Rev Drug Discov. 19 (3), 200-218 (2020).
- Barrow, A. D., Martin, C. J., Colonna, M. The natural cytotoxicity receptors in health and disease. Front Immunol. 10, 909(2019).
- Guan, X., et al. Tumor-associated NK cells facilitate tumor growth via NKp46 in immunocompetent murine hepatocellular carcinoma. Immunol Lett. 258, 8-19 (2023).
- Ali, E., et al. Prognostic role of macrophages and mast cells in the microenvironment of hepatocellular carcinoma after resection. BMC Cancer. 24 (1), 142(2024).
- Lin, C., et al. Tryptase expression as a prognostic marker in patients with resected gastric cancer. Br J Surg. 104 (8), 1037-1044 (2017).
- Hempel, H. A., et al. Low intratumoral mast cells are associated with a higher risk of prostate cancer recurrence. Prostate. 77 (4), 412-424 (2017).
- Rohr-Udilova, N., et al. Morphometric analysis of mast cells in tumor predicts recurrence of hepatocellular carcinoma after liver transplantation. Hepatol Commun. 5 (11), 1939-1952 (2021).
- Lin, S. Z., et al. Prediction of recurrence and survival in hepatocellular carcinoma based on two cox models mainly determined by FoxP3+ regulatory T cells. Cancer Prev Res. 6 (6), 594-602 (2013).
- Jia, Q., Wang, A., Yuan, Y., Zhu, B., Long, H. Heterogeneity of the tumor immune microenvironment and its clinical relevance. Exp Hematol Oncol. 11 (1), 24(2022).
- Pyo, J. -S., Son, B. K., Lee, H. Y., Oh, I. H., Chung, K. H. Prognostic implications of intratumoral and peritumoral infiltrating lymphocytes in pancreatic ductal adenocarcinoma. Curr Oncol. 28 (6), 4367-4376 (2021).
- Yusa, T., et al. Survival impact of immune cells infiltrating peritumoral area of hepatocellular carcinoma. Cancer Sci. 113 (12), 4048-4058 (2022).
- Halama, N., et al. Localization and density of immune cells in the invasive margin of human colorectal cancer liver metastases are prognostic for response to chemotherapy. Cancer Res. 71 (17), 5670-5677 (2011).
- Zwing, N., et al. Analysis of spatial organization of suppressive myeloid cells and effector T cells in colorectal cancer-A potential tool for discovering prognostic biomarkers in clinical research. Front Immunol. 11, 550250(2020).
- Soeratram, T. T. D., et al. Prognostic value of T-cell density in the tumor center and outer margins in gastric cancer. Mod Pathol. 36 (9), 100218(2023).
- Gonzàlez-Farré, M., et al. Characterization and spatial distribution of the immune cell infiltrate in triple-negative breast cancer: a novel classification based on plasma cells and CD8+ T cells. Hum Pathol. 139, 91-105 (2023).
- Hendry, S., et al. Assessing tumor-infiltrating lymphocytes in solid tumors: A practical review for pathologists and proposal for a standardized method from the International Immunooncology Biomarkers Working Group: Part 1: Assessing the host immune response, TILs in invasive breast carcinoma and ductal carcinoma in situ, metastatic tumor deposits and areas for further research. Adv Anat Pathol. 24 (5), 235-251 (2017).
- Brück, O., et al. Spatial immunoprofiling of the intratumoral and peritumoral tissue of renal cell carcinoma patients. Mod Pathol. 34 (12), 2229-2241 (2021).
- Knebel, M., et al. Prognostic impact of intra- and peritumoral immune cell subpopulations in head and neck squamous cell carcinomas - comprehensive analysis of the TCGA-HNSC cohort and immunohistochemical validation on 101 patients. Front Immunol. 14, 1172768(2023).
- Lee, S., et al. Interactive classification of whole-slide imaging data for cancer researchers. Cancer Res. 81 (4), 1171-1177 (2021).
- Ciga, O., et al. Overcoming the limitations of patch-based learning to detect cancer in whole slide images. Sci Rep. 11 (1), 8894(2021).
- Rizzardi, A. E., et al. Quantitative comparison of immunohistochemical staining measured by digital image analysis versus pathologist visual scoring. Diagn Pathol. 7, 42(2012).
- Masuda, S., Nakanishi, Y. Application of immunohistochemistry in clinical practices as a standardized assay for breast cancer. Acta Histochem Cytochem. 56 (1), 1-8 (2023).
- Matsutani, S., et al. Tumor-infiltrating immune cells in H&E-stained sections of colorectal cancer tissue as a reasonable immunological biomarker. Anticancer Res. 38 (12), 6721-6727 (2018).
- Väyrynen, J. P., et al. Prognostic significance of immune cell populations identified by machine learning in colorectal cancer using routine hematoxylin and eosin-stained sections. Clin Cancer Res. 26 (16), 4326-4338 (2020).
- Zhou, G., et al. Clinical significance of tumor-infiltrating lymphocytes investigated using routine H&E slides in small cell lung cancer. Radiat Oncol. 17 (1), 127(2022).
- Horny, H. -P., Sotlar, K., Valent, P. Mastocytosis. Immunol Allergy Clin North Am. 34 (2), 315-321 (2014).
- Mi, H., Ho, W. J., Yarchoan, M., Popel, A. S. Multi-scale spatial analysis of the tumor microenvironment reveals features of cabozantinib and nivolumab efficacy in hepatocellular carcinoma. Front Immunol. 13, 892250(2022).
- de Ruiter, E. J., et al. Assessing the prognostic value of tumor-infiltrating CD57+ cells in advanced stage head and neck cancer using QuPath digital image analysis. Virchows Archiv. 481 (2), 223-231 (2022).
- Fanucci, K. A., et al. Image analysis-based tumor infiltrating lymphocytes measurement predicts breast cancer pathologic complete response in SWOG S0800 neoadjuvant chemotherapy trial. NPJ Breast Cancer. 9 (1), 38(2023).
- Acs, B., et al. Ki67 reproducibility using digital image analysis: an inter-platform and inter-operator study. Lab Invest. 99 (1), 107-117 (2019).
- Sobottka, B., et al. Establishing standardized immune phenotyping of metastatic melanoma by digital pathology. Lab Invest. 101 (12), 1561-1570 (2021).
- Moratin, J., et al. Digital pathology scoring of immunohistochemical staining reliably identifies prognostic markers and anatomical associations in a large cohort of oral cancers. Front Oncol. 11, 712944(2021).
- Bankhead, P., et al. Integrated tumor identification and automated scoring minimizes pathologist involvement and provides new insights to key biomarkers in breast cancer. Lab Invest. 98 (1), 15-26 (2018).
- Humphries, M. P., Maxwell, P., Salto-Tellez, M. QuPath: The global impact of an open source digital pathology system. Comput Struct Biotechnol J. 19, 852-859 (2021).
- Koh, J. H., et al. Liver resection versus liver transplantation for hepatocellular carcinoma within Milan criteria: a meta-analysis of 18,421 patients. Hepatobiliary Surg Nutr. 11 (1), 78-93 (2022).
- Eriksen, A. C., et al. Computer-assisted stereology and automated image analysis for quantification of tumor infiltrating lymphocytes in colon cancer. Diagn Pathol. 12 (1), 65(2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유