Method Article
Techniques de laboratoire aseptiques: Méthodes de placage
Dans cet article
Résumé
Lorsque vous travaillez avec les médias et des réactifs utilisés pour les micro-organismes de la culture, une technique aseptique doit être pratiqué afin d'assurer la contamination est réduit au minimum. Une variété de méthodes de placage sont couramment utilisés pour isoler, de propager ou de bactéries et de phages énumérer, toutes les procédures qui incorporent qui maintiennent la stérilité des matériaux expérimentaux.
Résumé
Micro-organismes sont présents sur toutes les surfaces inanimées la création de sources de contamination possible omniprésents dans le laboratoire. Succès expérimentale repose sur la capacité d'un scientifique de stériliser les surfaces de travail et de l'équipement ainsi que d'éviter tout contact des instruments stériles et des solutions avec des surfaces non stériles. Nous présentons ici les étapes à suivre pour les méthodes de placage plusieurs couramment utilisés dans le laboratoire afin d'isoler, de propager, ou d'énumérer les micro-organismes comme les bactéries et de phages. Tous les cinq méthodes incorporer une technique aseptique, ou des procédures qui assurent la stérilité des matériaux expérimentaux. Les procédures décrites comprennent (1) série-placage des cultures bactériennes pour isoler des colonies isolées, (2) versez-placage et (3) propagation de placage à énumérer viables colonies bactériennes, (4) superpositions agar mou pour isoler des phages et plaques énumérer, et ( 5) réplique de placage pour transférer des cellules d'une plaque à l'autre dans une configuration spatiale identique. Ces procédures peuvent être effectuées à l'instant til paillasse de laboratoire, à condition qu'ils impliquent des souches non pathogènes de micro-organismes (niveau de biosécurité 1, BSL-1). Si vous travaillez avec BSL-2 organismes, ces manipulations doivent avoir lieu dans une enceinte de biosécurité. Consultez l'édition la plus récente de la prévention des risques dans les laboratoires microbiologiques et biomédicaux (BMBL) ainsi que les fiches signalétiques (FS) pour les substances infectieuses pour déterminer la classification de risque biologique, ainsi que les consignes de sécurité et des installations de confinement requis pour le micro-organisme en question. Les souches bactériennes et les stocks de phages peuvent être obtenues auprès des enquêteurs de recherche, des entreprises, et des collections gérées par des organisations particulières telles que l'American Type Culture Collection (ATCC). Il est recommandé que les souches non pathogènes être utilisé lors de l'apprentissage des méthodes de placage différents. En suivant les procédures décrites dans ce protocole, les élèves devraient être en mesure de:
- Effectuer les procédures de placage sans contaminatimédias ng.
- Isoler simples colonies bactériennes par la méthode strie de placage.
- Utiliser des méthodes versez-placage et la propagation de placage pour déterminer la concentration de bactéries.
- Effectuer des superpositions agar mou lorsque vous travaillez avec le phage.
- Transférer les cellules bactériennes d'une plaque à l'autre en utilisant la procédure réplique-plaquage.
- Compte tenu d'une tâche expérimentale, sélectionnez la méthode de placage appropriée.
Protocole
1. Préparer un espace de travail sécuritaire et stérile
- Être familier avec toutes les règles de laboratoire et les précautions de sécurité à prendre lorsqu'on travaille avec des micro-organismes. Indépendamment de la classification des risques biologiques, tous les matériaux entrant en contact avec des micro-organismes sont considérés comme des déchets infectieux et doivent être décontaminés avant d'être éliminés. Suivez les consignes de sécurité conformes à celles fournies par la santé environnementale institutionnelle et les ministères de sécurité, la mise en place de poubelles appropriées pour l'évacuation immédiate et appropriée de matériaux potentiellement contaminés (risques biologiques).
- Stériliser tous les instruments, de solutions, et les médias avant de les utiliser pour les procédures de placage.
- Dégagez tous les matériaux qui encombrent votre espace de travail sur le banc de laboratoire.
- Nettoyer la zone de travail avec un désinfectant pour minimiser la contamination possible.
- Mettre en place un bec Bunsen et de travailler lentement, soigneusement, et délibérément dans la zone champ stérile créée par le updrapi de la flamme.
- Si vous travaillez avec BSL-2 organismes, configurer votre espace de travail dans une armoire prévention des risques biotechnologiques. Un bec Bunsen ne peuvent pas être utilisés à l'intérieur du cabinet parce que la chaleur de la flamme perturbe l'écoulement de l'air indispensable à sa fonctionnalité.
- Disposer tous les approvisionnements nécessaires à la procédure sur le banc de laboratoire à proximité du champ stérile. Assurez-vous que tous les matériaux sont correctement étiquetés. L'organisation de la zone de travail afin de maximiser l'efficacité du travail et éviter les mouvements inutiles permettra de minimiser le temps d'exposition de matériel d'expérimentation aux contaminants aéroportés.
- Placez le bec Bunsen à votre droite sur le banc.
- Des plaques d'agar Placer ou des boîtes de Pétri à votre gauche.
- Disposer cultures cellulaires, des tubes, des flacons et des bouteilles dans le centre de la banquette.
- Desserrer les bouchons de tubes, flacons et bouteilles de sorte qu'ils peuvent être facilement ouverts avec une seule main lors des manipulations ultérieures.
- Se laver les mains soigneusement avec du savon antiseptique et chaleureusel'eau avant de manipuler des micro-organismes.
2. Procédure Plate Streak: Isolement des colonies bactériennes en utilisant la méthode des quadrants
La procédure strie-plaque est conçue pour isoler les cultures pures de bactéries, ou des colonies, à partir de populations mixtes par séparation mécanique simple. Des colonies isolées sont constitués de millions de cellules en croissance dans un cluster sur ou dans une plaque de gélose (Figure 1). Une colonie, à la différence d'une seule cellule, est visible à l'œil nu. En théorie, toutes les cellules dans une colonie proviennent d'une seule bactérie initialement déposée sur la plaque et sont donc considéré comme un clone, ou amas de cellules génétiquement identiques.
- Les bactéries existent dans une variété de formes et de tailles. Par exemple, individuels des cellules d 'Escherichia coli sont en forme de tige avec une longueur moyenne de 2 um et une largeur de 0,5 um tandis que les cellules de Streptococcus sont sphériques avec un diamètre moyen de 1 pm. Certaines bactéries (such comme E. coli) existent en tant que cellules individuelles tandis que d'autres forment des motifs distincts de liaison. Streptococcus, par exemple, pousser en paires ou des chaînes de forme ou de groupes de cellules. Il est généralement admis que d'une seule colonie provient d'une seule cellule subir une fission binaire, mais cette hypothèse n'est pas vrai pour les bactéries qui existent naturellement sous forme de paires, les chaînes, ou des groupes ou qui divisent par d'autres mécanismes. Alternativement, si les bactéries sont trop plaqué, puis se chevauchent de cellules peuvent se produire et augmenter la probabilité de deux ou plusieurs bactéries à l'origine de ce qui semble être une seule colonie. Pour éviter ces complications lors de la description ou l'énumération des cultures bactériennes en croissance sur un milieu solide, les colonies sont appelées unités formant colonies (UFC).
La procédure strie-plaque, un mélange de cellules est étalée sur la surface d'un semi-solide, à base de gélose milieu nutritif dans une boîte de Pétri telle que de moins en moins bactériennecellules sont déposées à des points éloignés les uns des sur la surface du support et, après incubation, développer en colonies. La méthode des quadrants pour isoler des colonies isolées à partir d'un mélange de cellules seront décrites ici.
- Streak-placage peut être accompli avec un certain nombre d'instruments différents (figure 2). Une boucle en métal peuvent être réutilisés plusieurs fois et est utilisé pour laisser de traces de placage des souches de laboratoire de routine. Jetables boucles en plastique sont disponibles dans le commerce et sont utilisés plus fréquemment lorsque vous travaillez avec BSL-2 souches dans une armoire prévention des risques biotechnologiques. De nombreux chercheurs préfèrent utiliser jetable, pré-stérilisés bâtons en bois ou des cure-dents plats pour strie de placage. Ce sont une alternative peu coûteuse aux boucles en plastique jetables et peut être particulièrement utile lorsque vous travaillez avec un échantillon de l'environnement tels que le sol qui contient probablement les bactéries sporulées.
- Jetables pré-stérilisés des bâtons et des cure-dents ne doivent pas être brûlé avec le jeu. bec Bunsens la prévention aérosolisation inutile de bactéries sporulées et la contamination croisée des surfaces de laboratoire ou les boîtes de gélose avec des spores.
- Étiquette sur le pourtour du fond (pas le couvercle) d'une plaque de gélose avec au moins votre nom, la date, le type de milieu de croissance, et le type d'organisme à être plaqué sur le support.
- Les plaques doivent être complètement sec sans condensation sur le couvercle et préchauffé à la température ambiante avant de série de placage. Si les plaques sont stockées à 4 ° C, retirez-les plusieurs heures ou même la veille. Répartissez-les dans de petits empilements décalés de pas plus de 2-3 plaques et les laisser sécher.
- L'échantillon à partir de laquelle la strie-plaque sera inoculé peut être soit une suspension de cellules dans le bouillon ou une colonie existante d'une autre plaque de gélose. Pour commencer, il est recommandé qu'un seul échantillon être utilisée pour inoculer une seule plaque. Pour gagner du temps et des matériaux, une seule plaque peut être utilisée pour de multiples samples mais seulement une fois que vous devenez compétent dans les rayures d'un échantillon sur une plaque.
- Pour le premier quadrant, l'échantillon est ramassé et s'étend sur environ un quart de la surface du support en utilisant une méthode rapide, lisse, de va-et-vient (Groupe A de la figure 3). Soulevez la moitié inférieure d'une plaque retournée sur le banc. Déplacer la boucle, bâton, ou cure-dent d'avant en arrière à plusieurs reprises à travers la surface de gélose de la jante au centre de la plaque.
- Si l'inoculum est une suspension de cellules, d'obtenir une anse avec une boucle en métal ou 5 à 10 pi avec une micropipette. Assurez-vous de tourbillon ou vortex de la suspension cellulaire avant de retirer une aliquote pour le placage. Les conditions d'asepsie tout en effectuant de cette étape, flammes non seulement la boucle, mais aussi le bord de la bouteille ou le tube avant et après avoir enlevé l'inoculum. En outre, ne pas toucher les parois du tube ou de bouteille avec la boucle ou le canon de la micropipette. Si l'inoculum de pipetage, distribution de la suspension cellulaire sur le appropRIATE place sur la plaque, puis utiliser une boucle, un bâton ou cure-dent pour l'étaler sur le premier quadrant de la plaque.
- Si l'inoculum est une colonie d'un autre plateau, toucher doucement la colonie avec la boucle, un bâton ou cure-dent, puis étalez-la sur le premier quadrant de la plaque. Assurez-vous de la boucle métallique est refroidie avant de toucher la colonie. Afin de garantir la boucle n'est pas trop chaud, le toucher légèrement sur la plaque de gélose dans une zone désignée où aucune rayures se produira. Seuls quelques cellules sont nécessaires de la colonie, et non pas toute la colonie.
- Si vous utilisez une boucle en métal, de la flamme à l'aide d'un bec Bunsen, avant d'obtenir l'inoculum de la plaque (Groupe B de la figure 3). Démarrer sur les 3-4 pouces de la boucle, avec le fil à la pointe du cône bleu, partie la plus chaude de la flamme. Le métal doit devenir rouge chaud. Déplacez le fil de sorte que la flamme s'approche de la boucle. Cette manipulation empêche aérosolisation de bactéries laissées sur la boucle de l'usage précédent.
- Flaming tue l'bactéries (mais pas les spores) et des courants de convection de l'air chauffé de prévenir d'autres contaminants dans l'air de s'installer sur le fil métallique lors des manipulations ultérieures.
- Si vous utilisez un cure-dent stérile plate, maintenez l'extrémité étroite doucement entre le pouce et l'annulaire à un angle de 10 à 20 ° au milieu, et d'utiliser l'extrémité la plus large à la strie des quadrants. Si vous utilisez une boucle ou d'un bâton en bois, tenez comme un crayon sous le même angle. N'appuyez pas sur si fort que les fouilles de la boucle, de bâton ou de cure-dent dans la gélose.
- Après avoir terminé le premier quadrant, inverser et mettre en bas de la plaque arrière dans le couvercle sur le banc. Éliminer le bâton ou cure-dent ou re-flamme de la boucle de métal tel que décrit à l'étape n ° 4.
- Tournez la boîte de Pétri de 90 ° à la série de la deuxième quadrant. Soulevez la moitié inférieure d'une plaque retournée sur le banc, puis touchez la boucle, un bâton ou cure-dent pour le premier quadrant près de l'extrémité de la ligne précédente. Avec le dos-et-vient modèle, traverser le lasmoitié des stries t dans le premier quadrant ensuite passer dans le quadrant vide seconde. Inverser et posa la plaque arrière dans le couvercle sur le banc une fois que le deuxième quadrant est rempli.
- La boucle, un bâton ou cure-dent ne doit jamais revenir dans la première moitié des stries dans le premier quadrant où la plupart de l'inoculum d'origine a été déposé.
- Une nouvelle boucle en plastique, bâton de bois ou de cure-dent doit être utilisé pour chaque quadrant.
- Répétez l'étape # 6 deux fois pour les troisième et quatrième quadrants.
- Assurez-vous de disposer de la baguette ou cure-dent ou re-flamme de la boucle de métal entre chaque quadrant.
- Évitez d'aller dans le premier quadrant quand striant la quatrième quadrant.
- Incuber la plaque à l'envers afin de condensation qui s'accumule sur le couvercle ne coule pas sur les colonies.
3. Verser la procédure Plate: Dénombrement des cellules bactériennes dans un échantillon mixte
Cette méthode est souvent utiliserd pour compter le nombre de micro-organismes dans un échantillon mélangé, qui est ajoutée à un milieu de gélose fondu avant sa solidification. Les résultats du processus dans les colonies répartis uniformément dans le milieu solide lorsque la dilution appropriée de l'échantillon est plaqué. Cette technique est utilisée pour effectuer des comptages de plaques viables, dans lequel le nombre total d'unités formatrices de colonies dans la gélose et sur la surface de l'agar-agar sur une seule plaque est énumérés. Dénombrement sur plaques des viables fournir aux scientifiques un moyen standardisé pour générer des courbes de croissance, pour calculer la concentration de cellules dans le tube à partir de laquelle l'échantillon a été plaquée, et à étudier l'effet de divers environnements ou des conditions de croissance sur la survie de la cellule bactérienne ou taux de croissance.
- Étiquette autour du bord du fond (pas le couvercle) de boîte de Pétri stérile sans vide avec au moins un nom, la date, le type de milieu de croissance, et le type d'organisme à être ajoutés au milieu de gélose fondue.
- Inclure le facteur de dilution si platIng dilutions en série, ou une série de dilutions répétées, ce qui entraîne une réduction systématique de la concentration de cellules dans l'échantillon. Préparation des dilutions en série est nécessaire si le nombre de cellules dans l'échantillon excède la capacité de la boîte de gélose, dans lequel la plage statistiquement significative est de 30 à 300 UFC. S'il ya plus de 300 ufc sur une plaque, puis les colonies sera bondé et se chevauchent.
- Procurez-vous un tube contenant 18 ml de gélose fondue.
- Le milieu de gélose doit être distribué dans les tubes à essai et pré-stérilisé en autoclave. Le même jour, il est nécessaire pour une expérience, l'agar-agar doit être fondu dans un bateau à vapeur pendant 30 minutes puis transférés dans un bain de 55 ° C l'eau. Seulement agar autant qu'il est nécessaire pour l'expérience devrait être fondu car il ne peut pas être réutilisé.
- Dix minutes avant de verser plaques, des tubes de gélose fondue doit être transféré à partir du bain d'eau à 55 ° C à un bloc de chaleur sur la main-d'oeuvreratoire banc fixé à 48 ° C. Une fois la gélose atteint cette température, il est prêt à verser. Si l'agar-agar est trop chaud, les bactéries présentes dans l'échantillon peut être tué. Si l'agar-agar est trop cool, le milieu peut être grumeleuse une fois solidifié.
- Obtenir votre échantillon, ce qui devrait être soit un bouillon de culture ou d'une suspension de cellules produites par les cellules de mélange provenant d'une colonie dans un buffer ou une solution saline.
- Les échantillons peuvent être dérivées à partir d'une série de dilutions d'un seul échantillon.
- Volume d'échantillon à plaquer devrait se situer entre 0,1 et 1,0 ml.
- Ouvrez le couvercle de la boîte de Pétri vide, et de distribuer votre échantillon dans le milieu de la plaque (Groupe A de la figure 4). Fermez le couvercle.
- Utiliser une technique aseptique tout au long de cette procédure.
- Utilisez une pipette sérologique ou micropipette pour transférer votre échantillon à la plaque. Contrôler le flux de l'échantillon afin de ne pas éclabousser de la plaque.
- Retirez le bouchon de la baignoiree de l'agar fondu, et passer le bord du tube ouvert à travers la flamme du bec Bunsen.
- Ouvrez le couvercle de la boîte de Pétri contenant l'échantillon et verser la gélose dans attentivement (Groupe B de la figure 4). Fermez le couvercle, puis mélanger l'échantillon avec l'agar-agar en remuant doucement la plaque.
- Laisser la gélose se solidifier avant de bien inverser la plaque pour l'incubation.
4. Procédure plaque préparée par étalement: Formation de colonies bactériennes discrets pour dénombrement sur plaques des, d'enrichissement, de sélection ou de dépistage
Cette méthode est généralement utilisé pour les micro-organismes séparés contenues dans un faible volume d'échantillon, qui est étalé sur la surface d'une plaque de gélose, conduisant à la formation de colonies discrètes réparties uniformément sur toute la surface de la gélose, lorsque la concentration appropriée de cellules est plaquée. En plus d'utiliser cette technique pour dénombrement sur plaques des viables, dans lequel le nombre total d'unités formant colonies sur un chanterplaque le est énumérée et utilisé pour calculer la concentration de cellules dans le tube à partir de laquelle l'échantillon a été plaqué, de placage propagation est couramment utilisée dans des expériences d'enrichissement, de sélection et de dépistage. Le résultat souhaité pour ces trois expériences est généralement le même que pour les chefs d'accusation de plaque, dans lequel une distribution de colonies discrètes formes à travers la surface de l'agar-agar. Cependant, le but n'est pas de s'assurer que toutes les cellules viables former des colonies. Au lieu de cela, seules les cellules au sein d'une population qui ont un génotype particulier devrait croître. La procédure plaque propagation peut être employé sur la technique des plaques d'écoulement pour une expérience de recensement si l'objectif final est d'isoler les colonies pour une analyse plus approfondie parce que les colonies se développer de manière accessible sur le surface de la gélose, alors qu'ils sont enfoncés dans la gélose à la procédure de plaque d'écoulement.
Il ya deux stratégies décrites ici pour la procédure plaque propagation. La première (méthode A) implique l'utilisation d'une plaque tournante et le verre ou en métal rod la forme d'un bâton de hockey. La seconde (méthode B), dénommé la «méthode Copacabana", implique secouent des billes de verre pré-stérilisés. À la fois faciliter encore la propagation des cellules à travers la surface de gélose.
Méthode A: la propagation de placage avec un tourne-disque et tige de verre ou de métal
- Étiquette sur le pourtour du fond (pas le couvercle) d'une plaque de gélose avec au moins votre nom, la date, le type de milieu de croissance, et le type d'organisme à être plaqué sur le support.
- Inclure le facteur de dilution si le placage des dilutions en série.
- Les plaques doivent être complètement sec sans condensation sur le couvercle et préchauffé à la température ambiante avant de la propagation de placage. Si les plaques sont stockées à 4 ° C, retirez-les plusieurs heures ou même la veille. Répartissez-les dans de petits empilements décalés de pas plus de 2-3 plaques et les laisser sécher.
- Centre de la plaque sur le plateau tournant (Figure 5).
- Vous obtenezl'échantillon r, qui devrait être un bouillon de culture ou d'une suspension de cellules produites par les cellules de mélange provenant d'une colonie dans un buffer ou une solution saline.
- Les échantillons peuvent être dérivées à partir d'une série de dilutions d'un seul échantillon.
- Le volume d'échantillon à revêtir doit être comprise entre 0,1 et 0,2 ml.
- Ouvrez le couvercle de la boîte de Petri, et de distribuer votre échantillon sur le centre de la gélose. Fermez le couvercle.
- Utiliser une technique aseptique tout au long de cette procédure.
- Utilisez une micropipette pour transférer votre échantillon à la plaque. Contrôler le flux de l'échantillon afin de ne pas éclabousser de la plaque.
- Tremper le verre ou d'une tige métallique tige (également appelé un épandeur) dans un bécher de 70% (v / v) d'éthanol.
- ATTENTION: Ne jamais tremper un épandeur à chaud dans un bécher d'alcool.
- L'éthanol ne doit toucher la partie inférieure de l'épandeur et le premier pouce de la tige.
- Égoutter et enflammer l'excès d'éthanol en le faisant passer à travers la flamme d'un Bunsfr brûleur.
- La flamme doit parcourir toute la longueur de l'épandeur et la tige qui est venu en contact avec de l'éthanol, puis éteindre rapidement.
- Si le bécher du feu de capture d'éthanol, ne paniquez pas! Placez un couvercle en verre sur le bécher, qui va vite éteindre le feu.
- Ouvrez le couvercle de la plaque de gélose, maintenir le couvercle dans votre main gauche avec votre pouce et l'index. Refroidir l'épandeur en le touchant à la gélose le long du bord proche de la jante.
- Ne touchez pas la gélose où les cellules ont été ajoutées. L'épandeur à chaud sera de tuer les cellules.
- Avec votre main gauche (tout en maintenant le couvercle de la plaque de gélose), faire tourner la table tournante lentement.
- Bien qu'il vaut mieux éviter, si vous devez mettre le couvercle vers le bas, placez-le face vers le bas sur une surface désinfectée au sein du champ stérile du bec Bunsen. Avec un couvercle qui fait face, il n'y a plus de chance de contamination par des mouvements d'objets ou des mains, la création de courants d'air qui provoquentmicroorganismes et des particules de poussière de descendre à la surface intérieure du couvercle.
- Avec votre main droite, maintenez la barre de flèche doucement sur la surface de la gélose et s'est progressivement étendue de l'échantillon uniformément sur toute la plaque. Déplacez la barre de flèche et d'autre de la plaque, comme la plaque tournante tourne.
- Laisser l'échantillon d'absorber complètement (au moins 5 minutes) avant de la retourner la plaque pour l'incubation.
Méthode B: Propagation de placage avec des perles de verre: la «méthode Copacabana"
- Étiquette sur le pourtour du fond (pas le couvercle) d'une plaque de gélose avec au moins votre nom, la date, le type de milieu de croissance, et le type d'organisme à être plaqué sur le support.
- Inclure le facteur de dilution si le placage des dilutions en série.
- Ouvrez le couvercle de la plaque de gélose, maintenir le couvercle dans votre main gauche avec votre pouce et l'index. Ensuite, ouvrez le tube en verre ou une bouteille contenant pré-stérilisés glperles ass. Avec votre main, la flamme le droit du bord du tube ou une bouteille, puis verser le liquide soigneusement 10-12 billes de verre stériles sur une plaque de gélose (Figure 6). Fermez le couvercle de la plaque, et la flamme de la jante du tube ou le flacon une fois de plus avant de remplacer le bouchon et le mettre de côté.
- Verser doucement les perles sur le tube ou une bouteille de quelques centimètres au-dessus de l'agar de sorte que les billes ne rebondissent sur la boîte de Petri.
- Utilisez des perles de verre qui sont de 4 mm de diamètre. Stériliser les bouteilles en verre ou tubes à essai dans l'autoclave à 121 ° C sur le cycle de séchage (réglage gravité) pendant 30 minutes.
- Un avantage de l'utilisation de billes au lieu d'un épandeur est qu'aucun des contenants ouverts de l'éthanol sont nécessaires pour flaming répétée.
- Ouvrez le couvercle de la plaque de gélose, et distribuer votre échantillon sur le centre de la gélose. Fermez le couvercle.
- Utiliser une technique aseptique tout au long de cette procédure.
- Aliquoter 100 à 150 pi d'échantillon par plaque. Cettevolume faciliter encore la propagation des cellules. Utilisez une micropipette pour transférer votre échantillon à la plaque. Contrôler le flux de l'échantillon afin de ne pas éclabousser de la plaque.
- Étaler l'échantillon en agitant doucement les billes sur la surface de la gélose 6 à 7 fois. Afin de garantir cellules répartie uniformément, utiliser un mouvement de secouage horizontal.
- Ne pas faire tourbillonner les perles ou encore toutes les cellules se terminera à bord de la plaque.
- ASTUCE: Si c'est fait correctement, la procédure ressemble à "secouer" maracas.
- Faites pivoter la plaque de 60 ° horizontalement, puis agiter à nouveau 6 à 7 fois.
- Faites pivoter la plaque de 60 ° une deuxième fois et horizontalement agiter à nouveau. A l'heure actuelle, vous devriez obtenir une répartition uniforme des cellules à travers la surface de gélose.
- Lorsque vous avez terminé la propagation de placage, l'échantillon doit être absorbée et la surface de la gélose doit être sec. Décanter les perles contaminés dans un bécher contenant de collecte nette eau de Javel à 10%.
- Ne pasjeter les perles dans la poubelle! Les billes utilisées seront rincés et autoclavés, re-stérilisation eux pour une utilisation répétée.
- REMARQUE: Si la surface de la gélose est encore humide après avoir serré la trois fois, permettre à la plaque de s'asseoir pendant plusieurs minutes afin de permettre au liquide d'être absorbé par l'agar-agar, puis répétez les étapes # 4-6 jusqu'à ce que le surface de la plaque semble sec.
- Retourner la plaque pour l'incubation.
5. Doux procédure gélose: la formation de plaques d'isolement et le dénombrement des phages (un essai sur plaque)
Cette technique est couramment utilisée pour détecter et quantifier bactériophage (phage), ou des virus bactériens qui varient en taille de 100 à 200 nm. Un microscope électronique est nécessaire pour voir les particules de phage individuels. Toutefois, la présence de particules de phage infectieux peut être détecté sous forme de plaques sur une plaque de gélose (figure 7A). Les phages ne peut pas répliquer en dehors de leurs cellules hôtes bactériennes, afin de propagation unela détection d exige phages de mélange et des cellules hôtes ainsi avant le placage. Pour la procédure de gélose molle, un petit volume, généralement dans la plage de 50 pl à 200 pl, d'une suspension de phage est distribué dans un tube contenant environ 10 8 bactéries (cellules hôtes) qui sont uniformément dispersées dans 2.5 à 3.0 ml d' douce (0,5 à 0,7% [w / v]), fondu gélose nutritive. Le mélange résultant est versé sur la surface d'un matériau dur (1,5 à 1,9% [p / v]) gélose nutritive. La plaque est basculé suffisamment pour que l'agar mou recouvre toute la surface de la gélose dur. Puis la plaque est placée sur une surface plane jusqu'à ce que la couche de top agar a eu le temps de se solidifier et ensuite peut être placé dans l'incubateur.
Au fil du temps, une suspension trouble des cellules bactériennes, dénommées une pelouse, devient visible à travers le milieu agar mou (figure 7B). Plaques se forment si un phage infecte une des cellules bactériennes, réplique à l'intérieur ecellule e, lyse de la cellule, puis libérant jusqu'à 100 phages descendance (aka, la taille de rafale). Les particules de phage de nouvelles diffuser dans la gélose molle, infecter les bactéries dans la région entourant la cellule bactérienne lysé. Après plusieurs cycles d'infection et une lyse, la suspension de cellules bactériennes dans le trouble agar mou disparaît, laissant une zone de compensation appelé une plaque. Chaque plaque contient plus de 10 9 particules de phage, tous génétiquement identiques à la particule de phage infectieux d'origine. Parce que se pose une plaque à partir d'une seule particule de phage, le nombre résultant d'unités formant des plages (pfu) peuvent être comptées de la concentration initiale, ou titre, de la suspension de phages peut être calculée. Ce type d'expérience, appelé un test de la plaque, fournit également aux scientifiques un moyen standardisé pour générer des courbes de croissance en une étape, pour enquêter sur la spécificité gamme d'hôtes, et pour transduire des cellules bactériennes pour des expériences génétiques.
- Préparer les bactéries indicatrices: Une culture en croissance exponentielle de la souche bactérienne hôte doit être préparé pour l'expérience gélose molle. Chaque hôte a ses exigences propres de croissance. Entre 0,3 ml et 0,5 ml d'une suspension de bactéries indicatrices est nécessaire pour chaque plaque. Si un flacon de bactéries indicatrices est préparé (par exemple, 25 ml), la culture devrait être divisé en aliquots plus petits (par exemple, des aliquotes de 5 ml déversés dans des stériles tubes à bouchon vissé) pour réduire au minimum la possibilité de contamination croisée. Les tubes peuvent être conservés dans la glace (à 4 ° C) jusqu'au moment de servir dans l'expérience. Utiliser une technique aseptique pour inoculer un bouillon nutritif stérile puis incuber selon les spécifications de la souche bactérienne. Les restes de cultures doivent être jetés à la fin de la journée.
- Préparer des tubes d'adsorption: Placez deux microtubes stériles dans un rack. Étiquetez le couvercle du tube "Phage" premier et le couvercle du tube "Control" seconde.
- Typiquedilutions ly série de lysats de phages sont préparés et étalés dans ce cas, des microtubes supplémentaires seraient ajoutés à la grille et étiquetés avec le facteur de dilution.
- Stocks de phages doit être préparée à partir de plaques simples et conservés à 4 ° C. Pour ventiler les particules de phage, les stocks devraient être réchauffé à la température ambiante avant de procéder à une expérience.
- Stocks de phages doit être manipulé avec soin - ne pas vortexer ou une pipette vigoureusement.
- Ajouter 50 ul de l'échantillon de phages pour le premier tube, puis ajouter 50 ul de tampon de phages pour le second tube, qui servira comme un contrôle négatif pour le dosage de plaque.
- Utiliser une technique aseptique tout au long de toutes les manipulations de phages et les bactéries.
- Ensuite, ajoutez 500 ul de bactéries indicatrices dans chaque tube d'adsorption.
- Les cellules bactériennes se déposent au fond d'un tube si assis sur la glace ou le banc pendant de longues périodes de temps. Si la culture n'est pas mélangered avant de retirer une partie aliquote de l'expérience, pas assez de cellules bactériennes seront transférés sur le tube d'adsorption. Il doit y avoir un nombre suffisant de colonies bactériennes formées dans la gélose molle (c.-à-une pelouse) pour la détection des plaques. Utilisez un mélangeur vortex pour doucement remettre en suspension les bactéries indicatrices dans une suspension homogène avant de transférer des aliquotes dans les tubes d'adsorption.
- Mélanger les cellules bactériennes et de phages par un léger effleurant les tubes.
- Incuber le phage / mélange bactéries pendant 15-20 minutes (pas plus de 30 minutes) à une température appropriée pour la souche indicatrice.
- Préparer des éléments nutritifs agar mou et des plaques d'agar durs: Bien que l'adsorption se produit, le lieu de deux tubes de gélose molle (préalablement fondu et stocké à 55 ° C) dans un bloc chauffant à votre paillasse de laboratoire fixé à 46-48 ° C.
- La gélose fondue douce doivent être ramenés à la température du bloc chauffant pendant 10-15 minutes. Si la douce fondueagar est assis plus de 15 minutes, il va commencer à se solidifier et former des amas quand on le verse sur la gélose dur. Si la gélose fondue douce se situe à moins de 10 minutes, il sera trop chaud lorsqu'il est ajouté au phage / bactérie mélange, tuant les cellules hôtes avant le placage. Par conséquent, une pelouse ne peuvent pas former et les plaques peu ou pas peut être détectable.
- Préparer supplémentaires tubes agar mou, si le placage des dilutions en série de l'lysat de phage.
- Étiquette sur le pourtour du fond (pas le couvercle) de deux plaques de gélose nutritive plaque dur avec au moins votre nom, la date, le type de milieu de croissance, et "Phage" ou "Control" correspondant à des tubes d'adsorption.
- Inclure des plaques supplémentaires marqués par le facteur de dilution si le placage des dilutions en série.
- Les plaques d'agar durs doivent être complètement sec sans condensation sur le couvercle et préchauffé à la température ambiante avant d'ajouter l'agar mou. Si les plaques sont stockées à 4 ° C, retirez-les plusieurs heures, voire de la day avant. Répartissez-les dans de petits empilements décalés de pas plus de 2-3 plaques et les laisser sécher. L'humidité excessive dans la couche d'agar dur entraîner une dilution de l'agar mou, permettant de diffuser phage plus facilement à travers l'agar mou pendant la formation de plaque. Par conséquent, la taille des plaques vont augmenter.
- Plaque d'adsorption des tubes un à la fois comme suit: Utilisez une micropipette P1000 à transférer aseptiquement le phage / bactérie mélange (qui devrait être d'environ 550 pi) à un tube de gélose molle alors rapidement faire tourner entre les paumes de vos mains pour mélanger le contenu. Ne PAS secouer le tube de sorte que les bulles d'air sont introduits. En tenant le tube dans votre main gauche, ouvrez le couvercle d'une boîte de gélose dur avec votre main droite et verser immédiatement la totalité du contenu du tube sur la surface d'une plaque de gélose dur. Avant de fermer le couvercle, secouez doucement la plaque, mais rapidement à répandre la gélose fondue douce sur toute la surface de la plaqueavant qu'il ait le temps de se solidifier. Eviter les éclaboussures de la gélose fondue doux sur les côtés de la boîte de Petri. Fermez le couvercle.
- Répéter l'étape # 9 pour tous les tubes d'adsorption, un tube à la fois.
- Placer les plaques sur une surface plane et de leur permettre de se tenir tranquille jusqu'à ce que la gélose molle est solidifié.
- Habituellement 30 minutes est suffisante.
- Inverser les plaques d'incubation.
- Plaques multiples peuvent être empilées et collées ensemble, puis inversé pour l'incubation.
Après incubation, les plaques peuvent être inspectés pour les plaques. Le contrôle négatif ne devrait avoir qu'un tapis de bactéries (pas de trous de plaques indicatives). Plaques varient en termes d'apparence taille, la forme et dans l'ensemble. Un lysotype donnée peut être isolé à partir d'un mélange hétérogène de plaques par poinçonnage soin du centre de l'un de plaque avec un cure-dent stérile et transfert de l'inoculum d'un t microcentrifugeuse stérileube contenant 100 à 1000 pi de bouillon ou de tampon phage. Ce lysat peut être plaqué utilisant la même procédure décrite ci-dessus. Au moins 3 à 6 successives simple plaque isolements sont nécessaires pour veiller à ce qu'un phage pur a été obtenu. Souvent, le lysat doit être dilué dans une large gamme (10 -1 à 10 -10) pour trouver un titre qui produit non-chevauchement des plaques sur une plaque. Le nombre varie en fonction de la taille de la plaque.
6. Procédure Plate Replica: transfert de cellules pour les mutants auxotrophes de dépistage et
Cette technique permet de comparer la croissance des cellules sur une plaque primaire à plaques secondaires, un moyen de génération à des cellules d'écran pour un phénotype sélectionnable. D'abord un primaire, ou le capitaine, la plaque est inoculé avec des cellules, soit par propagation-le placage d'une dilution qui produit des colonies isolées ou en les transférant à une plaque dans un modèle spatiale spécifiées par des marques de la grille. Plaques secondaires contenant les médias avec inhib la croissanceitors ou des médias qui manque d'un nutriment particulier sont inoculés avec des cellules provenant de colonies sur la plaque principale. La répartition spatiale des colonies est reproduit première pression sur une pièce de velours à la plaque principale. Cellules bactériennes adhérer à du velours car ils ont une plus grande affinité pour le velours de la gélose. L'empreinte de cellules sur le velours est ensuite transféré à de multiples plaques secondaires avec la croissance des cellules du motif réfléchissant colonie même que celui de la plaque principale. En d'autres termes, c'est comme avoir un tampon en caoutchouc, en reproduisant le modèle de croissance d'une plaque à l'autre. Cette technique est avantageuse car elle permet à un nombre relativement important de colonies à être projeté simultanément des phénotypes de nombreux en une seule expérience.
- Préparer la plaque primaire: Label autour du bord du fond (pas le couvercle) d'une plaque de gélose avec au moins votre nom, la date et le type de milieu de croissance.
- Marquez d'une grille sur le fond de la plaque avecau moins deux lignes verticales équidistantes et au moins deux lignes horizontales espacées de manière égale. Nombre des places qui en résultent.
- Utilisez un cure-dent pré-stérilisé pour inoculer chaque carré avec un échantillon de cellules. Pour chaque échantillon, la limande du centre de la place. Ne pas couvrir toute la place avec des cellules (Figure 8), ou bien l'échantillon proliférer lorsqu'ils sont incubés et contaminer carrés entourant.
- Chaque carré sera inoculé avec un échantillon différent, proviennent, soit de cultures en bouillon ou en colonies sur une autre plaque.
- Inverser et incuber la plaque principale, qui sera utilisé pour inoculer les divers milieux secondaires.
- Inoculer plaques secondaires: Stack la plaque primaire et toutes les plaques secondaires. Avec un marqueur, faire une marque d'orientation sur le côté des plaques. Assurez-vous que la marque est sur le côté inférieur de chaque plaque, pas le couvercle.
- Obtenir un chiffon de velours stérile et placez-le sur le bloc cylindrique (Figure 9). Verrouiller le tissu en velours en place avec le titulaire. Notez la marque d'orientation sur le bloc.
- Le bloc doit être la même taille que le fond de boîte de Pétri (10,2 cm de diamètre). Il devrait venir avec un anneau de verrouillage qui serre le tissu en velours sur le bloc de placage réplique tout.
- Le tissu velours (15,2 x 15,2 cm de côté) doivent être pré-stérilisés. Pile 10 ou 12 places propres, puis les envelopper dans du papier aluminium, puis les placer dans l'autoclave à 121 ° C sur le cycle de séchage (réglage gravité) pendant 30 minutes. Avant de les utiliser dans une expérience de placage réplique, assurez-vous qu'ils sont complètement secs en les plaçant dans un four chaud pendant plusieurs heures. Notez que les carrés de velours peut-être besoin d'être dé-implémentés avec lint avec du ruban adhésif avant de les stériliser.
- Carrés de velours peuvent être réutilisés. Après carrés de velours utilisés ont été décontaminés à l'autoclave, ils doivent être rincés à l'eau claire et re-stérilisés comme décrit ci-dessus.
- Le bloc cylindriquek et pince doit être désinfecté entre les utilisations avec une mémoire de rinçage dans 70% (v / v) d'éthanol ou agent de blanchiment 10%.
- Retirez le couvercle et retourner la plaque principale. Alignez le repère d'orientation sur la plaque avec la marque sur le bloc. Abaisser la plaque de sorte que la surface de la gélose est en contact avec le tissu de velours sur le bloc cylindrique. Légèrement, mais régulièrement appuyez vers le bas avec le bout des doigts sur le dos de la plaque principale, puis soulevez délicatement la plaque primaire loin du bloc. Remettre le couvercle sur la plaque.
- L'impression de velours de cellules provenant de la plaque primaire peut être utilisée pour inoculer environ 7-8 plaques secondaires avant une impression avec un velours de nouveaux besoins à prendre.
- Répéter l'étape 7 à chacune des plaques secondaires.
- Comme contrôle positif, la dernière plaque de la série devrait être un milieu de gélose dans laquelle toutes les souches testées devrait croître. De cette façon, vous pouvez confirmer que les cellules ont été transférées à l'ensemble du secondaire plAtes dans la série. Sinon, l'absence de croissance sur un milieu d'essai particulière peut être attribuée à la cellule de transfert insuffisante plutôt que d'un phénotype de la souche.
- Pour éviter les faux positifs, les plaques secondaires doivent être commandés au moins à partir de substrat plus favorable. Sinon, les éléments nutritifs peuvent être transférés entre les plaques permettant aux cellules de se développer sur un milieu défavorable.
- Inverser les plaques et incuber.
- Remarque: Lors de l'inspection des plaques secondaires pour la croissance, assurez-vous de faire la distinction entre la croissance et une empreinte. Ce dernier est un résultat négatif.
7. Nettoyage de l'espace de travail
- Éteignez le brûleur Bunsen, puis mis de côté toutes les fournitures y compris les cultures sur plaques ou en tubes, les médias supplémentaires, et d'autres réactifs.
- Ne pas laisser les vieilles cultures, que ce soit sur des plaques ou dans des tubes, à accumuler sur le banc de laboratoire ou dans des zones de stockage. Ces échantillons, qui sont des sources notoires de contaminationtels que les moisissures et les espèces bactériennes indésirables, doivent être jetés dès qu'ils ne sont plus nécessaires.
- Lieu contaminé matériel de laboratoire (gants, embouts de pipette, Kimwipes), verrerie (tubes, flacons, bouteilles), et les déchets dangereux (cultures bactériennes ou des solutions de phages, des plaques utilisées) dans le récipient d'élimination adéquat. Lorsque vous travaillez avec un E. non-pathogène coli souche (BSL-1), que non infectieux déchets dangereux est généré. Lors de ces mêmes procédures avec des organismes pathogènes (BSL-2 ou au-dessus), infectieuse déchets dangereux sont produits. Indépendamment de la classification des risques biologiques, les déchets dangereux doivent être autoclavés ou désinfectés avant d'être rejetée. Suivez les directives décrites dans le BMBL (5 e éd.) Ainsi que ceux fournis par votre santé environnementale institutionnelle et le département de la sécurité pour l'élimination immédiate et appropriée des risques biologiques générées lors d'une expérience.
- Nettoyer la zone de travail avec un désinfectant.
- Se laver les mainssoigneusement avec du savon antiseptique et de l'eau chaude avant de quitter le laboratoire.
8. Les résultats représentatifs
Streak-technique de la plaque. Un exemple d'application pour le placage série est illustré à la figure 1. Cette procédure est utilisée pour isoler les colonies bactériennes à partir de cultures cellulaires mixtes et est de loin l'une des techniques les plus importantes à maîtriser dans la microbiologie et de génétique moléculaire. Chaque colonie représente une population de cellules qui sont génétiquement identiques. Pour de nombreuses applications en aval, il est impératif de commencer par une seule colonie ou d'une culture bactérienne pure générée par l'inoculation des médias avec des cellules à partir d'une seule colonie. Par exemple, la morphologie des cellules individuelles au sein d'une colonie peut être inspecté à l'aide d'un microscope optique. L'identité génétique peut être attribué par le séquençage de la petite sous-unité ribosomique gène de l'ARN à partir d'ADN génomique isolé à partir d'une culture cellulaire a commencé avec une seule colonie.Et les caractéristiques métaboliques peut être décrite en soumettant les cellules à différents tests biochimiques et physiologiques. Seulement en effectuant de telles expériences avec des cultures pures peut-on être certain de les propriétés attribuées à un micro-organisme particulier. Les résultats ne sont pas masqués par la possibilité que la culture est contaminée. Des erreurs techniques peuvent se produire si la stérilité de l'instrument utilisé pour strier les cellules à travers la plaque n'est pas maintenu tout au long de la procédure. Oublier de la flamme d'une boucle ou de récupérer un cure-dent frais entre quadrants, il est difficile d'obtenir des colonies isolées. Certaines espèces bactériennes ne peut être isolée en culture pure car ils sont dépendants d'une association coopérative avec une autre espèce bactérienne pour les besoins de croissance de certains. Désigné sous le nom syntrophs, ces organismes ne peuvent être cultivées dans des conditions de co-culture, de sorte colonies (si elle est formée) sera toujours composé de deux ou plusieurs espèces. Un autre problème rencontré dans le laboratoire lors de l'exécution dustrie-plaque de la procédure par des bactéries provenant des échantillons environnementaux est que les cellules présentent des caractéristiques de croissance qui s'écartent à partir de souches de laboratoire traditionnels, tels que E. coli. Ces souches bactériennes peuvent produire des colonies qui sont filamenteuses (par opposition à grappes serrées de cellules) avec des branches qui s'étendent sur une grande partie d'une plaque de gélose, calcifiées et donc réfractaire à la pénétration par un instrument strie-plaque, ou entouré d'une capsule collant de sorte que les colonies individuelles ne peuvent être discernés. Ces caractéristiques font qu'il est difficile de purifier des colonies isolées par la technique de stries plaque.
Technique pour-plaque. Avec la technique d'écoulement à plaques, les colonies former au sein de la gélose, ainsi que sur la surface du milieu gélosé fournissant ainsi un moyen commode pour compter le nombre de cellules viables dans un échantillon. Cette procédure est utilisée dans une variété d'applications industrielles. Par exemple, il est essentiel pour une tre des eaux uséesusine de atment, qui est responsable du nettoyage des déchets liquides (par exemple, les eaux usées, le ruissellement des eaux pluviales) générés par les propriétés domestiques, commerciaux et industriels ainsi que les pratiques agricoles, d'analyser des échantillons d'eau à la suite du processus de purification extensive. Les eaux usées traitées (eau non potable) est réutilisé dans une variété de façons - pour l'irrigation des cultures non-alimentaires dans l'agriculture, pour le rinçage sanitaire dans les résidences, et dans les tours de refroidissement industrielles - de sorte qu'il doit être exempt de contamination chimique et microbienne. L'eau potable (eau potable) doit être purifié selon les normes de l'EPA et est testée en utilisant des méthodes microbiologiques qui permettent de placage énumération de certains agents pathogènes humains. Illustré à la figure 10 sont des colonies bactériennes résultant de cellules de bactéries présentes dans un échantillon d'eau prélevé à partir d'une fontaine publique. Il est peu probable bactéries pathogènes produites ces colonies, compte tenu des mesures de purification pour l'eau potable, mais les microbes sont partoutoù et même la contamination par des souches non pathogènes peut être seulement minimisé, et non pas d'éliminer entièrement. Comme autre exemple, une entreprise pharmaceutique doit évaluer le degré de contamination microbienne, ou la charge microbienne, d'un nouveau médicament en cours de production, de stockage et de transport. En échantillonnant le médicament pendant les diverses phases du processus et des échantillons de placage en utilisant la procédure pour-plaque, la charge microbienne, ou le nombre de bactéries contaminantes, peut être facilement déterminée. Les mesures de précaution peut alors être conçu pour minimiser ou éliminer la contamination microbienne. Une des erreurs les plus courantes techniques qui se produit lors de l'exécution de la technique pour-plaque est insuffisante mélange de l'échantillon avec les colonies sur gélose fondu provoquant à s'agglutiner rendant compte de la plaque inexactes. Une autre erreur fréquente est de verser le gélose fondue quand il est trop chaude, tuant un grand nombre des cellules bactériennes dans l'échantillon. Cette erreur sera également affecter la précision du dénombrement sur plaques des numéros de donner ce que sous-représenter les ee nombre total d'unités formant des colonies dans l'échantillon.
Spread-technique de la plaque. La technique d'étalement de plaque est analogue à la procédure pour-plaque dans son utilité en tant que moyen d'effectuer dénombrement sur plaques des viables. Cependant, parce que les colonies qui forment en utilisant la technique à étalement de plaque sont réparties uniformément sur la surface du milieu de gélose, des cellules provenant de colonies individuelles peut être isolé et utilisé dans des manipulations expérimentales suivantes (par exemple, que l'inoculum pour une strie-plaque ou une bouillon de culture). Trois applications courantes dans lequel la technique d'étalement de plaque est un élément important sont les expériences d'enrichissement, de sélection et de dépistage. Dans tous les trois demandes, du type cellulaire souhaité peut être séparé du mélange et ensuite soumis à un nombre quelconque de tests de biochimiques, physiologiques ou génétique.
Une expérience d'enrichissement consiste à plaquer une culture mixte sur un support ou l'incubation des plaques dans l'econditions qui favorisent la croissance ENVIRONNEMENTALE de ces micro-organismes au sein de l'échantillon qui démontrent les propriétés métaboliques souhaités, les caractéristiques de croissance, ou des comportements. Cette stratégie n'a pas inhiber la croissance d'autres organismes, mais les résultats à une augmentation du nombre de micro-organismes désirés par rapport aux autres dans la culture. Ainsi, les colonies qui se forment sur une plaque d'enrichissement susceptible présentent des propriétés phénotypiques qui reflètent le génotype désiré. Par exemple, si votre objectif est de cultiver bactéries fixatrices d'azote à partir d'un échantillon de l'environnement contenant un mélange de plus de 1000 espèces bactériennes différentes, puis plaquer l'échantillon sur un support déficient en azote viendra enrichir pour les bactéries qui peuvent produire ce composé à partir de la l'atmosphère en utilisant les capacités métaboliques fournis par une suite de gènes nécessaires à la fixation de l'azote.
Une expérience de sélection consiste à plaquer une culture mixte sur un support qui permet uniquement les cellules qui conmaintenir un gène particulier ou un ensemble de gènes de croissance. Ce type d'expérience est courante dans les laboratoires de biologie moléculaire lors de la transformation des souches bactériennes avec des plasmides contenant les gènes de résistance aux antibiotiques. Si votre objectif est de cultiver seulement des cellules recombinées, ou ceux qui ont réussi à pris le plasmide, puis plaquer l'échantillon sur un support qui a été complétée par une concentration appropriée de l'antibiotique sera sélectionner pour ces cellules qui présentent une résistance à ce médicament particulier.
Une expérience de dépistage consiste à plaquer une culture mixte sur un support qui permet toutes les cellules viables de croître, mais les cellules avec le génotype désiré peuvent être distinguées des autres cellules en fonction de leur phénotype. Encore une fois, ce type d'expérience est courante dans les laboratoires de biologie moléculaire lors de la réalisation des tests de mutagenèse ou le clonage de gènes dans des plasmides. Un exemple classique, comme le montre la figure 11, fait usage de la enco gène lacZding β-galactosidase; cette enzyme permet aux cellules à métaboliser X-Gal (5-bromo-4-chloro-3-indolyl-β-D-galactopyranoside), un analogue de substrat de son substrat naturel, le lactose. Le clivage de X-Gal par β-galactosidase des résultats dans un produit bleu insoluble. Ainsi, si un support contient X-gal, et un échantillon contenant des cellules soit avec un type sauvage (fonctionnelle) ou mutante (non-fonctionnel) gène lacZ sont étalées sur ce milieu, puis à la suite d'incubation cellules de type sauvage abritant une fonctionnelle lacZ gène apparaît en bleu-pigmentées colonies tandis que les cellules mutantes avec un gène non fonctionnel lacZ apparaîtra comme non pigmentées ("blanc") colonies.
Un problème technique la plus fréquemment rencontrée lors de la première à apprendre à effectuer la technique d'étalement de plaque est une distribution inégale des cellules à travers la surface agar. Lors de l'utilisation un plateau tournant et de la tige en verre, l'échantillon peut être absorbée trop rapidement de telle sorte que les colonies former seulement à proximité du centre de la plaque. Wpoule fait la «méthode Copacabana", les perles de verre sont tourbillonnait plutôt que ébranlée à travers la surface de la gélose. Par conséquent, de nombreuses colonies se développer le long du bord externe de la plaque. Dans les deux cas, la distribution résultant des colonies ne profite pas de la surface totale disponible de sorte cellules peuvent s'agglutiner et se développer dans les colonies qui se chevauchent rendant compte de la plaque inexactes ou distinction de types de cellules irréalisables.
Doux technique de superposition Agar. Un procédé semblable à la technique d'étalement de plaque utilisé pour compter des colonies bactériennes peut être utilisé pour calculer le nombre de phage. Alors qu'entre 30 et 300 cellules bactériennes sont étalés sur la surface de la gélose pour dénombrement sur plaques des (ufc / ml), entre 100 et 400 particules phagiques infectieuses sont mélangés avec 10 de 8 à 10 9 cellules hôtes pour la numération des plaques (UFP / ml) dans une couche de la gélose molle répartis sur la surface de gélose nutritive dur. Sauf preuve du contraire, il est généralement admis tchapeau d'une seule cellule bactérienne se divise et accumule un grand nombre de cellules génétiquement identiques dans un cluster unique appelé une colonie. Comme indiqué précédemment, cette hypothèse n'est pas valable lorsque les cellules se développent en grappes (c.-à-paires, tétrades, des chaînes ou grappes) ou d'afficher des caractéristiques de croissance tels que les capsules qui entravent la formation de colonies unique. Un hypothèse similaire est prévu pour la formation de plaque, en ce que chaque plaque représente l'activité d'un phage clé. Cette affirmation est vraie uniquement si un phage infecte une bactérie. Qu'advient-il si des particules de phage multiples infecter une seule bactérie? Ce problème est lié à un paramètre important statistique qui doit être pris en considération lors réaliser des expériences avec le phage - multiplicité d'infection (MOI) - décrivant le rapport des particules de phage infectieuses au nombre de cellules hôtes dans un échantillon. Parce que certains adsorber les cellules plus d'un phage tandis que les cellules d'autres adsorber qu'un seul ou aucun phage, une population de cellules d'accueil devraient être infectées à un faible moment d'inertie (≤ 1) to minimiser la probabilité qu'une cellule soit infecté par plus d'une particule de phage. Employant la plaque forming unit (pfu) comme une définition fonctionnelle permet d'éviter ces complications lors de l'exécution compte la plaque pour calculer le titre d'un stock de phages.
Comme le montre la figure 12, morphologie de la plaque varie pour le phage différente. Certains phages générer des petites plaques (partie A) tandis que d'autres donnent lieu à de grandes plaques (Groupe B). Un certain nombre de variables influent sur la taille des plaques. Il ya des raisons techniques qui contribuent à cette variabilité. Par exemple, les médias complets et une épaisseur de développement dur soutien des grandes plaques de gélose, car les cellules hôtes peuvent soutenir la croissance du phage pour une plus longue période de temps. Un haute densité de placage de cellules hôtes (> 10 9 cfu par la plaque) provoque une réduction de la taille de la plaque. En utilisant de plus faibles concentrations de gélose molle va augmenter le taux de diffusion des particules de phage dans la gélose molle et ainsi augmenter la taille des plaques. Rappelons eà ce taux de diffusion accrue peut se produire par inadvertance si les plaques d'agar durs ne sont pas sécher complètement de telle sorte que l'humidité de condensation ou d'excès dans le plat dilue l'agar mou dans la superposition. Ce contrôle technique va produire des résultats incohérents par rapport à la taille des plaques pour un phage particulier.
La taille des plaques est également lié à un certain nombre d'événements de cellules hôtes comprenant l'efficacité d'adsorption, la durée de la période de latence (l'intervalle de temps à partir du phage d'adsorption à la lyse de la cellule hôte), et la taille de salve (le nombre de descendance libéré par une seule infection). Un mélange hétérogène de tailles de plaque peut être observé si des particules de phage infecter des cellules hôtes lors des différentes phases de la croissance bactérienne. Par exemple, ceux qui adsorbent durant la phase exponentielle au début faire de plus gros plaques avec le phage plus descendance que ceux qui s'adsorbent en phase exponentielle tardive. En règle générale, un phage lytique produire des plaques claires, alors que les plaques de phages lysogènes troubles du formulaire. HoWever, certains phages lytiques produire des modèles intéressants, tels que "œil de bœuf» de la plaque montré dans la figure 12B. Ces plaques claires sont entourés par un halo trouble parce que ces cellules au niveau du bord de la plaque ne sont pas entièrement lysées ou peut être résistant à l'infection au phage. Un "taureau oeil" tendance observée avec le phage tempéré est une plaque avec un centre de trouble entouré par un anneau clair. Cette morphologie reflète la MOI et de la physiologie de la cellule hôte à l'égard de la décision lyse-lysogénie. Lorsque les cellules sont d'abord infectées par le phage, la MOI est faible et les cellules se développent rapidement car les nutriments sont abondants; ensemble ce qui facilite la croissance lytique. Comme les cellules de plus en plus sont lysées, la MOI augmente et une plaque se forme claires. Lysogènes dans le centre de la plaque, cependant, continuer à croître car ils sont à l'abri de lyse donnant lieu à une plaque claire avec un centre de trouble.
La technique de superposition peut être modifié pour des essais de plaque avec des virus eucaryotes. Dansles mêmes plaques manière bactériophages forme sur un tapis de cellules bactériennes dans l'agar mou, les virus eucaryotes former des plaques sur une monocouche de cellules couvertes par un gel. Un monocouche confluente est une feuille de culture de cellules côte à côte sur la surface d'un récipient de culture, en contact entre eux, mais pas de plus en plus au-dessus de l'autre. Pour mener à bien ce type de dosage de plaque, des aliquotes de virus sont ajoutés aux monocouches sensibles des cellules eucaryotes. Puis la monocouche est recouvert d'un support d'agarose à base nutritive - gel de cette limite la propagation de virus descendants libérés de cellules infectées à des cellules adjacentes dans la monocouche. En conséquence, une zone sphérique, ou de la plaque, est produit qui contient des cellules endommagées par la libération des virions. Pour faciliter la visualisation des plaques, des colorants qui tache les cellules vivantes peuvent être appliquées à la culture cellulaire avec un contraste entre les cellules infectées et non infectées.
La technique de superposition douce-agar est utilisé pour des expériences d'autres que les essais de plaque. Tout d'abord, il est sRINCIPALES de se rappeler que l'agar-agar dur en éléments nutritifs est une matrice de support qui permet la croissance des bactéries. Deuxièmement, le soft-agar utilisé pour le fond peut avoir une composition différente des éléments nutritifs que l'agar-agar dur. De cette façon, l'agar mou-peut servir comme un moyen de souches bactériennes de dosage pour des caractéristiques de croissance différentes ou des propriétés métaboliques. Par exemple, la technique de superposition est utilisée pour les bactéries d'écran pour la capacité de dégrader la cellulose (Teather et Wood, 1982). Des colonies isolées sont cultivées sur un milieu non sélectif agar dur puis soft-agar contenant 0,1% (p / v) de carboxyméthylcellulose (CMC) est répartie sur la surface de l'agar dur. Après incubation, les plaques sont inondés avec des tâches qui permet la visualisation des zones de compensation autour des colonies dans la gélose molle. La compensation est causée par des enzymes hydrolytiques secrétées par les bactéries décomposant la cellulose dans le milieu. Plus récemment, la technique de superposition a été utilisé pour détecter les bactéries qui inhibent la croissance des methanogeArchaea nic dans le rumen des animaux d'élevage (Gilbert et al. 2010). Les isolats bactériens provenant d'échantillons de l'environnement sont cultivées sur un milieu nutritif gélose dur, puis les colonies sont recouvertes de soft-agar contenant une culture de bactéries méthanogènes. Après incubation, les plaques sont inspectés pour les zones d'inhibition de croissance autour des colonies. Cette méthode identifie les souches bactériennes qui produisent des inhibiteurs des bactéries méthanogènes dans la gélose molle.
Les erreurs les plus courantes techniques qui se produisent avec la technique de superposition douce-agar sont en versant le fondu doux-agar, soit quand il est trop chaud ou trop froid. Si elle est trop chaude, les cellules bactériennes mélangé dans le milieu seront tués avant le placage. Si elle est trop cool, puis l'agar mou-vous former des amas quand on le verse sur la gélose dur. Dans les deux cas, les résultats seront ambiguë ou illisible, au mieux.
Réplique de plaque procédure. Transfert des cultures d'un type de milieu nutritifà l'autre pour tester les exigences de croissance devient très laborieux s'il ya plus que de simples quelques souches. Plaquage réplique est un procédé qui permet de dépistage simultanée d'un grand nombre de micro-organismes. Par exemple, après mutagenèse d'une culture de cellules de type sauvage, on peut se propager à plaques des dilutions de la culture pour obtenir des plaques avec des colonies isolées. Les plaques primaires contenir un milieu qui favorise la croissance de toutes les cellules, y compris de type sauvage prototrophes, qui synthétisent tous les composés nécessaires à la croissance et auxotrophes mutantes, qui portent une mutation génétique dans une voie de biosynthèse les rendant incapables de synthétiser des composés particuliers essentiels à la croissance. En étalant le mélange de cellules sur un support complet, les nutriments manquants peuvent être pris à partir de l'environnement. Pour distinguer entre les prototrophes et auxotrophes, les colonies peuvent être répliquées sur un milieu minimal. Seulement prototrophes sera en mesure de croître. Parce que la répartition spatiale de la plaque principale est préservée, Comparissur la plaque secondaire avec la plaque primaire permet une identification de colonies mutantes. Pour déterminer lequel composé des mutants ne sont plus capables de synthétiser, les colonies peut être répliqué sur un milieu minimal supplémenté avec des composés spécifiques (par exemple, des acides aminés, des sources de carbone, des vitamines, etc). De cette manière, des centaines de colonies peuvent être criblés en même temps en utilisant la procédure réplique-plaque. Une erreur technique qui pourrait se produire est l'utilisation des plaques de gélose qui sont trop humides, provoquant des colonies de salir ainsi la contamination de toutes les cultures sur la plaque. Cela produit des résultats qui sont entièrement fiables. Une autre erreur technique est à appliquer une pression trop lors du transfert de cellules provenant de la velours aux plaques secondaires. Encore une fois, après incubation des plaques secondaires, les colonies qui en résultent peuvent se chevaucher produire phénotypes de croissance attribuées à la contamination plutôt que auxotrophie.
Toutes les espèces microbiennes de type sauvage sont prototrophes, de sorte que la réplique-pprocédure de fin peut être utilisé pour cribler simultanément différentes souches de type sauvage pour les besoins de croissance caractéristiques. Comme le montre la Figure 13, "plots" de cellules provenant de quatre souches de Pseudomonas différentes bactéries ont été étalées en double exemplaire sur une plaque de grille-marquée contenant des milieux complets appelé YTA (partie A). Les souches ont été ensuite reproduite sur trois plaques secondaires et les panneaux B, C et D) composé d'un milieu minimal (MSA) supplémenté avec une source de carbone différente (acétamide, le lactose, et de la glycine, respectivement). Les résultats démontrent que deux des souches de Pseudomonas quatre (P. aeruginosa et P. stutzeri) sont incapables de croître sur ces trois sources de carbone. Comme un contrôle, les souches ont été reproduits sur une plaque en quatrième position avec support YTA pour confirmer cellules ont été transférées durant la procédure. Comme tous les quatre souches se développent sur la plaque de commande YTA, les déficits de croissance exposé sur les trois dernières plaques de la série sont fiables. Les résultats réplique-placage sont présentées au tableau 1. Une erreur couramment faite est l'interprétation d'une empreinte de la croissance sur une plaque secondaire comme un résultat positif. Par exemple, comparer le phénotype de P. aeruginosa à celle de P. stutzeri sur MSA + acétamide (panneau B). Ce dernier affiche une empreinte de la croissance, qui est un résultat négatif, et peut se produire si la plaque de nutriments précédente sont transférés avec les cellules parentales. Pas de croissance de nouvelles cellules se produit parce que les nutriments manquants ne sont pas disponibles à cellules de la descendance. Il est facile de confondre une empreinte avec une croissance réelle. En cas de doute, l'expérience devrait être répétée en utilisant une méthode alternative comme strie-placage des cellules de la plaque primaire sur des supports secondaires.
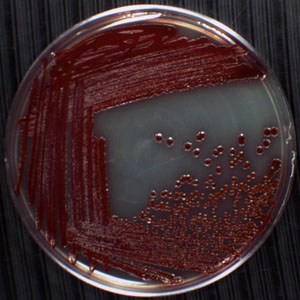
Figure 1. Exemple de colonies isolées sur une plaque. Les sphères roses près du centre de la plaque sont des colonies de Serratia marcescens, un bacille à Gram négatif, en forme de tige Protéobactérie dans la famille des entérobactéries. En raison de sa préférence pour les milieux humides, ce micro-organisme est communément trouvée de plus en plus dans les coins de baignoires, éviers dans les bassins, en coulis, et sur les rideaux de douche. S. marcescens est facile à reconnaître car il produit un pigment rougeâtre appelé prodigiosine. Les colonies sur cette plaque ont été générés en utilisant la technique strie-plaque, avec des colonies isolées figurant dans le quatrième quadrant d'incubation à la suite à 30 ° C pendant 24 heures. Les trois autres quadrants afficher une croissance confluente dans lequel les cellules déposée sur la surface de la gélose au point en colonies qui se chevauchent.

Instruments Figure 2. Utilisé pour strie-technique de la plaque. De haut en bas, sont des cure-dents (montré aplati pas rond), une boucle de fil de fer, une boucle en plastique jetable et des bâtons. Cure-dents sont généralement transférésun récipient en verre petite à l'extrémité large en bas, puis recouverts d'une feuille lorsque autoclave pour stériliser avant utilisation. Bâtons de bois sont transférés à 18 tubes à essai, puis autoclavés mm pour stériliser avant utilisation.
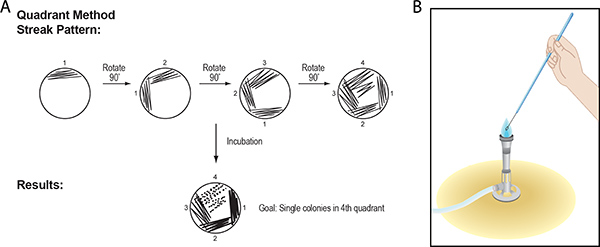
Figure 3. (A) Streak-technique de la plaque à l'aide de la méthode des quadrants. Une boucle de pré-stérilisés, un bâton ou cure-dent est utilisé pour étaler l'échantillon à travers un quart de la surface de la gélose avec une rapide, lisse, de va-et-quatrième mouvement de la jante au centre de la plaque. Cette action est répétée pour chacun des quatre quadrants de la plaque. Après l'incubation, la croissance des cellules apparaît sur le chemin de l'instrument utilisé pour déposer les cellules sur la plaque. La séparation mécanique des cellules dans un échantillon mixte utilisant cette technique devrait se traduire par des colonies isolées dans le quatrième quadrant (voir la figure 1 pour un exemple). Des colonies isolées sont considérés comme des unités formant colonie (ufc). (B) Quand une boucle en métal est utilisé pour strie de placage, il doit être stérilisé à l'aide de la flamme d'un brûleur Bunsen avant le contact avec l'inoculum ou le milieu gélose. Rappelons que la partie la plus chaude de la flamme est la pointe du cône bleu. En tenant le manche de l'instrument, placer le fil dans la flamme sur les 3-4 pouces de la boucle. Laissez suffisamment longtemps pour que le fil de devenir rouge. Déplacez le fil de sorte que la flamme s'approche de la boucle. Assurez-vous de la boucle métallique est refroidie avant de toucher l'inoculum.

Figure 4. Versez-technique de la plaque. (A) Un petit volume d'échantillon (entre 0,1 à 1,0 ml) est distribué de façon aseptique dans un plat vide, mais Petri stérile avec l'aide d'un 5,0 ml pipette sérologique. (B) agar fondu équilibrée à une température d'environ 48 ° C est ensuite versé dans la boîte de Pétri avec l'échantillon. Après la fermeture du couvercle, la plaque est doucement agité pour mélanger l'échantillon etgélose fondue. L'agar est autorisé à se solidifier pendant environ 30 minutes, puis les plaques sont inversées pour l'incubation.
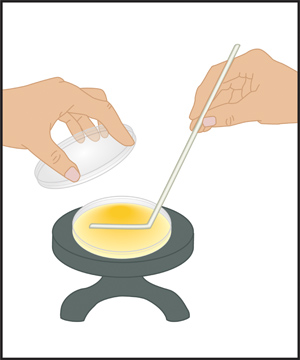
Figure 5. Spread-technique de la plaque avec une plaque tournante et d'un épandeur de verre. Après que la plaque gélose est placé sur un plateau tournant, un petit volume d'échantillon (0,1 à 0,2 ml) est distribué de manière aseptique sur le centre de la plaque à l'aide d'une micropipette. Barre d'écartement est stérilisé par trempage dans un bécher d'éthanol, puis faisant passer à travers la flamme du brûleur Bunsen pour enflammer l'excès d'éthanol. Avant de faire contact avec l'échantillon, l'épandeur doit être refroidi par le toucher de la gélose près du bord de la plaque. Barre d'écartement est légèrement déplacé d'avant en arrière à travers l'échantillon à travers la plaque tandis que le plateau tournant est tournant lentement. Cette action permet progressive mais encore la propagation de l'échantillon à travers la surface agar. Après la fermeture du couvercle, la plaque doit être réglé sur le u de paillassendisturbed pendant au moins 5 minutes pour permettre l'échantillon pour absorber complètement dans la gélose avant inversant la plaque pour l'incubation.
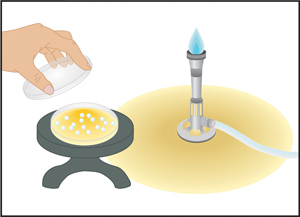
Figure 6. Spread-technique de la plaque avec des perles de verre (méthode Copacabana). Perles de verre qui ont été pré-stérilisés dans un autoclave sont versé sur la surface d'une plaque de gélose assis sur le dessus de table. Un petit volume d'échantillon (100 à 150 pi) est distribué aseptiquement sur le centre de la gélose en utilisant une micropipette. Avec le couvercle de la plaque de fermeture, un mouvement horizontal agitation est utilisé pour déplacer doucement les perles et d'autre de la plaque de 6 à 7 fois, étalement de l'échantillon. Cette action est répétée après rotation de la plaque 60 °. Le mouvement de secousse est répété une troisième fois suite à une autre rotation de 60 °. Une fois l'échantillon a complètement absorbé dans le milieu de gélose, les perles sont versés au loin dans un bécher contenant l'eau de Javel à 10%. Laplaques sont ensuite inversées pour l'incubation.
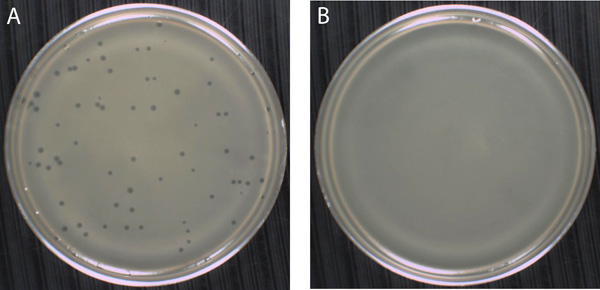
Figure 7. Technique de superposition Soft-agar utilisés pour isoler et dénombrer phage sur la base de la formation de plaques (aussi appelé un essai sur plaque). (A) La présence du phage peuvent être détectées comme des zones de compensation, ou des plaques, sur une suspension confluent de colonies bactériennes en croissance dans la gélose molle. Phage T4 est un virulent, phage d'ADN double brin qui infecte son hôte, Escherichia coli, causant des cellules hôtes pour lyser et libérer phage descendance. Après plusieurs tours de l'infection et une lyse, le voisin E. des cellules de E. dans la région immédiate entourant la cellule hôte infectée d'origine disparaît en laissant une plaque contenant des milliards de particules de phage T4. Phage T4 produit des plaquettes qui sont approximativement 1 mm de diamètre. Dans cette expérience, 200 pi d'une dilution 10 -5 de 2 x 10 8 pfu / ml de bouillon de Phage T4a été mélangé avec environ 300 pi de E. cellules indicatrices coli préparée comme une croissance exponentielle, la culture cellulaire à 37 ° C. La fois le phage et les bactéries ont été ajoutés à un tube EHA agar mou, mélangé, ensuite versée sur la surface d'une plaque de gélose EHA dur. Notez que ce n'était pas nécessaire pour permettre à des phages et les bactéries pour adsorber avant le placage dans ce cas. Après avoir laissé le agar mou à se solidifier au repos pendant 20 minutes, les plaques ont été inversées et incubées à 37 ° C pendant 24 heures. (B) En l'absence d'infecter des particules de phages, les résultats de la croissance bactérienne dans une suspension trouble de cellules dans la gélose molle dans laquelle les colonies discrètes ne sont pas visibles. Au lieu de cela, une pelouse, même des cellules bactériennes, dans ce cas E. coli, les formes tout au long de la couche agar mou ensemble.
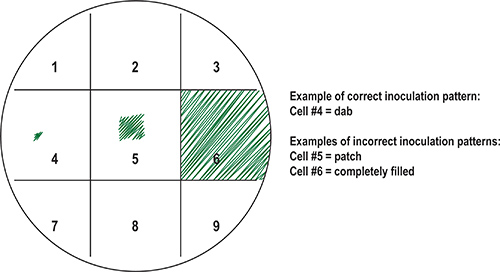
Figure 8. Préparation de la plaque primaire (maître) avec des échantillons bactériens. Pour conserver des échantillons organisés, le fond de la plaque peut être marqué sur une grille et des places numérotées résultant. Chaque échantillon peut être attribué un carré posé sur la grille. Montré en sont des exemples de schémas d'inoculation correctes par rapport incorrect. Idéalement, un petit nombre de cellules sont transférées dans le centre de la place en utilisant un outil d'inoculation stérile, comme une cure-dent pour "dab" l'échantillon (cellule n ° 4). Erreurs inoculation communs, tels que ceux décrits dans la cellule n ° 5 ("patch") et la cellule n ° 6 («remplissage»), le résultat en cas de prolifération des échantillons bactériens après incubation, par conséquent contaminer les places adjacentes.
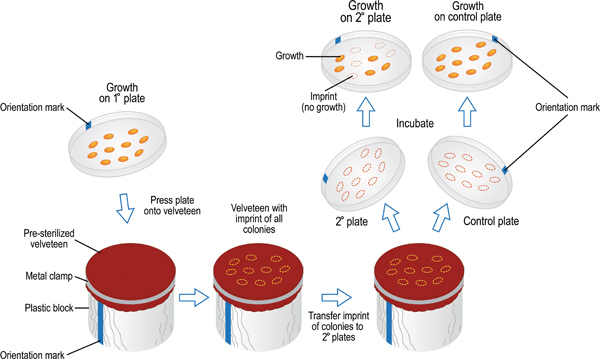
Figure 9. Replica-technique de la plaque utilisée pour transférer des cellules du primaire au secondaire pour plaques écrans phénotype. La marque sur la plaque primaire est aligné avec la marque sur le bloc de velours couverts, puis Lowe rouge pour permettre la surface de gélose à contacter la toile. Les cellules sont transférées à partir de la plaque à la légère, mais par velours régulièrement en appuyant sur la plaque primaire avec le bout des doigts. Cette action laissera une empreinte des échantillons de cellules sur le velours dans le même schéma spatial que la plaque principale. La même procédure est utilisée pour transférer des cellules de la velours sur une plaque secondaire. Comme beaucoup que 7-8 plaques secondaires peuvent être inoculés en utilisant l'impression même plaque principale sur le velours. La dernière plaque inoculé du velours devrait servir de contrôle positif. Il devrait être un milieu qui favorise la croissance de toutes les souches testées, en assurant le transfert de cellules suffisante eu lieu tout au long de toute la série de plaques. Après incubation, les plaques secondaires peuvent être inspectés et notés pour la croissance par rapport à une croissance nulle. Ainsi, plusieurs souches bactériennes peuvent être projetés simultanément sur des supports de croissance de plusieurs en une seule expérience.
_upload/3064/3064fig10.jpg "/>
Figure 10. Exemple de résultat en utilisant pour-technique de la plaque. Un échantillon de 1,0 ml d'eau recueillie à partir d'une fontaine publique a été distribué dans une boîte de Pétri stérile vide. Puis fondu, mais refroidi YTA a été versé dans la capsule avec l'échantillon. L'agar contenait également 100 pg / ml de cycloheximide pour empêcher la croissance des levures et moisissures qui peuvent avoir été présent dans l'échantillon d'eau. Après remuant doucement au mélange, la plaque a été posée sur une surface plane et l'agar-agar a été autorisé à se solidifier complètement. La plaque a été incubée à 37 ° C pendant 48 heures. Montré est le résultat de cette expérience. Notez la différence dans l'apparence des colonies de surface, qui sont grands et de forme circulaire, contre le sous-sol des colonies, qui sont très petits et irrégulièrement façonner parce que le milieu solidifié inhibe la propagation colonie en Afrique sub-surface.

Figure 11. </ Strong> Exemple de résultat en utilisant la propagation-technique de la plaque. La «méthode Copacabana" a été utilisé pour la plaque d'un mélange de cellules de E. coli pour une expérience de dépistage. Dans ce cas, le milieu de croissance (LB) contient X-Gal, de sorte que ces cellules exprimant une β-galactosidase fonctionnelles forment des colonies bleues tandis que les enzymes des cellules présentant une mutation dans le gène lacZ et donc incapables d'exprimer une forme de l'enzyme fonctionnelle β-galactosidase colonies blanches. Souvent considéré comme un «bleu / blanc écran", les deux types de colonies peuvent être facilement distingués les uns des autres sur la même plaque.

Figure 12. Exemple de résultat d'un essai sur plaque en utilisant une technique de superposition douce-agar. Montré sont des plaques formées sur la souche hôte Mycobacterium smegmatis mc 2 155 (ATCC 700084) par deux différents pHâges: (A) Destroyers mycobactériophage et (B) MSSS mycobactériophage. Ces phages ont été isolés par les étudiants dans le laboratoire de UCLA cours MIMG 103L au printemps 2010. M. smegmatis est un actinobactérie non-pathogènes et appartient à une famille des mycobactéries qui comprend quelques pathogènes connus pour provoquer des maladies graves telles que la tuberculose (M. tuberculosis, M. africanum, M. bovis) et la lèpre (Mycobacterium leprae). Environ 50 pi d'une dilution de 10 -2 destroyers et une dilution 10 -3 du MSSS chacun ont été incubées avec 500 pl de M. smegmatis pendant 20 minutes à 37 ° C, puis mélangé avec MBTA (agar mou) et versé sur MHA (agar dur) plaques. Après avoir laissé le agar mou à se solidifier au repos pendant 20 minutes, les plaques ont été inversées et incubées à 37 ° C pendant 48 heures. Notez les morphologies distinctes plaque produites par chaque phage. Destructeurs (A) forme petite (diamètre moyen d'environ 1 mm), des plages claires caractéristique d'un phage lytiquetandis que le MSSS (B) se développe "œil de bœuf» avec des centres de grande plaques claires entourées d'un halo trouble (diamètre moyen d'environ 3,2 mm). L'anneau brumeux peut être constituée de bactéries qui sont résistantes au phage infection. Ce modèle est distinct de celui formé par le phage lysogène, qui produisent des plaques turbides.
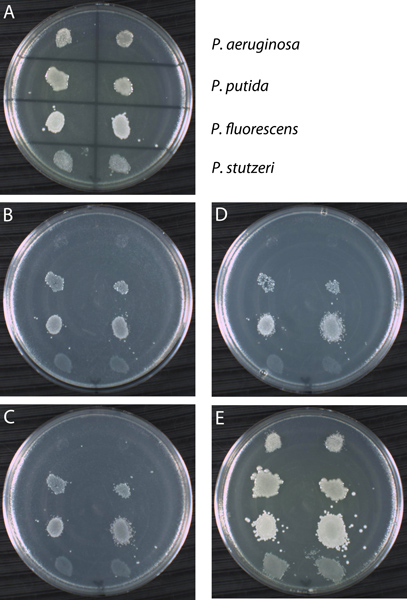
Figure 13. Exemple de résultat en utilisant réplique-plaque de la procédure. Quatre souches de Pseudomonas aeruginosa (P., P. putida, P. fluorescens et P. stutzeri) ont été testés en double exemplaire pour la croissance sur les trois différentes sources de carbone: acétamide, le lactose et la glycine. (A) La plaque primaire est un milieu complet (YTA) inoculés avec les quatre souches comme indiqué. Après incubation à 30 ° C pendant 24 heures, tous les quatre souches poussent sur YTA. La plaque principale a été utilisé pour la plaque réplique sur un minimum moidium (MSA) supplémenté avec une source de carbone unique: acétamide (B), du lactose (C), et la glycine (D). La dernière plaque de la série était un contrôle positif YTA plaque (E). Comme le montre, les souches présentent des modèles de croissance après l'incubation variables sur les plaques secondaires. Notez qu'il est parfois difficile de distinguer entre la croissance et une empreinte de cellules. Par exemple, comparer l'empreinte générée par P. stutzeri sur les plaques de MSA de trois à une croissance nulle sur les mêmes trois plaques par P. aeruginosa. Les deux sont des résultats négatifs par rapport aux modèles de croissance exposées par P. putida et P. fluorescens. Cependant, toutes les souches croître sur les cellules positives plaque de commande confirmant ont été transférés à tous les plaques secondaires dans la série. Les résultats de cette expérience sont présentées au tableau 1.
| YTA (Primaire) | MSA + acétamide | MSA + lactose | MSA + glycine | YTA (Contrôle) | |
| P. aeruginosa | + | - | - | - | + |
| P. putida | + | + | + | + | + |
| P. fluorescens | + | + | + | + | + |
| P. stutzeri | + | - | - | - | + |
Résumé Tableau 1. Des résultats de placage répliques. La croissance a indiqué que signe plus (+) et pas de croissance représenté comme un signe moins (-). YTA est un milieu complet (agar tryptone levure) et MSA est un milieu minimal (minimal agar sels). Les plaques de MSA ont été complétées avec une seule source de carbone comme indiqué.
Discussion
Culture de microorganismes implique un certain nombre de méthodes de revêtement, qui exigent tous des conditions d'asepsie que soit maintenue pendant toute la manipulation de cellules et les médias. Cinq procédures différentes ont été décrites dans le présent protocole. Bien que ces techniques de placage sont couramment utilisés pour manipuler les bactéries et les bactériophages, ils peuvent aussi être appliquée à la culture de cellules de mammifères et de micro-organismes eucaryotes couramment utilisé en génétique moléculaire comme la levure (c.-à-Saccharomyces cerevisiae, Candida albicans, Schizosaccharomyces pombe), les algues et les protozoaires ( c.-à-Volvox, Chlamydomonas, Amoeba, la paramécie), et les nématodes (c.-à-Caenorhabditis elegans). Il ya aussi de nombreux (et encore plus sophistiquée) des variations de chaque procédé de placage selon l'objectif expérimental ou de l'organisme à l'étude. Ainsi, il est important non seulement pour choisir la technique la plus appropriée pour un microorganisme donné l'expérience ou de la cible, mais aussi d'adapter la méthodologie t telschapeau les résultats expérimentaux s'occuper de manière appropriée la question de recherche ou un problème.
Quelques-unes des applications les plus courantes des techniques de placage abordés dans le présent protocole impliquent des avancées technologiques qui donnent des résultats à haut débit pour des expériences de découverte de dépistage et de drogues. Par exemple, centres de séquençage du génome d'utiliser la «méthode Copacabana" pour les bibliothèques clone propagation-placage, qui sont E. des cellules de E. transformé par des plasmides contenant des fragments d'ADN dérivées du génome d'un microorganisme. Parce que des dizaines de grandes plaques (appelé plateaux de dosage biologique) sont préparés à la fois, un système automatisé Agitateur de plaque est utilisée pour serrer les perles de verre pour le lot entier de plateaux. En outre, lors de la sélection des colonies de ces plaques après incubation, un sélecteur de colonie robotique est utilisée pour recueillir des cellules provenant de colonies appropriées que l'inoculum pour le bouillon LB dans 384 microplaques. Pour ce test de dépistage à haut débit, les principes de procédure de la propagation-ptechnique de la fin de s'appliquer, mais la technologie permet diverses mesures pour être automatisé et mis à l'échelle pour permettre à un grand nombre d'échantillons à analyser simultanément et dans un court laps de temps.
Les entreprises de biotechnologie et pharmaceutiques investissent des ressources considérables dans le développement de la technologie à haut débit pour les techniques les plus élémentaires de la microbiologie et la génétique moléculaire. Par exemple, il ya multi-canaux micropipettes pour effectuer les transferts de volume pour un maximum de 8 ou 12 échantillons à la fois. Il ya même des postes de travail robotiques qui manœuvre une tête 96-pipette à canaux! Ces efforts impliquent des équipes multidisciplinaires de chercheurs, biologistes appariement qui possèdent une expertise méthodologique avec les ingénieurs et les programmeurs informatiques, qui peuvent développer l'instrumentation nécessaire pour effectuer les opérations mécaniques associées aux expériences. Indépendamment de l'application de la recherche, l'objectif partagé par les entreprises en développement de ces technologies est le même - pour automatiser laboratoiresprocessus ry, outils, systèmes et instruments, les rendant moins de main-d'œuvre et plus efficace.
Déclarations de divulgation
Je n'ai rien à communiquer.
Remerciements
Un merci spécial à Cori Sanders aux conceptions IROC pour la préparation des illustrations et de Kris Reddi et Bhairav Shah à l'UCLA pour la mise en place des cultures d'échantillons et d'aider avec les chiffres. Le financement de ce projet a été fourni par HHMI (HHMI Grant n ° 52006944).
matériels
1. Yeast Tryptone Agar (YTA)
| Name | Company | Catalog Number | Comments |
| Yeast extract | 2.0 g | ||
| Tryptone | 10.0 g | ||
| Agar | 15.0 g | ||
| Distilled water | up to 1000.0 ml | ||
| pH 7.0 |
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
If preparing tubes for the pour-plate procedure, allow the agar to cool to ~55°C then add 2.0 ml of 50 mg/ml cycloheximide. Aseptically dispense 18.0 ml of the melted agar per 18 mm tube then store at 4°C. The agar will solidify and will need to be melted in a steamer or microwave prior to use.
2. Minimal Salts Agar (MSA) + 0.1% (w/v) carbon source
| NH4Cl | 1.0 g |
| NH2HPO4•2H2O | 2.14 g |
| KH2PO4 | 1.09 g |
| MgSO4•7H2O | 0.2 g |
| Carbon source* | 1.0 g |
| Trace salts solution** | 10.0 ml |
| Agar | 15.0 g |
| Distilled water | up to 1000.0 ml |
| pH 7.0 |
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
* Carbon sources used for experiments presented in Figure 13 include acetamide, lactose, and glycine.
** Trace salts solution is prepared in 0.1 N HCl as follows. It is added to the base before sterilization (autoclave at 121°C for 20 minutes).
| FeSO4•7H2O | 300.0 mg |
| MnCl2•4H2O | 180.0 mg |
| Co(NO3)2•6H2O | 130.0 mg |
| ZnSO4.7H2O | 40.0 mg |
| H2MoO4 | 20.0 mg |
| CuSO4•5H2O | 1.0 mg |
| CaCl2 | 1000.0 mg |
| HCl (0.1 N) | up to 1000.0 ml |
3. EHA soft agar (0.65 % w/v)
| Agar | 6.5 g | |
| Tryptone | 13.0 g | |
| NaCl | 8.0 g | |
| Na Citrate•2H2O | 2.0 g | |
| Glucose | 3.0 g | |
| Distilled water | up to 1000.0 ml |
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
4. EHA hard agar (1.2% w/v)
| Agar | 12.0 g |
| Tryptone | 13.0 g |
| NaCl | 8.0 g |
| Na Citrate•2H2O | 2.0 g |
| Glucose | 3.0 g |
| Distilled water | up to 1000.0 ml |
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
5. 1X Middlebrook Top Agar (MBTA soft agar, 0.5% w/v)
| 100 mM CaCl2 stock* | 1 ml |
| 7H9 liquid medium: Neat ** | 50 ml |
| 2XTA *** | 50 ml |
Melt 50 ml of 2XTA and allow it to cool to ~55°C. Using aseptic technique, add the CaCl2 and 7H9 broth to the melted agar. Aseptically dispense 4.5 ml of the mixture per 13 mm tube and store in a 55°C incubator ≤7 days. Cooling MBTA to room temperature or 4°C will cause the CaCl2 to precipitate out of solution.
* 100 mM CaCl2. stock must be stored at room temperature to prevent CaCl2 from precipitating out of solution.
** 7H9 liquid medium: Neat
| 7H9 broth base | 4.7 g |
| 40% glycerol stock | 5 ml |
| Distilled water | up to 900.0 ml |
Mix the base with water then add the glycerol while stirring. Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
*** 2X Middlebrook Top Agar (2XTA, 1.0% w/v)
| 7H9 broth base | 4.7 g |
| Agar | 1.0 g |
| Distilled water | up to 1000.0 ml |
Autoclave at 121°C for 20 minutes to sterilize. Dispense 50 ml aliquots into 100 ml bottles and store at 4°C.
6. Middlebrook 7H10 Agar Plates (MHA hard agar, 1.9% w/v)
| 7H10 agar base | 19.0 g |
| 40% glycerol stock | 12.5 ml |
| Distilled water | 887.5 ml |
Mix the agar base with water then add the glycerol while stirring. Heat the solution to boiling then stir for one minute to completely dissolve the base powder. Autoclave at 121°C for 20 minutes to sterilize. Allow the agar to cool to ~55°C then aseptically add the following reagents:
| AD supplement (pre-warmed to 37°C)* | 100 ml |
| 50 mg/ml Carbenicillin ** | 1.0 ml |
| 10 mg/ml Cycloheximide ** | 1.0 ml |
* AD supplement
| NaCl > | 17 g > |
| Albumin (Fraction V)> | 100 g> |
| Dextrose (D-Glucose)> | 40 g> |
| Distilled water> | up to 2000.0 ml> |
Filter-sterilize this solution; do not autoclave. Store at 4°C.
** Filter-sterilize and store these solutions at 4°C for ≤60 days.
7. LB agar (1.5% w/v) + X-Gal (60 μg/ml)
| Tryptone | 10.0 g |
| Yeast extract | 5.0 g |
| NaCl | 10.0 g |
| Agar | 15.0 g |
| Distilled water | up to 1000.0 ml |
| pH 7.5 at 25°C |
Autoclave at 121°C for 20 minutes to sterilize. Allow the agar to cool to ~55°C then aseptically add 3.0 ml of 20 mg/ml X-Gal solution. Freshly prepare X-gal stock by dissolving 400 mg X-Gal in 20 ml dimethylformamide (DMF).
Table of specific reagents:
| Name of the reagent | Company | Catalogue number |
| Yeast extract | Becton Dickenson | 212750 |
| Tryptone | Becton Dickenson | 211705 |
| Agar | Becton Dickenson | 214030 |
| NH4Cl | Acros Organics | 123340010 |
| NH2HPO4•2H2O | Sigma-Aldrich | 30435 |
| KH2PO4 | Fisher Scientific | BP 303-500 |
| MgSO4•7H2O | Sigma-Aldrich | 230391 |
| Acetamide | Sigma-Aldrich | A-0500 |
| Lactose | Fisher Scientific | L6-500 |
| Glycine | Sigma-Aldrich | G-7126 |
| FeSO4•7H2O | Sigma-Aldrich | F8048 |
| MnCl2•4H2O | Sigma-Aldrich | M-3634 |
| Co(NO3)2•6H2O | Sigma-Aldrich | 230375 |
| ZnSO4.7H2O | Sigma-Aldrich | Z4750 |
| H2MoO4 | Acros Organics | 213621000 |
| CuSO4•5H2O | Sigma-Aldrich | 209198 |
| CaCl2 | Sigma-Aldrich | C1016 |
| HCl | Fisher Scientific | A144-212 |
| Cycloheximide | Sigma | 038K1561 |
| Carbenicillin | Cellgro | 46-100-RG |
| NaCl | Fisher | S271-1 |
| Na Citrate•2H2O | Fisher | S279-500 |
| Glucose (Dextrose) | BD | 215530 |
| 7H9 broth base | BD | 271310 |
| 7H10 agar base | BD | 262710 |
| Glycerol | Shelton | IB15760 |
| Albumin (Fraction V) | Fisher | S71907 |
| X-Gal (5-bromo-4-chloro-3-indolyl-β-D-galactopyranoside) | Teknova | X1205 |
| Dimethylformamide (DMF) | Sigma | D4551 |
| Ethanol | Fisher | CDA19 |
| Chlorine Bleach | Chlorox | 02490/06644884 |
| CiDecon (disinfectant) | Decon Laboratories, Inc. | 8504 |
Table of specific equipment:
| Name of equipment | Company | Catalogue number | Experiment |
| Metal loops | American Educational Products | S17352 | Streak plating |
| Disposable plastic loops | Fisher | 22-363-602 | Streak plating |
| Wooden sticks | Fisher | 23-400-104 | Streak plating |
| Flat Toothpicks | American Educational Products | S67859 | Streak plating |
| Turn tables | Fisher | 08-758Q | Spread plating |
| Glass rods | Bellco Glass | NC9004380 | Spread plating |
| 4 mm glass beads | Fisher | 11-312B | Spread plating |
| Velveteen cloth | Bel-Art Products | 09-718-2 | Replica plating |
| Cylindrical block | Bel-Art Products | 09-718-1 | Replica plating |
Références
- Barker, K. . At the Bench: A Laboratory Navigator. , (1998).
- US Department of Health and Human Services (DHHS). . Biosafety in Microbiological And Biomedical Laboratories (BMBL). , (2009).
- Dulbecco, R., Vogt, M. Some problems of animal virology as studied by the plaque technique. Cold Spring Harbor Symp. Quant. Biol. 18, 273-279 (1953).
- Fields, B. N., Knipe, D. M., Chanock, R. M., Hirsch, M. S., Melnick, J. L., Monath, T. P., Roizman, B. . Fields Virology. 1, (1991).
- Gilbert, R. A., Ouwerkerk, D., Zhang, L. H., Klieve, A. V. Cooperative Research Center for Beef Genetic Technologies. In vitro detection and primary cultivation of bacteria producing materials inhibitory to ruminal methanogens. Journal of Microbiological Methods. 80, 217-218 (2010).
- Jett, B. D., Hatter, K. L., Huycke, M. M., Gilmore, M. S. Simplified agar plate method for quantifying viable bacteria. BioTechniques. 23, 648-650 (1997).
- Karam, J. D. . Molecular Biology of Bacteriophage T4. , (1994).
- Knipe, D. M., Howley, P. M., Griffin, D. E., Lamb, R. A., Martin, M. A., Roizman, B., Straus, S. E. . Fundamental Virology. , (2001).
- Lederberg, J., Lederberg, E. M. Replica plating and indirect selection of bacterial mutants. J. Bacteriol. 63, 399-406 (1952).
- Miller, J. H. . A Short Course in Bacterial Genetics: A Laboratory Manual and Handbook for Escherichia coli and related bacteria. , (1992).
- Mills, K. V., Gareau, J. R., Garcia, A. M. Low-cost modification to the Copacabana method for spreading transformation mixtures. BioTechniques. 39, 188 (2005).
- Jordan, T., et al. . RESEARCH: National Genomics Research Initiative Phage Resource Laboratory Manual. , (2008).
- Sambrook, J., Russell, D. W. . Molecular Cloning - A Laboratory Manual. , (2001).
- Sanders, E. R., Miller, J. H. I. . Microbiologist: A Discovery-based Course in Microbial Ecology and Molecular Evolution. , (2010).
- Slonczewski, J. L., Foster, J. W. . Microbiology - An Evolving Science. , (2011).
- Teather, R. M., Wood, P. J. Use of Congo Red-Polysaccharide Interactions in Enumeration and Characterization of Cellulolytic Bacteria from the Bovine Rumen. Applied and Environmental Microbiology. 43, 777-780 (1982).
- Wise, K. . Preparing Spread Plates Protocols. , (2006).
- Worthington, M., Luo, R. Q., Pelo, J. Copacabana method for spreading E. coli and yeast colonies. BioTechniques. 30, 738-742 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon