Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Протокол для использования гена набор обогащения анализ для выявления соответствующих животных модель для трансляционного исследования
В этой статье
Резюме
Мы предоставляем стандартизированный протокол для использования анализа набора обогащения гена транскриптомики данных для определения идеальной мыши модель для трансляционного исследования.
Этот протокол может использоваться с microarray ДНК и РНК последовательности данных и далее может быть распространено на другие данные омику, если доступны данные.
Аннотация
Недавние исследования, которые сравнивали транскриптомики наборов данных заболеваний человека с наборами данных из моделей мышей с использованием традиционных гена в геном Сравнение методов привело к противоречивым выводам относительно актуальности животных моделей для поступательного исследования. Основная причина расхождений между различными ген выражение анализа является произвольным фильтрация дифференциально выраженной генов. Кроме того сравнение единичных генов между видами и платформ часто ограничивается техническим дисперсия, ведущих к неправильному толкованию кон/несоответствие между данными от человека и животных моделей. Таким образом необходимы стандартизированные подходы для анализа систематических данных. Для преодоления субъективного гена фильтрации и неэффективной гена в геном сравнений, мы недавно продемонстрировали, что ген анализ набора обогащения (GSEA) имеет потенциал, чтобы избежать этих проблем. Поэтому мы разработали стандартный протокол для использования GSEA проводить различие между соответствующими и неуместные животных моделей для трансляционного исследования. Этот протокол не подходит предсказать как разработать новые модели систем априори–, как это требует существующих экспериментальных омику данных. Однако протокол описывает, как интерпретировать существующих данных в стандартной форме для того, чтобы выбрать наиболее подходящую модель животных, таким образом избегая ненужных экспериментов на животных и заблуждение трансляционного исследования.
Введение
Животные модели широко используются для изучения болезней человека, из-за их предполагаемого сходства для людей с точки зрения генетики, анатомии и физиологии. Кроме того Животные модели часто служат в качестве привратников по клинической терапии и может иметь огромное влияние на успех трансляционного исследования. Тщательный подбор оптимальной модели на животных может уменьшить количество заблуждение исследований на животных. Недавно актуальность животных моделей для трансляционного исследования состязательно обсуждался, особенно потому, что Анализируя же наборы данных, полученные из воспалительных заболеваний человека и связанных с ними мыши модели привело к противоречивым выводам 1,2. Эта дискуссия показала фундаментальной проблемой во время анализа данных омику: стандартизированные подходы для анализа систематических данных необходимы для того, чтобы уменьшить предвзятым гена выбор и увеличить надежность межвидовые сравнения 3.
Традиционно, анализ данных transcriptomics (и другие данные омику) делается на уровне одного гена и включает в себя первый шаг гена отбора на основе строгих отключения параметров (например, изменение раза > 2.0, значение p < 0,05). Однако настройка параметров первоначального производства часто является субъективной, произвольным и не биологически оправданной и может даже привести к противоположным выводам1,2. Кроме того первоначальный гена выбор обычно ограничивает анализ несколько высоко вверх - и downregulated генов и таким образом не достаточно чувствительны, чтобы включать большинство генов, которые высказывались дифференцировано в меньшей степени.
С ростом эпоху геномики в начале 2000-х и увеличение знания биологических путей и контексты были разработаны альтернативных статистических подходов, которые позволили обойти ограничения уровня анализа одного гена. Джин набор обогащения анализа (GSEA)4, который является одним из широко признанных методов для анализа данных, transcriptomics, делает использование групп a-priori определенных генов (например, сигнальные пути, проксимальной расположение на хромосоме и т.д.). GSEA сначала карты всех обнаруженных нефильтрованное генов в предполагаемой гена наборы (например, пути), независимо от их отдельных изменений в выражении. Таким образом этот подход также включает умеренно регулируется генов, которые в противном случае будут потеряны с уровня анализа одного гена. Аддитивные изменения выражения внутри ген наборы впоследствии выполняется с помощью работает сумма статистики.
Несмотря на его широкое применение в медицинских исследованиях GSEA и соответствующий набор обогащения подходы не учитываются само собой разумеется для анализа сложных омику данных. Здесь мы описываем протокол для сравнения данных омику из человеческих образцов с теми, от моделей мышей с целью выявления идеальная модель для трансляционного исследования. Мы демонстрируем применимости Протокола, основанный на коллекцию моделей мышей, которые используются для изображая человека воспалительных заболеваний. Однако этот анализ трубопровод не ограничивается сравнений человека мыши и допускающем дальнейшего исследования вопросов.
протокол
1. Скачать молекулярных подписей базы данных и программного обеспечения GSEA
- перейдите на официальный сайт широкий институт GSEA (http://software.broadinstitute.org/gsea/index.jsp) и зарегистрируйтесь, чтобы получить доступ к программному обеспечению GSEA инструмент и молекулярных подписи базы данных (MSigDB).
- Скачать javaGSEA настольных приложений или вариант альтернативного программного обеспечения (например, сценарий R).
Примечание: Все варианты реализации точно тот же алгоритм. GSEA программное обеспечение свободно доступно лицам в научных кругов и промышленности для целей внутреннего исследования. - Далее подробную информацию о GSEA программного обеспечения, зайдите на веб-сайт документации (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Main_Page) и руководство пользователя GSEA (http://software.broadinstitute.org/gsea/doc/ GSEAUserGuideFrame.html).
- скачать молекулярных подписи базы данных (MSigDB) с сайта GSEA, чтобы получить доступ к коллекции множества отдельных генов.
Примечание: MSigDB представляет собой совокупность аннотированный гена наборов для использования с GSEA программного обеспечения или других целей. Джин наборы могут быть разделены согласно сигнальные пути, генная онтология термины, СНГ регуляторные мотивы, экспериментальный подписей и другие. Гены от MSigDB всегда именуются по их официальным символом гена ХЬЮГО (Организация генома человека). Для сравнения пути регулирования между данного человека расстройства и различные мыши модели рекомендуется скачать ' все канонические пути, Джин символы ' файл (c2.cp.v5.2.symbols.gmt). Этот файл включает гена наборы, которые были аннотацией и организованы в сигнальных путей KEGG 5 , 6, Reactome , 7 8 и BioCarta 9. строка ' v5.2 ' представляет сведения о версии коллекции. Убедитесь в том загрузить последнюю версию файлов. MSigDB свободно доступен для лиц в научных кругов и промышленности для целей внутреннего исследования. Это не требуется для загрузки MSigDB, если подключение к Интернету предоставляется в ходе анализа. В этом случае MSigDB может быть выбран непосредственно в пользовательском интерфейсе GSEA. - Скачать ДНК чип (массив) аннотации файлы с веб-сайта GSEA для перевода зонд массив конкретных идентификаторов для общего Уго гена символы (например, Mouse430_2.chip).
Примечание: Это не нужно скачать чип аннотации ДНК, если подключение к Интернету предоставляется в ходе анализа. В этом случае ДНК чип аннотации могут быть выбраны непосредственно в интерфейсе пользователя GSEA. Протокол также может использоваться с РНК последовательности данных. В этом случае она не требуется для загрузки файлы заметок. Вместо этого, используйте средство preranked GSEA для анализа данных выражение гена (см. шаг 4.12).
2. Скачать экспериментальные данные выражения гена для человека расстройство и соответствующие модели животных
- выявлять экспериментальные ген выражение (transcriptomics) исследования для человека расстройство выбора (например, выражение гена профили лейкоциты, производные от больных с септическим расстройства, GSE9960). Модели
- аналогично, поиск нескольких животных, которые должны сопоставляться с человека исследования (например, профили выражение гена кровяных клеток, полученных от мышей после инъекции золотистый стафилококк (S. aureus), GSE20524). На этом этапе использовать предварительных знаний для предыскание Животные модели, которые могут быть пригодны для изображая человека ситуации.
- Для этой цели относятся к литературе и баз данных, таких как Омнибус выражение гена (GEO) базу данных 10 или 11 ArrayExpress и скачать нормализованных transcriptomics данных, представляющих интерес. Сохраните данные в виде текстовых файлов на локальном жестком диске. Для базы данных ГЕО рекомендуется скачать текстовых файлов с разделителями табуляциями серии матрицы. Также принять к сведению платформы (тип массива) используется для этого исследования, поскольку эта информация необходима для преобразования массива конкретных зонд идентификаторы для общего Уго гена символы.
Примечание: Обеспечить достаточно памяти для хранения данных, как transcriptomics наборы данных обычно составляют несколько сотен MB.
3. Обработки и форматирования данных
- перед импортом данных экспериментальных ген выражение в GSEA программное средство, рассмотрим структуру требуемых данных. Для каждого исследования вручную создать два разных файла: 1) ген выражение данных файл, содержащий значения измерений для различных генов и образцы и 2) файл фенотип, содержащий образец этикетки для группировки отдельных образцов (например, для обработки групп).
Для дальнейших деталей и структуру данных параметры перейдите к странице формат данных GSEA (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Data_formats).
Примечание: Как правило, все формы transcriptomics данных совместимы с протоколом, включая эксперименты microarray ДНК, РНК seq или чип seq исследования. В случае использования ДНК microarray эксперименты, файл данных выражение гена должен содержать идентификатор массива конкретных зонд или Уго гена символы для каждого гена (зонд идентификаторов будет переводиться на Уго гена символы во время анализа, см. шаги 1.5 и 4.10). В случае использования РНК seq или чип seq данных, вручную вычисляемое групповых метрик для ген выражение данных (например, среднее соотношение группы) должна использоваться вместо отдельных образцов данных. Эти метрики группы затем должны быть проанализированы с помощью инструмента preranked GSEA (см. шаг 4.12). Данные выражения гена должны быть нормализованы как обычно перед импортом в программное обеспечение GSEA. Тип нормализации (например, квартиль или Кубический сплайн) обычно слева для исследователя. - Ген выражение данных файла: используйте формат табуляцией текстового файла (*.txt) для описания набора данных выражение, как показано на рисунке 1A. Смотрите также поддерживается пример файла GSE20524_expression.txt.
Примечание: Файл данных выражение гена содержит значения выражения для всех обнаружению генов (или зондов), также для генов, которые не могут быть выражены дифференцированно. Поэтому файл обычно состоит из многих тысяч генов. Она организована как изображено на рисунке 1A. Первая строка содержит имя метки (например, ген символ или зонд ID) следуют идентификатор для каждой выборки в наборе данных (например, образец 1, образец 2 и т.д.). Остальная часть файла содержит выражение значения для каждого из генов и для каждой выборки в наборе данных. GSEA программное средство выполняет вычисления для групповых метрик (например, среднее соотношение группы или сигнал шум коэффициент), поэтому рекомендуется включать данные для каждого индивидуального образца. В качестве альтернативы, можно использовать внешне рассчитанные групповые метрики для данных выражение гена (см. рис. 1B). - Фенотип файла: Создайте отдельный файл для определения и маркировки группы составляют отдельные образцы, как изображено в < сильного класса= «xfig» > рисунок 2. Использование пробелов или знаков табуляции для разделения полей. Сохраните в формате файла CLS (определение класса C++). Смотрите также поддерживается пример файла GSE20524_pheno_infection.cls.
Примечание: Первая строка содержит общее количество образцов и далее количество групп ( рис. 2). В то время как количество образцов должен соответствовать файлу данных выражение гена (см. 3.2), количество групп зависит от дизайна исследования. Третье поле первой строки всегда ' 1 '.
Вторая строка в файле CLS содержит имя для каждой группы. Строка должна начинаться с знак фунта (#), пробел ( рис. 2).
Третья строка содержит метку группы для каждого образца. Метка группы может быть произвольное число или текст. Это только порядке меток определяет ассоциацию каждого образца для групп: Первая метка присваивается первой группы на второй линии; Второй уникальной метки присваивается второй группы и так далее. Убедитесь, что каждый образец той же группы имеет тот же знак на этот шаг, и что количество этикеток является таким же, как количество выборок, указанный в первой строке. Наконец, сохраните файл как табуляцией текстовый файл (*.txt) и вручную изменить расширение имени файла (*.cls). - -(необязательный) файлы базы данных набор генов: определить наборы пользовательских ген. Используйте формат файла с разделителями табуляциями GMT (ген матрица транспонированы) для наборов гена, как изображено на рисунке 3. Также увидеть поддерживаемые пример файла Gene_sets_Inflammation_BIOCARTA_KEGG_REACTOME.gmt.
Примечание: Определение наборов пользовательских ген может быть полезным например ограничить ген набор обогащения анализ путей особый интерес (например, иммунологии, сигнализации для исследования сепсис), или de novo, определение наборов собственных генов (например, активации и Ингибирует генов в исследованиях, которые должны сопоставляться). Файл организован как изображено на рисунке 3. В формате GMT каждая строка представляет набор генов ( рис. 3). Каждый набор генов описывается имя, описание и гены в наборе ген. Первый столбец содержит имена наборов уникальных генов. Вторая строка может содержать описание набора генов. Следующие столбцы содержат имена гена (официальный Уго гена символы) соответствующего набора генов. Наконец, сохраните файл как вкладка текстового файла с разделителями (*.txt) и вручную изменить расширение имени файла для (* .gmt).
4. Выполнение GSEA
- Открыть средство программного обеспечения GSEA (см. 1.2).
- Нажмите ' загрузки данных ' кнопку в левой части главного окна ( рис. 4A). Новая вкладка будет открыта для импорта необходимых данных файлов ( рис. 4В). Просмотр в новой вкладке в файл данных (*.txt) выражение гена (см. 3.2), файл фенотип (*.cls) (см. 3.3) и, при необходимости, чтобы наборы пользовательских гена (* .gmt) файл ( рис. 4B).
- В случае, если GSEA не удается подключиться к Интернету, также загрузки загруженный MSigDB (* .gmt) файлы (например, c2.cp.v5.2.symbols.gmt для пути, см. 1.4) и ДНК чип аннотации (массив) (* .chip) файлы (например, Mouse430_2.chip, разделе 1.5). Успешно импортированных данных появляются в ' загрузки данных ' секции ( рис. 4 c).
Примечание: Каждое исследование выражения Джин должны быть проанализированы с GSEA индивидуально. Сравнение между двух исследований (например, человека расстройство против модель мыши) будет осуществляться на этапе 5.
- В случае, если GSEA не удается подключиться к Интернету, также загрузки загруженный MSigDB (* .gmt) файлы (например, c2.cp.v5.2.symbols.gmt для пути, см. 1.4) и ДНК чип аннотации (массив) (* .chip) файлы (например, Mouse430_2.chip, разделе 1.5). Успешно импортированных данных появляются в ' загрузки данных ' секции ( рис. 4 c).
- Нажмите ' работать GSEA ' кнопку в левой части главного окна. Новая вкладка будет открыта для того, чтобы задать параметры для анализа ( рис. 4 d). Вкладка разделена на три части: Расширенные поля обязательные поля, основные поля и.
- В обязательные поля, сначала выберите выражение набора данных загружаются в шаге 4.2 ( рис. 4 d).
- Выбрать гена устанавливает базу данных, подключенной веб-сайта или из файла набора вручную импортированные гена ( рис. 4 d).
- Изменение фенотипа этикетки для выбора групп образцов, которые должны сравниваться друг с другом (например, S. aureus лечение против здорового управления) ( Рисунок 4 d).
- Распада dataset гена символы (= true) для того, чтобы перевести идентификаторы зонд в выражение набора данных для официальных Уго гена символы, используемые в базе наборы генов. Выберите значение false, если выражение dataset уже содержит ХЬЮГО гена символы ( рис. 4 d).
- Задать значение по умолчанию количество перестановок для 1000 ( рис. 4 d).
Примечание: Для более высоких чисел вычисляя время будет значительно увеличить. - Изменить тип перестановки в ' гена набор ', так как пермутирование фенотип рекомендуется только в тех случаях, когда существует более чем семь образцов в каждом фенотип ( рис. 4 d).
- Наконец, выберите чип платформой, используемой для генерации данных выражение гена, от подключенных веб-сайта или вручную импортированные ДНК чип файл аннотации (массив) ( Рисунок 4 d).
Примечание: Этот шаг необходим только, если зонд идентификаторы используются в наборе загруженные выражение. - В основных областях редактировать по крайней мере разделе анализ имя и сохранить результаты в этой папке, чтобы снова найти файл результатов ( рис. 4 d). Кроме того дополнительные статистические параметры могут быть изменены. Для дальнейших деталей на параметры и в разделе Расширенные поля, пожалуйста, перейдите на GSEA руководство пользователя (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html).
- (Опционально): В случае внешне рассчитанные групповые метрики для экспрессии генов, данные (например, среднее соотношение группы) должны использоваться вместо отдельных образцов данных, используйте средство preranked GSEA. Будет затем проведен анализ основан на простой список генов, назначен с предварительно вычисленные групповой метрики, которые используются для ранжирования генов. После загрузки файла выражения альтернативных гена перейти к главной навигации и щелкните дальше Инструменты/GseaPreranked. Аналогичным образом, Новая вкладка будет открыта для настройки параметров для анализа ( Рисунок 4E).
Примечание: Рекомендуется использовать средство preranked GSEA для исследований, которые не имеют отдельные примеры конкретных генов выражение данных. Это может быть случай, если специальные статистические данные или нормализации процедуры выполнялись на данных приводит к группе средние значения вместо отдельных образцов данных. Рекомендуется использовать средство preranked GSEA РНК последовательности данных. Нормализации данных выражение последовательности РНК и вычисления групповых метрик для образцов (например, журнал изменений раза), которые могут использоваться для ранжирования согласно их выражение генов. - Нажмите ' запустить ' кнопку на правой нижней части окна.
Примечание: Анализ затем может занять до нескольких минут в зависимости от скорости вычислений. Следить за ходом анализа в разделе Отчеты GSEA в левой нижней части окна. После окончания анализа, статус ' успех ' отображается в разделе Отчеты GSEA. - Нажмите на успешном анализ в разделе отчеты GSEA, чтобы открыть результаты анализа.
Примечание: Новое меню навигации откроется в окне браузера, который суммирует все результаты и параметров настройки ( Рисунок 5). Включает верхние два раздела меню навигации Джин набор обогащения результаты для определенных групп населения (например, обогащения в S. aureus рассматриваются примеры или образцы здорового управления). Первые строки обоих разделов Показать резюме статистика результатов. Джин наборы, которые значительно обогатили со скоростью ложь открытие (ФДР) ниже 25% рассматриваются как обогащенный следующее толкование. Более подробная информация о толковании анализа можно найти в руководстве пользователя GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html). - Нажмите на подробные обогащения результаты в excel формат для экспорта результатов анализа в электронную таблицу ( рис. 6A). Экспортировать результаты подробного обогащения в excel отдельно для обоих фенотипов ( рис. 5) и присоединиться к результатов данных в одном файле электронной таблицы. Для последующего сравнения между ген выражение данные нескольких исследований, поддерживать по крайней мере имя набора генов (столбец A), его нормализованных обогащения Оценка (РЭШ) (столбец F) и его значение ДРД (ложные обнаружения курс) (колонка H) ( Рисунок 6B ).
Примечание: Файл электронной таблицы содержит огромные данные для каждого из проанализированных гена набор, в том числе имя набора генов (столбец A), его размер (то есть, количество генов, обнаруженных в данных, выражение гена, столбец D), его нес (количественная мера направления и степени обогащения, столбец F), его значение номинального p (некомпенсированная, столбец G) и его значение ДРД (исправлениями для нескольких гипотез, колонка H). Для дальнейших сведений о толковании, пожалуйста, обратитесь к руководству пользователя GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html). - Повторить набор ген анализ обогащения (шаги 4.1 до 4.15) для второго исследования (например, S. aureus GSE9960) и для всех дальнейших исследований, которые должны сравниваться друг с другом. Включать столько человеческих клинических исследований и различных мыши модели можно определить оптимальный мыши модель для трансляционного исследования вопрос.
5. Сравнение результатов GSEA
- для определения оптимальной модели животных подражая сравнить человека ситуация GSEA результаты всех исследований друг к другу. Использовать результаты обогащения и ФДР значения для классификации пути (ген наборы) активирована (РЭШ > 0, ФДР < 25%), ингибированный (РЭШ < 0, ФДР < 25%) или ни один из обоих (ФДР > 25%). Для каждого сравнения двух исследований, подсчитать количество реализаций девяти возможных комбинаций пути регулирования как указано таблицей непредвиденных 3 x 3 ( рис. 7A).
- Оценить взаимосвязь между двух исследований путем расчета прогностичность положительного (ppv) и предсказательная ценность отрицательного результата (npv), который по определению является частью пути, которые показывают же регулирование (активированный или препятствует) в двух исследованиях .
- Расчет ppv и npv согласно следующей формулы (1) и (2):
(1)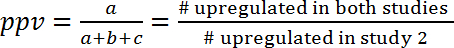
(2)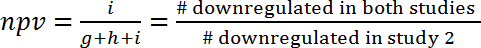
Примечание: Поскольку перекрытие может быть чисто coincidental, ppv и npv должны быть далее по сравнению с случайно ожидаемых значений. Этот подход позволяет Оценка количество информации, которые могут быть получены от одного исследования для прогнозирования последствий в другом исследовании. Например если регулирование процессов в две модели являются независимыми друг от друга (и только пересекаются случайно), и если в первой модели 10% путей были upregulated, чем ppv к Вторая модель будет также 10% и не было никаких дополнительных прирост inf информацию. С другой стороны если обе модели были связаны общих механизмов регулирования, то ppv (и npv) будет значительно больше, чем ожидалось, случайно. Например, для прогнозирования изменения выражения гена во время человека сепсис (GSE9960) от эффектов в мышиных инъекции модели S. aureus (GSE20524), ppv-43% (6/(6+8+0)) и npv является 61% (11/(0+7+11)). Другими словами 43% активированного путей в мышиных S. aureus инъекции модели (GSE20524) также активируются во время человека сепсис (GSE9960). Аналогичным образом, 61% тормозится путей в мышиных S. aureus инъекции модели (GSE20524) также мешают во время человека сепсис (GSE9960) ( рис. 7B). PPV и ЧПС также могут быть определены для обратной Созвездие (это означает, предсказывая исследования 1 для изучения 2).
- Расчет ppv и npv согласно следующей формулы (1) и (2):
- Для вычисления перекрытие случайно обратитесь к таблице случай 3 x 3 ( рис. 7) и вычисления ppvchance и npvchance согласно следующей формулы (3) и (4):
(3) < img alt = «Уравнение 3» src = «/files/ ftp_upload/55768/55768eq3.jpg» / >
(4)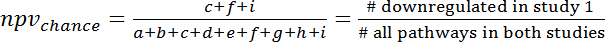
Примечание: например, для прогнозирования изменения выражения гена во время человека сепсис (GSE9960) от эффектов в мышиных S. aureus инъекции модель (GSE20524) ppvchance-13% (8/64) и npvchance и 22% (14/64). - Рассчитать прирост ppv против шанс путем вычитания ppvchance от ppv. Рассчитать соответственно для npv:
(5)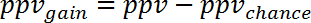
(6)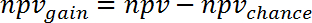
Примечание: например, для прогнозирования гена Выражение изменяется во время человека сепсис (GSE9960) от эффектов в мышиных инъекции модели S. aureus (GSE20524) изменения в ppv и npv против шанс-+ 30% (43% - 13%), + 39% (61% - 22%), соответственно. - Рассчитать прирост информации, которые могут быть получены из исследования 2 относительно изучения 1 путем усреднения ppvgain и npvgain:
(7)
- использовать резервный таблицы, определенной на шаге 5.1 пару исследований (study1.pathway, study2.pathway), чтобы вычислить значение p Хи test.
Хранить данные таблицы на случай непредвиденных обстоятельств в матрице X. Perform хи-квадрат тест, например, путем использования R функционировать chisq.test.
Примечание: например, сравнивая исследование выбранного человека сепсис (GSE9960) с мышиных S. aureus инъекции модель (GSE20524) показывает статистически значимого совпадения в регуляции воспалительной путь:
> chisq.test(X,simulate.p.value=F)$ p.Value
3.82e-07
6. Определение оптимальной модели животных
- GSEA сравнить результаты для всех комбинаций этих исследований, которые были отобраны для анализа.
Примечание: Также рекомендуется для сравнения (аналогичные) человека исследования друг с другом, а также различные исследования на животных друг к другу. Это сравнение может обеспечить понимание внутривидовых дисперсию клинические исследования (или заболевания) и различные животные модели. Ожидается, что клинические исследования должна показать получить приемлемого совпадения и важную информацию, потому что в противном случае клинические исследования может быть слишком разнородные найти животных модель, которая может имитировать человека ситуации. В этом случае рекомендуется включать только человеческие исследования, которые похожи друг на друга для выявления подходящих животных моделей. - Сортировать все комбинации прирост информации (шаг 5.5). Для сравнения несколько наборов данных, использовать матрицу и визуализировать результаты использования цветных heatmap или cThe cLike ( рис. 8).
- Выберите животных модель с высоким прирост информации. Для того чтобы оценить значение прироста информации, также учитывать критерий хи-квадрат тест (шаг 5.6).
Примечание: Животные модели следует только рассматривать как соответствующие если прирост информации является существенным, и если значение p хи-квадрат тест ниже уровень значимости. User-Defined порогов, как правило, будет зависеть от нескольких факторов: 1) предварительное исследование на переводимость результатов от модели на животных на людей (например, аналогичные физиологии), 2) ожидаемая польза для людей предполагаемого успеха, 3) практические знания применимость этого животного эксперимента и 4) ожидаемый боль, страдания или ущерб, причиненный в лабораторииОраторское искусство животных.
Результаты
GSEA рабочего процесса и скриншотов подражания данных демонстрируются. Рисунок 1 показывает файл данных выражение гена, содержащий данные транскриптомики интерес. Для каждого исследования описательного фенотип файл требуется показан на рис...
Обсуждение
Животные модели давно были применены для расследования заболевания механизмов и разработки новых терапевтических стратегий. Однако скептицизм относительно predictivity животных моделей начал распространяться после неудачи12клинических испытаний. Кроме того были подняты спо?...
Раскрытие информации
Авторы заявляют, что они не имеют никаких финансовых интересов.
Благодарности
Эта работа финансировалась Германского федерального института оценки риска (BfR).
Материалы
| Name | Company | Catalog Number | Comments |
| Excel | Microsoft Corporation |
Ссылки
- Seok, J., et al. Genomic responses in mouse models poorly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 110 (9), 3507-3512 (2013).
- Takao, K., Miyakawa, T. Genomic responses in mouse models greatly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 112 (4), 1167-1172 (2015).
- Weidner, C., Steinfath, M., Opitz, E., Oelgeschläger, M., Schönfelder, G. Defining the optimal animal model for translational research using gene set enrichment analysis. EMBO Mol Med. 8 (8), 831-838 (2016).
- Subramanian, A., et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proc Natl Acad Sci U S A. 102 (43), 15545-15550 (2005).
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., Tanabe, M. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44 (D1), D457-D462 (2016).
- Kanehisa, M., Goto, S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res. 28 (1), 27-30 (2000).
- Fabregat, A., et al. The Reactome pathway Knowledgebase. Nucleic Acids Res. 44 (D1), D481-D487 (2016).
- Croft, D., et al. The Reactome pathway knowledgebase. Nucleic Acids Res. 42 (Database issue), D472-D477 (2014).
- Nishimura, D. BioCarta. Biotech Software & Internet Report. 2 (3), 117-120 (2001).
- Edgar, R., Domrachev, M., Lash, A. E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Res. 30 (1), 207-210 (2002).
- Kolesnikov, N., et al. ArrayExpress update--simplifying data submissions. Nucleic Acids Res. 43 (Database issue), D1113-D1116 (2015).
- Cohen, J., et al. Sepsis: a roadmap for future research. Lancet Infect Dis. 15 (5), 581-614 (2015).
- Spinelli, L., Carpentier, S., Montanana Sanchis, F., Dalod, M., Vu Manh, T. P. BubbleGUM: automatic extraction of phenotype molecular signatures and comprehensive visualization of multiple Gene Set Enrichment Analyses. BMC Genomics. 16 (1), 814 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены