Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Microscopía de seguimiento de una sola molécula - Una herramienta para determinar los estados difusos de las moléculas citosólicas
En este artículo
Resumen
La microscopía de localización de una sola molécula 3D se utiliza para sondear las posiciones espaciales y las trayectorias de movimiento de las proteínas etiquetadas fluorescentes mente en células bacterianas vivas. El protocolo experimental y de análisis de datos descrito en este documento determina los comportamientos difusivos prevalentes de las proteínas citosólicas basadas en trayectorias agrupadas de una sola molécula.
Resumen
La microscopía de localización de una sola molécula sondea la posición y los movimientos de moléculas individuales en células vivas con decenas de nanómetros de resolución espacial y temporal de milisegundos. Estas capacidades hacen que la microscopía de localización de una sola molécula sea ideal para estudiar las funciones biológicas a nivel molecular en entornos fisiológicamente relevantes. Aquí, demostramos un protocolo integrado para la adquisición y procesamiento / análisis de datos de seguimiento de una sola molécula para extraer los diferentes estados difusivos que una proteína de interés puede exhibir. Esta información se puede utilizar para cuantificar la formación de complejos moleculares en células vivas. Proporcionamos una descripción detallada de un experimento de localización 3D de una sola molécula basado en cámara, así como los siguientes pasos de procesamiento de datos que producen las trayectorias de moléculas individuales. Estas trayectorias se analizan utilizando un marco de análisis numérico para extraer los estados difusos prevalentes de las moléculas etiquetadas fluorescentes y la abundancia relativa de estos estados. El marco de análisis se basa en simulaciones estocásticas de trayectorias de difusión Browniana intracelular que están limitadas espacialmente por una geometría celular arbitraria. Sobre la base de las trayectorias simuladas, las imágenes de una sola molécula en bruto se generan y analizan de la misma manera que las imágenes experimentales. De esta manera, las limitaciones de precisión y precisión experimentales, que son difíciles de calibrar experimentalmente, se incorporan explícitamente al flujo de trabajo de análisis. El coeficiente de difusión y las fracciones de población relativa de los estados difusos prevalentes se determinan ajustando las distribuciones de los valores experimentales mediante combinaciones lineales de distribuciones simuladas. Demostramos la utilidad de nuestro protocolo resolviendo los estados difusivos de una proteína que exhibe diferentes estados difusivos al formar complejos homo y hetero-oligoméricos en el citosol de un patógeno bacteriano.
Introducción
El examen del comportamiento difuso de las biomoléculas proporciona información sobre sus funciones biológicas. Las técnicas basadas en la microscopía de fluorescencia se han convertido en herramientas valiosas para observar biomoléculas en su entorno celular nativo. La recuperación de fluorescencia después del fotoblanqueo (FRAP) y la espectroscopia de correlación de fluorescencia (FCS)1 proporcionan comportamientos difusivos promediados por el conjunto. Por el contrario, la microscopía de localización de una sola molécula permite la observación de moléculas individuales etiquetadas fluorescentes con alta resolución espacial y temporal2,3,4. Observar moléculas individuales es ventajoso ya que puede existir una proteína de interés en diferentes estados difusivos. Por ejemplo, dos estados difusivos fácilmente distinguibles surgen cuando un regulador transcripcional, como El CueR en Escherichia coli, se difunde libremente en el citosol o se une a una secuencia de ADN y se inmoviliza en la escala temporal de la medición5 . El seguimiento de una sola molécula proporciona una herramienta para observar estos diferentes estados directamente, y no se requieren análisis sofisticados para resolverlos. Sin embargo, se vuelve más difícil resolver múltiples estados difusos y sus fracciones de población en casos en los que sus tasas difusas son más similares. Por ejemplo, debido a la dependencia del tamaño del coeficiente de difusión, los diferentes estados de oligomerización de una proteína se manifiestan como diferentes estados difusores6,7,8,9 , 10. Estos casos requieren un enfoque integrado en términos de adquisición, procesamiento y análisis de datos.
Un factor crítico que influye en las tasas difusas de las moléculas citosólicas es el efecto del confinamiento por el límite celular. Las restricciones impuestas al movimiento molecular por un límite de células bacterianas hacen que la tasa de difusión medida de una moléculacitos parezca más lenta que si el mismo movimiento hubiera ocurrido en un espacio no confinado. Para moléculas que difuminan muy lentamente, el efecto del confinamiento celular es insignificante debido a la falta de colisiones con el límite. En tales casos, puede ser posible resolver con precisión estados difusos ajustando las distribuciones de los desplazamientos moleculares, r,o coeficientes de difusión aparentes, D*,utilizando modelos analíticos basados en las ecuaciones para el movimiento Browniano ( difusión aleatoria)11,12,13. Sin embargo, para la rápida difusión de moléculas citosólicas, las distribuciones experimentales ya no se asemejan a las obtenidas para el movimiento Browniano no confinado debido a colisiones de moléculas difusoras con los límites celulares. Los efectos de confinamiento deben tenerse en cuenta para determinar con precisión los coeficientes de difusión no confinados de las moléculas etiquetadas fluorescentemente. Recientemente se han desarrollado varios enfoques para tener en cuenta los efectos de confinamiento (semi-)analíticamente 5,14,15,16 o numéricamente a través de simulaciones de Monte Carlo de Difusión Browniana6,10,16,17,18,19.
Aquí, proporcionamos un protocolo integrado para recopilar y analizar datos de microscopía de localización de una sola molécula con un enfoque particular en el seguimiento de una sola molécula. El objetivo final del protocolo es resolver estados difusos de proteínas citosólicas etiquetadas fluorescentemente en el interior, en este caso, células bacterianas en forma de varilla. Nuestro trabajo se basa en un protocolo anterior para el seguimiento de una sola molécula, en el que se demostró que existe una polimerasa de ADN, PolI, en un estado unido y sin enlazar el ADN mediante el análisis de difusión20. Aquí, ampliamos el análisis de seguimiento de una sola molécula a mediciones 3D y realizamos simulaciones computacionales más realistas para resolver y cuantificar múltiples estados difusivos presentes simultáneamente en las células. Los datos se adquieren utilizando un microscopio de fluorescencia de superresolución 3D construido en casa que es capaz de determinar la posición 3D de los emisores fluorescentes mediante imágenes con la función de difusión por puntos de doble hélice (DHPSF)21,22. Las imágenes de una sola molécula en bruto se procesan utilizando software escrito a medida para extraer las localizaciones 3D de una sola molécula, que luego se combinan en trayectorias de una sola molécula. Miles de trayectorias se agrupan para generar distribuciones de coeficientes de difusión aparentes. En un último paso, las distribuciones experimentales se ajustan a las distribuciones generadas numéricamente obtenidas a través de simulaciones de Montecarlo de movimiento Browniano en un volumen confinado. Aplicamos este protocolo para resolver los estados difusivos de la proteína del sistema de secreción Tipo 3 YscQ en Yersinia enterocoliticaviva. Debido a su naturaleza modular, nuestro protocolo es generalmente aplicable a cualquier tipo de experimento de seguimiento de una sola molécula o de una sola partícula en geometrías celulares arbitrarias.
Protocolo
1. Calibración de la función de dispersión por puntos de doble hélice
NOTA: Las imágenes descritas en esta y las siguientes secciones se adquieren utilizando un microscopio de fluorescencia invertida construido a medida, como se describe en Rocha et al.23. El mismo procedimiento es aplicable a diferentes implementaciones de microscopio diseñadas para la localización de una sola molécula y la microscopía de seguimiento2,3,4. Todo el software para la adquisición de imágenes y el procesamiento de datos descrito en este artículo está disponible (https://github.com/GahlmannLab2014/Single-Molecule-Tracking-Analysis.git).
- Preparación de almohadillas de agarosa para el montaje de muestras en resbalones de cubierta de vidrio para ser vistobajo el microscopio.
- Añadir 1,5-2% en peso bajo punto de fusión agarosa a 5 mL de tampón M2G (4,9 mM Na2HPO4, 3,1 mM KH2PO4, 7,5 mM NH4Cl, 0,5 mM MgSO4, 10 M FeSO4, 0,5 mM CaCl2 y 0,2% glucosa). Microondas durante varios segundos hasta que se disuelva toda la agarosa. No deje hervir la solución.
- Deje que la solución de agarosa se enfríe durante unos minutos (2-3 min).
- Pipetear 600 l de solución de agarosa sobre un resbalón de la cubierta de vidrio (22 mm x 22 mm). Coloque suavemente un segundo resbalón de la cubierta de vidrio en la parte superior de la agarosa. Esto crea una almohadilla de gel de agarosa delgada (0,5 mm) entre los dos resbalones de la cubierta de vidrio.
- Deje que las almohadillas de agarosa se sentén a solidificar durante 20 min.
- Separe suavemente los resbalones de la cubierta de vidrio. La almohadilla de agarosa se pegará a uno de ellos.
- Usando una cuchilla de afeitar, corta la almohadilla de agarosa en cuatro secciones cuadradas del mismo tamaño. Cada sección cuadrada se puede utilizar para una sola muestra.
- Pipetear 1,5 l de solución fluorescente de perla en una almohadilla de agarosa. La solución de abalorios es una dilución 1/100000 de la solución en stock en M2G (ver Tabla de Materiales).
- Invierta la almohadilla de agarosa y colóquela en un resbalón de la cubierta de vidrio que se haya limpiado en un limpiador de ozono durante 30 min. El tiempo de limpieza se elige para eliminar cualquier molécula fluorescente adherente a los cubreobjetos.
- Monte el vidrio de la cubierta de muestra en el soporte de muestra de un microscopio de fluorescencia invertido y fíjelo en su lugar con cinta adhesiva o clips portamuestras cargados por resorte.
- Agregue una gota de aceite de inmersión al objetivo del microscopio.
- Coloque el soporte de la muestra en un microscopio y fíjelo en su lugar.
- Inicializar la interfaz gráfica de usuario (GUI) para controlar la cámara del microscopio, la etapa de muestra y los láseres de excitación. Aquí, el software escrito a medida en MATLAB se utiliza para el control de instrumentos (Figura Suplementaria 1).
- Inicializar el software de la cámara HCImage Live. En la pestaña Captura de la sección Control de cámara, establezca el tiempo de exposición en 0.03 s. Haga clic en Live para comenzar una transmisión en vivo de la cámara.
- Encienda el láser haciendo clic en Abrir láser de 514 nm en la interfaz GUI para excitar las perlas fluorescentes en la almohadilla y ver la emisión de fluorescencia en la cámara utilizando el modo de transmisión en vivo (es decir, no hay datos guardados en el disco).
- Ajuste las posiciones X e Y de la etapa del microscopio haciendo clic en las flechas 'XY-Pos' bajo la sección 'Fase de microposicionamiento' de la GUI para colocar al menos un cordón fluorescente en el centro del campo de visión (FOV). El tamaño del paso se puede modificar haciendo clic en el cuadro desplegable debajo de las flechas.
- Ajuste la posición Z de la etapa del microscopio haciendo clic en las flechas Z-Pos bajo la sección Etapa de Nano-Posicionamiento de la GUI. Establezca la orientación de la función de propagación por puntos de doble hélice (DHPSF) del cordón fluorescente en vertical. Esta orientación vertical se define como el punto de partida en la calibración Z. El tamaño del paso se puede modificar haciendo clic en el cuadro desplegable debajo de las flechas.
- En HCImage Live, en Capture (Capturar) Modos de disparo, velocidad y registro, cambie el modo de disparo de Interno a Activador de nivel externo. Esto permitirá que la GUI de MATLAB controle la cámara.
- En Secuencia ( Sequence) Ajustesde escaneado , cambie el número de recuentos de fotogramas a 1200. Elija una carpeta de destino de guardado haciendo clic en el botón etiquetado .... Finalmente haga clic en Inicio. El número de recuentos de tramas se establece en 1200 para que se puedan recopilar 10 fotogramas para cada uno de los pasos de posición de 120 Z.
- Escanee a través de un rango de posiciones Z (30 pasos por encima y por debajo de la posición Z inicial en incrementos de 50 nm) y registre 10 fotogramas en cada paso utilizando un tiempo de exposición de 0.03 s. Comience el proceso automatizado haciendo clic en GO en la sección Z-Calibration de la GUI.
NOTA: Los parámetros para la calibración, incluida la longitud del paso, el número de pasos, el tiempo de exposición de la cámara y el número de fotogramas por paso, también se pueden ajustar aquí. Alrededor de 105 fotones se pueden adquirir de un cordón fluorescente en un solo marco utilizando tiempos de exposición de 0,03 s, lo que resulta en precisiónes de localización x,y,z de aproximadamente 1 nm. - Desactive la iluminación láser haciendo clic en Cerrar láser de 515 nm en la GUI. En HCImage Live, en Capture (Capturar) Modos de disparo, velocidad y registro, cambie el modo de disparo de nuevo a Interno. En Secuencia ( Sequence) Los ajustes de escaneado cambian el número de recuentos de fotogramas a 200. Elija una carpeta de destino de guardado haciendo clic en el botón etiquetado .... Haga clic en Iniciar para recopilar 200 fotogramas de imágenes oscuras con un tiempo de exposición de 0,03 s.
NOTA: Incluso en ausencia de luz que caiga sobre el detector, cada píxel leerá un número positivo (denominado el valor de desplazamiento oscuro), que puede variar ligeramente entre píxeles. El valor de desplazamiento oscuro puede cambiar con el tiempo. Por lo tanto, es necesario recoger marcos oscuros para cada calibración. - Ajuste el DHPSF con un modelo de doble gaussiana utilizando el software Easy-DHPSF24 para obtener las posiciones X e Y del cordón, así como una curva de calibración angular frente a Z.
- Inicializar el software Easy-DHPSF en MATLAB. En Configuración, establezca Canal en G y Método de conexión en MLE con el modelo DG. G se refiere a la cámara de canal verde, ya que la proteína fluorescente que se utiliza para la adquisición de datos emite con longitudes de onda verdes. MLE con modelo DG se refiere a la estimación de máxima verosimilitud con el modelo de doble gaussiana.
NOTA: El tamaño de píxel y la ganancia de conversión dependen de la configuración óptica específica y es posible que deba modificarse. - En la sección Calibrar DHPSF, haga clic en Ejecutar. Haga clic en Aceptar en la siguiente ventana emergente para mantener la configuración predeterminada.
- Seleccione la pila de imágenes guardada en los pasos 1.13-1.14. A continuación, seleccione la pila de imágenes con el fondo oscuro guardado en el paso 1.15. Por último, seleccione el archivo .txt que se guardó automáticamente durante el paso 1.14. Este archivo contiene la posición Z de la etapa durante todo el proceso de escaneo.
- En la siguiente ventana emergente, si el chip de la cámara completa no se utilizó para el campo de visión, introduzca las posiciones iniciales x0 e y0 en el chip. De lo contrario, la entrada x0 a 1 y y0 a 1.Esta información se puede encontrar en HCImage Live en la sección Binning and SubArrary.
- En la siguiente ventana, cambie el tamaño y la posición del cuadro que aparece sobre la imagen de las cuentas fluorescentes para que tenga un tamaño aproximado de 100 x 100 píxeles y se centre sobre una sola señal Fluorescente DHPSF. A continuación, haga doble clic para continuar.
NOTA: El DHPSF elegido debe aislarse de las otras señales DHPSF e idealmente ser el cordón más brillante posible. - Haga clic en el centro de la señal DHPSF, entre los dos lóbulos y, a continuación, pulse Intro. La siguiente ventana muestra una vista ampliada del DHPSF elegido. Elija con mayor precisión la ubicación del centro de la DHPSF haciendo clic.
NOTA: A continuación, el programa se ajustará al DHPSF y mostrará la imagen en bruto y la reconstrucción del ajuste. También emitirá imágenes de plantilla de la calibración Z correspondientes a una sección transversal DHPSF en diferentes posiciones Z. Estos se utilizarán más adelante para la adaptación de los datos experimentales. El programa emitirá la posición X,Y y Z estimada en cada fotograma. En un sistema óptico bien alineado, X e Y deben cambiar muy poco (desviación de 30 nm) a medida que cambia la posición Z. Si la variación de salida es mayor que 30 nm, la máscara de fase, ubicada en el plano de Fourier del sistema de imágenes(Figura 1), debe realinearse y repetirse los pasos 1.9-1.16. - Guarde la GUI Easy-DHPSF haciendo clic en el icono Guardar en la esquina superior izquierda de la GUI. Esto se puede cargar más adelante haciendo clic en el icono de la carga en el GUI.
NOTA: El procedimiento de calibración Z debe llevarse a cabo en cada día de un experimento para tener en cuenta los cambios de alineación en el microscopio que pueden haber ocurrido debido a fluctuaciones de temperatura o vibraciones mecánicas.
- Inicializar el software Easy-DHPSF en MATLAB. En Configuración, establezca Canal en G y Método de conexión en MLE con el modelo DG. G se refiere a la cámara de canal verde, ya que la proteína fluorescente que se utiliza para la adquisición de datos emite con longitudes de onda verdes. MLE con modelo DG se refiere a la estimación de máxima verosimilitud con el modelo de doble gaussiana.
2. Preparación del cultivo bacteriano
- Preparar medios de cultivo que apoyen el crecimiento celular bacteriano. Para Y. enterocolitica, utilice 5 ml de caldo BHI (Brain Heart Infusion) que contenga ácido nalidixico (35 g/ml) y ácido diaminopimelico de 2,6 (80 g/ml). Aquí, se utiliza una cepa Y. enterocolitica que tiene la proteína YscQ etiquetada con la proteína fluorescente eYFP23.
- Inocular medios con cultivos bacterianos de las existencias de congelador o cultivos de placas.
- Cultivar la cultura a 28 oC con temblores durante la noche.
- Diluir una pequeña cantidad (250 l) de cultivo de la noche saturada a 5 ml utilizando medios de cultivo frescos.
- Cultivar el cultivo a 28 oC con temblores durante 60-90 min.
- Inducir la expresión de proteína de fusión fluorescente. Para Y. enterocolitica, choque por calor las células a 37 oC en un agitador de agua para inducir el regulón de yop.
- Incubar las células durante 3 h adicionales a 37 oC con agitación.
- Centrífuga 1 ml de cultivo a 5.000 x g durante 3 min a temperatura ambiente. Descarta el sobrenadante.
- Lave el pellet 3 veces con 1 ml de soporte M2G.
- Vuelva a suspender las bacterias peletadas en 250 l de medios M2G.
- Añadir cuentas fluorescentes como marcadores fiduciarios. La solución de perlas fluorescentes debe añadirse en cantidades adecuadamente diluidas, de modo que solo haya 1-2 perlas por FOV cuando se vean en el microscopio.
- Pipetear o vórtice suavemente la suspensión para separar las células agregadas.
- Placa 1,5 l de suspensión en una almohadilla de agarosa de 1,5-2% hecha con M2G.
- Invierta la almohadilla de agarosa y colóquela en un resbalón de la cubierta del microscopio limpiado con ozono. El resbalón de la cubierta debe colocarse en un limpiador de ozono durante 30 minutos para reducir cualquier fondo de fluorescencia inherente.
3. Adquisición de datos
- Monte el vidrio de la cubierta de muestra en el soporte de muestra de un microscopio de fluorescencia invertido y fíjelo en su lugar con cinta adhesiva o clips portamuestras cargados por resorte.
- Agregue una gota de aceite de inmersión en el objetivo del microscopio, luego coloque el soporte de la muestra en el microscopio y fíjelo en su lugar.
- Inicializar la interfaz gráfica de usuario (GUI) para controlar la cámara del microscopio, la etapa de muestra y los láseres de excitación. Aquí, el software escrito a medida en MATLAB se utiliza para el control de instrumentos.
- Inicializar el software de la cámara HCImage Live. En la pestaña Captura de la sección Control de cámara, establezca el tiempo de exposición en 0.025 s. Haga clic en Live para comenzar una transmisión en vivo de la cámara.
- Ajuste las posiciones X e Y de la etapa del microscopio haciendo clic en las flechas XY-Pos bajo la sección Etapa de Microposicionamiento de la GUI para escanear alrededor de la muestra y encontrar un FOV con una población adecuadamente densa de células bacterianas.
NOTA: Para maximizar el rendimiento de los datos, las celdas deben ser lo más densas posible, sin superponerse ni tocar celdas. El FOV también debe incluir al menos 1 cordón fluorescente para ser utilizado como un marcador fiduciario, preferiblemente colocado en una esquina del FOV. - Ajuste la posición Z de la etapa del microscopio haciendo clic en las flechas Z-Pos bajo la sección De la etapa de nanoposicionamiento de la GUI, de modo que los lóbulos DHPSF del cordón fluorescente sean verticales.
- En Secuencia ( Sequence) Ajustesde escaneado , cambie el número de recuentos de fotogramas a 20.000. Elija una carpeta de destino de guardado haciendo clic en el botón etiquetado .... Finalmente haga clic en Iniciar para recoger hasta 20.000 fotogramas de cámara utilizando un corto tiempo de exposición de 0.025 s. eYFP photoblinking se inicia utilizando luz de excitación de alta intensidad a 514 nm19,25.
NOTA: Aquí, se utiliza una intensidad láser de 350 W/cm2 en el plano focal para el blanqueo inicial y la posterior toma de imágenes de moléculas EYFP individuales. No se utilizó la fotoactivación de moléculas de eYFP en longitudes de onda UV durante la toma de imágenes. Debe haber como máximo una señal de una sola molécula por célula bacteriana. Si la densidad de la señal de una sola molécula es demasiado alta inicialmente, continúe iluminando hasta que se produzca suficiente fotoblanqueo antes de comenzar la adquisición de datos. - Desactive la iluminación láser haciendo clic en Cerrar láser de 515 nm en la GUI. Recoge 200 fotogramas de imágenes oscuras utilizando el mismo tiempo de exposición.
- En la GUI, marque la casilla situada junto a Thorlabs LED y haga clic en Alternar espejo . Esto cambiará la vía de la vía de fluorescencia a la vía de contraste de fase.
- Inicializar el software de adquisición de datos IC Capture 2.4. Esto controla la cámara en la vía de contraste de fase. Pulse el botón Start/Stop Live Display para ver una transmisión en vivo desde la cámara. Haga clic en Capturar (Capture) Guardar imagen para recopilar una imagen de contraste de fase de las celdas en el campo de visión.
- Repita los pasos 3.5-3.10 para FOVs adicionales. En este caso, los datos adquiridos a partir de 500 células bacterianas en diez FoVs diferentes se utilizan para aumentar el número de trayectorias de una sola molécula disponibles para el análisis.
ADVERTENCIA: Las celdas montadas en las almohadillas de agarosa durante largos períodos de tiempo pueden comportarse de manera diferente que las células recién montadas. Además, la almohadilla de agarosa puede perder su integridad después de algún tiempo, lo que puede afectar negativamente a la calidad de los datos. Por lo general, como máximo se utilizan 3 FOV (30 min en el microscopio) por diapositiva de muestra.
4. Procesamiento de datos
NOTA: Una versión modificada del software Easy-DHPSF24 se utiliza en MATLAB para el análisis de los marcos de cámara sin procesar para extraer localizaciones de una sola molécula. Easy-DHPSF se utiliza específicamente para adaptarse a las localizaciones DHPSF en imágenes de una sola molécula. Se realizaron cambios personalizados para implementar la rutina de ajuste basada en la estimación de máxima verosimilitud (MLE) que tiene en cuenta las características de ruido dependientes de píxeles de las cámaras sCMOS modernas26. También se modificó para aceptar la salida de tipo de archivo de imagen del programa HCImage Live (.dcimg). Para obtener una explicación más detallada del software y los pasos individuales, consulte Lew etal.
- Inicializar la GUI Easy-DHPSF en MATLAB(Figura suplementaria 2). Cargue el archivo guardado en el paso 1.16.8.
- Determinación de los valores de umbral para cada una de las 7 plantillas de salida en el paso 1.16.7
- En la sección Calibrar identificación SM , haga clic en Ejecutar. Haga clic en Aceptar en las dos ventanas emergentes siguientes para mantener la configuración predeterminada.
- Abra la pila de imágenes que contiene los datos del primer FOV cuando se le solicite.
- Elija una pequeña gama de marcos para que coincidan con las plantillas. Típicamente las tramas 1001-2000 se utilizan para evitar las señales superpuestas densas en los primeros cientos de tramas. Haga clic en Aceptar en la siguiente ventana emergente para mantener la configuración predeterminada. Haga clic en Cancelar cuando se le solicite el archivo de registro de secuencia en la siguiente ventana.
- Abra la pila de imágenes con el fondo oscuro guardado en el paso 3.8. Haga clic en 'Aceptar' en la siguiente ventana emergente para dejar los parámetros para la estimación de fondo establecido en el valor predeterminado. El valor predeterminado es estimar el fondo utilizando un filtro mediano27 que cubre 100 fotogramas de cámara subsiguientes alrededor del fotograma actual.
- En la siguiente ventana, cambie el tamaño del cuadro superpuesto en la imagen para cubrir el FOV completo y, a continuación, haga doble clic para continuar.
- Defina la región de interés haciendo clic en varios puntos de la imagen para crear un polígono. La región de interés debe incluir la mayor parte del campo de visión posible, a la vez que se asegura de que las cuentas fluorescentes (objetos muy brillantes) de la imagen no se encuentran dentro del polígono y, a continuación, haga doble clic para continuar.
NOTA: El software intentará hacer coincidir las plantillas con la imagen y mostrará una imagen con posibles coincidencias en círculo. - Cuando el software se ha detenido, guardará muchas imágenes de coincidencias de plantilla encontradas y mostrará el valor de umbral correspondiente en una carpeta predefinida. Un valor de umbral más alto corresponde a una mejor coincidencia. Para cada número de plantilla, examine las coincidencias de ejemplo y determine el umbral más bajo que muestra una imagen de un DHPSF. Ingrese estos umbrales para cada una de las 7 plantillas bajo la sección Calibrar identificación SM en la GUI Easy-DHPSF.
NOTA: El programa intentará elegir automáticamente y los umbrales de entrada, sin embargo, estos son a menudo poco fiables y deben comprobarse manualmente. Los umbrales se eligen de tal manera que se pierden pocas señales verdaderas de una sola molécula, pero el número de candidatos falsos positivos para el ajuste sigue siendo manejable desde el punto de vista computacional. - Guarde la GUI Easy-DHPSF de nuevo haciendo clic en el icono Guardar en la esquina superior izquierda.
- Colocación del cordón fluorescente en el FOV para usarlo como marcador fiduciario
- En la sección Seguimiento de fiduciarios de la GUI Easy-DHPSF, haga clic en Ejecutar. Haga clic en Aceptar en las dos ventanas emergentes siguientes para mantener la configuración predeterminada.
- Arrastre el cuadro superpuesto en la imagen y celérelo sobre la señal DHPSF del cordón fluorescente y, a continuación, haga doble clic.
- Haga clic en el centro de la señal DHPSF, en el punto medio entre los dos lóbulos y, a continuación, pulse intro. Haga clic en Cancelar cuando se le solicite el archivo de registro de secuencia en la siguiente ventana.
NOTA: El software se ajustará al DHPSF en todos los marcos de la cámara y mostrará la imagen sin procesar y la imagen reconstruida. Cuando el software haya terminado, emitirá cifras con las posiciones X,Y y Z del cordón fluorescente durante la adquisición de la imagen. - Marque la casilla junto a Usar fiduciarios y guarde la GUI Easy-DHPSF de nuevo haciendo clic en el icono Guardar en la esquina superior izquierda.
- Busque y ajuste todas las localizaciones en todos los marcos de la cámara utilizando los umbrales de plantilla obtenidos en el paso 4.2.
- En la sección Localizar SMS DHPSF de la GUI Easy-DHPSF, haga clic en Ejecutar. Haga clic en Aceptar en las siguientes ventanas emergentes para mantener la configuración predeterminada. Haga clic en Cancelar cuando se le solicite el archivo de registro de secuencia en la siguiente ventana.
NOTA: El software encontrará y ajustará el DHPSF utilizando un modelo de doble gaussiana si la calidad de la coincidencia está por encima del umbral definido por el usuario. Mostrará la imagen sin procesar con círculos alrededor de las coincidencias de plantilla, así como una imagen del DHPSF reconstruido. - Guarde la GUI Easy-DHPSF de nuevo haciendo clic en el icono Guardar en la esquina superior izquierda.
- En la sección Localizar SMS DHPSF de la GUI Easy-DHPSF, haga clic en Ejecutar. Haga clic en Aceptar en las siguientes ventanas emergentes para mantener la configuración predeterminada. Haga clic en Cancelar cuando se le solicite el archivo de registro de secuencia en la siguiente ventana.
- Visualización de localizaciones de una sola molécula y filtrado de localizaciones no deseadas o poco fiables
- En la sección Localizaciones de DHPSF SM de salida, haga clic en Salida de filtro.
- Haga clic en Aceptar en las tres ventanas siguientes para realizar una interpolación de las posiciones fiduciales X, Y y Z a lo largo del tiempo. En la mayoría de los casos, las opciones predeterminadas son suficientes. Si la línea interpolada negra no refleja una interpolación razonable de la línea de posición roja, cambie los parámetros de interpolación en la ventana emergente.
NOTA: La línea interpolada se utiliza para corregir las localizaciones de una sola molécula. - Abra la imagen de contraste de fase correspondiente para el FOV que se está analizando cuando se le solicite. Haga clic en Aceptar en las dos ventanas emergentes siguientes para mantener la configuración predeterminada.
- En las dos ventanas emergentes siguientes, cambie los valores de filtro para permitir requisitos de localización de una sola molécula más estrictos o más indulgentes y, a continuación, haga clic en Aceptar.
- En la ventana que aparece, arrastre o cambie el tamaño del cuadro superpuesto en las imágenes para ver la región de interés deseada y haga doble clic para continuar.
- Se muestra una reconstrucción 3D de localizaciones de una sola molécula. Utilice las herramientas de figura para manipular la reconstrucción (rotar, hacer zoom, etc.). Haga clic en Continuar para mostrar otro cuadro de diálogo preguntando'¿Desea volver a trazar con un conjunto de parámetros diferente?'. Si los resultados son satisfactorios, haga clic en No. Si no lo están, haga clic en Sí.
NOTA: Las razones comunes para obtener resultados insatisfactorios incluyen que la imagen de contraste de fase no se superpone correctamente con los datos de localización o que los umbrales de plantilla iniciales eran demasiado bajos, lo que crea muchas localizaciones de falsos positivos. Si se ha seleccionado Sí, el software volverá al paso 4.5.4 para que se puedan definir nuevos valores de parámetro. Si se ha seleccionado No, los resultados se guardarán.
5. Postprocesamiento de datos
- Utilizando software escrito a medida en MATLAB, recorte la imagen de contraste de fase para que solo permanezca la región que contiene las células que se crearon imágenes bajo el microscopio de fluorescencia. Este paso es necesario porque la imagen de contraste de fase cubre un área mucho más grande que la imagen de fluorescencia. Recortar la imagen simplifica el siguiente paso.
- Segmente celdas individuales procesando la imagen de contraste de fase con el software OUFTI28 (Figura suplementaria 3)
- Inicializar OUFTI en MATLAB. Cargue la imagen de contraste de fase recortada del paso anterior haciendo clic en Cargar fase.
- Haga clic en Archivo para elegir y asignar un nombre a una ubicación de guardado para el archivo de salida.
- Seleccione Marcos independientes en el encabezado Detección y análisis.
- Haga clic en Cargar parámetros para cargar parámetros para la detección de celdas. Algunos ejemplos de parámetros son el área de celda aceptable, el ancho de celda y un umbral de división de celdas.
NOTA: Todos estos parámetros deben ajustarse para maximizar el rendimiento de los tamaños de celda específicos y la calidad de imagen que se utiliza. Es importante destacar que el parámetro de algoritmo debe establecerse en subpíxel para permitir una medición precisa de los contornos de celda. - Haga clic en Este marco para iniciar la segmentación de celdas. Los contornos de celda aparecerán sobre la imagen de contraste de fase cuando finalice el proceso.
- Mediante los controles de la sección Manual, divida celdas, agregue celdas o refinar contornos de celda para obtener contornos para las celdas que se segmentaron inexactamente durante el proceso automatizado.
- Para generar los contornos de celda, haga clic en Guardar análisis.
- Utilice software escrito a medida en MATLAB para superponer con precisión los contornos obtenidos en el paso anterior con las localizaciones de una sola molécula. Los siguientes subpasos detallan los pasos del software.
- Seleccione manualmente 5 pares de puntos de control en la ventana emergente estimando y haciendo clic en la posición de los polos celulares de las mismas cinco celdas tanto en los datos de localización de una sola molécula como en los contornos de celda, generados en el paso anterior. La posición del polo celular se puede estimar aproximadamente dibujando mentalmente un casco convexo alrededor de las localizaciones de una sola molécula pertenecientes a una célula y seleccionando el punto de curvatura más alta(Figura Suplementaria 4).
- Genere una función de transformación afín 2D utilizando la función cp2tform en MATLAB y utilícela para generar una superposición aproximada de los contornos de celda y las localizaciones de una sola molécula.
- Elimine las celdas que contienen menos de 10 localizaciones y elimine las celdas que se colocan parcialmente fuera del campo de visión. Elimine manualmente las celdas no deseadas adicionales en la ventana emergente haciendo clic dentro de su contorno de celda(Figura suplementaria 5).
- Utilice el centro de masa para todos los contornos de celda restantes y localizaciones de una sola molécula dentro de ellos para formar un conjunto más grande de pares de puntos de control, vuelva a calcular la función de transformación 2D y utilícela para generar una superposición final de los contornos de la célula y el localizaciones de una sola molécula.
- Asigne localizaciones que residan dentro del límite de un contorno de celdas a esa celda. Deseche las localizaciones que no se encuentren dentro de ningún esquema de celda(Figura 2a, Figura suplementaria 6).
6. Seguimiento de una sola molécula
NOTA: La siguiente sección se completa con software escrito a medida en MATLAB. En esta sección se describen los pasos que realiza el software.
- Para las localizaciones asignadas a la misma celda y en fotogramas de cámara posteriores, calcule la distancia euclidiana entre las localizaciones. Si la distancia entre las localizaciones está por debajo de un umbral de 2,5 m, vincule las localizaciones asignándolas a la misma trayectoria de una sola molécula.
NOTA: Es importante tener en cuenta solo las localizaciones dentro de una sola celda, de modo que las localizaciones en celdas adyacentes que cumplan los requisitos de umbral espacial y temporal no estén vinculadas. El umbral de 2,5 m se eligió como la distancia máxima que una molécula muy rápida (30 m2/s) podría viajar en la longitud del tiempo de exposición (0,025 s) más un búfer del 20%. - Descartar trayectorias inferiores a 4 localizaciones. Si dos o más localizaciones (es decir, dos o más emisores de fluorescencia) están presentes simultáneamente en una celda, deseche las trayectorias asociadas. Establecer la longitud de la vía mínima en 4 localizaciones permite promediar varias mediciones de distancia, lo que produce una estimación más precisa de los coeficientes de difusión.
- Calcule el desplazamiento cuadrado medio (MSD) para una trayectoria determinada:
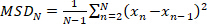 (1)
(1)
donde N es el número total de localizaciones en la trayectoria y xn es la posición de la molécula en el punto de tiempo n. - Calcular el coeficiente de difusión aparente, D* por
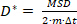 (2)
(2)
donde m 2 o 3 es la dimensionalidad de la medida y el tiempo de exposición de la cámara es el m.
NOTA: Un experimento típico producirá trayectorias de entre 5.000 y 100.000 euros en total, lo que resultará en una distribución aparente del coeficiente de difusión con tantos recuentos.
7. Simulación de Montecarlo del movimiento Browniano en un volumen confinado
NOTA: Crear bibliotecas de distribuciones simuladas de coeficientes de difusión aparentes mediante la realización de simulaciones de Monte Carlo de movimiento Browniano confinado a un volumen cilíndrico, utilizando 64 valores en el rango de 0.05-20 m2/s como parámetros de entrada (software disponibles de los autores bajo petición). Este rango fue elegido para cubrir el rango de coeficientes de difusión previamente estimados de proteínas fluorescentes (fusión) en bacterias. Se utilizan 64 coeficientes de difusión para muestrear este rango de manerasuficiente. Esta sección se realiza utilizando software escrito a medida en MATLAB y describe los pasos que el software toma automáticamente. Las células de Y. enterocolitica en forma de varilla utilizadas aquí se aproximan por un volumen cilíndrico de longitud l a 5 m y diámetro d a 0,8 m.
- Iniciar trayectorias individuales en una posición aleatoria en el volumen cilíndrico, y simular sus pasos de difusión aleatorios (es decir, Brownianos) utilizando un intervalo de tiempo de 100 ns (el intervalo de tiempo debe ser mucho más corto que el tiempo de exposición de la cámara para permitir suficiente muestra de la posición en el transcurso de un marco de cámara). Muestre cada paso de desplazamiento para una entrada D de la función de distribución gaussiana correspondiente mediante la reorganización de Eqn. 2 y agréguela a la posición anterior:
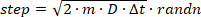 (3)
(3)
donde la función randn en MATLAB muestrea un número aleatorio de una distribución normal. Si un paso hace que la molécula se desplace fuera del volumen del cilindro, refleje la molécula dentro del cilindro en un ángulo aleatorio. - Para cada intervalo de tiempo, genere una imagen DHPSF que corresponda a la posición x,y,z instantánea del emisor simulado.
- Para que coincida con las condiciones experimentales, simule imágenes que contengan 1.000 fotones por localización, con un fondo láser de 13 fotones por píxel y ruido de Poisson. Además, añada un desplazamiento oscuro de 50 fotones por píxel y ruido de lectura gaussiano (1,5 fotones), de acuerdo con las mediciones experimentales de calibración de la cámara. Por último, multiplique la imagen por la ganancia dependiente de píxeles medida experimentalmente de la cámara sCMOS para obtener la imagen en unidades de recuento de detectores.
NOTA: Después de estas manipulaciones, la relación señal-ruido de la imagen final es de 2. - Genere imágenes borrosas en el movimiento que reflejen la posición cambiante de las moléculas sumando 50 imágenes DHPSF simuladas durante el tiempo de exposición utilizado durante la adquisición de datos experimentales. Para limitar el gasto computacional, solo se eligieron 50 posiciones muestreadas periódicamente para generar una imagen (en lugar de las 250.000 posiciones muestreadas durante un tiempo de exposición de 0,025 s en un intervalo de muestreo de 100 ns).
NOTA: La longitud (número de fotogramas) de las trayectorias simuladas debe coincidir con la longitud media de las trayectorias experimentales. En este caso, el número de fotogramas por trayectoria es 6.
- Para que coincida con las condiciones experimentales, simule imágenes que contengan 1.000 fotones por localización, con un fondo láser de 13 fotones por píxel y ruido de Poisson. Además, añada un desplazamiento oscuro de 50 fotones por píxel y ruido de lectura gaussiano (1,5 fotones), de acuerdo con las mediciones experimentales de calibración de la cámara. Por último, multiplique la imagen por la ganancia dependiente de píxeles medida experimentalmente de la cámara sCMOS para obtener la imagen en unidades de recuento de detectores.
- Genere 5.000 trayectorias para cada uno de los 64 coeficientes de difusión de entrada simulados.
- Analice los fotogramas de cámara simulados como se describe en la sección Procesamiento de datos.
- Vincule las localizaciones a trayectorias como se describe en la sección Seguimiento de molécula única.
- Interpolar la función de distribución acumulativa (CDF) para cada distribución simulada utilizando la interpolación B-spline de la orden 25. Las distribuciones interpoladas son necesarias para que se puedan consultar en puntos arbitrarios.
- Interpolar las curvas B-spline resultantes a lo largo del eje D(D es el verdadero coeficiente de difusión sin confinar que rige el movimiento de los emisores simulados) utilizando la función scatteredInterpolant en MATLAB (especificar el ' natural'). Esto proporciona una función 2D continua a partir de la cual se puede consultar cualquier distribución aparente del coeficiente de difusión correspondiente a un valor de coeficiente de difusión real en el rango de 0,05-20 m2/s.
8. Ajuste experimental de la distribución del coeficiente de difusión aparente
NOTA: Ajuste las distribuciones medidas experimentalmente de los coeficientes de difusión aparentes utilizando combinaciones lineales de las distribuciones simuladas generadas en la sección anterior(simulación Monte-Carlo del movimiento Browniano en un volumen confinado). Esta sección se realiza utilizando software escrito a medida en MATLAB y describe los pasos que el software toma automáticamente. Para obtener más información y ejemplos de solicitud, véase Rocha et al.29
- Realice un ajuste de mínimos cuadrados lineales restringidos (utilizando la función lsqlin en MATLAB) del CDF experimental utilizando una matriz muestreada periódicamente de CDF simulados de la biblioteca creada en la sección 7. La salida de este paso es un vector de parámetros que contiene los coeficientes de difusión y las fracciones de población de los estados difusivos prevalentes en la distribución experimental.
- Combine estados difusos con valores de coeficiente de difusión dentro del 20% uno del otro en un único estado difuso por promedio de peso basado en la fracción de población relativa. Este es el vector de parámetro sin salida.
NOTA: Para reducir la complejidad del modelo, los estados difusos con coeficientes de difusión inferiores a 0,5 m2/s se pueden mantener constantes durante todos los pasos siguientes. - Cree matrices de vectores de parámetros de ajuste de prueba con diferentes números de estados difusos, que van desde un único estado difuso hasta un número máximo de estados definido por el usuario.
- Usando el vector de parámetro inicial, combine estados difusos adyacentes a través de promedioponderado y divida estados difusos en dos estados con fracciones de población iguales y coeficientes de difusión 20% por encima y por debajo del valor original. Repita el proceso para todas las combinaciones de estados y las posibilidades de división.
- Utilice cada vector de parámetro de ajuste de prueba para inicializar un ajuste mínimo cuadrado no lineal de 5 subconjuntos separados de los datos (utilizando la función fmincon en MATLAB). Determine la calidad del ajuste buscando la suma residual de cuadrados entre el ajuste y la distribución correspondiente a los subconjuntos restantes (validación cruzada de datos).
- Utilice la suma residual media de cuadrados de los 5 accesorios separados para cada vector de prueba como la calidad general de ajuste, determinar el vector de prueba con la mejor calidad de ajuste para cada número de estados difusos.
- Determinar el número óptimo de estados mediante la identificación del vector de prueba para el que agregar un estado adicional no resulta en al menos una mejora del 5% en la calidad del ajuste.
- Utilice este vector de prueba para inicializar el ajuste de mínimos cuadrados no lineales del conjunto de datos completo.
- Estimar el error para los parámetros individuales mediante el remuestreo de los datos mediante el arranque y el reajuste con el mismo vector de prueba.
Resultados
En las condiciones experimentales descritas aquí (20.000 fotogramas, longitud de trayectoria mínima de 4 localizaciones) y dependiendo de los niveles de expresión de las proteínas de fusión etiquetadas fluorescentemente, aproximadamente 200-3.000 localizaciones que producen 10-150 trayectorias se pueden generar por celda(Figura 2a,b). Se hace necesario un gran número de trayectorias para producir una distribución bien muestreada de los...
Discusión
Un factor crítico para la aplicación exitosa del protocolo presentado es asegurar que las señales de una sola molécula estén bien separadas entre sí (es decir, deben ser escasas en el espacio y en el tiempo(Movimiento Suplementario 1)). Si hay más de una molécula de fluorescencia en una célula al mismo tiempo, entonces la localización podría asignarse incorrectamente a la trayectoria de otras moléculas. Esto se conoce como el problema de vinculación30. Se pueden elegi...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Alecia Achimovich y Ting Yan por la lectura crítica del manuscrito. Agradecemos a Ed Hall, científico del personal superior del grupo Advanced Research Computing Services de la Universidad de Virginia, por su ayuda para establecer las rutinas de optimización utilizadas en este trabajo. La financiación de este trabajo fue proporcionada por la Universidad de Virginia.
Materiales
| Name | Company | Catalog Number | Comments |
| 2,6-diaminopimelic acid | Chem Impex International | 5411 | Necessary for growth of Y. enterocolitica cells used. |
| 4f lenses | Thorlabs | AC508-080-A | f = 80mm, 2" |
| 514 nm laser | Coherent | Genesis MX514 MTM | Use for fluorescence excitation |
| agarose | Inivtrogen | 16520100 | Used to make gel pads to mount liquid bacterial sample on microscope. |
| ammonium chloride | Sigma Aldrich | A9434 | M2G ingredient. |
| bandpass filter | Chroma | ET510/bp | Excitation pathway. |
| Brain Heart Infusion | Sigma Aldrich | 53286 | Growth media for Y. enterocolitica. |
| calcium chloride | Sigma Aldrich | 223506 | M2G ingredient. |
| camera | Imaging Source | DMK 23UP031 | Camera for phase contrast imaging. |
| dielectric phase mask | Double Helix, LLC | N/A | Produces DHPSF signal. |
| disodium phosphate | Sigma Aldrich | 795410 | M2G ingredient. |
| ethylenediaminetetraacetic acid | Fisher Scientific | S311-100 | Chelates Ca2+. Induces secretion in the T3SS. |
| flip mirror | Newport | 8892-K | Allows for switching between fluorescence and phase contrast pathways. |
| fluospheres | Invitrogen | F8792 | Fluorescent beads. 540/560 exication and emission wavelengths. 40 nm diameter. |
| glass cover slip | VWR | 16004-302 | #1.5, 22mmx22mm |
| glucose | Chem Impex International | 811 | M2G ingredient. |
| immersion oil | Olympus | Z-81025 | Placed on objective lens. |
| iron(II) sulfate | Sigma Aldrich | F0518 | M2G ingredient. |
| long pass filter | Semrock | LP02-514RU-25 | Emission pathway. |
| magnesium sulfate | Fisher Scientific | S25414A | M2G ingredient. |
| microscope platform | Mad City Labs | custom | Platform for inverted microscope. |
| nalidixic acid | Sigma Aldrich | N4382 | Y. enterocolitica cells used are resistant to nalidixic acid. |
| objective lens | Olympus | 1-U2B991 | 60X, 1.4 NA |
| Ozone cleaner | Novascan | PSD-UV4 | Used to eliminate background fluorescence on glass cover slips. |
| potassium phosphate | Sigma Aldrich | 795488 | M2G ingredient. |
| Red LED | Thorlabs | M625L3 | Illuminates sample for phase contrast imaging. 625nm. |
| sCMOS camera | Hamamatsu | ORCA-Flash 4.0 V2 | Camera for fluorescence imaging. |
| short pass filter | Chroma | ET700SP-2P8 | Emission pathway. |
| Tube lens | Thorlabs | AC508-180-A | f=180 mm, 2" |
| Yersinia enterocolitica dHOPEMTasd | N/A | N/A | Strain AD4442, eYFP-YscQ |
| zero-order quarter-wave plate | Thorlabs | WPQ05M-514 | Excitation pathway. |
Referencias
- Kapanidis, A. N., Uphoff, S., Stracy, M. Understanding Protein Mobility in Bacteria by Tracking Single Molecules. Journal of Molecular Biology. , (2018).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, T. Y., et al. Quantifying Multistate Cytoplasmic Molecular Diffusion in Bacterial Cells via Inverse Transform of Confined Displacement Distribution. Journal of Physical Chemistry B. 119 (45), 14451-14459 (2015).
- Mohapatra, S., Choi, H., Ge, X., Sanyal, S., Weisshaar, J. C. Spatial Distribution and Ribosome-Binding Dynamics of EF-P in Live Escherichia coli. mBio. 8 (3), (2017).
- Stracy, M., et al. Single-molecule imaging of UvrA and UvrB recruitment to DNA lesions in living Escherichia coli. Nature Communications. 7, 12568 (2016).
- Persson, F., Lindén, M., Unoson, C., Elf, J. Extracting intracellular diffusive states and transition rates from single-molecule tracking data. Nature Methods. 10 (3), 265-269 (2013).
- Bakshi, S., Choi, H., Weisshaar, J. C. The spatial biology of transcription and translation in rapidly growing Escherichia coli. Frontiers in Microbiology. 6, 636 (2015).
- Mustafi, M., Weisshaar, J. C. Simultaneous Binding of Multiple EF-Tu Copies to Translating Ribosomes in Live Escherichia coli. mBio. 9 (1), (2018).
- Michalet, X., Berglund, A. J. Optimal diffusion coefficient estimation in single-particle tracking. Physical Review E. 85 (6), (2012).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 82 (4 Pt 1), 041914 (2010).
- Backlund, M. P., Joyner, R., Moerner, W. E. Chromosomal locus tracking with proper accounting of static and dynamic errors. Physical Review E Statistical, Nonlinear, and Soft Matter Physics. 91 (6), 062716 (2015).
- Stracy, M., et al. Live-cell superresolution microscopy reveals the organization of RNA polymerase in the bacterial nucleoid. Proceedings of the National Academy of Sciences of the United States of America. 112 (32), E4390-E4399 (2015).
- Plochowietz, A., Farrell, I., Smilansky, Z., Cooperman, B. S., Kapanidis, A. N. In vivo single-RNA tracking shows that most tRNA diffuses freely in live bacteria. Nucleic Acids Research. 45 (2), 926-937 (2017).
- Koo, P. K., Mochrie, S. G. Systems-level approach to uncovering diffusive states and their transitions from single-particle trajectories. Physical Review E. 94 (5-1), 052412 (2016).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proceedings of the National Academy of Sciences of the United States of America. 110 (20), 8063-8068 (2013).
- Bakshi, S., Bratton, B. P., Weisshaar, J. C. Subdiffraction-Limit Study of Kaede Diffusion and Spatial Distribution in Live Escherichia coli. Biophysical Journal. 101 (10), 2535-2544 (2011).
- Bakshi, S., Siryaporn, A., Goulian, M., Weisshaar, J. C. Superresolution imaging of ribosomes and RNA polymerase in live Escherichia coli cells. Molecular Microbiology. 85 (1), 21-38 (2012).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing Protein-DNA Interactions in Live Bacterial Cells Using Photoactivated Single-molecule Tracking. JoVE. (85), e51177 (2014).
- Pavani, S. R. P., Piestun, R. Three dimensional tracking of fluorescent microparticles using a photon-limited double-helix response system. Optics Express. 16 (26), 22048-22057 (2008).
- Pavani, S. R. P., et al. Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 2995-2999 (2009).
- Rocha, J. M., et al. Single-molecule tracking in live Yersinia enterocolitica reveals distinct cytosolic complexes of injectisome subunits. Integrative Biology. 10 (9), 502-515 (2018).
- Lew, M. D., von Diezmann, A. R. S., Moerner, W. E. Easy-DHPSF open-source software for three-dimensional localization of single molecules with precision beyond the optical diffraction limit. Protocol Exchange. , (2013).
- Biteen, J. S., et al. Super-resolution imaging in live Caulobacter crescentus cells using photoswitchable EYFP. Nature Methods. 5 (11), 947-949 (2008).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nature Methods. 10 (7), 653-658 (2013).
- Hoogendoorn, E., et al. The fidelity of stochastic single-molecule super-resolution reconstructions critically depends upon robust background estimation. Scientific Reports. 4, 3854 (2014).
- Paintdakhi, A., et al. Oufti: An integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular microbiology. 99 (4), 767-777 (2016).
- Rocha, J. M., Corbitt, J., Yan, T., Richardson, C., Gahlmann, A. Resolving Cytosolic Diffusive States in Bacteria by Single-Molecule Tracking. bioRxiv. , 483321 (2018).
- Lee, A., Tsekouras, K., Calderon, C., Bustamante, C., Presse, S. Unraveling the Thousand Word Picture: An Introduction to Super-Resolution Data Analysis. Chemical Reviews. 117 (11), 7276-7330 (2017).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chemical Biology. , (2008).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chemistry & Biology. 15 (2), 128-136 (2008).
- Bisson-Filho, A. W., et al. Treadmilling by FtsZ filaments drives peptidoglycan synthesis and bacterial cell division. Science. 355 (6326), 739-743 (2017).
- Douglass, K. M., Sieben, C., Archetti, A., Lambert, A., Manley, S. Super-resolution imaging of multiple cells by optimised flat-field epi-illumination. Nature Photonics. 10 (11), 705-708 (2016).
- Zhao, Z., Xin, B., Li, L., Huang, Z. L. High-power homogeneous illumination for super-resolution localization microscopy with large field-of-view. Optics Express. 25 (12), 13382-13395 (2017).
- Yan, T., Richardson, C. J., Zhang, M., Gahlmann, A. Computational Correction of Spatially-Variant Optical Aberrations in 3D Single Molecule Localization Microscopy. bioRxiv. , 504712 (2018).
- Gahlmann, A., Moerner, W. E. Exploring bacterial cell biology with single-molecule tracking and super-resolution imaging. Nature Reviews Microbiology. 12 (1), 9-22 (2014).
- Liu, Z., Lavis, L. D., Betzig, E. Imaging live-cell dynamics and structure at the single-molecule level. Mol Cell. 58 (4), 644-659 (2015).
- Berglund, A. J. Statistics of camera-based single-particle tracking. Physical Review E. 82 (1), 011917 (2010).
- Parry, B. R., et al. The bacterial cytoplasm has glass-like properties and is fluidized by metabolic activity. Cell. 156 (1-2), 183-194 (2014).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proceedings of the National Academy of Sciences of the United States of America. 108 (31), E365-E373 (2011).
- Niu, L. L., Yu, J. Investigating intracellular dynamics of FtsZ cytoskeleton with photoactivation single-molecule tracking. Biophysical Journal. 95 (4), 2009-2016 (2008).
- Coquel, A. S., et al. Localization of protein aggregation in Escherichia coli is governed by diffusion and nucleoid macromolecular crowding effect. PLoS Computational Biology. 9 (4), e1003038 (2013).
- Nenninger, A., Mastroianni, G., Mullineaux, C. W. Size Dependence of Protein Diffusion in the Cytoplasm of Escherichia coli. Journal of Bacteriology. 192 (18), 4535-4540 (2010).
- Dix, J. A., Verkman, A. S. Crowding effects on diffusion in solutions and cells. Annual Review of Biophysics. 37, 247-263 (2008).
- Elliott, L. C., Barhoum, M., Harris, J. M., Bohn, P. W. Trajectory analysis of single molecules exhibiting non-brownian motion. Physical Chemistry Chemical Physics. 13 (10), 4326-4334 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados