Method Article
Fraccionamiento subcelular de células de leucemia linfocítica crónica primaria para monitorear el tráfico de proteínas nucleares/citoplasmáticas
En este artículo
Resumen
Este protocolo permite la optimización y posterior generación eficiente de fracciones nucleares y citoplasmáticas a partir de células de leucemia linfocítica crónica primarias. Estas muestras se utilizan para determinar la localización de proteínas, así como los cambios en el tráfico de proteínas que tienen lugar entre los compartimentos nuclear y citoplasmático tras la estimulación celular y el tratamiento farmacológico.
Resumen
La exportación nuclear de macromoléculas a menudo se desregula en las células cancerosas. Las proteínas supresoras de tumores, como p53, pueden quedar inactivas debido a que la localización celular aberrante interrumpe su mecanismo de acción. La supervivencia de las células de la leucemia linfocítica crónica (LLC), entre otras células cancerosas, está asistida por la desregulación de los enclavados nucleares a citoplasmáticos, al menos en parte mediante la desregulación del receptor de transporte XPO1 y la activación constitutiva de Vías de señalización mediadas por PI3K. Es esencial entender el papel de las proteínas individuales en el contexto de su ubicación intracelular para obtener una comprensión más profunda del papel de dichas proteínas en la patobiología de la enfermedad. Además, la identificación de procesos que subyacen a la estimulación celular y el mecanismo de acción de inhibidores farmacológicos específicos, en el contexto del tráfico de proteínas subcelulares, proporcionará una comprensión más completa del mecanismo de Acción. El protocolo descrito aquí permite la optimización y posterior generación eficiente de fracciones nucleares y citoplasmáticas a partir de células de leucemia linfocítica crónica primarias. Estas fracciones se pueden utilizar para determinar los cambios en el tráfico de proteínas entre las fracciones nuclear y citoplasmática tras la estimulación celular y el tratamiento farmacológico. Los datos pueden cuantificarse y presentarse en paralelo con imágenes inmunofluorescentes, proporcionando así datos robustos y cuantificables.
Introducción
El transporte de macromoléculas entre el núcleo y el citoplasma se ha establecido durante mucho tiempo para desempeñar un papel clave en la función celular normal y a menudo se desregula en las células cancerosas1,2. Esta desregulación puede ser el resultado de una sobreexpresión/mutación de proteínas que controlan la exportación nuclear. Una de estas proteínas Exportin-1 (XPO1), es un receptor de transporte que exporta >200 proteínas que contienen señal de exportación nuclear (NES) en el citoplasma desde el núcleo2. XPO1-cargos incluyen p53, miembros de la familia FOXO e IB, contribuyendo a su inactivación mediante la inhibición de su mecanismo de acción1,2,3. Se puede producir más deslocalización proteica cuando las señales microambientales afectan a las células cancerosas, lo que conduce a la activación de vías de señalización intracelularcomo la vía fosfatidil-inositol-3-quinasa (PI3K)/Akt, lo que resulta en inactivación de los miembros de la familia FOXO y posterior exportación desde el núcleo4,5. Dicha deslocalización de proteínas supresoras tumorales se ha visto implicada en la progresión de una serie de tumores hematológicos y sólidos1,2,6.
El desarrollo de inhibidores de moléculas pequeñas para uso clínico en neoplasias malignas hematológicas (leucemia mieloide aguda (LMA)/CLL), que se unen a la función XPO1 e inhiben selectivamente, subraya la importancia de desarrollar técnicas adecuadas para impacto de los agentes farmacológicos en el cierre de proteínas entre los compartimentos nuclear y citoplasmático6,7,8. Las técnicas de diagnóstico por imágenes han avanzado significativamente permitiendo la identificación de proteínas en compartimentos subcelulares tras la estimulación externa de los tratamientos farmacológicos, sin embargo, la importancia de las técnicas paralelas robustas y de apoyo es fundamental para informar a un público científico de la validez de un resultado.
Los linfocitos en reposo y las células malignas de la CLL-B aisladas de muestras de sangre de pacientes representan un desafío en la generación de fracciones nucleares y citoplasmáticas debido a la alta relación nuclear: citoplasma. La optimización de las condiciones experimentales para generar datos experimentales sólidos y fiables es, por supuesto, fundamental para planificar futuros programas experimentales. El método descrito aquí permite la cuantificación de proteínas en las fracciones nucleares y citoplasmáticas y determina cómo estas proteínas pueden verse afectadas por la estimulación celular y/o el tratamiento farmacológico.
Protocolo
El uso de muestras primarias de pacientes con LlAns descritas aquí ha sido aprobado por el West of Scotland Research Ethics Service, NHS Greater Glasgow y Clyde (Reino Unido) y todo el trabajo se llevó a cabo de acuerdo con las directrices aprobadas.
1. Aislamiento de células CLL de muestras de sangre del paciente
-
Las muestras de sangre periférica de pacientes con LL consentidos previamente se reciben de la clínica en tubos de recolección de sangre EDTA, acompañados por el recuento de glóbulos blancos (CMI). Purificar las muestras de CLL de sangre periférica según el CMI. Para el CMI < 40 x 106 células/mL, proceda al paso 1.1.1; para el CMI a 40 x 106 células/ml, proceda al paso 1.1.2.
- Vierta el contenido de todos los tubos sanguíneos EDTA en un tubo centrífugo cónico de 50 ml y agregue 50 ml de cóctel de enriquecimiento de células B humanas por 1 ml de sangre. Incubar a temperatura ambiente (RT) durante 20 min. Proceder al paso 1.1.2.
- Diluir la muestra en una proporción de 1:1 con tampón de lavado RT CLL (salina tamponada con fosfato (PBS), 0,5% de suero bovino fetal (FBS) y 2 mM EDTA).
- Medios de gradiente de densidad Aliquot RT en un tubo centrífugo cónico de tamaño adecuado para la muestra (10 ml en un tubo de 50 ml para 30 ml de muestra o 4 ml en un tubo de 15 ml para 10 ml de muestra).
- Coloque cuidadosamente la muestra en la parte superior de los medios de gradiente de densidad y centrífuga a 400 x g durante 30 minutos a RT.
NOTA: Asegúrese de que la centrífuga está en RT antes de que las muestras se coloquen en la centrífuga como un cambio de temperatura resultará en un pobre enriquecimiento de las células mononucleares, y apagar el freno en la centrífuga, ya que el frenado repentino puede interrumpir la interfaz líquida. - Cosecha suavemente la capa blanca de células mononucleares que se acumulan en la interfaz de los medios de gradiente de densidad y el tampón de lavado CLL, en un nuevo tubo de centrífuga cónica de 50 ml utilizando una pipeta Pasteur de plástico.
- Agregue 40 ml de tampón de lavado CLL a la monocapa aislada para lavar las células y centrifugar a 300 x g durante 10 min a RT.
- Deseche el sobrenadante, resuspenda el pellet moviendo la parte inferior del tubo y, a continuación, repita el paso de lavado descrito en el paso 1.5.
- Deseche el sobrenadante, resuspenda el pellet como se describe en el paso 1.6, luego resuspenda el pellet en un volumen establecido de tampón de lavado CLL (hasta 40 ml, dependiendo del tamaño del pellet celular).
- Cuente las células usando el azul del trypan y un hemocitómetro. A continuación, proceda a la citometría de flujo para comprobar la pureza de las células CLL.
NOTA: En esta etapa las células CLL se pueden cultivar a una concentración de 10 x 106 células/ml en medios para ser utilizados en experimentos, y/o crioconservados en 10% de sulfóxido de dimetil (DMSO)/FBS para el trabajo futuro en concentraciones de hasta 100 x 106 células/vial.
2. Citometría de flujo de células CLL
- Etiqueta Tubos de poliestireno de fondo redondo de 12 mm x 75 mm, tal como se describe en la Tabla 1.
- En los tubos 2 – 5, poner una gota de cuentas de compensación y almacenar en el hielo. Añadir 1 l del anticuerpo apropiado (anti-CD5, CD19, CD23 o CD45, como se indica en la Tabla 1) a los tubos 2 – 5, e incubar sobre hielo durante 20 minutos, protegido de la luz colocando papel de aluminio sobre el cubo de hielo.
NOTA: Estos tubos sirven como controles de compensación para configurar la plantilla de citometría de flujo. -
Coloque hasta 1 x 106 células CLL en tubos 1, 6 y 7, agregue 2 ml de tampón FACS (PBS + 2% FBS) a cada tubo y centrífuga a 300 x g durante 5 minutos a RT para lavar las células. Deseche el sobrenadante y almacene los tubos que contienen gránulos celulares en hielo.
- Resuspenda los gránulos celulares y añada la combinación adecuada de anticuerpos a las células en el tubo 7 como se indica en la Tabla 1,en un volumen final de 100 l con tampón FACS. Los anticuerpos se utilizan a una concentración adecuada de acuerdo con las directrices del fabricante.
- Resuspenda los gránulos celulares en los tubos 2 y 6 en 100 ml de tampón FACS.
- Incubar las células sobre hielo, junto a las cuentas manchadas en los tubos 2 – 5, protegidos de la luz durante 20 min.
- Después de la incubación, añadir 2 ml de tampón FACS a todos los tubos y centrifugar a 300 x g durante 5 minutos en RT para lavar las células. Deseche el sobrenadante y resuspenda los pellets de perlas/células moviendo suavemente los tubos.
- Resuspenda los tubos 1 - 5 en 100 l de tampón FACS y colóquelos en hielo hasta que estén listos para analizar en el citometro de flujo.
- Diluir la solución DAPI a 0,05-0,2 g/ml en el búfer FACS inmediatamente antes de su uso. La concentración óptima puede variar y se recomienda la valoración.
- Resuspenda los tubos 6 y 7 con 100 ml de solución DAPI diluida e incubar los tubos sobre hielo durante un mínimo de 5 minutos para permitir que las células se manta.
NOTA: No es necesario lavarmás más, ya que la DAPI debe estar presente en el búfer para que las células muertas permanezcan etiquetadas. Una vez que se ha agregado DAPI, las celdas deben analizarse en el catómetro de flujo dentro de 4 h. - Analice las células utilizando un catómetro de flujo.
3. Preparación de fracciones subcelulares a partir de células CLL
NOTA: Al planificar la configuración experimental, incluya un pozo de células no estimuladas/no tratadas a partir de las cuales se puede generar todo el extracto celular.
- Realizar la estimulación deseada y / o tratamiento farmacológico de la línea celular DEC1 CLL o células primarias aisladas De CLL utilizando 10 – 20 x 106 células/condición. Las celdas se utilizarán para el fraccionamiento subcelular (pasos 3.4 y 3.5) o para generar extracto de celda entera (paso 3.6).
- Preparación de soluciones/tubos: Preparar todas las soluciones/buffers recién el día del fraccionamiento, antes de cosechar las células. Almacene las soluciones en hielo hasta que sea necesario y utilícelo dentro de las 4 h de la preparación.
- Solución inhibidora de PBS/fosfatasa: Preparar los inhibidores de la fosfatasa en PBS diluyendo los inhibidores de la fosfatasa 1:20 en 1x PBS (es decir, 0,5 ml de inhibidores de la fosfatasa en 9,5 ml de 1x PBS).
NOTA: Asegúrese de que los inhibidores de la fosfatasa no se hayan precipitado. Si hay un precipitado presente, calentar a 50 oC durante 10 min. - Tampón hipotónico: Preparar 1 x tampón hipotónico haciendo una dilución 1:10 de tampón hipotónico 10x en agua destilada (es decir, 50 l de tampón hipotónico 10x en 450 oL de dH2O).
- DTT de 10 mM (TDT): Preparar 10 mM de TDT haciendo una dilución de 1:100 de TDT de 1M con agua destilada (es decir, 10 ml de TDT de 1 M en 990 ml de dH2O).
NOTA: La TDT es muy lábil, así que prepáralo de forma fresca cada vez. Evite ciclos repetidos de congelación/descongelación. - Búfer de lisis completa: determine cuánto búfer se necesita para cada experimento. Cada muestra requiere 50 l de tampón de lisis completo, por lo que añadir 5 ml de TDT de 10 mM (paso 3.2.3) a 44,5 l de tampón de lisis y, a continuación, añadir 0,5 ml de cóctel inhibidor de proteasa. Esta cantidad se puede escalar verticalmente dependiendo del número de muestras en el experimento.
- Etiquetar cuatro conjuntos de tubos de microfusión de 1,5 ml para cada estimulación y/o tratamiento farmacológico para las células recién estimuladas (paso 3.3), las fracciones citoplasmáticas recién generadas (paso 3.4.3), las fracciones nucleares recién generadas (paso 3.5.3) y toda la célula (paso 3.6.3). Enfríe previamente estos tubos de microfúcya en hielo hasta que sea necesario.
- Solución inhibidora de PBS/fosfatasa: Preparar los inhibidores de la fosfatasa en PBS diluyendo los inhibidores de la fosfatasa 1:20 en 1x PBS (es decir, 0,5 ml de inhibidores de la fosfatasa en 9,5 ml de 1x PBS).
- Transfiera las células a tubos de microfusión de 1,5 ml y pellets etiquetados individualmente centrifugando a 200 x g durante 5 min a 4 oC. Retire el sobrenadante y resuspenda las células en 1 ml de inhibidores de PBS/fosfatasa heladas (paso 3.2.1). Peletizar las células por centrifugación a 200 x g durante 5 min a 4oC. Retire el sobrenadante y mantenga los gránulos celulares en hielo.
- Preparación de fracciones citoplasmáticas: Resuspenda suavemente los gránulos celulares que se utilizarán para el fraccionamiento subcelular en 50 ml de tampón hipotónico 1x (paso 3.2.2). Incubar las células sobre hielo durante 15 minutos para permitir que las células se hinchen.
NOTA: El volumen de tampón hipotónico utilizado se puede aumentar empíricamente dependiendo del número de celda.- Añadir 0,8 - 2,5 l (1:20 a 1:60) de detergente en cada muestra y vórtice en el ajuste más alto durante 10 s.
- Para determinar la concentración óptima de detergente que se debe utilizar para un tipo de celda específico para aislar fracciones nucleares y citoplasmáticas, realice un gradiente de detergente inicialmente. Un rango de 1:20 a 1:60 (es decir, de 2,5 a 0,8 ml de detergente en 50 ml de tampón hipotónico) debe ser adecuado.
NOTA: Si se ajusta el volumen del tampón hipotónico en el paso 3.4, asegúrese de que se mantiene la relación de detergente adecuada. - Verifique la lisis celular observando las células utilizando un microscopio de contraste de fase antes y después de la adición de detergente. Las células enteras aparecen más grandes con un núcleo denso y oscuro. El citoplasma aparecerá como un halo brillante alrededor del núcleo.
NOTA: La lisis adecuada se confirma aún más mediante el uso de la hincha occidental para analizar proteínas específicas dentro de las fracciones lysed generadas a partir del gradiente del detergente.
- Para determinar la concentración óptima de detergente que se debe utilizar para un tipo de celda específico para aislar fracciones nucleares y citoplasmáticas, realice un gradiente de detergente inicialmente. Un rango de 1:20 a 1:60 (es decir, de 2,5 a 0,8 ml de detergente en 50 ml de tampón hipotónico) debe ser adecuado.
- Una vez lysed, centrifugar las muestras a 14.000 x g durante 30 s a 4oC.
- Transfiera cuidadosamente el sobrenadante a un tubo de microfófugo preenfriado y etiquetado. Esta fracción citoplasmática se puede almacenar a -80 oC hasta que sea necesario para un análisis posterior. El pellet restante contiene la fracción nuclear (paso 3.5).
NOTA: Evite ciclos repetidos de congelación/descongelación de las muestras.
- Añadir 0,8 - 2,5 l (1:20 a 1:60) de detergente en cada muestra y vórtice en el ajuste más alto durante 10 s.
- Preparación de fracciones nucleares: Resuspenda cada gleta nuclear en 50 ml de tampón de lisis completo (paso 3.2.4) pipeteando hacia arriba y hacia abajo.
NOTA: El volumen del búfer de lisis completo se puede ajustar empíricamente según el número de celda inicial.- Añadir 2,5 ml de detergente para solubilizar las proteínas asociadas con la membrana nuclear y el vórtice en el ajuste más alto durante 10 s. Incubar las muestras sobre hielo durante 30 min.
- Vórtice en el ajuste más alto durante 30 s, luego centrifugar las muestras a 14.000 x g durante 20 min a 4 oC.
- Transfiera el sobrenadante a un tubo de microfófugo preenfriado y etiquetado. Esta fracción nuclear se puede almacenar a -80 oC hasta que sea necesario para un análisis posterior.
NOTA: Evite ciclos repetidos de congelación/descongelación de las muestras.
- Preparación de lisados de células enteras (WCL) a partir de células CLL
NOTA: La preparación de todo el extracto celular se puede llevar a cabo al mismo tiempo que la preparación de las fracciones nucleares (paso 3.5).- Resuspenda todos los gránulos de extracto celular en 100 ml de tampón de lisis completo (preparado en el paso 3.2.4) pipeteando hacia arriba y hacia abajo, luego agregue 5 s l de detergente para asegurar la lisis celular completa. Incubar las muestras sobre hielo durante 30 min.
- Vórtice en el ajuste más alto durante 30 s, luego centrifugar las muestras a 14.000 x g durante 20 min a 4 oC.
- Transfiera el sobrenadante a un tubo de microfófugo preenfriado. Este lisato de células enteras se puede almacenar a -80 oC hasta que sea necesario para un análisis posterior.
NOTA: Evite ciclos repetidos de congelación/descongelación de las muestras.
4. Análisis aguas abajo de fracciones subcelulares
NOTA: En este protocolo, el análisis de las fracciones celulares generadas se llevó a cabo mediante la hinchazón occidental utilizando protocolos estándar, cargando números de celda/carril iguales (equivalentes a 10 g de proteína) para fracciones nucleares y citoplasmáticas.
- Cuantificación del tráfico de proteínas entre fracciones nucleares y citoplasmáticas: Realice análisis cuantitativos de manchas occidentales a través de la cuantificación de la intensidad de la señal o la densitometría utilizando software de análisis de manchas occidentales disponible libremente.
- Importación de imágenes: Las imágenes western blot generadas a partir de diferentes instrumentos en desarrollo deben importarse como archivos JPG, PNG o TIFF. Se recomienda un archivo RAW de profundidad de 16 bits. Para importar una imagen, haga clic en el icono de software y mantenga el puntero sobre Importar. A continuación, haga clic en Imágenes de terceros. Seleccione el archivo de imagen y haga clic en Abrir.
- Visualización de la imagen: En la cinta imagen, haga clic en el botón Elegir del grupo Visualización. El cuadro de diálogo Ajustar visualización se abrirá para habilitar más ajustes si es necesario. Implemente mejoras adicionales, como Brillo o Contraste, con los controles deslizantes ajustables de la ficha LUTs de imagen.
- Análisis de datos (deselección de canal): haga clic en la cinta de opciones Análisis. Para analizar solo un canal, anule la selección de los canales que no se están analizando. Haga clic en la miniatura No mostrar canal de un canal en los LUT de imagen, dejando solo el canal deseado mostrado. Las imágenes importadas como archivos JPG, PNG o TIFF pueden requerir la deselección de varios canales RGB no deseados.
-
Adición de formas: Para cuantificar la intensidad de la señal, haga clic en Agregar rectángulo para agregar un rectángulo a la imagen. Haga clic en el centro de una entidad (por ejemplo, una banda de proteína) para colocar un rectángulo alrededor de ella. Como alternativa, para dibujar manualmente una forma, elija Dibujar rectángulo. Después de agregar todas las formas deseadas, haga clic en Seleccionar para devolver el cursor a la herramienta de selección.
NOTA: Agregue varias formas en un orden lógico, ya que los datos se ordenan por un número de identificador que se genera secuencialmente. -
Resta de fondo: Para restar ruido de fondo, haga clic en el primer botón del grupo Fondo y seleccione Mediana en el menú desplegable. Establezca el ancho del borde en 3 en el diálogo Fondo y seleccione los segmentos que se utilizarán para el cálculo de fondo. Al elegir los segmentos que desea utilizar, seleccione los segmentos que mejor representen el fondo de la imagen.
NOTA: El ruido de fondo puede afectar a la cuantificación de la señal, por lo que debe restarse para calcular con precisión la señal a partir de las formas de interés. -
Recortar señal y recortar fondo - OPCIONAL: Los archivos importados en formatos JPG, PNG o TIFF pueden mostrar saturación de píxeles: regiones resaltadas/brillantes dentro de una banda de proteínas. Recortar señal y fondo de recorte (Bkgnd) elimina los píxeles saturados del análisis. Para ver estos valores, agregue Recortar señal y Recortar Bkgnd a una tabla haciendo clic en el botón Columnas a la derecha de la vista de tabla.
NOTA: La saturación de píxeles puede llevar a una cuantificación poco fiable. Los píxeles saturados solo se pueden eliminar si se saturan menos del 5% de los píxeles de una forma.
-
Adición de formas: Para cuantificar la intensidad de la señal, haga clic en Agregar rectángulo para agregar un rectángulo a la imagen. Haga clic en el centro de una entidad (por ejemplo, una banda de proteína) para colocar un rectángulo alrededor de ella. Como alternativa, para dibujar manualmente una forma, elija Dibujar rectángulo. Después de agregar todas las formas deseadas, haga clic en Seleccionar para devolver el cursor a la herramienta de selección.
- Exportar datos: Haga clic en la pestaña Formas situada encima de la tabla. Para la densitometría, se requieren valores en la columna Signal. Señal es la suma de los valores de intensidad de píxel (Total) para una forma menos el producto del Bkgnd y el área. Haga clic en el botón Informe. Haga clic en Guardar como o Iniciar hoja de cálculo.
Señal : Total – (Bkgnd x Area)
NOTA: La pestaña formas proporciona una tabla de valores cuantitativos, incluidos Signal, Total, Area y Bkgrnd. - Cuantificación de la expresión proteica: Dentro de la hoja de cálculo guardada, calcular la expresión normalizada de la proteína de interés para cada carril o variable dividiendo la Señal obtenida para la proteína de interés por la Señal para la banda de control de carga de proteínas correspondiente.
NOTA: Las comparaciones entre las cantidades de una proteína normalizada de interés entre fracciones nucleares y citoplasmáticas no se pueden comparar directamente debido a los diferentes controles de carga utilizados para distinguir las fracciones nucleares y citoplasmáticas. Sin embargo, las comparaciones dentro de fracciones individuales, por ejemplo, después del tratamiento farmacológico, son apropiadas. - Exportar imagen para su publicación o presentación: Haga clic en la pestaña Imágenes que se encuentra encima de la tabla y, a continuación, haga clic en la imagen que desea exportar. Si utiliza la imagen para una presentación de diapositivas u otros formatos digitales, haga clic en el icono de software, mantenga el puntero sobre Exportar y haga clic en Imagen para medios digitales. Guarde la imagen como un archivo JPG, PNG o TIFF, según sea necesario.
Resultados
Al planificar experimentos en células CLL primarias, si los ensayos requieren un gran número de celdas (>50 x 106 células), hay una preferencia para usar células CLL recién aisladas, en lugar de células crioconservadas que requieren descongelación, sin embargo esto no siempre es Posible. Esto se debe a que el proceso de congelación/descongelación puede dar lugar a la muerte de hasta el 50% de las células CLL, aunque esto depende de la muestra. El enriquecimiento de células CLL con un CMI >40 x 106/mL utilizando centrifugación de densidad como se describe aquí (pasos 1.3 – 1.5) permite una alta recuperación celular con alta pureza (-95%) células CLL primarias. En la muestra mostrada, se recuperaron el CMI a 177 x 106/ml: a partir de una muestra de sangre de 30 ml 5 x 109 células, lo que representa un rendimiento celular del 94% del total de células. El análisis de esta muestra por citometría de flujo reveló una pureza de células CLL de >95% como lo indica la expresión de superficie dual de los marcadores de células CLL CD19 y CD5 después de realizar una velocidad en FSC/SSC, células únicas que fueron DAPI negativas (células viables)(Figura 1).
La optimización del procedimiento de fraccionamiento subcelular se llevó a cabo utilizando una gama de proporciones de detergente (1:20 a 1:60) durante la preparación de la fracción citoplasmática (paso 3.4). A partir de entonces, se prepararon las fracciones nucleares y los WCL (pasos 3.5 y 3.6 respectivamente). Los inmunoblotes se realizaron en las fracciones resultantes de la línea de células CLL MEC1(Figura 2A)y células CLL primarias(Figura 2B). Las manchas fueron sondeadas para los marcadores de fracción Lamin A/C (nuclear; 74/63 kDa) y la tubulina (citoplasmática; 55 kDa) para confirmar el fraccionamiento celular exitoso. El fraccionamiento indica que el nivel óptimo de detergente para las células MEC1 es una dilución 1:60(Figura 2A),en comparación con una dilución de 1:30 óptima para las células Primarias de la LLC(Figura 2B),como se indica mediante un enriquecimiento de proteína nuclear y la falta de proteína citoplasma en las fracciones yviceversa. Los WCL representan la proteína total y actúan como un control positivo de los anticuerpos utilizados para sondear las fracciones subcelulares. Es importante elegir proteínas apropiadas como marcadores de fracción: la Figura 2C muestra inmunoblots de fracciones nucleares/citoplasmas preparadas a partir de células MEC1 en las que el ARN polimerasa II (Rpb1 CTD; 250 kDa) y Lamin A/C fueron borrados como marcadores de fracciones nucleares, mientras que la tubulina y la tubulina (50 kDa) se utilizaron como marcadores citoplasma. Está claro que la tubulina se enriquece en el citoplasma, sin embargo la expresión es evidente en el núcleo, como se ha demostrado anteriormente9.
Una vez optimizadas las condiciones experimentales, se puede realizar un experimento. En los ejemplos mostrados, la localización subcelular de FOXO1 en fracciones nucleares y citoplasmáticas se determinó tras la estimulación de células con el receptor de antígeno de células B (BCR) en presencia o ausencia del inhibidor dual mTORC1/2 AZD8055, en células MEC1 ( Figura 3A) y células CLL primarias(Figura 3B)5,10. En ambos ejemplos, la generación de fracciones nucleares y citoplasmáticas altamente enriquecidas se logró como lo indica la expresión casi exclusiva de Lamin en la fracción nuclear y la tubulina en las fracciones citoplasmáticas. En ambos tipos de células, la expresión de FOXO1 se redujo en el citoplasma tras el tratamiento con AZD8055 en comparación con NDC, acompañado de un aumento de la expresión de FOXO1 en el compartimento nuclear, demostrando así la translocación de proteínas(Figura 3). Para eliminar la subjetividad de la interpretación de datos, se cuantificaron los inmunoblots individuales de cinco muestras de LLC primarias dentro de fracciones subcelulares (paso 4; Figura 4A), utilizando las proteínas nucleares o citoplasmas respectivas como controles de carga interna para cada muestra, y luego normalizando cada fracción al control no estimulado (EE.UU.) sin control de drogas (NDC), como se indica. El gráfico resultante muestra las tendencias del movimiento FOXO1 entre las fracciones nuclear y citoplasmática, con AZD8055 reduciendo los niveles de expresión de FOXO1 en el citoplasma, al mismo tiempo que aumenta la expresión en el núcleo. Además, una elevación en la expresión citoplasmática FOXO1 es evidente en el entrecruzado BCR.
| Tubo | Nombre del tubo | Células/Abalorios | Antígeno | Fluoróforo |
| 1 | Sin mancha | Células | Na | Na |
| 2 | Mancha única | perlas | CD5 | Fitc |
| 3 | Mancha única | perlas | CD19 | PE-Cy7 |
| 4 | Mancha única | perlas | CD23 | Apc |
| 5 | Mancha única | perlas | CD45 | APC-Cy7 |
| 6 | Mancha única | Células | Viabilidad | Dapi |
| 7 | CLL Stain | Células | CD5, CD19, CD23, CD45 y viabilidad | FITC, PE-Cy7, APC, APC-Cy7 y DAPI |
Tabla 1: Tabla que muestra el conjunto ideal de tubos de muestra necesarios para la citometría de flujo de células CLL. Cada experimento debe incluir todos los controles adecuados para un análisis preciso de los resultados obtenidos.
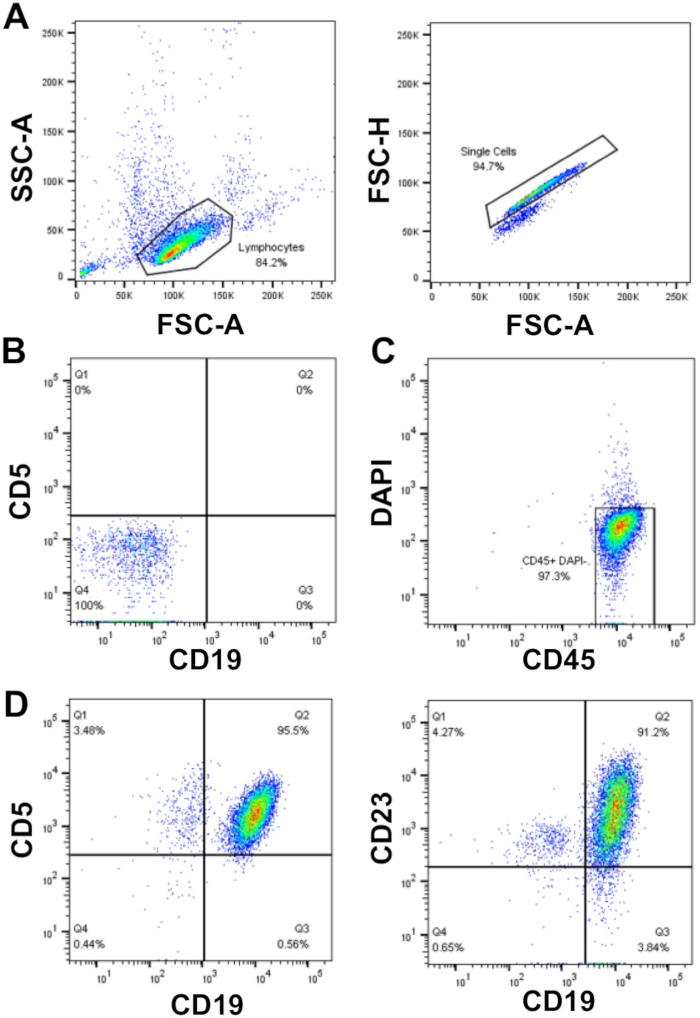
Figura 1: Gráfica representativa del análisis de la citometría de flujo de pacientes con LLC enriquecida. Las células mononucleares-CLL enriquecidas a partir de la sangre periférica de un paciente de LLC individual fueron cerradas usando FSC-A vs. SSC-A, y luego se excluyeron los dobletes usando FSC-A vs FSC-H (A). Se utilizaron células no retenidas (tubo 1) y controles de compensación (tubos 2-6) para configurar el citómetro de flujo para detectar células y compensar entre los canales fluorescentes, asegurando así que las señales de fluorescencia se detectaran correctamente. (B) Un ejemplo de tinción negativa (células no manchadas; tubo 1) en los canales de fluorescencia CD19 y CD5. Las células positivas en vivo (DAPI negativa) y CD45 estaban cerradas (C) y la proporción de CD19+CD5+ (95,5%) y CD19+CD23+ (91,2%) células dentro de la DAPI-CD45+ población se determinó (D). Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Optimización del fraccionamiento nuclear/citoplasma. Las fracciones citoplasmáticas y nucleares, y los lysatos de células enteras (ClL), se prepararon a partir de gránulos celulares (10 – 20 x 106 células) de la línea celular de la CLL (A) MEC1 o (B) células primarias de LlC enriquecidas a partir de la sangre periférica de los pacientes, como se describe en Paso 3. La optimización del fraccionamiento subcelular se llevó a cabo utilizando una gama de proporciones de detergente (1:20 a 1:60) al preparar la fracción citoplasmática (como se describe en el paso 3.4). Las muestras resultantes fueron inmunoblsidos y sondeados con anticuerpos anti-Lamina A/C (nuclear) y anti-tubulina (citoplasmática) para confirmar el fraccionamiento celular exitoso junto con la ClM. Los marcadores de peso molecular se muestran a la izquierda de la mancha (M). * indica las condiciones óptimas de detergente para la lisis celular. (C) Inmunoblot de fracciones nucleares y citoplasmáticas de células MEC1 con condiciones de control (NDC) o tratamiento farmacológico (8055) en presencia de ausencia de estimulación (+ o – reticulación BCR respectivamente). Las manchas fueron sondeadas con anticuerpos antiRbp1 CTD (clon 4H8; reconociendo la subunidad B1 del ARN polimerasa II), anti-Lamin A/C, anti-tubulina o anti-tubulina (clon GTU-88), para identificar fracciones subcelulares. Haga clic aquí para ver una versión más grande de esta figura.
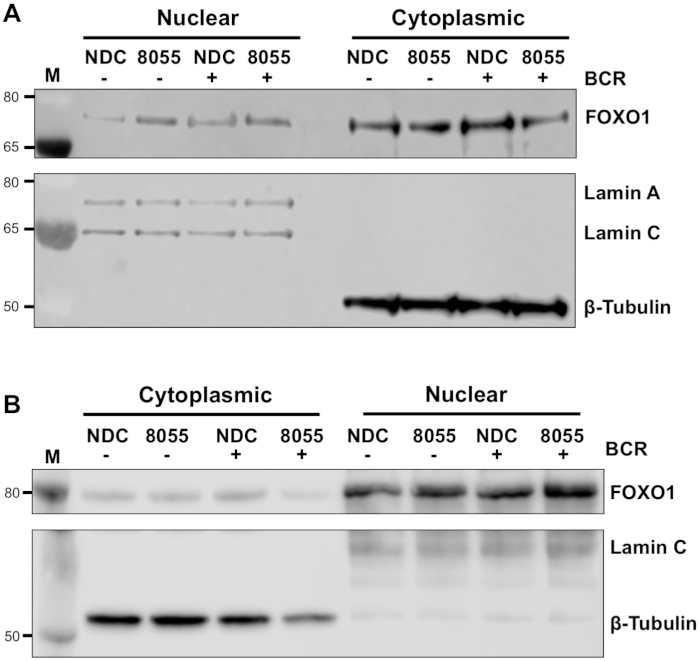
Figura 3: El fraccionamiento subcelular demuestra el cierre de FOXO1 entre el núcleo y el citoplasma en la LL. (A) las células MEC-1 y (B) las células CLL primarias se pretrataron durante 30 min con 100 nM AZD8055 (8055) o se dejaron sin tratar (NDC) como se indica y luego el BCR fue ligado durante 1 h o a la izquierda de los EE.UU. Luego se prepararon e inmunoblólaste fracciones nucleares y citoplasmáticas. Tras la confirmación del fraccionamiento sondeando con anticuerpos anti-Lamin A/C (nuclear) y anti-tubulina (citoplasmática), el efecto tanto del tratamiento farmacológico como de la ligadura BCR se evaluó en la expresión de la proteína FOXO1, utilizando un anticuerpo anti-FOXO1. M indica marcador de peso molecular. Haga clic aquí para ver una versión más grande de esta figura.
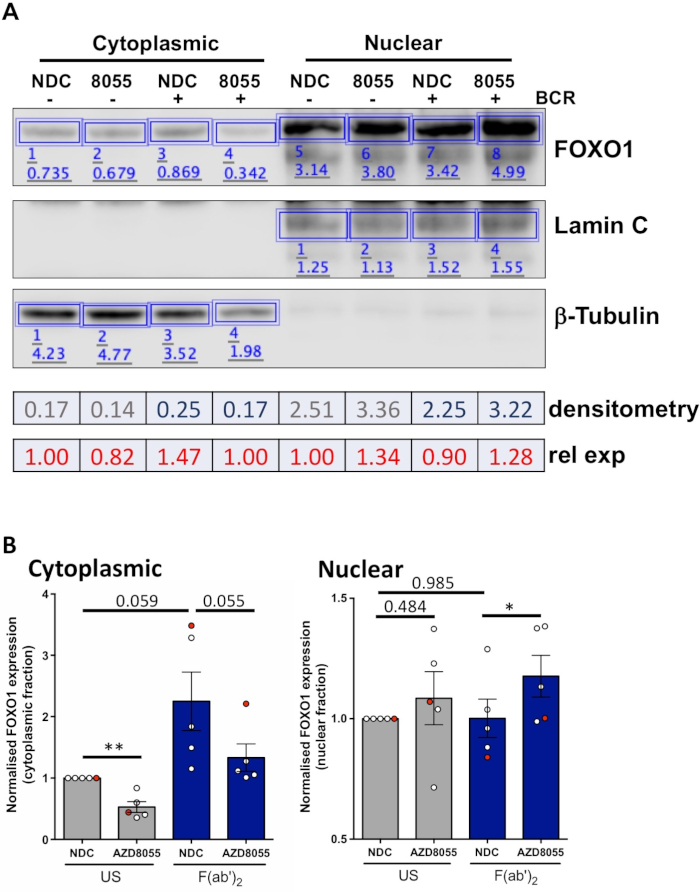
Figura 4: Un ejemplo trabajado de análisis cuantitativo de manchas occidentales (densitometría). (A) La densitometría se realizó utilizando el software de análisis de manchas occidentales disponible en línea. Brevemente, dentro de la cinta Análisis, se dibujaron rectángulos alrededor de las bandas de proteínas de la imagen para calcular la intensidad de la señal. Se representa la densitometría de una imagen representativa de la mancha occidental de una muestra de paciente de LLA que se sometió a fraccionamiento citoplasmático/nuclear. Las fracciones citolasmáticas y nucleares se distinguen por la expresión de marcadores citoplasma (a-tubulina) y nuclear (Lamin A/C). La expresión normalizada de FOXO1 para una condición determinada se puede calcular dividiendo la señal obtenida para FOXO1 por la señal correspondiente para el a-Tubulino o Lamin A/C, dependiendo de la fracción que se analice. La expresión relativa FOXO1 (en relación con el control del vehículo estadounidense), se puede calcular dividiendo la expresión FOXO1 normalizada de una condición dada por la expresión normalizada FOXO1 del control del vehículo estadounidense de una fracción celular dada. (B) Gráfico que muestra los niveles de expresión FOXO1 en las fracciones citoplasmáticas (izquierda) o nuclear (derecha) normalizadas al control US-NDC dentro de cada fracción celular. El punto rojo en el gráfico es el ejemplo trabajado que se muestra. Estos datos muestran el cambio de pliegue promedio en la expresión FOXO1 en comparación con los valores de US-NDC a SEM. P fueron determinados por la prueba t emparejada de los estudiantes de dos colas. n 5 muestras individuales de pacientes con LLC. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El protocolo descrito proporciona un método rápido y eficiente para la generación de fracciones nucleares y citoplasmáticas a partir de células CLL primarias, y la cuantificación posterior del tráfico de proteínas entre las fracciones nucleares y citoplasmáticas tras la estimulación celular y el tratamiento farmacológico. Los datos presentados demuestran la capacidad de detectar el tráfico de proteínas específicas, por ejemplo, FOXO1, entre las fracciones nuclear y citoplasmática, tras el tratamiento con un inhibidor mTOR dual AZD8055 en presencia/ausencia de reticulación BCR a través de F( ab')2 estimulación de fragmentos(Figura 3 y Figura 4). El acoplamiento de estos experimentos con la cuantificación de las manchas occidentales a partir de muestras individuales de pacientes con LLC, permite análisis objetivos de los datos generados y demuestra la robustez del ensayo descrito para cuantificar los cambios globales en la localización de proteínas en Células CLL aisladas de cohortes de pacientes(Figura 4). De los datos se desprende claramente que un promedio de cinco muestras de pacientes en las fracciones citoplasmáticas alcanzó un significado cercano. Dada la heterogeneidad clínica de los pacientes con LLC11, estos análisis normalmente se llevarían a cabo en cohortes de pacientes más grandes, y/o se centrarían en subgrupos de pronóstico específicos de pacientes para obtener una comprensión más completa de la respuesta celular de la LL células a tratamientos farmacológicos específicos.
Los datos presentados demuestran la importancia de elegir marcadores proteicos que residan exclusivamente en las fracciones citoplasmáticas o nucleares, ya que la pureza del fraccionamiento será confirmada por estos marcadores. Se eligió la tubulina para la confirmación de la fracción citoplasmática, y Lamin A/C como marcador nuclear. Otras proteínas que se utilizan comúnmente son GAPDH y -tubulina para identificar la fracción citoplasmática o Brg1 (SMARCA4), TFIID y RNA Polimerasa II para la pureza de la fracción nuclear4,5. Sin embargo, se debe tener cuidado al elegir proteínas que están altamente enriquecidas en fracciones específicas, y que no están presentes en ambas fracciones (por ejemplo, -tubulina) (Figura 2C)9. De hecho, GAPDH y actina, aunque generalmente se consideran proteínas citoplasmáticas pueden localizarse al núcleo12,13,destacando la importancia de elegir un marcador de fracción que no se reubique cuando la estimulación o el tratamiento es aplicado a las células. Además, es importante confirmar que el marcador de proteína elegido se expresa en la célula de interés mediante la ejecución de la WCL junto con los fraccionamientos subcelulares.
En el experimento representativo mostrado, se utilizó el mismo número de células CLL para cada condición (tratamiento de estimulación/drogas), y a partir de entonces se prepararon inmediatamente las muestras de fraccionamiento. La carga de 10 g de proteína/carril fraccionado proporciona material suficiente para la detección de las proteínas de interés. Dado que estas muestras sólo se sometieron a un tratamiento y estimulación farmacológica a corto plazo (hasta 4 h), se asumió que el nivel de proteína seguiría siendo el mismo en cada muestra, y no se realizó un ensayo proteico. Sin embargo, si los tratamientos celulares se extienden (18 - 72 h), el nivel de muerte celular o proliferación en las células puede alterar significativamente la calidad y la cantidad de proteína extraída, dependiendo de la estimulación de fármacos/células aplicada, alterando así los niveles de proteína en el tratado /muestras estimuladas. En estos casos para tratamientos farmacológicos a largo plazo, es aconsejable llevar a cabo la cuantificación de proteínas utilizando un ensayo de Bradford o equivalente, antes de la hincha occidental para asegurar que la misma cantidad de proteína se ejecuta en cada carril del inmunoblot. La presencia de detergentes puede interferir con ensayos proteicos específicos14, esta interferencia se puede reducir diluyendo muestras de proteínas de fracción celular. Además, utilice el tampón de lisis completo como el espacio en blanco, utilizando la misma dilución que en las muestras que se están probando.
Para proporcionar pruebas justificativas de los hallazgos descritos aquí, se podrían realizar experimentos paralelos mediante microscopía de fluorescencia para analizar la ubicación de FOXO1 dentro de las células CLL para permitir la visualización de estos hallazgos5. Además, las fracciones subcelulares generadas también se pueden utilizar para ensayos de actividad enzimática o análisis proteómicos en análisis posteriores.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores quieren agradecer a la Dra. Natasha Malik por revisar críticamente el manuscrito. Este estudio fue financiado por una beca de proyecto Bloodwise otorgada a AMM (18003). Las instalaciones de análisis de FACS fueron financiadas por la Fundación Howat. MWM fue financiado por una beca de doctorado del Centro de Investigación de la Leucemia Friends of Paul O'Gorman, JC fue financiado por el Centro de Investigación de Leucemia De Paul O'Gorman y JH fue financiado por una beca de proyecto Bloodwise (18003).
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge Tubes | Griener Bio one | 616201 | |
| 3 mL Pasteur Pipettes | Griener Bio one | 612398 | |
| 12 mm x 75 mm FACS Tubes | Elkay | 2052-004 | |
| 15 mL Tube | Griener Bio one | 188271 | |
| 50 mL Tube | Griener Bio one | 227261 | |
| anti-CD5 FITC antibody | BD Biosciences | 555352 | phenotypic surface marker |
| anti-CD19 PE Cy7 antibody | BD Biosciences | 557835 | phenotypic surface marker |
| anti-CD23 APC antibody | BD Biosciences | 558690 | phenotypic surface marker |
| anti-CD45 APC Cy7 antibody | BD Biosciences | 557833 | phenotypic surface marker |
| anti-β-Tubulin antibody | Cell Signaling | 2146 | cytoplasmic marker |
| anti-γ-Tubulin Mouse antibody (clone GTU-88) | Sigma-Aldrich | T5326 | |
| anti-FoxO1 (C29H4) Rabbit antibody | Cell Signaling | 2880 | |
| anti-Lamin A/C antibody | Cell Signaling | 2032 | nuclear marker |
| anti-mouse IgG, HRP-linked Antibody | Cell Signaling | 7076 | Secondary antibody |
| anti-rabbit IgG, HRP-linked Antibody | Cell Signaling | 7074 | Secondary antibody |
| anti-Rpb1 CTD antibody (clone 4H8) | Cell Signaling | 2629 | nuclear marker |
| BDFACS Canto II | BD Biosciences | By Request | Flow Cytometer |
| DAPI Solution | BD Biosciences | 564907 | live/dead discriminator |
| DMSO | Sigma | D2650 | |
| EDTA | Sigma | EDS | |
| Fetal Bovine Serum | Thermofisher | 10500064 | |
| GraphPad Prism 6 | GraphPad Software | Software | |
| Histopaque1077 density gradient media | Sigma | H8889 | |
| HyperPAGE 10 - 190 kDa protein marker | Bioline | BIO-33066 | Molecular weight marker |
| Image Studio Lite (version 5.2.5) | LI-COR | www.licor.com | Software |
| Labnet VX100 | Fisher Scientific | Vortex | |
| Nucelar Extract Kit | Active Motif | 40010 | |
| OneComp eBeads | Thermofisher | 01-111-42 | |
| Trypan Blue Solution | Thermofisher | 15250061 | |
| PageRuler Plus Prestained Protein Ladder | Thermo Fisher Scientific | 26619 | Molecular weight marker |
| PBS Tablets | Fisher Scientific | BR0014G | |
| RosetteSep Human B Cell Enrichment Cocktail | Stem Cell Technologies | 15064 | |
| Sigma 3-16P | SciQuip | Centrifuge | |
| Sigma 1-15PK | SciQuip | Centrifuge |
Referencias
- Kau, T. R., Way, J. C., Silver, P. A. Nuclear transport and cancer: from mechanism to intervention. Nature Reviews Cancer. 4, 106-117 (2004).
- Turner, J. G., Dawson, J., Sullivan, D. M. Nuclear export of proteins and drug resistance in cancer. Biochemical Pharmacology. 83, 1021-1032 (2012).
- Nakamura, N., et al. Forkhead transcription factors are critical effectors of cell death and cell cycle arrest downstream of PTEN. Molecular and Cellular Biology. 20, 8969-8982 (2000).
- Calnan, D. R., Brunet, A. The FoxO code. Oncogene. 27, 2276-2288 (2008).
- Cosimo, E., et al. AKT/mTORC2 inhibition activates FOXO1 function in CLL cells reducing B cell receptor-mediated survival. Clinical Cancer Research. 25, 1574-1587 (2019).
- Mahipal, A., Malafa, M. Importins and exportins as therapeutic targets in cancer. Pharmacology & Therapeutics. 164, 135-143 (2016).
- Hing, Z. A., et al. Next-generation XPO1 inhibitor shows improved efficacy and in vivo tolerability in hematological malignancies. Leukemia. 30, 2364-2372 (2016).
- Lapalombella, R., et al. Selective inhibitors of nuclear export show that CRM1/XPO1 is a target in chronic lymphocytic leukemia. Blood. 120, 4621-4634 (2012).
- Hořejší, B., et al. Nuclear γ-tubulin associates with nucleoli and interacts with tumor suppressor protein C53. Journal of Cellular Physiology. 227, 367-382 (2012).
- McCaig, A. M., Cosimo, E., Leach, M. T., Michie, A. M. Dasatinib inhibits B cell receptor signalling in chronic lymphocytic leukaemia but novel combination approaches are required to overcome additional pro-survival microenvironmental signals. British Journal of Haematology. 153, 199-211 (2011).
- Fischer, K., Hallek, M. Optimizing frontline therapy of CLL based on clinical and biological factors. Hematology. American Society of Hematology. Education Program. 2017, 338-345 (2017).
- Butera, G., et al. Regulation of autophagy by nuclear GAPDH and its aggregates in cancer and neurodegenerative disorders. International Journal of Molecular Sciences. 20, 2062 (2019).
- Virtanen, J. A., Vartiainen, M. K. Diverse functions for different forms of nuclear actin. Current Opinion in Cell Biology. 46, 33-38 (2017).
- Friedenauer, S., Berlet, H. H. Sensitivity and variability of the Bradford protein assay in the presence of detergents. Analytical Biochemistry. 178, 263-268 (1989).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados