È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Saggi di trasferimento di energia di Förster Resonance risolti nel tempo per la misurazione di proteine STAT fosforilate endogene in cellule umane
In questo articolo
Riepilogo
I protocolli di analisi basati su cellule di trasferimento di energia di risonanza di Förster risolti nel tempo sono descritti per la quantificazione semplice, specifica, sensibile e robusta del trasduttore di segnale fosforilato endogeno e dell'attivatore della trascrizione (STAT) 1/3/4/5/6 proteine in lisati cellulari in un formato a 384 pozzetti.
Abstract
La via di segnalazione Janus chinasi (JAK)/trasduttore di segnale e attivatore della trascrizione (STAT) svolge un ruolo cruciale nel mediare le risposte cellulari alle citochine e ai fattori di crescita. Le proteine STAT sono attivate dalla fosforilazione tirosina mediata principalmente dai JAB. L'attivazione anomala delle vie di segnalazione STAT è implicata in molte malattie umane, in particolare il cancro e le condizioni immuno-correlate. Pertanto, la capacità di monitorare la fosforilazione della proteina STAT all'interno dell'ambiente di segnalazione cellulare nativo è importante sia per la ricerca accademica che per la scoperta di farmaci. I formati di analisi tradizionali disponibili per quantificare le proteine STAT fosforilate includono il western blotting e il saggio immunoassorbinte enzimatico (ELISA). Questi metodi eterogenei sono laboriosi, a bassa produttività e spesso non affidabili (specifici) nel caso del western blotting. Sono disponibili metodi omogenei (senza lavaggio) ma rimangono costosi.
Qui vengono forniti protocolli dettagliati per la misurazione sensibile, robusta ed economica in un formato a 384 pozzetti di livelli endogeni di STAT1 fosforilato (Y701), STAT3 (Y705), STAT4 (Y693), STAT5 (Y694/Y699) e STAT6 (Y641) in lisati cellulari da cellule aderenti o in sospensione utilizzando la nuova piattaforma THUNDER time-resolved Förster resonance energy transfer (TR-FRET). Il flusso di lavoro per il test cellulare è semplice, veloce e progettato per lo screening ad alto rendimento (HTS). Il protocollo di analisi è flessibile, utilizza un campione a basso volume (15 μL), richiede una sola fase di aggiunta del reagente e può essere adattato ad applicazioni a bassa produttività e ad alta produttività. Ogni test immunologico sandwich fosfo-STAT viene convalidato in condizioni ottimizzate con agonisti e inibitori noti e genera i valori attesi di farmacologia e fattore Z. Poiché i saggi TR-FRET sono raziometrici e non richiedono passaggi di lavaggio, forniscono una riproducibilità molto migliore rispetto agli approcci tradizionali. Insieme, questa suite di saggi fornisce nuovi strumenti economici per un'analisi più completa di specifiche proteine STAT fosforilate dopo il trattamento cellulare e lo screening e la caratterizzazione di modulatori specifici e selettivi della via di segnalazione JAK / STAT.
Introduzione
La via di segnalazione JAK/STAT svolge un ruolo chiave nel mediare le risposte cellulari a diverse citochine, interferoni, fattori di crescita e molecole correlate1,2. Il legame di questi ligandi a specifici recettori della superficie cellulare provoca l'attivazione di JAK, che a loro volta attivano le proteine STAT mediante fosforilazione di specifici residui di tirosina. La fosforilazione STAT provoca la loro dimerizzazione e traslocazione nel nucleo, dove esercitano il loro effetto sulla trascrizione dei geni bersaglio regolati. La famiglia STAT è composta da sette membri: STAT1, STAT2, STAT3, STAT4, STAT5a, STAT5b e STAT6. I membri svolgono un ruolo complesso ed essenziale nella regolazione dei processi cellulari fisiologici, tra cui proliferazione, differenziazione, apoptosi, angiogenesi e regolazione del sistema immunitario. L'attivazione anomala delle vie di segnalazione STAT è implicata in molte malattie umane, in particolare il cancro e le condizioni immuno-correlate3,4. Pertanto, la capacità di valutare la fosforilazione della proteina STAT all'interno dell'ambiente di segnalazione cellulare nativo è importante sia per la ricerca accademica che per la scoperta di farmaci.
Ad oggi, i metodi convenzionali utilizzati per misurare i livelli di proteine fosforilate intracellulari, compresi gli ST, sono basati su anticorpi e includono western blotting, ELISA e citometria a fosfoflusso. Questi metodi eterogenei sono laboriosi, dispendiosi in termini di tempo, soggetti a errori, a bassa produttività e spesso inaffidabili (ad esempio, problemi di specificità) nel caso del western blotting5. Al contrario, i saggi omogenei richiedono meno passaggi sperimentali, utilizzano volumi di campione più piccoli e sono suscettibili di HTS. Esistono cinque piattaforme di immunodosaggio omogenee basate su cellule disponibili in commercio che possono essere utilizzate per monitorare quantitativamente la fosforilazione JAK-dipendente degli ST nei lisati cellulari: SureFire, HTRF, LANCE, LanthaScreen e Lumit. Ognuna di queste piattaforme ha i suoi vantaggi e svantaggi.
SureFire si basa sulla tecnologia di canalizzazione dell'ossigeno luminescente, che utilizza perline di donatore e accettore rivestite per catturare specificamente un paio di anticorpi, uno dei quali è biotinilato. In presenza di proteine fosforilate, i due anticorpi portano le perle del donatore e dell'accettore nelle immediate vicinanze, consentendo la generazione di un segnale chemiluminescente6. Sebbene versatile e sensibile, questa tecnologia è costosa, è influenzata dalla biotina nel terreno di coltura, è molto sensibile alla temperatura ambiente e alla luce e richiede un lettore speciale per il rilevamento. HTRF e LANCE sono entrambi basati sulla tecnologia TR-FRET che utilizza complessi ioni di lantanidi luminescenti di lunga durata (chelati di europio o terbio o criptato di europio) come molecole donatrici e fluorofori rosso lontano come molecole accettori7. Quando due anticorpi protein-specifici etichettati con molecole donatori o accettori vengono portati nelle immediate vicinanze, si verifica FRET, causando un aumento della fluorescenza dell'accettore e una diminuzione della fluorescenza del donatore. Questi segnali fluorescenti di lunga durata possono essere misurati in modo tempo e raziometrico per ridurre le interferenze del test e aumentare la qualità dei dati. Altri vantaggi di TR-FRET sono che non è sensibile alla luce, consente letture ripetute e presenta una lunga stabilità del segnale. Mentre TR-FRET è ampiamente implementato in HTS grazie alla sua versatilità, sensibilità e elevata robustezza, tutte le piattaforme di analisi commerciali basate su TR-FRET sono costose, precludendo così la sua ampia adozione in laboratori accademici e piccoli laboratori industriali. Il test LanthaScreen utilizza anche una lettura basata su TR-FRET, ma si basa su una linea cellulare U2OS ingegnerizzata che esprime stabilmente la proteina fluorescente verde (GFP)-STAT1 combinata con un anticorpo STAT1 fosfo-specifico marcato con terbio8. Oltre ad essere limitato in termini di scelta delle proteine di segnalazione, questo metodo richiede l'acquisto di costose linee cellulari trasfettate, riducendone l'applicabilità e aumentando la possibilità di artefatti sperimentali. Lumit è una piattaforma di immunodosaggio bioluminescente generico che utilizza anticorpi secondari (anti-topo e anti-coniglio) chimicamente etichettati con le piccole e grandi subunità NanoBit di NanoLuc Luciferase9. Il legame di due anticorpi primari alla proteina bersaglio porta gli anticorpi secondari in prossimità per formare un enzima attivo che genera un segnale di luminescenza. Mentre la luminescenza è generalmente una lettura sensibile e robusta, il requisito di anticorpi primari allevati in due specie diverse limita le scelte per la progettazione del saggio. Inoltre, l'uso di anticorpi secondari in matrici di campioni complessi può essere soggetto a interferenze di analisi.
Pertanto, esiste ancora la necessità di una piattaforma di analisi basata su cellule affidabile, rapida ma conveniente per misurare le singole proteine STAT fosforilate e totali in modo compatibile con HTS. Per rispondere a questa esigenza, è stata sviluppata una nuova piattaforma di analisi immunologica basata su cellule ad alto rendimento basata su una tecnologia TR-FRET avanzata (THUNDER) e progettata per consentire una misurazione semplice, sensibile, robusta ed economica delle proteine intracellulari espresse endogenamente (fosforilate o totali) nei lisati cellulari. I vantaggi di questa tecnologia derivano dalla combinazione di una coppia FRET donatore/accettore che presenta un'eccezionale compatibilità spettrale e un segnale TR-FRET, anticorpi rigorosamente convalidati e tamponi di lisi ottimizzati. Questi test sono formattati come test immunologici sandwich e utilizzano un flusso di lavoro semplice in tre fasi (Figura 1). Le cellule vengono prima trattate per modulare la fosforilazione proteica e poi lisate con lo specifico tampone di lisi fornito nel kit. La proteina STAT fosforilata target o totale nel lisato cellulare viene rilevata in una singola fase di aggiunta e incubazione del reagente con una coppia di anticorpi marcati con fluoroforo che riconoscono epitopi distinti sulla proteina bersaglio (Figura 2). Un anticorpo è etichettato con un donatore di chelato di europio (Eu-Ab1), mentre il secondo anticorpo è etichettato con un fluoroforo accettore rosso lontano (FR-Ab2). I due anticorpi marcati si legano alla proteina in soluzione, portando le due etichette nelle immediate vicinanze. L'eccitazione del donatore chelato di europio a 320 o 340 nm innesca un FRET all'accettore, che emette un segnale TR-FRET di lunga durata a 665 nm proporzionale alla concentrazione di proteina bersaglio (fosforilata o totale) nel lisato cellulare.
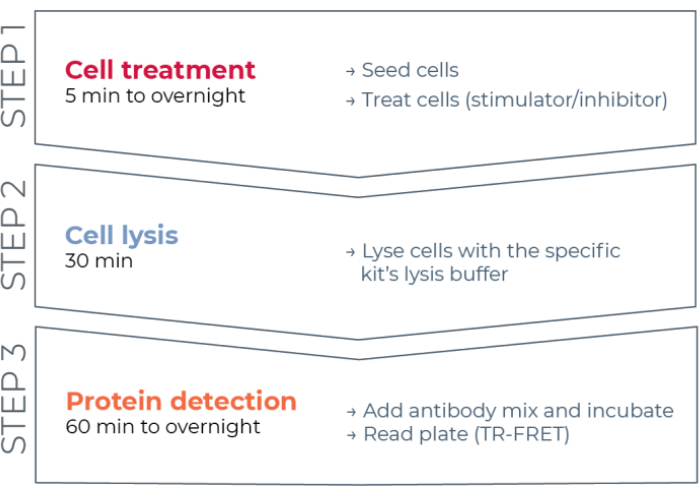
Figura 1: Flusso di lavoro del saggio TR-FRET. Il flusso di lavoro consiste in tre fasi: trattamento cellulare, lisi cellulare e rilevamento delle proteine utilizzando TR-FRET. Nel protocollo di analisi a due piastre, i lisati vengono trasferiti su una piastra di rilevamento bianca a 384 pozzetti, mentre nel protocollo a una piastra, tutti i passaggi sono condotti nella stessa piastra di rilevamento bianca a 384 pozzetti (protocollo all-in-one-well). Indipendentemente dal protocollo di analisi utilizzato, il rilevamento delle proteine viene eseguito nello stesso volume totale (20 μL per pozzetto). Abbreviazione: TR-FRET = trasferimento di energia di risonanza di Förster risolto nel tempo. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Principio del test immunologico sandwich TR-FRET. Un anticorpo è etichettato con il donatore di chelato di europio (Eu-Ab1) e il secondo con l'accettore di fluoroforo piccolo rosso lontano (FR-Ab2). I due anticorpi marcati si legano specificamente a epitopi distinti sulla proteina bersaglio (fosforilata o totale) nel lisato cellulare, portando i due fluorofori nelle immediate vicinanze. L'eccitazione del donatore chelato di europio a 320 o 340 nm innesca un FRET dal donatore alle molecole accettori, che a loro volta emettono un segnale a 665 nm. Questo segnale è proporzionale alla concentrazione di proteine nel lisato cellulare. In assenza della proteina bersaglio specifica, i fluorofori donatore e accettore sono troppo distanti l'uno dall'altro perché si verifichi FRET. Abbreviazioni: FRET = Förster resonance energy transfer; TR-FRET = FRET risolto nel tempo; Ab = anticorpo; FR = rosso lontano; Eu - Chelato di europio; P = fosforilazione. Fare clic qui per visualizzare una versione più grande di questa figura.
Qui vengono forniti protocolli dettagliati per misurare, in un formato a 384 pozzetti, i livelli intracellulari di STAT1 fosforilato (Y701), STAT3 (Y705), STAT4 (Y693), STAT5 (Y694/Y699) e STAT6 (Y641), insieme a STAT1, STAT3, STAT5 e STAT6 totali, in lisati cellulari da cellule aderenti o in sospensione utilizzando la piattaforma THUNDER TR-FRET. Questi protocolli definiscono i passaggi per il trattamento cellulare, la lisi e il rilevamento di proteine bersaglio basate su TR-FRET utilizzando un protocollo di trasferimento a due piastre o un protocollo all-in-one-well a una piastra. Questi saggi basati su cellule sono applicati per determinare il profilo farmacologico di attivatori e inibitori noti della via JAK/STAT. La robustezza e l'idoneità dei saggi selezionati per HTS sono dimostrate. Infine, vengono discussi esperimenti chiave per l'ottimizzazione del test, insieme a raccomandazioni per la risoluzione dei problemi del test.
Protocollo
1. Coltura cellulare
- Mantenere le cellule in un incubatore e coltura a CO2 umidificata a 37 °C / 5% con DMEM integrato con siero bovino fetale al 10% (FBS) (cellule HeLa e A431) o RPMI integrato con il 15% fbS (cellule U266B1). Coltiva le cellule fino a quando non raggiungono il 70-80% di confluenza, quindi tripsinilizzale e passale o usale per i test.
NOTA: i terreni di coltura contenevano rosso fenolo. Nessuna fame sierica è stata condotta per nessuna linea cellulare prima di condurre i test.
2. Titolazione dello stimolatore o dell'inibitore utilizzando il protocollo di dosaggio a due piastre con cellule aderenti
NOTA: questa procedura descrive come determinare le potenze dello stimolatore o dell'inibitore generando una curva concentrazione-risposta da una serie di diluizione del composto in esame.
- Semina cellulare
- Erogare 50 μL di cellule alla densità pre-ottimizzata (40.000 cellule HeLa/pozzetto sia per STAT3 che per STAT6; 75.000 cellule A431/pozzetto per STAT5) in una piastra trattata con coltura tissutale a 96 pozzetti nel terreno di coltura appropriato. Incubare durante la notte a 37 °C/5% DI CO2.
NOTA: è necessario determinare la densità cellulare ottimale e il tempo di incubazione della coltura.
- Erogare 50 μL di cellule alla densità pre-ottimizzata (40.000 cellule HeLa/pozzetto sia per STAT3 che per STAT6; 75.000 cellule A431/pozzetto per STAT5) in una piastra trattata con coltura tissutale a 96 pozzetti nel terreno di coltura appropriato. Incubare durante la notte a 37 °C/5% DI CO2.
- Diluizioni di composti in esame
- Preparare serie intermedie di diluizione 2x di composti di prova diluendo in serie i composti (diluizioni a intervalli di mezzo log) attraverso 12 pozzetti di una piastra di polipropilene a 96 pozzetti in un mezzo privo di siero.
NOTA: si raccomanda di condurre una curva concentrazione-risposta a 12 punti, intervallo di mezzo log, almeno duplicata per una stima accurata dell'EC50 o dell'IC50. - In alternativa, per i composti di prova idrofobici solubili in dimetilsolfossido (DMSO), eseguire le diluizioni iniziali in DMSO al 100% e quindi diluire la serie di diluizione composta in mezzo privo di siero.
NOTA: la tolleranza del saggio al DMSO deve essere stabilita prima di eseguire una titolazione del composto di prova nel veicolo DMSO. È importante mantenere uguali concentrazioni di solvente tra cellule trattate e non trattate. Inoltre, quando si testano le diluizioni seriali dei composti, le concentrazioni di solvente devono rimanere sempre costanti in tutta la serie di diluizione.
- Preparare serie intermedie di diluizione 2x di composti di prova diluendo in serie i composti (diluizioni a intervalli di mezzo log) attraverso 12 pozzetti di una piastra di polipropilene a 96 pozzetti in un mezzo privo di siero.
- Trattamento cellulare
- Per la stimolazione cellulare, aggiungere 50 μL di mezzo privo di siero da solo (cellule non trattate) o contenente lo stimolatore (2x).
- Incubare per il tempo pre-ottimizzato a temperatura ambiente (RT) o 37 °C (interferone (IFN) α2b/20 min a RT per STAT3; fattore di crescita epidermico (EGF)/10 min a RT per STAT5; interleuchina (IL)-4/20 min a RT per STAT6). Passare quindi alla sezione 2.4.
NOTA: è necessario determinare la temperatura di incubazione ottimale. - Per l'inibizione cellulare, aggiungere 25 μL di mezzo libero da siero da solo (cellule non trattate) o contenente l'inibitore (4x).
- Incubare per il tempo pre-ottimizzato a RT o 37 °C (inibitore JAK 1/30 min a RT per STAT3 e STAT6; Erlotinib/15 min a RT per STAT5).
- Aggiungere 25 μL di mezzo privo di siero da solo (cellule non trattate) o contenente lo stimolatore (4x) al suo EC80.
- Incubare per il tempo pre-ottimizzato a RT o 37 °C (stesse condizioni del passaggio 2.3.2).
- Lisi cellulare
- Preparare il buffer di lisi integrato 1x specifico del kit come indicato dal produttore.
NOTA: È obbligatorio integrare il tampone di lisi 1x con il cocktail inibitore della fosfatasi 100x diluito ad una concentrazione finale di 1x. Il 1x Tampone di Lisi Integrato contiene 1 mM di fluoruro di sodio, 2 mM di ortovanadato di sodio e 2 mM di beta-glicerofosfato. Altri inibitori della fosfatasi non sono necessari e devono essere evitati. I tamponi di lisi e gli inibitori della fosfatasi diversi da quelli inclusi nel kit non sono raccomandati in quanto potrebbero contenere ingredienti che potrebbero interferire con la misurazione. - Rimuovere con cura ed eliminare il terreno di coltura cellulare aspirando il surnatante.
- Aggiungere immediatamente 50 μL di 1x tampone di lisi integrato.
NOTA: il volume del buffer di lisi (25-50 μL) può essere ottimizzato. - Incubazione per 30 minuti a RT sotto agitazione (agitatore a piastre orbitali impostato a 400 giri / min; agitazione moderata).
NOTA: il tempo di incubazione della lisi (30-60 min) può essere ottimizzato. I lisati possono essere utilizzati immediatamente per il rilevamento di proteine bersaglio o congelati a -80 °C.
- Preparare il buffer di lisi integrato 1x specifico del kit come indicato dal produttore.
- Rilevamento TR-FRET
- Preparare il 4x Antibody Detection Mix in 1x Detection Buffer come indicato dal produttore.
- In questa fase di trasferimento, pipettare con cura 15 μL di lisato cellulare dalla piastra di coltura a 96 pozzetti a un pozzo di una micropiastra bianca a basso volume da 384 pozzetti.
- Aggiungere 15 μL di lisato di controllo positivo e 15 μL di 1x tampone di lisi (controllo negativo) per separare i pozzetti di analisi.
- Aggiungere 5 μL di 4x Antibody Detection Mix (Eu-Ab1/FR-Ab2 per il rilevamento della fosfo-proteina o Eu-Ab3/FR-Ab4 per il rilevamento della proteina totale) a ciascuno dei pozzetti di analisi.
- Coprire la piastra con un sigillante a piastre e incubare per 1 ora fino a notte a RT, a seconda del test (vedere la scheda tecnica corrispondente).
NOTA: il tempo di lettura ottimale deve essere ottimizzato per ogni test e linea cellulare. La piastra può essere letta più volte senza un effetto negativo sulle prestazioni del test. - Rimuovere il sigillante per piastre adesive e leggere la piastra su un lettore di micropiastre compatibile TR-FRET.
NOTA: si raccomandano fluorometri a base di filtro, anche se è possibile utilizzare alcuni strumenti monocromatori. Verificare che sia installato il modulo ottico appropriato (filtri e specchio) per TR-FRET. Utilizzare una lunghezza d'onda di eccitazione di 320 o 340 nm per eccitare il chelato di europio. Leggere i saggi sia a 615 nm (o 620 nm) che a 665 nm per rilevare sia l'emissione dall'europio donatore che dal fluoroforo accettore, rispettivamente. Le impostazioni dello strumento dipenderanno dal particolare lettore. I dati qui presentati sono stati ottenuti utilizzando l'eccitazione basata su lampada, il ritardo di 90 μs, il tempo di integrazione di 300 μs e 100 lampi per pozzetto. Il test fosfo-STAT4, tuttavia, è stato letto utilizzando l'eccitazione laser per generare rapporti segnale-sfondo (S / B) più elevati.
3. Titolazione dello stimolatore o dell'inibitore utilizzando il protocollo di dosaggio a due piastre con cellule in sospensione
- Diluizione dei composti in esame
- Preparare serie intermedie di diluizione 2x di composti in esame come descritto nei passaggi 2.2.1 e 2.2.2.
- Semina e trattamento cellulare
- Erogare 20 μL di cellule alla densità pre-ottimizzata (200.000 cellule U266B1/pozzetto per STAT1; 400.000 cellule U266B1/pozzetto per STAT4) in una piastra trattata con coltura tissutale a 96 pozzetti nel terreno di coltura appropriato. Procedere direttamente al trattamento cellulare o incubare 2-4 ore a 37 °C, 5% CO2.
NOTA: questo passaggio deve essere ottimizzato per diversi tipi di celle. - Per la stimolazione cellulare, aggiungere 20 μL di mezzo privo di siero da solo (cellule non trattate) o contenente lo stimolatore (2x).
- Incubare per il tempo pre-ottimizzato a RT o 37 °C (IFNα2b/15 min a RT per STAT1; IFNα2b/25 min a 37 °C per STAT4). Procedere quindi alla sezione 3.3.
NOTA: è necessario determinare la temperatura di incubazione ottimale. - Per l'inibizione cellulare, aggiungere 10 μL di mezzo libero da siero da solo (cellule non trattate) o contenente l'inibitore (4x).
- Incubare per il tempo pre-ottimizzato a RT o 37 °C (inibitore JAK 1/30 min a RT per STAT1 e STAT4).
- Aggiungere 10 μL di mezzo privo di siero da solo (cellule non trattate) o contenente lo stimolatore (4x) al suo EC80.
- Incubare per il tempo pre-ottimizzato a RT o 37 °C (stesse condizioni del punto 3.2.3).
- Erogare 20 μL di cellule alla densità pre-ottimizzata (200.000 cellule U266B1/pozzetto per STAT1; 400.000 cellule U266B1/pozzetto per STAT4) in una piastra trattata con coltura tissutale a 96 pozzetti nel terreno di coltura appropriato. Procedere direttamente al trattamento cellulare o incubare 2-4 ore a 37 °C, 5% CO2.
- Lisi cellulare
- Preparare il buffer di lisi integrato 5x specifico del kit come indicato dal produttore.
NOTA: È obbligatorio integrare il tampone di lisi 5x con il cocktail inibitore della fosfatasi 100x diluito fino a una concentrazione finale di 5x. Il 5x Tampone di Lisi Integrato contiene 5 mM di fluoruro di sodio, 10 mM di ortovanadato di sodio e 10 mM di beta-glicerofosfato. Altri inibitori della fosfatasi non sono necessari e devono essere evitati. - Aggiungere 10 μL di 5x tampone di lisi integrato.
- Incubazione per 30 minuti a RT sotto agitazione (agitatore a piastre orbitali impostato a 400 giri / min; agitazione moderata).
NOTA: il tempo di incubazione della lisi (30-60 min) può essere ottimizzato. I lisati possono essere utilizzati immediatamente per il rilevamento di proteine bersaglio o congelati a -80 °C.
- Preparare il buffer di lisi integrato 5x specifico del kit come indicato dal produttore.
- Rilevamento TR-FRET
- Dopo la lisi cellulare, eseguire la fase di rilevamento TR-FRET come descritto nel paragrafo 2.5 per il protocollo di analisi a 2 piastre per le cellule aderenti.
4. Titolazione dello stimolatore utilizzando il protocollo di analisi a una piastra con cellule aderenti o in sospensione
- Diluizione dei composti in esame
- Preparare serie intermedie di diluizione di composti di prova a 3x diluendo in serie i composti (diluizioni a intervalli di mezzo log) attraverso 12 pozzetti di una piastra di polipropilene a 96 pozzetti in un mezzo privo di siero.
- Semina e trattamento cellulare
- Erogare 8 μL di cellule alla densità pre-ottimizzata (160.000 cellule U266B1/pozzetto per STAT4; 80.000 cellule HeLa/pozzetto per STAT6), nel terreno di coltura privo di siero appropriato, in una piastra bianca a basso volume da 384 pozzetti. Procedere direttamente al trattamento cellulare o incubare 2-4 ore a 37 °C, 5% CO2.
NOTA: Il requisito di un periodo di incubazione in coltura cellulare prima del trattamento deve essere determinato per diversi tipi di cellule. - Per la stimolazione cellulare, aggiungere 4 μL di mezzo privo di siero da solo (cellule non trattate) o contenente lo stimolatore (3x).
- Incubare per il tempo pre-ottimizzato a temperatura ambiente o 37 °C (IFNα2b/25 min a 37°C per STAT4; IL-4/20 min a 37°C per STAT6). Passare quindi alla sezione 4.3.
- Erogare 8 μL di cellule alla densità pre-ottimizzata (160.000 cellule U266B1/pozzetto per STAT4; 80.000 cellule HeLa/pozzetto per STAT6), nel terreno di coltura privo di siero appropriato, in una piastra bianca a basso volume da 384 pozzetti. Procedere direttamente al trattamento cellulare o incubare 2-4 ore a 37 °C, 5% CO2.
- Lisi cellulare
- Preparare il buffer di lisi integrato 5x specifico del kit come indicato dal produttore.
NOTA: È obbligatorio integrare il tampone di lisi 5x con il cocktail inibitore della fosfatasi 100x diluito fino a una concentrazione finale di 5x. - Aggiungere 3 μL di 5x Buffer di Lisi Integrato.
- Incubare per 30 minuti a RT sotto agitazione (agitatore a piastra orbitale a 400 giri / min).
NOTA: il tempo di incubazione della lisi (30-60 min) può essere ottimizzato. I lisati possono essere utilizzati immediatamente o congelati a 80 °C.
- Preparare il buffer di lisi integrato 5x specifico del kit come indicato dal produttore.
- Rilevamento TR-FRET
- Aggiungere 15 μL di lisato di controllo positivo (non diluito) e 15 μL di 1x tampone di lisi integrato (controllo negativo) a pozzi di analisi separati.
- Aggiungere 5 μL di 4x Antibody Detection Mix (Eu-Ab1/FR-Ab2 per la fosfo-proteina o Eu-Ab3-/FR-Ab4 per la proteina totale) preparato in 1x Detection Buffer a ciascuno dei pozzi di analisi.
- Coprire la piastra con un sigillante a piastre e incubare per 1 ora fino a notte a RT, a seconda del test.
NOTA: il tempo di lettura ottimale deve essere ottimizzato per ogni test e linea cellulare. La piastra può essere letta più volte senza un effetto negativo sulle prestazioni del test. - Rimuovere il sigillante per piastre adesive. Leggere la piastra su un lettore di micropiastre compatibile con TR-FRET.
5. Analisi dei dati
- Calcolare il rapporto TR-FRET per ciascun pozzo utilizzando la seguente formula (1):
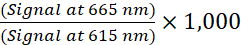 (1)
(1)
NOTA: poiché il segnale TR-FRET viene letto in modalità risolta nel tempo, la sottrazione di sfondo di solito non è necessaria. Se viene condotta la sottrazione di fondo, utilizzare i pozzetti privi di cellule contenenti il buffer di lisi integrato 1x (controllo negativo) per la sottrazione di sfondo. Determinare il rapporto medio TR-FRET dai pozzetti privi di cellule e quindi sottrarre questo valore dal rapporto TR-FRET di ciascun pozzo. - Per le curve concentrazione-risposta, analizzare i dati secondo una regressione non lineare utilizzando l'equazione logistica a 4 parametri (curva dose-risposta sigmoidale con pendenza variabile) e una ponderazione dei dati 1/Y2 per generare valori EC50 o IC50 .
- Per l'esperimento del fattore Z' analizzare i dati secondo la seguente formula (2)10
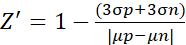 (2)
(2)
Dove μ e σ sono i valori medi e le deviazioni standard per il controllo positivo (p; cellule stimolate) e il controllo negativo (n; cellule non trattate), rispettivamente.
Risultati
Ogni test THUNDER TR-FRET è stato validato farmacologicamente trattando cellule aderenti (HeLa o A431) o in sospensione (U266B1) con attivatori o inibitori specifici della via JAK/STAT e quindi misurando i livelli di specifiche ST fosforilate e totali, se applicabile. I saggi sono stati condotti in formato 384 pozzetti utilizzando il protocollo di trasferimento a due piastre e condizioni di analisi pre-ottimizzate. La Figura 3, la Figura 4,
Discussione
Rispetto ai metodi convenzionali per l'analisi delle fosfoproteine come il western blotting e i metodi basati su ELISA, il flusso di lavoro per un test cellulare THUNDER TR-FRET è semplice e veloce, utilizza un campione a basso volume (15 μL), è progettato per HTS in un formato a 384 pozzetti ed è altamente suscettibile all'automazione. Il protocollo di analisi è flessibile e può essere facilmente adattato ad applicazioni a throughput medio e alto. I saggi possono essere eseguiti utilizzando un protocollo di trasfe...
Divulgazioni
Interessi in competizione: Jaime Padros, Mireille Caron e Geneviève Chatel sono dipendenti di BioAuxilium Research, che produce i kit di analisi THUNDER TR-FRET utilizzati in questo studio. Inoltre, Jaime Padros e Mireille Caron sono azionisti di BioAuxilium Research. Ciò non altera l'adesione degli autori a tutte le politiche di JoVE sulla condivisione di dati e materiali.
Riconoscimenti
Nessuno.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well microplate, clear, flat bottom, polystyrene, tissue culture-treated, sterile | Corning | 3595 | This is the plate for culturing cells when using the two-plate assay protocol. Other cell culture 96-well plates can be used |
| 384-well microplate, white, low-volume | PerkinElmer | 6007290 | This is the plate for TR-FRET detection when using the two- or one-plate assay protocols. Other low-volume, white 384-well plates can be used |
| A431 cell line | ATCC | CRL-1555 | |
| Adhesive microplate seal | PerkinElmer | 6050185 | |
| DMSO | Fisher | D159-4 | |
| Dulbecco’s modified Eagle medium (DMEM) | Wisent | 319-005-CL | THUNDER TR-FRET is compatible with culture medium containing phenol red |
| EnVision Xcite Multilabel plate reader | PerkinElmer | 2104-0020A | The assays can be performed on a variety of plate readers equipped with the TR-FRET option |
| Erlotinib hydrochloride | Sigma | CDS022564 | |
| Falcon tissue culture treated flasks | Fisher | 13-680-65 | |
| Fetal bovine serum (FBS) | Wisent | 098-150 | |
| HeLa cell line | ATCC | CCL-2 | |
| JAK Inhibitor 1 | Cayman Chemical | 15146 | |
| Orbital plate shaker | Many options available | Not applicable | |
| Recombinant human EGF | PeproTech | AF-100-15 | |
| Recombinant human IFNα2b | ProSpec | CYT-460 | |
| Recombinant human IL-4 | R&D Systems | 204-IL | |
| Roswell Park Memorial Institute 1640 medium (RPMI) | Wisent | 350-007-CL | THUNDER TR-FRET is compatible with culture medium containing phenol red |
| THUNDER Phospho-STAT1 (Y701) + Total STAT1 TR-FRET Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT1PT-500 | |
| THUNDER Phospho-STAT3 (Y705) + Total STAT3 Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT3PT-500 | |
| THUNDER Phospho-STAT4 (Y693) TR-FRET Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT4P-500 | |
| THUNDER Phospho-STAT5 (Y694/Y699) + Total STAT5 TR-FRET Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT5PT-500 | |
| THUNDER Phospho-STAT6 (Y641) + Total STAT6 TR-FRET Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT6PT-500 | |
| Trypsin/EDTA 0.05% | Wisent | 325-542-CL | |
| U266B1 cell line | ATCC | TIB-196 | |
| Ultrapure water | NA | NA | Use Milli-Q grade water (18 MΩ.cm) to dilute Lysis Buffer and Detection Buffer |
Riferimenti
- Villarino, A., Kanno, Y., O'Shea, J. Mechanisms and consequences of Jak-STAT signaling in the immune system. Nature Immunology. 18 (4), 374-384 (2017).
- Hammarén, H. M., Virtanen, A. T., Raivola, J., Silvennoinen, O. The regulation of JAKs in cytokine signaling and its breakdown in disease. Cytokine. 118, 48-63 (2019).
- O'Shea, J. J., et al. The JAK-STAT pathway: impact on human disease and therapeutic intervention. Annual Review of Medicine. 66, 311-328 (2015).
- Verhoeven, Y., et al. et al. potential and controversy of targeting STAT family members in cancer. Seminars in Cancer Biology. 60 (2), 41-56 (2020).
- Gilda, J. E., et al. et al. blotting inaccuracies with unverified antibodies: need for a Western blotting minimal reporting standard (WBMRS). PLoS One. 10 (8), 0135392 (2015).
- Binder, C., et al. et al. and utilization of the SureFire phospho-STAT5 assay for a cell-based screening campaign. Assay and Drug Development Technologies. 6 (1), 27-37 (2008).
- Ayoub, M. A., et al. Homogeneous time-resolved fluorescence-based assay to monitor extracellular signal-regulated kinase signaling in a high-throughput format. Frontiers in Endocrinology. 5, 94 (2014).
- Robers, M. B., Machleidt, T., Carlson, C. B., Bi, K. Cellular LanthaScreen and beta-lactamase reporter assays for high-throughput screening of JAK2 inhibitors. Assay and Drug Development Technologies. 6 (4), 519-529 (2008).
- Hwang, B., Engel, L., Goueli, S. A., Zegzouti, H. A. Homogeneous bioluminescent immunoassay to probe cellular signaling pathway regulation. Communications Biology. 3 (8), 1-12 (2020).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high-throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Osmond, R. I. W., Das, S., Crouch, M. F. Development of cell-based assays for cytokine receptor signaling, using an AlphaScreen SureFire assay format. Analytical Biochemistry. 403 (1-2), 94-101 (2010).
- Haan, C., et al. Jak1 has a dominant role over Jak3 in signal transduction through γc-containing cytokine receptors. Chemistry & Biology. 18 (3), 314-323 (2011).
- Kim, Y., Apetri, M., Luo, B., Settleman, J. E., Anderson, K. S. Differential effects of tyrosine kinase inhibitors on normal and oncogenic EGFR signaling and downstream effectors. Molecular Cancer Research. 13 (4), 765-774 (2015).
- Qian, J., et al. Comparison of two homogeneous cell-based kinase assays for JAK2 V617F: SureFire pSTAT5 and GeneBLAzer fluorescence resonance energy transfer assays. ASSAY and Drug Development Technologies. 10 (2), 212-217 (2012).
- Iversen, P. W., et al., Markossian, S., et al. HTS assay validation. Assay Guidance Manual. , (2012).
- Muelas, M. W., Ortega, F., Breitling, R., Bendtsen, C., Westerhoff, H. V. Rational cell culture optimization enhances experimental reproducibility in cancer cells. Scientific Reports. 8, 3029 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon