Method Article
Análisis espaciotemporal bidimensional automatizado de sondas MÓVILES FRET de una sola molécula
En este artículo
Resumen
Este artículo presenta un método para el análisis espaciotemporal de sondas móviles basadas en la transferencia de energía de resonancia de Förster (smFRET) de una sola molécula utilizando microscopía de fluorescencia de campo amplio. El kit de herramientas de software recientemente desarrollado permite la determinación de trazas de tiempo smFRET de sondas en movimiento, incluida la eficiencia FRET correcta y las posiciones moleculares, como funciones del tiempo.
Resumen
La transferencia de energía de resonancia de Förster de molécula única (smFRET) es una técnica versátil que informa sobre distancias en el rango de subnanométrico a nanómetro. Se ha utilizado en una amplia gama de experimentos biológicos biofísicos y moleculares, incluida la medición de fuerzas moleculares, la caracterización de la dinámica conformacional de biomoléculas, la observación de la colocalización intracelular de proteínas y la determinación de los tiempos de interacción receptor-ligando. En una configuración de microscopía de campo amplio, los experimentos generalmente se realizan utilizando sondas inmovilizadas en superficie. Aquí, se presenta un método que combina el seguimiento de una sola molécula con experimentos smFRET de excitación alterna (ALEX), lo que permite la adquisición de trazas de tiempo smFRET de sondas móviles unidas a la superficie en membranas plasmáticas o bicapas lipídicas soportadas por vidrio. Para el análisis de los datos registrados, se desarrolló una colección automatizada de software de código abierto que apoya (i) la localización de señales fluorescentes, (ii) el seguimiento de partículas individuales, (iii) la determinación de cantidades relacionadas con FRET, incluidos los factores de corrección, (iv) la verificación estricta de los rastros de smFRET y (v) la presentación intuitiva de los resultados. Los datos generados se pueden utilizar convenientemente como entrada para una mayor exploración a través de un software especializado, por ejemplo, para la evaluación del comportamiento de difusión de las sondas o la investigación de las transiciones FRET.
Introducción
La transferencia de energía de resonancia de Förster (FRET) ha sido un importante impulsor en la investigación biológica molecular y biofísica, ya que permite la investigación de procesos a resolución subnanométrica. Como la eficiencia de la transferencia de energía entre los fluoróforos donantes y aceptores depende en gran medida de la distancia entre colorantes en el rango subnanométrico a nanométrico, se ha utilizado eficazmente como regla espectroscópica para explorar la conformación estática y dinámica de biomoléculas1,2,3,4. Además, el fenómeno FRET ha sido ampliamente utilizado para estudios de colocalización de proteínas asociadas a membrana e intracelulares a granel5,6. En las últimas dos décadas, el método se adaptó para el monitoreo de eventos smFRET7, lo que ayudó a aumentar sustancialmente la resolución temporal y espacial y resolvió incluso subpoblaciones raras en muestras heterogéneas. Equipados con estas técnicas, se obtuvieron conocimientos únicos sobre la dinámica de la maquinaria molecular, como la tasa de procesamiento de transcripciones de la ARN polimerasa II8, la velocidad de replicación de las ADN polimerasas9,10, la tasa de translocación de nucleosomas11, la tasa de empalme y estancamiento de transcripciones de los espliceosomas ensamblados12, la actividad de las subpoblaciones ribosómicas13 y la velocidad de marcha de los motores de kinesina14 , por nombrar algunos. Se han cuantificado las duraciones de la interacción receptor-ligando15 y las fuerzas moleculares16.
Los estudios smFRET basados en la intensidad generalmente se basan en la emisión sensibilizada para medir la eficiencia de FRET: un divisor de haz en la ruta de emisión separa espacialmente la luz que se origina en los fluoróforos donantes y aceptores sobre la excitación del donante, lo que permite la cuantificación de las intensidades de fluorescencia individuales. La eficiencia se puede calcular posteriormente como la fracción de fotones emitidos por el aceptor con respecto al recuento total de fotones17. Además, la excitación del aceptor después de la excitación del donante (ALEX) permite medir la estequiometría de los eventos FRET, ayudando en la discriminación entre las verdaderas señales FRET bajas de las señales que surgen, por ejemplo, de sondas con un fluoróforo aceptor fotoblanqueado18.
Los experimentos FRET de una sola molécula se llevan a cabo comúnmente de una de dos maneras. Primero, una pequeña región en el volumen de la muestra se ilumina usando un microscopio confocal. Las moléculas de sonda individual en solución se excitan cuando se difunden dentro del volumen focal. Con esta técnica, se pueden utilizar detectores rápidos de conteo de fotones, lo que permite una resolución de tiempo de menos de microsegundos. En segundo lugar, las sondas se inmovilizan específicamente en superficies y se monitorean a través de microscopía de campo amplio, a menudo utilizando la configuración de reflexión interna total (TIR) para minimizar la fluorescencia de fondo. La inmovilización de la sonda permite tiempos de grabación mucho más largos que el uso del primer enfoque. Además, el campo de visión más amplio permite el monitoreo de múltiples sondas en paralelo. La necesidad de una cámara hace que este método sea lento en comparación con el descrito anteriormente. La resolución de tiempo se limita al rango de milisegundos a segundo.
Si se requieren trazas de tiempo largo, por ejemplo, para estudiar procesos dinámicos en una escala de tiempo de milisegundos a segundos, el primer método no es aplicable, ya que las ráfagas de fluorescencia suelen ser demasiado cortas. El segundo enfoque falla cuando la inmovilización no es factible, por ejemplo, en experimentos de células vivas con sondas que se difunden dentro de la membrana celular. Además, se ha observado que los sistemas modelo biológicos pueden variar drásticamente su respuesta en función de la movilidad de la superficie contactada16.
Si bien en el pasado se han realizado experimentos combinados de seguimiento smFRET y de una sola partícula que registran sondas FRET móviles19, no existe un software disponible públicamente para la evaluación de los datos. Esto impulsó el desarrollo de una nueva plataforma de análisis, que permite la determinación de múltiples propiedades de sondas fluorescentes móviles, incluida la eficiencia smFRET y la estequiometría, las posiciones con precisión de subpíxeles y las intensidades de fluorescencia como funciones del tiempo. Se establecieron métodos para filtrar los rastros resultantes mediante el examen del comportamiento de blanqueamiento paso a paso, las distancias vecinas más cercanas, las intensidades de emisión y otros rasgos para elegir exclusivamente moléculas de sonda únicas correctamente sintetizadas y funcionales. El software también admite técnicas experimentales y analíticas recientemente acordadas en un estudio multilaboratorio para producir datos smFRET confiables y cuantitativos17. En particular, la implementación se adhiere a los procedimientos validados para el cálculo de la eficiencia FRET y la estequiometría. Las intensidades de fluorescencia sobre la excitación del donante en el canal de emisión del donante IDD y el canal de emisión aceptor IDA se utilizan para el cálculo de la eficiencia aparente de FRET Eapp utilizando Eq (1).
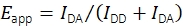 (1)
(1)
Con la ayuda de la intensidad de fluorescencia en el canal de emisión aceptor sobre la excitación aceptora IAA, la estequiometría aparente se calcula utilizando Eq (2).
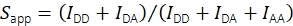 (2)
(2)
La eficiencia FRET E y la estequiometría S se pueden derivar de Eapp y Sapp considerando cuatro factores de corrección.
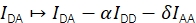
α describe la fuga de fluorescencia del donante en el canal de emisión del aceptor y se puede determinar utilizando una muestra que contiene solo fluoróforos del donante o analizando partes de las trayectorias donde el aceptor ha sido blanqueado. δ corrige la excitación directa del aceptor por la fuente de luz de excitación del donante y se puede medir utilizando una muestra con solo fluoróforos aceptores o analizando partes de trayectorias donde el donante ha sido blanqueado.
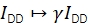 .
.
γ escala IDD para rectificar las eficiencias de detección divergentes en los canales de emisión de donantes y aceptores y las diferentes eficiencias cuánticas de los fluoróforos. El factor se puede calcular analizando el aumento de la intensidad del donante tras el blanqueo del aceptor en trayectorias con altas eficiencias FRET20 o estudiando una muestra con múltiples estados FRET discretos.
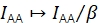
β escala IAA para corregir eficiencias dispares de excitación de donantes y aceptores. Si se determinara γ mediante análisis de blanqueo de aceptores, se podría calcular β a partir de una muestra de relación donante-aceptor conocida21. De lo contrario, la muestra FRET de varios estados también produce β.
Juntas, las correcciones permiten el cálculo de la eficiencia FRET corregida utilizando Eq (3).
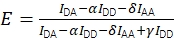 (3)
(3)
y la estequiometría corregida usando Eq (4).
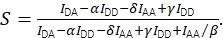 (4)
(4)
Idealmente, la estequiometría corregida para una relación donante-aceptor de 1:1 da S = 0,5. En la práctica, una relación señal-ruido reducida produce una dispersión de los valores medidos de S, lo que dificulta la discriminación de las señales solo de donante (S = 1) y las señales de solo aceptor (S = 0). Las trazas de tiempo resultantes pueden utilizarse como insumo para un análisis más detallado de las trayectorias de una sola molécula para obtener información como los perfiles de fuerza espaciotemporal16, la movilidad de los eventos de molécula única22 o la cinética de transición entre diferentes estados1.
El siguiente protocolo describe los parámetros y procedimientos experimentales para los experimentos de seguimiento smFRET, así como el principio de funcionamiento detrás del análisis de datos utilizando el paquete de software recientemente desarrollado. Para la adquisición de datos experimentales, se recomienda utilizar una configuración de microscopía que cumpla con los siguientes requisitos: i) capacidad de detectar la emisión de moléculas de colorante individuales; ii) iluminación de campo amplio: en particular para experimentos con células vivas, se recomienda la configuración de reflexión interna total (TIR23,24,25); iii) separación espacial de la luz de emisión según la longitud de onda, de modo que la fluorescencia donante y aceptora se proyecte en diferentes regiones del mismo chip de cámara25 o cámaras diferentes; iv) modulación de fuentes de luz para excitación donante y aceptora con precisión de milisegundos, por ejemplo, utilizando láseres directamente modulables o modulación a través de moduladores acústico-ópticos. Esto permite la iluminación estroboscópica para minimizar el fotoblanqueo de los fluoróforos, así como la excitación alterna para determinar las estequiometrías; v) salida de un archivo por secuencia de imagen grabada en un formato que puede ser leído por el paquete PIMS Python26. En particular, se admiten archivos TIFF de varias páginas.
Protocolo
1. Requisitos previos de software
- Instale la distribución de Python miniconda27 (versión mínima requerida de Python: 3.7).
- Abra un mensaje de Anaconda en el menú Inicio de Windows, o abra un terminal y ejecute conda activate si usa Linux o macOS.
- Habilite el repositorio de paquetes conda-forge mantenido por la comunidad28 ejecutando los siguientes comandos:
conda config --add channels conda-forge
conda config --set channel_priority estricto
conda update --all - Instale los paquetes de Python necesarios ejecutando:
conda install opencv trackpy lmfit ipympl scikit-learn pyqt sdt-python jupyterlab - Familiarícese con JupyterLab, la interfaz de usuario del software de análisis (consulte la documentación del software29).
- Instale el sistema de control de versiones git , que se utilizará más adelante para descargar y actualizar el software de análisis. Si utiliza Linux, utilice el software de gestión de paquetes de la distribución para descargar y actualizar. De lo contrario, ejecute:
conda instalar git - Opcionalmente, instale el paquete de Python sidecar para mostrar los conjuntos de datos después de filtrar los pasos durante el análisis:
conda instalar sidecar
2. Medición de muestras
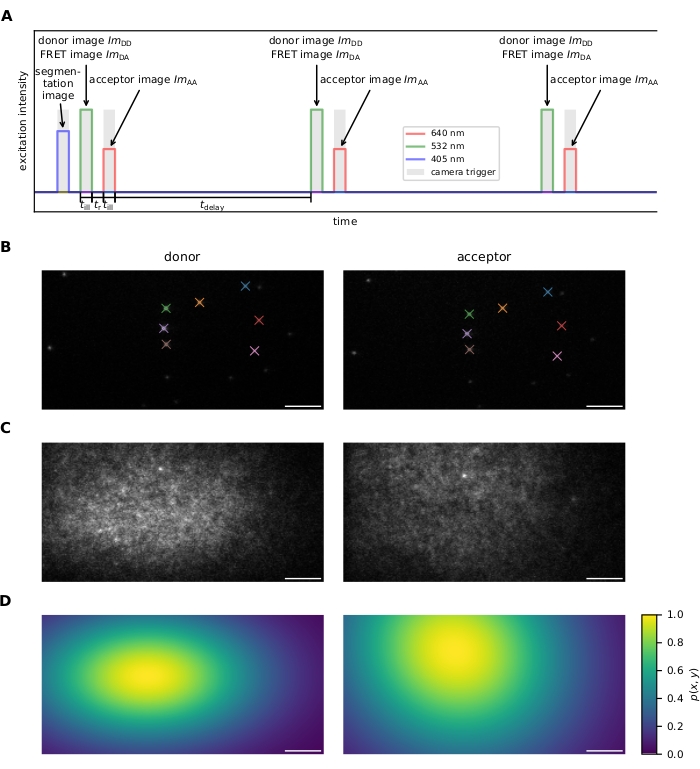
Figura 1: Adquisición de imágenes. (A) Secuencia de excitación. Después de grabar una imagen opcional de una célula cargada con tinte utilizando el láser de 405 nm, el donante y el aceptor se excitan alternativa y repetidamente para el tiempo de iluminación con láseres de 532 nm y 640 nm, respectivamente. El tiempo transcurrido entre la excitación del donante y del aceptor debe ser lo suficientemente largo como para permitir la lectura de la imagen por parte de la cámara. El tiempo de retardo tdelay se puede utilizar para ajustar la velocidad de fotogramas de adquisición y, por lo tanto, el lapso de tiempo de observación antes del fotoblanqueo. Este panel se modifica de 16. (B) Los marcadores fiduciarios se utilizan para el cálculo de las transformaciones de coordenadas entre los dos canales de emisión. Los fiduciales coincidentes se indican por color. Se deben grabar varias imágenes desplazadas para garantizar que se cubra todo el campo de visión. (C) Los perfiles láser para la corrección de campo plano se registran utilizando una muestra densamente etiquetada. El perfil del aceptador se registra y se fotoblanquea, seguido de la adquisición del perfil del donante. Se deben tomar múltiples imágenes en diferentes regiones de la muestra, promediarlas y suavizarlas para mitigar la influencia de las imperfecciones de la muestra (por ejemplo, el punto brillante en la parte central superior de la imagen). (D) Mapa de corrección de campo plano p(x,y) calculado a partir de 20 perfiles láser registrados como se describe en C. Abreviaturas: FRET = transferencia de energía de resonancia de Förster; ImDD = imagen de emisión del donante sobre la excitación del donante; ImDA = imagen de emisión del aceptor sobre la excitación del donante; ImAA = imagen de emisión del aceptor sobre la excitación del donante. Barras de escala = 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
- Cuando utilice una cámara de dispositivo de carga acoplada multiplicadora de electrones (EMCCD), permita que la ganancia EM observe señales de una sola molécula con altas relaciones señal-ruido (consulte las instrucciones del fabricante).
- Secuencia de excitación (ver Figura 1A para más detalles).
- Opcionalmente, grabe una imagen para la segmentación para restringir el análisis de datos a ciertas regiones en el campo de visión. Por ejemplo, excite las células cargadas con Fura-2 utilizando un láser de 405 nm y capture su emisión alrededor de 510 nm para evaluar solo las sondas ubicadas en las interfaces entre las células y las bicapas lipídicas (SLB) soportadas. En consecuencia, espere a que el tiempo permita la lectura de la cámara.
NOTA: En las cámaras EMCCD, tr depende del número de líneas en la región de interés elegida (ROI). Por lo tanto, elegir un ROI pequeño puede ser ventajoso porque reduce el retraso entre fotogramas y el tamaño de los datos registrados. Además, habilitar el modo de transferencia de trama permite una mayor reducción de tr. - Excitar alternativamente los fluoróforos donantes y aceptores repetidamente.
- Excita al donante durante un tiempo de iluminación (5-10 ms suele ser lo suficientemente corto como para evitar el desenfoque de movimiento) al tiempo que activa la cámara.
- Espere el tiempo para permitir la lectura de la cámara.
- Excita el aceptor mientras activas la cámara.
- Espera un tiempo tdelay.
NOTA: Esto debe ser más largo que tr para habilitar la lectura por la cámara, pero de lo contrario se puede elegir arbitrariamente. Equilibrará los requisitos de resolución temporal y longitud de traza. - Repita los pasos 2.2.2.1-2.2.2.4. Elija que el número de repeticiones sea lo suficientemente grande como para garantizar el fotoblanqueo de al menos un fluoróforo por sonda dentro del campo de visión, lo que permite el análisis de fotoblanqueo paso a paso para la discriminación de señales de una sola molécula de agregados.
NOTA: La elección de intensidades láser de excitación y till apropiadas comúnmente requiere cierta experimentación: cuanto más largos sean los tiempos de iluminación y mayores sean las intensidades del láser, mejor será la relación señal-ruido en las imágenes resultantes, pero cuanto más cortas sean las trazas de tiempo resultantes.
- Opcionalmente, grabe una imagen para la segmentación para restringir el análisis de datos a ciertas regiones en el campo de visión. Por ejemplo, excite las células cargadas con Fura-2 utilizando un láser de 405 nm y capture su emisión alrededor de 510 nm para evaluar solo las sondas ubicadas en las interfaces entre las células y las bicapas lipídicas (SLB) soportadas. En consecuencia, espere a que el tiempo permita la lectura de la cámara.
- Grabe un número suficiente de películas para cada muestra.
3. Mediciones adicionales para la determinación de los factores de corrección
- Registrar una serie de marcadores fiduciarios colocados aleatoriamente visibles en ambos canales de emisión para el registro de imágenes (es decir, encontrar la transformación que mapea las coordenadas del canal de emisión donante en el canal de emisión aceptor y viceversa). Véase la Figura 1B.
NOTA: El registro de imágenes es realizado por el software; consulte el paso 6.1.4. - Mida el perfil de intensidad de las fuentes de luz de excitación donante y aceptora para la corrección del campo plano (es decir, la corrección de la excitación no homogénea en todo el campo de visión). Con este fin, prepare una muestra con una alta densidad de sondas FRET y primero adquiera una imagen tras la excitación del aceptor, seguida del fotoblanqueo del aceptor y el posterior registro de una imagen tras la excitación del donante. Para aumentar la estabilidad, repita varias veces en diferentes regiones de la muestra. Véase la figura 1C,D. Alternativamente, registre una muestra decorada solo con la molécula donante y una segunda muestra decorada solo con los fluoróforos aceptores.
NOTA: La corrección de campo plano es realizada por el software de análisis; consulte el paso 8.1.2. - Registre una muestra de una sola molécula (como en la sección 2) de una sonda sin un fluoróforo aceptor para determinar la fuga de emisión del donante en el canal aceptor.
NOTA: La fuga del donante también se puede calcular a partir de los rastros de tiempo de las sondas reales después del blanqueo del aceptor. Si se registra un número suficiente de tales eventos, no es necesaria ninguna medición adicional. Ambas opciones son compatibles con el software de análisis; ver Información complementaria, sección 3.15. - Adquirir grabaciones de una sonda sin un fluoróforo donante para la cuantificación de la excitación del aceptor directo por la fuente de luz de excitación del donante.
NOTA: La excitación del aceptor directo también se puede derivar de los rastros de tiempo de las sondas reales después del blanqueamiento del donante. Si se registra un número suficiente de tales eventos, no es necesaria ninguna medición adicional. Ambas opciones son compatibles con el software de análisis; ver Información complementaria, sección 3.15. - Registre una muestra de una sola molécula con dos eficiencias FRET distintas para corregir las diferentes eficiencias de detección de los canales de emisión donante y aceptor y los diferentes rendimientos cuánticos de los colorantes.
NOTA: Tales muestras podrían ser, por ejemplo, uniones de Holliday1, que fluctúan entre dos conformaciones, o varillas de ADN que tienen pares FRET unidos a distancias diferentes y bien definidas. Si las sondas presentan eficiencias FRET altas y suficientemente constantes, la corrección también se puede calcular a partir de eventos de blanqueo del aceptor de los rastros de tiempo de las sondas, en cuyo caso no se necesitan mediciones adicionales. Ambas opciones son compatibles con el software de análisis; ver Información complementaria, sección 3.15.
4. Algoritmos de localización de una sola molécula
NOTA: Varios pasos de análisis requieren la localización de una sola molécula. Elija entre un algoritmo de ajuste gaussiano30 y un cálculo del centro de masa31, dependiendo de la densidad de la señal, el fondo y la relación señal-ruido.
- Para realizar el ajuste gaussiano, elija el algoritmo 3D-DAOSTORM30 a través de las respectivas interfaces de usuario.
NOTA: 3D-DAOSTORM está diseñado para distinguir señales uniformes con funciones de dispersión de puntos superpuestas. Si bien esto es generalmente una ventaja, viene con una advertencia: las señales únicas y brillantes se identifican ocasionalmente como dos adyacentes, lo que puede confundir el algoritmo de seguimiento y dar como resultado la detección de dos trayectorias cortas en lugar de una sola larga.
Establezca los siguientes parámetros (para obtener más información, consulte la documentación de la biblioteca sdt-python32, que proporciona la implementación del algoritmo).- radio: Establezca el valor de σ inicial de la función de ajuste gaussiano en píxeles en función del tamaño efectivo del píxel.
-
umbral: Establezca una amplitud mínima (es decir, el valor de píxel más brillante, corregido para el fondo local estimado) para que se ajuste un máximo de intensidad local.
NOTA: El umbral es posiblemente el parámetro más importante. Si se establece demasiado bajo, el ruido puede considerarse una señal de fluorescencia, y las señales brillantes pueden equiparse con dos gaussianos. Si se establece demasiado alto, las señales de atenuación no son adecuadas. -
modelo: Establecido en 2d para adaptarse a gaussianos circulares.
NOTA: Los otros modelos no son aplicables a los datos smFRET. -
encontrar filtro: Aplique un filtro antes de encontrar máximos locales para reducir el ruido, lo cual es útil en situaciones de baja relación señal-ruido. Esto puede ser i) identidad: sin filtro; ii) Crocker-Grier: filtro de paso de banda del algoritmo crocker-Grier31,33; o iii) Gaussiano: un desenfoque gaussiano con σ establecido por el parámetro sigma.
NOTA: Para Crocker-Grier, el parámetro feat. size debe ser aproximadamente el radio de una función de dispersión de puntos en píxeles.
NOTA: La conexión se realiza utilizando datos sin procesar sin filtrar. -
distancia mínima: Ajuste dos señales prospectivas separadas por menos de píxeles de distancia mínima por un solo gaussiano.
NOTA: Esto puede ayudar en el escenario antes mencionado donde una señal brillante se detecta erróneamente como dos señales adyacentes. - rango de tamaño: Seleccione el σ mínimo y máximo de los ajustes para eliminar las detecciones de señales espurias debidas al ruido.
- Elija el algoritmo crocker-Grier a través de las respectivas interfaces de usuario para realizar el cálculo del centro de masa (un algoritmo refinado31 basado en la idea de Crocker y Grier33).
NOTA: Este algoritmo es muy robusto incluso en escenarios de baja cantidad de señal a ruido y en el tratamiento de señales con un rango de intensidades, pero no puede adaptarse a moléculas con funciones de dispersión de puntos superpuestas con precisión.- radio: Establezca el radio (en píxeles) de un disco lo suficientemente grande como para contener toda la función de dispersión de puntos.
-
trilla de señal: Establezca la amplitud mínima (píxel más brillante por encima del fondo estimado) para un máximo de intensidad local que se analizará.
NOTA: Si se establece demasiado bajo, el ruido puede considerarse una señal de fluorescencia. Si se establece demasiado alto, las señales de atenuación no son adecuadas. -
trilla de masa: Establezca la intensidad total mínima (suma de los valores de píxeles corregidos en segundo plano) de una señal que se va a analizar.
NOTA: Se aplican las mismas consideraciones que las anteriores.
5. Inicialización del software
- Descargar scripts de análisis. En un símbolo del sistema anaconda , navegue a una carpeta para guardar el análisis (mediante el comando cd ) y ejecutar
git clone https://github.com/schuetzgroup/fret-analysis.git carpeta de destino- Reemplace la carpeta de destino por un nombre descriptivo como 2021-06-14_Force-FRET-experiment.
NOTA: El software de análisis terminará en esta carpeta; asegúrese de que esta carpeta no existe de antemano. Se recomienda descargar una copia de los scripts de análisis para cada experimento. De esta manera, es posible revisar el análisis más adelante, recordar los parámetros utilizados y realizar cambios.
- Reemplace la carpeta de destino por un nombre descriptivo como 2021-06-14_Force-FRET-experiment.
- Copiar cuadernos jupyter (01. Tracking.ipynb, 02. Análisis.ipynb, 03. Plots.ipynb) en la carpeta recién creada (en adelante, la carpeta raíz). Si es la primera vez que usa el software, consígalos de la subcarpeta de blocs de notas de la carpeta raíz.
NOTA: Si ya se han analizado conjuntos de datos similares, copiar los blocs de notas de un experimento anterior puede ser una opción conveniente, ya que los parámetros pueden haber cambiado solo ligeramente. - Inicie el servidor jupyterLab ejecutando el siguiente comando en el símbolo del sistema Anaconda para abrir una ventana del navegador web que muestre JupyterLab.
laboratorio de jupyter
NOTA: El navegador es solo la interfaz, mientras que el proceso que se ejecuta en el símbolo del sistema Anaconda está haciendo el trabajo real. Como consecuencia, cerrar la ventana del navegador solo tiene un efecto mínimo; la sesión se puede restaurar accediendo a http://localhost:8888. Sin embargo, interrumpir el proceso de JupyterLab en el mensaje o cerrar el mensaje terminará el análisis, lo que provocará la pérdida de trabajo no guardado. - En la ventana del navegador JupyterLab, utilice el panel izquierdo para desplazarse a la carpeta raíz. Haga doble clic en 01. Tracking.ipynb para iniciar el primer bloc de notas. Después de iniciar, busque una nueva pestaña para que aparezca, que muestre cuadros, las llamadas celdas, de código Python.
NOTA: Todos los blocs de notas de Jupyter cuentan con comentarios que describen la funcionalidad de cada celda de código. Además, la documentación de cada llamada al método se puede mostrar colocando el cursor de texto inmediatamente antes de la apertura ( y presione Mayús + Tab. - Consulte la Figura 2 para obtener una descripción general del proceso de análisis de datos.
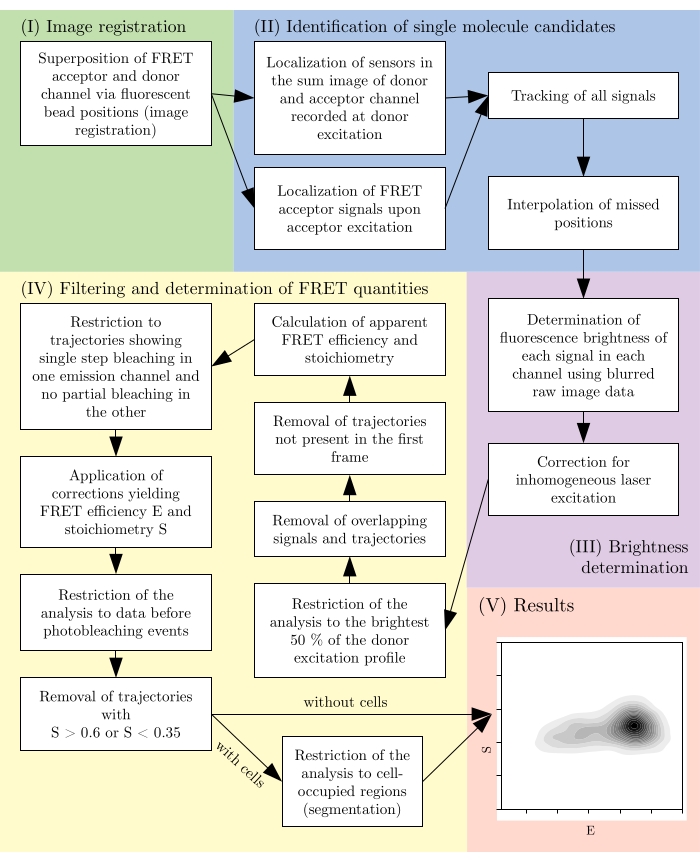
Figura 2: Descripción general de una canalización de análisis típica. Tenga en cuenta que los pasos de filtrado están sujetos a adaptación de acuerdo con el diseño experimental. Esta cifra se modifica de 16. Abreviatura: FRET = transferencia de energía de resonancia de Förster. Haga clic aquí para ver una versión más grande de esta figura.
NOTA: Los datos de muestra para probar el software se pueden descargar desde https://github.com/schuetzgroup/fret-analysis/releases/tag/example_files
6. Localización, seguimiento y análisis de intensidad de fluorescencia de moléculas individuales (01. Tracking.ipynb).
- Utilice el botón 01. Tracking.ipynb Jupyter notebook para el análisis confiable de los valores de intensidad de fluorescencia de señales de molécula única, que se adapta para la cuantificación precisa, particularmente de señales débiles que a menudo ocurren en las mediciones FRET (por ejemplo, debido a señales bajas de donantes en eventos FRET altos y viceversa).
NOTA: Con este fin, se implementa la integración directa de las intensidades de píxeles en los datos sin procesar con corrección para el fondo local. Para obtener capturas de pantalla de cada paso de análisis y la descripción de los parámetros de llamada a la función, consulte el Información complementaria.- Especifique la secuencia de iluminación para permitir la selección de fotogramas de excitación donantes y aceptores, así como fotogramas para la segmentación de imágenes a partir de secuencias de imágenes grabadas.
NOTA: Como el software permite el procesamiento de datos grabados con protocolos de iluminación arbitrarios, es necesario indicar qué fotograma de una secuencia de imagen se adquirió mientras se excita qué tipo de fluoróforo; consulte Información complementaria, sección 1.2, paso 3. Se conservan los números de fotograma de la secuencia de imágenes original. - Describir y cargar conjuntos de datos. Analice múltiples conjuntos de datos a la vez, siempre que se hayan grabado utilizando la misma configuración de iluminación. Asigne un identificador y un patrón que coincida con los respectivos nombres de archivo de secuencia de imágenes a cada conjunto de datos. Además, defina conjuntos de datos específicos para fines especiales, como registros de marcadores fiduciarios para el registro de imágenes, perfiles de luz de excitación para la corrección de campo plano y, opcionalmente, muestras solo para donantes y solo para aceptores para determinar los factores de corrección.
- Seleccione los canales de emisión en imágenes raw si ambos canales se grabaron con una sola cámara. Para ello, utilice el widget gráfico apropiado para seleccionar las regiones apropiadas para la emisión de donantes y aceptores.
- Localice marcadores fiduciarios en ambos canales de emisión y realice el registro de imágenes. Utilice la interfaz de usuario proporcionada para encontrar los parámetros adecuados para el algoritmo de localización tanto para el donante como para el aceptor de los canales de emisión. Consulte la sección 4 para obtener información sobre los algoritmos de localización admitidos.
NOTA: Los marcadores fiduciarios distribuidos aleatoriamente se pueden identificar a través de los canales de emisión por la distribución espacial de sus vecinos más cercanos (Figura 1B). Una implementación personalizada del algoritmo propuesto para la microscopía de iluminación de plano selectivo34 en la biblioteca sdt-python hace coincidir automáticamente la posición de cada marcador en el canal de emisión del donante con la posición en el canal de emisión del aceptor. Se encuentra una transformación T que asigna las coordenadas del canal de emisión donante a las coordenadas del canal de emisión aceptor a través de un ajuste lineal de mínimos cuadrados de una transformación afín a las posiciones de los marcadores35. RANSAC se utiliza para tener en cuenta los valores atípicos, como las posiciones erróneamente coincidentes del paso anterior. - Localice las sondas FRET de forma independiente en la excitación del donante y del aceptor en todos los fotogramas y combine los resultados en una tabla que contenga el número de fotograma original, las coordenadas de 2 dimensiones y un identificador que haga referencia al archivo de imagen de origen.
NOTA: Con este fin, el software proporciona interfaces de usuario para encontrar las opciones adecuadas para el algoritmo de localización.- Localice las sondas FRET sobre la excitación del donante en la suma de las imágenes obtenidas de la emisión del donante ImDD y la emisión del aceptor ImDA, que apenas dependen de la eficiencia FRET. Para obtener información sobre las opciones del algoritmo de localización, consulte la sección 4.
NOTA: Cada imagen de suma se calcula transformando ImDD utilizando la transformación T obtenida previamente del registro de la imagen y agregada pixelwise a ImDA. - Localice las sondas en la excitación del aceptor en el canal de emisión del aceptor ImAA (consulte la sección 4 para obtener más información sobre los algoritmos de localización).
- Localice las sondas FRET sobre la excitación del donante en la suma de las imágenes obtenidas de la emisión del donante ImDD y la emisión del aceptor ImDA, que apenas dependen de la eficiencia FRET. Para obtener información sobre las opciones del algoritmo de localización, consulte la sección 4.
- Realizar seguimiento y medición de la intensidad de fluorescencia.
- Especifique la secuencia de iluminación para permitir la selección de fotogramas de excitación donantes y aceptores, así como fotogramas para la segmentación de imágenes a partir de secuencias de imágenes grabadas.
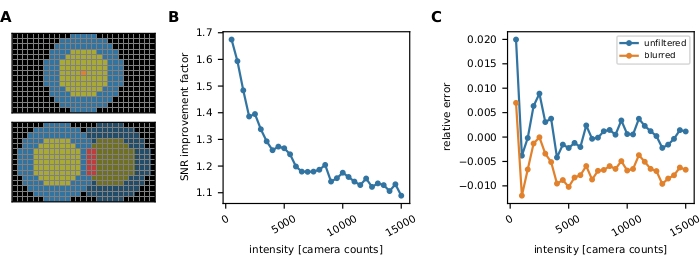
Figura 3: Medición de la intensidad de una sola molécula. (A) Para un fluoróforo ubicado en el píxel naranja, su intensidad no corregida Iuncorr se determina sumando las intensidades de todos los píxeles dentro de un disco (píxeles amarillos y naranjas) lo suficientemente grande como para cubrir todos los píxeles afectados por la señal: 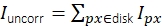 . El fondo local se calcula como la media de los píxeles en un anillo (píxeles azules) alrededor del disco:
. El fondo local se calcula como la media de los píxeles en un anillo (píxeles azules) alrededor del disco: 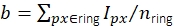 , donde nring es el número de píxeles en el anillo. La intensidad de fluorescencia I es el resultado de restar el fondo de la intensidad no corregida, I = Iuncorr - b × ndisk, donde ndisk es el número de píxeles en el disco. El radio del círculo se especifica a través del parámetro feat_radius del método de seguimiento. El ancho del anillo viene dado por el parámetro bg_frame. Si la función de dispersión de puntos de una señal se superpone con el anillo de fondo de otra (panel inferior), los píxeles afectados (rojo) se excluyen del análisis de fondo local. Si dos funciones de dispersión de puntos se superponen, las intensidades de fluorescencia no se pueden calcular de manera confiable y, por lo tanto, se descartan. (B, C) Las simulaciones muestran que la aplicación de un desenfoque gaussiano con una desviación estándar de 1 píxel mejora la relación señal-ruido hasta un factor cercano a 2 a bajas intensidades de fluorescencia (B) e introduce apenas ningún error (ligera subestimación de menos del 1%, (C))16. Además, el error relativo (es decir, (Imeas - Itruth)/Itruth, donde Itruth es la verdad fundamental e Imeas es el resultado del análisis) es constante en todo el rango de intensidad y, por lo tanto, se cancela para cantidades ratiométricas como las eficiencias FRET y las estequiometrías. Todas las tramas se basan en trabajos publicados anteriormente16. Abreviaturas: SNR = relación señal-ruido; FRET = Transferencia de energía de resonancia de Förster. Haga clic aquí para ver una versión más grande de esta figura.
, donde nring es el número de píxeles en el anillo. La intensidad de fluorescencia I es el resultado de restar el fondo de la intensidad no corregida, I = Iuncorr - b × ndisk, donde ndisk es el número de píxeles en el disco. El radio del círculo se especifica a través del parámetro feat_radius del método de seguimiento. El ancho del anillo viene dado por el parámetro bg_frame. Si la función de dispersión de puntos de una señal se superpone con el anillo de fondo de otra (panel inferior), los píxeles afectados (rojo) se excluyen del análisis de fondo local. Si dos funciones de dispersión de puntos se superponen, las intensidades de fluorescencia no se pueden calcular de manera confiable y, por lo tanto, se descartan. (B, C) Las simulaciones muestran que la aplicación de un desenfoque gaussiano con una desviación estándar de 1 píxel mejora la relación señal-ruido hasta un factor cercano a 2 a bajas intensidades de fluorescencia (B) e introduce apenas ningún error (ligera subestimación de menos del 1%, (C))16. Además, el error relativo (es decir, (Imeas - Itruth)/Itruth, donde Itruth es la verdad fundamental e Imeas es el resultado del análisis) es constante en todo el rango de intensidad y, por lo tanto, se cancela para cantidades ratiométricas como las eficiencias FRET y las estequiometrías. Todas las tramas se basan en trabajos publicados anteriormente16. Abreviaturas: SNR = relación señal-ruido; FRET = Transferencia de energía de resonancia de Förster. Haga clic aquí para ver una versión más grande de esta figura.
- Elija las opciones adecuadas para el algoritmo trackpy36 utilizado para vincular las localizaciones de sondeo FRET en trayectorias. En particular, establezca la distancia máxima de búsqueda de un fotograma a otro y el número de fotogramas consecutivos para los que una señal puede pasar desapercibida, lo que puede ocurrir debido al blanqueo o a la falta de localizaciones.
NOTA: Estos vacíos se llenan mediante interpolación entre las posiciones anteriores y subsiguientes. Estas posiciones interpoladas se marcan y solo se utilizan más tarde para leer los valores de intensidad, pero no para el análisis de difusión. Las pistas se analizan en el sistema de coordenadas del canal de emisión aceptor. Para el análisis de la intensidad de la fluorescencia (paso 6.1.6.2), las pistas se transforman adicionalmente en el sistema de coordenadas del canal de emisión donante utilizando la transformación inversa T-1 obtenida mediante el registro de imágenes (véase el paso 6.1.4). - Seleccione opciones para el algoritmo de cálculo de la intensidad de fluorescencia (consulte la Figura 3A para obtener más información). Especifique i) el radio de un disco que, cuando se centra en la posición de una señal, contiene todos los píxeles afectados por esa señal y ii) el ancho de un anillo alrededor de cada disco utilizado para determinar el fondo local.
NOTA: Para reducir el ruido en las mediciones de intensidad obtenidas, se aplica a las imágenes un desenfoque gaussiano con una desviación estándar de 1 píxel (Figura 3B,C).
- Utilice la funcionalidad del software de análisis para procesar datos de imágenes auxiliares de secuencias de imágenes.
- Extraiga imágenes adicionales grabadas para facilitar la segmentación (ver paso 2.2.1, marcado por s en la secuencia de excitación (ver Información suplementaria, sección 1.2, paso 3).
- Determine los perfiles de luz de excitación del donante y del aceptor en todo el campo de visión a partir de imágenes grabadas en una muestra densamente etiquetada (consulte el paso 3.2).
NOTA: La media píxel por píxel se calcula a partir de las imágenes para calcular los perfiles de luz. Se resta la línea base de la cámara. Las imágenes se difuminan utilizando un filtro gaussiano para reducir los efectos debidos a las impurezas de la muestra. Finalmente, las imágenes resultantes se dividen píxeles por su valor máximo para obtener las coordenadas de mapeo p(x,y) del perfil en el intervalo [0,1].
7. Visualización de trayectorias FRET (opcional)
- Utilice la aplicación inspector para mostrar pistas de una sola molécula en datos de imagen sin procesar y las intensidades de fluorescencia correspondientes y las eficiencias y estequiometrías FRET aparentes.
NOTA: Esta es una herramienta valiosa para evaluar la validez de los parámetros elegidos y aceptar o rechazar manualmente los rastros de tiempo individuales. Consulte Información complementaria para obtener una captura de pantalla e información de uso detallada.
8. Análisis y filtrado de datos de molécula única (02. Análisis.ipynb)
- Utilice el botón 02. Analysis.ipynb Jupyter cuaderno para el análisis y filtrado de los datos de una sola molécula obtenidos a través del 01. Tracking.ipynb cuaderno. Consulte los pasos a continuación para obtener una canalización de análisis típica.
NOTA: Diferentes preguntas científicas y diseños experimentales pueden requerir ajustes de la configuración. El uso de cuadernos Jupyter permite una fácil adaptación al omitir, reorganizar y modificar los pasos de análisis. Para obtener capturas de pantalla de cada paso de análisis y descripción de los parámetros de llamada a la función, consulte Información complementaria.- Realice los pasos iniciales de filtrado.
- Descarte las señales con funciones de dispersión de puntos superpuestas, ya que es difícil determinar sus intensidades de fluorescencia de manera confiable.
- En el caso de iluminación no homogénea, acepte solo señales ubicadas en regiones bien iluminadas dentro del campo de visión para garantizar una buena relación señal-ruido.
- Si estudia fret intramolecular, restrinja el análisis a aquellas trayectorias presentes desde el comienzo de la secuencia de imágenes para garantizar que todos los pasos de blanqueamiento se registren y puedan evaluarse adecuadamente más adelante durante el análisis de fotoblanqueo paso a paso.
NOTA: Al realizar experimentos con sondas FRET intermoleculares, en las que los fluoróforos donantes y aceptores no forman parte de un complejo preformado, puede que no sea factible restringir el análisis a las trayectorias inicialmente presentes.
- Ejecute la corrección de campo plano, que utiliza los perfiles de fuente de luz de excitación obtenidos en el paso 6.1.7.2 para invertir las variaciones de intensidad de fluorescencia dependientes de la posición causadas por una iluminación no homogénea.
NOTA: La intensidad de fluorescencia I(x,y) de una sonda en la posición (x,y) se corrige a través de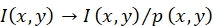 ; ver Figura 1C,D.
; ver Figura 1C,D. - Calcular la eficiencia aparente de FRET Eapp (es decir, la fracción de energía transmitida desde el fluoróforo donante al fluoróforo aceptor) y la estequiometría aparente Sapp (es decir, el número de fluoróforos donantes dividido por el número total de fluoróforos dentro de un punto limitado por difracción).
NOTA: Al trazar E vs. S para cada punto de datos, es posible distinguir las alteraciones en las eficiencias FRET medidas debido al cambio en la distancia donante-aceptor de las alteraciones debidas a cambios en la estequiometría18. Esto permite la diferenciación entre E = 0 debido a la separación del colorante de E = 0 debido a la ausencia de un aceptor activo. Los gráficos E-S se utilizan a lo largo del análisis como una herramienta para la evaluación de la calidad; ver Figura 4 como ejemplo. - Realizar análisis escalonados del fotoblanqueo para la discriminación entre sondas moleculares individuales y agregados. Elija aceptar una de las siguientes opciones.
NOTA: Con este fin, el software de análisis aplica una implementación personalizada32 del algoritmo de detección de puntos de cambio PELT37 por separado a la intensidad de fluorescencia sobre la excitación del donante (IDD + IDA) y la excitación aceptora (IAA).- Elija la opción 1, en la que el fluoróforo aceptor blanquea en un solo paso mientras que el donante no muestra blanqueamiento parcial (es decir, no hay un paso de blanqueamiento a una intensidad distinta de cero).
NOTA: Esta opción rechaza además las trayectorias en las que el donante blanquea ante el aceptor en un solo paso. La opción 1 es la opción preferida en caso de altas tasas de fotoblanqueo de aceptación. - Elija la opción 2, en la que el donante blanquea en un solo paso mientras no hay un blanqueo parcial del aceptor.
NOTA: Esta opción rechaza aún más las trayectorias en las que el donante blanquea después del aceptor en un solo paso. La opción 2 es la opción preferida en caso de altas tasas de fotoblanqueo de donantes. - Elija la opción 3, en la que cualquiera de los blanqueadores fluoróforos en un solo paso mientras que el otro no blanquea parcialmente.
NOTA: La opción 3 ofrece una mayor flexibilidad que las opciones 1 y 2 y sería la preferencia sugerida para el análisis de datos. - Elija la opción 4, en la que los fluoróforos donantes y aceptores muestran fotoblanqueo de un solo paso o ningún fotoblanqueo.
NOTA: Se prefiere la opción 4 en caso de bajas tasas de fotoblanqueo.
- Elija la opción 1, en la que el fluoróforo aceptor blanquea en un solo paso mientras que el donante no muestra blanqueamiento parcial (es decir, no hay un paso de blanqueamiento a una intensidad distinta de cero).
- Calcule los factores de corrección para la fuga de emisión del donante en el canal aceptor α, la δ de excitación del aceptor directo, las eficiencias de detección γ y las eficiencias de excitación β 17.
- Utilice los factores de corrección para calcular la eficiencia FRET E a partir de la eficiencia aparente Eapp y la estequiometría S a partir de la estequiometría aparente Sapp.
- Realice más pasos de filtrado. Seleccione solo los puntos de datos anteriores al primer evento de blanqueo en cada trayectoria. Además, acepte solo trayectorias con al menos el 75% de los puntos de datos que satisfagan 0.35 < S < 0.6 para restringir el análisis a sondas de una sola molécula (los números son ajustables).
NOTA: Los límites superior e inferior para deben elegirse de acuerdo con la propagación de la población de interés frente a las poblaciones que se excluirán del análisis (por ejemplo, poblaciones solo para donantes y solo aceptantes). Según la experiencia, 0.35 < S < 0.6 resultó ser una buena opción para muchas situaciones experimentales. - Realizar la segmentación de imágenes a través de métodos de umbral global o adaptativo35 en las imágenes auxiliares apropiadas (consulte los pasos 2.2.1 y 6.1.7) para restringir el análisis a regiones distintas dentro del campo de visión.
NOTA: Esto permite, por ejemplo, la evaluación exclusiva de sondas ubicadas en una interfaz celda-SLB o en una estructura modelada.
- Realice los pasos iniciales de filtrado.
9. Trazado de resultados y análisis posteriores (03. Plot.ipynb)
NOTA: Consulte Información complementaria para obtener capturas de pantalla del bloc de notas de Jupyter y descripción de los parámetros de llamada a la función.
- Cree gráficos E-S para verificar que las señales de estequiometría incorrecta se hayan identificado y eliminado correctamente.
- Trazar histogramas de las eficiencias de FRET para proporcionar una visión general bien establecida de las distribuciones de eficiencia de FRET. Agrupe los histogramas para una comparación conveniente de los resultados de diferentes experimentos.
- Evalúe los datos más a fondo (por ejemplo, análisis de difusión, conversión de eficiencias FRET a fuerzas en experimentos utilizando sensores de fuerza molecular o análisis de transición) dentro del cuaderno aprovechando las bibliotecas científicas de Python.
NOTA: Los datos también se pueden exportar en muchos formatos de archivo como entrada a otro software de análisis.
Resultados
Se puede extraer una variedad de información de bajo y alto nivel de las pistas smFRET dependiendo de la cuestión científica del experimento. Aquí se presentan ejemplos de pipelines de análisis con sondas analógicas y digitales: un sensor de fuerza molecular basado en péptidos16 y una sonda de ADN con conmutación estocástica de su conformación38, respectivamente. Consulte la Figura 5 para conocer el diseño y el principio de funcionamiento de estas sondas.
Después de que los algoritmos de localización y seguimiento se hayan ejecutado como se describe en el protocolo, el paquete ofrece múltiples herramientas de visualización de datos para optimizar los parámetros iniciales y los pasos de filtro posteriores: (i) visualización de eventos smFRET individuales, (ii) segmentación de imágenes opcional para analizar datos en ciertas regiones de interés, (iii) monitoreo de pasos de filtro a través de gráficos de eficiencia FRET vs. estequiometría (E-S). La visualización de los datos de una sola molécula se presenta en la Figura 6.
Finalmente, los eventos FRET filtrados están representados por una gráfica E-S y un histograma de eficiencia FRET (Figura 4). El gráfico E-S es una herramienta útil para optimizar los pasos de filtrado antes mencionados e investigar el resultado final. Los sensores FRET parcialmente blanqueados o etiquetados incompletamente pueden excluirse por su valor de estequiometría. Los parámetros de movilidad se pueden investigar trazando una trayectoria de trayectoria individual en una gráfica x-y (Figura 6) o una gráfica de desplazamiento cuadrado medio (MSD) (Figura 4). El primer método es especialmente útil para discriminar el móvil de los eventos inmovilizados, mientras que el segundo se utiliza para calcular el coeficiente de difusión.
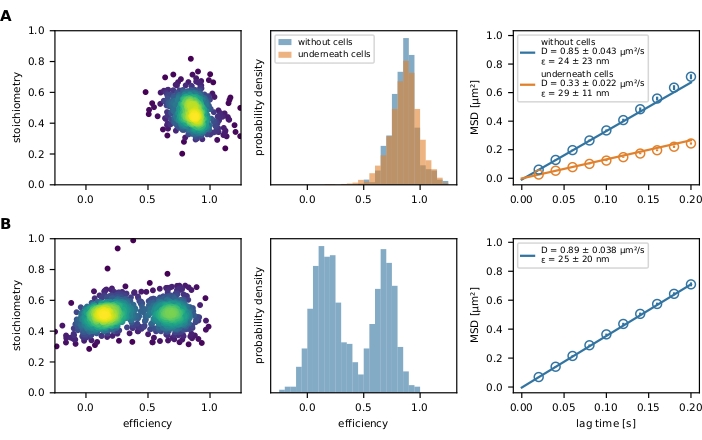
Figura 4: Resultado ejemplar. (A) La eficiencia FRET se traza frente a la estequiometría (gráfica E-S) para una población del sensor de fuerza molecular (panel izquierdo) que decora una bicapa lipídica soportada por vidrio y tensada por una célula T. Solo una nube de población es visible. El histograma respectivo de las eficiencias FRET ejemplifica la diferencia entre una población de sensores de fuerza en presencia y ausencia de células (panel medio). No se puede observar ningún cambio hacia eficiencias FRET más bajas de la población de sensores en presencia de células T, lo que indica poco o ningún estiramiento dependiente de la fuerza del módulo del sensor. La gráfica MSD de estas condiciones experimentales confirma que la población del sensor de fuerza debajo de una célula T se mueve considerablemente más lento que sus contrapartes no unidas (panel derecho). (B) El mismo análisis se realizó con el sensor de ADN de la unión Holliday que decora una bicapa lipídica fluida soportada por vidrio. La gráfica E-S muestra claramente dos poblaciones, que también son evidentes en el histograma de eficiencia FRET. La gráfica MSD indica la presencia de una población de sensores de movimiento rápido. Abreviaturas: FRET = transferencia de energía de resonancia de Förster; MSD = desplazamiento cuadrático medio. Haga clic aquí para ver una versión más grande de esta figura.
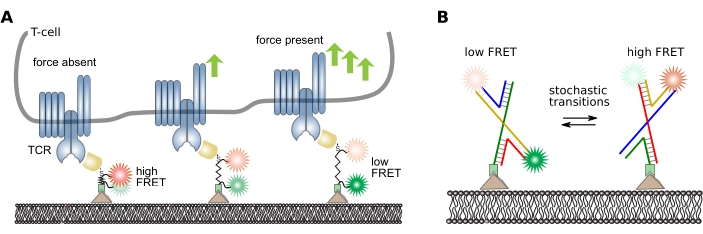
Figura 5: Diseño y principio de funcionamiento de sondas FRET intramoleculares. (A) Sensor peptídico analógico para cuantificación de fuerzas moleculares mecánicas. Los fluoróforos donante y aceptor están unidos covalentemente a cada extremo de la columna vertebral peptídica. El módulo sensor está específicamente unido a un ligando específico, que a su vez se une a un receptor de superficie residente en la célula de interés (aquí, un fragmento de anticuerpo que reconoce específicamente la cadena beta del receptor de células T). Tras la unión receptor-ligando, se ejerce fuerza, y el módulo sensor se extiende y, finalmente, retrocede después de la escisión del enlace. Este panel se modifica de 16. (B) Sensor de ADN digital para cuantificación de transiciones FRET. El sensor FRET está compuesto por cuatro hebras de ADN que forman una unión Holliday. El fluoróforo donante y aceptor están unidos covalentemente a dos hebras. Las uniones Holliday cambian con frecuencia su conformación dependiendo de las condiciones de amortiguación circundantes. La conmutación estocástica de estas conformaciones se puede monitorear cuantificando la eficiencia FRET de sondas individuales. Abreviaturas: TCR = receptor de células T; FRET = Transferencia de energía de resonancia de Förster. Haga clic aquí para ver una versión más grande de esta figura.
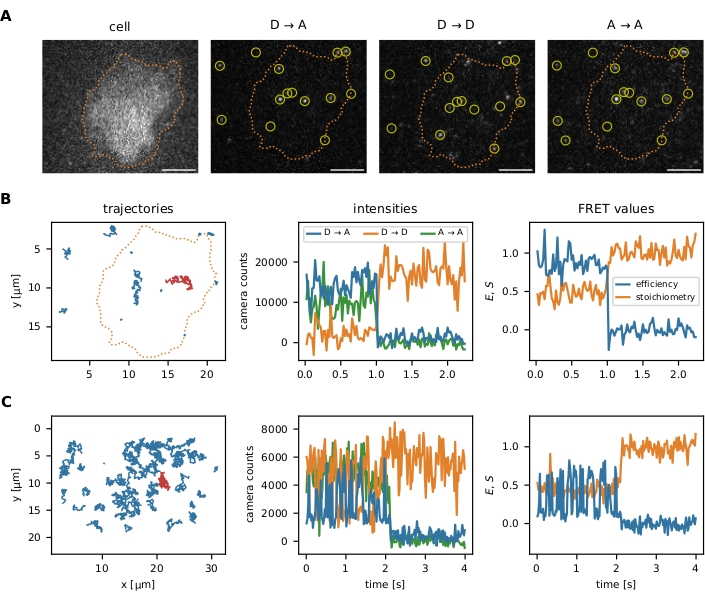
Figura 6: Ejemplos de localización y seguimiento de sondas FRET. (A) La eficiencia FRET y la estequiometría de eventos individuales se calculan cuantificando la intensidad del fluoróforo del donante sobre la excitación del donante (D → D), el fluoróforo aceptor sobre la excitación del donante (D → A) y el fluoróforo aceptor sobre la excitación del aceptor (A → A). El filtrado del vecino más cercano evita el sesgo al superponer las funciones de propagación de puntos de los emisores cercanos. La segmentación de imágenes permite al usuario elegir ciertos eventos smFRET localizados dentro de un área de interés (por ejemplo, una celda o un micropatrones). Como ejemplo de segmentación de imágenes, las células T se tiñeron con Fura-2 (que se muestra a la izquierda) y se sometieron a umbrales adaptativos para identificar los bordes de las celdas (línea punteada naranja). Barras de escala = 5 μm. (B) trayectorias smFRET utilizando el sensor de fuerza molecular. Las trayectorias individuales se pueden trazar en el plano x-y , visualizando su comportamiento de difusión y localización (panel izquierdo). Además, las intensidades de cada trayectoria se pueden trazar a lo largo del tiempo para identificar transiciones FRET o pasos de blanqueo (el panel central muestra la trayectoria roja desde el panel izquierdo). La eficiencia FRET resultante y la estequiometría se pueden visualizar de manera similar (panel derecho). (C) trayectorias smFRET utilizando el sensor de ADN de unión Holliday. HBSS + 12 mM MgCl2 se utilizó como tampón durante las mediciones. Aparte del paso de blanqueamiento del aceptor aparente cerca del final de la secuencia de estos ejemplos, se puede determinar la frecuencia de las transiciones FRET para cada sensor. Las uniones holliday cambian su conformación con una alta frecuencia, mientras que el sensor de fuerza molecular no exhibe transiciones FRET. Esta información permite ajustar las condiciones experimentales, como el retardo entre los fotogramas, para aumentar o reducir el número de transiciones observadas. Abreviaturas: FRET = transferencia de energía de resonancia de Förster; smFRET = FRET de molécula única; HBSS = Solución salina equilibrada de Hank. Haga clic aquí para ver una versión más grande de esta figura.
Información complementaria: Localización y seguimiento de moléculas individuales (01. Tracking.ipynb). Haga clic aquí para descargar este archivo.
Discusión
Este artículo detalla una tubería para los registros automatizados y el análisis cuantitativo de datos smFRET que se originan en moléculas de sonda móviles pero atadas a la superficie. Complementa los dos enfoques predominantes de los experimentos smFRET, que involucran sondas inmovilizadas en la superficie o sondas que se difunden en solución dentro y fuera de un volumen de excitación confocal17. Proporciona la eficiencia FRET correcta y las posiciones moleculares en función del tiempo. Por lo tanto, se puede utilizar como insumo para programas de análisis especializados, por ejemplo, para cuantificar la cinética de transición1, histogramas FRET39 o difusión bidimensional22.
El software se publica bajo una licencia libre y de código abierto aprobada por la Open Source Initiative que otorga al usuario el derecho perpetuo de uso, modificación y redistribución gratuitos. Github fue elegido como una plataforma de desarrollo y distribución para que sea lo más fácil posible obtener el software y participar en el proceso de desarrollo informando errores o contribuyendo con code40. Escrito en Python, el software no depende de componentes propietarios. La elección de los cuadernos Jupyter como interfaces de usuario facilita la inspección de datos en cada paso del análisis y permite adaptar y ampliar la tubería específicamente para el sistema experimental en cuestión. La biblioteca sdt-python32 sirve como base e implementa funcionalidad para evaluar datos de microscopía de fluorescencia, como localización de molécula única, análisis de difusión, análisis de intensidad de fluorescencia, registro de canales de color, análisis de colocalización y manejo de ROI.
En principio, el seguimiento de una sola partícula se puede realizar en sistemas unidimensionales, bidimensionales o tridimensionales. Aquí, la tubería de análisis de una sola molécula se adaptó al estudio de los sistemas móviles 2D. Esta elección refleja la disponibilidad de sistemas simples, como las bicapas lipídicas (SLB) con soporte planar, para presentar sondas fluorescentes móviles. Tales sistemas de bicapa lipídica se componen típicamente de dos o más partes de fosfolípidos, donde la fracción a granel determina los parámetros fisicoquímicos clave del SLB (como la fase y la viscosidad), y la fracción menor proporciona sitios de unión para biomoléculas. Estos sitios de unión pueden ser fosfolípidos biotinilados para plataformas de proteínas a base de avidina o estreptavidina o fosfolípidos conjugados de níquel-NTA para plataformas de proteínas con etiquetas de histidina41. La elección de la plataforma adecuada para vincular las proteínas al SLB depende de la cuestión científica. Los lectores pueden consultar la literatura16,38,42 para obtener ejemplos de estrategias empleadas con éxito. La densidad de las sondas en la muestra debe ser lo suficientemente baja como para evitar la superposición de funciones de dispersión de puntos; por lo general, se recomiendan menos de 0,1 moléculas por μm2. Consulte la sección de resultados representativos (en particular, la Figura 6) para ver un ejemplo que muestra una densidad de sonda adecuada. El método de análisis también es aplicable a moléculas de proteínas marcadas fluorescentemente que se difunden en la membrana plasmática de células vivas.
Un aspecto crítico de los experimentos smFRET es la producción y caracterización de las propias sondas FRET. Al elegir fluoróforos para un par FRET, su radio de Förster debe coincidir con las distancias entre tintes esperadas43. Se prefieren los tintes resistentes al fotoblanqueo, ya que producen trazas de larga duración. Sin embargo, para tasas de blanqueamiento elevadas, se puede utilizar una especie de fluoróforo para reconocer eventos multieminentes que se originan a partir de moléculas colocalizadas a través del análisis de fotoblanqueo paso a paso; consulte el paso 8.1.4 de la sección Protocolo. Los pares de fluoróforos deben estar unidos covalentemente a las moléculas de interés, formando pares FRET intra o intermoleculares.
La combinación de smFRET con otras técnicas fácilmente disponibles puede aumentar su resolución espacial más allá del límite de difracción (a través de STED44). El algoritmo de seguimiento smFRET presentado aquí amplía la aplicabilidad del enfoque a nuevos entornos experimentales y sistemas de modelos. Esto incluye estudios de (i) cambios cinéticos en la estequiometría de biomoléculas móviles, (ii) asociación dinámica de biomoléculas móviles, (iii) la velocidad de reacciones enzimáticas de reactivos de libre difusión, y (iv) la cinética de cambios conformacionales de biomoléculas móviles. Los dos primeros ejemplos requieren sistemas modelo que muestren FRET intermolecular, es decir, donante y aceptor se conjugan para separar entidades biomoleculares de interés. Estos últimos ejemplos pueden hacer uso de biosensores portadores de donante y aceptor dentro de la misma entidad molecular (FRET intramolecular).
Los sensores intramoleculares basados en FRET pueden proporcionar información sobre los cambios conformacionales intrínsecos de las biomoléculas1,2,3,4, los cambios conformacionales causados por la carga de fuerza endógena o externa (sensores de fuerza molecular16) o las concentraciones de iones en el nanoambiente, como el calcio45 y el pH46 . Dependiendo del sistema modelo y la plataforma de anclaje preferida, tales eventos smFRET se pueden rastrear en 2D o 3D: (i) el seguimiento plano de los eventos smFRET se puede emplear para la cuantificación de los tiempos de interacción receptor-ligando dentro de una membrana plasmática, la asociación de cascadas de amplificación de señal ancladas a la membrana y los cambios de estequiometría de los receptores de superficie; ii) el seguimiento del volumen de los eventos smFRET puede utilizarse para cualquier sonda FRET intra o intermolecular en células vivas o en sistemas reconstituidos in vitro.
El método de seguimiento smFRET se desarrolló principalmente teniendo en cuenta las sondas FRET intramoleculares. Estas sondas cuentan con un número fijo y bien conocido de etiquetas fluorescentes, un hecho que se explotó para rechazar datos de moléculas aglomeradas y sintetizadas incorrectamente (por ejemplo, etiquetadas incompletamente), así como de sondas donde uno de los fluoróforos ha sido fotoblanqueado. Sin embargo, ajustando los pasos de filtrado, el método también se puede aplicar a sondas FRET intermoleculares. Por ejemplo, en lugar de aceptar solo moléculas con un solo donante y un solo fluoróforo aceptor, se podrían examinar las trayectorias espaciales de los colorantes donante y aceptor y seleccionar, por ejemplo, para la codifusión de las trayectorias donante-aceptor.
Como el algoritmo 3D-DAOSTORM tiene soporte para determinar la posición de una señal a lo largo del eje óptico a través del astigmatismo debido a una lente cilíndrica en la trayectoria del haz de emisión, los experimentos 3D podrían integrarse fácilmente en la tubería de análisis. En este caso, la señal aceptora sobre la excitación aceptora serviría para determinar la estequiometría y la posición axial. El software de análisis también se puede emplear para evaluar datos de experimentos con sondas inmovilizadas utilizando su gran grado de automatización y esquemas de filtrado. De hecho, los conjuntos de datos de eficiencia smFRET de las uniones Holliday inmovilizadas en bicapas en fase gel38 se analizaron utilizando una versión temprana del software.
Divulgaciones
Los autores declaran que no hay conflictos de intereses.
Agradecimientos
Este trabajo fue apoyado por los proyectos P30214-N36, P32307-B del Fondo Austriaco para la Ciencia (FWF) y por el Fondo de Ciencia y Tecnología de Viena (WWTF) LS13-030.
Materiales
| Name | Company | Catalog Number | Comments |
| 1,2-dioleoyl-sn-glycero-3-[(N-(5-amino-1-carboxypentyl)iminodiacetic acid)succinyl] (nickel salt) (Ni-NTA-DOGS) | Avanti Polar Lipids | 790404P | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Avanti Polar Lipids | 850375P | |
| 1-palmitoyl-2-oleoyl-glycero-3-phosphocholine (POPC) | Avanti Polar Lipids | 850457P | |
| α Plan-FLUAR 100x/1.45 oil objective | Zeiss | 000000-1084-514 | |
| Axio Observer microscope body | Zeiss | ||
| Bandpass filter | Chroma Technology Corp | ET570/60m | donor emission filter |
| Bandpass filter | Chroma Technology Corp | ET675/50m | acceptor emission filter |
| conda-forge | conda-forge community | community-maintaned Python package repository for Anaconda/miniconda | |
| Coverslips 60 mm x 24 mm #1.5 | MENZEL | ||
| Dichroic mirror | Semrock Inc | FF640-FDi01-25×36 | separation of donor and acceptor emission |
| Dichroic mirror (quad band) | Semrock Inc | Di01-R405/488/532/635-25×36 | separation of excitation and emission light |
| DPBS | Sigma-Aldrich | D8537 | |
| FCS | Sigma-Aldrich | F7524 | for imaging buffer |
| fret-analysis | Schütz group at TU Wien | Python package for smFRET data analysis; version 3 | |
| Fura-2 AM | Thermo Fisher Scientific | 11524766 | |
| HBSS | Sigma-Aldrich | H8264 | for imaging buffer |
| iBeam Smart 405-S 405 nm laser | Toptica Photonics AG | ||
| iXon Ultra 897 EMCCD camera | Andor Technology Ltd | ||
| Lab-Tek chambers (8 wells) | Thermo Fisher Scientific | 177402PK | for sample preparation and imaging |
| Millenia Prime 532 nm laser | Spectra Physics | ||
| miniconda | Anaconda Inc. | Python 3 distribution. Min. version: 3.7 | |
| Monovalent streptavidin (plasmids for bacterial expression) | Addgene | 20860 & 20859 | |
| OBIS 640 nm laser | Coherent Inc | 1185055 | |
| Optosplit II | Cairn Research | ||
| Ovalbumin | Sigma-Aldrich | A5253 | for imaging buffer |
| Plasma cleaner | Harrick Plasma | PDC-002 | |
| sdt-python | Schütz group at TU Wien | Python library for data analysis; version 17 | |
| TetraSpek bead size kit | Thermo Fisher Scientific | T14792 | Randomly distributed, immobilized fiducial markers for image registration |
| USC500TH Ultrasound bath | VWR | for SUV formation |
Referencias
- McKinney, S. A., Déclais, A. -. C., Lilley, D. M. J., Ha, T. Structural dynamics of individual holliday junctions. Nature Structural Biology. 10 (2), 93-97 (2002).
- Wang, S., Vafabakhsh, R., Borschel, W. F., Ha, T., Nichols, C. G. Structural dynamics of potassium-channel gating revealed by single-molecule FRET. Nature Structural & Molecular Biology. 23 (1), 31-36 (2015).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2016).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), 235 (2018).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47 (1), 819-846 (1978).
- Wu, P. G., Brand, L. Resonance energy transfer: Methods and applications. Analytical Biochemistry. 218 (1), 1-13 (1994).
- Qiao, Y., Luo, Y., Long, N., Xing, Y., Tu, J. Single-molecular förster resonance energy transfer measurement on structures and interactions of biomolecules. Micromachines. 12 (5), 492 (2021).
- Malkusch, N., Dörfler, T., Nagy, J., Eilert, T., Michaelis, J. smFRET experiments of the RNA polymerase II transcription initiation complex. Methods. 120, 115-124 (2017).
- Lee, J. -. B., et al. Single-molecule views of MutS on mismatched DNA. DNA repair. 20, 82-93 (2014).
- Phelps, C., Israels, B., Jose, D., Marsh, M. C., von Hippel, P. H., Marcus, A. H. Using microsecond single-molecule FRET to determine the assembly pathways of T4 ssDNA binding protein onto model DNA replication forks. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), E3612-E3621 (2017).
- Deindl, S., Zhuang, X. Monitoring conformational dynamics with single-molecule fluorescence energy transfer: Applications in nucleosome remodeling. Methods in Enzymology. 513, 59-86 (2012).
- Crawford, D. J., Hoskins, A. A., Friedman, L. J., Gelles, J., Moore, M. J. Single-molecule colocalization FRET evidence that spliceosome activation precedes stable approach of 5' splice site and branch site. Proceedings of the National Academy of Sciences of the United States of America. 110 (17), 6783-6788 (2013).
- Wang, Y., Xiao, M., Li, Y. Heterogeneity of single molecule FRET signals reveals multiple active ribosome subpopulations. Proteins. 82 (1), 1-9 (2014).
- Mori, T., Vale, R. D., Tomishige, M. How kinesin waits between steps. Nature. 450 (7170), 750-754 (2007).
- Huppa, J. B., et al. TCR-peptide-MHC interactions in situ show accelerated kinetics and increased affinity. Nature. 463 (7283), 963-967 (2010).
- Göhring, J., et al. Temporal analysis of T-cell receptor-imposed forces via quantitative single molecule FRET measurements. Nature Communications. 12 (1), 2502 (2021).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669 (2018).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences of the United States of America. 101 (24), 8936-8941 (2004).
- Sakon, J. J., Weninger, K. R. Detecting the conformation of individual proteins in live cells. Nature Methods. 7 (3), 203-205 (2010).
- McCann, J. J., Choi, U. B., Zheng, L., Weninger, K., Bowen, M. E. Optimizing methods to recover absolute FRET efficiency from immobilized single molecules. Biophysical Journal. 99 (3), 961-970 (2010).
- Lee, N. K., et al. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophysical Journal. 88 (4), 2939-2953 (2005).
- Asher, W. B., et al. Single-molecule FRET imaging of GPCR dimers in living cells. Nature Methods. 18 (4), 397-405 (2021).
- Joo, C., Ha, T. . Single-molecule FRET with total internal reflection microscopy. (12), 1223-1237 (2012).
- Joo, C., Ha, T. Objective-type total internal reflection microscopy (excitation) for single-molecule FRET. Cold Spring Harbor Protocols. 11, 1189-1191 (2012).
- Joo, C., Ha, T. Objective-type total internal reflection microscopy (emission) for single-molecule FRET. Cold Spring Harbor Protocols. 11, 1192-1194 (2012).
- Allan, D. B., Caswell, T., van der Wel, C. M., Dimiduk, T. . Soft-matter/pims: PIMS v0.5. , (2020).
- Anaconda Inc. . Miniconda. , (2021).
- conda-forge community. . The conda-forge project: community-based software distribution built on the conda package format and ecosystem. , (2015).
- . . JupyterLab Contributors Notebooks - JupyterLab documentation. , (2021).
- Babcock, H., Sigal, Y. M., Zhuang, X. A high-density 3D localization algorithm for stochastic optical reconstruction microscopy. Optical Nanoscopy. 1 (6), (2012).
- Gao, Y., Kilfoil, M. L. Accurate detection and complete tracking of large populations of features in three dimensions. Optics Express. 17 (6), 4685 (2009).
- Schrangl, L. . sdt-python: Python library for fluorescence microscopy data analysis (v17.1). , (2021).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. Journal of Colloid and Interface Science. 179 (1), 298-310 (1996).
- Preibisch, S., Saalfeld, S., Schindelin, J., Tomancak, P. Software for bead-based registration of selective plane illumination microscopy data. Nature Methods. 7 (6), 418-419 (2010).
- Bradski, G. The OpenCV library. Dr. Dobb's Journal: Software Tools for the Professional Programmer. 25 (11), 120-123 (2000).
- Allan, D. B., Caswell, T., Keim, N. C., van der Wel, C. M., Verweij, R. W. Soft-matter/trackpy: Trackpy v0.5.0. Zenodo. , 4682814 (2021).
- Killick, R., Fearnhead, P., Eckley, I. A. Optimal detection of changepoints with a linear computational cost. Journal of the American Statistical Association. 107 (500), 1590-1598 (2012).
- Schrangl, L., Göhring, J., Schütz, G. J. Kinetic analysis of single molecule FRET transitions without trajectories. The Journal of Chemical Physics. 148 (12), 123328 (2018).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Schrangl, L. Single-molecule FRET analysis software (3.0). Zenodo. , (2021).
- Nye, J. A., Groves, J. T. Kinetic control of histidine-tagged protein surface density on supported lipid bilayers. Langmuir. 24 (8), 4145-4149 (2008).
- Platzer, R., et al. Unscrambling fluorophore blinking for comprehensive cluster detection via photoactivated localization microscopy. Nature Communications. 11 (1), 4993 (2020).
- Johnson, I., Spence, M. . The molecular probes handbook: A guide to fluorescent probes and labeling technologies. , (2010).
- Szalai, A. M., et al. Super-resolution imaging of energy transfer by intensity-based STED-FRET. Nano Letters. 21 (5), 2296-2303 (2021).
- Miyawaki, A., et al. Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature. 388 (6645), 882-887 (1997).
- Zhai, B., Zhai, S., Hao, R., Xu, J., Liu, Z. A FRET-based two-photon probe for in vivo tracking of pH during a traumatic brain injury process. New Journal of Chemistry. 43 (43), 17018-17022 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados