Method Article
Analyse spatio-temporelle bidimensionnelle automatisée de sondes FRET mobiles à molécule unique
Dans cet article
Résumé
Cet article présente une méthode d’analyse spatio-temporelle de sondes mobiles à transfert d’énergie de résonance de Förster (smFRET) à molécule unique utilisant la microscopie à fluorescence à grand champ. La boîte à outils logicielle nouvellement développée permet de déterminer les traces temporelles smFRET des sondes en mouvement, y compris l’efficacité FRET correcte et les positions moléculaires, en tant que fonctions du temps.
Résumé
Le transfert d’énergie par résonance Förster à molécule unique (smFRET) est une technique polyvalente qui rend compte des distances de l’ordre du nanomètre au nanomètre. Il a été utilisé dans un large éventail d’expériences biophysiques et de biologie moléculaire, y compris la mesure des forces moléculaires, la caractérisation de la dynamique conformationnelle des biomolécules, l’observation de la colocalisation intracellulaire des protéines et la détermination des temps d’interaction récepteur-ligand. Dans une configuration de microscopie à grand champ, les expériences sont généralement effectuées à l’aide de sondes immobilisées en surface. Ici, une méthode combinant le suivi d’une seule molécule avec des expériences smFRET à excitation alternée (ALEX) est présentée, permettant l’acquisition de traces temporelles smFRET de sondes liées à la surface, mais mobiles dans des membranes plasmiques ou des bicouches lipidiques supportées par du verre. Pour l’analyse des données enregistrées, une collection logicielle automatisée et open source a été développée pour soutenir (i) la localisation des signaux fluorescents, (ii) le suivi des particules uniques, (iii) la détermination des quantités liées aux FRET, y compris les facteurs de correction, (iv) la vérification rigoureuse des traces smFRET et (v) la présentation intuitive des résultats. Les données générées peuvent facilement être utilisées comme entrée pour une exploration ultérieure via un logiciel spécialisé, par exemple pour l’évaluation du comportement de diffusion des sondes ou l’étude des transitions FRET.
Introduction
Le transfert d’énergie par résonance de Förster (FRET) a été un moteur majeur de la recherche en biologie moléculaire et biophysique, car il permet d’étudier les processus à une résolution inférieure au nanomètre. Comme l’efficacité du transfert d’énergie entre les fluorophores donneurs et accepteurs dépend fortement de la distance inter-colorants dans la gamme sub-nanométrique à nanomètre, il a été efficacement utilisé comme règle spectroscopique pour explorer la conformation statique et dynamique des biomolécules1,2,3,4. De plus, le phénomène FRET a été largement utilisé pour les études de colocalisation des protéines membranaires et intracellulaires à un niveau global5,6. Au cours des deux dernières décennies, la méthode a été adaptée pour surveiller les événements smFRET7, ce qui a permis d’augmenter considérablement la résolution temporelle et spatiale et de résoudre même des sous-populations rares dans des échantillons hétérogènes. Grâce à ces techniques, des connaissances uniques ont été acquises sur la dynamique de la machinerie moléculaire, telles que le taux de traitement des transcriptions de l’ARN polymérase II8, la vitesse de réplication des ADN polymérases9,10, le taux de translocation des nucléosomes11, le taux d’épissage et de décrochage des épissures de transcription des spliceosomes assemblés12, l’activité des sous-populations ribosomiques13 et la vitesse de marche des moteurs de kinésine14 , pour n’en nommer que quelques-uns. Les durées d’interaction récepteur-ligand15 et les forces moléculaires16 ont été quantifiées.
Les études smFRET basées sur l’intensité s’appuient généralement sur une émission sensibilisée pour mesurer l’efficacité fret : un séparateur de faisceau dans le chemin d’émission sépare spatialement la lumière provenant des fluorophores donneurs et accepteurs lors de l’excitation du donneur, ce qui permet de quantifier les intensités de fluorescence individuelles. L’efficacité peut ensuite être calculée comme la fraction de photons émise par l’accepteur par rapport au nombre total de photons17. En outre, l’excitation de l’accepteur après l’excitation du donneur (ALEX) permet de mesurer la stœchiométrie des événements FRET, ce qui contribue à la discrimination entre les signaux FRET faibles réels provenant de signaux provenant, par exemple, de sondes dotées d’un fluorophore accepteur photoblanchi18.
Les expériences FRET à molécule unique sont généralement menées de deux manières. Tout d’abord, une petite région du volume de l’échantillon est éclairée à l’aide d’un microscope confocal. Les molécules à sonde unique en solution sont excitées lorsqu’elles diffusent dans le volume focal. Avec cette technique, des détecteurs à comptage rapide de photons peuvent être utilisés, permettant une résolution temporelle inférieure à la microseconde. Deuxièmement, les sondes sont spécifiquement immobilisées sur les surfaces et surveillées par microscopie à grand champ, souvent en utilisant une configuration de réflexion interne totale (TIR) pour minimiser la fluorescence de fond. L’immobilisation de la sonde permet des temps d’enregistrement beaucoup plus longs que l’utilisation de la première approche. De plus, le plus grand champ de vision permet la surveillance de plusieurs sondes en parallèle. Le besoin d’une caméra rend cette méthode lente par rapport à celle décrite ci-dessus. La résolution temporelle est limitée à la plage de milliseconde à seconde.
Si de longues traces temporelles sont nécessaires, par exemple pour étudier des processus dynamiques sur une échelle de temps de milliseconde à seconde, la première méthode n’est pas applicable, car les sursauts de fluorescence sont généralement trop courts. La deuxième approche échoue lorsque l’immobilisation n’est pas possible, par exemple dans des expériences sur des cellules vivantes mettant en vedette des sondes diffusant dans la membrane cellulaire. En outre, il a été observé que les systèmes modèles biologiques peuvent varier considérablement leur réponse en fonction de la mobilité de la surface contactée16.
Bien que des expériences combinées de suivi smFRET et d’une seule particule enregistrant des sondes FRET mobiles aient été réalisées dans le passé19, il n’existe aucun logiciel accessible au public pour l’évaluation des données. Cela a incité au développement d’une nouvelle plate-forme d’analyse, qui permet de déterminer de multiples propriétés des sondes fluorescentes mobiles, y compris l’efficacité smFRET et la stœchiométrie, les positions avec une précision inférieure au pixel et les intensités de fluorescence en fonction du temps. Des méthodes de filtrage des traces résultantes en examinant le comportement de blanchiment par étapes, les distances du plus proche voisin, les intensités d’émission et d’autres traits ont été établies pour choisir exclusivement des molécules mono-sonde correctement synthétisées et fonctionnelles. Le logiciel prend également en charge les techniques expérimentales et analytiques récemment convenues dans le cadre d’une étude multilaboratoire visant à produire des données smFRET quantitatives fiables17. En particulier, la mise en œuvre respecte les procédures validées pour le calcul de l’efficacité FRET et de la stœchiométrie. Les intensités de fluorescence lors de l’excitation du donneur dans l’IDD du canal d’émission du donneur et le canal d’émission de l’accepteur IDA sont utilisées pour le calcul de l’efficacité FRET apparente Eapp en utilisant Eq (1).
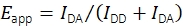 (1)
(1)
À l’aide de l’intensité de fluorescence dans le canal d’émission de l’accepteur lors de l’excitation de l’accepteur IAA, la stœchiométrie apparente est calculée à l’aide de Eq (2).
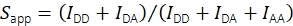 (2)
(2)
L’efficacité FRET E et la stœchiométrie S peuvent être dérivées d’Eapp et de Sapp en tenant compte de quatre facteurs de correction.
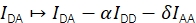
α décrit la fuite de fluorescence du donneur dans le canal d’émission de l’accepteur et peut être déterminée à l’aide d’un échantillon contenant uniquement des fluorophores donneurs ou en analysant des parties de trajectoires où l’accepteur a été blanchi. δ corrige l’excitation directe de l’accepteur par la source lumineuse d’excitation du donneur et peut être mesurée à l’aide d’un échantillon avec uniquement des fluorophores accepteurs ou en analysant des parties de trajectoires où le donneur a été blanchi.
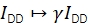 .
.
γ met à l’échelle l’IDD pour rectifier les rendements de détection divergents dans les canaux d’émission donneur et accepteur et les différentes efficacités quantiques des fluorophores. Le facteur peut être calculé en analysant l’augmentation de l’intensité du donneur lors du blanchiment de l’accepteur dans des trajectoires à haut rendement FRET20 ou en étudiant un échantillon présentant plusieurs états FRET discrets.
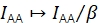
β met à l’échelle l’IAA pour corriger les efficacités disparates de l’excitation du donneur et de l’accepteur. Si γ était déterminée par une analyse de blanchiment de l’accepteur, β pouvait être calculée à partir d’un échantillon de rapport donneur-accepteur connu21. Sinon, l’échantillon FRET multi-états donne également β.
Ensemble, les corrections permettent de calculer l’efficacité FRET corrigée à l’aide de Eq (3).
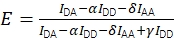 (3)
(3)
et la stœchiométrie corrigée à l’aide de Eq (4).
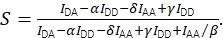 (4)
(4)
Idéalement, la stœchiométrie corrigée pour un rapport donneur-accepteur de 1:1 donne S = 0,5. En pratique, un rapport signal/bruit réduit produit une répartition des valeurs mesurées de S, ce qui entrave la discrimination entre les signaux du donneur uniquement (S = 1) et les signaux de l’accepteur uniquement (S = 0). Les traces temporelles résultantes peuvent être utilisées comme entrée pour une analyse plus détaillée des trajectoires d’une seule molécule afin d’obtenir des informations telles que les profils de force spatio-temporelle16, la mobilité des événements à molécule unique22 ou la cinétique de transition entre différents états1.
Le protocole suivant décrit les paramètres expérimentaux et les procédures pour les expériences de suivi smFRET, ainsi que le principe de fonctionnement de l’analyse des données à l’aide de la suite logicielle nouvellement développée. Pour l’acquisition de données expérimentales, il est recommandé d’utiliser une installation de microscopie répondant aux exigences suivantes: i) capacité de détecter l’émission de molécules de colorant uniques; ii) éclairage grand champ: en particulier pour les expériences sur cellules vivantes, la configuration par réflexion interne totale (TIR23,24,25) est recommandée; iii) séparation spatiale de la lumière d’émission en fonction de la longueur d’onde telle que la fluorescence du donneur et de l’accepteur soit projetée sur différentes régions de la même puce de caméra25 ou sur différentes caméras; iv) modulation des sources lumineuses pour l’excitation du donneur et de l’accepteur avec une précision de l’ordre de la milliseconde, par exemple, à l’aide de lasers directement modulables ou modulation via des modulateurs acousto-optiques. Cela permet un éclairage stroboscopique pour minimiser le photoblanchiment des fluorophores ainsi qu’une excitation alternée pour déterminer les stœchiométries; v) sortie d’un fichier par séquence d’images enregistrée dans un format qui peut être lu par le package Python PIMS26. En particulier, les fichiers TIFF multipages sont pris en charge.
Protocole
1. Prérequis logiciels
- Installez la distribution Python miniconda27 (version minimale requise de Python : 3.7).
- Ouvrez une invite Anaconda dans le menu Démarrer de Windows ou ouvrez un terminal et exécutez conda activate si vous utilisez Linux ou macOS.
- Activez le référentiel de paquets conda-forge géré par la communauté28 en exécutant les commandes suivantes :
conda config --ajouter des canaux conda-forge
conda config --set channel_priority strict
conda mise à jour --all - Installez les packages Python requis en exécutant :
conda install opencv trackpy lmfit ipympl scikit-learn pyqt sdt-python jupyterlab - Familiarisez-vous avec JupyterLab, l’interface utilisateur du logiciel d’analyse (voir la documentation du logiciel29).
- Installez le système de contrôle de version git , qui sera utilisé ultérieurement pour télécharger et mettre à jour le logiciel d’analyse. Si vous utilisez Linux, utilisez le logiciel de gestion des paquets de la distribution pour télécharger et mettre à jour. Sinon, exécutez :
conda installer git - Installez éventuellement le package Python sidecar pour afficher les jeux de données après les étapes de filtrage pendant l’analyse :
conda installer sidecar
2. Mesure des échantillons
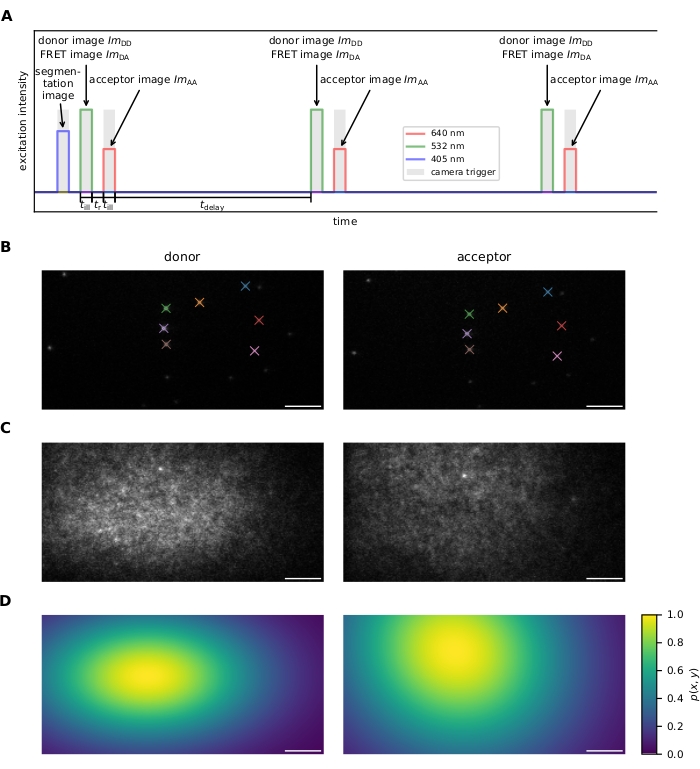
Figure 1 : Acquisition d’images. (A) Séquence d’excitation. Après avoir enregistré une image facultative d’une cellule chargée de colorant à l’aide du laser 405 nm, le donneur et l’accepteur sont excités alternativement et à plusieurs reprises pour le temps d’éclairage à l’aide de lasers 532 nm et 640 nm, respectivement. Le temps entre l’excitation du donneur et de l’accepteur doit être suffisamment long pour permettre la lecture de l’image par la caméra. Le délai de temps peut être utilisé pour ajuster la fréquence d’images d’acquisition et, par conséquent, la durée d’observation avant le photoblanchiment. Ce panneau est modifié à partir de 16. (B) Les marqueurs fiducaux sont utilisés pour le calcul des transformations de coordonnées entre les deux canaux d’émission. Les fiducials correspondants sont indiqués par couleur. Plusieurs images décalées doivent être enregistrées pour s’assurer que tout le champ de vision est couvert. (C) Les profils laser pour la correction de champ plat sont enregistrés à l’aide d’un échantillon densément étiqueté. Le profil de l’accepteur est enregistré et photoblanchi, suivi de l’acquisition du profil du donneur. Plusieurs images doivent être prises dans différentes régions de l’échantillon, moyennées et lissées pour atténuer l’influence des imperfections de l’échantillon (par exemple, le point lumineux au centre-haut de l’image). (D) Carte de correction de champ plat p(x,y) calculée à partir de 20 profils laser enregistrés comme décrit dans C. Abréviations : FRET = transfert d’énergie de résonance de Förster ; ImDD = image d’émission du donneur lors de l’excitation du donneur; ImDA = image d’émission d’accepteur lors de l’excitation du donneur; ImAA = image d’émission d’accepteur lors de l’excitation du donneur. Barres d’échelle = 5 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
- Lorsque vous utilisez une caméra EMCCD (Electron-Multiplying Charge-Coupled Device), activez le gain EM pour observer des signaux à molécule unique à des rapports signal/bruit élevés (reportez-vous aux instructions du fabricant).
- Séquence d’excitation (voir la figure 1A pour plus de détails).
- Enregistrez éventuellement une image à des fins de segmentation afin de limiter l’analyse des données à certaines régions du champ de vision. Par exemple, excitez les cellules chargées de Fura-2 à l’aide d’un laser de 405 nm et capturez leur émission autour de 510 nm pour évaluer uniquement les sondes situées dans les interfaces entre les cellules et les bicouches lipidiques (SLB) prises en charge. Par conséquent, attendez le temps tr pour permettre la lecture de la caméra.
REMARQUE: Sur les caméras EMCCD, tr dépend du nombre de lignes dans la région d’intérêt (ROI) choisie. Par conséquent, le choix d’un faible retour sur investissement peut être avantageux car il réduit le délai entre les trames et la taille des données enregistrées. De plus, l’activation du mode de transfert de trame permet une réduction supplémentaire de tr. - Exciter alternativement les fluorophores donneurs et accepteurs à plusieurs reprises.
- Excitez le donneur pour un temps d’éclairage (5-10 ms est généralement assez court pour éviter le flou de mouvement) tout en déclenchant la caméra.
- Attendez le temps tr pour autoriser la lecture de la caméra.
- Excitez l’accepteur tout en déclenchant la caméra.
- Attendez un tdelay de temps.
REMARQUE: Cela doit être plus long que tr pour permettre la lecture par la caméra, mais peut autrement être choisi arbitrairement. Il doit équilibrer les exigences en matière de résolution temporelle et de longueur de trace. - Répétez les étapes 2.2.2.1 à 2.2.2.4. Choisissez le nombre de répétitions suffisamment important pour assurer le photoblanchiment d’au moins un fluorophore par sonde dans le champ de vision, ce qui permet une analyse par photoblanchiment par étapes pour la discrimination des signaux d’une seule molécule à partir d’agrégats.
REMARQUE: Le choix des intensités laser d’excitation et d’attelage appropriées nécessite généralement une certaine expérimentation: plus les temps d’éclairage sont longs et plus les intensités laser sont élevées, meilleur est le rapport signal/bruit dans les images résultantes, mais plus les traces de temps résultantes sont courtes.
- Enregistrez éventuellement une image à des fins de segmentation afin de limiter l’analyse des données à certaines régions du champ de vision. Par exemple, excitez les cellules chargées de Fura-2 à l’aide d’un laser de 405 nm et capturez leur émission autour de 510 nm pour évaluer uniquement les sondes situées dans les interfaces entre les cellules et les bicouches lipidiques (SLB) prises en charge. Par conséquent, attendez le temps tr pour permettre la lecture de la caméra.
- Enregistrez un nombre suffisant de films pour chaque échantillon.
3. Mesures supplémentaires pour la détermination des facteurs de correction
- Enregistrez une série de marqueurs fiduciaux placés au hasard visibles dans les deux canaux d’émission pour l’enregistrement de l’image (c.-à-d. trouver la transformation qui mappe les coordonnées du canal d’émission du donneur sur le canal d’émission de l’accepteur et vice versa). Voir la figure 1B.
REMARQUE: L’enregistrement de l’image est effectué par le logiciel; voir l’étape 6.1.4. - Mesurer le profil d’intensité des sources lumineuses d’excitation du donneur et de l’accepteur pour la correction du champ plat (c.-à-d. corriger l’excitation inhomogène dans le champ de vision). À cette fin, préparez un échantillon comportant une densité élevée de sondes FRET et acquérez d’abord une image lors de l’excitation de l’accepteur, suivie d’un photoblanchiment de l’accepteur et de l’enregistrement ultérieur d’une image lors de l’excitation du donneur. Pour une stabilité accrue, répétez plusieurs fois dans différentes régions de l’échantillon. Voir la figure 1C,D. Alternativement, enregistrez un échantillon décoré avec seulement la molécule donneuse et un deuxième échantillon décoré avec seulement les fluorophores accepteurs.
REMARQUE: La correction du champ plat est effectuée par le logiciel d’analyse; voir l’étape 8.1.2. - Enregistrer un échantillon d’une seule molécule (comme à la section 2) d’une sonde sans fluorophore accepteur pour déterminer l’émission du donneur qui fuit dans le canal accepteur.
REMARQUE: Les fuites du donneur peuvent également être calculées à partir des traces temporelles réelles des sondes après le blanchiment de l’accepteur. Si un nombre suffisant de tels événements est enregistré, aucune mesure supplémentaire n’est nécessaire. Les deux options sont prises en charge par le logiciel d’analyse; voir Renseignements supplémentaires, section 3.15. - Acquérir des enregistrements d’une sonde sans fluorophore donneur pour la quantification de l’excitation directe de l’accepteur par la source lumineuse d’excitation du donneur.
REMARQUE: L’excitation directe de l’accepteur peut également être dérivée des traces temporelles réelles des sondes après le blanchiment du donneur. Si un nombre suffisant de tels événements est enregistré, aucune mesure supplémentaire n’est nécessaire. Les deux options sont prises en charge par le logiciel d’analyse; voir Renseignements supplémentaires, section 3.15. - Enregistrez un échantillon à molécule unique présentant deux efficacités FRET distinctes pour corriger les différentes efficacités de détection des canaux d’émission donneur et accepteur et les rendements quantiques différents des colorants.
REMARQUE: Ces échantillons pourraient être, par exemple, des jonctions Holliday1, qui fluctuent entre deux conformations, ou des tiges d’ADN qui ont des paires FRET attachées à des distances différentes et bien définies. Si les sondes présentent des rendements FRET élevés et suffisamment constants, la correction peut également être calculée à partir des événements de blanchiment de l’accepteur des traces temporelles des sondes, auquel cas aucune mesure supplémentaire n’est nécessaire. Les deux options sont prises en charge par le logiciel d’analyse; voir Renseignements supplémentaires, section 3.15.
4. Algorithmes de localisation d’une seule molécule
REMARQUE: Plusieurs étapes d’analyse nécessitent une localisation d’une seule molécule. Choisissez entre un algorithme d’ajustement gaussien30 et un calcul du centre de masse31, en fonction de la densité du signal, de l’arrière-plan et du rapport signal/bruit.
- Pour effectuer un ajustement gaussien, choisissez l’algorithme 3D-DAOSTORM30 via les interfaces utilisateur respectives.
REMARQUE: 3D-DAOSTORM est conçu pour distinguer les signaux pairs avec des fonctions d’étalement de points qui se chevauchent. Bien qu’il s’agisse généralement d’un avantage, il s’accompagne d’une mise en garde: des signaux uniques et lumineux sont parfois identifiés comme deux signaux adjacents, ce qui peut confondre l’algorithme de suivi et entraîner la détection de deux trajectoires courtes au lieu d’une seule longue.
Définissez les paramètres suivants (pour plus de détails, consultez la documentation de la bibliothèque sdt-python32, qui fournit l’implémentation de l’algorithme).- rayon : définissez la valeur initiale σ de la fonction d’ajustement gaussien en pixels en fonction de la taille effective des pixels.
-
seuil : Définissez une amplitude minimale (c.-à-d. la valeur de pixel la plus brillante, corrigée pour l’arrière-plan local estimé) pour qu’une intensité locale maximale soit ajustée.
REMARQUE: Le seuil est sans doute le paramètre le plus important. S’il est réglé trop bas, le bruit peut être considéré comme un signal de fluorescence, et les signaux lumineux peuvent être équipés de deux gaussiens. S’ils sont réglés trop haut, les signaux sombres ne sont pas adaptés. -
modèle: Réglez sur 2d pour s’adapter aux Gaussiens circulaires.
REMARQUE : Les autres modèles ne s’appliquent pas aux données smFRET. -
trouver un filtre : appliquez un filtre avant de trouver des maxima locaux pour réduire le bruit, ce qui est utile dans les situations de faible rapport signal/bruit. Cela peut être i) l’identité: pas de filtre; ii) Crocker-Grier: filtre passe-bande de l’algorithme Crocker-Grier31,33; ou iii) gaussien : un flou gaussien avec σ défini par le paramètre sigma.
REMARQUE: Pour Crocker-Grier, le paramètre feat. size doit être à peu près le rayon d’une fonction d’étalement de points en pixels.
REMARQUE : Le raccord est effectué à l’aide de données brutes non filtrées. -
distance min. : Ajuster deux signaux prospectifs séparés par moins de min. distance de pixels par un seul gaussien.
REMARQUE: Cela peut aider dans le scénario susmentionné où un signal lumineux est détecté à tort comme deux signaux adjacents. - Plage de taille: Sélectionnez le σ minimum et maximum des ajustements pour supprimer les détections de signaux fallacieux dus au bruit.
- Choisissez l’algorithme Crocker-Grier via les interfaces utilisateur respectives pour effectuer le calcul du centre de masse (un algorithme affiné31 basé sur l’idée de Crocker et Grier33).
REMARQUE: Cet algorithme est très robuste même dans les scénarios de faible rapport signal-bruit et dans le traitement des signaux présentant une gamme d’intensités, mais ne peut pas s’adapter avec précision aux molécules dont les fonctions d’étalement de points se chevauchent.- rayon : définissez le rayon (en pixels) d’un disque suffisamment grand pour contenir la fonction d’étalement de points entiers.
-
signal thresh.: Définissez l’amplitude minimale (pixel le plus lumineux au-dessus de l’arrière-plan estimé) pour une intensité locale maximale à analyser.
REMARQUE: S’il est réglé trop bas, le bruit peut être considéré comme un signal de fluorescence. S’ils sont réglés trop haut, les signaux sombres ne sont pas adaptés. -
mass thresh.: Définissez l’intensité totale minimale (somme des valeurs de pixels corrigées de l’arrière-plan) d’un signal à analyser.
REMARQUE: Les mêmes considérations que ci-dessus s’appliquent.
5. Initialisation du logiciel
- Télécharger les scripts d’analyse. Dans une invite Anaconda , accédez à un dossier pour enregistrer l’analyse (à l’aide de la commande cd ) et exécuter
git clone https://github.com/schuetzgroup/fret-analysis.git dossier cible- Remplacez le dossier cible par un nom descriptif tel que 2021-06-14_Force-FRET-experiment.
REMARQUE: Le logiciel d’analyse se retrouvera dans ce dossier; assurez-vous que ce dossier n’existe pas au préalable. Il est recommandé de télécharger une copie des scripts d’analyse pour chaque expérience. De cette façon, il est possible de revoir l’analyse plus tard, de rappeler les paramètres utilisés et d’apporter des modifications.
- Remplacez le dossier cible par un nom descriptif tel que 2021-06-14_Force-FRET-experiment.
- Copier les blocs-notes Jupyter (01. Tracking.ipynb, 02. Analyse.ipynb, 03. Plots.ipynb) dans le dossier nouvellement créé (ci-après dénommé le dossier racine). Si c’est la première fois que vous utilisez le logiciel, procurez-les à partir du sous-dossier notebooks du dossier racine.
REMARQUE: Si des jeux de données similaires ont déjà été analysés, la copie des blocs-notes à partir d’une expérience précédente peut être une option pratique, car les paramètres peuvent n’avoir que légèrement changé. - Démarrez le serveur JupyterLab en exécutant la commande suivante dans l’invite Anaconda pour ouvrir une fenêtre de navigateur Web affichant JupyterLab.
laboratoire jupyter
REMARQUE: Le navigateur est uniquement l’interface, tandis que le processus en cours d’exécution dans l’invite Anaconda fait le travail réel. En conséquence, la fermeture de la fenêtre du navigateur n’a qu’un effet minime; la session peut être restaurée en accédant à http://localhost:8888. Toutefois, l’interruption du processus JupyterLab dans l’invite ou la fermeture de l’invite mettra fin à l’analyse, entraînant la perte du travail non enregistré. - Dans la fenêtre du navigateur JupyterLab, utilisez le volet gauche pour accéder au dossier racine. Double-cliquez sur 01. Tracking.ipynb pour lancer le premier bloc-notes. Après le lancement, recherchez un nouvel onglet à apparaître, qui affiche des boîtes, appelées cellules, de code Python.
REMARQUE : Tous les blocs-notes Jupyter comportent des commentaires décrivant les fonctionnalités de chaque cellule de code. En outre, la documentation pour chaque appel de méthode peut être affichée en plaçant le curseur de texte immédiatement avant l’ouverture ( et appuyez sur Maj + Tab. - Voir la figure 2 pour une vue d’ensemble du processus d’analyse des données.
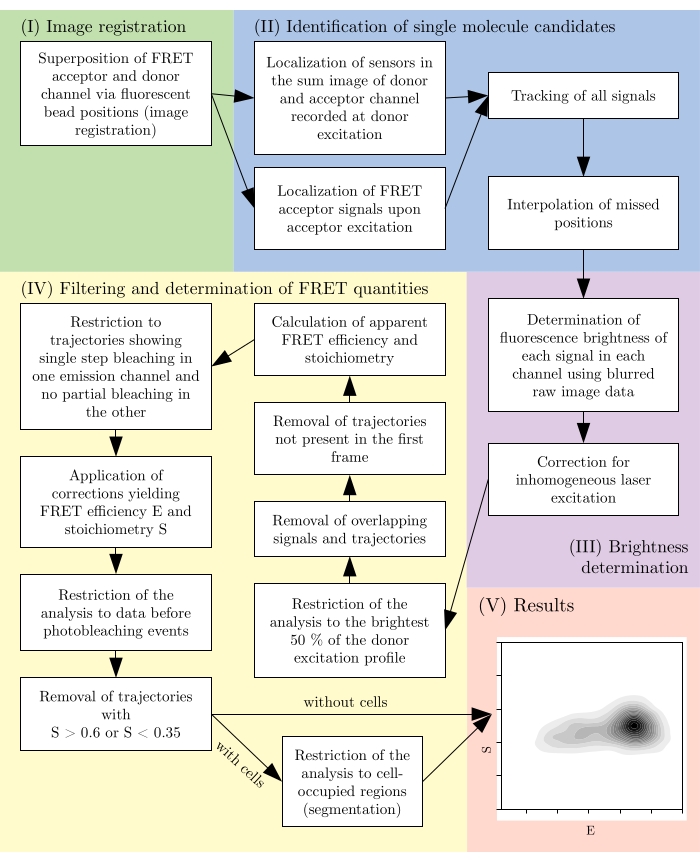
Figure 2 : Vue d’ensemble d’un pipeline d’analyse typique. Notez que les étapes de filtrage sont sujettes à adaptation selon la conception expérimentale. Ce chiffre est modifié par rapport à 16. Abréviation : FRET = Transfert d’énergie de résonance de Förster. Veuillez cliquer ici pour voir une version agrandie de cette figure.
REMARQUE: Des exemples de données pour essayer le logiciel peuvent être téléchargés à partir de https://github.com/schuetzgroup/fret-analysis/releases/tag/example_files
6. Localisation, suivi et analyse de l’intensité de fluorescence de molécules individuelles (01. Tracking.ipynb).
- Utilisez le 01. Suivi.ipynb Ordinateur portable Jupyter pour l’analyse fiable des valeurs d’intensité de fluorescence des signaux à molécule unique, qui est adapté à la quantification précise, en particulier des signaux faibles qui se produisent souvent dans les mesures FRET (par exemple, en raison de faibles signaux donneurs lors d’événements FRET élevés et vice versa).
REMARQUE: À cette fin, l’intégration directe des intensités de pixels dans les données brutes avec correction pour l’arrière-plan local est implémentée. Pour obtenir des captures d’écran de chaque étape d’analyse et une description des paramètres d’appel de fonction, reportez-vous à la section Informations supplémentaires.- Spécifiez la séquence d’éclairage pour permettre la sélection des images d’excitation du donneur et de l’accepteur, ainsi que des images pour la segmentation d’image à partir de séquences d’images enregistrées.
REMARQUE: Comme le logiciel permet le traitement de données enregistrées avec des protocoles d’éclairage arbitraires, il est nécessaire d’indiquer quelle image dans une séquence d’images a été acquise tout en excitant quel type de fluorophore; voir Informations supplémentaires, section 1.2, étape 3. Les numéros d’image de la séquence d’images d’origine sont conservés. - Décrire et charger des jeux de données. Analysez plusieurs ensembles de données à la fois, à condition qu’ils aient été enregistrés à l’aide des mêmes paramètres d’éclairage. Attribuez un identificateur et un modèle qui correspondent aux noms de fichiers de séquence d’images respectifs à chaque jeu de données. En outre, définissez des ensembles de données spécifiques à des fins spéciales, tels que des enregistrements de marqueurs fiducaux pour l’enregistrement d’images, des profils lumineux d’excitation pour la correction de champ plat et, éventuellement, des échantillons à donneur uniquement et à accepteur uniquement pour déterminer les facteurs de correction.
- Sélectionnez les canaux d’émission dans les images brutes si les deux canaux ont été enregistrés à l’aide d’une seule caméra. Pour cela, utilisez le widget graphique approprié pour sélectionner les régions appropriées pour l’émission du donneur et de l’accepteur.
- Localisez les marqueurs fiduciaires dans les deux canaux d’émission et effectuez l’enregistrement des images. Utilisez l’interface utilisateur fournie pour trouver les paramètres appropriés pour l’algorithme de localisation des canaux d’émission donneur et accepteur. Reportez-vous à la section 4 pour plus d’informations sur les algorithmes de localisation pris en charge.
REMARQUE : Les marqueurs fiducaux répartis de manière aléatoire peuvent être identifiés entre les canaux d’émission par la distribution spatiale de leurs voisins les plus proches (Figure 1B). Une implémentation personnalisée de l’algorithme proposé pour la microscopie à éclairage plan sélectif34 dans la bibliothèque sdt-python fait automatiquement correspondre la position de chaque marqueur dans le canal d’émission du donneur avec la position dans le canal d’émission de l’accepteur. Une transformation T mappant les coordonnées du canal d’émission donneur aux coordonnées du canal d’émission accepteur se trouve via un ajustement linéaire des moindres carrés d’une transformation affine aux positions des marqueurs35. RANSAC est utilisé pour tenir compte des valeurs aberrantes, telles que les positions mal appariées de l’étape précédente. - Localisez les sondes FRET indépendamment de l’excitation du donneur et de l’accepteur dans toutes les images et fusionnez les résultats dans une table contenant le numéro d’image d’origine, les coordonnées à 2 dimensions et un identificateur faisant référence au fichier image source.
REMARQUE: À cette fin, le logiciel fournit des interfaces utilisateur pour trouver les options appropriées pour l’algorithme de localisation.- Localiser les sondes FRET lors de l’excitation du donneur dans la somme des images obtenues à partir de l’émission du donneur ImDD et de l’émission de l’accepteur ImDA, qui ne dépendent guère de l’efficacité FRET. Pour plus d’informations sur les options de l’algorithme de localisation, reportez-vous à la section 4.
REMARQUE: Chaque image de somme est calculée en transformant ImDD à l’aide de la transformation T précédemment obtenue lors de l’enregistrement de l’image et ajoutée en pixels à ImDA. - Localiser les sondes lors de l’excitation de l’accepteur dans le canal d’émission de l’accepteur ImAA (voir la section 4 pour plus de détails sur les algorithmes de localisation).
- Localiser les sondes FRET lors de l’excitation du donneur dans la somme des images obtenues à partir de l’émission du donneur ImDD et de l’émission de l’accepteur ImDA, qui ne dépendent guère de l’efficacité FRET. Pour plus d’informations sur les options de l’algorithme de localisation, reportez-vous à la section 4.
- Effectuer un suivi et une mesure de l’intensité de fluorescence.
- Spécifiez la séquence d’éclairage pour permettre la sélection des images d’excitation du donneur et de l’accepteur, ainsi que des images pour la segmentation d’image à partir de séquences d’images enregistrées.
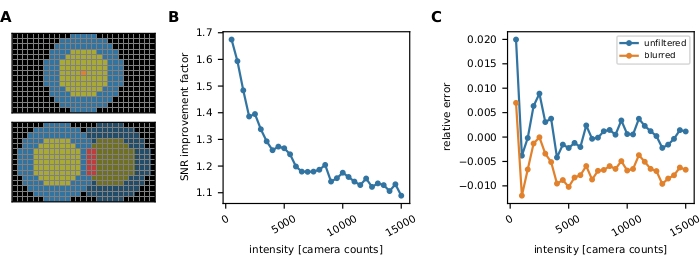
Figure 3 : Mesure de l’intensité d’une seule molécule. (A) Pour un fluorophore situé au pixel orange, son intensité non corrigée est déterminée en additionnant les intensités de tous les pixels à l’intérieur d’un disque (pixels jaunes et oranges) suffisamment grand pour couvrir tous les pixels affectés par le signal: 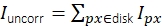 . L’arrière-plan local est calculé comme la moyenne des pixels dans un anneau (pixels bleus) autour du disque:
. L’arrière-plan local est calculé comme la moyenne des pixels dans un anneau (pixels bleus) autour du disque: 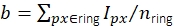 , où nring est le nombre de pixels dans l’anneau. L’intensité de fluorescence I est le résultat de la soustraction de l’arrière-plan de l’intensité non corrigée, I = Iuncorr - b × ndisk, où ndisk est le nombre de pixels dans le disque. Le rayon du cercle est spécifié via le paramètre feat_radius de la méthode de suivi. La largeur de l’anneau est donnée par le paramètre bg_frame. Si la fonction d’étalement de points d’un signal chevauche l’anneau d’arrière-plan d’un autre (panneau inférieur), les pixels affectés (rouge) sont exclus de l’analyse d’arrière-plan local. Si deux fonctions d’étalement ponctuel se chevauchent, les intensités de fluorescence ne peuvent pas être calculées de manière fiable et sont donc écartées. (B, C) Les simulations montrent que l’application d’un flou gaussien avec un écart-type de 1 pixel améliore le rapport signal/bruit jusqu’à un facteur proche de 2 à de faibles intensités de fluorescence (B) et n’introduit pratiquement aucune erreur (légère sous-estimation de moins de 1%, (C))16. De plus, l’erreur relative (c’est-à-dire (Imeas - Itruth)/Itruth, où Itruth est la vérité de terrain et Imeas est le résultat de l’analyse) est constante sur toute la plage d’intensité et s’annule donc pour les grandeurs ratiométriques telles que les efficacités FRET et les stœchiométries. Toutes les parcelles sont basées sur des travaux publiés précédemment16. Abréviations : SNR = rapport signal/bruit; FRET = Transfert d’énergie de résonance de Förster. Veuillez cliquer ici pour voir une version agrandie de cette figure.
, où nring est le nombre de pixels dans l’anneau. L’intensité de fluorescence I est le résultat de la soustraction de l’arrière-plan de l’intensité non corrigée, I = Iuncorr - b × ndisk, où ndisk est le nombre de pixels dans le disque. Le rayon du cercle est spécifié via le paramètre feat_radius de la méthode de suivi. La largeur de l’anneau est donnée par le paramètre bg_frame. Si la fonction d’étalement de points d’un signal chevauche l’anneau d’arrière-plan d’un autre (panneau inférieur), les pixels affectés (rouge) sont exclus de l’analyse d’arrière-plan local. Si deux fonctions d’étalement ponctuel se chevauchent, les intensités de fluorescence ne peuvent pas être calculées de manière fiable et sont donc écartées. (B, C) Les simulations montrent que l’application d’un flou gaussien avec un écart-type de 1 pixel améliore le rapport signal/bruit jusqu’à un facteur proche de 2 à de faibles intensités de fluorescence (B) et n’introduit pratiquement aucune erreur (légère sous-estimation de moins de 1%, (C))16. De plus, l’erreur relative (c’est-à-dire (Imeas - Itruth)/Itruth, où Itruth est la vérité de terrain et Imeas est le résultat de l’analyse) est constante sur toute la plage d’intensité et s’annule donc pour les grandeurs ratiométriques telles que les efficacités FRET et les stœchiométries. Toutes les parcelles sont basées sur des travaux publiés précédemment16. Abréviations : SNR = rapport signal/bruit; FRET = Transfert d’énergie de résonance de Förster. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Choisissez les options appropriées pour le trackpy36algorithm utilisé pour lier les localisations de sonde FRET aux trajectoires. En particulier, définissez la distance de recherche maximale d’une image à l’autre et le nombre d’images consécutives pour lesquelles un signal peut passer inaperçu, ce qui peut se produire en raison d’un blanchiment ou de localisations manquées.
NOTE: Ces lacunes sont comblées par interpolation entre les postes précédents et suivants. Ces positions interpolées sont marquées et utilisées uniquement plus tard pour lire les valeurs d’intensité, mais pas pour l’analyse de diffusion. Les pistes sont analysées dans le système de coordonnées du canal d’émission de l’accepteur. Pour l’analyse de l’intensité de fluorescence (étape 6.1.6.2), les pistes sont en outre transformées en système de coordonnées du canal d’émission du donneur à l’aide de la transformation inverse T-1 obtenue par enregistrement d’image (voir étape 6.1.4). - Sélectionnez les options de l’algorithme de calcul de l’intensité de fluorescence (voir la figure 3A pour plus de détails). Spécifiez i) le rayon d’un disque qui, lorsqu’il est centré sur la position d’un signal, contient tous les pixels affectés par ce signal et ii) la largeur d’un anneau autour de chaque disque utilisé pour déterminer l’arrière-plan local.
REMARQUE: Pour réduire le bruit dans les mesures d’intensité obtenues, un flou gaussien avec un écart type de 1 pixel est appliqué aux images (Figure 3B, C).
- Utilisez la fonctionnalité du logiciel d’analyse pour traiter les données d’image auxiliaires à partir de séquences d’images.
- Extraire des images supplémentaires enregistrées pour faciliter la segmentation (voir l’étape 2.2.1, marquée par s dans la séquence d’excitation (voir Informations supplémentaires, section 1.2, étape 3).
- Déterminer les profils lumineux d’excitation du donneur et de l’accepteur dans le champ de vision à partir d’images enregistrées sur un échantillon densément étiqueté (voir l’étape 3.2).
REMARQUE: La moyenne pixel par pixel est calculée à partir des images pour calculer les profils lumineux. La ligne de base de la caméra est soustraite. Les images sont floues à l’aide d’un filtre gaussien pour réduire les effets dus aux impuretés de l’échantillon. Enfin, les images résultantes sont divisées en pixels par leur valeur maximale pour obtenir les coordonnées de mappage du profil p(x,y) sur l’intervalle [0,1].
7. Visualisation des trajectoires FRET (facultatif)
- Utilisez l’application d’inspecteur pour afficher des traces d’une seule molécule dans des données d’image brutes et les intensités de fluorescence correspondantes et les efficacités FRET et stœchiométries apparentes.
REMARQUE: Il s’agit d’un outil précieux pour évaluer la validité des paramètres choisis et accepter ou rejeter manuellement les traces temporelles individuelles. Voir Informations supplémentaires pour une capture d’écran et des informations d’utilisation détaillées.
8. Analyse et filtrage des données unimoléculaires (02. Analyse.ipynb)
- Utilisez le 02. Analyse.ipynb Jupyter ordinateur portable pour l’analyse et le filtrage des données monomoléculaires obtenues via le 01. Suivi.ipynb carnet de notes. Consultez les étapes ci-dessous pour obtenir un pipeline d’analyse typique.
REMARQUE: Différentes questions scientifiques et conceptions expérimentales peuvent nécessiter des ajustements des paramètres. L’utilisation des blocs-notes Jupyter permet une adaptation facile en omettant, réorganisant et modifiant les étapes d’analyse. Pour des captures d’écran de chaque étape d’analyse et une description des paramètres d’appel de fonction, reportez-vous à la section Informations supplémentaires.- Effectuez les étapes de filtrage initiales.
- Écartez les signaux dont les fonctions d’étalement ponctuel se chevauchent, car il est difficile de déterminer leurs intensités de fluorescence de manière fiable.
- Dans le cas d’un éclairage non homogène, n’acceptez que les signaux situés dans des régions bien éclairées du champ de vision pour assurer un bon rapport signal/bruit.
- Si vous étudiez le FRET intramoléculaire, limitez l’analyse aux trajectoires présentes dès le début de la séquence d’images pour vous assurer que toutes les étapes de blanchiment sont enregistrées et peuvent être correctement évaluées ultérieurement lors de l’analyse de photoblanchiment par étapes.
REMARQUE: Lors d’expériences avec des sondes FRET intermoléculaires, dans lesquelles les fluorophores donneurs et accepteurs ne font pas partie d’un complexe préformé, il peut ne pas être possible de limiter l’analyse aux trajectoires initialement présentes.
- Exécuter la correction de champ plat, qui utilise les profils de source lumineuse d’excitation obtenus à l’étape 6.1.7.2 pour inverser les variations d’intensité de fluorescence dépendantes de la position causées par un éclairage inhomogène.
REMARQUE: L’intensité de fluorescence I(x,y) d’une sonde à la position (x,y) est corrigée via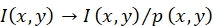 ; voir Figure 1C,D.
; voir Figure 1C,D. - Calculer l’efficacité FRET apparente Eapp (c.-à-d. la fraction d’énergie transmise du fluorophore donneur au fluorophore accepteur) et la stœchiométrie apparente Sapp (c.-à-d. le nombre de fluorophores donneurs divisé par le nombre total de fluorophores dans un endroit limité par diffraction).
REMARQUE: En traçant E vs S pour chaque point de données, il est possible de distinguer les altérations de l’efficacité FRET mesurées dues à la variation de la distance donneur-accepteur des altérations dues aux modifications de la stœchiométrie18. Cela permet la différenciation entre E = 0 en raison de la séparation du colorant de E = 0 en raison de l’absence d’un accepteur actif. Les diagrammes E-S sont utilisés tout au long de l’analyse comme outil d’évaluation de la qualité; voir la figure 4 à titre d’exemple. - Effectuer une analyse par étapes du photoblanchiment pour la discrimination entre les sondes unimoléculaires et les agrégats. Choisissez d’accepter l’une des options suivantes.
REMARQUE: À cette fin, le logiciel d’analyse applique une implémentation personnalisée32 de l’algorithme de détection de point de changement PELT37 séparément à l’intensité de fluorescence lors de l’excitation du donneur (IDD + IDA) et de l’excitation de l’accepteur (IAA).- Choisissez l’option 1, dans laquelle le fluorophore accepteur blanchit en une seule étape alors que le donneur ne montre aucun blanchiment partiel (c’est-à-dire qu’il n’y a pas d’étape de blanchiment à intensité non nulle).
REMARQUE: Cette option rejette en outre les trajectoires où le donneur blanchit avant l’accepteur en une seule étape. L’option 1 est le choix privilégié en cas de taux élevés de photoblanchiment acceptant. - Choisissez l’option 2, dans laquelle le donneur blanchit en une seule étape alors qu’il n’y a pas de blanchiment partiel de l’accepteur.
REMARQUE: Cette option rejette en outre les trajectoires où le donneur blanchit après l’accepteur en une seule étape. L’option 2 est le choix privilégié en cas de taux élevés de photoblanchiment des donneurs. - Choisissez l’option 3, dans laquelle l’un ou l’autre fluorophore blanchit en une seule étape tandis que l’autre ne blanchit pas partiellement.
REMARQUE : L’option 3 offre une plus grande souplesse que les options 1 et 2 et constituerait la préférence suggérée pour l’analyse des données. - Choisissez l’option 4, dans laquelle les fluorophores donneurs et accepteurs présentent un photoblanchiment en une seule étape ou aucun photoblanchiment.
REMARQUE: L’option 4 est préférable en cas de faibles taux de photoblanchiment.
- Choisissez l’option 1, dans laquelle le fluorophore accepteur blanchit en une seule étape alors que le donneur ne montre aucun blanchiment partiel (c’est-à-dire qu’il n’y a pas d’étape de blanchiment à intensité non nulle).
- Calculez les facteurs de correction des fuites d’émission du donneur dans le canal accepteur α, les δ d’excitation directe de l’accepteur, les rendements de détection γ et les rendements d’excitation β 17.
- Utilisez les facteurs de correction pour calculer l’efficacité FRET E à partir de l’efficacité apparente Eapp et la stœchiométrie S à partir de la stœchiométrie apparente Sapp.
- Effectuez d’autres étapes de filtrage. Sélectionnez uniquement les points de données avant le premier événement de blanchiment dans chaque trajectoire. De plus, n’acceptez que les trajectoires avec au moins 75% des points de données satisfaisant 0,35 < S < 0,6 pour limiter l’analyse aux sondes à molécule unique (les nombres sont réglables).
REMARQUE : Les limites supérieure et inférieure doivent être choisies en fonction de la répartition de la population d’intérêt par rapport aux populations à exclure de l’analyse (p. ex., les populations réservées aux donneurs et aux accepteurs seulement). D’après l’expérience, 0,35 < S < 0,6 s’est avéré être un bon choix pour de nombreuses situations expérimentales. - Effectuer une segmentation d’image via des méthodes de seuilage globales ou adaptatives35 sur les images auxiliaires appropriées (voir étapes 2.2.1 et 6.1.7) pour limiter l’analyse à des régions distinctes dans le champ de vision.
REMARQUE: Cela permet, par exemple, l’évaluation exclusive des sondes situées dans une interface cellule-SLB ou sur une structure à motifs.
- Effectuez les étapes de filtrage initiales.
9. Traçage des résultats et analyse plus approfondie (03. Plot.ipynb)
REMARQUE : Reportez-vous à la section Informations supplémentaires pour obtenir des captures d’écran du bloc-notes Jupyter et une description des paramètres d’appel de fonction.
- Créez des tracés E-S pour vérifier que les signaux de stœchiométrie incorrecte ont été correctement identifiés et supprimés.
- Tracez des histogrammes des efficacités FRET pour fournir une vue d’ensemble bien établie des distributions d’efficacité FRET. Regroupez les histogrammes pour une comparaison pratique des résultats de différentes expériences.
- Évaluez davantage les données (par exemple, l’analyse de diffusion, la conversion des efficacités FRET en forces dans des expériences utilisant des capteurs de force moléculaire ou l’analyse de transition) dans le bloc-notes en tirant parti des bibliothèques scientifiques Python.
REMARQUE: Les données peuvent également être exportées dans de nombreux formats de fichiers en entrée vers d’autres logiciels d’analyse.
Résultats
Une variété d’informations de bas et de haut niveau peuvent être extraites des pistes smFRET en fonction de la question scientifique de l’expérience. Ici, des exemples de pipelines d’analyse avec des sondes analogiques et numériques sont présentés: un capteur de force moléculaire à base de peptide16 et une sonde à ADN avec commutation stochastique de sa conformation38, respectivement. Reportez-vous à la Figure 5 pour connaître la conception et le principe de fonctionnement de ces sondes.
Une fois les algorithmes de localisation et de suivi exécutés comme décrit dans le protocole, le package offre plusieurs outils de visualisation de données pour optimiser les paramètres initiaux et les étapes de filtrage suivantes: (i) visualisation des événements smFRET individuels, (ii) segmentation d’image facultative pour analyser les données dans certaines régions d’intérêt, (iii) surveillance des étapes de filtrage via des graphiques d’efficacité FRET vs stœchiométrie (E-S). La visualisation des données d’une seule molécule est présentée à la figure 6.
Enfin, les événements FRET filtrés sont représentés par un diagramme E-S et un histogramme d’efficacité FRET (Figure 4). Le tracé E-S est un outil utile pour optimiser les étapes de filtrage susmentionnées et étudier le résultat final. Les capteurs FRET partiellement blanchis ou incomplètement étiquetés peuvent être exclus par leur valeur stœchiométrique. Les paramètres de mobilité peuvent être étudiés en traçant une trajectoire individuelle dans un diagramme x-y (figure 6) ou un diagramme de déplacement carré moyen (MSD) (figure 4). La première méthode est particulièrement utile pour distinguer les événements mobiles des événements immobilisés, tandis que la seconde est utilisée pour calculer le coefficient de diffusion.
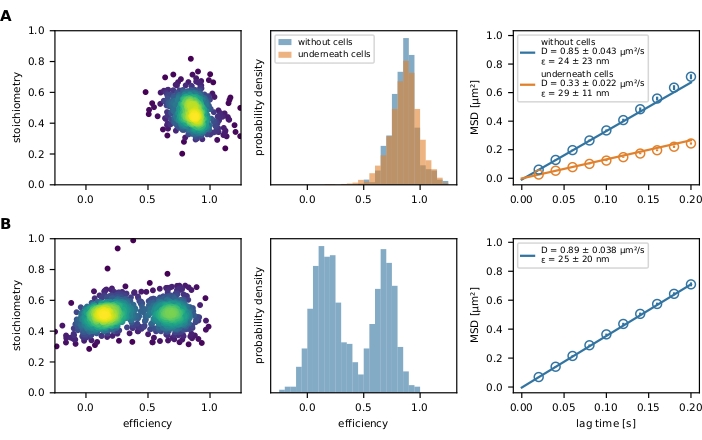
Figure 4 : Rendement exemplaire. (A) L’efficacité FRET est tracée par rapport à la stœchiométrie (diagramme E-S) pour une population du capteur de force moléculaire (panneau de gauche) décorant une bicouche lipidique supportée par du verre et tendue par une cellule T. Un seul nuage de population est visible. L’histogramme respectif des efficacités FRET illustre la différence entre une population de capteurs de force en présence et en l’absence de cellules (panneau du milieu). Aucun changement vers des rendements FRET plus faibles de la population de capteurs en présence de lymphocytes T ne peut être observé, ce qui indique peu ou pas d’étirement dépendant de la force du module de capteur. Le diagramme MSD de ces conditions expérimentales confirme que la population de capteurs de force sous une cellule T se déplace considérablement plus lentement que leurs homologues non liés (panneau de droite). (B) La même analyse a été effectuée avec un capteur d’ADN de jonction Holliday décorant une bicouche lipidique fluide supportée par du verre. Le graphique E-S montre clairement deux populations, qui sont également apparentes dans l’histogramme d’efficacité FRET. Le diagramme MSD indique la présence d’une population de capteurs en mouvement rapide. Abréviations : FRET = transfert d’énergie de résonance de Förster ; MSD = déplacement carré moyen. Veuillez cliquer ici pour voir une version agrandie de cette figure.
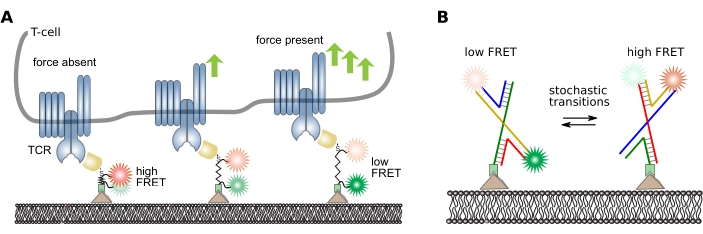
Figure 5: Conception et principe de fonctionnement des sondes FRET intramoléculaires. (A) Capteur peptidique analogique pour la quantification des forces moléculaires mécaniques. Les fluorophores donneurs et accepteurs sont attachés de manière covalente à chaque extrémité de l’épine dorsale peptidique. Le module de capteur est spécifiquement attaché à un ligand spécifique, qui à son tour lie un récepteur de surface résident dans la cellule d’intérêt (ici, un fragment d’anticorps reconnaissant spécifiquement la chaîne bêta du récepteur des cellules T). Lors de la liaison récepteur-ligand, une force est exercée et le module de capteur s’étend et finit par reculer après le clivage de la liaison. Ce panneau est modifié à partir de 16. (B) Capteur d’ADN numérique pour la quantification des transitions FRET. Le capteur FRET est composé de quatre brins d’ADN formant une jonction Holliday. Le fluorophore donneur et l’accepteur sont attachés de manière covalente à deux brins. Les jonctions Holliday changent fréquemment de conformation en fonction des conditions tampons environnantes. La commutation stochastique de ces conformations peut être surveillée en quantifiant l’efficacité FRET des sondes individuelles. Abréviations : TCR = récepteur des lymphocytes T; FRET = Transfert d’énergie de résonance de Förster. Veuillez cliquer ici pour voir une version agrandie de cette figure.
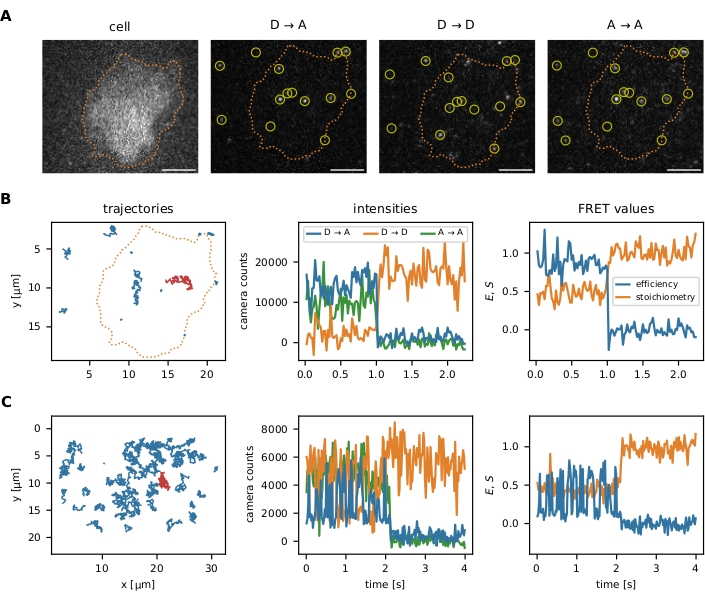
Figure 6 : Exemples de localisation et de suivi des sondes FRET. (A) L’efficacité FRET et la stœchiométrie des événements individuels sont calculées en quantifiant l’intensité du fluorophore donneur lors de l’excitation du donneur (D → D), du fluorophore accepteur lors de l’excitation du donneur (D → A) et du fluorophore accepteur lors de l’excitation de l’accepteur (A → A). Le filtrage du plus proche voisin empêche les biais en chevauchant les fonctions d’étalement ponctuel des émetteurs proches. La segmentation d’image permet à l’utilisateur de choisir certains événements smFRET localisés dans une zone d’intérêt (par exemple, une cellule ou un micromotif). À titre d’exemple de segmentation d’image, les lymphocytes T ont été colorés avec Fura-2 (affiché à gauche) et soumis à un seuil adaptatif pour identifier les bords de la cellule (ligne pointillée orange). Barres d’échelle = 5 μm. (B) trajectoires smFRET à l’aide du capteur de force moléculaire. Les trajectoires individuelles peuvent être tracées dans le plan x-y, en visualisant leur comportement de diffusion et leur localisation (panneau de gauche). De plus, les intensités de chaque trajectoire peuvent être tracées au fil du temps pour identifier les transitions FRET ou les étapes de blanchiment (le panneau du milieu montre la trajectoire rouge à partir du panneau de gauche). L’efficacité FRET et la stœchiométrie résultantes peuvent être visualisées de la même manière (panneau de droite). (C) trajectoires smFRET à l’aide du capteur d’ADN de jonction Holliday. HBSS + 12 mM MgCl2 a été utilisé comme tampon pendant les mesures. Outre l’étape apparente de blanchiment de l’accepteur vers la fin de la séquence de ces exemples, la fréquence des transitions FRET pour chaque capteur peut être déterminée. Les jonctions Holliday changent leur conformation avec une fréquence élevée, tandis que le capteur de force moléculaire ne présente pas de transitions FRET. Ces informations permettent d’ajuster les conditions expérimentales, comme le délai entre les trames, pour augmenter ou réduire le nombre de transitions observées. Abréviations : FRET = transfert d’énergie de résonance de Förster ; smFRET = FRET monomoléculaire; HBSS = solution saline équilibrée de Hank. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Informations supplémentaires : Localisation et suivi de molécules uniques (01. Tracking.ipynb). Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Cet article détaille un pipeline pour les enregistrements automatisés et l’analyse quantitative des données smFRET provenant de molécules de sonde mobiles mais attachées à la surface. Il complète les deux approches prédominantes des expériences smFRET, impliquant soit des sondes immobilisées en surface, soit des sondes diffusant en solution dans et hors d’un volume d’excitation confocale17. Il fournit l’efficacité FRET correcte et les positions moléculaires en fonction du temps. Il peut donc être utilisé comme entrée pour des programmes d’analyse spécialisés, par exemple pour quantifier la cinétique de transition1, les histogrammes FRET39 ou la diffusion bidimensionnelle22.
Le logiciel est publié sous une licence libre et open-source approuvée par l’Open Source Initiative qui accorde à l’utilisateur le droit perpétuel à l’utilisation, à la modification et à la redistribution gratuites. Github a été choisi comme plate-forme de développement et de distribution pour faciliter au maximum l’obtention du logiciel et la participation au processus de développement en signalant des bogues ou en contribuant au code40. Écrit en Python, le logiciel ne dépend pas de composants propriétaires. Le choix des ordinateurs portables Jupyter comme interfaces utilisateur facilite l’inspection des données à chaque étape d’analyse et permet d’adapter et d’étendre le pipeline spécifiquement pour le système expérimental en question. La bibliothèque sdt-python32 sert de base et implémente des fonctionnalités pour évaluer les données de microscopie à fluorescence, telles que la localisation d’une seule molécule, l’analyse de diffusion, l’analyse de l’intensité de fluorescence, l’enregistrement des canaux de couleur, l’analyse de colocalisation et la gestion du retour sur investissement.
En principe, le suivi d’une seule particule peut être effectué dans des systèmes unidimensionnels, bidimensionnels ou tridimensionnels. Ici, le pipeline d’analyse monomoléculaire a été adapté à l’étude des systèmes mobiles 2D. Ce choix reflète la disponibilité de systèmes simples, tels que les bicouches lipidiques (SLB) à support planaire, pour présenter des sondes fluorescentes mobiles. Ces systèmes de bicouches lipidiques sont généralement composés de deux fractions de phospholipides ou plus, où la fraction en vrac détermine les paramètres physico-chimiques clés du SLB (tels que la phase et la viscosité), et la fraction mineure fournit des sites de fixation pour les biomolécules. Ces sites de fixation peuvent être des phospholipides biotinylés pour les plateformes protéiques à base d’avidine ou de streptavidine ou des phospholipides conjugués nickel-NTA pour les plateformes protéiques avec des marqueurs histidine41. Le choix de la plate-forme appropriée pour relier les protéines au SLB dépend de la question scientifique. Les lecteurs peuvent se référer à la littérature16,38,42 pour des exemples de stratégies utilisées avec succès. La densité des sondes dans l’échantillon doit être suffisamment faible pour éviter le chevauchement des fonctions d’étalement ponctuel; typiquement, moins de 0,1 molécule par μm2 sont recommandés. Voir la section des résultats représentatifs (en particulier, la figure 6) pour un exemple montrant une densité de sonde appropriée. La méthode d’analyse est également applicable aux molécules de protéines marquées par fluorescence qui diffusent dans la membrane plasmique des cellules vivantes.
Un aspect critique des expériences smFRET est la production et la caractérisation des sondes FRET elles-mêmes. Lors du choix des fluorophores pour une paire FRET, leur rayon de Förster doit correspondre aux distances inter-colorants attendues43. Les colorants résistants au photoblanchiment sont préférés car ils donnent de longues traces temporelles. Cependant, pour des taux de blanchiment élevés, une espèce de fluorophore peut être utilisée pour reconnaître les événements multi-émetteurs provenant de molécules colocalisées par analyse de photoblanchiment par étapes; voir l’étape 8.1.4 dans la section protocole. Les paires de fluorophores doivent être attachées de manière covalente et spécifique au site aux molécules d’intérêt, formant des paires FRET intra- ou intermoléculaires.
La combinaison de smFRET avec d’autres techniques facilement disponibles peut augmenter sa résolution spatiale au-delà de la limite de diffraction (via STED44). L’algorithme de suivi smFRET présenté ici élargit l’applicabilité de l’approche à de nouveaux paramètres expérimentaux et systèmes modèles. Cela comprend des études sur (i) les changements cinétiques dans la stœchiométrie des biomolécules mobiles, (ii) l’association dynamique des biomolécules mobiles, (iii) le taux de réactions enzymatiques des réactifs à diffusion libre, et (iv) la cinétique des changements conformationnels des biomolécules mobiles. Les deux premiers exemples nécessitent des systèmes modèles qui montrent fret intermoléculaire, c’est-à-dire que le donneur et l’accepteur sont conjugués à des entités biomoléculaires distinctes d’intérêt. Ces derniers exemples peuvent utiliser des biocapteurs transportant donneur et accepteur au sein de la même entité moléculaire (FRET intramoléculaire).
Les capteurs intramoléculaires à base de FRET peuvent fournir un aperçu des changements conformationnels intrinsèques des biomolécules1,2,3,4, des changements conformationnels causés par une charge de force endogène ou externe (capteurs de force moléculaire16) ou des concentrations d’ions dans le nano-environnement tels que le calcium45 et le pH46 . Selon le système de modèle et la plate-forme d’ancrage préférée, ces événements smFRET peuvent être suivis en 2D ou en 3D: (i) le suivi planaire des événements smFRET peut être utilisé pour la quantification des temps d’interaction récepteur-ligand dans une membrane plasmique, l’association de cascades d’amplification du signal ancré dans la membrane et les changements stœchiométriques des récepteurs de surface; (ii) le suivi volumique des événements smFRET peut être utilisé pour toutes les sondes FRET intra- ou intermoléculaires dans des cellules vivantes ou dans des systèmes reconstitués in vitro.
La méthode de suivi smFRET a été développée principalement avec des sondes FRET intramoléculaires à l’esprit. Ces sondes comportent un nombre fixe et bien connu d’étiquettes fluorescentes, un fait qui a été exploité pour rejeter les données de molécules agglomérées et mal synthétisées (par exemple, incomplètement marquées), ainsi que de sondes où l’un des fluorophores a été photoblanchi. Cependant, en ajustant les étapes de filtrage, la méthode peut également être appliquée aux sondes FRET intermoléculaires. Par exemple, au lieu d’accepter uniquement des molécules comportant un seul donneur et un seul fluorophore accepteur, on pourrait examiner les trajectoires spatiales des colorants donneur et accepteur et sélectionner, par exemple, pour la co-diffusion des trajectoires donneur-accepteur.
Comme l’algorithme 3D-DAOSTORM prend en charge la détermination de la position d’un signal le long de l’axe optique via l’astigmatisme dû à une lentille cylindrique dans le chemin du faisceau d’émission, les expériences 3D pourraient être facilement intégrées dans le pipeline d’analyse. Dans ce cas, le signal d’accepteur lors de l’excitation de l’accepteur servirait à déterminer la stœchiométrie et la position axiale. Le logiciel d’analyse peut également être utilisé pour évaluer les données d’expériences comportant des sondes immobilisées en utilisant son grand degré d’automatisation et de filtrage. En fait, les ensembles de données d’efficacité smFRET des jonctions Holliday immobilisées sur des bicouches en phase gel38 ont été analysés à l’aide d’une première version du logiciel.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par les projets P30214-N36, P32307-B du Fonds autrichien pour la science (FWF) et par le Fonds viennois pour la science et la technologie (WWTF) LS13-030.
matériels
| Name | Company | Catalog Number | Comments |
| 1,2-dioleoyl-sn-glycero-3-[(N-(5-amino-1-carboxypentyl)iminodiacetic acid)succinyl] (nickel salt) (Ni-NTA-DOGS) | Avanti Polar Lipids | 790404P | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Avanti Polar Lipids | 850375P | |
| 1-palmitoyl-2-oleoyl-glycero-3-phosphocholine (POPC) | Avanti Polar Lipids | 850457P | |
| α Plan-FLUAR 100x/1.45 oil objective | Zeiss | 000000-1084-514 | |
| Axio Observer microscope body | Zeiss | ||
| Bandpass filter | Chroma Technology Corp | ET570/60m | donor emission filter |
| Bandpass filter | Chroma Technology Corp | ET675/50m | acceptor emission filter |
| conda-forge | conda-forge community | community-maintaned Python package repository for Anaconda/miniconda | |
| Coverslips 60 mm x 24 mm #1.5 | MENZEL | ||
| Dichroic mirror | Semrock Inc | FF640-FDi01-25×36 | separation of donor and acceptor emission |
| Dichroic mirror (quad band) | Semrock Inc | Di01-R405/488/532/635-25×36 | separation of excitation and emission light |
| DPBS | Sigma-Aldrich | D8537 | |
| FCS | Sigma-Aldrich | F7524 | for imaging buffer |
| fret-analysis | Schütz group at TU Wien | Python package for smFRET data analysis; version 3 | |
| Fura-2 AM | Thermo Fisher Scientific | 11524766 | |
| HBSS | Sigma-Aldrich | H8264 | for imaging buffer |
| iBeam Smart 405-S 405 nm laser | Toptica Photonics AG | ||
| iXon Ultra 897 EMCCD camera | Andor Technology Ltd | ||
| Lab-Tek chambers (8 wells) | Thermo Fisher Scientific | 177402PK | for sample preparation and imaging |
| Millenia Prime 532 nm laser | Spectra Physics | ||
| miniconda | Anaconda Inc. | Python 3 distribution. Min. version: 3.7 | |
| Monovalent streptavidin (plasmids for bacterial expression) | Addgene | 20860 & 20859 | |
| OBIS 640 nm laser | Coherent Inc | 1185055 | |
| Optosplit II | Cairn Research | ||
| Ovalbumin | Sigma-Aldrich | A5253 | for imaging buffer |
| Plasma cleaner | Harrick Plasma | PDC-002 | |
| sdt-python | Schütz group at TU Wien | Python library for data analysis; version 17 | |
| TetraSpek bead size kit | Thermo Fisher Scientific | T14792 | Randomly distributed, immobilized fiducial markers for image registration |
| USC500TH Ultrasound bath | VWR | for SUV formation |
Références
- McKinney, S. A., Déclais, A. -. C., Lilley, D. M. J., Ha, T. Structural dynamics of individual holliday junctions. Nature Structural Biology. 10 (2), 93-97 (2002).
- Wang, S., Vafabakhsh, R., Borschel, W. F., Ha, T., Nichols, C. G. Structural dynamics of potassium-channel gating revealed by single-molecule FRET. Nature Structural & Molecular Biology. 23 (1), 31-36 (2015).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2016).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), 235 (2018).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47 (1), 819-846 (1978).
- Wu, P. G., Brand, L. Resonance energy transfer: Methods and applications. Analytical Biochemistry. 218 (1), 1-13 (1994).
- Qiao, Y., Luo, Y., Long, N., Xing, Y., Tu, J. Single-molecular förster resonance energy transfer measurement on structures and interactions of biomolecules. Micromachines. 12 (5), 492 (2021).
- Malkusch, N., Dörfler, T., Nagy, J., Eilert, T., Michaelis, J. smFRET experiments of the RNA polymerase II transcription initiation complex. Methods. 120, 115-124 (2017).
- Lee, J. -. B., et al. Single-molecule views of MutS on mismatched DNA. DNA repair. 20, 82-93 (2014).
- Phelps, C., Israels, B., Jose, D., Marsh, M. C., von Hippel, P. H., Marcus, A. H. Using microsecond single-molecule FRET to determine the assembly pathways of T4 ssDNA binding protein onto model DNA replication forks. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), E3612-E3621 (2017).
- Deindl, S., Zhuang, X. Monitoring conformational dynamics with single-molecule fluorescence energy transfer: Applications in nucleosome remodeling. Methods in Enzymology. 513, 59-86 (2012).
- Crawford, D. J., Hoskins, A. A., Friedman, L. J., Gelles, J., Moore, M. J. Single-molecule colocalization FRET evidence that spliceosome activation precedes stable approach of 5' splice site and branch site. Proceedings of the National Academy of Sciences of the United States of America. 110 (17), 6783-6788 (2013).
- Wang, Y., Xiao, M., Li, Y. Heterogeneity of single molecule FRET signals reveals multiple active ribosome subpopulations. Proteins. 82 (1), 1-9 (2014).
- Mori, T., Vale, R. D., Tomishige, M. How kinesin waits between steps. Nature. 450 (7170), 750-754 (2007).
- Huppa, J. B., et al. TCR-peptide-MHC interactions in situ show accelerated kinetics and increased affinity. Nature. 463 (7283), 963-967 (2010).
- Göhring, J., et al. Temporal analysis of T-cell receptor-imposed forces via quantitative single molecule FRET measurements. Nature Communications. 12 (1), 2502 (2021).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669 (2018).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences of the United States of America. 101 (24), 8936-8941 (2004).
- Sakon, J. J., Weninger, K. R. Detecting the conformation of individual proteins in live cells. Nature Methods. 7 (3), 203-205 (2010).
- McCann, J. J., Choi, U. B., Zheng, L., Weninger, K., Bowen, M. E. Optimizing methods to recover absolute FRET efficiency from immobilized single molecules. Biophysical Journal. 99 (3), 961-970 (2010).
- Lee, N. K., et al. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophysical Journal. 88 (4), 2939-2953 (2005).
- Asher, W. B., et al. Single-molecule FRET imaging of GPCR dimers in living cells. Nature Methods. 18 (4), 397-405 (2021).
- Joo, C., Ha, T. . Single-molecule FRET with total internal reflection microscopy. (12), 1223-1237 (2012).
- Joo, C., Ha, T. Objective-type total internal reflection microscopy (excitation) for single-molecule FRET. Cold Spring Harbor Protocols. 11, 1189-1191 (2012).
- Joo, C., Ha, T. Objective-type total internal reflection microscopy (emission) for single-molecule FRET. Cold Spring Harbor Protocols. 11, 1192-1194 (2012).
- Allan, D. B., Caswell, T., van der Wel, C. M., Dimiduk, T. . Soft-matter/pims: PIMS v0.5. , (2020).
- Anaconda Inc. . Miniconda. , (2021).
- conda-forge community. . The conda-forge project: community-based software distribution built on the conda package format and ecosystem. , (2015).
- . . JupyterLab Contributors Notebooks - JupyterLab documentation. , (2021).
- Babcock, H., Sigal, Y. M., Zhuang, X. A high-density 3D localization algorithm for stochastic optical reconstruction microscopy. Optical Nanoscopy. 1 (6), (2012).
- Gao, Y., Kilfoil, M. L. Accurate detection and complete tracking of large populations of features in three dimensions. Optics Express. 17 (6), 4685 (2009).
- Schrangl, L. . sdt-python: Python library for fluorescence microscopy data analysis (v17.1). , (2021).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. Journal of Colloid and Interface Science. 179 (1), 298-310 (1996).
- Preibisch, S., Saalfeld, S., Schindelin, J., Tomancak, P. Software for bead-based registration of selective plane illumination microscopy data. Nature Methods. 7 (6), 418-419 (2010).
- Bradski, G. The OpenCV library. Dr. Dobb's Journal: Software Tools for the Professional Programmer. 25 (11), 120-123 (2000).
- Allan, D. B., Caswell, T., Keim, N. C., van der Wel, C. M., Verweij, R. W. Soft-matter/trackpy: Trackpy v0.5.0. Zenodo. , 4682814 (2021).
- Killick, R., Fearnhead, P., Eckley, I. A. Optimal detection of changepoints with a linear computational cost. Journal of the American Statistical Association. 107 (500), 1590-1598 (2012).
- Schrangl, L., Göhring, J., Schütz, G. J. Kinetic analysis of single molecule FRET transitions without trajectories. The Journal of Chemical Physics. 148 (12), 123328 (2018).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Schrangl, L. Single-molecule FRET analysis software (3.0). Zenodo. , (2021).
- Nye, J. A., Groves, J. T. Kinetic control of histidine-tagged protein surface density on supported lipid bilayers. Langmuir. 24 (8), 4145-4149 (2008).
- Platzer, R., et al. Unscrambling fluorophore blinking for comprehensive cluster detection via photoactivated localization microscopy. Nature Communications. 11 (1), 4993 (2020).
- Johnson, I., Spence, M. . The molecular probes handbook: A guide to fluorescent probes and labeling technologies. , (2010).
- Szalai, A. M., et al. Super-resolution imaging of energy transfer by intensity-based STED-FRET. Nano Letters. 21 (5), 2296-2303 (2021).
- Miyawaki, A., et al. Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature. 388 (6645), 882-887 (1997).
- Zhai, B., Zhai, S., Hao, R., Xu, J., Liu, Z. A FRET-based two-photon probe for in vivo tracking of pH during a traumatic brain injury process. New Journal of Chemistry. 43 (43), 17018-17022 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon